Method Article
Un protocollo per la valutazione funzionale del Whole-Protein Saturazione mutagenesi biblioteche Utilizzando High-Throughput Sequencing
In questo articolo
Riepilogo
Vi presentiamo un protocollo per la valutazione funzionale delle biblioteche unico sito di saturazione di mutagenesi complete di proteine che utilizzano high-throughput sequencing. È importante sottolineare che questo approccio utilizza coppie di primer ortogonali al multiplex di costruzione di biblioteche e sequenziamento. Risultati rappresentativi utilizzando TEM-1 β-lattamasi selezionato ad un dosaggio clinicamente rilevante di ampicillina sono forniti.
Abstract
Site-directed mutagenesis has long been used as a method to interrogate protein structure, function and evolution. Recent advances in massively-parallel sequencing technology have opened up the possibility of assessing the functional or fitness effects of large numbers of mutations simultaneously. Here, we present a protocol for experimentally determining the effects of all possible single amino acid mutations in a protein of interest utilizing high-throughput sequencing technology, using the 263 amino acid antibiotic resistance enzyme TEM-1 β-lactamase as an example. In this approach, a whole-protein saturation mutagenesis library is constructed by site-directed mutagenic PCR, randomizing each position individually to all possible amino acids. The library is then transformed into bacteria, and selected for the ability to confer resistance to β-lactam antibiotics. The fitness effect of each mutation is then determined by deep sequencing of the library before and after selection. Importantly, this protocol introduces methods which maximize sequencing read depth and permit the simultaneous selection of the entire mutation library, by mixing adjacent positions into groups of length accommodated by high-throughput sequencing read length and utilizing orthogonal primers to barcode each group. Representative results using this protocol are provided by assessing the fitness effects of all single amino acid mutations in TEM-1 at a clinically relevant dosage of ampicillin. The method should be easily extendable to other proteins for which a high-throughput selection assay is in place.
Introduzione
Mutagenesi è stato a lungo utilizzato in laboratorio per studiare le proprietà dei sistemi biologici e la loro evoluzione, e per la produzione di proteine mutanti o organismi con funzioni avanzate e innovative. Mentre i primi approcci fatti valere metodi che producono mutazioni casuali negli organismi, l'avvento della tecnologia del DNA ricombinante ha permesso ai ricercatori di introdurre modifiche a selezionare il DNA in modo specifico del sito, vale a dire., Sito-diretta mutagenesi 1,2. Con le tecniche attuali, tipicamente utilizzando oligonucleotidi mutageni in una reazione a catena della polimerasi (PCR), è relativamente facile per creare e valutare piccolo numero di mutazioni (ad es., Mutazioni puntiformi) in un dato gene 3,4. E 'molto più difficile tuttavia quando gli approcci obiettivo, per esempio, la creazione e la valutazione di tutti i possibili single-site (o di ordine superiore) mutazioni.
Mentre molto si è appreso dai primi studi che tentano di valutare un gran numero di mutations nei geni, le tecniche utilizzate erano spesso laboriosa, ad esempio imponendo la valutazione di ogni mutazione indipendentemente con dialogo soppressore ceppi 5-7, o sono stati limitati nella loro capacità quantitativa causa della bassa profondità sequenziamento di Sanger sequenziamento 8. Le tecniche utilizzate in questi studi sono stati in gran parte soppiantato con metodi che utilizzano high-throughput tecnologia di sequenziamento 9-12. Questi concettualmente semplici approcci comportano la creazione di una libreria comprendente un gran numero di mutazioni, sottoponendo la libreria da uno schermo o di selezione per la funzione, e quindi profonde sequenziamento (es., Dell'ordine di> 10 6 sequenziamento legge) biblioteca ottenuta prima e dopo la selezione. In questo modo, i fenotipiche o idoneità effetti di un gran numero di mutazioni, rappresentati come il cambiamento nella frequenza popolazione di ciascun mutante, può essere valutato simultaneamente e quantitativamente.
Abbiamo già introdotto un semp(. cioè, tutta-proteina librerie saturazione di mutagenesi) approccio per valutare le biblioteche di tutte le possibili singole mutazioni aminoacidiche di proteine applicabili alle geni con una lunghezza superiore alla lunghezza di sequenziamento leggi 11,13: primo, ogni posizione amminoacido è randomizzato dal sito-mutageno PCR. Durante questo processo, il gene viene suddiviso in gruppi composti da posizioni contigue con una lunghezza totale alloggiati dalla piattaforma sequenziamento. I prodotti mutageni PCR per ciascun gruppo sono poi combinati, e ogni gruppo sottoposti indipendentemente per la selezione e high-throughput sequencing. Mantenendo una corrispondenza tra la posizione di mutazioni nella sequenza e la lunghezza di sequenza di lettura, questo approccio ha il vantaggio di massimizzare profondità sequenziamento: mentre si potrebbe semplicemente sequenziare tali librerie in finestre brevi senza divisione in gruppi (ad esempio, da un fucile standard. approccio sequenziamento), più letture ottenuta sarebbe wild-type e quindi il maggioranza di sequenziamento sprecato (ad esempio, per un intero proteico libreria saturazione mutagenesi di una proteina di 500 aminoacidi sequenziato in 100 aminoacidi (300 bp) finestre, al minimo 80% di letture sarà la sequenza wild-type).
Qui, un protocollo è presentato che utilizza high-throughput sequenziamento per la valutazione funzionale del tutto-proteina librerie saturazione di mutagenesi, utilizzando l'approccio di cui sopra (illustrato in Figura 1). È importante sottolineare che, si introduce l'uso di primer ortogonali nel processo di clonazione biblioteca in codice a barre di ciascun gruppo sequenza, che consente loro di essere multiplexing in una libreria, sottoposti contemporaneamente a screening o selezione, e poi de-multiplex per sequenziamento profondo. Poiché i gruppi di sequenza non sono sottoposti a selezione indipendente, questo riduce il carico di lavoro ed assicura che ogni mutazione sperimenta lo stesso livello di selezione. TEM-1 β-lattamasi, un enzima che conferisce resistenza ad alto livelloantibiotici β-lattamici (ad es., ampicillina) nei batteri è utilizzato come sistema modello 14-16. Un protocollo è descritto per la valutazione di un intero proteico libreria mutagenesi saturazione del TEM-1 in E. coli sotto selezione a livello sierico approssimativo per una dose clinica di ampicillina (50 ug / ml) 17,18.
Protocollo
Nota: Vedere la Figura 1 per una sintesi del protocollo. Diversi passaggi e reagenti nel protocollo richiedono misure di sicurezza (indicate con "ATTENZIONE"). Consultare le schede di sicurezza dei materiali prima dell'utilizzo. Tutte le fasi del protocollo vengono eseguiti a temperatura ambiente a meno che altro indicato.
1. Preparare la cultura media e Piastre
- Preparare e sterilizzare in autoclave 1 L di acqua purificata, 100 ml Super ottimale Broth (SOB; Tabella 1), 1 L Luria-Bertani brodo (LB; tabella 2) e 1 L LB-agar (Tabella 3). Preparare a parte e sterilizzare tre palloni di coltura ciascuna contenente 1 L LB.
Nota: Durante il protocollo "acqua" si riferisce a autoclave-sterilizzato acqua purificata; SOB, LB e LB-agar si riferiscono alle soluzioni autoclave-sterilizzati. - Preparare un 12 mg / ml di brodo Tet sciogliendo 0,12 g di tetraciclina cloridrato in 10 ml di etanolo al 70%. Sterilizzare utilizzando un filtro da 0,2 micron e conservarea 4 ° C al riparo dalla luce.
- Raffreddare LB-agar a 50 ° C e si aggiunge 1 ml di Tet magazzino (concentrazione finale di 12 mg / ml Tet). Versare in piastre di Petri e raffreddare a temperatura ambiente al riparo dalla luce. Conservare a 4 ° C al riparo dalla luce.
2. Costruzione del Tutto-gene Saturazione mutagenesi Biblioteca
Nota: Primer; PCR completato, digerisce restrizione e legature; e campioni di DNA purificati possono essere conservati a -20 ° C.
- Progettare primer mutagenesi
- Per mutagenize ogni posizione aminoacido a tutte le possibili aminoacidi, la progettazione di un paio di primer mutagenesi complementari (senso / avanti e antisenso / indietro) per ogni posizione amminoacido con le seguenti linee guida:
- Sostituire il codone corrispondente amminoacido da mutagenizzata da NNS (dove N è una miscela di tutti e quattro basi nucleotidiche e S è una miscela di citosina (C) e guanina (G)) e il centro nella primer, affiancato da circa 15 nucleotidi su ogni lato.
- Assicurarsi che il 5 'e 3' estremità terminano in C o G e che la temperatura di fusione (T m) è di circa 70 ° C 3. Utilizzare lo script di NNS_PrimerDesign.m computazionale (Vedere file di codice supplementare) per la progettazione NNS primer mutagenesi secondo queste linee guida.
- Ordine primer da una fonte commerciale. Per facilità d'uso, li hanno sintetizzato in formato piastra a 96 pozzetti e pre-diluito in acqua a 50 micron, con una serie di piastre contenenti i primer mutagenesi senso e un altro i primer antisenso.
- Riempire un bacino pipetta con acqua e utilizzare una pipetta multicanale per trasferire 95 microlitri di 263 pozzi nel corso di tre piastre da 96 pozzetti. Diluire il primer 20 volte a 2,5 micron utilizzando una pipetta multicanale per trasferire 5 ml dalle piastre a 96 pozzetti contenenti i primer senso di mutagenesi ai pozzi cognate delle piastre contenenti acqua.
- Ripeterepasso protocollo 2.1.3 per diluire il primer antisenso mutagenesi.
- Per mutagenize ogni posizione aminoacido a tutte le possibili aminoacidi, la progettazione di un paio di primer mutagenesi complementari (senso / avanti e antisenso / indietro) per ogni posizione amminoacido con le seguenti linee guida:
- Sintesi di NNS sub-librerie per ogni posizione aminoacido da due fasi PCR mutagenesi sito-diretta
- Eseguire le primo turno PCR mutageni. Per ciascun primer mutagenesi, preparare un 25 microlitri reazione di PCR utilizzando pBR322_AvrII plasmide come modello e primer AatII_F o AvrII_R (primer senso di mutagenesi accoppiati con primer AvrII_R, e antisenso primer mutagenesi accoppiati con primer AatII_F; totale di 526 PCR). Vedere la Tabella dei Materiali per AatII_F e AvrII_R sequenze.
- Preparare una PCR "Master Mix" aggiungendo i reagenti riportati nella tabella 4 a un tubo da 15 ml. Trasferire in un bacino di pipetta. Utilizzare una pipetta multicanale per trasferire 15 microlitri di 263 pozzi nel corso di tre piastre a 96 pozzetti PCR. Utilizzare una pipetta multicanale per trasferire 10 ml dalle piastre a 96 pozzetti contenenti i primer diluito senso di mutagenesi ai pozzi cognate in le piastre PCR.
- Coprire ciascuna piastra PCR con un sigillo piastra a 96 pozzetti. Centrifugare a 200 xg per 2 min.
- Trasferire le piastre PCR per termociclatore ed eseguire il seguente programma: 98 ° C per 30 sec; 20 cicli: 98 ° C per 10 sec, 55 ° C per 20 sec, 72 ° C per 1 min; 72 ° C per 2 min; tenere a 4 ° C.
- protocollo Ripetere i punti 2.2.1.1 - 2.2.1.3 per le piastre a 96 pozzetti contenenti i primer antisenso mutagenesi diluiti.
- Eseguire il secondo turno PCR mutageni. Per ogni posizione aminoacido, preparare un 25 microlitri PCR reazione usando primer AatII_F e AvrII_R, e il misto e diluito primo turno prodotti di PCR mutageni come modello (per un totale di 263 PCR).
- Riempire un bacino pipetta con acqua e utilizzare una pipetta multicanale per trasferire 198 ml per 263 pozzi nel corso di tre piastre da 96 pozzetti.
- Unire e diluire 100 volte i prodotti di PCR mutageni per ogni posizione amminoacido utilizzando una pipetta multicanale perprimo trasferimento 1 ml delle piastre a 96 pozzetti PCR contenenti i prodotti di PCR derivanti dai primer senso mutagenesi ai pozzetti cognate delle piastre contenenti acqua. Quindi ripetere il trasferimento per i prodotti di PCR derivanti dai primer antisenso mutagenesi.
- Preparare una PCR "Master Mix" aggiungendo i reagenti da tabella 5 ad un tubo da 15 ml. Trasferire in un bacino di pipetta. Utilizzare una pipetta multicanale per trasferire 24 microlitri di 263 pozzi nel corso di tre piastre a 96 pozzetti PCR. Utilizzare una pipetta multicanale per trasferire 1 ml dalle piastre a 96 pozzetti contenenti il miscelato e diluito primo turno prodotti di PCR mutageni ai pozzetti affini nelle piastre PCR.
- Coprire ciascuna piastra PCR con un sigillo piastra a 96 pozzetti. Centrifugare a circa 200 xg per 2 min. piastre di trasferimento di termociclatore ed eseguire lo stesso programma come al punto 2.2.1.3 protocollo.
- Analizzare i risultati del secondo turno PCR mutageni mediante elettroforesi su gel.Assicurarsi che tutti i prodotti sono di dimensioni corrette e assenza di contaminazione dei prodotti.
- Aggiungere 2 ml di 2x colorante di caricamento del gel ad un bacino pipetta e quindi utilizzare una pipetta multicanale per trasferire 6 ml per 263 pozzi nel corso di tre piastre da 96 pozzetti. Utilizzare una pipetta multicanale per trasferire 6 ml dalle piastre a 96 pozzetti PCR che contiene il secondo turno prodotti di PCR mutageni ai pozzi cognate delle piastre a 96 pozzetti contenenti colorante.
- Preparare un gel 1,5% con 0,2 mg / ml di etidio bromuro (ATTENZIONE).
- Carico scala del DNA nel primo e nell'ultimo corsie di ogni riga. Quindi utilizzare una pipetta multicanale per caricare 10 ml di campioni dal punto 2.2.3.1 del protocollo.
- Attivare il gel a 100 V per 40 minuti e l'immagine su un transilluminatore UV.
- protocollo Ripetere i punti 2.2.3.2 - 2.2.3.4 fino a quando tutti i campioni vengono analizzati.
- Misurare accuratamente la concentrazione di ogni NNS sub-biblioteca prodotto di PCR utilizzando un reagente quantificazione dsDNA.
- Trasferimentocirca 15 ml EB buffer per un bacino pipetta. Utilizzare una pipetta multicanale per trasferire 49 microlitri di 263 pozzi nel corso di tre a 96 pozzetti, chiare piastre test inferiore nera con pareti.
- Utilizzare una pipetta multicanale per trasferire 1 ml di ogni seconda fase prodotto di PCR (passo protocollo 2.2.2) per i pozzi cognate delle piastre test a 96 pozzetti.
- Preparare una curva standard concentrazione di DNA diluendo lambda DNA del fago a 2 ng / ml in 300 microlitri di buffer EB e poi fare dieci diluizioni due volte (per un totale di 11 concentrazioni). Trasferire 50 microlitri per primi undici colonne di una fila di una delle piastre a 96 pozzetti test dal passo precedente che contiene nessun campione; alla colonna dodicesimo aggiungere 50 ml di tampone EB (Bianco reagente).
- Preparare dsDNA reagente quantificazione con l'aggiunta di 75 ml di reagente (vedi tabella dei materiali) ad un tubo da 15 ml, quindi aggiungere 15 ml di tampone EB. Mescolare invertendo il tubo e quindi il trasferimento a un bacino pipetta. Proteggere il reagente dalla luce.
- Utilizzare una pipetta multicanale per trasferire 50 ml di preparato reagente di quantificazione dsDNA in ciascun pozzetto delle piastre test. Mescolare pipettando su e giù. Incubare le piastre a temperatura ambiente per 5 minuti al riparo dalla luce.
- Misurare la fluorescenza di ogni campione usando un lettore per micropiastre e lunghezze d'onda fluoresceina standard (eccitazione 485 nm, emissione di 520 nm; 0,1 sec).
- Sottrarre il valore di fluorescenza del bianco reagente da tutti i campioni. Generare una curva standard dalle misure di fluorescenza dei campioni fago lambda. Calcolare la concentrazione di ogni campione usando le rispettive misure di fluorescenza e la curva standard.
- Eseguire le primo turno PCR mutageni. Per ciascun primer mutagenesi, preparare un 25 microlitri reazione di PCR utilizzando pBR322_AvrII plasmide come modello e primer AatII_F o AvrII_R (primer senso di mutagenesi accoppiati con primer AvrII_R, e antisenso primer mutagenesi accoppiati con primer AatII_F; totale di 526 PCR). Vedere la Tabella dei Materiali per AatII_F e AvrII_R sequenze.
- Clonazione di NNS sub-librerie in vettori di selezione
- Mescolare 100 ng di ciascun NNS sub-biblioteca prodotto di PCR in cinque NNS gruppi sub-biblioteca. Le istruzioni del fabbricante, ripulire campioni utilizzando un kit di purificazione del DNA e quindi misurare la concentrazione utilizzando un DSDNA quantificazione reagente.
Nota: Ogni gruppo è composto da circa 53 contigui aminoacidi posizioni distanziate lungo la sequenza di TEM-1 (gruppi NNS sub-biblioteca 1-5 sono composti da posizioni 26-78, 79-132, 133-183, 184-236, e 237 - 290, rispettivamente numerazione secondo Ambler et al 19).. - Creare clonazione vettori per ogni gruppo di sub-biblioteca NNS.
- Preparare cinque 100 microlitri PCR secondo la tabella 6, usando primer AvrII_F e AatII_OP1_R - AatII_OP5_R, e plasmidi pBR322_OP1-5 come modello (AatII_OP1_R in coppia con pBR322_OP1, etc.).
- Trasferimento a termociclatore ed eseguire il seguente programma: 98 ° C per 30 sec; 25 cicli: 98 ° C per 10 sec, 55 ° C per 20 sec, 72 ° C per 1,5 minuti; 72 ° C per 2 min; tenere a 4 ° C. Vedere T in grado di Materiali per le sequenze di AatII_R e AvrII_F.
- Preparare un gel 1% con 0,2 mg / ml di etidio bromuro (ATTENZIONE).
- Aggiungere 20 ml di 6x dye gel loading ad ogni campione PCR. Caricare prima corsia del gel con DNA ladder; caricare intero volume di ciascun campione, saltando almeno un pozzetto tra i campioni.
- Eseguire gel a 100 V per 50 min.
- Visualizza gel utilizzando un illuminatore UV-lunghezza d'onda (ATTENZIONE). fette accise che contengono il prodotto di PCR a ~ 3.500 bp; trasferire per separare i tubi microcentrifuga. fette gel possono essere conservati a -20 ° C.
- Le istruzioni del fabbricante, purificare campioni utilizzando un kit di estrazione gel e la concentrazione misura utilizzando un reagente quantificazione dsDNA.
- Per entrambi i gruppi NNS sub-biblioteca (step protocollo 2.3.1) e vettori di clonazione (step protocollo 2.3.2), istituito restrizione digerisce con gli enzimi AatII e AvrII secondo il T in grado 7. Incubare a 37 ° C per 1 ora. Le istruzioni del fabbricante, ripulire campioni utilizzando un kit di purificazione del DNA e quindi misurare la concentrazione utilizzando una quantita dsDNAreagente zione.
- Impostare reazioni legatura seguente Tabella 8 per ciascun gruppo NNS restrizione digerito sub-biblioteca di restrizione digerito clonazione cognate vettore (gruppo NNS sub-biblioteca 1 con pBR322_OP1, etc.). Incubare a temperatura ambiente per 1 ora. Pulire reazioni utilizzando un kit di purificazione del DNA secondo le istruzioni del fabbricante; eluire DNA con 20 ml di acqua.
- Trasformare l'insieme delle reazioni legatura purificati nella libreria-efficiente E. coli di elettroporazione.
- Scongelare electrocompetent E. coli e poi le cellule luogo e reazioni legatura purificati su ghiaccio.
- Trasferire 10 ml scongelati cellule per ogni reazione legatura purificato e poi il trasferimento al elettroporazione cuvetta. Elettroporazione a 1,8 kV.
- Recuperare le cellule dalla risospensione in 1 ml SOB. Incubare per 1 ora a 37 ° C.
- Risospendere 10 ml di ogni cultura recupero in 990 ml LB; diffondere 100 ml su LB-agar piatti containing 12 mg / ml Tet. Incubare le piastre O / N (~ 16 ore) a 37 ° C.
- Per ogni cultura recupero, preparare un pallone di coltura da 250 ml con 50 ml di LB e 50 ml Tet magazzino. Trasferimento a pallone i restanti ~ 1 ml di cultura recupero. Incubare O / N (~ 16 ore) a 37 ° C con vigorosa scuotimento (~ 200 rpm).
- Contare il numero di colonie su ogni piatto. Calcolare il numero di trasformanti di successo come
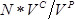 , dove
, dove  è il numero di colonie,
è il numero di colonie, 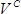 è il volume di cultura di recupero (1000 ml) e
è il volume di cultura di recupero (1000 ml) e 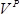 è il volume della coltura recupero placcato (1 ml).
è il volume della coltura recupero placcato (1 ml).
Nota: per garantire una copertura completa di tutte le mutazioni, come regola generale il numero di trasformanti di successo dovrebbe essere ≥100 volte rispetto al numero di mutazioni attesi. Ogni sotto-biblioteca NNS ha ~ 53 posizioni, in modo che il numero previsto di mutazioni è 53 posizioni × 32 codoni / posizione ≈ 1.7 × 10 3; per dare una dimensione biblioteca ≥100 volte (≥1.7 × 10 5) ci dovrebbe essere ≥170 colonie su ogni piatto. - Secondo le indicazioni del produttore, isolare il DNA plasmidico da culture utilizzando un kit di purificazione plasmide e quindi misurare le concentrazioni utilizzando un reagente quantificazione dsDNA. Mescolare 100 ng di ogni plasmide. Questo crea la libreria di saturazione di mutagenesi finale tutto-proteina.
- Mescolare 100 ng di ciascun NNS sub-biblioteca prodotto di PCR in cinque NNS gruppi sub-biblioteca. Le istruzioni del fabbricante, ripulire campioni utilizzando un kit di purificazione del DNA e quindi misurare la concentrazione utilizzando un DSDNA quantificazione reagente.
3. Selezione del TEM-1 Whole-proteina saturazione Mutagenesi Library per la resistenza agli antibiotici
- Preparazione della cultura preselezione.
- Diluire il plasmide dal passaggio del protocollo 2.3.7 a 0.5 ng / ml in acqua e trasferire 20 microlitri di una provetta. Eseguire trasformazione, ricupero, placcatura e O / N crescita come precedentemente descritto al punto 2.3.5 del protocollo, ad eccezione di trasferimento 1 ml di cultura recupero SOB a 999 ml LB.
- Contare il numero di colonie. Per garantire la copertura completa di tutte le mutazioni non ci dovrebbe essere ≥100 colonie, indicando ≥10 6 trasformanti di successo (100 × 263 × 32 posizioni codoni / posizione ≈ 10 6).
- Misurare la concentrazione della cultura 50 ml O / N.
- Preparare un vuoto LB con l'aggiunta di 1 ml di LB ad una cuvetta spettrofotometro. Misurare OD600 su uno spettrofotometro.
- Diluire la O / N cultura di 10 volte da risospensione 100 ml in 900 ml LB. Misurare la OD600. Sottrarre la lettura OD600 del vuoto e moltiplicare per 10 per dare la OD600 del / della cultura O N.
- Pre-riscaldare le tre palloni di coltura dal punto di protocollo 1.1 per ~ 30 minuti a 37 ° C. Diluire il / la cultura O N per OD600 = 0,1 e aggiungere 1 ml di un pallone (OD600 finale = 0,001). Tla sua è la "cultura pre-selezione".
- Incubare la "cultura pre-selezione" a 37 ° C con vigorosa agitazione (200 rpm). Periodicamente monitorare la crescita misurando OD600 come al punto 3.1.3 protocollo (non è necessario diluire cultura 10 volte) fino OD600 = 0,1 (~ 2,5 ore).
- Trasferire 100 ml di cultura pre-selezione per due provette da 50 ml coniche. Centrifugare a 4000 xg per 6 minuti a 4 ° C. Rimuovere la maggior parte del surnatante e combinare in un unico tubo conico 15. Ripetere la centrifugazione e rimuovere tutti surnatante. Conservare a -20 ° C.
- La selezione per la resistenza ampicillina.
- Mentre la cultura pre-selezione è in incubazione, preparare un 50 mg / ml scorta di Ampère in acqua sciogliendo 0,5 g di sodio ampicillina in 10 ml di acqua. Sterilizzare utilizzando un filtro da 0,2 micron e conservare a 4 ° C.
- Per gli altri due recipienti, aggiungere Tet ad una concentrazione finale di 12 mg / ml e un volume di pre-selezione coltura tale tha t la OD600 finale = 0,001. Per un pallone, aggiungere 1 ml Amp, per una concentrazione finale di 50 mg / ml - questa è la "cultura di selezione".
- Incubare le culture a 37 ° C con vigorosa agitazione (200 rpm). Monitorare la crescita della coltura per le quali non ampicillina è stata aggiunta nel passaggio precedente, fino OD600 = 0,1 (~ 2,5 ore). In questo momento, misurare anche la OD600 della cultura selezione.
- Dividere la OD600 della cultura selezione in 0,1 e moltiplicare per 100 ml. Trasferire questo volume (~ 400 ml) a 50 ml provette coniche e centrifugare a 4000 xg per 6 minuti a 4 ° C. Rimuovere la maggior parte del surnatante e combinare in un unico tubo conico 15. Ripetere la centrifugazione e rimuovere tutti surnatante.
- Secondo le indicazioni del produttore, isolare il DNA plasmidico dal pre-selezione (step del protocollo 3.1.6) e la selezione (passo protocollo 3.2.4) pellet cellulari cultura e quindi misurare le concentrazioni utilizzando un reagente quantificazione dsDNA.
- Preparazione dei campioni per high-throughput sequencing
- Preparare 25 microlitri PCR di de-multiplex dei gruppi NNS sub-libreria con primer ortogonali
- Preparare una master mix PCR base alla tabella 9; trasferire 23 ml a dieci provette PCR.
- Aggiungere 1 ml di 0,5 ng DNA plasmidico / ml purificato dalla cultura pre-selezione per PCR tubi 1 - 5 e dalla cultura di selezione ai tubi 6 - 10.
- Mescolare 50 ml di 50 micron in avanti primer ortogonali OP1_F - OP5_F con il rispettivo inverso primer ortogonali OP1_R - OP5_R. Nello stesso ordine, trasferire 1 ml di provette PCR 1 - 5 e 6 - 10. Vedere tabella dei materiali per le sequenze di OP1_F - OP5_F e OP1_R - OP5_R.
- Trasferimento provette PCR per termociclatore. Eseguire il seguente programma: 98 ° C per 30 sec; 20 cicli: 98 ° C per 10 sec, 55 °; C per 20 sec, 72 ° C per 1,5 minuti; 72 ° C per 2 min; tenere a 4 ° C.
- Preparare 25 microlitri PCR per isolare ciascuno dei gruppi di sub-biblioteca NNS
- Diluire 100 volte i PCR dieci dal punto 4.1.1.4 protocollo per il trasferimento di 1 ml di ciascuna di separare provette PCR e l'aggiunta di 99 ml di acqua. Mix, Pipettare quindi fuori 99 ml e scartare.
- Mescolare 50 ml di 50 micron primer forward Group1_F - Group5_F con il rispettivo primer Group1_R inverso - Group5_R. Nello stesso ordine, di trasferimento 1 ml per PCR tubi 1-5 e 6 - 10. Vedere tabella dei materiali per le sequenze di Group1_F - Group5_F e Group1_R - Group5_R.
- Preparare una master mix PCR base alla tabella 9, trasferire 23 ml ad ogni provetta PCR. Trasferimento provette PCR per termociclatore; eseguire lo stesso programma come al punto 2.2.1.3 protocollo.
- Effettuare gli ultimi 25 microlitri PCR per aggiungere sequenze di indicizzazione
- diliuto 100 volte il PCR dieci dal punto 4.1.2.3 protocollo per il trasferimento di 1 ml di ciascuna di separare provette PCR e l'aggiunta di 99 ml di acqua. Mix, Pipettare quindi fuori 99 ml e scartare.
- Preparare una master mix PCR base alla tabella 9, trasferire 23 ml ad ogni provetta PCR.
- Per i tubi con template provenienti da NNS sub-biblioteca gruppi 1-5, trasferire 0,5 ml per primer forward tubo 501_F - 505_F rispettivamente. Per i tubi con modello risultante dalle preselezione e selezione delle culture, trasferire 0,5 ml per provetta primer 701_R e 702_R inversa, rispettivamente. Vedere la Tabella dei Materiali per sequenze di 501_F - 505_F, e 701_R e 702_R.
- Trasferimento provette PCR al termociclatore ed eseguire il programma dal punto 2.2.1.3.
- Mescolare e purificare campioni
- Le concentrazioni di misura utilizzando un reagente quantificazione dsDNA secondo le istruzioni del produttore. Mescolare 100 ng di ogni int prodotto di PCROA singolo tubo microcentrifuga.
- Preparare un gel di agarosio al 2% con 0,2 mg / ml di etidio bromuro (ATTENZIONE).
- Aggiungere 6x colorante di caricamento del gel ai prodotti campione misto PCR. Caricare prima corsia del gel con DNA ladder; caricare intero volume del campione.
- Eseguire gel a 100 V per 50 min. Visualizza gel utilizzando un illuminatore UV-lunghezza d'onda (ATTENZIONE). fetta Excise contenente il prodotto di PCR a ~ 360 bp; trasferimento a microcentrifuga tubo. fetta gel può essere conservato a -20 ° C.
- Le istruzioni del fabbricante, purificare campione utilizzando un kit di estrazione gel e misurare la concentrazione utilizzando un reagente quantificazione dsDNA. Questo è il campione finale per high-throughput sequencing.
- Sequenza su una piattaforma di sequenziamento high-throughput (vedi tabella di materiali per piattaforma utilizzata in questo protocollo).
- Calcola concentrazione del campione in Nm come
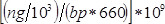 , Dove
, Dove 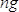 è la concentrazione del campione in ng / ml e
è la concentrazione del campione in ng / ml e  è la lunghezza della sequenza del DNA campione (~ 360 bp). Diluire il campione di 4 Nm in un tampone EB.
è la lunghezza della sequenza del DNA campione (~ 360 bp). Diluire il campione di 4 Nm in un tampone EB. - Seguire le istruzioni del produttore per denaturare campione e portare a 9:00 in tampone di ibridazione.
- Carico 600 ml di campione in cartuccia del reattivo. Sequenza seguendo le istruzioni del produttore e le istruzioni sullo schermo.
- Calcola concentrazione del campione in Nm come
- Preparare 25 microlitri PCR di de-multiplex dei gruppi NNS sub-libreria con primer ortogonali
- Analisi dei dati di sequenziamento
- Scarica Flash (Fast Lunghezza Regolazione del corto legge) 20, inserire nella cartella con i file fastq.gz ottenuti da sequenziamento.
- Utilizzare Flash per unire i file fastq.gz corrispondenti al-end accoppiato legge per ogni coppia di indici (avanti e indietro legge per ogni gruppo di sub-biblioteca NNS per la preselezione e selezione delle culture).
- Aprire Prompt dei comandi e passare alla cartella in fase protocollo 4.2.1. Partecipa ogni coppia di letture con il comando: il flash , dove mates1.fastq.gz e mates2.fastq.gz sono i file che contengono la diretta e inversa legge, rispettivamente.
- Dopo l'adesione ogni coppia di legge, posizionare il file di output out.extendedFrags.fastq in cartelle separate per i risultati di pre-selezione o di selezione culture. Rinominare il file di output out.extendedFrags.fastq secondo il gruppo di sub-libreria NNS a cui corrisponde (es., 1.fastq, 2.fastq, etc.).
- Eseguire il NNS_DataAnalyzer.m script di calcolo (Vedere File Codice supplementare) da ogni cartella per calcolare i conteggi per ogni singola mutazione aminoacido, e la conta per la wild-type, per ogni gruppo di sub-biblioteca NNS.
- Calcolare l'effetto di fitness
 di ogni mutazione
di ogni mutazione  in ogni posizione
in ogni posizione  come base dieci logaritmo del rapporto di conteggi ottenuti nella selezione (
come base dieci logaritmo del rapporto di conteggi ottenuti nella selezione (  ) Rispetto al pre-selezione (
) Rispetto al pre-selezione ( 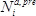 ) Condizioni, rispetto al wild-type:
) Condizioni, rispetto al wild-type:  .
.
Risultati
La mappa plasmide per cinque plasmidi pBR322 modificati contenenti siti di priming ortogonali (pBR322_OP1 - pBR322_OP5) è mostrato in Figura 2A. Per verificare se i primer ortogonali sono specifici, PCR sono state eseguite utilizzando ciascuna coppia di primer ortogonali individualmente, insieme a tutti e cinque i plasmidi pBR322_OP1-5, o con tutti i plasmidi meno il plasmide corrispondente alla coppia di primer ortogonale. Il prodotto è stato ottenuto corretta solo quando il plasmide corrispondente è stato incluso, e nessun prodotto di qualsiasi dimensione è stata ottenuta in sua assenza (Figura 2B).
Un esperimento rappresentativo è stata eseguita seguendo il protocollo descritto in questo testo (Figura 1). A seguito di trasformazione (sezione del protocollo 4.2), 6.2 × 10 6 legge dalla condizione di pre-selezione e 6,3 × 10 6 legge dal 50 ug / ml ampicillina conditi selezionein sono stati ottenuti. I conteggi ottenuti per ogni amino acido mutazione dalla condizione di pre-selezione visualizzare una caratteristica log-normale distribuzione (figura 3A) 13. Almeno un conteggio dal sequenziamento della cultura pre-selezione per 98,9% di mutazioni (58 avuto conteggi), e maggiore di 100 conteggi per 91,2% (465 avevano meno di 100 conteggi) sono stati ottenuti. Figura 3B illustra l'effetto di fitness relativa () per ciascuna mutazione in ciascuna posizione del TEM-1; la distribuzione dei è mostrato in Figura 3C. In Selezione a 50 ug / ml di ampicillina, la maggior parte delle mutazioni hanno un effetto di fitness neutro o quasi neutro (≈ 0, corrispondenti a pixel bianchi in Figura 3B e la grande picco nella Figura 3C). Una piccola frazione di mutazioni a questa concentrazione ha effetti sostanziali sulla forma fisica (<< 0, corrispondente al pixel blu in figura 3B e la coda di sinistra della figura 3C); exp ectedly, questi includono le mutazioni all'interno dei residui del sito attivo altamente conservati (S70, K73, S130, D131, N132, K234 e G236) 13,14. Al contrario, alcuni mutazioni aumentano notevolmente fitness su quella del TEM-1 (>> 0, corrispondenti a pixel rossi in Figura 3B), come potrebbe essere previsto dal TEM-1 è molto efficace a ampicillina idrolisi ( 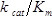 ≈ 10 7 M -1 s -1) 14.
≈ 10 7 M -1 s -1) 14.
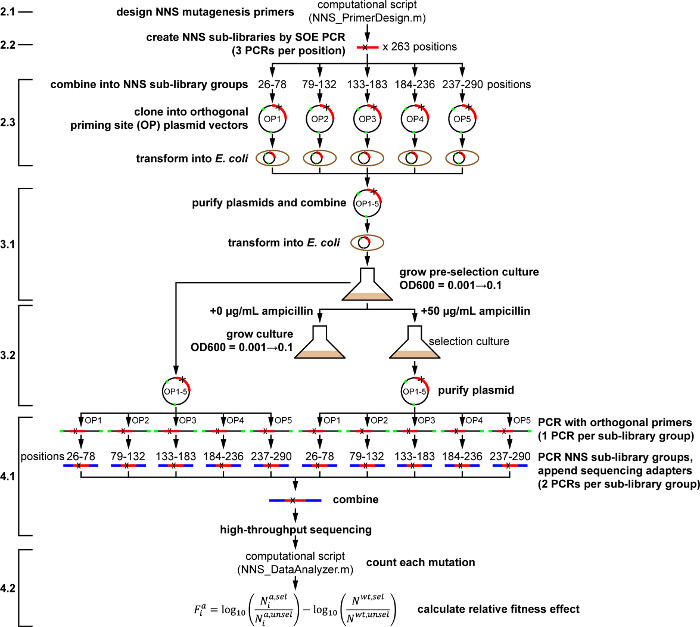
Figura 1. Schema di Whole-proteina saturazione Mutagenesi Protocollo per TEM-1 β-lattamasi sotto ampicillina selezione. Azioni come descritto nel protocollo sono indicati in grassetto. passaggi del protocollo numerati sono mostrati a sinistra, per riferimento al testo principale.k "> Clicca qui per vedere una versione più grande di questa figura.

Figura 2. Convalida dei Orthogonal adescamento sito plasmidi vettori. Mappa (A) plasmidi per i cinque plasmidi pBR322 modificati contenenti siti di priming ortogonali (pBR322_OP1 - pBR322_OP5). Posizione e la direzione dei siti priming ortogonali sono indicati. Sedi di diversi siti di restrizione sono etichettati; tutto-proteina libreria saturazione mutagenesi TEM-1 (che comprende l'intero TEM-1 gene e promotore) viene clonato in-tra i siti di restrizione AatII e AvrII. Abbreviazioni: tet gene di resistenza alla tetraciclina, ORI origine di replicazione (B) Ogni coppia di primers ortogonali (OP1-OP5) è stato testato in un PCR contenente tutti i cinque plasmidi pBR322_OP1-5 (+), o con tutti i plasmidi meno rispettivo plasmide (. ˗). Viene mostrato un bromuro di etidiomacchiato gel di agarosio (1% w / v) Pellicola ciascuna reazione di PCR, dimensioni separate mediante elettroforesi. Il prodotto formato previsto è 1.628 bp; la prima corsia è una scala di DNA, la dimensione delle norme pertinenti sono indicati. Si prega di cliccare qui per vedere una versione più grande di questa figura.

Figura 3. Risultati di TEM-1 β-lattamasi Whole-proteina Saturazione esperimento. (A) istogramma che mostra la distribuzione di conteggi per ciascuna mutazione amminoacido ottenuto da high-throughput sequenziamento della biblioteca dalla condizione pre-selezione. Per semplicità, mutazioni con valori pari a zero (53 mutazioni) sono mostrate come aventi un conteggio di uno. Effetti (B) Fitness di tutti i singoli mutazioni aminoacidiche nella TEM-1 sotto selezione a 50 ug / ml di ampicillina. Mostrato sono i datimatrice contenente l'effetto di fitness relativa () raffigurato colorimetricamente con blu che rappresenta effetto deleterio, rosso un effetto positivo e bianco nessun effetto idoneità rispetto al wild-type. Mutazioni per i quali non conteggi sono stati ottenuti dalla cultura pre-selezione sono di colore nero. Righe raffigurano posizioni lungo la sequenza primaria e colonne indicano la mutazione ad uno dei venti amminoacidi o codone di stop (indicato dal codice di una sola lettera, * è codone di stop); la struttura secondaria di TEM-1 è indicato con motivi sinistra e diversi altamente conservati all'interno del sito attivo sono indicate a destra. (C) Istogramma mostra la distribuzione dei relativi effetti di fitness. Vengono riportati i risultati di tali mutazioni con> 100 conteggi ottenuti dal sequenziamento della cultura pre-selezione. Cliccate qui per vedere una versione più grande di questa figura.
| Reagente | Messa o Volume | Commento |
| Typtone | 2 g | |
| Estratto di lievito | 0,5 g | |
| Cloruro di sodio | 0,06 g | |
| Cloruro di potassio | 0,02 g | |
| Solfato di magnesio | 0,24 g | |
| Acqua purificata | 100 ml |
Tabella 1. Super ottimale Broth (SOB). I nomi dei reagenti e quantità utilizzate nella preparazione di 100 ml SOB (Protocollo passo 1.1).
| Reagente | Messa o Volume | Commento |
| Typtone | 10 g | |
| Estratto di lievito | 5 g | |
| Cloruro di sodio | 10 g | |
| Acqua purificata | 1 L |
Tabella 2. Luria-Bertani Broth (LB). I nomi dei reagenti e quantità utilizzata nella preparazione di 1 L LB (passo protocollo 1.1).
| Reagente | Messa o Volume | Commento |
| Typtone | 10 g | |
| Estratto di lievito | 5 g | |
| Cloruro di sodio | 10 g | |
| agar | 15 g | |
| Acqua purificata | 1 L |
I nomi dei reagenti Tabella 3. LB-agar. E quantità utilizzate nella preparazione LB-agar (passo protocollo 1.1).
| Reagente | Volume | Commento |
| tampone 5x PCR | 1.450 ml | |
| additivo PCR | 1.450 ml | |
| 2 dNTP mM | 725 ml | 2 mm ogni nucleotide |
| 50 micron AatII_F o AvrII_R innesco | 145 ml | |
| 1 ng / ml plasmide pBR322_AvrII | 145 ml | |
| 2 unità / ml DNA polimerasi | 72,5 ml | |
| acqua | 363 ml |
Tabella 4. primo turno mutageni PCR Master Mix. I nomi dei reagenti e quantità per la preparazione della miscela master per il primo turno mutageno PCR (passo protocollo 2.2.1). Quantità totale è sufficiente per 290 microlitri 25 reazioni.
| Reagente | Volume | Commento |
| tampone 5x PCR | 1.450 ml | |
| additivo PCR | 1.450 ml | |
| 2 dNTP mM | 725 ml | 2 mm ogni nucleotide |
| 50 micron AatII_F Primer | 145 ml | |
| 50 micron AvrII_R Primer | 145 ml | |
| 2 unità / ml DNA polimerasi | 72,5 ml | |
| acqua | 2.973 ml |
Tabella 5. secondo turno mutageni PCR Master Mix. I nomi dei reagenti e quantità per la preparazione della miscela master per il secondo turno mutageno PCR (passo protocollo 2.2.2). Quantità totale è sufficiente per 290 microlitri 25 reazioni.
| Reagente | Volume | Commento |
| tampone 5x PCR | 20 microlitri | |
| additivo PCR | 20 microlitri | |
| 2 dNTP mM | 10 microlitri | 2 mm ogni nucleotide |
| 50 micron AvrII_F Primer | 2 ml | |
| 50 micron AatII_OP1_R - AatII_OP5_R fondo | 2 ml | un fondo per reazione, in coppia con rispettivi plasmide |
| 1 ng / ml pBR322_OP1-5 plasmide | 2 ml | un plasmide per reazione |
| 2 unità / ml DNA polimerasi | 1 ml | |
| acqua | 43 ml |
Tabella 6. Clonazione vettore PCR. I nomi dei reagenti e quantità per la preparazione della PCR per rendere i vettori di clonazione (step protocollo 2.3.2).
| Reagente | Volume | Commento |
| 10x tampone enzima di restrizione | 5 ml | |
| 4 unità / AvrII microlitri | 2,5 ml | |
| 20 unità / AatII microlitri | 0,5 ml | |
| DNA di restrizione digest | volume per 500 ng | |
| acqua | a 50 microlitri volume totale |
Tabella 7. Restriction Digest. I nomi dei reagenti e quantità per digest restrizione di vettori di clonazione e gruppi sub-biblioteca NNS (step protocollo 2.3.3).
| Reagente | Volume | Commento |
| tampone ligasi T4 DNA 10x | 5 ml | |
| NNS DNA purificato restrizione digerito sub-library gruppo | volume per 48 ng | |
| purificato DNA clonazione vettore restrizione digerito | volume per 52 ng | |
| 400 unità / ml di T4 DNA ligasi | 1 ml | |
| acqua | a 20 microlitri volume totale |
Tabella 8. legature nomi reagenti e quantità per legature di vettori di clonazione con NNS gruppi sub-libreria di restrizione digerito in un 1: 3. Vettore: inserire rapporto molare (passo protocollo 2.3.4).
| Reagente | Volume | Commento |
| tampone 5x PCR | 55 ml | |
| additivo PCR | 55 ml | |
| 2 dNTP mM | 27,5 ml | 2 mm ogni nucleotide |
| 2 unità / ml DNA polimerasi | 2,75 ml | |
| acqua | 113 ml |
Tabella 9. PCR Reagenti per la preparazione dei campioni per high-throughput Sequencing. I nomi dei reagenti e quantità per la preparazione di PCR Master Mix utilizzati per la de-multiplexing con primer ortogonali (4.1.1), isolando i gruppi sub-biblioteca NNS (step protocollo 4.1.2) e l'aggiunta di sequenze di indicizzazione (step protocollo 4.1.3). Quantità totale è sufficiente per 11 25 reazioni microlitri.
Discussione
Qui un protocollo è descritto per eseguire la valutazione funzionale di tutto il contenuto proteico librerie saturazione di mutagenesi, utilizzando la tecnologia di sequenziamento high-throughput. Un aspetto importante del metodo è l'uso di primer ortogonali durante la clonazione. In breve, ogni posizione aminoacido è randomizzati per mutageno PCR, e mescolati tra loro in gruppi di posizioni la cui azione combinata sequenza lunghezza è ospitato da high-throughput sequencing. Questi gruppi sono clonati in vettori plasmidici contenenti coppie di siti priming ortogonali, mescolati insieme e sottoposti a selezione, quindi de-multiplex utilizzando i primers ortogonali, e successivamente profonda sequenziato. Dal momento che le mutazioni sono confinati all'interno del sequenziamento leggono limite di lunghezza, questo approccio massimizza il numero di letture utili contenente mutazioni per i geni di dimensioni più lungo del sequenziamento lettura lunghezza. Inoltre, questa tecnica consente di "one-batch" selezione simultanea o dell'intero mutatiobiblioteca nal, riducendo il carico di lavoro, nonché la possibilità che le mutazioni sperimentano diversi livelli di selezione. In pratica, i passaggi critici del protocollo in gran parte dell'organizzazione riguardano: durante il processo di clonazione (passo protocollo 2.3) si deve garantire la corretta miscelazione di prodotti di PCR mutageni in gruppi e la loro successiva clonazione nel ortogonale sito di adescamento corretta vettore; durante la preparazione del campione per il sequenziamento (passo protocollo 4.1), i primer ortogonali corretti, così come primer per isolare ciascuno dei gruppi NNS sub-biblioteca e aggiungere sequenze di indicizzazione, deve essere utilizzato.
Le tre fasi principali del protocollo - costruzione biblioteca, selezione, e sequenziamento - possono essere modificati in vari aspetti. Durante biblioteca costruzione uno potrebbe introdurre mutazioni utilizzando una varietà di tecniche, per esempio, da soggetto a errori PCR, o costruendo il gene utilizzando oligonucleotidi sintetizzati mediante drogaggio in una piccola frazione del nucleotide alternativas 21. Si potrebbe costruire la biblioteca di includere mutazioni doppi o di ordine superiore all'interno segmenti della proteina (ad esempio, gruppi NNS sub-biblioteca), o in genotipo sfondi alternati 22. È importante sottolineare che tutte le modifiche al passo di costruzione biblioteca tuttavia devono soddisfare il criterio che la corrispondenza tra la posizione di mutazioni nella sequenza e il sequenziamento leggi lunghezza viene mantenuta. Questo criterio esclude pertanto l'applicazione del protocollo verso studi approfonditi di mutazioni multiple attraverso una proteina. Modifiche alla seconda parte del protocollo includono condizioni di selezione alternativi: (es., Temperatura, livelli di nutrienti) diversi tipi β-lattamici (o combinazioni) e concentrazioni, condizioni di stress esterni, tipo di host (ad esempio, diversi tipi di batteri), o diversi tempi di campionamento (ore o giorni). Ad esempio, nel lavoro precedente abbiamo esaminato gli effetti di fitness di tutte le mutazioni di acido singolo aminoin TEM-1 in diverse concentrazioni di ampicillina, e sotto la terza generazione cefalosporina cefotaxime 13. Per quanto riguarda il terzo gradino del protocollo, al momento non consiglia di deviare dalla scelta della piattaforma di sequenziamento qui utilizzato (vedi tabella dei materiali). Mentre il sequenziamento leggere le lunghezze sono infatti attualmente più in altre piattaforme, il numero di letture ottenibile è attualmente di gran lunga inferiore; in generale la precisione che l'effetto di una mutazione può essere determinato è proporzionale al numero di letture ottenute (vedi equazione nella fase protocollo 4.2.5).
In gran parte per semplicità, il protocollo utilizza TEM-1 β-lattamasi come sistema modello, ma la metodologia descritta qui può essere esteso ad altri sistemi per i quali una selezione di alto-rendimento o test di screening è a posto. La costruzione di tali dosaggi è comunque spesso non banale: in primo luogo, deve essere stabilita una strategia per la compartimentazione del gene (mutazione) e proteine insieme, Ad esempio all'interno di una cella, goccia di liquido (come in una piattaforma microfluidica), o mediante phage display. In secondo luogo, e soprattutto, una connessione quantitativa tra la funzione delle proteine e un fenotipo selezionabile, o idoneità, deve essere stabilita. Per gli enzimi coinvolti nel metabolismo o resistenza agli antibiotici, la capacità delle cellule di crescere in nutrienti drop-out o supporto antibiotici è spesso una funzione diretta dell'attività enzimatica. Un approccio più sintetico potrebbe essere utilizzata in altri sistemi, ad esempio collegando proteina-proteina legante affinità gene reporter (per es., Proteina fluorescente) espressione in batteri o lieviti 11,23, o utilizzando un substrato enzimatico fluorogenico in un sistema microfluidica 24 . Infine, un tale test deve essere scalabile, per affrontare la dimensione di una libreria mutagenesi complesso proteico.
In sintesi, un approccio basato su sequenziamento high-throughput per la valutazione funzionale di tutto il contenuto proteico librerie saturazione di mutagenesi è described qui. Centrale per l'approccio è la costruzione della libreria di mutagenesi all'interno segmenti lungo il gene, e l'utilizzazione di codici a barre di primer ortogonali per contrassegnare ciascun segmento di multiplexing e de-multiplexing biblioteca. Prevediamo che questo protocollo potrebbe essere facilmente applicato ad altre proteine per le quali è stata sviluppata una selezione di alto-rendimento appropriato o schermo.
Divulgazioni
The authors declare they have no competing financial interests
Riconoscimenti
R.R. acknowledges support from the National Institutes of Health (RO1EY018720-05), the Robert A. Welch Foundation (I-1366), and the Green Center for Systems Biology.
Materiali
| Name | Company | Catalog Number | Comments |
| Typtone | Research Products Intl. Corp. | T60060-1000.0 | |
| Yeast extract | Research Products Intl. Corp. | Y20020-500.0 | |
| Sodium chloride | Fisher Scientific | BP358-212 | |
| Potassium chloride | Sigma-Aldrich | P9333-500G | |
| Magnesium sulfate | Sigma-Aldrich | M7506-500G | |
| Agar | Fisher Scientific | BP1423-500 | |
| Tetracycline hydrochloride | Sigma-Aldrich | T7660-5G | |
| Petri plates | Corning | 351029 | |
| MATLAB | Mathworks | http://www.mathworks.com/products/matlab/ | |
| Oligonucleotide primers | Integrated DNA Technologies | https://www.idtdna.com/pages/products/dna-rna/custom-dna-oligos | 25 nmol scale, standard desalting |
| pBR322_AvrII | available upon request | pBR322 plasmid modified to contain AvrII restriction site downstream of the TEM-1 gene | |
| pBR322_OP1 – pBR322_OP5 | available upon request | five modified pBR322 plasmids each containing a pair of orthogonal priming sites | |
| Q5 high-fidelity DNA polymerase | New England Biolabs | M0491L | includes 5x PCR buffer and PCR additive (GC enhancer) |
| 15 ml conical tube | Corning | 430025 | |
| Multichannel pipettes (Eppendorf ResearchPlus) | Eppendorf | ||
| PCR plate, 96 well | Fisher Scientific | 14230232 | |
| 96 well plate seal | Excel Scientific | F-96-100 | |
| Veriti 96-well thermal cycler | Applied Biosystems | 4375786 | |
| 6x gel loading dye | New England Biolabs | B7024S | |
| Agarose | Research Products Intl. Corp. | 20090-500.0 | |
| Ethidium bromide | Bio-Rad | 161-0433 | |
| UV transilluminator (FOTO/Analyst ImageTech) | Fotodyne Inc. | http://www.fotodyne.com/content/ImageTech_gel_documentation | |
| EB buffer | Qiagen | 19086 | |
| 96-well black-walled, clear bottom assay plates | Corning | 3651 | |
| Lambda phage DNA | New England Biolabs | N3011S | |
| PicoGreen dsDNA reagent | Invitrogen | P7581 | dsDNA quantitation reagent, used in protocol step 2.2.4 |
| Victor 3 V microplate reader | PerkinElmer | ||
| DNA purification kit | Zymo Research | D4003 | |
| Microcentrifuge tubes | Corning | 3621 | |
| Long-wavelength UV illuminator | Fisher Scientific | FBUVLS-80 | |
| Agarose gel DNA extraction buffer | Zymo Research | D4001-1-100 | |
| AatII | New England Biolabs | R0117S | |
| AvrII | New England Biolabs | R0174L | |
| T4 DNA ligase | New England Biolabs | M0202S | |
| EVB100 electrocompetent E. coli | Avidity | EVB100 | |
| Electroporator (E. coli Pulser) | Bio-Rad | 1652102 | |
| Electroporation cuvettes | Bio-Rad | 165-2089 | |
| Spectrophotometer (Ultrospec 3100 pro) | Amersham Biosciences | 80211237 | |
| 50 ml conical tubes | Corning | 430828 | |
| Plasmid purification kit | Macherey-Nagel | 740588.25 | |
| 8 well PCR strip tubes | Axygen | 321-10-551 | |
| Qubit dsDNA HS assay kit | Invitrogen | Q32854 | dsDNA quantitation reagent |
| Qubit assay tubes | Invitrogen | Q32856 | |
| Qubit fluorometer | Invitrogen | Q32866 | |
| Ampicillin sodium salt | Akron Biotechnology | 50824296 | |
| MiSeq reagent kit v2 (500 cycles) | Illumina | MS-102-2003 | |
| MiSeq desktop sequencer | Illumina | http://www.illumina.com/systems/miseq.html | alternatively, one could sequence on Illumina HiSeq platform |
| FLASh software | John Hopkins University - open source | http://ccb.jhu.edu/software/FLASH/ | software to merge paired-end reads from next-generation sequencing data |
| AatII_F | GATAATAATGGTTTCTTAGACG TCAGGTGGC | ||
| AvrII_R | CTTCACCTAGGTCCTTTTAAAT TAAAAATGAAG | ||
| AvrII_F | CTTCATTTTTAATTTAAAAGGA CCTAGGTGAAG | ||
| AatII_OP1_R | ACCTGACGTCCGTATTTCAAC TGTCCGGTCTAAGAAACCATT ATTATCATGACATTAAC | ||
| AatII_OP2_R | ACCTGACGTCCGCTCACGGA GTGTACTAATTAAGAAACCATT ATTATCATGACATTAAC | ||
| AatII_OP3_R | ACCTGACGTCGTACGTCTGA ACTTGGGACTTAAGAAACCA TTATTATCATGACATTAAC | ||
| AatII_OP4_R | ACCTGACGTCCCGTTCTCGAT ACCAAGTGATAAGAAACCATT ATTATCATGACATTAAC | ||
| AatII_OP5_R | ACCTGACGTCGTCCGTCGGA GTAACAATCTTAAGAAACCAT TATTATCATGACATTAAC | ||
| OP1_F | GACCGGACAGTTGAAATACG | ||
| OP1_R | CGACGTACAGGACAATTTCC | ||
| OP2_F | ATTAGTACACTCCGTGAGCG | ||
| OP2_R | AGTATTAGGCGTCAAGGTCC | ||
| OP3_F | AGTCCCAAGTTCAGACGTAC | ||
| OP3_R | GAAAAGTCCCAATGAGTGCC | ||
| OP4_F | TCACTTGGTATCGAGAACGG | ||
| OP4_R | TATCACGGAAGGACTCAACG | ||
| OP5_F | AGATTGTTACTCCGACGGAC | ||
| OP5_R | TATAACAGGCTGCTGAGACC | ||
| Group1_F | ACACTCTTTCCCTACACGAC GCTCTTCCGATCTNNNNNGC ATTTTGCCTACCGGTTTTTGC | ||
| Group1_R | GTGACTGGAGTTCAGACGTG TGCTCTTCCGATCTNNNNNTC TTGCCCGGCGTCAAC | ||
| Group2_F | ACACTCTTTCCCTACACGAC GCTCTTCCGATCTNNNNNGA ACGTTTTCCAATGATGAGCAC | ||
| Group2_R | GTGACTGGAGTTCAGACGTG TGCTCTTCCGATCTNNNNNGT CCTCCGATCGTTGTCAGAAG | ||
| Group3_F | ACACTCTTTCCCTACACGAC GCTCTTCCGATCTNNNNNAG TAAGAGAATTATGCAGTGCTGCC | ||
| Group3_R | GTGACTGGAGTTCAGACGTG TGCTCTTCCGATCTNNNNNTC GCCAGTTAATAGTTTGCGC | ||
| Group4_F | ACACTCTTTCCCTACACGAC GCTCTTCCGATCTNNNNNCC AAACGACGAGCGTGACAC | ||
| Group4_R | GTGACTGGAGTTCAGACGTG TGCTCTTCCGATCTNNNNNGC AATGATACCGCGAGACCC | ||
| Group5_F | ACACTCTTTCCCTACACGAC GCTCTTCCGATCTNNNNNCG GCTGGCTGGTTTATTGC | ||
| Group5_R | GTGACTGGAGTTCAGACGTG TGCTCTTCCGATCTNNNNNTAT ATGAGTAAACTTGGTCTGACAG | ||
| 501_F | AATGATACGGCGACCACCGA GATCTACACTATAGCCTACAC TCTTTCCCTACACGAC | ||
| 502_F | AATGATACGGCGACCACCGA GATCTACACATAGAGGCACA CTCTTTCCCTACACGAC | ||
| 503_F | AATGATACGGCGACCACCGA GATCTACACCCTATCCTACAC TCTTTCCCTACACGAC | ||
| 504_F | AATGATACGGCGACCACCGA GATCTACACGGCTCTGAACA CTCTTTCCCTACACGAC | ||
| 505_F | AATGATACGGCGACCACCGA GATCTACACAGGCGAAGACA CTCTTTCCCTACACGAC | ||
| 701_R | CAAGCAGAAGACGGCATAC GAGATCGAGTAATGTGACTG GAGTTCAGACGTG | ||
| 702_R | CAAGCAGAAGACGGCATAC GAGATTCTCCGGAGTGACTG GAGTTCAGACGTG |
Riferimenti
- Hutchison, C. A., et al. Mutagenesis at a specific position in a DNA sequence. J Biol Chem. 253 (18), 6551-6560 (1978).
- Mullis, K. B., Faloona, F. A. Specific synthesis of DNA in vitro via a polymerase-catalyzed chain reaction. Methods Enzymol. 155, 335-350 (1987).
- Papworth, C., Bauer, J. C., Braman, J., Wright, D. A. Site-directed mutagenesis in one day with >80% efficiency. Strategies. 9 (3), 3-4 (1996).
- Higuchi, R., Krummel, B., Saiki, R. K. A general method of in vitro preparation and specific mutagenesis of DNA fragments: study of protein and DNA interactions. Nucleic Acids Res. 16 (15), 7351-7367 (1988).
- Rennell, D., Bouvier, S. E., Hardy, L. W., Poteete, A. R. Systematic mutation of bacteriophage T4 lysozyme. J Mol Biol. 222 (1), 67-88 (1991).
- Markiewicz, P., Kleina, L. G., Cruz, C., Ehret, S., Miller, J. H. Genetic studies of the lac repressor. XIV. Analysis of 4000 altered Escherichia coli lac repressors reveals essential and non-essential residues, as well as 'spacers' which do not require a specific sequence. J Mol Biol. 240 (5), 421-433 (1994).
- Kleina, L. G., Miller, J. H. Genetic studies of the lac repressor. XIII. Extensive amino acid replacements generated by the use of natural and synthetic nonsense suppressors. J Mol Biol. 212 (2), 295-318 (1990).
- Huang, W., Petrosino, J., Hirsch, M., Shenkin, P. S., Palzkill, T. Amino acid sequence determinants of beta-lactamase structure and activity. J Mol Biol. 258 (4), 688-703 (1996).
- Fowler, D. M., et al. High-resolution mapping of protein sequence-function relationships. Nat Methods. 7 (9), 741-746 (2010).
- Hietpas, R. T., Jensen, J. D., Bolon, D. N. Experimental illumination of a fitness landscape. Proc Natl Acad Sci U S A. 108 (19), 7896-7901 (2011).
- McLaughlin, R. N., Poelwijk, F. J., Raman, A., Gosal, W. S., Ranganathan, R. The spatial architecture of protein function and adaptation. Nature. 491 (7422), 138-142 (2012).
- Deng, Z., et al. Deep sequencing of systematic combinatorial libraries reveals beta-lactamase sequence constraints at high resolution. J Mol Biol. 424 (3-4), 150-167 (2012).
- Stiffler, M. A., Hekstra, D. R., Ranganathan, R. Evolvability as a Function of Purifying Selection in TEM-1 beta-Lactamase. Cell. 160 (5), 882-892 (2015).
- Matagne, A., Lamotte-Brasseur, J., Frere, J. M. Catalytic properties of class A beta-lactamases: efficiency and diversity. Biochem J. 330 (Pt2), 581-598 (1998).
- Salverda, M. L., De Visser, J. A., Barlow, M. Natural evolution of TEM-1 beta-lactamase: experimental reconstruction and clinical relevance. FEMS Microbiol Rev. 34 (6), 1015-1036 (2010).
- Weinreich, D. M., Delaney, N. F., Depristo, M. A., Hartl, D. L. Darwinian evolution can follow only very few mutational paths to fitter proteins. Science. 312 (5770), 111-114 (2006).
- Stewart, S. M., Fisher, M., Young, J. E., Lutz, W. Ampicillin levels in sputum, serum, and saliva. Thorax. 25 (3), 304-311 (1970).
- Giachetto, G., et al. Ampicillin and penicillin concentration in serum and pleural fluid of hospitalized children with community-acquired pneumonia. Pediatr Infect Dis J. 23 (7), 625-629 (2004).
- Ambler, R. P., et al. A standard numbering scheme for the class A beta-lactamases. Biochem J. 276 (Pt 1), 269-270 (1991).
- Magoc, T., Salzberg, S. L. FLASH: fast length adjustment of short reads to improve genome assemblies). Bioinformatics. 27 (21), 2957-2963 (2011).
- Melamed, D., Young, D. L., Gamble, C. E., Miller, C. R., Fields, S. Deep mutational scanning of an RRM domain of the Saccharomyces cerevisiae poly(A)-binding protein. RNA. 19 (11), 1537-1551 (2013).
- Bank, C., Hietpas, R. T., Jensen, J. D., Bolon, D. N. A systematic survey of an intragenic epistatic landscape. Mol Biol Evol. 32 (1), 229-238 (2015).
- Dove, S. L., Joung, J. K., Hochschild, A. Activation of prokaryotic transcription through arbitrary protein-protein contacts. Nature. 386 (6625), 627-630 (1997).
- Romero, P. A., Tran, T. M., Abate, A. R. Dissecting enzyme function with microfluidic-based deep mutational scanning. Proc Natl Acad Sci U S A. 112 (23), 7159-7164 (2015).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon