Method Article
Purificação e microRNA Profiling de Exosomes derivados do sangue e Meios de Cultura
Neste Artigo
Resumo
A presença de microRNAs (miRNAs estáveis) em exossomos gerou enorme interesse como um novo modo de comunicação intercelular, pela sua utilidade potencial como biomarcadores e como uma rota para a intervenção terapêutica. Aqui demonstramos purificação exosome de mídia e cultura de sangue, seguido por PCR quantitativo para identificar os miRNAs sendo transportados.
Resumo
MiRNAs estáveis estão presentes em todos os fluidos do corpo e em alguns miRNAs circulantes são protegidos da degradação por sequestração em pequenas vesículas chamadas exosomes. Exosomes podem fundir com a membrana plasmática que resulta na transferência de RNA e proteínas da célula-alvo. As suas funções biológicas incluem resposta imune, a apresentação de antigénio, e a comunicação intracelular. Entrega de miRNAs que podem regular a expressão do gene em células receptoras através do sangue, abriu novas possibilidades para intervenção alvo. Além de oferecer uma estratégia para a entrega de medicamentos ou agentes terapêuticos RNA, o conteúdo exosomal podem servir como biomarcadores que podem ajudar no diagnóstico, determinar as opções de tratamento e prognóstico.
Aqui vamos descrever o procedimento para analisar quantitativamente miRNAs e RNAs mensageiro (mRNA) de exossomos secretados no sangue e meio de cultura de células. Purificados exosomes será caracterizado através da análise de western blot para exosmarcadores Omal e PCR para mRNAs de interesse. Microscopia eletrônica de transmissão (TEM) e immunogold rotulagem será usado para validar exosomal morfologia e integridade. O RNA total será purificada a partir destes exosomes para assegurar que podemos estudar tanto mRNA e miRNA partir da mesma amostra. Depois de validar a integridade do RNA por Bioanalyzer, vamos realizar um meio de transferência quantitativa PCR em tempo real (qPCR) para identificar o miRNA exosomal usando Taqman Matriz de Baixa Densidade (TLDA) cartas e estudos de expressão gênica de transcrições de interesse.
Estes protocolos podem ser utilizados para quantificar as alterações na miRNAs exosomal em pacientes, modelos de roedor e meios de cultura de células antes e após a intervenção farmacológica. Conteúdo exosomal variar devido à fonte de origem e as condições fisiológicas de células que secretam exossomos. Essas variações podem fornecer uma visão sobre como as células e os sistemas de lidar com o estresse ou perturbações fisiológicas. Nossos dados representativos mostrar variations em miRNAs apresentam em exossomos purificados a partir do sangue mouse, sangue humano e os meios de cultura de células humanas.
Aqui vamos descrever o procedimento para analisar quantitativamente miRNAs e RNAs mensageiro (mRNA) de exossomos secretados no sangue e meio de cultura de células. Purificados exosomes será caracterizado através da análise de western blot para marcadores exosomal e PCR para mRNAs de interesse. Microscopia eletrônica de transmissão (TEM) e immunogold rotulagem será usado para validar exosomal morfologia e integridade. O RNA total será purificada a partir destes exosomes para assegurar que podemos estudar tanto mRNA e miRNA partir da mesma amostra. Depois de validar a integridade do RNA por Bioanalyzer, vamos realizar um meio de transferência quantitativa PCR em tempo real (qPCR) para identificar o miRNA exosomal usando Taqman Matriz de Baixa Densidade (TLDA) cartas e estudos de expressão gênica de transcrições de interesse.
Estes protocolos podem ser utilizados para quantificar as alterações na miRNAs exosomal in pacientes, modelos de roedores e meios de cultura de células antes e depois da intervenção farmacológica. Conteúdo exosomal variar devido à fonte de origem e as condições fisiológicas de células que secretam exossomos. Essas variações podem fornecer uma visão sobre como as células e os sistemas de lidar com o estresse ou perturbações fisiológicas. Nossos dados representativos mostram variações em miRNAs apresentam em exossomos purificados a partir do sangue mouse, sangue humano e os meios de cultura de células humanas
Introdução
MiRNAs noncoding Curtas modular a expressão do gene por ligação ao ARNm alvo. Semente sequência complementaridade de pares de bases ~ 7 permite miRNA se liguem ao ARNm alvo, resultando na inibição da tradução ou na redução na estabilidade do mRNA, o que pode resultar na diminuição da expressão da proteína alvo 1. Pesquisas na última década tem se mostrado inequivocamente um papel fundamental para miRNAs em mediar funções celulares. Também tem havido considerável esforço dirigido para dissecar miRNA alterações moleculares subjacentes a diversas doenças mediadas por 2,3. Além disso, a recente identificação de miRNAs estáveis em fluidos corporais 4-6 pavimentou o caminho para a sua utilização como novos biomarcadores passíveis de diagnóstico clínico.
Um modo de transporte miRNA em fluidos corporais é via exossomos, pequenas vesículas que transportam mRNAs, proteínas, mediadores lipídicos e miRNAs em células receptoras via cir arterial sistêmicaion 7-14. Isto resulta na modulação da expressão de genes em células do receptor e representa um novo mecanismo de comunicação celular. Por exemplo, as células são capazes de modular processos de imuno-regulação através da secreção e / ou absorventes exosomes contendo biomoléculas envolvidas na inflamação, tais como a interleucina-1β (IL1β), factor de necrose tumoral α (TNF), factor de crescimento transformante-β5 (TGFβ5) e os miRNAs que regulam estes genes 13. Como expressão de miRNA aberrante é uma característica comum em uma variedade de doenças humanas, estas moléculas oferecem novas oportunidades para a descoberta e validação de novos alvos terapêuticos 2.
Circulating miRNAs estão presentes em todos os fluidos corporais e é sabido que a composição do exosomes é diferente baseada nas células de origem a partir do qual eles foram libertados. Assim, eles oferecem um caminho para estudar o estado fisiológico das células e como as células alteram a sinalização atéts em resposta ao estresse, incluindo doenças. Estudar as alterações na composição exosome pode fornecer insights sobre a transdução de sinal e investigar a sua utilidade potencial como biomarcadores ou rotas de intervenção terapêutica.
Aqui vamos demonstrar a purificação de exossomos a partir de várias fontes baseadas em protocolos publicados. Estes exosomes será usado para o isolamento de ARN seguida por qPCR para identificar e medir os níveis de miRNAs presentes nas exosomes.
Protocolo
Todos os experimentos com amostras de sangue de humanos e roedores foram executados em conformidade com todas as diretrizes, regulamentos e agências reguladoras. Seres humanos foram registrados após o consentimento informado aprovado pela Drexel University College of Medicine Institutional Review Board e todos os procedimentos de estudos realizados com animais foram aprovados pelo Institutional Animal Care e do Comitê Use Drexel.
1. Purificação exossomo de sangue (~ 5,5 h)
DICAS
- Nós ressuspender exossomos a partir de um tubo de sangue (~ 10 ml de sangue humano e ~ 2 ml de sangue mouse) em 300 mL de tampão de lise RNA a partir do kit de isolamento miRNA miRvana.
- Para purificar exosomes de meios de cultura de células, o meio é centrifugado a 500 xg durante 10 min para remover as células e também filtrada através de um filtro de 0,22 um, após centrifugação a 12.000 x g. A purificação exosome de meios de cultura celularnão necessita de diluição em PBS, PBS ou a solução de lavagem do segundo passo de ultracentrifugação.
- Se forem utilizados os exosomes purificadas para estudos de proteómica, a adição de um passo de centrifugação em gradiente de sacarose após o passo de ultracentrifugação vai diminuir a quantidade de agregados de proteína e contaminantes 15.
- A morfologia observada de exosomes pode variar de acordo com a fonte e as etapas de processamento para a ETM. Não observamos a morfologia em forma de copo para exossomos, que foi anteriormente atribuído a diferenças no processamento da amostra 9,15. Nossas imagens TEM, incluindo a rotulagem immunogold são semelhantes aos relatos anteriores de diferentes fontes, incluindo sangue, biliar e cultura cortical derivado exosomes 9,16,17.
- Inverter EDTA tubo de revestimento que contém o sangue de 5x e coloque na posição vertical à temperatura ambiente por 10-60 min.
- Centrifugar a 2.000 xg durante 15 min a 4 ° C.
- Recolher a fase superior, o plasma, em tubos de 15 ml e guardar em gelo.
- Dilui-se fluido com um volume igual de PBS. Centrifugar 30 min a 2000 xg, a 4 ° C.
- Transferir para tubos de centrífuga, adicionar 1X PBS, para um volume total de 24 ml e centrifugar 45 min a 12000 xg, a 4 ° C.
- Transfira para os tubos de ultracentrifugação e centrifugar 2 horas a 110.000 xg, a 4 ° C.
- Ressuspender o pellet em PBS e centrifugar 1 hora a 100.000 x g a 4 ° C.
- Ressuspender o sedimento em tampão apropriado (tampão de lise de ARN).
- Ressuspender em PBS ou tampão RIPA para microscopia eletrônica e análise de western blot, respectivamente.
2. Purificação de RNA usando um kit de isolamento miRvana miRNA modificação (~ 1 hora)
DICAS
- Preparar a área para isolar RNA, usando equipamentos de proteção individual (EPI), limpando superfícies e pipetas com RNase Zap, e usando pontas com filtro estéreis e tubos. Embora o protocolo do fornecedor dá a opção de purificação só miRNA, sempre isolar RNA totais, incluindo mRNA e miRNA para que possamos estudar tanto a partir da mesma amostra.
- Adicionando um passo de tratamento de DNase na coluna remove vestígios de ADN que podem estar presentes. Dependendo da fonte de exosomes, o DNA foi relatado que estavam ausentes em exosomes purificadas a partir de algumas fontes, incluindo o rato e as linhas de mastócitos humanos 8 mas as sequências de ADN cromossómico foram detectados em exosomes purificadas a partir de meios de cultura de cardiomiócitos 18.
- Nós reduziu o volume de solução de lavagem de 1-350 ul (Passo 2.4), porque o fornecedor disponibiliza apenas suficiente uma solução de lavagem para uma adição de 700 ul. Se o volume não seja reduzido, não há solução suficiente deixada para utilizar todo o conteúdo do kit. Demais etapas foram realizadas de acordo com as recomendações do fabricante.
- Adicione 1/10 volume de aditivo miRNA homogeneizado (30 uL) e incubar em gelo durante 10 min.
- Extrai-se com um volume de ácido-fenol: clorofórmio, igual ao volume de lisado inicial. Centrifugar e recuperar a fase superior num novo tubo.
- Adicionar 1,25 volumes de etanol a 100% (375 ul). Vortex. Aplicar para filtro de cartucho e centrifugar 15 segundos a 10.000 x g.
- Adicione metade do volume de miRNA solução de lavagem recomendado 1 (350 mL) e centrifugar 15 segundos a 10.000 x g.
- Adicionar 10 ul de DNase 1 a 70 ul de tampão de RDD (Qiagen) para cada amostra. Adicionar 80 ul para a coluna e incubar 15 min temperatura ambiente.
- Adicionar uma solução de lavagem (350 ul) e de centrifugação de 15 segundos a 10.000 x g.
- Adicionar solução de lavagem de 2/3 (500 mL) e centrifugar 15 s a 10.000 x g. REPEAT.
- Eluir com 40 ul de 95 ° C sem nuclease H2O
- Determinar a concentração de ARN e diluir o ARN a 100 ng / mL.
3. miRNA Profiling de Exosomes de Blood Usando Taqman Low Density Array (TLDA) Cartões (~ 6,5 hr)
DICAS
- Preparar a área para isolar RNA, vestindo PPE, limpando superfícies e pipetas com RNase Zap, e usando pontas com filtro estéreis e tubos.
- Pré-amplificação passo (pré-amplificador) é recomendado para 1-350 ng de RNA (350-1,000 amostras ng não requerem passo de pré-amplificador, após a síntese de cDNA). É importante determinar se o pré-amplificador é necessário para a sua amostra porque você deve tratar todas as amostras em condições de igualdade. Passo para pré-amplificador adiciona custo e os valores de Ct mais baixo tem um cut off aceitável (32 em vez de 40).
- É útil para preparar a reacção de cDNA em tubos em vez de uma tira de chapa de acordo com o número de cartas que pode ser executado num único dia. Isto irá minimizar a congelação-descongelação de amostras de cDNA que não serão processados no mesmo dia.
- Exossomos provenientes de diferentes fontes têm difealugar quantidades de RNA. Este protocolo permite que um máximo de 3 ul de molde de ARN para a reacção de síntese de cDNA. Dependendo da fonte, a quantidade de RNA exosomal que pode ser isolado talvez limitada. Enquanto um tecido típico renderia o suficiente RNA que cDNA seria feito a partir de 100 ng de RNA em 3 mL de volume, exossomos de sangue humano ou de meios de cultura de células humanas, muitas vezes contêm menos RNA e cDNA deve ser feita a partir de material menos de partida. Rato exosomes sangue contêm níveis mais elevados de RNA e de pelo menos 100 ng em 3 ul pode ser obtida.
- Para preparar cDNA a partir de RNA usando Pools Megaplex purificada primers da transcriptase reversa (Grupo A e Grupo B contêm primers para os miRNAs em cartões microfluídicos matriz A e B), descongelar os componentes de reação no gelo.
- Rótulo dois tubos de 1,5 ml RNase para reação de RT com tanque primers ou Grupo B primários e adicionar componentes a fim gráfico. Adicionar pelo menos um volume adicional de componentes de dois ou mais reacções ou fazer uma mistura principal, tal como recomendado por tele fornecedor.
| Componentes da reacção RT | Concentração Componente | Volume de uma amostra (total V / amostra = 4,5 mL) |
| Água livre de nuclease | 0,20 mL | |
| Megaplex tampão RT (10X) | 1X | 0,80 mL |
| dNTPs (100 mM) | 4,4 mM | 0,20 mL |
| MgCl2 (25 mM) | 5mM | 0,90 mL |
| Inibidor de RNase (20U/μl) | 2 U | 0,10 mL |
| Megaplex RT Primers A ou B (10X) | 1X | 0,80 mL |
| MultiScribe transcriptase reversa | 75 U | 1,50 mL |
- Inverter tubos de 6 vezes e centorifuge brevemente. Em seguida, pipeta de 4,5 ul da mistura de reacção de RT em 8 MicroAmp Tiras de tubo ou uma placa de reacção óptica de 96 poços MicroAmp.
- Adicionam-se 100 ng de RNA e ajustar o volume para 3 mL de água tratada com DEPC ou adicionar 3 ul de RNA total, misture com a ponta da pipeta, e fechar os tubos ou de placas. Girar rapidamente e incubar no gelo por 5 min.
- Carregar a reacção para dentro da máquina de PCR e executá-la sob as seguintes condições (como recomendado pelo protocolo da Applied Biosystems Megaplex Piscinas):
| Etapa | Temp | Tempo |
| Ciclo (40x) | 16 ° C | 2 min |
| 42 ° C | 1 min | |
| 50 ° C | 1 seg | |
| Manter | 85 ° C | 5 min |
| Manter | 4 ° C, | ∞ |
- Armazenar de cDNA a -20 ° C (boa durante pelo menos uma semana), ou continuar a etapa de pré-amplificação que podem ser armazenadas durante uma semana.
- Descongele primers pré-amplificador para o Grupo A e Grupo B no gelo e inverter para misturar. Swirl Taqman Pré-Amp mestre misturar e manter em gelo também.
- Seguir o gráfico de adição de pelo menos um volume adicional de componentes de dois ou mais reacções ou fazer uma mistura principal, tal como recomendado pelo fornecedor.
| Componente Reação Pré-Amp | Volume de uma amostra |
| Água livre de nuclease | 7,5 mL |
| Taqman Mix Master PreAmp (2X) | 12,5 mL |
| Primers pré-amplificador Megaplex A ou B (10x) | 2,5 mL |
- Pipetar 2,5 mL RT product MicroAmp em tiras de 8-tubo ou uma placa de reacção óptica de 96 poços e dispensar MicroAmp 22,5 ul da mistura de reacção PreAmp para cada poço.
- Selar a placa, incubar no gelo 5 min e em seguida, coloque a amostra na máquina de PCR. Executar sob as seguintes condições.
| Etapa | Temp | Tempo |
| Manter | 95 ° C | 10 min |
| Manter | 55 ° C | 2 min |
| Manter | 72 ° C | 2 min |
| Ciclo (12x) | 95 ° C | 15 seg |
| 60 ° C | 4 min | |
| Manter | 99,9 ° C | 10 min |
| Manter | 4 ° C, | ∞ |
- Após a reacção do pré-amplificador é terminado, centrifugar brevemente os tubos ou as placas e, em seguida, adicionar 75 ul de 0,1 x TE pH 8,0 a cada poço ou tubo.
- Seal e misture e girar rapidamente. Armazenar o produto diluído durante uma semana a -20 ° C, ou continuar a carregar a placa.
- Combinar o seguinte num tubo de 1,5 ml RNase em gelo.
| Componente | Volume de um cartão |
| TaqMan Universal PCR Master Mix, No AmpErase UNG (2X) | 450 mL |
| Produto pré-amplificador diluído | 9 mL |
| Água livre de nuclease | 441 mL |
- Inverter o tubo de misturar e centrifugar brevemente. Pipetar 100 ul da mistura de reacção de PCR em cada porta do cartão de matriz MicroRNA Taqman.
- Centrifugar o cartão de 2 x 1.000 xg por 1 min. Selar a placa e executá-lo usando o modelo fornecido com os primers no manual da Applied Biosystems e os Taqman configurações de matriz densidade 384 bem baixa. Usamos Applied Biosystems 7900HT Rápido Real-Time PCR System.
4. Análise por ARNm Taqman (~ 1,5 h)
- Para preparar a reacção de qPCR, misturar os componentes da reacção, no fim do gráfico. Todas as amostras serão analisados com sondas de iniciadores para o gene de interesse, bem como rRNA 18S como normalizador do gene.
| Reacção de componentes Taqman | Volume de uma amostra |
| Água livre de nuclease | 7 ul (ajuste com base na amostra individual) |
| Taqman rápido Master Mix (2X) | 10 ul |
| Sondas Taqman iniciadores (20x) | 1 ul |
| cDNA a partir de 100 ng de ARN | 2 & mu, L (ajuste com base na amostra individual) |
- Executar a placa de 96 poços utilizando o bloco de 96 poços rápida, tal como recomendado pelo fabricante (Applied Biosystems 7900HT Rápido Real-Time PCR System).
Resultados
Após o isolamento exosomes de meios de cultura de sangue ou de células, a pureza das exosomes pode ser testado por microscopia electrónica (ME) e Western blot (Figuras 1A e 1B). Confirmámos a nossa preparação exosome partir de várias fontes com EM e western blot utilizando anticorpos múltiplos. Figura 1A mostra imagens EM confirmando que exosomes estão intactas com um diâmetro de 30 ~ -100 nm e contêm CD81 por imuno rotulagem. Marcadores exosomal comumente utilizados são Hsp70 e tetraspannin glicoproteínas família CD63, CD81 e CD9 14. Após a confirmação da integridade dos exosomes purificadas, foi realizada western blot para a Hsp70 na Figura 1B e revelou que contêm Hsp70 exosomes sozinho enquanto meios (controlo negativo) não. O traçado mostra que Bioanalyzer exosomes isoladas de meios de cultura de células RAW conter uma variedade de RNAs, mas que não contêm grandes quantidades de ARN ribossómico que são típicas no ARN de todacelular (Figura 1C). Figura 1D mostra a análise de qPCR de ARNm a partir de sangue inteiro e exosomes derivado do sangue. Nós somos capazes de isolar 500 ng de RNA exosomal de ~ 10 ml de sangue humano usando kit de isolamento miRNA miRvana. Os resultados indicam a presença de qPCR de mRNAs que codificam o factor de crescimento endotelial vascular e TNFa A (VEGFA) em exosomes purificado, bem como em todo o sangue.
Além qPCR para estudos de expressão de genes, o RNA total foi também utilizado para perfis de miRNA. O miRNA TLDA cartão versão 3.0 contém sondas Primer para ~ 758 miRNAs incluindo o controle endógeno RNA U6. Com a adição da fase de pré-amplificação, o fornecedor recomenda 30 ng de ARN para rodar uma placa de TLDA. Isto é útil para a análise de RNA a partir de exosomes purificados a partir de sangue ou de soro humano, que normalmente têm rendimentos mais baixos do que aqueles purificada a partir de sangue de camundongos. A análise miRNA mostrado na Figura 2 foi realizada com 250 ng de ARN de sangue de camundongosexossomos e 15-30 ng de RNA exosomal de sangue ou células endoteliais aórticas humanas (HAOEC). Nossos resultados indicam a presença de 89 miRNAs em exossomos obtidos a partir de sangue mouse, 209 miRNAs em exossomos de sangue humano, e 199 miRNAs em exossomos de meios de cultura de células humanas. Os dados representativos para os seis miRNAs é apresentado como o valor delta Ct normalizado ao ARN U6 endógeno para o sangue de roedores (Figura 2A), de sangue humano (Figura 2B) e HAOEC meios de cultura celular (Figura 2C). Enquanto miR-126 e miR-200c estavam ausentes em exossomos a partir do sangue de roedores ou mídia HAOEC respectivamente, os restantes quatro miRNAs estão presentes em todas as três amostras em quantidades variadas. Relativo ao exosomes purificadas de meio de cultura celular, o miR-223 é expresso em níveis mais elevados em exosomes de amostras de sangue humano e de roedores.

Figura 1. Transmission microscopia electrónica (TEM), a análise de Western Blot e de qRT-PCR para ARNm utilizando exosomes purificadas a partir de várias fontes. A) imagem TEM de sangue rato exossomos ressuspendido em 1% de glutaraldeído (esquerda) ou exossomos derivados de células RAW ressuspendido em paraformaldeído a 4%, marcado com 10 nm de ouro e de coelho anti-CD81 (à direita) e viu em Formvar grades de carbono revestidos por EM . análise (barra de escala = 100 nm) B) análise de Western de HSP70 em exossomos purificados a partir do sangue do mouse ou mídia exosome livres de + / -. 24 horas de incubação com células RAW 264.7 murino e ressuspendidas em tampão RIPA C) análise Bioanalyzer de RNA total de purificado de exossomos derivados de cultura de células RAW 264.7 media D) Total RNA purificadas de sangue total e exossomos obtidos a partir de um controle humano representante foi utilizado para comparar os níveis de TNF e VEGFA de expressão. Clique aquipara ver a figura maior.
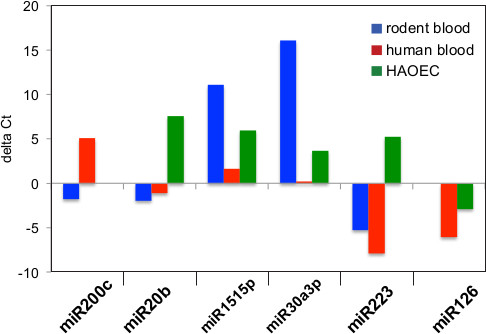
Figura 2. A expressão relativa da fracção miRNA encontrada em exosomes. Ciclo Limiar (valor CT) é uma medida relativa da concentração de um indivíduo miRNA na reacção de PCR e de menor valor TC indica maior expressão. valores qPCR para seis miRNAs exosomal detectáveis foram normalizados para RNA U6 e os valores de delta Ct foram representados graficamente para exossomos de três fontes. Um valor negativo indica a maior expressão nesta figura. Exossomos a partir do sangue de roedores (A) e sangue humano (B) expressa HSA-miR-223 em níveis superiores de controle, enquanto que exossomos de HAOEC meios de cultura de células (C) expressou HSA-miR-223 em níveis inferiores de controle. HSA-miR-226 estava ausente de roedores exosomes sangue e HSA-miR-200c estava ausente de exossomos HAOEC.
Discussão
Neste protocolo, vamos mostrar a quantificação dos miRNAs e mRNAs de exossomos purificados por centrifugação diferencial dos meios de sangue e cultura. Exosomes têm diversos componentes que dependem da sua origem e estão envolvidos em várias funções biológicas, incluindo a resposta imunológica, a apresentação de antigénio, a comunicação intracelular, bem como a transferência de RNA e proteínas 9,11,12,19,20. Embora a forma e tamanho é um determinante de pureza exossomo, uma série de artigos que mostram dados de EM exosomes indicam que estas vesículas podem variar em tamanho e morfologia com base na fonte a partir da qual eles são segregados, bem como o método utilizado para a fixação e imagiologia 9 , 15. O tamanho aceite para exosomes de 30-100 nm, uma gama média, que inclui uma pequena população de exosomes com um diâmetro maior, dependendo da fonte de 21,22, uma razão que são utilizados vários métodos para validar exosomal pureza. Secretadas miRNAs têm muitas featur requisitoes de bons biomarcadores 23, além de ser potenciais alvos terapêuticos para várias doenças 2,3,24. Este método de purificação fornece uma forma não invasiva para caracterizar mudanças biológicas temporais no conteúdo exosomal de uma variedade de fluidos corporais, enquanto miRNA profiling por qPCR fornece uma visão abrangente do conteúdo miRNA 2. Mais de 4500 proteínas, 1,639 mRNAs, 764 e 194 miRNAs lípidos são conhecidas para associar exosomes 25 ( www.exocarta.org ) e, em alguns casos, as alterações nos níveis destas biomoléculas já tem sido associada a estados de doença. Em nossos estudos, os exossomos purificados a partir do sangue humano tinha muitos miRNAs em comum com exossomos purificadas de mídia das linhas de cultura de células humanas, mas diferenças significativas de miRNAs exosomal encontradas no sangue do mouse. Embora haja exemplos de miRNAs que têm como alvo um único ARNm, muitas miRNAs funcionar parcialmente para reduzir os níveis de multiple metas levando a uma forte resposta fisiológica. Caracterizar tanto mRNA e miRNA que residem em exossomos fornece uma visão única sobre exosome mediada transferência de informação eo papel dos miRNAs circulando na mediar mudanças na expressão gênica. Purificação e caracterização de exosomes de uma variedade de fontes, será benéfico em identificar assinaturas moleculares associados com essas vesículas secretoras.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Este estudo foi apoiado por fundos de Rita Allen doação da Fundação para Seena Ajit. Os autores gostariam de agradecer Erika Balogh e Dr. Soumitra Ghoshroy da Universidade da Carolina do Sul Centro de Microscopia Eletrônica para o uso do instrumento, assistência técnica e científica.
Materiais
| Name | Company | Catalog Number | Comments |
| EDTA coated vacutainer (10 ml tubes) | BD Diagnostics | 366643 | For exosome purification from human blood |
| EDTA coated vacutainer (2 ml tubes) | BD Diagnostics | 367841 | For exosome purification from mouse blood |
| PAXgene Blood RNA Tube | BD Diagnostics | 762165 | For miRNA isolation from total blood |
| miRVana microRNA isolation kit | Ambion | AM1561 | |
| Acid-Phenol: CHCl3 | Ambion | 9721G | |
| DNase 1 | Qiagen | 79254 | |
| TaqMan Universal PCR Master Mix, No AmpErase UNG | Applied Biosystems | 4326614 | For TLDA cards |
| Megaplex RT rodent Pool Set v3.0 | Applied Biosystems | 4444746 | |
| Megaplex preamp rodent Pool set v3.0 | Applied Biosystems | 4444747 | |
| Taqman Array Rodent MicroRNA A+B set V3.0 | Applied Biosystems | 4444909 | |
| Megaplex RT Human Pool set V3.0 | Applied Biosystems | 4444745 | |
| Megaplex Preamp human pool set v3.0 | Applied Biosystems | 4444748 | |
| Taqman Array Human MicroRNA A+B Cards Set v3.0 | Applied Biosystems | 4444913 | |
| Taqman MicroRNA RT kit | Applied Biosystems | 4366596 | |
| Taqman fast universal PCR master mix | Applied Biosystems | 4366072 | For mRNA qRT-PCR |
| Tumor necrosis factor (primer probe) | Applied Biosystems | Hs01113624_g1 | |
| Vascular endothelial growth factor A (primer probe) | Applied Biosystems | Hs00900055_m1 | |
| Maxima First Strand cDNA Synthesis Kit for RT-qPCR | Thermo Scientific | K1642 | For mRNA |
| HSP70 antibody | Abcam | ab94368 | For western blot |
| Anti-rabbit IgG-Gold | Sigma | G7402 | For electron microscopy |
| Rabbit-anti CD81 | Sigma | SAB3500454 | |
| Nickel 300 mesh carbon formvar grids | Electron Microscopy Sciences | FCF300-Ni | |
| Copper 300 mesh carbon formvar grids | Electron Microscopy Sciences | FCF300-Cu | |
| Table 1. Table of specific reagents. | |||
Referências
- Bartel, D. P. MicroRNAs: target recognition and regulatory functions. Cell. 136, 215-233 (2009).
- Mendell, J. T., Olson, E. N. MicroRNAs in stress signaling and human disease. Cell. 148, 1172-1187 (2012).
- Esteller, M. Non-coding RNAs in human disease. Nature reviews. Genetics. 12, 861-874 (2011).
- Mitchell, P. S., et al. Circulating microRNAs as stable blood-based markers for cancer detection. Proceedings of the National Academy of Sciences of the United States of America. 105, 10513-10518 (2008).
- Chen, X., et al. Characterization of microRNAs in serum: a novel class of biomarkers for diagnosis of cancer and other diseases. Cell research. 18, 997-1006 (2008).
- Gilad, S., et al. Serum microRNAs are promising novel biomarkers. PloS one. 3, e3148 (2008).
- Mittelbrunn, M., et al. Unidirectional transfer of microRNA-loaded exosomes from T cells to antigen-presenting cells. Nature. 2, 282 (2011).
- Valadi, H., et al. Exosome-mediated transfer of mRNAs and microRNAs is a novel mechanism of genetic exchange between cells. Nat Cell Biol. 9, 654-659 (2007).
- Gyorgy, B., et al. Membrane vesicles, current state-of-the-art: emerging role of extracellular vesicles. Cell Mol Life Sci. 68, 2667-2688 (2011).
- Mathivanan, S., Ji, H., Simpson, R. J. Exosomes: extracellular organelles important in intercellular communication. J. Proteomics. 73, 1907-1920 (2010).
- Ramachandran, S., Palanisamy, V. Horizontal transfer of RNAs: exosomes as mediators of intercellular communication. Wiley Interdiscip Rev. RNA. , (2011).
- Record, M., Subra, C., Silvente-Poirot, S., Poirot, M. Exosomes as intercellular signalosomes and pharmacological effectors. Biochem Pharmacol. 81, 1171-1182 (2011).
- Thery, C., Ostrowski, M., Segura, E. Membrane vesicles as conveyors of immune responses. Nat Rev Immunol. 9, 581-593 (2009).
- Ludwig, A. K., Giebel, B. Exosomes: small vesicles participating in intercellular communication. The international journal of biochemistry & cell biology. 44, 11-15 (2012).
- Thery, C., Amigorena, S., Raposo, G., Clayton, A. Isolation and characterization of exosomes from cell culture supernatants and biological fluids. Curr. Protoc. Cell Biol. Chapter 3, Unit 3 22 (2006).
- Masyuk, A. I., et al. Biliary exosomes influence cholangiocyte regulatory mechanisms and proliferation through interaction with primary cilia. American journal of physiology. Gastrointestinal and liver physiology. 299, G990-G999 (2010).
- Fauré, J., et al. Exosomes are released by cultured cortical neurones. Molecular and Cellular Neuroscience. 31, 642-648 (2006).
- Waldenstrom, A., Genneback, N., Hellman, U., Ronquist, G. Cardiomyocyte microvesicles contain DNA/RNA and convey biological messages to target cells. PloS one. 7, e34653 (2012).
- Simpson, R. J., Lim, J. W., Moritz, R. L., Mathivanan, S. Exosomes: proteomic insights and diagnostic potential. Expert Rev Proteomics. 6, 267-283 (2009).
- Turchinovich, A., Weiz, L., Langheinz, A., Burwinkel, B. Characterization of extracellular circulating microRNA. Nucleic acids research. 39, 7223-7233 (2011).
- El-Andaloussi, S., et al. Exosome-mediated delivery of siRNA in vitro and in vivo. Nature. 7, 2112-2126 (2012).
- Singh, P. P., Smith, V. L., Karakousis, P. C., Schorey, J. S. Exosomes isolated from mycobacteria-infected mice or cultured macrophages can recruit and activate immune cells in vitro and in vivo. J. Immunol. 189, 777-785 (2012).
- Etheridge, A., Lee, I., Hood, L., Galas, D., Wang, K. Extracellular microRNA: A new source of biomarkers. Mutat. Res. , (2011).
- Stenvang, J., Silahtaroglu, A. N., Lindow, M., Elmen, J., Kauppinen, S. The utility of LNA in microRNA-based cancer diagnostics and therapeutics. Seminars in cancer biology. 18, 89-102 (2008).
- Mathivanan, S., Fahner, C. J., Reid, G. E., Simpson, R. J. ExoCarta 2012: database of exosomal proteins, RNA and lipids. Nucleic acids research. 40, D1241-D1244 (2012).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados