Method Article
Purificazione e microRNA profiling di Exosomes derivati dal sangue e cultura dei media
In questo articolo
Riepilogo
La presenza di microRNA stabili (miRNA) in exosomes ha generato enorme interesse come un romanzo modalità di comunicazione intercellulare, per la loro potenziale utilità come biomarcatori e come via per l'intervento terapeutico. Qui mostriamo purificazione exosome dal sangue e cultura mediatica seguita da PCR quantitativa per identificare miRNA trasportati.
Abstract
MiRNA stabili sono presenti in tutti i fluidi corporei e di alcuni microRNA circolanti sono protetti dalla degradazione di sequestro in piccole vescicole chiamate exosomes. Exosomes possono fondersi con la membrana plasmatica con conseguente trasferimento di RNA e proteine per la cellula bersaglio. Le loro funzioni biologiche includono la risposta immunitaria, presentazione dell'antigene, e la comunicazione intracellulare. Consegna dei miRNA che possono regolare l'espressione genica nelle cellule riceventi attraverso il sangue ha aperto nuove strade per l'intervento di destinazione. Oltre ad offrire una strategia per la consegna di farmaci o RNA agenti terapeutici, contenuti exosomal possono servire come biomarcatori che possono aiutare nella diagnosi, determinare le opzioni di trattamento e la prognosi.
Qui descriveremo la procedura per analizzare quantitativamente miRNA e RNA messaggero (mRNA) da exosomes secreti nel sangue e nei mezzi di coltura cellulare. Exosomes purificati saranno caratterizzati mediante analisi Western Blot per exosmarcatori Omal e PCR per mRNA di interesse. Microscopia elettronica a trasmissione (TEM) e l'etichettatura immunogold verranno utilizzati per convalidare la morfologia e l'integrità exosomal. L'RNA totale saranno purificati da questi exosomes per assicurare che siamo in grado di studiare sia l'mRNA e miRNA dallo stesso campione. Dopo la convalida integrità dell'RNA da Bioanalyzer, eseguiremo una produttività quantitativa real time PCR media (qPCR) per identificare il miRNA exosomal utilizzando Taqman Low Density Array (TLDA) carte e studi di espressione genica per le trascrizioni di interesse.
Questi protocolli possono essere usati per quantificare variazioni miRNAs exosomal in pazienti, modelli di roditori e terreni di coltura cellulare prima e dopo l'intervento farmacologico. Contenuto Exosomal variabili per la fonte di origine e le condizioni fisiologiche di cellule che secernono exosomes. Queste variazioni possono fornire informazioni su come le cellule e sistemi di far fronte allo stress o perturbazioni fisiologiche. I nostri dati rappresentativi mostrano variations in miRNA presenti in exosomes purificate dal sangue del mouse, sangue umano e mezzi di coltura di cellule umane.
Qui descriveremo la procedura per analizzare quantitativamente miRNA e RNA messaggero (mRNA) da exosomes secreti nel sangue e nei mezzi di coltura cellulare. Exosomes purificati saranno caratterizzati mediante analisi western blot per i marcatori exosomal e PCR per mRNA di interesse. Microscopia elettronica a trasmissione (TEM) e l'etichettatura immunogold verranno utilizzati per convalidare la morfologia e l'integrità exosomal. L'RNA totale saranno purificati da questi exosomes per assicurare che siamo in grado di studiare sia l'mRNA e miRNA dallo stesso campione. Dopo la convalida integrità dell'RNA da Bioanalyzer, eseguiremo una produttività quantitativa real time PCR media (qPCR) per identificare il miRNA exosomal utilizzando Taqman Low Density Array (TLDA) carte e studi di espressione genica per le trascrizioni di interesse.
Questi protocolli possono essere usati per quantificare variazioni miRNA exosomal in pazienti, modelli di roditori e terreni di coltura delle cellule prima e dopo l'intervento farmacologico. Contenuto Exosomal variabili per la fonte di origine e le condizioni fisiologiche di cellule che secernono exosomes. Queste variazioni possono fornire informazioni su come le cellule e sistemi di far fronte allo stress o perturbazioni fisiologiche. I nostri dati rappresentativi mostrano variazioni di miRNA presenti in exosomes purificate dal sangue del mouse, sangue umano e mezzi di coltura di cellule umane
Introduzione
Brevi non codificanti miRNA modulano l'espressione genica legandosi al mRNA bersaglio. Seme sequenza di complementarità ~ 7 coppie di basi miRNA consente di legarsi al mRNA bersaglio conseguente inibizione della traduzione o nella riduzione della stabilità del mRNA, entrambi i quali possono portare a diminuzione dell'espressione della proteina bersaglio 1. La ricerca negli ultimi dieci anni ha inequivocabilmente dimostrato un ruolo fondamentale per il miRNA nel mediare le funzioni cellulari. C'è stato anche un notevole sforzo diretto verso la dissezione miRNA cambiamenti molecolari alla base di diverse malattie mediate 2,3. Inoltre, la recente identificazione di miRNA stabili in fluidi corporei 4-6 aperto la strada per il loro uso come nuovi biomarcatori suscettibili di diagnosi clinica.
Un modo di trasporto miRNA nei fluidi corporei è via exosomes, piccole vescicole che trasportano mRNA, proteine, mediatori lipidici, e miRNA alle cellule riceventi tramite cir arteriosa sistemicaione 7-14. Ciò si traduce in modulazione dell'espressione genica in cellule riceventi e rappresenta un nuovo meccanismo di comunicazione cellulare. Per esempio, le cellule possono modulare processi immuno-regolatori secernendo e / o exosomes assorbenti contenenti biomolecole coinvolte nella infiammazione, come interleuchina-1β (IL1β), fattore di necrosi tumorale-α (TNFa), fattore di crescita trasformante-β5 (TGFβ5), e i miRNA che regolano questi geni 13. Come espressione di miRNA aberrante è una caratteristica comune in una varietà di malattie umane, queste molecole offrono nuove interessanti opportunità per la scoperta e la validazione di nuovi bersagli terapeutici 2.
MicroRNA circolanti sono presenti in tutti i fluidi corporei ed è noto che la composizione del exosomes è diverso in base alle celle di origine da cui sono stati rilasciati. Così essi offrono una via per studiare lo stato fisiologico delle cellule e come le cellule cambiano a segnalazione anchets in risposta a stress comprese malattie. Studiare le alterazioni nella composizione exosome può fornire indicazioni in trasduzione del segnale e studiare il loro potenziale utilità come biomarcatori o percorsi di intervento terapeutico.
Qui dimostreremo la purificazione exosomes provenienti da fonti multiple basate su protocolli pubblicati. Questi exosomes saranno utilizzati per l'isolamento dell'RNA seguito da qPCR per identificare e misurare i livelli di miRNA presenti nelle exosomes.
Protocollo
Tutti gli esperimenti usando campioni di sangue da uomo e roditori sono stati eseguiti in conformità a tutte le direttive, i regolamenti e le agenzie di regolamentazione. Soggetti umani sono stati arruolati dopo aver dato il consenso informato come approvato dalla Drexel University College of Medicine Institutional Review Board e di tutte le procedure per gli studi condotti su animali sono stati approvati dalla Institutional Animal Care di Drexel e utilizzo Comitato.
1. Purificazione Exosome dal sangue (~ 5,5 ore)
CONSIGLI
- Noi Risospendere exosomes da una provetta di sangue (~ 10 ml di sangue umano e ~ 2 ml di sangue del mouse) in 300 ml di RNA tampone di lisi del kit di isolamento miRNA Mirvana.
- Per purificare exosomes da mezzi di coltura cellulare, il supporto viene centrifugato a 500 xg per 10 minuti per rimuovere le cellule ed anche filtrato attraverso un filtro di 0,22 micron dopo centrifugazione a 12.000 x g. La purificazione exosome da terreni di coltura cellularenon richiede una diluizione in PBS, il lavaggio PBS o il secondo passo ultracentrifugazione.
- Se vengono utilizzati i exosomes purificati per studi di proteomica, aggiungendo un gradiente di saccarosio fase di centrifugazione dopo il passo ultracentrifugazione diminuirà la quantità di aggregati proteici e contaminanti 15.
- La morfologia osservata di exosomes può variare a seconda sia della fasi di lavorazione per la TEM sorgente e. Non abbiamo osservato la morfologia a forma di coppa per exosomes che è stato precedentemente attribuite a differenze di trattamento del campione 9,15. Le nostre immagini TEM compreso l'etichettatura immunogold sono simili alle precedenti relazioni provenienti da diverse fonti, tra cui sangue, delle vie biliari e della cultura corticale derivato exosomes 9,16,17.
- Capovolgere tubo rivestito EDTA contenente il sangue 5x e posto in posizione verticale a temperatura ambiente per 10-60 min.
- Centrifugare a 2000 xg per 15 min a 4 ° C.
- Raccogliere lo strato superiore, il plasma, in 15 ml provette e tenere in ghiaccio.
- Diluire fluido con un volume uguale di PBS. Centrifugare 30 min a 2000 xga 4 ° C.
- Trasferimento di centrifugare provette, aggiungere 1X PBS per un volume totale di 24 ml e centrifugare 45 min a 12.000 xg a 4 ° C.
- Trasferimento a ultracentrifuga tubi e centrifugare 2 ore a 110.000 xg a 4 ° C.
- Risospendere il pellet in PBS e centrifugare 1 ora a 100.000 xg a 4 ° C.
- Risospendere il pellet in tampone appropriato (tampone di lisi RNA).
- Risospendere in PBS o RIPA buffer per la microscopia elettronica e western blot, rispettivamente.
2. RNA Purification Utilizzando un Mirvana Isolation Kit miRNA Modified (~ 1 ora)
CONSIGLI
- Preparare l'area per l'isolamento di RNA da indossare dispositivi di protezione individuale (DPI), pulendo le superfici e pipette con RNasi Zap, e l'utilizzo di puntali con filtro sterili e tubi. Anche se il protocollo dal venditore dà la possibilità di purificare solo miRNA, abbiamo sempre isolare l'RNA totale tra mRNA e miRNA in modo da poter studiare sia dallo stesso campione.
- Aggiunta di una fase di trattamento DNasi on-column rimuove le tracce di DNA che possono essere presenti. A seconda della sorgente di exosomes, il DNA è stato riportato essere assente in exosomes purificate da alcune fonti tra mouse e linee di mastociti umani 8 ma sequenze di DNA cromosomico è stato isolato exosomes purificati da supporti di cardiomiociti in coltura 18.
- Abbiamo ridotto il volume della soluzione di lavaggio 1-350 microlitri (Passo 2.4) perché il venditore fornisce abbastanza unica soluzione di lavaggio 1 per un 700 microlitri aggiunta. Se il volume non è ridotta non vi è sufficiente per la soluzione utilizzando l'intero contenuto del kit. Passaggi rimanenti sono state eseguite secondo le raccomandazioni del fabbricante.
- Aggiungere 1/10 VOLume di miRNA Additivo Omogenato (30 ml) e incubare in ghiaccio per 10 min.
- Estrarre con un volume di acido-fenolo: cloroformio pari al volume iniziale lisato. Centrifugare e recuperare la fase superiore in una nuova provetta.
- Aggiungere 1,25 volumi 100% di etanolo (375 ml). Vortex. Applicare al filtro a cartuccia e centrifugare 15 sec a 10.000 x g.
- Aggiungere la metà del volume di raccomandata miRNA soluzione di lavaggio 1 (350 ml) e centrifugare 15 sec a 10.000 x g.
- Aggiungere 10 microlitri DNasi 1-70 microlitri tampone RDD (Qiagen) per ogni campione. Aggiungere 80 microlitri di colonna e incubare 15 min a temperatura ambiente.
- Aggiungi Soluzione di lavaggio 1 (350 ml) e centrifugare 15 sec a 10.000 x g.
- Aggiungi Soluzione di lavaggio 2/3 (500 microlitri) e centrifugare 15 sec a 10.000 x g. REPEAT.
- Eluire con 40 microlitri 95 ° C nucleasi H 2 O.
- Determinare la concentrazione di RNA e diluire l'RNA a 100 ng / ml.
3. miRNA Profiling di Exosomes da Blood Utilizzando Taqman Low Density Array (TLDA) Cards (~ 6,5 ore)
CONSIGLI
- Preparare l'area per l'isolamento di RNA da indossare PPE, asciugandosi le superfici e pipette con RNasi Zap, e l'utilizzo di puntali con filtro sterili e tubi.
- Pre-amplificazione fase (pre-amp) è consigliato per 1-350 ng RNA (350-1,000 campioni ng non richiedono fase di pre-amp dopo sintesi del DNA). E 'importante determinare se pre-amp è necessario per il vostro campione, perché è necessario trattare tutti i campioni in condizioni di parità. Fase di pre-amp aggiunge costi e dei valori Ct hanno un basso cut off accettabile (32 invece di 40).
- È utile preparare la reazione cDNA in tubi striscia invece di una piastra a seconda del numero di carte che possono essere eseguiti in un solo giorno. Questo ridurrà al minimo congelamento-scongelamento dei campioni di cDNA che non saranno trattate il giorno stesso.
- Exosomes raccolti da diverse fonti hanno diffeaffittare quantità di RNA. Questo protocollo consente un massimo di 3 microlitri stampo di RNA per la reazione di sintesi di cDNA. A seconda della sorgente, la quantità di RNA exosomal che può essere isolata forse limitato. Mentre un tessuto tipico produrrebbe abbastanza RNA che cDNA sarebbe composto da 100 ng di RNA in 3 volumi microlitri, exosomes dal sangue o dal mezzo di coltura di cellule umane spesso contengono meno RNA e cDNA deve essere effettuato da meno materiale di partenza. Exosomes sangue topo presentano livelli di RNA e almeno 100 ng in 3 ml possono essere ottenuti.
- Per preparare cDNA da RNA purificato utilizzando Piscine Megaplex trascrittasi inversa primer (Pool A e Pool B contenere primer per i miRNA su schede di microfluidica array A e B), scongelare i componenti di reazione in ghiaccio.
- Etichetta due provette da 1,5 ml RNasi-free per la reazione RT con piscina A primer o Pool B primer e aggiungere componenti in modo grafico. Aggiungi almeno un volume aggiuntivo di componenti per 2 o più reazioni o fare un master mix come consigliato da tegli venditore.
| Componente reazione RT | Concentrazione Componente | Volume per 1 campione (totale V / campione = 4,5 ml) |
| Acqua priva di nucleasi | 0.20 ml | |
| Megaplex RT Buffer (10X) | 1X | 0,80 ml |
| dNTP (100 mm) | 4.4 mM | 0.20 ml |
| MgCl2 (25 mM) | 5 mM | 0,90 ml |
| RNase Inhibitor (20U/μl) | 2 U | 0,10 ml |
| Megaplex RT Primer A o B (10 volte) | 1X | 0,80 ml |
| MultiScribe trascrittasi inversa | 75 U | 1,50 ml |
- Invertire i tubi 6 volte e centrifuge brevemente. Poi, pipetta 4,5 microlitri mix di reazione RT in MicroAmp strisce da 8 provette o una piastra di reazione ottica a 96 pozzetti MicroAmp.
- Aggiungere 100 ng di RNA e di regolare il volume a 3 ml con acqua trattata DEPC o aggiungere 3 RNA totale microlitri, mescolate con la punta della pipetta, e sigillare le provette o la piastra. Spin brevemente e incubare in ghiaccio per 5 min.
- Caricare la reazione nella macchina di PCR e correre con le seguenti condizioni (come raccomandato dal Biosystems protocollo Piscine Megaplex Applicata):
| Tappa | Temp | Volta |
| Ciclo (40x) | 16 ° C | 2 min |
| 42 ° C | 1 min | |
| 50 ° C | 1 sec | |
| Tenere | 85 ° C | 5 min |
| Tenere | 4 ° C | ∞ |
- Conservare cDNA a -20 ° C (buono per almeno una settimana) o passare alla fase di pre-Amp, che possono essere conservati per una settimana.
- Scongelare primer preamplificazione per la Pool A e Pool B sul ghiaccio e miscelare. Swirl Taqman Pre-Amp Master Mix e tenere in ghiaccio anche.
- Seguire la tabella aggiungendo almeno un volume aggiuntivo di componenti per 2 o più reazioni o fare un master mix come raccomandato dal produttore.
| Componente Reazione Pre-Amp | Volume per 1 campione |
| Acqua priva di nucleasi | 7,5 microlitri |
| TaqMan PreAmp Master Mix (2X) | 12,5 ml |
| Megaplex Primer PreAmp A o B (10 volte) | 2,5 microlitri |
- Pipettare 2,5 ml RT produttorict in MicroAmp Strips 8-tubo o una piastra di reazione ottica a 96 pozzetti MicroAmp ed erogare 22,5 ml mix di reazione PreAmp in ciascun pozzetto.
- Sigillare la piastra, incubare in ghiaccio 5 min e quindi caricare il campione nella macchina PCR. Eseguire le seguenti condizioni.
| Tappa | Temp | Volta |
| Tenere | 95 ° C | 10 min |
| Tenere | 55 ° C | 2 min |
| Tenere | 72 ° C | 2 min |
| Ciclo (12x) | 95 ° C | 15 sec |
| 60 ° C | 4 min | |
| Tenere | 99,9 ° C | 10 min |
| Tenere | 4 ° C | ∞ |
- Dopo la reazione preamplificatore è finito, centrifugare brevemente le provette o la piastra e poi aggiungere 75 ml 0,1 X. TE pH 8.0 per ciascun bene o un tubo.
- Sigillare e mescolare e centrifugare brevemente. Conservare il prodotto diluito per una settimana a -20 ° C o proseguire per caricare la piastra.
- Unire la segue in un RNase-free 1,5 ml tubo in ghiaccio.
| Componente | Volume per 1 scheda |
| TaqMan Universal PCR Master Mix, No AmpErase UNG (2X) | 450 microlitri |
| PREAMP prodotto diluito | 9 ml |
| Acqua priva di nucleasi | 441 microlitri |
- Capovolgere la provetta per mescolare e centrifugare brevemente. Dispensare 100 ul miscela di reazione PCR in ogni porta della scheda Array MicroRNA Taqman.
- Centrifugare la scheda 2 x 1.000 xg per 1 minuto. Sigillare la carta ed eseguirlo utilizzando il modello fornito con i primer nel manuale Applied Biosystems e le Taqman impostazioni Array 384 e bassa densità. Usiamo Applied Biosystems 7900HT veloce Real-Time PCR.
4. mRNA Analisi per Taqman (~ 1,5 ore)
- Per preparare reazione di qPCR, miscelare i componenti di reazione nell'ordine nel grafico. Tutti i campioni saranno analizzati con sonde di primer per il gene di interesse e 18S rRNA come il gene normalizzatore.
| Componente Reazione Taqman | Volume per 1 campione |
| Acqua priva di nucleasi | 7 ml (regolare sulla base del campione individuale) |
| Taqman Veloce Master Mix (2X) | 10 microlitri |
| Sonde TaqMan Primer (20x) | 1 microlitri |
| cDNA da 100 ng di RNA | 2 & mu, L (regolare sulla base del campione individuale) |
- Esegui il pozzo piastra 96 tramite il blocco a 96 pozzetti veloce come consigliato dal produttore (Biosystems 7900HT veloce Real-Time PCR System Applicata).
Risultati
Dopo aver isolato exosomes da sangue o terreni di coltura cellulare, la purezza dei exosomes può essere testata mediante microscopia elettronica (EM) e western blot (Figure 1A e 1B). Abbiamo confermato la nostra preparazione exosome da varie fonti con EM e western blot utilizzando anticorpi multipli. Figura 1A mostra EM immagini confermando che exosomes sono intatte con un diametro di ~ 30 -100 nm e contengono CD81 mediante l'etichettatura immunogold. Marcatori exosomal comunemente utilizzati sono glicoproteine famiglia Hsp70 e tetraspannin CD63, CD81 e CD9 14. Dopo aver verificato l'integrità dei exosomes purificati, abbiamo effettuato western blot per Hsp70 in Figura 1B e ha dimostrato che exosomes contengono Hsp70, mentre solo i media (controllo negativo) non lo fa. La traccia Bioanalizzatore mostra che exosomes isolati da mezzi di coltura di cellule RAW contengono una varietà di RNA, ma non contengono grandi quantità di RNA ribosomiale che sono tipici RNA da tuttacella (Figura 1C). Figura 1D mostra analisi qPCR di mRNA da sangue intero e exosomes derivato dal sangue. Siamo in grado di isolare 500 ng di RNA exosomal da ~ 10 ml di sangue umano usando Mirvana Kit di isolamento miRNA. I risultati qPCR indicano la presenza di mRNA codificante il fattore di crescita endoteliale vascolare A e TNFa (VEGFA) in purificato exosomes così come in sangue intero.
Oltre alla qPCR per studi di espressione genica, l'RNA totale è stato utilizzato anche per miRNA profiling. Il miRNA TLDA scheda versione 3.0 contiene sonde primer per ~ 758 miRNA tra cui il controllo endogeno RNA U6. Con l'aggiunta della fase di pre-amplificazione, il fornitore consiglia 30 ng di RNA per eseguire una scheda TLDA. Ciò è utile per analizzare RNA da exosomes purificate dal sangue o siero umano che tipicamente hanno rendimenti bassi di quelli purificati da sangue topo. L'analisi miRNA mostrata in figura 2 è stata effettuata con 250 ng di RNA da sangue topoexosomes e 15-30 ng RNA exosomal dal sangue o cellule endoteliali aortiche umane (HAOEC). I nostri risultati indicano la presenza di 89 miRNA in exosomes raccolte dal sangue del mouse, 209 miRNA in exosomes sangue umano, e di 199 miRNA in exosomes da terreni di coltura di cellule umane. Dati rappresentativi per sei miRNA è indicato come delta Ct valore normalizzato al endogeno U6 RNA per il sangue dei roditori (Figura 2A), il sangue umano (Figura 2B) e HAOEC cella cultura dei media (Figura 2C). Mentre miR-126 e miR-200c erano assenti in exosomes dal sangue di roditori o supporti HAOEC rispettivamente, i restanti quattro miRNA sono presenti in tutti e tre i campioni in quantità variabili. Rispetto al exosomes purificati da colture cellulari, miR-223 è espressa ad alti livelli in exosomes da campioni di sangue umano e di roditori.

Figura 1. Trmicroscopia elettronica ansmission (TEM), western blot e RT-PCR quantitativa per mRNA utilizzando exosomes purificati da varie fonti. A) Immagine TEM di sangue del mouse exosomes risospeso in 1% glutaraldeide (a sinistra) o RAW cellulari exosomes derivate risospese in paraformaldeide al 4%, marcato con 10 nm d'oro e di coniglio anti-CD81 (a destra) e depositata su griglie di carbonio rivestite Formvar per EM . analisi (scala bar = 100 nm) B) analisi Western di HSP70 in exosomes purificate dal sangue del mouse o del supporto exosome-liberi + / -. 24 ore di incubazione con cellule RAW 264.7 murine e risospese in tampone RIPA C) analisi Bioanalizzatore di RNA totale purificati da exosomes derivati da RAW 264.7 cella cultura dei media D) L'RNA totale purificato da sangue intero e exosomes ottenuto da un controllo umano rappresentante è stato utilizzato per confrontare i livelli di espressione di TNFa e VEGFA. Clicca quiper ingrandire la figura.
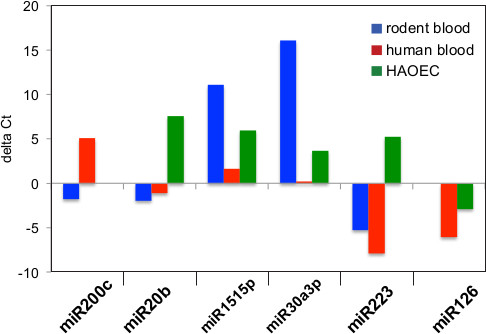
Figura 2. Ciclo Espressione relativa frazione di miRNA si trovano in exosomes. Soglia (valore CT) è una misura relativa della concentrazione di un miRNA individuale nella reazione PCR e valore più basso CT indica maggiore espressione. valori qPCR per sei miRNA exosomal rilevabili sono stati normalizzati per U6 RNA ei valori delta Ct sono stati rappresentati graficamente per exosomes da tre fonti. Un valore negativo indica una superiore espressione in questa figura. Exosomes dal sangue di roditori (A) e di sangue umano (B) espressi hsa-miR-223 a livelli più elevati di controllo, mentre exosomes da HAOEC cella cultura dei media (C) espresso hsa-miR-223 a livelli inferiori di controllo. Hsa-miR-226 era assente da roditori exosomes sangue e hsa-miR-200c era assente da exosomes HAOEC.
Discussione
In questo protocollo, mostriamo la quantificazione dei miRNA e mRNA da exosomes purificati mediante centrifugazione differenziale di sangue e di cultura dei media. Exosomes hanno diverse componenti che dipendono dalla loro origine e sono coinvolti in una serie di funzioni biologiche, compresa la risposta immunitaria, presentazione dell'antigene, comunicazione intracellulare, e il trasferimento di RNA e proteine 9,11,12,19,20. Mentre la dimensione e la forma è un determinante di purezza exosome, una serie di documenti che mostrano EM dati di exosomes indicano che queste vescicole possono variare in dimensione e morfologia basato sulla sorgente da cui sono secreti nonché il metodo utilizzato per il fissaggio e l'imaging 9 , 15. Il formato accettato per exosomes di 30-100 nm è una gamma media che comprende una piccola popolazione di exosomes di diametro maggiore a seconda della fonte 21,22, uno dei motivi che più metodi sono utilizzati per convalidare la purezza exosomal. Secrete miRNA hanno molti featuring requisitoes di buone biomarker 23 oltre ad essere potenziali bersagli terapeutici per varie malattie 2,3,24. Questo metodo di purificazione fornisce un modo non invasivo per caratterizzare cambiamenti biologici temporali nel contenuto exosomal da una varietà di fluidi corporei mentre profiling miRNA da qPCR fornisce una panoramica completa dei contenuti miRNA 2. Oltre 4.500 proteine, 1639 mRNA, miRNA 764 e 194 lipidi sono noti da associare exosomes 25 ( www.exocarta.org ) e, in alcuni casi, i cambiamenti nei livelli di queste biomolecole è già stato collegato a stati di malattia. Nei nostri studi, i exosomes purificate da sangue umano hanno avuto molti miRNA in comune con exosomes purificati da un supporto di linee di cellule umane in coltura, ma differenze significative dal miRNA exosomal trovati nel sangue del mouse. Mentre ci sono esempi di miRNA che colpiscono un solo mRNA, molti miRNA funzione per ridurre in parte i livelli di multipLe rivolge portando ad una forte risposta fisiologica. Caratterizzare sia mRNA e miRNA che risiedono in exosomes fornisce una visione unica exosome mediato trasferimento di informazioni e il ruolo dei microRNA circolanti nel mediare i cambiamenti nell'espressione genica. Purificazione e caratterizzazione di exosomes da una varietà di fonti sarà utile nell'identificare firme molecolari associati a queste vescicole secretorie.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo studio è stato sostenuto da fondi da Rita Allen sovvenzione della Fondazione per Seena Ajit. Gli autori desiderano ringraziare Erika Balogh e Dr. Soumitra Ghoshroy dall'Università di Centro Electron Microscopy South Carolina per l'uso dello strumento, l'assistenza scientifica e tecnica.
Materiali
| Name | Company | Catalog Number | Comments |
| EDTA coated vacutainer (10 ml tubes) | BD Diagnostics | 366643 | For exosome purification from human blood |
| EDTA coated vacutainer (2 ml tubes) | BD Diagnostics | 367841 | For exosome purification from mouse blood |
| PAXgene Blood RNA Tube | BD Diagnostics | 762165 | For miRNA isolation from total blood |
| miRVana microRNA isolation kit | Ambion | AM1561 | |
| Acid-Phenol: CHCl3 | Ambion | 9721G | |
| DNase 1 | Qiagen | 79254 | |
| TaqMan Universal PCR Master Mix, No AmpErase UNG | Applied Biosystems | 4326614 | For TLDA cards |
| Megaplex RT rodent Pool Set v3.0 | Applied Biosystems | 4444746 | |
| Megaplex preamp rodent Pool set v3.0 | Applied Biosystems | 4444747 | |
| Taqman Array Rodent MicroRNA A+B set V3.0 | Applied Biosystems | 4444909 | |
| Megaplex RT Human Pool set V3.0 | Applied Biosystems | 4444745 | |
| Megaplex Preamp human pool set v3.0 | Applied Biosystems | 4444748 | |
| Taqman Array Human MicroRNA A+B Cards Set v3.0 | Applied Biosystems | 4444913 | |
| Taqman MicroRNA RT kit | Applied Biosystems | 4366596 | |
| Taqman fast universal PCR master mix | Applied Biosystems | 4366072 | For mRNA qRT-PCR |
| Tumor necrosis factor (primer probe) | Applied Biosystems | Hs01113624_g1 | |
| Vascular endothelial growth factor A (primer probe) | Applied Biosystems | Hs00900055_m1 | |
| Maxima First Strand cDNA Synthesis Kit for RT-qPCR | Thermo Scientific | K1642 | For mRNA |
| HSP70 antibody | Abcam | ab94368 | For western blot |
| Anti-rabbit IgG-Gold | Sigma | G7402 | For electron microscopy |
| Rabbit-anti CD81 | Sigma | SAB3500454 | |
| Nickel 300 mesh carbon formvar grids | Electron Microscopy Sciences | FCF300-Ni | |
| Copper 300 mesh carbon formvar grids | Electron Microscopy Sciences | FCF300-Cu | |
| Table 1. Table of specific reagents. | |||
Riferimenti
- Bartel, D. P. MicroRNAs: target recognition and regulatory functions. Cell. 136, 215-233 (2009).
- Mendell, J. T., Olson, E. N. MicroRNAs in stress signaling and human disease. Cell. 148, 1172-1187 (2012).
- Esteller, M. Non-coding RNAs in human disease. Nature reviews. Genetics. 12, 861-874 (2011).
- Mitchell, P. S., et al. Circulating microRNAs as stable blood-based markers for cancer detection. Proceedings of the National Academy of Sciences of the United States of America. 105, 10513-10518 (2008).
- Chen, X., et al. Characterization of microRNAs in serum: a novel class of biomarkers for diagnosis of cancer and other diseases. Cell research. 18, 997-1006 (2008).
- Gilad, S., et al. Serum microRNAs are promising novel biomarkers. PloS one. 3, e3148 (2008).
- Mittelbrunn, M., et al. Unidirectional transfer of microRNA-loaded exosomes from T cells to antigen-presenting cells. Nature. 2, 282 (2011).
- Valadi, H., et al. Exosome-mediated transfer of mRNAs and microRNAs is a novel mechanism of genetic exchange between cells. Nat Cell Biol. 9, 654-659 (2007).
- Gyorgy, B., et al. Membrane vesicles, current state-of-the-art: emerging role of extracellular vesicles. Cell Mol Life Sci. 68, 2667-2688 (2011).
- Mathivanan, S., Ji, H., Simpson, R. J. Exosomes: extracellular organelles important in intercellular communication. J. Proteomics. 73, 1907-1920 (2010).
- Ramachandran, S., Palanisamy, V. Horizontal transfer of RNAs: exosomes as mediators of intercellular communication. Wiley Interdiscip Rev. RNA. , (2011).
- Record, M., Subra, C., Silvente-Poirot, S., Poirot, M. Exosomes as intercellular signalosomes and pharmacological effectors. Biochem Pharmacol. 81, 1171-1182 (2011).
- Thery, C., Ostrowski, M., Segura, E. Membrane vesicles as conveyors of immune responses. Nat Rev Immunol. 9, 581-593 (2009).
- Ludwig, A. K., Giebel, B. Exosomes: small vesicles participating in intercellular communication. The international journal of biochemistry & cell biology. 44, 11-15 (2012).
- Thery, C., Amigorena, S., Raposo, G., Clayton, A. Isolation and characterization of exosomes from cell culture supernatants and biological fluids. Curr. Protoc. Cell Biol. Chapter 3, Unit 3 22 (2006).
- Masyuk, A. I., et al. Biliary exosomes influence cholangiocyte regulatory mechanisms and proliferation through interaction with primary cilia. American journal of physiology. Gastrointestinal and liver physiology. 299, G990-G999 (2010).
- Fauré, J., et al. Exosomes are released by cultured cortical neurones. Molecular and Cellular Neuroscience. 31, 642-648 (2006).
- Waldenstrom, A., Genneback, N., Hellman, U., Ronquist, G. Cardiomyocyte microvesicles contain DNA/RNA and convey biological messages to target cells. PloS one. 7, e34653 (2012).
- Simpson, R. J., Lim, J. W., Moritz, R. L., Mathivanan, S. Exosomes: proteomic insights and diagnostic potential. Expert Rev Proteomics. 6, 267-283 (2009).
- Turchinovich, A., Weiz, L., Langheinz, A., Burwinkel, B. Characterization of extracellular circulating microRNA. Nucleic acids research. 39, 7223-7233 (2011).
- El-Andaloussi, S., et al. Exosome-mediated delivery of siRNA in vitro and in vivo. Nature. 7, 2112-2126 (2012).
- Singh, P. P., Smith, V. L., Karakousis, P. C., Schorey, J. S. Exosomes isolated from mycobacteria-infected mice or cultured macrophages can recruit and activate immune cells in vitro and in vivo. J. Immunol. 189, 777-785 (2012).
- Etheridge, A., Lee, I., Hood, L., Galas, D., Wang, K. Extracellular microRNA: A new source of biomarkers. Mutat. Res. , (2011).
- Stenvang, J., Silahtaroglu, A. N., Lindow, M., Elmen, J., Kauppinen, S. The utility of LNA in microRNA-based cancer diagnostics and therapeutics. Seminars in cancer biology. 18, 89-102 (2008).
- Mathivanan, S., Fahner, C. J., Reid, G. E., Simpson, R. J. ExoCarta 2012: database of exosomal proteins, RNA and lipids. Nucleic acids research. 40, D1241-D1244 (2012).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon