Method Article
레이저 캡처 미세 절 RNA-시퀀싱 용 공간 및 측두 조직 -특정 유전자 발현 분석
요약
여기에 제시된 것은 식물 조직의 레이저 포획 미세 절(LCM)을 위한 프로토콜입니다. LCM은 오염없는 방식으로 조직의 영역을 격리하기위한 현미경 기술입니다. 절차는 조직 고정, 파라핀 포함, 단면, LCM 및 RNA 추출을 포함한다. RNA는 다운스트림 조직 특이적, 전사의 현세적으로 해결된 분석에서 사용된다.
초록
복잡한 다세포 유기체의 발달은 상이한 전사 단면도가 있는 명백한 세포 모형에 의해 지배됩니다. 발달 과정을 제어하는 전사 적 규제 네트워크를 식별하려면 이러한 개별 세포 유형의 공간 및 측두성 유전자 발현 프로파일을 측정할 필요가 있습니다. 따라서 유전자 발현의 현세-측량 제어에 대한 통찰력은 생물학적 및 발달 과정이 어떻게 규제되는지 이해하는 데 필수적입니다. 여기서는 발아 중에 시간 과정을 통해 3개의 보리 배아 기관에서 소수의 세포를 분리하는 레이저 포획 미세 절(LCM) 방법을 설명하고 전사체 프로파일링을 실시한다. 이 방법은 조직 고정, 조직 처리, 파라핀 포함, 단면, LCM 및 RNA 추출 다음으로 실시간 PCR 또는 RNA-seq로 구성됩니다. 이 방법을 통해 종자 장기 전사의 공간적 및 측두프로파일을 다양한 수의 세포(수십~수백 개)에서 얻을 수 있게 되어 일반적인 벌크 조직 분석보다 훨씬 더 큰 조직 특이성을 제공합니다. 이러한 데이터에서 우리는 전사 적 규제 네트워크를 정의하고 비교할 뿐만 아니라 개별 조직에 대한 후보 규제 전사 요인을 예측할 수 있었습니다. 이 방법은 최소한의 최적화를 가진 다른 플랜트 조직에 적용되어야 합니다.
서문
식물 개발 및 성장은 복잡한 세포 환경에 존재하는 다른 세포 내의 전사 규제 네트워크의 조정 된 작용을 포함한다. 이러한 규제 네트워크의 활동을 이해하기 위해, 우리는 발달 단계에 걸쳐 다른 세포 모형 내의 공간 및 측두성 유전자 발현의 지식을 요구합니다. 그러나, 유전자 발현의 분석은 세포의 작은 숫자를 격리하고 분석의 기술적 인 도전 때문에 전체 기관 또는 대량 조직 샘플에서 더 일반적으로 수행됩니다. 여기서 설명하는 방법은 LCM과 RNA-seq를 결합하여 공간 및 측두조직 특이적 전사 분석을 얻을 수 있게 하였다.
LCM은 에머트 벅과 동료에머트벅에 의해 이십 년 전에 개발되었다 1 . 이 기술을 통해 연구자들은 좁은 빔 레이저1을사용하여 직접 현미경 시각화 및 조작을 사용하여 단일 세포 또는 세포 클러스터를 환경으로부터 정확하게 분리할 수 있었습니다. 그 이후로 이 방법은 암 생물학 및 병리학2,,3에서널리 사용되고 있다. ,많은 식물 연구 그룹은 또한 다른 식물 종 및 다른 조직 유형,4,,5,,6,7,8,9,10,11과함께 사용하기 위해,LCM을적응시켰다. 최근에는, 몇몇 논문은 또한 종자 발달 및 발아 도중 배아, 내배식 및 그밖 종자 구조물을 공부하기 위하여 eudicot 및 단장화장 씨앗에LCM을 이용했습니다10,,12,13. 마이크로 파이펫팅, 세포 선별, 자기 분리 및 미세 유체 플랫폼과 같은 다른 일반적으로 사용되는 단일 세포 격리 방법의 대부분은 세포를 해리하기 위해 효소 소화 또는 기계적 균질화에 의존한다. 이는 데이터해석(14,15)을혼동하는 기술 적 유물을 도입하여 유전자발현을방해할 수 있다. 이러한 방법은 또한 해리된 세포를 공간 위치 및 진정한 세포 유형에 연관시키기 위해 각 세포 유형에 대한 마커 유전자에 대한 이전 지식을 요구합니다. 기술의 추가 그룹은 전체 세포 대신 세포 체형 구조의 친화성 기반 절연에 따라 달라집니다, 예를 들어 INTACT (세포 유형에서 태그 핵의 격리) 및 TRAP (리보솜 친화 정화 번역)16,,17. 그러나 핵 이나 리보좀의 선호도 라벨링 및 정제는 잘 확립 된 변형 프로토콜이없는 식물 종에서 기술적으로 도전적입니다. LCM은 짧은기간동안 이산 세포를 단기간에 격리할 수 있도록 하는 정상 조직/장기 문맥 내에서 세포의 직접 시각화에 의한 성적증명서 수준 및 종래의 조직학적 식별을 보존하기 위해 빠른 조직 고정을활용한다.19
여기에 제시된 프로토콜은 조직학적으로 식별될 수 있는 대부분의 세포에 적용될 수 있는 시리얼 씨앗의 조직 섹션에서 특정 세포 또는 세포 유형의 분리를 위한 최적화된 방법입니다. LCM은 접촉없는 세포 격리 방법을 제공하여 오염을 크게 줄이고 회수 된 RNA의 무결성을 증가시킵니다. 더욱이, 이 방법은 소량의 생물학적 물질로 시작하는 대규모 게놈 넓은 연구에 대한 LCM의 힘을 보여줍니다. 우리는 또한 다운스트림 전사/전사 분석을 위한 충분한 입력 물질을 생성하기 위한 RNA의 선형 증폭을 기술합니다.
이 LCM RNA-seq 프로토콜에는 조직 샘플, 탈수, 파라핀 침투, 임베딩, 단면, LCM, RNA 추출, RNA 증폭, RNA 정량화 및 qRT-PCR 및/또는 RNA-seq(그림1)의고정을 포함하는 공간 및 측두조직 특이적 전사에 대한 10가지 주요 단계가 있습니다.
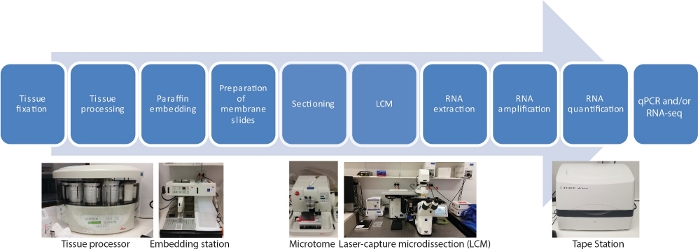
그림 1: LCM의 순서도는 RNA-seq 또는 qRT-PCR이 그 뒤를 따릅니다. LCM은 현미경 시각화 하에서 레이저 빔을 사용하여 고정 조직 섹션에서 세포를 수집하는 공간적으로 정확하고 접촉이 없는 기술입니다. 이 과정은 조직 샘플의 고정으로 시작하여 에탄올과 자일렌의 그라데이션 시리즈를 사용하여 탈수로 시작하여 파라핀 침투로 완성됩니다. 이 과정은 티슈 프로세서를 사용하여 완전히 자동화될 수 있습니다. 조직이 파라핀으로 침투하면 포함 스테이션을 사용하여 용융 파라핀이있는 금형에 내장됩니다. 절제는 원하는 두께로 설정된 마이크로토메를 사용하여 수행됩니다. 슬라이드는 RNA가 포획된 세포에서 추출되기 직전에 제조되고 LCM이 수행됩니다. RNA 추출은 qRT-PCR 및/또는 RNA-seq 이전에 RNA 증폭의 2라운드에 의해 직접 뒤따릅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
프로토콜
최종 제품은 RNA이기 때문에 RNases와 함께 작업을 오염하지 않도록주의하십시오. 장갑을 착용하는 것은 필수입니다. 다이틸 파이로카보네이트(DEPC) -처리된 물, 완충제 등을 사용하십시오. 자동 클래브 버퍼와 사용하기 전에 유리 제품을 굽습니다.
1. 조직 고정
- 종 과 조직 유형에 따라 선택의 고정을 준비; 보리 씨앗의 경우 농부의 고정 (75 % 에탄올, 25 % 빙하 아세트산 (v / v)을 사용합니다.
- 조직을 수확하기 전에 얼음에 고정을 식히세요.
- 관심있는 식물 재료를 수집하고 필요한 경우 선택한 포함 금형에 맞게 적절한 크기의 조각으로 해부하십시오. 보리 씨앗의 경우, 고정 용액의 침투를 돕고 포함 금형에 맞게 반 세로로 씨앗을 잘라.
- 조직을 얼음-차가운 고정물의 최소 10배 부피로 잠급합니다. 보리 씨앗의 경우, 씨앗을 반으로 가라앉히면 고정물에 담급됩니다.
- 진공 침투를 사용하여 고정 침투를 가속화하십시오. 조직은 고정의 진공 침투 후 침몰해야합니다. 보리 씨앗의 경우 진공 침투 30분을 사용하십시오.
- 고정 및 인큐베이션을 4°C에서 교체하여 고정제가 조직을 완전히 침투할 수 있도록 합니다. 보리 씨앗의 경우 밤새 샘플을 배양하십시오 (~12-16h).
참고: 희석제 또는 작은 조직은 조직에 고정의 높은 확산 비율로 인해 더 짧은 고정 시간이 필요합니다. - 고정에서 조직을 제거하고 카세트로 조직을 전송한 다음 조직 처리를 시작합니다.
참고: 잎 조직과 같은 작거나 깨지기 쉬운 조직은 고정 중에 손상되지 않도록 고정하기 위해 카세트에 넣을 수 있습니다. 생검 가방, 패드 또는 랩은 조직 고정 및 조직 처리 단계 동안 카세트 내부에 조직을 안전하게 고정하는 데 사용할 수 있습니다.
2. 조직 처리
- 최소 10개의 용액 챔버와 2개의 가열된 파라핀 챔버를 갖춘 2단계에서 자동 티슈 프로세서를 사용합니다(재료표참조).
- 각 챔버에 적절한 양의 솔루션이 있는지 확인하십시오. 티슈 프로세서를 몇 번 사용한 후 솔루션을 교체하십시오.
- 조직으로 카세트를 금속 바구니에 넣습니다. 금속 바구니를 챔버 1 위의 홀더에 부착합니다. 홀더는 지정된 프로그램에 따라 카세트를 챔버로 "덩크앤딥"합니다.
- 각 챔버에 대한 시간 설정을 포함하는 티슈 프로세서의 제어판에서 버튼 클릭을 수행하여 프로그램을 설정합니다.
- "시작"버튼을 눌러 처리 프로그램을 시작합니다. 다음 프로그램은 보리 씨앗을 위해 설계되었으며 하룻밤 동안 실행됩니다 (~18h)
- 에탄올의 그라데이션 계열(75%, 85%, 100%, 100%, 100%, 100% 및 100% (v/v) 에탄올에서 각각 1시간 30분 동안 카세트를 찍어 탈수를 수행한다.
- 에탄올을 사용하여 클리어링을 수행: 자일렌 그라데이션 1 시간 30 분 각각 75:25, 50:50, 25:75 (에탄올: 자일렌 %, v/v). 그런 다음 카세트를 1 시간 30 분 마다 100 % 자일렌 다음 100 % 자일렌을 찍어.
- 용융 파라핀에서 1 시간 30 분 동안 55-60 °C에서 파라핀 침투를 수행하십시오.
참고: 파라핀 히터 챔버의 온도는 티슈 프로세서의 뒷면에 설정할 수 있습니다.
- 다음 날 아침, 티슈 프로세서에서 카세트를 제거하고 파라핀 포함으로 진행합니다.
참고: 프로그램 시간은 다른 조직 유형마다 다를 수 있습니다. 진공 및/또는 동요는 조직 처리 중에 조직 프로세서의 제어판에"V"및/또는"동요"버튼을 눌러 선택한 솔루션의 침투를 가속화하는 데 사용할 수 있습니다.
3. 파라핀 포함
- 이 단계에서 포함 기계를 사용합니다(재료 표참조).
- 포함 기계를 미리 설정하여 저장소의 파라핀이 완전히 녹을 수 있도록 포함되기 최소 몇 시간 전에 켜집니다.
- 시작하기 전에 콜드 플레이트를 켭니다.
- 미세 한 집게를 사용하여 위치에 샘플을 잡고 금형에 용융 파라핀을 분배하여 금형에 샘플을 포함. 각 실험 목적에 대한 적절한 샘플 방향을 보장합니다. 보리 씨앗의 경우 종자 세로를 절단 방향으로 방향을 지정하여 세로 섹션을 얻습니다.
참고: 내장 형은 다른 크기로 제공됩니다. 적절한 크기를 선택하여 샘플을 올바르게 배치하고 임베디드할 수 있도록 합니다. 샘플의 방향은 실험적 요구에 따라 고려해야 합니다. 세로 단면이 필요한 경우, 시료는 절단 방향에 대한 종방향방향을 지향해야 하는 반면, 가로 단면의 경우 시료는 절단 방향과 평행하게 방향을 지정해야 한다. - 깨끗한 카세트를 금형에 놓고 샘플을 카세트에 담을 수 있는 전체 카세트를 완전히 덮을 수 있는 충분한 파라핀을 보장합니다.
- 금형을 콜드 플레이트에 놓고 파라핀이 금형에서 블록을 방출하기 전에 파라핀을 완전히 설정(10-20분)할 수 있도록 합니다.
- 블록을 단면으로 진행하거나 블록을 저장을 위해 블록을 4°C로 전송합니다.
참고: 프로토콜은 여기에서 일시 중지할 수 있습니다. 임베디드 블록은 최대 3개월 동안 4°C로 저장할 수 있습니다.
4. 폴리에틸렌 나프탈레이트(PEN) 멤브레인 슬라이드 준비
- RNase 비활성화 용액에 침수 PEN 멤브레인 슬라이드 3 s 다음 슬라이드에 RNases를 제거하기 위해 DEPC 처리 물에 두 개의 간단한 세차재. 37°C 인큐베이터에서 슬라이드를 건조하여 남은 용액을 제거합니다.
- 30분 동안 라미나르 플로우 캐비닛에 UV 램프를 사용하여 슬라이드를 UV처리하여 파라핀 접착력을 향상시킵니다.
5. 단면
- 단면 단계에서 마이크로토메를 사용합니다(재료 표참조).
- 새 블레이드를 칼 홀더에 넣고, 적극적으로 단면하지 않을 때는 항상 칼 가드를 유지하십시오.
주의: 마이크로톤 블레이드는 매우 날카롭고 부적절하게 처리하면 심각한 피해를 입을 수 있습니다.
참고 : 마이크로 톤에 두 개의 잠금 메커니즘이 있으며, 하나는 기계의 측면에 있고 다른 하나는 바퀴의 손잡이에 있습니다. 둘 다 적극적으로 단면화하지 않을 때 참여해야 합니다. - 만지지 않고 가능한 한 시료에 가깝게 칼 블록을 조정합니다. 마이크로토메 암이 칼 블록과 완전히 접촉하지 않도록 하면 마이크로토메에 치명적인 구조적 손상을 입힙니다.
- 시작하기 전에 콜드 플레이트를 켭니다. 파라핀 블록을 단면화 하기 전에 콜드 플레이트에 보관하고 단면 중에 필요한 경우 블록을 다시 냉각하여 블록이 연화되지 않도록 하십시오.
- 시작 전에 DEPC 처리 된 물과 42 °C로 가열하여 수조를 채웁니다.
- 마이크로토메를 사용하여 원하는 깊이(관심 있는 섹션)와 파라핀 블록(6-10 μm)으로 블록을 트림; 잘 단면된 블록은 블레이드 가장자리에 '리본'을 형성합니다. 보리 씨앗의 경우 두께가 8 μm인 섹션입니다.
- 미세 페인트 브러시 나 미세 한 집게를 사용하여 마이크로 토메에서 수조로 리본을 부드럽게 전달하여 리본이 수면에 평평하도록합니다.
- 슬라이드를 45° 각도로 잡고 위쪽으로 움직이며 리본을 물 밖으로 밀어 내밀어 슬라이드에 조심스럽게 보풀이 없는 조직으로 여분의 물을 제거합니다.
- 37°C에서 30분 동안 건조 슬라이드를 사용하여 파라핀 아래에 남은 물을 제거합니다.
- 파라핀 제거 또는 탈수 조건 하에서 닫힌 상자에 4°C에 저장(며칠 이내에 사용)을 진행합니다.
- 자일렌에서 각각 20s에 3x 슬라이드를 세척하고, 100% (v/v) 에탄올에서 30s의 2x 워시와 70%(v/v) 에탄올로 30s의 2x 워시를 세척하여 파라핀을 제거합니다.
- 파라핀을 제거한 후 즉시 레이저 캡처 마이크로절로 진행합니다.
참고: 저온절면은 LCM과 성공적으로 결합된 대체 방법입니다. 극저온 절위를 위한 견본 준비는 다릅니다.
6. 레이저 캡처 마이크로 절제
- 레이저 포획 미세 절 현미경 (재료의 표참조)을 사용하여 분리 및 건조 된 조직 섹션에서 세포를 미세 하 게 합니다.
- 사용 가능한 세 슬롯에 슬라이드를 로드합니다.
- 수집 튜브의 특수 접착제 캡을 사용하여 캡처된 샘플을 수집합니다. 액체("건조" 수집)없이 캡처하면 RNase 활성이 최소화됩니다. 수집 튜브를 사용 가능한 슬롯에 로드합니다.
- 스테이지를 이동하여 잘라야 하는 샘플 영역을 찾습니다. 이 작업은 LCM 컴퓨터의 마우스 또는 조이스틱 또는 키보드의 화살표 키를 사용하여 수행할 수 있습니다.
- 절삭 속도를 최적화하기 위해, 에너지와 초점을 절단, 레이저 압력 촉매 (LPC) 에너지및 초점, 먼저 멤브레인 슬라이드에 조직이없는 빈 세그먼트에 절단. 보리 시드의 경우, 절단 속도 = 18, CutEnergy = 52 CutFocus = 63, LPCEnergy = 78, LPC 포커스 = 10배율에서 61.
참고: 절단 초점과 에너지를 다른 슬라이드에 대 한 조정 해야, 다른 조직, 그리고 캡처 된 지역 하지만 일반적인 규칙은 발사 전력 절삭 힘 보다 높은 레이저 발사에 대 한 defocus해야. 객관적인 렌즈의 배율이 높을수록 레이저의 초점이 작아지고 에너지가 높아지다. - 드로잉 도구를 사용하여 관심 영역을 요약하여 셀을 선택합니다.
- 함수 툴바에서 투석기 세포에 위의 검은 색 세그먼트에서 절단하여 얻은 최적화된 매개 변수를 기반으로 접착제 캡에 로보LPC 기능을 선택합니다.
참고: LCM 파라미터는 조직 유형뿐만 아니라 단면, 조직 경도 및 객관적인 렌즈의 두께마다 다릅니다. 따라서 실제 시료를 절단하기 전에 조직 시편이 없는 일반 멤브레인 영역에 각 슬라이드를 최적화하는 것이 가장 좋습니다. - 플래그 도구를 사용하여 관심 영역을 표시하여 요소 목록에서 해당 플래그를 선택하여 즉시 찾을 수 있습니다.
- "CapCheck"버튼을 사용하여 접착제 캡을 검사하여 샘플이 캡처되었는지 확인합니다. 전형적으로, 캡당 10-15개의 섹션(~200개의 세포의 LCM)이 RNA 추출을 위해 요구된다.
- 캡처된 샘플을 얼음 위에 보관하십시오. RNA 분해를 피하기 위해 RNA 추출을 위해 즉시 진행하십시오.
참고: 일부 LCM 현미경 검사법은 형광 마커로 표지된 세포의 포획을 허용하는 형광광광을 갖추고 있습니다.
7. RNA 추출
- LCM 후 RNA 추출을 위한 낮은 입력 RNA 절연(재료 표참조) 키트를 사용합니다. 이러한 키트는 10개 미만의 세포로부터 고품질 총 RNA를 일관되게 복구하도록 설계되었습니다.
- 온컬럼 DNase 처리를 포함하는 제조업체의 지침에 따라 캡처된 세포 유형으로부터 총 RNA를 분리한다.
참고: 튜브가 반전되고 플릭되는 RNA 추출의 첫 번째 단계는 뚜껑에 포착된 샘플이 추가된 추출 버퍼와 접촉하도록 하는 데 매우 중요합니다.
8. RNA 증폭
- RNA-seq 라이브러리 합성을 위한 충분한 aRNA를 생성하기 위해 체외 전사에 의해 추출된 RNA로부터 aRNA 증폭을 위한 안티센스 RNA(aRNA) 증폭 키트(재료표 참조)를 사용한다.
- 제조업체의 지침에 따라 aRNA 증폭 키트를 사용하여 2회 증폭을 수행합니다.
참고: 키트가 지시한 온도에 열 순환기와 뚜껑을 예열하는 것이 중요합니다(재료 표참조) 제조업체. RNA 증폭 대신에 다른 접근법은 추출된 RNA로부터 직접 라이브러리를 합성하기 위해 낮은 입력 라이브러리 준비 키트를 사용하는 것이다.
9. RNA 정량화
- 자동화된 전기 전광 시스템을 사용하여 aRNA를 정량화하고 자격을 갖추게 됩니다(재료 표참조).
참고: 자동화된 전기포고시스템은 샘플(1-2 μL)이 적고 각 개별 샘플에 대해 젤과 같은 이미지와 전기페로그램을 제공하기 때문에 선호됩니다.
10. qRT-PCR 및/또는 RNA-seq
- qRT-PCR을 위한 aRNA로부터 cDNA를 합성하거나 표준 RNA-seq 라이브러리 키트를 사용하여 RNA-seq 라이브러리를 만듭니다.
결과
우리는 우리의 LCM RNA-seq 프로토콜10을사용하여 발아 하는 동안 보리 씨앗에서 공간 및 측두 조직 특이적인 전사를 생성 했다. 이 연구는 발아 중 48시간 코스(0-48h, 7시간 포인트)를 통해 3개의 배아 기관(plumule, radicle tip, scutellum)에서 소수의 세포에 LCM RNA-seq를 적용하여 수행하였다(도2A,B).

그림 2: LCM에 의해 보리 씨앗에서 수집된 세포. (A)보리 씨앗의 세로 단면및, 인셋, 세포가 포획된 영역을 나타내는 확대된 만화. 블루 = 플럼, 마젠타 = 래디클, 그린 = 스커텔럼. (B)LCM의 플럼, 래디클 및 스큐렐럼. 상단 패널, 레이저 압력 발사 전에 조직 (LPC). 하단 패널, LPC 후 조직. 세포는 샘플 당 3개의 생물학적 복제를 가진 각 견본의 5개의 연속적인 세로 단면도에서 포착되었습니다. 각 복제는 약 200개의 셀(~0.05 - 0.15 mm2 총 면적)으로 구성되었다. Bar = 100 μm. E = 내정자, C = 분쇄 된 세포 층, SE = scutellar 상피, S = scutellum. 권한10을사용하여 식물 저널의 이전 데이터에서 재현된 그림입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
고정 및 포함은 제대로 고정된 조직 샘플이 조직 손상및 RNA 양 및 품질의 손실을 초래할 수 있기 때문에 중요한 단계입니다. 이러한 단계는 다른 종 및 조직 유형19,,20,21에대해 최적화되고 조정되어야 한다., 여기서 우리는 종자 조직 및 세포로 고정제의 침투를 향상시키기 위하여 진공 침투를 이용했습니다. 탈수및 파라핀 침윤은 조직 샘플의 손상을 피하기 위해 점차적으로 수행됩니다. 우리는 각 단계에서 적용 된 교반 및 진공이있는 자동화 된 조직 프로세서를 사용하여 전체 탈수 및 파라핀 침투 과정을 가속화했습니다. 이것은 인간 적인 수술에 의해 수행된 장기간조직 처리 단계 도중 생기는 RNA 분해의 리스크를 높게 감소시킵니다. 단면화하기 전에 파라핀 접착력을 보장하고 RNase가 없는지 확인하기 위해 멤브레인 슬라이드를 치료하는 것이 중요합니다. 멤브레인 슬라이드가 제대로 준비되지 않으면 샘플 섹션이 멤브레인에 부착되지 않으므로 LCM 발사 단계에 영향을 미치고 샘플이 멤브레인에서 분리되기 때문에 접착제 캡으로의 조직 전달을 감소시킵니다(그림 1참조).
LCM RNA-seq 프로토콜의 또 다른 중요한 단계는 LCM 파라미터 최적화입니다. 최적화할 다섯 가지 주요 매개 변수가 있습니다: (1) 절삭 속도; (2) 절삭 에너지; (3) 절단 초점; (4) LPC 에너지; 및 (5) LPC 초점. 선택된 부위를 정밀하게 절단하고 선택된 부위의 가장자리를 태우지 않고 주변 조직에서 벗어나기 위해 절단 파라미터를 올바르게 조정하는 것이중요하다(도 3A,B). 적절한 LPC 에너지와 초점은 슬라이드에서 수집 튜브의 특수 접착제 캡에 선택한 영역을 안정적으로 투석하는 데 필수적이며, 항상 캡에 포착된 샘플을 검사하여 충분한 재료가 수집되도록합니다(그림 3C). LCM의 효율은 1시간 이내에 1000개의 세포를 포획할 수 있게 해주며, 이는 시료 처리 중 RNA 분해 가능성을 크게 줄입니다. 접착제 캡이 달린 수집 튜브를 사용하면 액체를 캡처하지 않고도 "건조" 컬렉션을 사용할 수 있으므로 가능한 RNases 활동을 크게 줄일 수 있습니다. 모든 복제를 수집한 직후에 포획된 샘플을 얼음에 보관하고 RNA 추출을 수행하는 것이 중요합니다.
RNA 추출 및 증폭은 제조업체의 지시에 따라 시판되는 키트를 사용하여 수행됩니다. 자동화된 전기전광 시스템을 이용한 RNA 증폭 전에 총 RNA의 정량화는 양질의 RNA 샘플에서 18S 및 28S 리보소말 하위 단위의 뚜렷한 전기전전기 대역 및 형광 피크를 검출할것이다(도 3D). 엽록과 미토콘드리아 리보소말 RNA에 대응하는 추가 피크는 18S 및 28S 피크 전에 발견될 것이다. 식물 종에서 여러 RNA 피크로 인해 RIN (RNA 무결성 번호) 값은 전형적으로 9 (포유류 조직에서 예상되는 RIN 값)보다 낮으며 RNA무결성(22)을정확하게 반영하지 않는다. 명확한 18S 및 28S 피크가 RNA 분해의 표시 없이 볼 때 RNA 증폭을 계속하는 것이 적절하다. 우리의 보리 종자 실험에서 회수된 RNA의 양은 3개의 다른 조직 모형 (깃털, 방사형 및 scutellum) 사이, 또한 수확되는 세포의 대략 동등한 수에도 불구하고, 발아의 초기 그리고 늦은 단계 사이에서 변화했습니다. 보리 씨앗의 양은 대략 200 세포 (세포 당 0.5 ~ 100 pg)에서 100 pg에서 20 ng까지 다양했습니다. RNA 증폭에 권장되는 최소 입력 RNA의 양은 100 PG였다. 추출된 RNA의 양은 개발19,,21의그 단계에서 상이한 수확된 세포로부터의 추출 효율과 특정 세포 형의 총 성적증명서 풍부에 따라 달라집니다. LCM RNA-seq 실험을 계획할 때 이 점을 고려하는 것이 유용하지만, 이러한 이전 정보는 모든 경우에 사용할 수 없습니다.
성공적으로 합성된 aRNA는 2차례의 증폭 후 약 300개의 뉴클레오티드를 가진 100~1000개의 뉴클레오티드에서 100~1000개의 뉴클레오티드의 유니모달, 대칭 크기 분포를 나타낸다(도 3E). 우리의 보리 종자 실험에서, 우리는 RNA 증폭 10의 2 라운드 후에 aRNA의500pg 에서 2 μg를 얻었다. 증폭 및 증폭되지 않은 RNA 사이의 비교는 RNA 증폭이 재현 가능하고 선형적이며 성적증명서데이터(23,24,25)에체계적인 바이어스를 도입하지 않는다는 것을 입증했다.,, 출력 aRNA는 공간 및 측두조직 특이적 유전자 발현 분석을 위해 qRT-PCR 및/또는 RNA-seq를 위해 사용될 수 있다. RNA-seq 후 유효성 검사는 aRNA의 분석에서 결과를 확증하기 위해 수행되어야 합니다. 이는 시투 혼성화에서 RNA를 이용하여 RNA-seq 데이터로부터 세포형 마커 유전자의 발현을 검사함으로써 수행될 수 있다.
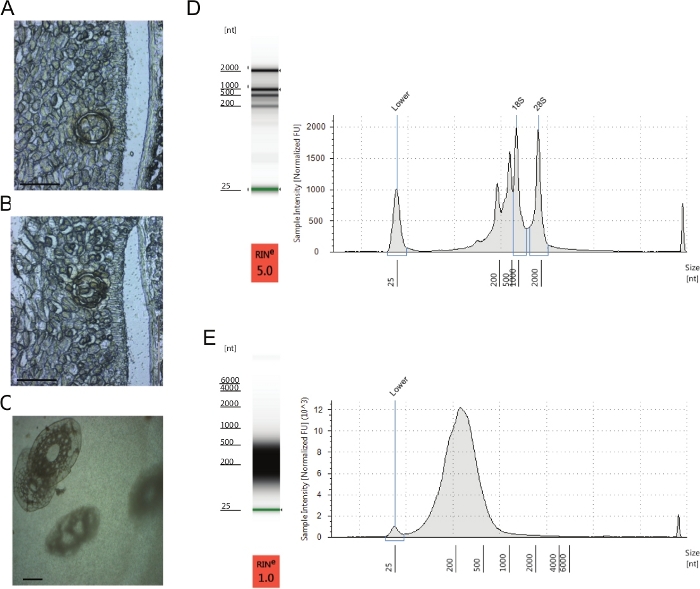
그림 3: LCM의 대표적인 결과. (A)적절한 절삭 에너지와 초점으로 절단 후 조직. 컷이 만들어진 위치를 명확하게 알 수 있습니다. 선택한 영역은 주변 재료에서 제거됩니다. 바 = 100 μm.(B)잘못된 절단 에너지와 초점절단 후 조직. 선택한 영역은 여전히 주변 재료와 연결되어 있습니다. Bar = 100 μm.(C)수집 튜브의 특수 접착제 캡에서 캡처 된 세포를 검사합니다. Bar = 100 μm.(D)RNA 증폭 전 총 RNA 프로파일의 예(왼쪽, 젤형 이미지; 오른쪽, 전기페로그램) 뚜렷한 전기전구대 및 18S 및 28S 리보소말 서브유닛의 형광 피크. 라벨이 부착되지 않은 피크는 엽록소 및 미토콘드리아 리보좀을 포함한 추가 리보솜 RNA에 해당합니다. (E)RNA 증폭 후 전형적인 aRNA 프로파일의 예(왼쪽, 젤 형 이미지; 오른쪽, 전자 페로그램). 100에서 1000개의 뉴클레오티드까지의 크기의 분포가 약 300개의 뉴클레오티드로 검출되었다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
RNA-seq 분석은 생물학적 삼중생물의 모든 샘플에서 수행되었다. 발아의 48h 이상 상이한 조직에서 발현된 유전자의 다차원 스케일링(MDS) 플롯은 동일한 시점부터 샘플 사이와 단일 조직의 샘플 간의 유사성을 더 큰 유사성을 보여 주지만 다른조직(도 4A). 우리는 발아 도중 시간이 지남에 따라 개별 조직에서 얼마나 광범위하게 유전자 발현이 분별하게 통제되었는지 결정하기 위하여 착수했습니다. 분화 유전자(DEGs)의 수는 각 조직에서 발아하는 과정을 통해 점진적으로 증가하여, 그 조직의 0h타임포인트(도 4B)에비해 증가하였다. 25%(910) 페뮬레, 34%(1876) 및 스큐렐럼 DEGs의 41%(2562)는 그 조직에서 단독으로 분화되는 것으로 나타났다(즉, 이들 유전자는 다른 조직, 도 4C)에서차별화적으로 발현되지 않은 것으로 나타났다. 종합하면, 이러한 결과는 LCM RNA-seq가 식물 발달 도중 시간적 및 공간 적인 유전자 발현을 이해하는 데 어떻게 사용될 수 있는지 를 보여줍니다.

그림 4: 보리 발아의 48 h를 통해 다른 조직의 뚜렷한 분리를 보여주는 LCM 견본의 RNA 라이브러리의 분석. (A)발아의 48h 이상의 상이한 조직에서 발현된 유전자의 다차원 스케일링(MDS) 플롯. 각 점은 1개의 샘플을 나타내며, 2점 사이의 거리는 해당 RNA 샘플의 선행 로그 폴드 체인지(FC)를 반영한다. x, y 및 z 축은 첫 번째, 두 번째 및 세 번째 차원 로그FC를 나타냅니다. P = 플럼, R = 래디클, S = 스커텔럼, 0-48h = 발아의 h, R1-3 = 복제 1-3. (B)3가지 조직 유형에 대해 시간점 0h에 비해 6시간 포인트에 대해 상당한 상승 조절(red) 및 하향 조절(blue) 분별발유전자(DEGs)의 수. (C)3가지 조직 유형에서 DEGs 수를 나타내는 벤 다이어그램. 중첩 세트의 유전자는 2개 또는 3개의 조직 모형에 있는 차등 발현을 보여줍니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
토론
많은 조직 별 유전자 발현 연구는 시간이 많이 소요되는, 노동 집약적인 시료의 손 해부에 의해 제한되었으며, 오염위험이 높으며 인간 수술이 수확하기에 충분히 손재주적이라는 시료만 활용할 수 있습니다. LCM은 현미경 시각화 하에서 기계적으로 작동하는 레이저 빔을 사용하여 고정 조직 섹션에서 세포를 수집하는 정확하고 접촉없는 기술입니다.
좋은 샘플 준비는 LCM에 매우 중요합니다. 이 과정은 조직 형태와 RNA의 무결성 사이의 균형을 유지하기 위해 시료의 적절한 고정 및 삽입에 의존합니다. 여기에 제시된 프로토콜은 고정 작업의 긴 고정 시간과 진공 침투를 포함하는 보리 씨앗에 대한 최적화된 고정 및 포함 단계를 제공합니다. 다른 조직 또는 종의 분석은 조건의 최적화를 요구할 것입니다, 이들은 전형적으로 조직 사이 다릅니다. 이 프로토콜의 또 다른 중요한 양은 RNA-seq 라이브러리 건설을 위해 충분한 양의 RNA가 생성되도록 보장하는 RNA 증폭 단계입니다. 우리는 RNA 증폭이 고품질 라이브러리에서 초래하지만, 대안적으로 낮은 입력 라이브러리 준비 방법이25를고용 할 수 있음을 발견한다. 표적 세포의 조직학적 식별은 우리의 방법의 중요한 단계이며 희귀하거나 특성이 낮은 세포 유형에 대한 문제를 제공 할 수 있습니다. 표적 세포 인식은 기존의 조직학적 얼룩 또는 면역형광 얼룩을 전처리 단계의 일부로 사용하거나, 대안적으로, 형광으로 표지된 세포 특이적마커(26,,27)를발현하는 형질대사를 사용하여 개선될 수 있다.
LCM 기술은 세포 형 특이적 성적증명서 분석1을위해 동물 유래 샘플에 먼저 사용되기 위해 개발되었다. 그러나, 게놈, 프로테오믹 및 후성 유전학 분석의 넓은 범위는 이후에 개발되었습니다. 식물 생물학적 연구에 적용을 위한 이러한 방법을 적응하는 것은 지속적인 도전18,,19,,20,,28. Schad 외. 애교성 혈관 번들에서 단백질과 대사산물은 2-D 젤 전기 포고및 질량 분광법29,,30LCM를사용하여 분석될 수 있음을 입증했다. 최근에는 LCM을 이용한 LCM 해부 된 석상 및 카펠에 대한 크로마틴 면역 침전(ChIP)-qPCR 분석을 수행하여 조직 균질성이 세포 별 후생유전학 적 사건의 해결을 향상시킨다는 것을 확립하였다31. Turco 외. 또한 혈관과 비혈관 조직 사이의 조직 별 차이를 강조하는, 사탕수수에서 혈관화 동안 DNA 메틸화 이벤트를 연구하기 위해 쌍황피티 시퀀싱LCM을 결합했다32. 흥미롭게도, 몇몇 연구 결과는 식물 세균 및 식물 병원체 상호 작용33,,34,35를 탐구하기 위하여LCM을적용했습니다. LCM이 다른 감염 단계에서 세포를 시각화하고 포획하는 능력은 감염된 세포의 공간 및 측두화 반응에 대한 정보를 제공했습니다.
여기에 제시된 RNA-seq와 결합된 우리의 LCM 프로토콜은 별개의 세포 모형의 유전자 발현의 spatio-측두전 글로벌 프로파일링을 용이하게 할 것입니다. LCM은 크롬라틴 및 단백질에 대한 향상된 정제 방법과 향상된 질량 분광계 기구를 통해 식물의 세포 유형 별 에피소드 및 프로테오믹 연구를 용이하게 하는 새로운 도구가 될 것입니다.
공개
저자는 공개 할 것이 없습니다.
감사의 말
이 작품은 식물 에너지 생물학 (CE140100008)에서 JW에 우수의 호주 연구 위원회 센터에 의해 지원되었다. M.G.L은 라 트로브 대학 시작 보조금에 의해 지원되었다. 우리는 높은 처리량 시퀀싱 및 데이터 분석에 대한 지원에 대한 La Trobe 유전체학 플랫폼에 감사드립니다. 우리는 우리의 실험실에서 LCM을 설립에 대한 전문가의 조언을 준 교수 매튜 터커 에게 감사드립니다.
자료
| Name | Company | Catalog Number | Comments |
| Acetic acid 100 % ACS/R. | AnalaR NORMAPUR (BioStrategies) | VWRC20104.323 | |
| AdhesiveCap 200 opaque | Zeiss | 415190-9181-000 | |
| Clear base moulds 8 X 10 | Leica | 3803015 | |
| Diethyl pyrocarbonate | Sigma-Aldrich | 40718-25ML | |
| High Sensitivity RNA ScreenTape | Agilent | 5067-5579 | |
| Lowprofile disp.blades DB80LS | Leica | 14035843489 | |
| MembraneSlide 1.0 PEN | Zeiss | 415190-9041-000 | |
| MessageAmp II aRNA Amplification Kit | Ambion (ThermoFisher) | AMB17515 | |
| On-Column DNase I Digestion Set | Sigma-Aldrich | DNASE70 | |
| Ovation RNA-Seq System V2 | NuGen (Integrated Science) | 7102-08 | |
| Paraffin (Surgipath Paraplast) | Leica | 39601006 | |
| PicoPure RNA Isolation Kit | ABI (ThermoFisher) | KIT0214 | |
| RNaseZap RNase Decontamination Solution | Ambion (ThermoFisher) | AM9780 | |
| Xylene | AnalaR NORMAPUR (BioStrategies) | VWRC28975.360 | |
| Leica Benchtop Tissue Processor | Leica Biosystems | TP1020 | |
| Leica Heated Paraffin Embedding Module | Leica Biosystems | EG1150H | |
| Leica Cold Plate | Leica Biosystems | EG1150C | |
| Safemate Class 2 Biological Safety Cabinets | LAF Technologies Pty Ltd | Safemate 1.5 | |
| Leica Fully Automated Rotary Microtome | Leica Biosystems | RM2265 | with PALMRobo v 4.6 software |
| Zeiss PALM MicroBeam LCM system | Zeiss miscroscopy | ||
| TapeStation | Agilent | TapeStation 2200 |
참고문헌
- Emmert-Buck, M. R., et al. Laser capture microdissection. Science. 274 (5289), 998-1001 (1996).
- Alevizos, I., et al. Oral cancer in vivo gene expression profiling assisted by laser capture microdissection and microarray analysis. Oncogene. 20 (43), 6196-6204 (2001).
- Cong, P., et al. In situ localization of follicular lymphoma: description and analysis by laser capture microdissection. Blood, The Journal of the American Society of Hematology. 99 (9), 3376-3382 (2002).
- Blokhina, O., et al. Laser capture microdissection protocol for xylem tissues of woody plants. Frontiers in Plant Science. 7, 1965(2017).
- Casson, S., Spencer, M., Walker, K., Lindsey, K. Laser capture microdissection for the analysis of gene expression during embryogenesis of Arabidopsis. The Plant Journal. 42 (1), 111-123 (2005).
- Chen, Z., et al. LCM-seq reveals the crucial role of LsSOC1 in heat-promoted bolting of lettuce (Lactuca sativa L.). The Plant Journal. 95 (3), 516-528 (2018).
- Jiao, Y., et al. A transcriptome atlas of rice cell types uncovers cellular, functional and developmental hierarchies. Nature Genetics. 41 (2), 258-263 (2009).
- Kivivirta, K., et al. A protocol for laser microdissection (LMD) followed by transcriptome analysis of plant reproductive tissue in phylogenetically distant angiosperms. Plant Methods. 15 (1), 1-11 (2019).
- Li, P., et al. The developmental dynamics of the maize leaf transcriptome. Nature Genetics. 42 (12), 1060-1067 (2010).
- Liew, L. C., et al. Temporal tissue-specific regulation of transcriptomes during barley (Hordeum vulgare) seed germination. The Plant Journal. 101 (3), 700-715 (2020).
- Matas, A. J., et al. Tissue-and cell-type specific transcriptome profiling of expanding tomato fruit provides insights into metabolic and regulatory specialization and cuticle formation. The Plant Cell. 23 (11), 3893-3910 (2011).
- Sakai, K., et al. Combining laser-assisted microdissection (LAM) and RNA-seq allows to perform a comprehensive transcriptomic analysis of epidermal cells of Arabidopsis embryo. Plant Methods. 14 (1), 10(2018).
- Zhan, J., et al. RNA Sequencing of Laser-Capture Microdissected compartments of the maize kernel identifies regulatory modules associated with endosperm cell differentiation. The Plant Cell. 27 (3), 513-531 (2015).
- Hwang, B., Lee, J. H., Bang, D. Single-cell RNA sequencing technologies and bioinformatics pipelines. Experimental & Molecular Medicine. 50 (8), 1-14 (2018).
- Zeb, Q., Wang, C., Shafiq, S., Liu, L. Single-Cell Omics. , Elsevier. 101-135 (2019).
- Deal, R. B., Henikoff, S. The INTACT method for cell type-specific gene expression and chromatin profiling in Arabidopsis thaliana. Nature Protocols. 6 (1), 56(2011).
- Heiman, M., Kulicke, R., Fenster, R. J., Greengard, P., Heintz, N. Cell type-specific mRNA purification by translating ribosome affinity purification (TRAP). Nature Protocols. 9 (6), 1282(2014).
- Bevilacqua, C., Ducos, B. Laser microdissection: A powerful tool for genomics at cell level. Molecular Aspects of Medicine. 59, 5-27 (2018).
- Nelson, T., Tausta, S. L., Gandotra, N., Liu, T. Laser microdissection of plant tissue: what you see is what you get. Annual Reviews in Plant Biology. 57, 181-201 (2006).
- Day, R. C., Grossniklaus, U., Macknight, R. C. Be more specific! Laser-assisted microdissection of plant cells. Trends in Plant Science. 10 (8), 397-406 (2005).
- Takahashi, H., et al. A method for obtaining high quality RNA from paraffin sections of plant tissues by laser microdissection. Journal of Plant Research. 123 (6), 807-813 (2010).
- Schroeder, A., et al. The RIN: an RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7 (1), 3(2006).
- Ferreira, E. N., et al. Linear mRNA amplification approach for RNAseq from limited amount of RNA. Gene. 564 (2), 220-227 (2015).
- Schneider, J., et al. Systematic analysis of T7 RNA polymerase based in vitro linear RNA amplification for use in microarray experiments. BMC Genomics. 5 (1), 29(2004).
- Shanker, S., et al. Evaluation of commercially available RNA amplification kits for RNA sequencing using very low input amounts of total RNA. Journal of Biomolecular Techniques. 26 (1), 4(2015).
- Bhattacherjee, V., et al. Laser capture microdissection of fluorescently labeled embryonic cranial neural crest cells. Genesis. 39 (1), 58-64 (2004).
- Clément-Ziza, M., Munnich, A., Lyonnet, S., Jaubert, F., Besmond, C. Stabilization of RNA during laser capture microdissection by performing experiments under argon atmosphere or using ethanol as a solvent in staining solutions. RNA. 14 (12), 2698-2704 (2008).
- Blokhina, O., et al. Parenchymal Cells Contribute to Lignification of Tracheids in Developing Xylem of Norway Spruce. Plant Physiology. 181 (4), 1552-1572 (2019).
- Schad, M., Lipton, M. S., Giavalisco, P., Smith, R. D., Kehr, J. Evaluation of two-dimensional electrophoresis and liquid chromatography-tandem mass spectrometry for tissue-specific protein profiling of laser-microdissected plant samples. Electrophoresis. 26 (14), 2729-2738 (2005).
- Schad, M., Mungur, R., Fiehn, O., Kehr, J. Metabolic profiling of laser microdissected vascular bundles of Arabidopsis thaliana. Plant Methods. 1 (1), 2(2005).
- Latrasse, D., et al. The quest for epigenetic regulation underlying unisexual flower development in Cucumis melo. Epigenetics & Chromatin. 10 (1), 22(2017).
- Turco, G. M., et al. DNA methylation and gene expression regulation associated with vascularization in Sorghum bicolor. The New Phytologist. 214 (3), 1213-1229 (2017).
- Gomez, S. K., Harrison, M. J. Laser microdissection and its application to analyze gene expression in arbuscular mycorrhizal symbiosis. Pest Management Science: Formerly Pesticide Science. 65 (5), 504-511 (2009).
- Roux, B., et al. An integrated analysis of plant and bacterial gene expression in symbiotic root nodules using laser-capture microdissection coupled to RNA sequencing. The Plant Journal. 77 (6), 817-837 (2014).
- Tang, W., Coughlan, S., Crane, E., Beatty, M., Duvick, J. The application of laser microdissection to in planta gene expression profiling of the maize anthracnose stalk rot fungus Colletotrichum graminicola. Molecular Plant-Microbe Interactions. 19 (11), 1240-1250 (2006).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유