Method Article
Analyse de l’ARN de microdissection à capture laser pour analyse d’expression génique spécifique aux tissus spatiaux et temporels chez les plantes
Dans cet article
Résumé
Présenté ici est un protocole pour la microdésection laser-capture (LCM) des tissus végétaux. LCM est une technique microscopique pour isoler les zones de tissu d’une manière sans contamination. La procédure comprend la fixation des tissus, l’incorporation de paraffine, la sectionnement, l’extraction de LCM et d’ARN. L’ARN est utilisé dans l’analyse spécifique aux tissus en aval et résolue temporellement des transcriptomes.
Résumé
Le développement d’un organisme multicellulaire complexe est régi par des types de cellules distincts qui ont des profils transcriptionnels différents. Pour identifier les réseaux de régulation transcriptionnels qui régissent les processus de développement, il est nécessaire de mesurer les profils d’expression des gènes spatiaux et temporels de ces différents types de cellules. Par conséquent, il est essentiel de mieux comprendre le contrôle spatio-temporel de l’expression génique pour mieux comprendre comment les processus biologiques et développementaux sont réglementés. Ici, nous décrivons une méthode de microdissection de capture laser (LCM) pour isoler un petit nombre de cellules de trois organes d’embryon d’orge au cours d’un cours de temps pendant la germination suivie du profilage de transcription. La méthode consiste en la fixation des tissus, le traitement des tissus, l’incorporation de paraffine, la sectionnement, l’extraction de LCM et d’ARN suivie d’une PCR ou d’un RNA-seq en temps réel. Cette méthode nous a permis d’obtenir des profils spatiaux et temporels des transcriptomes d’organes de semence à partir de différents nombres de cellules (des dizaines à des centaines), fournissant une spécificité tissulaire beaucoup plus grande que les analyses typiques des tissus en vrac. À partir de ces données, nous avons pu définir et comparer les réseaux de réglementation transcriptionnels ainsi que prévoir les facteurs de transcription réglementaires des candidats pour les tissus individuels. La méthode doit s’appliquer à d’autres tissus végétaux avec un minimum d’optimisation.
Introduction
Le développement et la croissance des plantes impliquent l’action coordonnée des réseaux de régulation transcriptionnels au sein de différentes cellules qui existent dans un environnement cellulaire complexe. Pour comprendre l’activité de ces réseaux de réglementation, nous avons besoin de la connaissance de l’expression des gènes spatiaux et temporels dans différents types de cellules à travers les stades de développement. Cependant, les analyses de l’expression des gènes sont plus couramment effectuées dans des organes entiers ou des échantillons de tissus en vrac en raison du défi technique d’isoler et d’analyser un petit nombre de cellules. La méthode que nous décrivons ici a permis d’obtenir l’analyse spatiale et temporelle de transcritome tissulaire-spécifique en couplant LCM avec l’ARN-seq.
LCM a été développé il y a deux décennies par Emmert-Buck et ses collègues1. La technique a permis aux chercheurs d’isoler précisément les cellules individuelles ou les grappes de cellules de leur environnement en utilisant une visualisation microscopique directe et la manipulation avec un faisceau étroit laser1. Depuis lors, la méthode a été largement utilisée dans la biologie du cancer et la pathologie2,3. De nombreux groupes de recherche végétale ont également adapté LCM pour l’utilisation avec différentes espèces végétales et différents types de tissus4,5,6,7,8,9,10,11. Récemment, plusieurs articles ont également utilisé LCM sur les graines eudicot et monocoques pour étudier l’embryon, les endospermes et d’autres structures de semences pendant le développement des semences et la germination10,12,13. La plupart des autres méthodes d’isolement unicellulaire couramment utilisées telles que le micro-pipetage, le tri cellulaire, la séparation magnétique et les plates-formes microfluidiques dépendent de la digestion enzymatique ou de l’homogénéisation mécanique pour dissocier les cellules. Cela peut perturber l’expression des gènes, en introduisant des artefacts techniques qui confondent l’interprétation des données14,15. Ces méthodes exigent également une connaissance préalable des gènes marqueurs pour chaque type de cellule pour relier les cellules dissociées à leur emplacement spatial et au vrai type de cellule. Un autre groupe de techniques dépend de l’isolement basé sur l’affinité des structures subcellulaires au lieu de cellules entières, par exemple INTACT (Isolation des noyaux marqués dans les types de cellules) et TRAP (Transducing Ribosome Affinity Purification)16,17. Cependant, l’étiquetage et la purification des noyaux ou des ribosomes sont techniquement difficiles chez les espèces végétales qui n’ont pas de protocoles de transformation bien établis. LCM profite de la fixation rapide des tissus pour préserver les niveaux de transcription et l’identification histologique conventionnelle par visualisation directe des cellules dans leur contexte normal de tissu/organe, ce qui permet aux cellules discrètes d’être isolées dans un court laps de temps18,19.
Le protocole présenté ici est une méthode optimisée pour l’isolement de cellules spécifiques ou de types de cellules des sections tissulaires des graines de céréales, qui peuvent être appliquées à la plupart des cellules qui peuvent être identifiées histologiquement. LCM fournit une méthode sans contact d’isolement cellulaire, réduisant considérablement la contamination et augmentant l’intégrité de l’ARN récupéré. En outre, la méthode illustre la puissance du LCM sur les études à grande échelle du génome à partir de petites quantités de matériaux biologiques. Nous décrivons également l’amplification linéaire de l’ARN pour produire suffisamment de matériel d’entrée pour les analyses de transcription/transcription.
Il y a dix étapes principales dans ce protocole d’ARN-seq de LCM pour les transcriptomes spatiaux et temporels spécifiques aux tissus, y compris la fixation des échantillons de tissu, la déshydratation, l’infiltration de paraffine, l’incorporation, la section, le LCM, l’extraction d’ARN, l’amplification d’ARN, la quantification de l’ARN et le qRT-PCR et/ou l’ARN-seq (Figure 1).
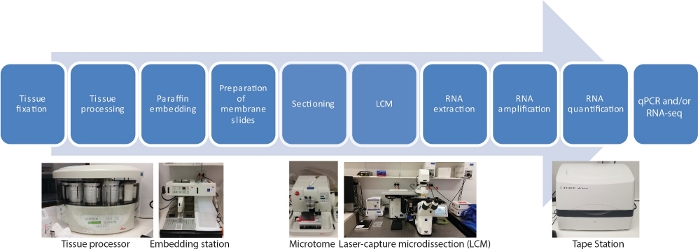
Figure 1 : Organigramme de LCM suivi de l’ARN-seq ou du qRT-PCR. LCM est une technique spatialement précise et sans contact pour recueillir les cellules à partir de sections de tissus fixes à l’aide d’un faisceau laser sous visualisation microscopique. Le processus commence par la fixation d’échantillons de tissus, suivi d’une déshydratation à l’aide d’une série de gradients d’éthanol et de xylène, et s’est terminé par une infiltration de paraffine. Le processus peut être entièrement automatisé à l’aide d’un processeur de tissus. Une fois que le tissu est infiltré avec de la paraffine, il est incorporé dans un moule avec de la paraffine en fusion à l’aide d’une station d’intégration. La section est effectuée à l’aide de microtomes réglés à l’épaisseur désirée. Les diapositives sont préparées et LCM effectuée immédiatement avant que l’ARN ne soit extrait des cellules capturées. L’extraction de l’ARN est suivie directement de deux cycles d’amplification de l’ARN avant qRT-PCR et/ou ARN-seq. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
Protocole
Comme le produit final est l’ARN, prenez soin d’éviter de contaminer le travail avec RNases. Porter des gants est un must. Utiliser le pyrocarbonate de diéthyle (DEPC) -eau traitée, tampons, etc. Tampons autoclave et cuire la verrerie avant utilisation.
1. Fixation des tissus
- Préparer le fixatif de choix en fonction des espèces et des types de tissus; pour les semences d’orge, utiliser le fixatif de Farmer (75 % d’éthanol, 25 % d’acide acétique glaciaire (v/v)).
- Refroidir le fixatif sur la glace avant la récolte des tissus.
- Recueillir le matériel végétal d’intérêt et, si nécessaire, le disséquer en morceaux de taille appropriée pour s’insérer dans le moule d’incorporation sélectionné. Pour les graines d’orge, couper la graine en deux longitudinalement pour aider à la pénétration de la solution fixative et pour s’insérer dans le moule d’incorporation.
- Submerger le tissu dans au moins 10x de volume de fixatif de glace. Pour les graines d’orge, submerger les graines coupées en deux dans le fixatif.
- Utilisez l’infiltration sous vide pour accélérer la pénétration du fixatif. Les tissus doivent couler après l’infiltration sous vide du fixatif. Pour les graines d’orge, utiliser 30 min d’infiltration sous vide.
- Remplacer le fixatif et l’incuber à 4 °C pour permettre au fixatif de pénétrer complètement le tissu. Pour les graines d’orge, incuber les échantillons pendant la nuit (~12-16 h).
REMARQUE : Les tissus plus minces ou petits exigeront un temps de fixation plus court en raison du taux de diffusion plus élevé du fixatif dans le tissu. - Retirer le tissu du fixatif et transférer le tissu dans les cassettes, puis commencer le traitement des tissus.
REMARQUE : Les tissus petits ou fragiles, tels que les tissus foliaires, peuvent être placés dans des cassettes pour les fixer afin de s’assurer qu’ils ne sont pas endommagés pendant la fixation. Des sacs de biopsie, des tampons ou des enveloppements pourraient être utilisés pour maintenir le tissu solidement à l’intérieur des cassettes pendant les étapes de fixation des tissus et de traitement des tissus.
2. Traitement des tissus
- Utiliser un processeur de tissu automatisé à l’étape 2 avec un minimum de 10 chambres de solution et 2 chambres de paraffine chauffées (voir tableau des matériaux).
- Vérifiez qu’il existe des quantités suffisantes de solutions dans chaque chambre; remplacer les solutions après quelques utilisations du processeur tissulaire.
- Placer les cassettes avec du tissu dans le panier métallique. Attachez le panier en métal à son support au-dessus de la chambre 1. Le support tournera et « dunk and dip » les cassettes dans les chambres, suivant le programme désigné.
- Définissez le programme en effectuant des clics de bouton sur les panneaux de commande du processeur de tissu qui impliquent la durée de réglage de chaque chambre.
- Appuyez sur le bouton «Démarrer» pour démarrer le programme de traitement. Le programme suivant est conçu pour les graines d’orge et fonctionne toute la nuit (~18 h)
- Effectuer la déshydratation en trempant la cassette pendant 1 h 30 min chacun dans la série de gradient d’éthanol (75%, 85%, 100%, 100%, et 100% (v/v) éthanol).
- Effectuer le déblaiement à l’éthanol : gradient de xylène pendant 1 h 30 min chacun à 75:25, 50:50, 25:75 (éthanol : xylène %, v/v). Puis tremper la cassette pendant 1 h 30 min chacun de 100% xylène puis 100% xylène.
- Effectuer l’infiltration de paraffine à 55-60 °C pendant 1 h 30 min deux fois dans la paraffine fondue.
REMARQUE : La température des chambres chauffantes de paraffine peut être réglée à l’arrière du processeur de tissu.
- Le lendemain matin, retirez les cassettes du processeur de tissu et passez à l’incorporation de paraffine.
REMARQUE : Le temps de programme peut varier d’un type de tissu à l’autre. Le vide et/ou l’agitation peuvent être utilisés pendant le traitement des tissus pour accélérer l’infiltration de solutions sélectionnées en appuyant sur les boutons «V» et/ou «agitation» sur le panneau de commande du processeur tissulaire.
3. Incorporation de paraffine
- Utilisez une machine d’intégration à cette étape (voir Tableau des matériaux).
- Prédéfinir la machine d’incorporation pour allumer au moins quelques heures avant l’intégration afin de laisser le temps à la paraffine dans les réservoirs de fondre complètement.
- Allumez la plaque froide avant de commencer.
- Incorporer des échantillons dans des moules en maintenant les échantillons en position à l’aide de forceps fins et en distribuant de la paraffine fondue dans le moule. Assurer une bonne orientation des échantillons à chaque fin expérimentale. Pour les semences d’orge, orienter la graine longitudinale vers la direction de coupe pour obtenir des sections longitudinales.
REMARQUE : Les moules d’incorporation sont disponibles dans différentes tailles. Sélectionnez une taille appropriée pour permettre à l’échantillon d’être positionné et intégré correctement. L’orientation des échantillons doit être prise en compte en fonction des besoins expérimentaux. Si des sections longitudinales sont nécessaires, l’échantillon doit être orienté longitudinale vers la direction de coupe, tandis que pour les sections transversales, l’échantillon doit être orienté parallèlement à la direction de coupe. - Placez une cassette propre sur le moule et assurez-vous que suffisamment de paraffine couvre entièrement toute la cassette pour tenir l’échantillon sur la cassette.
- Placez le moule sur la plaque froide et laissez la paraffine se régler complètement (10-20 min) avant de libérer le bloc de la moisissure.
- Procéder à la sectionnage ou le transfert des blocs à 4 °C pour le stockage.
REMARQUE : Le protocole peut être mis en pause ici. Les blocs intégrés peuvent être stockés à 4 °C pendant une période pouvant aller jusqu’à trois mois.
4. Préparation de lames membranaires de polyéthylène naphtalate (PEN)
- Submerger les lames de membrane PEN dans la solution de désactivation RNase pendant 3 s suivies de deux brèves lavages dans l’eau traitée par DEPC pour enlever les RNases sur les lames. Sécher les lames dans un incubateur de 37 °C pour enlever la solution laissée.
- Traitement uv les lames à l’aide d’une lampe UV dans une armoire à débit laminaire pendant 30 min pour améliorer les propriétés hydrophiles pour l’adhérence améliorée de paraffine.
5. Sectionnage
- Utilisez un microtome dans l’étape de sectionnage (voir Tableau des matériaux).
- Placez une nouvelle lame dans le support du couteau, et gardez toujours la garde du couteau en l’état lorsqu’il n’est pas activement sectionné
ATTENTION : Les lames de microtome sont extrêmement pointues et peuvent causer des dommages importants lorsqu’elles sont manipulées de manière inappropriée.
REMARQUE : Il y a deux mécanismes de verrouillage sur le microtome, l’un est sur le côté de la machine et l’autre sur la poignée de la roue. Les deux doivent être engagés lorsqu’ils ne sont pas activement sectionnant. - Ajuster le bloc du couteau pour qu’il soit le plus près possible de l’échantillon sans toucher. Assurez-vous que le bras microtome n’entre jamais en plein contact avec le bloc de couteau car cela causera des dommages structurels catastrophiques au microtome.
- Allumez la plaque froide avant de commencer. Gardez les blocs de paraffine sur la plaque froide avant de sectioner et de refroidir les blocs si nécessaire pendant la section pour empêcher les blocs de ramollir.
- Remplissez le bain d’eau avec de l’eau traitée par dePC et chauffez à 42 °C avant de commencer.
- Couper les blocs à la profondeur désirée (où la section qui vous intéresse) et les blocs de paraffine de section à l’épaisseur désirée (6-10 μm) à l’aide du microtome; un bloc bien sectionné formera un « ruban » au bord de la lame. Pour les graines d’orge, section d’une épaisseur de 8 μm.
- Transférer délicatement les rubans du microtome vers le bain d’eau à l’aide d’un pinceau fin ou de forceps fins, en veillant à ce que le ruban soit plat à la surface de l’eau.
- Tenez une lame à un angle de 45°, à l’aide d’un mouvement vers le haut, soulevez un ruban hors de l’eau sur la glissière et retirez soigneusement l’excès d’eau avec un tissu sans peluche.
- Glisse sèche pendant 30 min à 37 °C pour éliminer toute eau restante sous la paraffine.
- Procéder à l’enlèvement ou au stockage à 4 °C dans une boîte fermée dans des conditions de déshydratation (à utiliser dans les jours).
- Retirer la paraffine en lavant les lames 3x pour 20 s chacune en xylène, suivie de 2x lavages de 30 s dans 100% (v/v) d’éthanol et 2x lavages de 30 s dans 70% (v/v) éthanol.
- Passez immédiatement à la microdésection de capture au laser après l’enlèvement de la paraffine.
REMARQUE : Cryosection est une méthode alternative qui a été couplée avec succès avec LCM. La préparation de l’échantillon pour la cryosection diffère.
6. Microdésection de capture laser
- Utilisez un microscope à microdissection à capture laser (voir tableau des matériaux)pour microdissectiser les cellules provenant de sections de tissus dé-paraffinisés et séchés.
- Chargez les diapositives sur les trois emplacements disponibles.
- Utilisez les bouchons adhésifs spéciaux des tubes de collecte pour recueillir les échantillons capturés. La capture sans liquide (collecte « sèche ») minimise l’activité de RNase. Chargez les tubes de collecte dans les fentes disponibles.
- Déplacez l’étape pour localiser la région de l’échantillon qui doit être coupée. Cela peut être fait à l’aide de la souris ou du joystick de la machine LCM, ou les touches fléchées sur le clavier.
- Pour optimiser la vitesse de coupe, la réduction de l’énergie et de la mise au point, la pression laser catapultant l’énergie (LPC) de l’énergie et de se concentrer, d’abord coupé sur un segment vierge libre de tissu sur la lame de la membrane. Pour les semences d’orge, vitesse de coupe = 18, CutEnergy = 52 CutFocus = 63, LPCEnergy = 78, LPC focus = 61 à 10x grossissement.
REMARQUE : La mise au point de coupe et l’énergie doivent être ajustées pour les différentes diapositives, les différents tissus et les zones capturées, mais les règles générales sont la puissance de catapultation est plus élevée que la puissance de coupe et le laser doit être défocus pour catapulter. Plus le grossissement de la lentille objective est élevé, plus le focus du laser est petit et plus l’énergie est élevée. - Utilisez les outils De dessin pour sélectionner des cellules en décrivant la zone d’intérêt.
- Sélectionnez la fonction RoboLPC de la barre d’outils de la fonction pour catapulter les cellules dans les bouchons adhésifs en fonction des paramètres optimisés obtenus en coupant sur un segment noir ci-dessus.
REMARQUE : Les paramètres de LCM varient d’un type de tissu à l’autre ainsi que l’épaisseur de la section, la dureté des tissus et les lentilles objectives. Par conséquent, il est préférable d’optimiser chaque diapositive sur une zone de membrane ordinaire sans spécimen de tissu avant de couper l’échantillon réel. - Utilisez les outils Indicateur pour marquer les régions d’intérêt pour les localiser immédiatement en sélectionnant cet indicateur dans la liste des éléments.
- Vérifiez par "CapCheck" pour inspecter le bouchon adhésif pour confirmer que des échantillons ont été capturés. En règle générale, le LCM de 10 à 15 sections (~200 cellules) par bouchon est nécessaire pour l’extraction de l’ARN.
- Gardez les échantillons capturés sur la glace. Procéder immédiatement à l’extraction de l’ARN afin d’éviter la dégradation de l’ARN.
REMARQUE : Certaines microscopies LCM sont équipées d’une lumière fluorescente qui permet de capturer des cellules étiquetées avec des marqueurs fluorescents.
7. Extraction de l’ARN
- Utilisez un kit d’isolation de l’ARN à faible entrée (voir Tableau des matériaux)pour l’extraction de l’ARN après LCM. Ces kits sont conçus pour récupérer l’ARN total de haute qualité de manière cohérente à partir de moins de dix cellules.
- Isoler l’ARN total des types de cellules capturées selon les instructions du fabricant, y compris le traitement DNase à la colonne.
REMARQUE : La première étape de l’extraction de l’ARN où le tube est inversé et cligné est cruciale pour s’assurer que les échantillons capturés sur le couvercle sont en contact avec le tampon d’extraction ajouté.
8. Amplification de l’ARN
- Utiliser un kit d’amplification de l’ARN antisens (ARN) (voir tableau des matériaux) pour l’amplification de l’ARN extrait par transcription in vitro afin de produire suffisamment d’ARN pour la synthèse de la bibliothèque ARN-seq.
- Effectuer deux tours d’amplification à l’aide du kit d’amplification de l’ARN selon les instructions du fabricant.
REMARQUE : Il est important de préchauffer le thermocycler et le couvercle à la température indiquée par le fabricant du kit (voir tableau des matériaux). Une autre approche au lieu de l’amplification de l’ARN consiste à utiliser un kit de préparation de la bibliothèque à faible entrée pour synthétiser la bibliothèque directement à partir de l’ARN extrait.
9. Quantification de l’ARN
- Quantifier et qualifier l’ARN à l’aide d’un système automatisé d’électrophorèse (voir tableau des matériaux).
REMARQUE : Un système automatisé d’électrophorèse est préférable car il nécessite moins d’échantillon (1-2 μL) et fournit une image et un électrophérogramme en forme de gel pour chaque échantillon individuel.
10. qRT-PCR et/ou ARN-seq
- Synthétisez l’ADN cD À partir de l’ARN pour qRT-PCR ou pour faire des bibliothèques ARN-seq à l’aide de kits de bibliothèque STANDARD ARN-seq.
Résultats
Nous avons généré des transcriptomes spécifiques aux tissus spatiaux et temporels à partir de graines d’orge pendant la germination à l’aide de notre protocole LCM ARN-seq10. L’étude a été réalisée en appliquant l’ARN-seq LCM à un petit nombre de cellules de trois organes embryonnaires (plumule, pointe radicule, scutellum) tous les 8 h sur un cours de 48 h pendant la germination (0-48 h, 7 points de temps) (Figure 2A,B).

Figure 2 : Cellules récoltées à partir de graines d’orge par LCM. (A) Section longitudinale d’une graine d’orge et, encart, d’un dessin animé agrandi indiquant les régions à partir desquelles les cellules ont été capturées. Bleu = plumule, magenta = radicle, vert = scutellum. (B) LCM de plumule, radicule et scutellum. Panneau supérieur, tissu avant catapultage de pression laser (LPC). Panneau inférieur, tissu après LPC. Les cellules ont été capturées à partir de 5 sections longitudinales consécutives de chaque échantillon avec trois répliques biologiques par échantillon. Chaque réplique se composait d’environ 200 cellules (~0,05 - 0,15 mm2 superficie totale). Barre = 100 μm. E = endosperme, C = couche de cellules concassées, SE = épithélium scutellar, S = scutellum. Figure reproduite à partir de données antérieures dans The Plant Journal avec l’autorisation10. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
La fixation et l’incorporation sont des étapes critiques car l’échantillon de tissu mal fixé peut entraîner des dommages tissulaires et la perte de la quantité et de la qualité de l’ARN. Ces étapes doivent être optimisées et ajustées pour les différentes espèces et types de tissus19,20,21. Ici, nous avons utilisé l’infiltration sous vide pour améliorer la pénétration des fixatifs dans les tissus de semences et les cellules. La déshydratation et l’infiltration de paraffine sont effectuées progressivement pour éviter les dommages aux échantillons de tissus. Nous avons accéléré le processus entier de déshydratation et d’infiltration de paraffine en utilisant un processeur de tissu automatisé avec agitation et vide appliqué à chaque étape. Cela réduit fortement le risque de dégradation de l’ARN se produisant pendant les étapes prolongées de traitement des tissus menées par un agent humain. Avant la section, il est crucial de traiter la lame membranière pour assurer l’adhérence de la paraffine et pour s’assurer qu’elle est exempte de RNase. Si les lames membranaires ne sont pas préparées correctement, la section de l’échantillon n’adhérera pas à la membrane, ce qui affectera l’étape de catapulsation du LCM et diminuera le transfert des tissus vers les bouchons adhésifs parce que l’échantillon se séparera de la membrane (voir la figure 1).
Une autre étape critique du protocole LCM RNA-seq est l’optimisation des paramètres LCM. Il y a cinq paramètres principaux à optimiser : (1) vitesse de coupe; (2) la réduction de l’énergie; (3) mise au point de coupe; (4) LPC énergie; et (5) LPC focus. Il est important d’ajuster correctement les paramètres de coupe afin de couper précisément la zone sélectionnée et de déloger des tissus environnants sans brûler le bord de la zone sélectionnée (Figure 3A,B). L’énergie et la concentration appropriées du PLC sont essentielles pour catapulter de façon fiable la zone sélectionnée de la lame aux bouchons adhésifs spéciaux des tubes de collecte, et toujours inspecter les échantillons capturés dans les bouchons pour s’assurer qu’un nombre suffisant de matériaux sont recueillis (Figure 3C). L’efficacité du LCM permet de capturer 1000 cellules en une heure, ce qui réduit considérablement la probabilité de dégradation de l’ARN pendant le traitement de l’échantillon. L’utilisation de tubes de collecte avec des bouchons adhésifs permet la collecte « sèche » sans aucun liquide de capture, réduisant considérablement les activités possibles de RNases. Il est important de garder les échantillons capturés sur la glace et de faire l’extraction de l’ARN immédiatement après la collecte de toutes les répliques.
L’extraction et l’amplification de l’ARN sont effectuées à l’aide de kits disponibles dans le commerce en suivant les instructions des fabricants. La quantification de l’ARN total avant l’amplification de l’ARN à l’aide d’un système automatisé d’électrophorèse permettra de détecter des bandes électrophorétiques distinctes et des pics fluorescents de sous-unités ribosomales 18S et 28S dans des échantillons d’ARN de bonne qualité (Figure 3D). D’autres pics correspondant à l’ARN ribosomal chloroplaste et mitochondrial seront trouvés avant les pics 18S et 28S. En raison de plusieurs pics d’ARN chez les espèces végétales, les valeurs rin (numéro d’intégrité de l’ARN) sont généralement inférieures à 9 (la valeur prévue du RIN provenant des tissus des mammifères) et ne reflètent pas exactement l’intégrité de l’ARN22. Il convient de continuer à amplifier l’ARN lorsque des pics 18S et 28S clairs sont visibles sans signe de dégradation de l’ARN. La quantité d’ARN récupérée dans notre expérience sur les semences d’orge variait entre trois types de tissus différents (plumule, radicule et scutellum) et, également entre les stades précoces et tardifs de la germination, malgré un nombre à peu près égal de cellules récoltées. Les quantités de graines d’orge variaient de 100 pg à 20 ng d’environ 200 cellules (0,5 à 100 pg par cellule). La quantité minimale d’ARN d’entrée recommandée pour l’amplification de l’ARN était de 100 pg. La quantité d’ARN extraite dépend de l’efficacité de l’extraction des différentes cellules récoltées et de l’abondance totale de transcription dans le type particulier de cellules à ce stade de développement19,21. Il est utile d’envisager cela lors de la planification d’une expérience LCM ARN-seq, bien que ces informations préalables ne seront pas disponibles dans tous les cas.
L’ARN synthétisé avec succès présentera une distribution de taille unimodale et symétrique de 100 à 1000 nucléotides avec un pic autour de 300 nucléotides après deux cycles d’amplification (Figure 3E). Dans notre expérience de semences d’orge, nous avons obtenu 500 pg à 2 μg d’ARN après deux cycles d’amplification de l’ARN10. La comparaison entre l’ARN amplifié et l’ARN non amplifié a démontré que l’amplification de l’ARN est reproductible, linéaire et n’introduit pas de biais systématique pour les données de transcription23,24,25. L’ARN de sortie peut être utilisé pour qRT-PCR et/ou ARN-seq pour l’analyse d’expression génétique spatiale et temporelle spécifique aux tissus. La validation après ARN-seq doit être effectuée pour corroborer les résultats de l’analyse de l’ARN. Cela peut être fait en examinant l’expression des gènes marqueurs de type cellulaire à partir des données ARN-seq à l’aide de l’hybridation in situ de l’ARN.
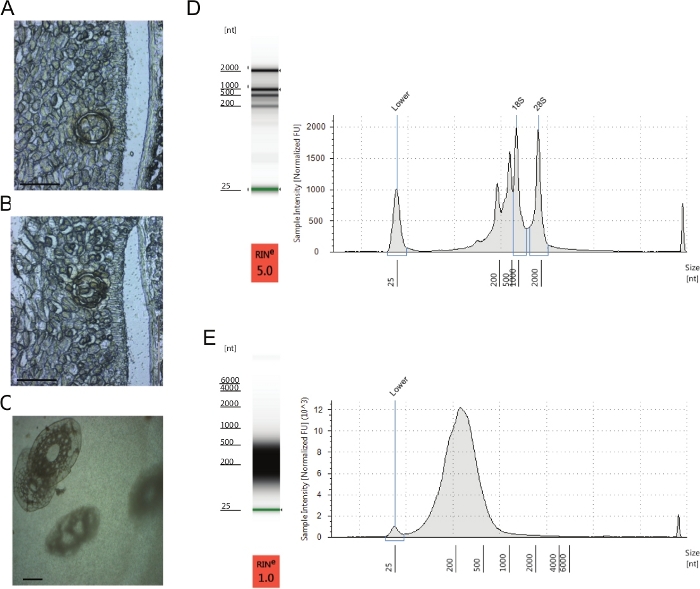
Figure 3 : Résultat représentatif de LCM. (A) Tissu après coupe avec l’énergie de coupe appropriée et de se concentrer. Une ligne fine claire peut être vue où la coupe a été faite. La zone sélectionnée sera délogée des matériaux environnants. Barre = 100 μm. (B) Tissu après coupe avec une énergie de coupe incorrecte et mise au point. La zone sélectionnée est toujours reliée aux matériaux environnants. Barre = 100 μm. (C) Examen des cellules capturées dans des bouchons adhésifs spéciaux des tubes de collecte. Barre = 100 μm. (D) Exemple de profil total de l’ARN avant l’amplification de l’ARN (image à gauche, semblable à un gel; droite, électrophérogramme) avec bandes électrophorériques distinctes et pics fluorescents de sous-unités ribosomales 18S et 28S. Les pics non étiquetés correspondent à l’ARN ribosomal supplémentaire, y compris le chloroplaste et les ribosomes mitochondriaux. (E) Exemple d’un profil d’ARN typique après amplification de l’ARN (image à gauche, semblable à un gel; à droite, électrophérogramme). Une répartition des tailles de 100 à 1000 nucléotides, avec un pic autour de 300 nucléotides, a été détectée. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
Des analyses ARN-seq ont été effectuées sur tous les échantillons dans des triples biologiques. Une parcelle multidimensionnelle de gènes exprimées dans les différents tissus de plus de 48 h de germination illustre une plus grande similitude entre les échantillons d’un seul tissu qu’entre les échantillons provenant du même point de temps mais des tissus différents (Figure 4A). Nous avons ensuite entrepris de déterminer dans quelle mesure l’expression des gènes a été réglementée différemment dans les tissus individuels au fil du temps pendant la germination. Le nombre de gènes exprimés différemment (DEG) a augmenté progressivement au cours de la germination dans chaque tissu, par rapport au délai de 0 h de ce tissu (figure 4B). Vingt-cinq pour cent (910) de plumule, 34 % (1876) de radicule et 41 % (2562) de scutellum DEGs se sont révélés exclusivement exprimés différemment dans ce tissu (c.-à-d. que ces gènes n’étaient pas exprimés différemment dans les autres tissus, figure 4C). Pris ensemble, ces résultats démontrent comment LCM ARN-seq peut être utilisé pour comprendre l’expression temporelle et spatiale des gènes au cours du développement des plantes.

Figure 4 : Analyse des bibliothèques d’ARN d’échantillons de LCM montrant une séparation distincte des différents tissus sur 48 h de germination d’orge. (A) Parcelle multidimensionnelle de gènes exprimées dans les différents tissus sur 48 h de germination. Chaque point représente 1 échantillon, et la distance entre 2 points reflète le changement de pli de journal de premier plan (FC) des échantillons d’ARN correspondants. L’axe x, y et z représente la première, la deuxième et la troisième dimension logFC. P = plumule, R = radicle, S = scutellum, 0-48h = h de germination, R1-3 = Réplique 1-3. (B) Nombre de gènes importants à haute réglementation (rouge) et régulés (bleus) exprimés différemment (DEG) pendant six points de temps par rapport au point de temps 0 h pour trois types de tissus. (C) Diagramme de Venn montrant le nombre de DEGs dans trois types de tissu. Les gènes dans les ensembles qui se chevauchent montrent l’expression différentielle dans deux ou trois types de tissus. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
Discussion
De nombreuses études d’expression génétique spécifiques aux tissus ont été limitées par la dissection à la main d’échantillons, qui prend beaucoup de temps, qui demande beaucoup de main-d’œuvre, présente un risque élevé de contamination et ne peut utiliser que des échantillons qu’un agent humain est suffisamment habile pour récolter. LCM est une technique précise et sans contact pour recueillir les cellules à partir de sections de tissus fixes à l’aide d’un faisceau laser actionné mécaniquement sous visualisation microscopique.
Une bonne préparation de l’échantillon est essentielle pour le LCM. Le processus repose sur la fixation et l’incorporation appropriées d’échantillons pour maintenir un bon équilibre entre la morphologie des tissus et l’intégrité de l’ARN. Le protocole présenté ici fournit des étapes optimisées de fixation et d’incorporation pour les graines d’orge, ce qui comprend un temps de fixation plus long et l’infiltration sous vide des fixatifs. L’analyse de différents tissus ou espèces nécessitera l’optimisation des conditions, car celles-ci diffèrent généralement d’un tissu à l’autre. Un autre aspect important de ce protocole est l’étape d’amplification de l’ARN qui garantit que des quantités suffisantes d’ARN sont générées pour la construction de la bibliothèque ARN-seq. Nous constatons que l’amplification de l’ARN entraîne des bibliothèques de haute qualité, mais alternativement faibles méthodes de préparation de bibliothèque d’entrée pourraient être utilisées25. L’identification histologique des cellules cibles est une étape critique de notre méthode et peut fournir des défis pour les types de cellules rares ou mal caractérisés. La reconnaissance des cellules cibles peut être améliorée en utilisant des taches histologiques traditionnelles ou des taches d’immunofluorescence dans le cadre de l’étape de prétraitement ou, alternativement, en utilisant des lignes transgéniques qui expriment des marqueurs cellulaires fluorescents26,27.
La technique LCM a d’abord été développée pour être utilisée sur des échantillons d’origine animale pour l’analyse de transcription spécifique à la cellule1. Cependant, un large éventail d’analyses génomiques, protéomiques et épigénétiques ont été développés par la suite. L’adaptation de ces méthodes pour l’application dans les études biologiques végétales est un défi permanent18,19,20,28. Schad et coll. ont démontré que les protéines et métabolites des faisceaux vasculaires d’Arabidopsis pouvaient être analysés à l’aide de LCM avec l’électrophorèse de gel 2-D et la spectrométrie de masse29,30. Récemment, Latrasse et coll. ont effectué une analyse de l’immunoprécipitation de la chromatine (ChIP)-qPCR sur les étamines et les carpels disséqués par LCM à l’aide de LCM, établissant que l’homogénéité tissulaire améliore la résolution des événements épigénétiques spécifiques aux cellules31. Turco et coll. ont également couplé LCM avec le séquençage de bisulfite pour étudier les événements de méthylation de l’ADN pendant la vascularisation dans le sorgho, soulignant la différence tissulaire spécifique entre les tissus vasculaires et non vasculaires32. Fait intéressant, plusieurs études ont appliqué LCM pour explorer les interactions plante-microbe et plante-pathogène33,34,35. La capacité de LCM à visualiser et capturer des cellules à différents stades d’infection a fourni des informations sur les réponses spatiales et temporelles des cellules infectées.
Notre protocole LCM couplé à l’ARN-seq présenté ici facilitera le profilage global spatio-temporel de l’expression génique de types de cellules distincts. Avec des méthodes de purification améliorées pour la chromatine et les protéines ainsi que des instruments de spectrométrie de masse améliorés, LCM sera un outil émergent pour faciliter les études épigénomiques et protéomiques spécifiques à la cellule chez les plantes.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Ces travaux ont été appuyés par le Australian Research Council Centre of Excellence in Plant Energy Biology (CE140100008) à JW. M.G.L a reçu le soutien d’une subvention de départ de l’Université La Trobe. Nous remercions la Plate-forme de génomique la Trobe pour son soutien dans le séquençage à haut débit et l’analyse des données. Nous remercions le professeur agrégé Matthew Tucker pour les conseils d’experts sur l’établissement de LCM dans notre laboratoire.
matériels
| Name | Company | Catalog Number | Comments |
| Acetic acid 100 % ACS/R. | AnalaR NORMAPUR (BioStrategies) | VWRC20104.323 | |
| AdhesiveCap 200 opaque | Zeiss | 415190-9181-000 | |
| Clear base moulds 8 X 10 | Leica | 3803015 | |
| Diethyl pyrocarbonate | Sigma-Aldrich | 40718-25ML | |
| High Sensitivity RNA ScreenTape | Agilent | 5067-5579 | |
| Lowprofile disp.blades DB80LS | Leica | 14035843489 | |
| MembraneSlide 1.0 PEN | Zeiss | 415190-9041-000 | |
| MessageAmp II aRNA Amplification Kit | Ambion (ThermoFisher) | AMB17515 | |
| On-Column DNase I Digestion Set | Sigma-Aldrich | DNASE70 | |
| Ovation RNA-Seq System V2 | NuGen (Integrated Science) | 7102-08 | |
| Paraffin (Surgipath Paraplast) | Leica | 39601006 | |
| PicoPure RNA Isolation Kit | ABI (ThermoFisher) | KIT0214 | |
| RNaseZap RNase Decontamination Solution | Ambion (ThermoFisher) | AM9780 | |
| Xylene | AnalaR NORMAPUR (BioStrategies) | VWRC28975.360 | |
| Leica Benchtop Tissue Processor | Leica Biosystems | TP1020 | |
| Leica Heated Paraffin Embedding Module | Leica Biosystems | EG1150H | |
| Leica Cold Plate | Leica Biosystems | EG1150C | |
| Safemate Class 2 Biological Safety Cabinets | LAF Technologies Pty Ltd | Safemate 1.5 | |
| Leica Fully Automated Rotary Microtome | Leica Biosystems | RM2265 | with PALMRobo v 4.6 software |
| Zeiss PALM MicroBeam LCM system | Zeiss miscroscopy | ||
| TapeStation | Agilent | TapeStation 2200 |
Références
- Emmert-Buck, M. R., et al. Laser capture microdissection. Science. 274 (5289), 998-1001 (1996).
- Alevizos, I., et al. Oral cancer in vivo gene expression profiling assisted by laser capture microdissection and microarray analysis. Oncogene. 20 (43), 6196-6204 (2001).
- Cong, P., et al. In situ localization of follicular lymphoma: description and analysis by laser capture microdissection. Blood, The Journal of the American Society of Hematology. 99 (9), 3376-3382 (2002).
- Blokhina, O., et al. Laser capture microdissection protocol for xylem tissues of woody plants. Frontiers in Plant Science. 7, 1965(2017).
- Casson, S., Spencer, M., Walker, K., Lindsey, K. Laser capture microdissection for the analysis of gene expression during embryogenesis of Arabidopsis. The Plant Journal. 42 (1), 111-123 (2005).
- Chen, Z., et al. LCM-seq reveals the crucial role of LsSOC1 in heat-promoted bolting of lettuce (Lactuca sativa L.). The Plant Journal. 95 (3), 516-528 (2018).
- Jiao, Y., et al. A transcriptome atlas of rice cell types uncovers cellular, functional and developmental hierarchies. Nature Genetics. 41 (2), 258-263 (2009).
- Kivivirta, K., et al. A protocol for laser microdissection (LMD) followed by transcriptome analysis of plant reproductive tissue in phylogenetically distant angiosperms. Plant Methods. 15 (1), 1-11 (2019).
- Li, P., et al. The developmental dynamics of the maize leaf transcriptome. Nature Genetics. 42 (12), 1060-1067 (2010).
- Liew, L. C., et al. Temporal tissue-specific regulation of transcriptomes during barley (Hordeum vulgare) seed germination. The Plant Journal. 101 (3), 700-715 (2020).
- Matas, A. J., et al. Tissue-and cell-type specific transcriptome profiling of expanding tomato fruit provides insights into metabolic and regulatory specialization and cuticle formation. The Plant Cell. 23 (11), 3893-3910 (2011).
- Sakai, K., et al. Combining laser-assisted microdissection (LAM) and RNA-seq allows to perform a comprehensive transcriptomic analysis of epidermal cells of Arabidopsis embryo. Plant Methods. 14 (1), 10(2018).
- Zhan, J., et al. RNA Sequencing of Laser-Capture Microdissected compartments of the maize kernel identifies regulatory modules associated with endosperm cell differentiation. The Plant Cell. 27 (3), 513-531 (2015).
- Hwang, B., Lee, J. H., Bang, D. Single-cell RNA sequencing technologies and bioinformatics pipelines. Experimental & Molecular Medicine. 50 (8), 1-14 (2018).
- Zeb, Q., Wang, C., Shafiq, S., Liu, L. Single-Cell Omics. , Elsevier. 101-135 (2019).
- Deal, R. B., Henikoff, S. The INTACT method for cell type-specific gene expression and chromatin profiling in Arabidopsis thaliana. Nature Protocols. 6 (1), 56(2011).
- Heiman, M., Kulicke, R., Fenster, R. J., Greengard, P., Heintz, N. Cell type-specific mRNA purification by translating ribosome affinity purification (TRAP). Nature Protocols. 9 (6), 1282(2014).
- Bevilacqua, C., Ducos, B. Laser microdissection: A powerful tool for genomics at cell level. Molecular Aspects of Medicine. 59, 5-27 (2018).
- Nelson, T., Tausta, S. L., Gandotra, N., Liu, T. Laser microdissection of plant tissue: what you see is what you get. Annual Reviews in Plant Biology. 57, 181-201 (2006).
- Day, R. C., Grossniklaus, U., Macknight, R. C. Be more specific! Laser-assisted microdissection of plant cells. Trends in Plant Science. 10 (8), 397-406 (2005).
- Takahashi, H., et al. A method for obtaining high quality RNA from paraffin sections of plant tissues by laser microdissection. Journal of Plant Research. 123 (6), 807-813 (2010).
- Schroeder, A., et al. The RIN: an RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7 (1), 3(2006).
- Ferreira, E. N., et al. Linear mRNA amplification approach for RNAseq from limited amount of RNA. Gene. 564 (2), 220-227 (2015).
- Schneider, J., et al. Systematic analysis of T7 RNA polymerase based in vitro linear RNA amplification for use in microarray experiments. BMC Genomics. 5 (1), 29(2004).
- Shanker, S., et al. Evaluation of commercially available RNA amplification kits for RNA sequencing using very low input amounts of total RNA. Journal of Biomolecular Techniques. 26 (1), 4(2015).
- Bhattacherjee, V., et al. Laser capture microdissection of fluorescently labeled embryonic cranial neural crest cells. Genesis. 39 (1), 58-64 (2004).
- Clément-Ziza, M., Munnich, A., Lyonnet, S., Jaubert, F., Besmond, C. Stabilization of RNA during laser capture microdissection by performing experiments under argon atmosphere or using ethanol as a solvent in staining solutions. RNA. 14 (12), 2698-2704 (2008).
- Blokhina, O., et al. Parenchymal Cells Contribute to Lignification of Tracheids in Developing Xylem of Norway Spruce. Plant Physiology. 181 (4), 1552-1572 (2019).
- Schad, M., Lipton, M. S., Giavalisco, P., Smith, R. D., Kehr, J. Evaluation of two-dimensional electrophoresis and liquid chromatography-tandem mass spectrometry for tissue-specific protein profiling of laser-microdissected plant samples. Electrophoresis. 26 (14), 2729-2738 (2005).
- Schad, M., Mungur, R., Fiehn, O., Kehr, J. Metabolic profiling of laser microdissected vascular bundles of Arabidopsis thaliana. Plant Methods. 1 (1), 2(2005).
- Latrasse, D., et al. The quest for epigenetic regulation underlying unisexual flower development in Cucumis melo. Epigenetics & Chromatin. 10 (1), 22(2017).
- Turco, G. M., et al. DNA methylation and gene expression regulation associated with vascularization in Sorghum bicolor. The New Phytologist. 214 (3), 1213-1229 (2017).
- Gomez, S. K., Harrison, M. J. Laser microdissection and its application to analyze gene expression in arbuscular mycorrhizal symbiosis. Pest Management Science: Formerly Pesticide Science. 65 (5), 504-511 (2009).
- Roux, B., et al. An integrated analysis of plant and bacterial gene expression in symbiotic root nodules using laser-capture microdissection coupled to RNA sequencing. The Plant Journal. 77 (6), 817-837 (2014).
- Tang, W., Coughlan, S., Crane, E., Beatty, M., Duvick, J. The application of laser microdissection to in planta gene expression profiling of the maize anthracnose stalk rot fungus Colletotrichum graminicola. Molecular Plant-Microbe Interactions. 19 (11), 1240-1250 (2006).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon