Method Article
Drosophila melanogasterにおける遺伝子座特異的リボソームRNA転写の一塩基多型感受性FISH検出
要約
このプロトコルは、Drosophila melanogasterのXまたはY染色体リボソームDNA遺伝子座に由来するリボソームRNA転写産物を区別するための一塩基多型感受性蛍光in situハイブリダイゼーション(SNP-FISH)法について説明しています。
要約
リボソームの機能のために十分なリボソームRNA(rRNA)が転写されるように、ゲノムにはrRNAをコードする配列の数百のタンデム重複が含まれており、リボソームDNA(rDNA)遺伝子座と呼ばれる領域を構成しています。多くの生物のゲノムには、異なる染色体に分布する複数のrDNA遺伝子座が含まれており、rRNAは複数のrDNA遺伝子座から転写される場合もあれば、単一の遺伝子座のみから転写される場合もあります。rRNA転写源の変化は、しばしばリボソームの苦痛を示しています。しかし、rRNAの均質性は、rRNAが複数のrDNA遺伝子座から転写されたのか、それとも単一のrDNA遺伝子座から転写されたのかを区別するのを妨げます。ここでは、一塩基多型感受性蛍光 in situ ハイブリダイゼーション(SNP-FISH)を使用して、X染色体とY染色体上の Drosophila melanogasterrDNA 遺伝子座間のrRNA転写を区別する方法について説明します。この方法は、遺伝子座特異的なrRNAバリアントを活用して、シングルセルの分解能でrRNA転写のソースを簡単に検出できます。このアッセイは、任意の ショウジョウバエ 細胞タイプに適用でき、他のシステムでの使用にも適合させることができます。
概要
リボソームの機能に必要な18S、5.8S、および28SリボソームRNA(rRNA)は、45S pre-rRNAと呼ばれる単一のシストロンで共転写されます。3つのrRNAは、45SプレrRNA内で2つの内部転写スペーサー配列(ITS)によって分離され、5'末端と3'末端で外部転写スペーサー配列(ETS)に隣接しています。これらのスペーサー配列はすべて、成熟rRNAがリボソームに取り込まれるために、45SプレrRNAから除去されます(1を参照)。リボソームの活性をサポートするために必要なrRNA産生に対する高い需要を満たすために、すべての真核生物のゲノムには45S配列の数百のコピーが含まれています。これらのコピーはタンデムリピートでクラスター化され、リボソームDNA(rDNA)遺伝子座と呼ばれるゲノム領域を形成します。ほとんどの真核生物のゲノムには、別々の染色体に広がる複数のrDNA遺伝子座が含まれています(2を参照)。45Sコピーの冗長性が高いということは、通常、転写に必要な数よりもはるかに多くの45Sコピーが存在することを意味し、45Sコピーの大部分は転写的に沈黙し、ヘテロクロマチン3に埋もれています。転写活性はrDNA遺伝子座間で均一ではなく、rDNA遺伝子座転写の変動は生物間で広く観察されており4,5,6、45S転写が個々のrDNA遺伝子座で異なる制御を受けていることを示しています。遺伝子座全体にわたるrDNAサイレンシングは、植物、無脊椎動物、および脊椎動物において観察されており7,8,9,10、遺伝子座全体にわたるrDNAサイレンシングがrRNA投与量を調節するための主要なメカニズムであることを示唆している11。rRNA転写はリボソーム産生における重要な調節ステップであり、翻訳活性を調節するrRNA転写の変化は、正常な分化と疾患状態の両方の重要な特徴である12。rDNA遺伝子座の転写の変化は、総rDNAコピー数13の減少にも関連しています。したがって、遺伝子座特異的なrDNA転写の変化は、細胞生理機能の破壊の重要な指標となる可能性があります。
遺伝子座特異的なサイレンシングがrRNA転写調節の主要な源であるように見えますが、このサイレンシングを確立し、どのrDNA遺伝子座が転写活性を持つかを指示するメカニズムはほとんどわかっていません。rDNA配列の均質性は、個々のrDNA遺伝子座転写の評価を困難にし、遺伝子座特異的なrDNA転写活性の広範な解析を妨げます。この課題は、同じゲノム内のrDNAコピー間の構造的および一塩基配列の変異を利用することで克服できる可能性があります4,5,6,13。これらの変異体の一部は、個々の遺伝子座のほとんどまたはすべてのコピー間で共有され、rDNA遺伝子座を区別する複数の変異体の固有のrDNA遺伝子座ハプロタイプを作成します。実際、テロメア・テロメア・コンソーシアムによるrDNA変異体の特定の遺伝子座への最近のマッピングにより、ヒトゲノム14における遺伝子座特異的な共有rDNA変異体が明らかになった。このようなrDNA遺伝子座マップは、シーケンシングデータセットからの遺伝子座特異的転写の同定を可能にする可能性があります。ただし、これらのマップの可用性は現在限られており、作成は容易ではありません。rDNA遺伝子座はDrosophila melanogaster(X染色体とY染色体ごとに1つの遺伝子座)15の性染色体にのみ存在するため、Y染色体のrDNA遺伝子座はXX人の女性には存在しない。Y染色体遺伝子座からのこの自然な分離は、XとYのrDNA遺伝子座の間の一塩基多型(SNP)変異体を、男性(XY)には存在するが女性(XX)には存在しない任意の変異体としてショートリードシーケンシングで容易に識別できることを意味します。以前に同定されたSNPは、遺伝子型が未定のショウジョウバエ株で、男性と女性のDNAの簡単なPCRおよびサンガーシーケンシングを通じて迅速にテストできます。さらに、X染色体がrDNA遺伝子座を持たないショウジョウバエ株が利用できるということは、個々のXおよびYのrDNA遺伝子座を個別に単離して、さらに正確なrDNA SNP特性評価を行うことができることを意味します。ショウジョウバエrDNA遺伝子座間のSNP変異体のこの容易な同定は、単一のXおよびYのrDNA遺伝子座SNPハプロタイプを決定することを可能にする13。X染色体rDNA遺伝子座も、通常、ショウジョウバエの男性では完全に沈黙していますが、両方のX染色体遺伝子座は女性で転写されます10,17、ショウジョウバエは遺伝子座全体のrDNAサイレンシングを研究するための有用なシステムとなっています。
蛍光 in situ ハイブリダイゼーション(FISH)は、組織の個々の細胞内の特定のRNAまたはDNA配列の存在を視覚化するための強力なツールです。FISH標識は、フルオロフォアに結合したアンチセンスオリゴヌクレオチドプローブを介してRNAまたはDNA配列を標的とします。FISHプローブは、通常、可視化するのに十分な安定したターゲット結合を提供するために~20ヌクレオチドの長さである必要がありますが、この長さのプローブは、1つのヌクレオチドのみが異なる非ターゲット配列にも容易に結合できます (図1A)。逆に、一塩基特異性を付与するほど短いプローブは、可視化に十分な安定性を持って結合しません。特異性と安定性のバランスをとるために、SNP感受性FISH(SNP-FISH)は、~26ヌクレオチド長のアンチセンス蛍光プローブと、プローブ18の5'末端を除くすべてに結合する非蛍光センスマスクオリゴヌクレオチドを組み合わせたものです。このマスクは、プローブの最も多くの5'ヌクレオチド10個のみを一本鎖に残し、ターゲット結合に利用できます (図1B)。プローブのこの短い一本鎖部分は、ターゲットに特異性を与えますが、これらの10ヌクレオチドと1つでもミスマッチを持つRNAとの交差反応性を防ぎます (図1B)。プローブの5'端がそのターゲットに結合すると、パッシブストランド変位によりプローブからマスクが剥がれ、3'領域がターゲットに結合し、安定したターゲット標識が作成されます (図1B)18。したがって、SNP-FISHは、それぞれが異なるSNPを補完するが共通のマスクを共有する異なる蛍光色素を持つプローブのペアを使用して、単一のSNPが異なるRNAを異なる方法で視覚化できます (図1C)。 この方法では、単一の蛍光色素標識プローブのみを標的SNPにリクルートしますが、共有rDNAハプロタイプ内の複数のSNPに複数のプローブマスクセットをプールするために使用できるため、単一のrRNAのSNP感度検出を増幅できます (図1D)。さらに、rRNAは非常に豊富に転写されるため、RNAあたりの蛍光標識の数が少ないFISH実験では、rRNA転写産物を簡単に視覚化できます。このようにRNAあたりの蛍光色素の要件が低いため、SNP-FISHは、少数のユニークなSNPのみを持つ特定の遺伝子座からのrRNAを可視化できます。この方法は、さまざまな ショウジョウバエ 組織および発生段階における遺伝子座特異的なrRNA転写を容易に検出できる17。これは、老化中に排他的なY-rDNA転写とX染色体およびY染色体rDNA遺伝子座の共発現の間で変化する ショウジョウバエ の雄性生殖幹細胞に特に効果的です13。ここでは、 ショウジョウバエ 精巣の異なるXおよびY rRNA転写産物を可視化するためのSNP-FISHプロトコルを提供し、遺伝子座特異的なrRNAサイレンシングを評価するという全体的な目標を達成します。この方法では、一般的な y1w1 実験室用 ショウジョウバエ 株 (図1D)のX染色体とY染色体上のrDNA遺伝子座間で以前に特徴付けられた4つのSNPを使用します。
プロトコル
1. バッファーおよび試薬の調製
注:ステップ1、3、および4全体でRNaseフリー技術を使用し、認定されたRNaseフリー試薬を使用する必要があります。
- ケミカルフードで、16%ホルムアルデヒド10 mLと4 mLのRNaseフリー10x PBSを26 mLのRNaseフリー水に加え、1x PBS固定溶液に40 mLの4%ホルムアルデヒドを調製します。1 mLアリコートに分配し、調製後30分以内に-20°Cで保存します。固定液は-20°Cで最大6ヶ月間保存できます。
注意: 溶液にはホルムアルデヒドが含まれています。手袋と適切なPPEで取り扱い、安全に廃棄してください。 - 1 mLの20x RNaseフリー生理食塩水-クエン酸ナトリウム(SSC)、1 gのデキストラン硫酸塩、100 μLの100 mg/mL 出芽酵母 tRNA、100 μLの200 mMバナジルリボヌクレオシド複合体、1 mLの5%RNaseフリーBSA、1 mLの脱イオンホルムアミドを6 mLのRNaseフリー水と混合して、10 mLのハイブリダイゼーションバッファーを調製します。1.5 mLマイクロチューブに1 mLアリコートに分配し、-20°Cで最大1ヶ月間保存します。
- 5 mLの20x RNase-free SSC、5 mLの脱イオンホルムアミド、50 μLのTriton-Xを40 mLのRNaseフリー水に加えて、50 mLの洗浄バッファーを調製します。室温で暗所で最大1か月間保管してください。
- 100 μL の 1M Tris-HCl pH 7.5-8.0、20 μL の 0.5M EDTA、50 μL の 5M NaCl を 9.8 mL の水に加えて、10 mL の DNA 分離バッファーを調製します。
2. XおよびY特異的なrRNA SNPのサンプルジェノタイピング
- 交配していないホモ接合Xの雌と、同じX染色体を持つ雄を0.2 mLのチューブに集め、氷の上で冷やします。
- サンプルあたり50 μLのDNA分離バッファーと0.5 μLの20 mg/mLプロテイナーゼKを組み合わせます。
- 50 μLのDNA分離バッファー/プロテイナーゼKをショウジョウバエのサンプルに加え、ピペットチップを使用してサンプルをホモジナイズします。
- サンプルを37°Cで30分間インキュベートし、続いて95°Cで2分間インキュベートします。40 mLのサンプル(動物の破片を避けてください)を清潔な1.5 mLチューブに移します。
- 次のプライマーペアの10 μM濃度を使用してPCRすることにより、各DNAサンプルから各SNP標的領域を個別に増幅します:18S SNP Forward + Reverse;ITS1 SNP フォワード + リバース。28S SNPフォワード+リバース。プライマー配列と予想されるPCRアンプリコンのサイズを 表1に示します。
- ゲル電気泳動を使用して、1%アガロース1x TAEゲル上でPCRサンプル全体を分離し、予想されるPCR産物を確認します。予想される製品サイズを 表1に示します。
- 市販のゲル抽出キットを使用して、ゲルからPCR産物を単離します。
- サンガーシーケンシングによるPCR産物のシーケンシング。各ターゲットのFプライマーとRプライマーの個別の反応を使用して、サンプルを両方向にシーケンシングします。
- 交配していない雌のDNAサンプルの 表2 の位置にあるX染色体SNPバリアントを決定します。予想されるXハプロタイプを 表2に記載する。
- オスDNAサンプルシーケンシングクロマトグラムのSNP位置を観察します。各SNP位置ですでに決定されたX SNPバリアントに基づいて、Y染色体SNPバリアントを推測します。予想されるYハプロタイプを 表2に記載する。
| プライマー名 | 順序 | 予想されるアンプリコンのサイズ |
| 18S SNP F | ガクタカットggttgcaacgg | 652bpの |
| 18S SNP R | TTCACCTCTCGCGTCGTAAT | |
| ITS1 SNP F | CTTGCGTGTTACGGTTGTTTC | 955bpの |
| ITS1 SNP R | ACAGCATGGACTGCGATATG | |
| 28S SNP F | ATGCGTAGAAGTGTTTGGCG | 598bpの |
| 28S SNP R | GCCGACTTCCCTTACCTACA |
表1:rDNA SNPをシーケンシングするためのプライマー配列。 プライマーペアは、18S、ITS1、および28S rDNA領域で以前に特性評価されたSNPを含むrDNA領域を増幅します。プライマーオリゴヌクレオチド配列と予想されるPCRアンプリコンサイズがリストされています。
| ハプロタイプ | SNPポジション | 順序 | ||
| X rDNA | 18秒 | 1603-ATACTTGTATTTTTTCATTG-1625 | ||
| ITS1-1 | 2873-CGTTAATAAATATTTGTAATT-2895 | |||
| ITS1-2 | 3115-GAAAATCGAAGAAACAAAATT-3137 | |||
| 28Sの | 5932-AACAAAAATGCCTAACTATAT-5954 | |||
| Y rDNAの | 18秒 | 1603-ATACTTGTATCTTTTCATTG-1624 | ||
| ITS1-1 | 2873-CGTTAATAAACATTTGTAATT-2895 | |||
| ITS1-2 | 3115-GAAAATCGAAAAAACAAAATT-3137 | |||
| 28Sの | 5932-AACAAAAAATGGCTAACTATAT-5954 | |||
表2:予想されるrDNAハプロタイプ。 y1w1 ショウジョウバエ株のX染色体およびY染色体rDNA遺伝子座で予想されるSNP。SNP は太字と下線で示しています。コンセンサス45S rRNA配列(補足ファイル1)に基づいて記載されている位置。
3. 精巣の解剖、固定、透過処理
- 実体顕微鏡、ワークステーション、解剖皿、および70%エタノール(または別のRNase処理)で鉗子を解剖します。
- 実体顕微鏡で ショウジョウバエを 解剖し、1x RNAseフリーPBSで精巣を分離します。1対の鉗子で動物の腹部の中央をつかみ、別の鉗子で腹部の端をつかんで動物をそっと引き離します。精巣は動物から解離し、鉗子を使用してさらに分離することができます。サンプルごとに30〜40個の精巣を収集します。
- 鉗子を使用して精巣をピックアップし、解剖皿から0.5 mLの1x RNaseフリーPBSを含む1.5 mLマイクロチューブに移します。
- PBSを吸引し、1mLの固定液を加えます。室温で、ニューティングシェーカーで30分間インキュベートします。
- 1x RNaseフリーPBS1 mLを2回、ニューティングシェーカーで各5分間洗浄します。PBSを1倍吸引し、RNaseを含まない70%エタノール(EtOH)を1 mL加えます。4°Cで一晩、ニューティングシェーカーでインキュベートします。
4. SNP感受性リボソームRNAフィッシュ
- エタノールを吸引し、1mLの洗浄バッファーを加えます。室温で3分間、ゲートシェーカーでインキュベートし、続いて2分間チューブを直立させて休ませます。
- プローブとマスクをハイブリダイゼーションバッファーと混合し、サンプルあたり総容量を100 μLにします。プローブ、マスク配列、および蛍光色素標識を 表3に示します。4つのSNPすべてを使用する場合は、ハイブリダイゼーションバッファーを80 μL、10 μM Y-SNPプローブを各1 μL(合計4 μL)、10 μM X-SNPプローブを各1 μL(合計4 μL)、10 μMコモンマスクを各3 μL(合計12 μL)調製します
- 洗浄バッファーを吸引し、100 μLのハイブリダイゼーション溶液を加えます。チューブの端に透明フィルムを貼り、アルミホイルで包んで光から保護し、37°Cの水浴で少なくとも24時間インキュベートします。
- ハイブリダイゼーション溶液を吸引せずに、1 mLの洗浄バッファーをサンプルに加えます。37°Cの水浴中で、暗所で30分間インキュベートします。
- 洗浄バッファーを吸引し、1 mLの洗浄バッファーをサンプルに加えます。37°Cの水浴中で、暗所で30分間インキュベートします。
- 洗浄バッファーを吸引し、50 μLの封入剤を添加します。サンプルは、すぐにスライドにマウントするか、マウント前に4°Cで最大1週間保存できます。
| SNPポジション | オリゴヌクレオチド | 順序 | 3'標識フルオロフォア | |
| 18秒 | X SNPプローブ | AAAAAATACAAGTATTTAATCACATA | アレクサ488 | |
| Y SNPプローブ | AAAAGATACAAGTATTTAATCACATA | アレクサ647 | ||
| マスク | タットガッタアタクト | |||
| ITS1-1 | X SNPプローブ | AAATATTTATTAACGGTAAGGATATT | アレクサ488 | |
| Y SNPプローブ | AAATGTTTATTAACGGTAAGGATATT | アレクサ647 | ||
| マスク | AATATCCTTACCGTTA | |||
| ITS1-2 | X SNPプローブ | GTTTCTTCGATTTTCATGTTCGAAAC | アレクサ488 | |
| Y SNPプローブ | GTTTTTTCGATTTTCATGTTCGAAAC | アレクサ647 | ||
| マスク | GTTTCGAACATGAAAA | |||
| 28Sの | X SNPプローブ | TTAGGCATTTTTTTTTTTTACTTGAAAA | アレクサ488 | |
| Y SNPプローブ | TTAGCCATTTTTTTTTTTTACTTGAAAA | アレクサ647 | ||
| マスク | TTTTCAAGTAAAACAA | |||
表3:rDNA SNP-FISHのプローブおよびマスク配列。 SNP の位置は太字で強調表示されています。マスクオリゴヌクレオチドに結合するプローブ部分に下線が引かれています。適切な3'標識蛍光色素の例を挙げていますが、適合する蛍光色素であれば何でも使用できます。
5. イメージング用サンプルの調製
- 解剖実体顕微鏡の下で作業し、ワイドオリフィスピペットを使用してサンプルを顕微鏡スライドに移します。
- 鉗子を使用して、イメージング時にサンプルを見つけやすくするために、精巣を一列に並べます(特定の向きは必要ありません)。サンプルをカバースリップで覆います。カバースリップの端をティッシュワイプで拭き取り、余分な封入剤を取り除きます。
- カバースリップの端をマニキュアで密封します。スライドを室温で10分間暗闇に置いて、マニキュアを乾かします。スライドは、共焦点イメージングにすぐに使用することも、イメージング前に4°Cで最大1か月間保存することもできます。
結果
SNPジェノタイピングのシーケンシング結果により、X遺伝子座とYrDNA遺伝子座の間のSNPの違いが検出されることが期待されます。これらのSNPは、シーケンシングクロマトグラムのSNP位置を直接評価することで検出されます (図2)。女性サンプルのシーケンシングでは、SNP位置に単一のシーケンシングシグナルがあることが予想され、2つのX染色体間のSNPホモ接合性を示します (図2A)。オスサンプルのシーケンシング結果は、SNP位置にダブルピークを持つことが予想されます (図2B)。女性サンプルのシーケンシングから決定されたX染色体の遺伝子型に基づいて、非XバリアントはY染色体rDNA SNPであると推測されます(つまり、 図2の18S SNPシーケンシングではX = T、Y = C)。あるいは、雄サンプルのSNP位置にある単一のシーケンシングピークは、そのSNP位置における雄と雌のrDNA遺伝子座間のホモ接合性を示し、その位置はrRNA SNP-FISHには使用できません。
SNP FISHのプロトコルに従って、サンプルをスライドにマウントし、密封されたカバースリップの下に置いた後、共焦点顕微鏡で観察することができます。表3に示すプローブを用いて、Y由来のrRNAシグナルをAlexa Fluor 647の発光で検出し、X由来のrRNAシグナルをAlexa Fluor 488の発光で検出します。rRNAシグナルは核小体で観察されることが予想され、核内のDAPI不良穴として特定できます(図3A)。核小体は、その大きな核と核小体のために、生殖細胞で特に同定が容易です(図3A)。微弱なシグナルは通常、細胞質にも見られますが(図3)、これは非特異的なシグナルであり、相補的なSNPを含むrRNAを含まないサンプルでも検出できます(つまり、Y rDNAを欠く細胞のYシグナルまたはX rDNAを欠く細胞のXシグナル;図 3B-C)。さらに、XおよびY rRNAの人工的共標識は、XまたはY rDNA遺伝子座を欠くサンプルであっても、体細胞ハブで検出できることがあります(図3B-C、黄色の矢印)。したがって、核シグナルのみを使用して遺伝子座特異的な転写を評価する必要があります。ほとんどの細胞、特に体細胞は、Y rRNAシグナルのみを有すると予想され、これはrRNAがY rDNA遺伝子座からのみ転写されることを示している(図3A、黄色の点線の円、および赤い矢印)10,17。しかし、XおよびY rRNAの共発現は、生殖細胞、特に生殖幹細胞13で検出されることが多い(図3A、白い点線の円)。共発現細胞のXおよびY rRNAシグナルは、隣接して1つの核小体を形成する場合(図3A上の細胞)または分離して2つの核小体を形成する場合(図3A下の細胞)があります。図 3 の例は、rDNA SNP-FISH から 2 つの SNP(2 つの ITS1 SNP)のみを対象とするプローブセットを使用したもので、rDNA SNP-FISH に必要な SNP は 2 つだけであることを示しています。しかし、4つのSNPすべてを標的とするプローブを使用すると、より頑健なシグナルが観察される13。
ネガティブコントロールは、特にメソッドを最初にトラブルシューティングする際に、プローブが間違ったSNPターゲットと交差反応しないことを確認するために、このアッセイの重要なインクルージョンです。X染色体陰性コントロールには、X染色体rDNAを欠くあらゆる状態、例えば、rDNA欠失を有するX染色体を有する男性が含まれる。X染色体ネガティブコントロールは、Y rRNAシグナルのみを検出し、X rRNAは検出しないことが期待されます (図3B)。Y染色体陰性コントロールには、Y染色体rDNAを欠く任意の状態が含まれます。これらの状態の例としては、XXの女性組織、Y染色体を欠く男性の組織、またはrDNA欠失15を持つY染色体を持つ男性の組織があります。Y染色体ネガティブコントロールは、X rRNAシグナルのみを検出し、検出可能なY rRNAシグナルは検出しないことが期待されます (図3C)。これらのコントロールは、プローブの特異性を判断するためだけでなく、信号の背景やアーチファクトを決定するためにも重要であることに注意してください。このようなコントロールに特異性を提供する新規のプローブまたはアッセイ条件は、遺伝子座特異的なrDNA転写を検出するために正確に使用できます。
遺伝的、発生的、または環境条件が異なると、rDNAがY染色体rDNA遺伝子座13,17から排他的に転写される可能性が変わる可能性がある。単一の遺伝子座または複数の遺伝子座からのrDNA転写の頻度を定量化し、サンプル間で直接比較することができます。rDNA遺伝子座の転写の違いを定量的に比較するためには、各細胞をY-dominant、Co-dominant、またはX-dominantに個別に分類する必要があります。細胞がY優性およびX優性として分類されるのは、それぞれのシグナルのみが検出された場合のみです。YとXの両方のrRNAシグナルを持つ細胞は、一方のシグナルが他方のシグナルよりもはるかに弱い場合でも、共優性と見なされます。したがって、非常に強いXシグナルとYシグナルを含む細胞は、強いYと弱いXシグナルを持つ細胞、または強いXシグナルと弱いYシグナルを持つ細胞と定性的には同じと見なされます。各カテゴリのすべてのサンプルにわたるすべてのセルの合計パーセンテージは、カイ二乗検定によってサンプル間で比較できます。このアッセイは、特定のrDNA遺伝子座が転写されるかどうかを定性的に判断するのには適していますが、個々のSNP-FISHシグナルの蛍光強度を直接定量する場合、サンプル間で一貫した評価は観察されていません。したがって、サンプル間のFISHシグナル強度や、単一細胞内のXおよびY rDNAシグナルの相対強度を定量的に評価することはお勧めしません。この蛍光強度の不一致の原因は不明ですが、すべての標的rRNAに結合しないマスクプローブの非効率的な結合が原因である可能性があります。
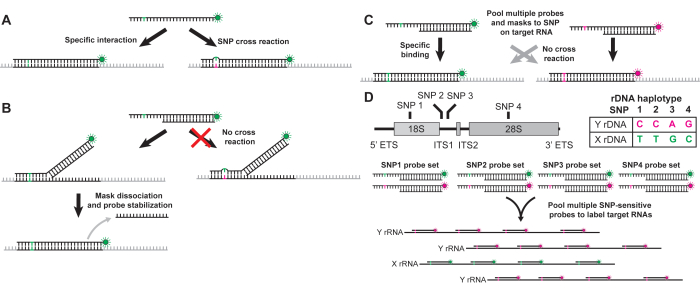
図1:SNP-FISH法の図。 (A)従来のFISHアンチセンスオリゴヌクレオチドプローブは、ヌクレオチドが1つだけ異なる非標的RNAと交差反応します。(B)SNP-FISH法では、オリゴヌクレオチドプローブの3'領域に結合した相補型マスクオリゴヌクレオチドを使用します。マスクは、標的配列に結合するために10ヌクレオチドのみを残します。マスクされていないプローブ領域は、1つ以上のヌクレオチドが異なる非標的配列に安定して結合しません。マスクは、プローブがターゲットに結合した後、プローブから解離するため、プローブとターゲット間の安定した結合が可能になります。(C)共通のマスクと組み合わせた異なる標識ペアのSNPプローブは、交差反応することなく、単一のヌクレオチドで異なるターゲットに特異的に結合します。(D)複数のプローブをプールして、異なるrRNAハプロタイプを特異的に標識することができます。 y1w1 Drosophila melanogaster 株のXとY rDNA遺伝子座の間で4つのSNPの違いが特徴付けられています。18S rRNAに1つのSNP、28S rRNAに1つ、pre-rRNAのITS1部分に2つのSNPがあります。SNP-FISHに用いたXおよびY特異的なrRNAハプロタイプを表示します。4つのrRNA SNPでX rDNAバリアントを標的とするプローブは共通の蛍光色素(緑色)に結合し、Y rDNA変異体を標的とするプローブは異なる蛍光色素(マゼンタ)に結合します。各 SNP に共通のマスクが使用されます (合計 4 つ)。rRNA上の各SNP位置に結合するSNP特異的プローブは、最大4つのFISHオリゴヌクレオチドプローブを使用して、XおよびY rDNA遺伝子座のrRNAを特異的に標識できます。 この図の拡大版を表示するには、ここをクリックしてください。

図2:ホモ接合型およびヘテロ接合型のrDNA SNPシーケンシング結果の例。 例:(A)女性および(B)男性サンプルからのDNAの18S SNPシーケンシングからのクロマトグラムシーケンシング。18S SNP の位置はアスタリスク (*) で示されます。女性サンプルのSNP位置の単一のチミン(T)信号は、X染色体rDNA遺伝子座のSNP位置にチミンがあることを示しています。男性サンプルのSNP位置での二重シグナルは、Y染色体rDNA遺伝子座のSNP位置にあるシトシンを示しています(チミンがX染色体遺伝子座にあることがわかっていることから推測されます)。シーケンシング結果は、プラスミドエディタソフトウェアであるApEを使用して表示しました19。 この図の拡大版を表示するには、ここをクリックしてください。

図3:2つのSNP-FISHプローブセットのみを使用した ショウジョウバエ 精巣でのSNP-FISHの結果の例。 (A)XとY rDNAの両方、(B)Y rDNAのみ、または(C)X rDNAのみを持つ動物の精巣におけるSNP FISHの例。これらの例では、2つのITS1 rRNA SNPを標識するプローブセットのみを使用しており、rRNA SNP FISHはわずか2つの標的SNPで機能することを示しました。生殖細胞は大きな丸い核によって識別され、体細胞は小さくて丸い核によって識別されます。生殖幹細胞は、アスタリスク(*)でマークされた体細胞ハブのすぐ隣の位置によって識別されます。XおよびYの両方のrDNA遺伝子座からrDNAを転写する生殖細胞は、XおよびY核FISHシグナル(白い点線の円、A-A'')によって同定されます。Y rDNA遺伝子座のみからDNAを転写する生殖細胞は、Y核FISHシグナル(黄色の点線)のみを持っています。Yのみを発現する体細胞は赤い矢印でマークされています。XおよびY rDNA遺伝子座の人工的共標識は、体細胞ハブ(黄色の矢印)で観察できます。スケールバーは10μmです。 この図の拡大版を表示するには、ここをクリックしてください。
補足ファイル1:X染色体遺伝子座のrRNA配列。このファイルをダウンロードするには、ここをクリックしてください。
ディスカッション
ここでは、SNP-FISHを用いて、 Drosophila melanogaster 組織のX染色体およびY染色体rDNA遺伝子座に由来する45S rRNA転写産物を鑑別する方法について述べる。このプロトコールで最も重要なステップは、SNP-FISHターゲットとして使用する45S SNPの正確なジェノタイピングです。私たちは、4つの既知の ショウジョウバエ 45S SNPを遺伝子型決定するためのプライマーとプロトコルを提供していますが、他のシーケンシング法では、アッセイに代替的に使用できる新規SNPが明らかになる場合があります。X染色体とY染色体のrDNA遺伝子座の間で同一であることが判明したSNP位置(すなわち、男性DNAサンプルに単一のシーケンシングピークがある)は、SNP FISHには使用できません。一部のSNP位置は、単一のrDNA遺伝子座内で不均一であることが判明する場合があります(つまり、シーケンシング結果では、XXサンプルに対して単一のSNPバリアントが得られなかったり、XYサンプルの2番目のシーケンシングピークが非常に小さいだけであったりします)。単一のrDNA遺伝子座内で不均一性を持つSNPも、バリアントの1つが2つのrDNA遺伝子座間で共有され、単一の遺伝子座または複数の遺伝子座からの転写を区別できなくなるため、このアッセイには適していません。さらに、雌の精子貯蔵20により、交配した雌から単離されたDNAは、Y染色体rDNAを含んでいてもよい。したがって、交配していない雌をXXシーケンシング解析に使用することが重要です。重要なことに、この方法では、少なくとも2つの遺伝子座特異的プローブが各rRNA分子に結合して検出できるようにするために、少なくとも2つの染色体特異的SNPが必要であり、その適用は、それらの間に少なくとも2つの識別SNPがあるrDNA遺伝子座に限定されます。より多くのSNPが利用できるため、より多くのプローブが各rRNAに結合でき、シグナルの有効性を高めることができます。興味深いことに、この方法では、細胞質に輸出される18Sおよび28SのrRNAのSNPを標的とする2つのプローブペアにもかかわらず、核小体内のrRNAのみを標識します(2つのITS1標的部位を含むrRNAは核小体にのみ存在します)。細胞質のFISHシグナルの欠如は、2つの非排他的な可能性を示唆しています:1)18Sおよび28Sプローブだけでは細胞質rRNAを検出するには不十分であり、これはおそらくrRNAが核小体よりも細胞質全体に分散しているため、または2)成熟リボソームサブユニットに統合されたrRNAへのプローブ結合効率は、未処理の45S rRNAよりも低いです。18S および 28S rRNA 内に SNP を追加することで、細胞質 rRNA の SNP 感受性 FISH 検出が可能になる場合があります。
遺伝子座特異的なrRNA転写を評価する他の方法には、特定の遺伝子座6に割り当てられたrRNAバリアントの発現を区別するrRNAシーケンシングアプローチが含まれる。同様に、qPCRは、独自の検出を可能にするのに十分なほど明確なrRNAバリアントの発現を鑑別的に評価できます5,21が、これらのバリアントのサイレンシングがrDNA遺伝子座全体のサイレンシングを表しているかどうかは不明です。これらの方法は、特定のrRNAバリアントの発現を定量するのに効果的ですが、シングルセルの分解能では実行できません。ショウジョウバエ組織におけるX染色体rDNAサイレンシングの不均一性は、rDNA遺伝子座転写17に強い細胞間変動があることを示唆しており、この変動性を説明できる技術の必要性を強調しています。H3.3ヒストンバリアント10やPol I転写因子UBF22など、rDNA遺伝子座での活性転写に関連するイメージング機能を利用して、遺伝子座特異的なrRNA転写をシングルセルの分解能で同定しています。しかし、これらの方法では、特定のrDNA遺伝子座で発生する転写を区別するために、有糸分裂細胞の凝縮した染色体が必要です。ショウジョウバエ細胞では、X染色体およびY染色体のrDNA遺伝子座は染色体形状10によって同定され得るが、ヒト細胞における転写活性rDNA遺伝子座の同定には、染色体特異的標識との共染色も必要である22。SNP-FISH法は、染色体特異的な共標識や転写マーカーの標識を必要とせず、任意の細胞周期段階または有糸分裂後の細胞で評価できるため、多様な組織や実験条件での使用に柔軟性があります。
このrRNA SNP-FISH法は、 ショウジョウバエ のrRNAを区別する他のSNPと併用するために改変することも、他の生物のrDNA遺伝子座を区別するSNPにも適用できる可能性があります。この方法を2つ以上のrDNA遺伝子座を持つ生物に適用するには、遺伝子座が、他のrDNA遺伝子座には存在せず、その遺伝子座のすべての45Sコピー間で共有される少なくとも2つのSNPを含む固有のrDNAバリアントハプロタイプを持つ必要があります。この要件は、この分析法が特定の1つの遺伝子座からの転写のみを他の遺伝子座と比較して特定できることを意味します(つまり、遺伝子座1のrRNA SNPと遺伝子座2および3が共有するSNPを比較します)。しかし、各rDNA遺伝子座が適合するハプロタイプを持っている場合、複数の実験を組み合わせて、各遺伝子座の転写を一度に1つずつ個別に評価することができます。このプロトコルで使用されるハイブリダイゼーション温度(37°C)の比較的低いストリンジェンシーは、特にプローブの短い、マスクされていない部分を考えると、強力なプローブ結合を可能にするが、より高い温度(50-75°C)でのハイブリダイゼーションは、いくつかのFISHプローブ23のシグナルを増強することが示されている。ハイブリダイゼーション温度が高いと、マスク鎖の変位が増大し、プローブの結合が増加する可能性がありますが、温度が高すぎるとプローブとマスクの結合が不安定になり、SNP特異性が失われる可能性があります。このため、二本鎖DNAターゲットを溶かしてプローブ結合を可能にするために必要な温度は、プローブマスク結合を不安定にし、SNP感受性ターゲット特異性を排除するため、SNP-FISHがDNAを特異的に標識するとは考えていません。それでも、新しいrRNA SNPプローブのハイブリダイゼーション温度を最適化すると、特にSNP部位が2つしか利用できない場合に、シグナルが増加する可能性があります。X染色体rDNAサイレンシングの喪失は 、ショウジョウバエ13のrDNAコピー数の減少と関連しているため、他のシステムにおける遺伝子座特異的なrRNA転写を特徴付けるSNP-FISHアッセイの開発は、rDNAおよびリボソーム機能の完全性を評価するための有用なツールとして役立つ可能性があります。さらに、この方法は ショウジョウバエの欠失バリアントと共に使用するために改変されており、この単一の構造的rRNAバリアントは検出に適していた(おそらくプローブマスクなしでより大きな標的結合親和性のため)17。構造的なrRNAバリアントをSNPバリアントと組み合わせられる可能性があることで、他のシステムへの応用の可能性が広がります。rDNA遺伝子座の転写を制御するメカニズムは依然としてほとんど不明であるため、rRNA SNP-FISHの柔軟性と他のシステムへの適応性は、rRNA転写を研究するための将来の研究のための強力なツールとなります。
開示事項
著者には、開示すべき利益相反はありません。
謝辞
ブルーミントン・ショウジョウバエ・ストック・センター、京都ショウジョウバエ・ストック・センター、FlyBaseのリソースに感謝します。この研究は、ストーニーブルック大学生化学・細胞生物学部とルネッサンス医学部(JON)のスタートアップ資金によって支援されました。
資料
| Name | Company | Catalog Number | Comments |
| PTC Tempo 96 Thermal Cycler | Bio Rad | 12015382 | Any thermal cycler can be used |
| 0.2 mL 8 strip flatcap PCR tubes | VWR | 89133-912 | Any compatable tubes can be used |
| 1 kb DNA ladder | NEB | N3232S | To use when checking sequencing PCR amplicon size by gel electrophoresis |
| 1.5 mL graduated microcentrifuge tubes | USA Scientific | 1615-5510 | Any certified RNase free tube will do |
| 2 mM dNTP Mix | ThermoFisher Scientific | R0241 | |
| 50x TAE Buffer | Bio Rad | 1610743 | Used for gel electrophoresis. Any TAE buffer can be used. |
| 5M NaCl | Any NaCl can be used or prepared from any source | ||
| Agarose | VWR | 97064-250 | Any Agarose can be used |
| ApE - A plasmid Editor software | N/A | N/A | https://jorgensen.biology.utah.edu/wayned/ape/ |
| Clear nail polish | Any nail polish from any retailer can be used | ||
| Cover glass, No. 1 Thickness | Thomas Scientific | 6672A38 | |
| Deionized Formamide | Fisher Scientific | NC9569627 | |
| Dextran Sulfate Sodium | Sigma Aldrich | D8906-10G | |
| DreamTaq Green PCR Master Mix | ThermoFisher Scientific | K1081 | |
| Dumont #5 forceps | Fine Science Tools | 11252-20 | Used for dissecting samples |
| EDTA (0.5M), Ph 8.0 | ThermoFisher Scientific | R1021 | Any comparable product can be used |
| Ethanol | VWR | 89125-172 | Any 200 proof Ethanol can be used. Used for dilution to 70% ethanol for permeabilization and cleaning for Rnase-free conditions |
| Ethidium Bromide | ThermoFisher Scientific | 15585011 | |
| Horizontal Mini Gel Electrophoresis System | Fisher Scientific | 14-955-170 | Any gel electrophoresis system can be used |
| Kimwipes | Fisher Scientific | 06-666 | |
| Micro-Test Staining Dish | Electron Microscopy Sciences | 71564 | Used for dissecting samples |
| Nutating shaker | Sigma Aldrich | BMSB3D1020 | Any nutating shaker can be used |
| Parafilm | USA Scientific | 3023-4526 | |
| Phosphate-Buffered Saline (10X) pH 7.4, RNase-free | Life Technologies | AM9624 | |
| Pierce 16% Formaldehyde (w/v), Methanol-free | Life Technologies | 28908 | |
| Precision General Purpose Water Bath | Life Technologies | TSGP02 | Any water bath can be used |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | Any gel extraction kit can be used |
| Recombinant Proteinase K Solution (20 mg/mL) | Invitrogen | AM2546 | Any comparable product can be used |
| Rnase-free UltraPure BSA | ThermoFisher Scientific | AM2618 | |
| S. cerevisiae tRNA | Sigma Aldrich | R8759 | |
| SSC (20X), RNase-free | Fisher Scientific | AM9763 | |
| Triton-X 100 | Life Technologies | A16046.AE | |
| UltaPure 1M Tris-HCl Buffer, pH 7.5 | ThermoFisher Scientific | 15567027 | Any comparable product can be used |
| UltraPure DNase/RNase-Free Distilled Water | Life Technologies | 10977023 | |
| Vanadyl ribonucleoside complex | NEB | S1402S | |
| VECTASHIELD mounting media with DAPI | Vector Laboratories | H-1200-10 | |
| VWR Superfrost Microscope Slide | VWR | 48311-601 | |
| y[1]w[1] Drosophila melanogaster line | Bloomington Drosophila Stock Center | 1495 | |
| Zeiss LSM 980 confocal microscope | Zeiss microscopy | Any confocal microscope with compatable emission and detection can be used | |
| Zeiss Stemi 2000-C Stereo Microscope and light source | Microscope Central | 455053 | Any steromicroscope can be used |
参考文献
- Baßler, J., Hurt, E. Eurokaryotic ribosome assembly. Ann Rev Biochem. 88 (1), 1-26 (2018).
- Hall, A. N., Morton, E., Queitsch, C. First discovered, long out of sight, finally visible: ribosomal DNA. Trend Genet. 38 (6), 587-597 (2022).
- Srivastava, R., Srivastava, R., Ahn, S. H. The epigenetic pathways to ribosomal DNA silencing. Microbiol Mol Biol Rev. 80 (3), 545-563 (2016).
- Kuo, B. A., Gonzalez, I. L., Gillespie, D. A., Sylvester, J. E. Human ribosomal RNA variants from a single individual and their expression in different tissues. Nucl Acid Res. 24 (23), 4817-4824 (1996).
- Tseng, H., et al. Mouse ribosomal RNA genes contain multiple differentially regulated variants. PLoS ONE. 3 (3), e1843(2008).
- Locati, M. D., et al. Expression of distinct maternal and somatic 5.8S, 18S, and 28S rRNA types during zebrafish development. RNA. 23 (8), 1188-1199 (2017).
- Roussel, P., André, C., Comai, L., Hernandez-Verdun, D. The rDNA transcription machinery is assembled during mitosis in active NORs and absent in inactive NORs. J Cell Biol. 133 (2), 235-246 (1996).
- Pontes, O., et al. Postembryonic establishment of megabase-scale gene silencing in nucleolar dominance. PLoS ONE. 2 (11), e1157(2007).
- Earley, K. W., et al. Mechanisms of HDA6-mediated rRNA gene silencing: suppression of intergenic Pol II transcription and differential effects on maintenance versus siRNA-directed cytosine methylation. Gene Dev. 24 (11), 1119-1132 (2010).
- Greil, F., Ahmad, K. Nucleolar dominance of the Y chromosome in Drosophila melanogaster. Genetics. 191 (4), 1119-1128 (2012).
- Preuss, S., Pikaard, C. S. rRNA gene silencing and nucleolar dominance: Insights into a chromosome-scale epigenetic on/off switch. Biochim Biophys Acta. 1769 (5-6), 383-392 (2007).
- Sharifi, S., Bierhoff, H. Regulation of RNA pPolymerase I transcription in development, disease, and aging. Ann Rev Biochem. 87 (1), 1-23 (2018).
- Lu, K. L., Nelson, J. O., Watase, G. J., Warsinger-Pepe, N., Yamashita, Y. M. Transgenerational dynamics of rDNA copy number in Drosophila male germline stem cells. eLife. 7, e32421(2018).
- Nurk, S., et al. The complete sequence of a human genome. Science. 376 (6588), 44-53 (2022).
- Ritossa, F. M. Unstable redundancy of genes for ribosomal RNA. Proc Natl Acad Sci. 60 (2), 509-516 (1968).
- Nelson, J. O., Slicko, A., Yamashita, Y. M. The retrotransposon R2 maintains Drosophila ribosomal DNA repeats. Proc Natl Acad Sci. 120 (23), e2221613120(2023).
- Warsinger-Pepe, N., Li, D., Yamashita, Y. M. Regulation of nucleolar dominance in Drosophila melanogaster. Genetics. 214 (4), (2020).
- Levesque, M. J., Ginart, P., Wei, Y., Raj, A. Visualizing SNVs to quantify allele-specific expression in single cells. Nat Meth. 10 (9), 865-867 (2013).
- Davis, M. W., Jorgensen, E. M. ApE, a plasmid editor: A freely available DNA manipulation and visualization program. Front Bioinfo. 2, 818619(2022).
- Qazi, M. C. B., Heifetz, Y., Wolfner, M. F. The developments between gametogenesis and fertilization: ovulation and female sperm storage in Drosophila melanogaster. Dev Biol. 256 (2), 195-211 (2003).
- Rogers, M. J., et al. Structural features of the large subunit rRNA expressed in Plasmodium falciparum sporozoites that distinguish it from the asexually expressed subunit rRNA. RNA. 2 (2), 134-145 (1996).
- Potapova, T., et al. Epigenetic control and inheritance of rDNA arrays. bioRxiv. , (2024).
- Tang, Y. Z., Gin, K. Y. H., Lim, T. H. High-temperature fluorescent in situ hybridization for detecting Escherichia coli in seawater samples, using rRNA-targeted oligonucleotide probes and flow cytometry. Appl Environ Microbiol. 71 (12), 8157-8164 (2005).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved