Method Article
FISH sensible au polymorphisme mononucléotidique Détection de la transcription de l’ARN ribosomique spécifique du locus chez Drosophila melanogaster
Dans cet article
Résumé
Le protocole décrit la méthode d’hybridation par fluorescence in situ sensible aux polymorphismes nucléotidiques uniques (SNP-FISH) pour distinguer les transcrits d’ARN ribosomique dérivés du locus de l’ADN ribosomique du chromosome X ou Y chez Drosophila melanogaster.
Résumé
Afin de garantir la transcription d’un nombre suffisant d’ARN ribosomique (ARNr) pour la fonction des ribosomes, les génomes contiennent des centaines de duplications en tandem des séquences qui codent pour les ARNr, composant des régions appelées loci d’ADN ribosomique (ADNr). Les génomes de nombreux organismes contiennent plus d’un locus d’ADNr répartis sur différents chromosomes, et l’ARNr peut être transcrit à partir de plusieurs locus d’ADNr ou d’un seul locus. Les changements dans les sources de transcription de l’ARNr indiquent souvent une détresse ribosomique. Cependant, l’homogénéité de l’ARNr empêche de distinguer si l’ARNr est transcrit à partir de plusieurs locus d’ADNr ou d’un seul locus d’ADNr. Ici, nous décrivons une méthode qui utilise l’hybridation in situ en fluorescence sensible au polymorphisme mononucléotidique (SNP-FISH) pour distinguer la transcription de l’ARNr entre les loci de l’ADNr de Drosophila melanogaster sur les chromosomes X et Y. Cette méthode exploite des variants d’ARNr spécifiques au locus pour permettre une détection facile de la source de transcription de l’ARNr avec une résolution unicellulaire. Ce test peut être appliqué à n’importe quel type de cellule de drosophile et peut être adapté pour être utilisé dans d’autres systèmes.
Introduction
Les ARN ribosomiques (ARNr) 18S, 5.8S et 28S nécessaires à la fonction des ribosomes sont co-transcrits dans un seul cistron appelé pré-ARNr 45S. Les trois ARNr sont séparés au sein du pré-ARNr 45S par deux séquences d’espacement transcrites internes (ITS) et flanqués de séquences d’espacement transcrites externes (ETS) aux extrémités 5' et 3'. Toutes ces séquences d’espacement sont retirées du pré-ARNr 45S pour que les ARNr matures soient incorporés dans les ribosomes (voir1). Afin de répondre à la forte demande de production d’ARNr nécessaire pour soutenir l’activité des ribosomes, tous les génomes eucaryotes contiennent des centaines de copies de la séquence 45S. Ces copies sont regroupées en répétitions en tandem, formant des régions génomiques appelées loci d’ADN ribosomique (ADNr). La plupart des génomes eucaryotes contiennent plusieurs loci d’ADNr répartis sur des chromosomes distincts (voir2). La redondance élevée des copies 45S signifie qu’il y a généralement beaucoup plus de copies 45S que nécessaire pour la transcription, et la majorité des copies 45S sont transcriptionnellement silencieuses et enfouies dans l’hétérochromatine3. L’activité transcriptionnelle n’est pas uniforme entre les loci de l’ADNr, et la variation de la transcription du locus de l’ADNr est largement observée entre les organismes 4,5,6, indiquant que la transcription 45S est régulée de manière différentielle au niveau des locus individuels de l’ADNr. Le silençage de l’ADNr à l’échelle du locus a été observé chez les plantes, les invertébrés et les vertébrés7, 8, 9, 10, ce qui suggère que le silençage de l’ADNr à l’échelle du locus est un mécanisme majeur de régulation de la dose d’ARNr11. La transcription de l’ARNr est une étape régulatrice clé dans la production de ribosomes, et les modifications de la transcription de l’ARNr qui modulent l’activité traductionnelle sont une caractéristique importante de la différenciation normale et des conditions pathologiques12. Les modifications de la transcription du locus de l’ADNr sont également associées à des réductions du nombre total de copies d’ADNr13. Ainsi, une transcription altérée de l’ADNr spécifique du locus peut être un indicateur important d’une physiologie cellulaire perturbée.
Bien que le silençage spécifique du locus semble être une source majeure de régulation transcriptionnelle de l’ARNr, les mécanismes qui établissent ce silençage et déterminent quels loci de l’ADNr sont transcriptionnellement actifs sont largement inconnus. L’homogénéité des séquences d’ADNr rend difficile l’évaluation de la transcription individuelle des locus de l’ADNr, ce qui empêche une analyse généralisée de l’activité transcriptionnelle de l’ADNr spécifique au locus. Il peut être possible de surmonter ce défi en tirant parti de la variation structurelle et de la séquence nucléotidique unique entre les copies d’ADNr au sein d’un même génome 4,5,6,13. Certaines de ces variantes peuvent être partagées entre la plupart ou toutes les copies d’un locus individuel, créant ainsi des haplotypes uniques de locus d’ADNr de plusieurs variantes qui distinguent un locus d’ADNr. En effet, la cartographie récente de variants de l’ADNr à des loci spécifiques par le consortium télomère-télomère a révélé des variants d’ADNr partagés spécifiques à un locus dans le génome humain14. De telles cartes de locus d’ADNr peuvent permettre l’identification de la transcription spécifique du locus à partir d’ensembles de données de séquençage ; Cependant, la disponibilité de ces cartes reste actuellement limitée et n’est pas facile à produire. Étant donné que les locus de l’ADNr ne résident que sur les chromosomes sexuels de Drosophila melanogaster (un locus sur chaque chromosome X et Y)15, le locus de l’ADNr du chromosome Y est absent chez les femelles XX. Cet isolement naturel du locus du chromosome Y signifie que les variants du polymorphisme nucléotidique unique (SNP) entre les loci X et Y de l’ADNr peuvent être facilement identifiés en séquençage à lecture courte comme n’importe quel variant présent chez les mâles (XY) mais absent chez les femelles (XX). Les SNP précédemment identifiés peuvent être rapidement testés dans des souches de drosophile non génotypées par simple PCR et séquençage de Sanger de l’ADN des mâles et des femelles. De plus, la disponibilité de souches de drosophile avec un chromosome X qui n’a pas de locus d’ADNr16 signifie que les loci individuels de l’ADNr X et Y peuvent être isolés individuellement pour une caractérisation encore plus précise des SNP de l’ADNr. Cette identification facile des variants SNP entre les loci de l’ADNr de la drosophile permet de déterminer les haplotypes uniques de SNP13 des locus X et Y de l’ADNr. Les loci de l’ADNr du chromosome X sont également généralement complètement silencieux chez les mâles drosophiles, mais les deux loci du chromosome X sont transcrits chez les femelles10,17, ce qui fait de la drosophile un système utile pour étudier le silençage de l’ADNr à l’échelle du locus.
L’hybridation in situ en fluorescence (FISH) est un outil puissant pour visualiser la présence de séquences d’ARN ou d’ADN spécifiques dans les cellules individuelles d’un tissu. Les marquages FISH ciblent des séquences d’ARN ou d’ADN grâce à des sondes oligonucléotidiques antisens conjuguées à des fluorophores. Les sondes FISH doivent généralement avoir une longueur de ~20 nucléotides afin de fournir une liaison cible suffisamment stable pour être visualisées, mais des sondes aussi longues peuvent également se lier facilement à des séquences non cibles qui ne diffèrent que d’un seul nucléotide (Figure 1A). À l’inverse, les sondes suffisamment courtes pour conférer une spécificité mononucléotidique ne se lient pas de manière suffisamment stable pour la visualisation. Pour équilibrer spécificité et stabilité, FISH-SNP-SENSITIVE (SNP-FISH) combine une sonde fluorescente antisens de ~26 nucléotides de longueur avec un oligonucléotide de masque de détection non fluorescent qui se lie à tous sauf à l’extrémité 5' de la sonde18. Ce masque ne laisse que les 10 nucléotides les plus 5' de la sonde monocaténaires et disponibles pour la liaison à la cible (Figure 1B). Cette courte partie monocaténaire de la sonde confère une spécificité à la cible, mais empêche la réactivité croisée avec des ARN qui présentent ne serait-ce qu’un seul décalage avec ces 10 nucléotides (Figure 1B). Une fois que l’extrémité 5' de la sonde se lie à sa cible, le déplacement passif des brins enlève le masque de la sonde, ce qui permet à la région 3' de se lier à la cible et de créer un étiquetage stable de la cible (Figure 1B)18. Par conséquent, SNP-FISH peut visualiser différemment les ARN qui diffèrent par un seul SNP en utilisant une paire de sondes qui ont des fluorophores différents qui complètent chacun un SNP différent mais partagent un masque commun (Figure 1C). Bien que cette méthode ne recrute qu’une seule sonde conjuguée au fluorophore sur le SNP cible, le regroupement de plusieurs ensembles de masques de sonde sur plusieurs SNP au sein d’un haplotype d’ADNr partagé peut être utilisé pour amplifier la détection sensible au SNP d’un seul ARNr (Figure 1D). De plus, en raison de l’abondance de transcription de l’ARNr, les transcrits de l’ARNr peuvent être facilement visualisés dans les expériences FISH en utilisant un petit nombre de marqueurs fluorescents par ARN. Ce faible besoin de fluorophore par ARN signifie que SNP-FISH peut visualiser les ARNr d’un locus spécifique avec seulement quelques SNP uniques. Cette méthode permet de détecter facilement la transcription de l’ARNr spécifique du locus dans une variété de tissus et de stades de développement de la drosophile 17. Il est particulièrement efficace dans les cellules souches germinales mâles de la drosophile, qui alternent entre la transcription exclusive de l’ADNr et la co-expression des loci de l’ADNr des chromosomes X et Y au cours du vieillissement13. Ici, nous fournissons un protocole SNP-FISH pour visualiser des transcrits distincts d’ARNr X et Y dans le testicule de la drosophile afin d’atteindre l’objectif global d’évaluer le silençage de l’ARNr spécifique au locus. Cette méthode utilise quatre SNP préalablement caractérisés entre les loci de l’ADNr sur les chromosomes X et Y de la souche commune de drosophile de laboratoire y1w1 (Figure 1D).
Protocole
1. Préparation des tampons et des réactifs
REMARQUE : La technique sans RNase doit être utilisée tout au long des étapes 1, 3 et 4, et des réactifs certifiés sans RNase doivent être utilisés.
- Dans une hotte chimique, préparez 40 ml de formaldéhyde à 4 % dans 1 solution de fixation PBS en ajoutant 10 ml de formaldéhyde à 16 % et 4 ml de PBS sans RNase à 26 ml d’eau sans RNase. Répartir dans des aliquotes de 1 mL et conserver à -20 °C dans les 30 minutes suivant la préparation. La solution de fixation peut être conservée à -20 °C jusqu’à 6 mois.
ATTENTION : La solution contient du formaldéhyde. Manipulez-le avec des gants et un EPI approprié et jetez-le en toute sécurité. - Préparez 10 mL de tampon d’hybridation en mélangeant 1 mL de 20 citrate salin et sodique (SSC) sans RNase, 1 g de sulfate de dextran, 100 μL d’ARNt de Saccharomyces cerevisiae , 100 μL de 200 mM de complexe de blastonucléoside Vanadyl, 1 mL de BSA sans RNase à 5 %, 1 mL de formamide désionisé combiné à 6 mL d’eau sans RNase. Distribuer dans des aliquotes de 1 mL dans des tubes de microfuge de 1,5 mL et conserver à -20 °C jusqu’à 1 mois.
- Préparez 50 ml de tampon de lavage en ajoutant 5 ml de SSC 20x sans RNase, 5 ml de formamide désionisé, 50 μL de Triton-X à 40 ml d’eau sans RNase. Conserver à température ambiante dans l’obscurité jusqu’à 1 mois.
- Préparez 10 ml de tampon d’isolement de l’ADN en ajoutant 100 μL de 1M de Tris-HCl pH 7,5-8,0, 20 μL d’EDTA 0,5M et 50 μL de 5M NaCl à 9,8 mL d’eau.
2. Génotypage de l’échantillon pour les SNP à ARNr spécifiques de l’X et de l’Y
- Prélever des individus homozygotes non accouplés, femelles et mâles porteurs du même chromosome X dans des tubes de 0,2 mL et les refroidir sur de la glace.
- Combinez 50 μL de tampon d’isolement d’ADN avec 0,5 μL de 20 mg/mL de protéinase K par échantillon.
- Ajouter 50 μL de tampon d’isolement d’ADN / protéinase K à l’échantillon de drosophile et homogénéiser l’échantillon à l’aide d’une pointe de pipette.
- Incuber les échantillons à 37 °C pendant 30 minutes, puis à 95 °C pendant 2 minutes. Transférez 40 ml d’échantillon (évitez les débris d’animaux) dans un tube propre de 1,5 ml.
- Amplifier séparément chaque région cible SNP de chaque échantillon d’ADN par PCR en utilisant une concentration de 10 μM des paires d’amorces suivantes : SNP 18S avant + inverse ; ITS1 SNP Marche avant + marche arrière ; 28S SNP avant + arrière. La séquence d’amorces et la taille attendue de l’amplicon de la PCR sont indiquées dans le tableau 1.
- Utilisez l’électrophorèse sur gel pour séparer l’ensemble de l’échantillon de PCR sur un gel d’agarose à 1 % 1x TAE afin de confirmer le produit de PCR attendu. Les tailles de produit prévues sont indiquées dans le tableau 1.
- Isolez le produit PCR du gel à l’aide de n’importe quel kit d’extraction de gel disponible dans le commerce.
- Séquencez les produits de PCR par séquençage de Sanger. Séquencez les échantillons dans les deux sens en utilisant des réactions individuelles distinctes pour l’amorce F et R pour chaque cible.
- Déterminer la variante SNP du chromosome X aux positions du tableau 2 dans un échantillon d’ADN féminin non accouplé. L’haplotype X attendu est décrit dans le tableau 2.
- Observez les positions SNP dans le chromatogramme de séquençage d’échantillon d’ADN masculin. Déduire la variante SNP du chromosome Y en fonction de la variante SNP X déjà déterminée à chaque position SNP. L’haplotype Y attendu est décrit dans le tableau 2.
| Nom de base | Séquence | Taille d’amplicon attendue |
| 18S SNP F | GACTACCATGGTTGCAACGG | 652 pb |
| 18S SNP R | TTCACCTCTCGCGTCGTAAT | |
| ITS1 SNP F | CTTGCGTGTTACGGTTGTTTC | 955 pb |
| ITS1 SNP R | ACAGCATGGACTGCGATATG | |
| 28S SNP F | ATGCGTAGAAGTGTTTGGCG | 598 pb |
| 28S SNP R | GCCGACTTCCCTTACCTACA |
Tableau 1 : Séquences d’amorces pour le séquençage des SNP d’ADNr. Les amorces s’apparient pour amplifier les régions d’ADNr qui contiennent des SNP précédemment caractérisés dans les régions d’ADNr 18S, ITS1 et 28S. La séquence d’oligonucléotides de l’amorce et la taille attendue de l’amplicon de la PCR sont indiquées.
| Haplotype | SNP Position | Séquence | ||
| X ADNr | Années 18 | 1603-ATACTTGTATTTTTTCATATG-1625 | ||
| ITS1-1 | 2873-CGTTAATAAATATTTGTAATT-2895 | |||
| ITS1-2 | 3115-GAAAATCGAAGAAACAAAATT-3137 | |||
| 28S | 5932-AACAAAAATGCCTAACTATATAT-5954 | |||
| ADNr Y | Années 18 | 1603-ATACTTGTATCTTTTCATATG-1624 | ||
| ITS1-1 | 2873-CGTTAATAAACATTTGTAATT-2895 | |||
| ITS1-2 | 3115-GAAAATCGAAAAAACAAAATT-3137 | |||
| 28S | 5932-AACAAAAATGGCTAACTATATAT-5954 | |||
Tableau 2 : Haplotypes d’ADNr attendus. SNP attendus aux loci de l’ADNr des chromosomes X et Y dans la souche de drosophile y1w1 . SNP indiqué en gras et souligné. Position répertoriée sur la base de la séquence consensuelle de l’ARNr 45S (fichier supplémentaire 1).
3. Dissection, fixation et perméabilisation des testicules
- Nettoyez le stéréomicroscope, le poste de travail, la parabole et la pince à dissection avec de l’éthanol à 70 % (ou un autre traitement RNase).
- Sous un stéréomicroscope, disséquez la drosophile pour isoler les testicules dans 1x PBS sans RNAse. Saisissez le milieu de l’abdomen de l’animal avec une paire de pinces et saisissez l’extrémité de l’abdomen avec une autre paire de pinces pour séparer doucement l’animal. Les testicules se dissocient de l’animal et peuvent être séparés à l’aide de pinces. Prélever 30 à 40 testicules par échantillon.
- À l’aide d’une pince, prélevez les testicules et transférez-les de la boîte de dissection dans un tube de microfuge de 1,5 ml contenant 0,5 ml de PBS 1x sans RNase.
- Aspirez le PBS et ajoutez 1 ml de solution de fixation. Incuber à température ambiante sur un agitateur à noix pendant 30 min. Fixateur aspirant.
- Lavez 2 fois avec 1 ml de PBS sans RNase pendant 5 minutes chacun sur un agitateur à nutation. Aspirez 1 fois PBS et ajoutez 1 mL d’éthanol à 70 % (EtOH) sans RNase. Incuber à 4 °C pendant la nuit sur un agitateur à noix.
4. ARN ribosomique sensible aux SNP FISH
- Aspirez l’éthanol et ajoutez 1 mL de tampon de lavage. Incuber à température ambiante pendant 3 min sur un agitateur à nutation, suivi de 2 min de tube posé à la verticale.
- Mélangez des sondes et des masques avec un tampon d’hybridation, ce qui donne 100 μL de volume total par échantillon. Les séquences de sonde, de masque et les étiquettes de fluorophore sont répertoriées dans le tableau 3. Lorsque vous utilisez les 4 SNP, préparez 80 μL de tampon d’hybridation, 1 μL de sonde Y-SNP de 10 μM (4 μL au total), 1 μL de sonde X-SNP de 10 μM (4 μL au total) et 3 μL de masque commun de 10 μM (12 μL au total)
- Aspirer le tampon de lavage, puis ajouter 100 μL de solution d’hybridation. Appliquez un film transparent sur les bords du tube, enveloppez-le dans du papier d’aluminium à l’abri de la lumière et incubez dans un bain-marie à 37 °C pendant au moins 24 h.
- Ajouter 1 mL de tampon de lavage à l’échantillon sans aspirer la solution d’hybridation. Incuber au bain-marie à 37 °C pendant 30 min dans l’obscurité.
- Aspirez le tampon de lavage, puis ajoutez 1 mL de tampon de lavage à l’échantillon. Incuber au bain-marie à 37 °C pendant 30 min dans l’obscurité.
- Aspirez le tampon de lavage et ajoutez 50 μL de support de montage. Les échantillons peuvent être immédiatement montés sur des lames ou stockés jusqu’à 1 semaine à 4 °C avant d’être montés.
| SNP Position | Oligonucléotide | Séquence | Fluorophore conjugué 3' | |
| Années 18 | Sonde SNP X | AAAAAATACAAGTATTTAATCACATA | Alexa 488 | |
| Sonde SNP Y | AAAAGATACAAGTATTTAATCACATA | Alexa 647 | ||
| Masque | TATGTGATTAAATACT | |||
| ITS1-1 | Sonde SNP X | AAATATTTATTAACGGTAAGGATATT | Alexa 488 | |
| Sonde SNP Y | AAATGTTTATTAACGGTAAGGATATT | Alexa 647 | ||
| Masque | AATATCCTTACCGTTA | |||
| ITS1-2 | Sonde SNP X | GTTTCTTCGATTTTCATGTTCGAAAC | Alexa 488 | |
| Sonde SNP Y | GTTTTTTCGATTTTCATGTTCGAAAC | Alexa 647 | ||
| Masque | GTTTCGAACATGAAAA | |||
| 28S | Sonde SNP X | TTAGGCATTTTTGTTTTACTTGAAAA | Alexa 488 | |
| Sonde SNP Y | TTAGCCATTTTTGTTTTACTTGAAAA | Alexa 647 | ||
| Masque | TTTTCAAGTAAAACAA | |||
Tableau 3 : Séquences de sondes et de masques pour l’ADNr SNP-FISH. Les positions du SNP sont mises en évidence en gras. La partie de la sonde qui se lie à l’oligonucléotide du masque est soulignée. Des exemples de fluorophores conjugués 3' appropriés sont répertoriés, mais tous les fluorophores compatibles peuvent être utilisés.
5. Préparation des échantillons pour l’imagerie
- En travaillant sous un stéréomicroscope à dissection, utilisez une pipette à large orifice pour transférer l’échantillon sur une lame de microscope.
- À l’aide d’une pince, disposez les testicules en ligne pour trouver facilement les échantillons lors de l’imagerie (aucune orientation spécifique n’est requise). Recouvrez l’échantillon d’une lamelle. Épongez les bords de la lamelle avec un chiffon pour enlever l’excès de support de montage.
- Scellez les bords de la lamelle avec du vernis à ongles. Laissez glisser dans l’obscurité à température ambiante pendant 10 minutes pour que le vernis à ongles sèche. Les lames peuvent être utilisées immédiatement pour l’imagerie confocale ou être stockées jusqu’à un mois à 4 °C avant l’imagerie.
Résultats
Les résultats de séquençage du génotypage des SNP devraient permettre de détecter des différences SNP entre les loci de l’ADNr X et Y. Ces SNP sont détectés en évaluant directement les positions des SNP dans les chromatogrammes de séquençage (Figure 2). Le séquençage des échantillons féminins devrait avoir un seul signal de séquençage à la position SNP, indiquant l’homozygotie des SNP entre les deux chromosomes X (Figure 2A). Les résultats du séquençage des échantillons mâles devraient présenter un double pic à la position SNP (figure 2B). D’après le génotype du chromosome X déterminé à partir du séquençage de l’échantillon féminin, on déduit que la variante non-X est un SNP d’ADNr du chromosome Y (c’est-à-dire X = T, Y = C pour le séquençage du SNP 18S dans la figure 2). Alternativement, un seul pic de séquençage à une position SNP dans l’échantillon mâle indiquerait une homozygotie entre les loci d’ADNr mâles et femelles à cette position SNP, et cette position ne serait pas utilisable pour l’ARNr SNP-FISH.
Conformément au protocole pour SNP FISH, les échantillons peuvent être visualisés en les collant sur des lames et placés sous une lamelle scellée, suivie d’une microscopie confocale. À l’aide des sondes répertoriées dans le tableau 3, le signal d’ARNr dérivé de l’Y est détecté par l’émission d’Alexa Fluor 647 et le signal d’ARNr dérivé de l’X est détecté par l’émission d’Alexa Fluor 488. On s’attend à ce que des signaux d’ARNr soient observés dans le nucléole, qui peut être identifié comme le trou pauvre en DAPI dans le noyau (Figure 3A). Le nucléole est particulièrement facile à identifier dans les cellules germinales en raison de son grand noyau et de son nucléole (Figure 3A). Le signal faible peut généralement être trouvé dans le cytoplasme (Figure 3), mais il s’agit d’un signal non spécifique qui peut également être détecté dans des échantillons sans ARNr contenant un SNP complémentaire (c’est-à-dire, signal Y dans les cellules dépourvues d’ADNr Y ou signal X dans les cellules dépourvues d’ADNr X ; Figure 3B-C). De plus, un co-marquage artificiel de l’ARNr X et Y peut parfois être détecté dans le moyeux somatique, même dans des échantillons dépourvus de loci d’ADNr X ou Y (Figure 3B-C, flèches jaunes). Ainsi, seuls les signaux nucléaires doivent être utilisés pour évaluer la transcription spécifique au locus. La plupart des cellules, en particulier les cellules somatiques, ne devraient avoir qu’un signal d’ARNr Y, ce qui indique que l’ARNr est exclusivement transcrit à partir du locus de l’ADNr Y (Figure 3A, cercle pointillé jaune et flèche rouge)10,17. Cependant, la co-expression de l’ARNr X et Y est souvent détectée dans les cellules germinales, en particulier les cellules souches germinales13 (Figure 3A, cercles pointillés blancs). Les signaux d’ARNr X et Y dans les cellules co-exprimant peuvent être adjacents et former un seul nucléole (figure 3A, cellule supérieure) ou être séparés, formant deux nucléoles (figure 3A, cellule inférieure). Les exemples de la figure 3 sont tirés de l’utilisation d’ensembles de sondes ciblant seulement deux SNP (les deux SNP ITS1), démontrant que seulement deux SNP sont nécessaires pour l’ADNr SNP-FISH. Cependant, des signaux plus robustes sont observés lors de l’utilisation de sondes ciblant les quatre SNP13.
Les contrôles négatifs sont une inclusion importante pour ce test afin de confirmer que les sondes ne réagissent pas de manière croisée avec la mauvaise cible SNP, en particulier lors du premier dépannage de la méthode. Les contrôles négatifs du chromosome X comprennent toute condition qui n’a pas d’ADNr du chromosome X, comme les hommes porteurs d’un chromosome X avec une délétion de l’ADNr. On s’attend à ce que les témoins négatifs du chromosome X ne détectent que le signal de l’ARNr Y et aucun ARNr X (Figure 3B). Les contrôles négatifs du chromosome Y comprennent toute affection qui n’a pas d’ADNr du chromosome Y. Quelques exemples de ces conditions sont tous les tissus féminins XX, les tissus d’hommes sans chromosome Y ou les hommes hébergeant un chromosome Y avec une délétionde l’ADNr 15. On s’attend à ce que les témoins négatifs du chromosome Y ne détectent que le signal de l’ARNr X et n’aient aucun signal d’ARNr Y détectable (Figure 3C). Notez que ces commandes sont non seulement importantes pour déterminer la spécificité de la sonde, mais aussi pour déterminer l’arrière-plan ou les artefacts du signal. Toute nouvelle sonde ou condition de test qui fournit une spécificité dans de tels contrôles peut être utilisée avec précision pour détecter la transcription de l’ADNr spécifique au locus.
Différentes conditions génétiques, développementales ou environnementales peuvent modifier la probabilité que l’ADNr soit exclusivement transcrit à partir du locus de l’ADNr du chromosome Y13,17. La fréquence de transcription de l’ADNr à partir d’un seul locus ou de plusieurs locus peut être quantifiée et comparée directement entre les échantillons. Afin de comparer quantitativement les différences de transcription du locus de l’ADNr, chaque cellule doit être classée individuellement comme dominante Y, codominante ou dominante X. Les cellules ne sont classées comme dominantes Y et X que si seul ce signal respectif est détecté. Toute cellule avec des signaux d’ARNr Y et X est considérée comme codominante, même si un signal est beaucoup plus faible que l’autre. Ainsi, les cellules contenant des signaux X et Y très forts sont qualitativement considérées comme les mêmes cellules que les cellules avec des signaux Y forts et X faibles ou des signaux X forts et Y faibles. Le pourcentage total de toutes les cellules de tous les échantillons de chaque catégorie peut être comparé entre les échantillons par le test du chi carré. Bien que ce test fonctionne bien pour déterminer qualitativement si un locus d’ADNr particulier est transcrit ou non, nous n’avons pas observé d’évaluations cohérentes entre les échantillons lors de la quantification directe de l’intensité de fluorescence des signaux SNP-FISH individuels. Par conséquent, nous ne recommandons pas d’effectuer des évaluations quantitatives de l’intensité du signal FISH entre les échantillons ou de l’intensité relative des signaux X et Y de l’ADNr au sein d’une seule cellule. La source de cette incohérence dans l’intensité de la fluorescence n’est pas claire, bien qu’elle puisse être due à la liaison inefficace de sondes masquées qui ne se lient pas à tous les ARNr cibles.
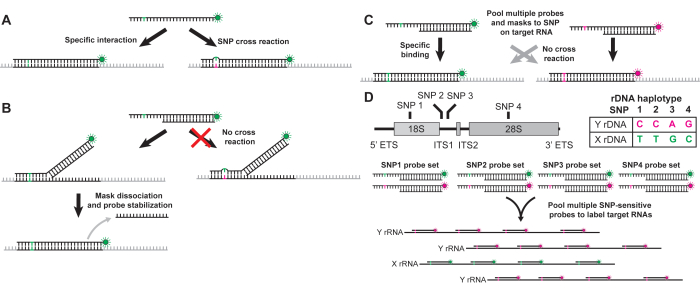
Figure 1 : Schéma de la méthode SNP-FISH. (A) Les sondes oligonucléotidiques antisens FISH traditionnelles réagissent de manière croisée avec des ARN non cibles qui ne diffèrent que par un nucléotide. (B) La méthode SNP-FISH utilise des oligonucléotides de masque complémentaires liés à la région 3' de la sonde oligonucléotidique. Le masque ne laisse que 10 nucléotides libres de se lier à la séquence cible. Une région de sonde non masquée ne se lie pas de manière stable à des séquences non cibles qui diffèrent d’un ou plusieurs nucléotides. Le masque se dissocie de la sonde après que la sonde se lie à la cible, ce qui permet une liaison stable entre la sonde et la cible. (C) Des sondes SNP appariées étiquetées différemment combinées à un masque commun lient spécifiquement des cibles qui diffèrent par un seul nucléotide sans réaction croisée. (D) Plusieurs sondes peuvent être regroupées pour marquer spécifiquement des haplotypes d’ARNr distincts. Quatre différences SNP ont été caractérisées entre les loci de l’ADNr X et Y dans la souche y1w1 de Drosophila melanogaster. Un SNP dans l’ARNr 18S, un dans l’ARNr 28S et deux dans la partie ITS1 du pré-ARNr. Les haplotypes d’ARNr spécifiques X et Y utilisés pour SNP-FISH sont affichés. Les sondes ciblant le variant X de l’ADNr au niveau des quatre SNP de l’ARNr sont conjuguées à un fluorophore commun (vert), et les sondes ciblant les variants de l’ADNr Y sont conjuguées à un fluorophore différent (magenta). Un masque commun est utilisé pour chaque SNP (quatre au total). La liaison d’une sonde spécifique au SNP à chaque position du SNP sur l’ARNr permet de marquer spécifiquement l’ARNr des loci X et Y de l’ADNr avec un maximum de quatre sondes oligonucléotidiques FISH. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Exemples de résultats de séquençage SNP de l’ADNr homozygote et hétérozygote. Exemple de chromatogrammes de séquençage à partir de 18S Séquençage SNP de l’ADN d’échantillons (A) féminins et (B) masculins. Position SNP 18S indiquée par un astérisque (*). Le signal unique de thymine (T) pour la position SNP dans l’échantillon femelle indique une thymine à la position SNP dans le locus de l’ADNr du chromosome X. Le double signal à la position SNP divisé entre thymine et cytosine (C) dans l’échantillon mâle indique une cytosine à la position SNP dans le locus de l’ADNr du chromosome Y (déduit en sachant que la thymine est au locus du chromosome X). Les résultats du séquençage ont été visualisés à l’aide d’ApE, un logiciel d’édition de plasmides19. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 3 : Exemples de résultats SNP-FISH chez la drosophile testicule en utilisant seulement deux ensembles de sondes SNP-FISH. Exemples de SNP FISH dans les testicules d’animaux avec (A) à la fois de l’ADNr X et Y, (B) seulement de l’ADNr Y, ou (C) seulement de l’ADNr X. Ces exemples n’ont utilisé que des ensembles de sondes marquant les deux SNP d’ARNr ITS1, démontrant que le SNP à ARNr FISH fonctionne avec aussi peu que deux SNP cibles. Les cellules germinales sont identifiées par leur grand noyau rond, et les cellules somatiques par leur noyau plus petit et moins rond. Les cellules souches germinales sont identifiées par leur position directement à côté du moyeu somatique, marquée d’un astérisque (*). Les cellules germinales qui transcrivent l’ADNr à partir des loci X et Y de l’ADNr sont identifiées par des signaux nucléaires FISH X et Y (cercles pointillés blancs, A-A''). Les cellules germinales qui transcrivent l’ADN à partir du seul locus de l’ADNr Y n’ont qu’un signal nucléaire Y FISH (cercle pointillé jaune). Les cellules somatiques exprimant uniquement Y sont marquées d’une flèche rouge. Un co-marquage artificiel des loci d’ADNr X et Y peut être observé dans le moyeu somatique (flèche jaune). La barre d’échelle est de 10 μm. Veuillez cliquer ici pour voir une version plus grande de cette figure.
Fichier supplémentaire 1 : Séquence d’ARNr du locus du chromosome X. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Ici, nous décrivons une méthode permettant d’utiliser SNP-FISH pour distinguer les transcrits de l’ARNr 45S dérivés des loci de l’ADNr des chromosomes X et Y dans les tissus de Drosophila melanogaster . L’étape la plus critique de ce protocole est le génotypage précis des SNP 45S à utiliser comme cibles SNP-FISH. Nous fournissons des amorces et un protocole pour génotyper quatre SNP 45S connus de la drosophile , mais d’autres méthodes de séquençage peuvent révéler de nouveaux SNP qui pourraient être utilisés alternativement pour le test. Les positions SNP identiques entre les loci de l’ADNr des chromosomes X et Y (c.-à-d. qu’il y a un seul pic de séquençage dans les échantillons d’ADN mâle) ne peuvent pas être utilisées pour les SNP FISH. Certaines positions SNP peuvent s’avérer hétérogènes au sein d’un seul locus d’ADNr (c’est-à-dire que les résultats de séquençage ne donnent pas une seule variante SNP pour les échantillons XX ou seulement un deuxième pic de séquençage très mineur dans les échantillons XY). Les SNP qui sont hétérogènes au sein d’un seul locus d’ADNr ne conviennent pas non plus à ce test, car l’une des variantes serait partagée entre les deux locus d’ADNr, ce qui rendrait la transcription d’un seul locus ou de plusieurs locus indiscernables. De plus, en raison de la conservation des spermatozoïdesfemelles 20, l’ADN isolé des femelles accouplées peut contenir de l’ADNr du chromosome Y. Ainsi, il est essentiel que des femelles non accouplées soient utilisées pour l’analyse de séquençage XX. Il est important de noter que cette méthode nécessite au moins deux SNP spécifiques aux chromosomes pour permettre à au moins deux sondes spécifiques au locus de se lier à chaque molécule d’ARNr pour la détection, limitant ainsi son application aux loci d’ADNr avec au moins deux SNP distincts entre eux. La disponibilité d’un plus grand nombre de SNP permet à un plus grand nombre de sondes de se lier à chaque ARNr et peut fournir une plus grande efficacité du signal. Il est intéressant de noter que cette méthode ne marque l’ARNr que dans le nucléole, bien que deux paires de sondes ciblent les SNP dans les ARNr 18S et 28S qui sont exportés vers le cytoplasme (l’ARNr contenant les deux sites cibles ITS1 n’existe que dans le nucléole). L’absence de signal FISH cytoplasmique suggère deux possibilités non exclusives : 1) les sondes 18S et 28S seules sont insuffisantes pour détecter l’ARNr cytoplasmique, peut-être parce que l’ARNr est plus dispersé dans le cytoplasme que dans le nucléole, ou 2) l’efficacité de liaison de la sonde est plus faible aux ARNr intégrés dans les sous-unités ribosomiques matures qu’à l’ARNr 45S non traité. Des SNP supplémentaires dans les ARNr 18S et 28S peuvent permettre la détection FISH sensible aux SNP des ARNr cytoplasmiques.
D’autres méthodes pour évaluer la transcription de l’ARNr spécifique au locus comprennent des approches de séquençage de l’ARNr qui différencient l’expression des variants de l’ARNr attribués à desloci 6 spécifiques. De même, la qPCR peut évaluer de manière différentielle l’expression des variants de l’ARNr qui sont suffisamment distincts pour permettre une détection unique 5,21, bien qu’il ne soit pas clair si le silençage de ces variants représente le silençage d’un locus d’ADNr entier. Bien que ces méthodes puissent être efficaces pour quantifier l’expression de variants spécifiques de l’ARNr, elles ne peuvent pas être réalisées avec une résolution unicellulaire. L’hétérogénéité du silençage de l’ADNr du chromosome X dans les tissus de la drosophile suggère qu’il existe une forte variation d’une cellule à l’autre dans la transcription du locus de l’ADNr17 et souligne la nécessité de techniques permettant de tenir compte de cette variabilité. Les caractéristiques d’imagerie associées à la transcription active au niveau des loci de l’ADNr, telles que le variant10 de l’histone H3.3 ou le facteur de transcription Pol I UBF22, ont été utilisées pour identifier la transcription de l’ARNr spécifique du locus avec une résolution unicellulaire. Cependant, ces deux méthodes nécessitent les chromosomes condensés des cellules mitotiques afin de distinguer la transcription se produisant à un locus d’ADNr particulier. Dans les cellules de drosophile, les loci de l’ADNr au niveau des chromosomes X et Y peuvent être identifiés par la forme du chromosome10, mais l’identification des loci de l’ADNr transcriptionnellement actifs dans les cellules humaines nécessite également une co-coloration avec des marqueurs spécifiques des chromosomes22. La méthode SNP-FISH ne nécessite ni co-marquage spécifique au chromosome ni marquage de marqueurs transcriptionnels et peut être évaluée à n’importe quel stade du cycle cellulaire ou cellules post-mitotiques, ce qui offre une flexibilité d’utilisation dans divers tissus et conditions expérimentales.
Cette méthode d’ARNr peut être modifiée pour être utilisée avec d’autres SNP qui distinguent les ARNr de la drosophile ou éventuellement être appliquée aux SNP qui distinguent les loci d’ADNr dans d’autres organismes. L’application de cette méthode à des organismes ayant plus de deux locus d’ADNr nécessitera qu’un locus ait un haplotype unique de variante d’ADNr contenant au moins deux SNP qui ne sont présents sur aucun autre locus d’ADNr et qui sont partagés entre toutes les copies 45S de ce locus. Cette exigence signifie que la méthode ne pourra spécifier la transcription qu’à partir d’un locus spécifique par rapport à tous les autres (c’est-à-dire les SNP d’ARNr du locus 1 par rapport aux SNP partagés par les locus 2 et 3). Cependant, si chaque locus d’ADNr a un haplotype compatible, plusieurs expériences pourraient être combinées pour évaluer individuellement la transcription de chaque locus un par un. La stricteur relativement faible de la température d’hybridation (37 °C) utilisée dans ce protocole permet une forte liaison de la sonde, en particulier compte tenu de la partie courte et non masquée de la sonde, bien qu’il ait été démontré que l’hybridation à des températures plus élevées (50-75 °C) améliore le signal de certaines sondes FISH23. Des températures d’hybridation plus élevées peuvent améliorer le déplacement des brins du masque et augmenter la liaison de la sonde, mais des températures trop élevées peuvent déstabiliser la liaison sonde-masque et perdre la spécificité SNP. Pour cette raison, nous ne nous attendons pas à ce que SNP-FISH marque spécifiquement l’ADN, car les températures nécessaires pour faire fondre les cibles d’ADN double brin afin de permettre la liaison de la sonde déstabiliseraient également la liaison du masque de sonde et élimineraient la spécificité des cibles sensibles aux SNP. Néanmoins, l’optimisation de la température d’hybridation pour les nouvelles sondes SNP d’ARNr peut augmenter le signal, en particulier lorsque seulement deux sites SNP sont disponibles. La perte de silençage de l’ADNr du chromosome X est associée à la réduction du nombre de copies d’ADNr chez la drosophile13, de sorte que la mise au point de tests SNP-FISH pour caractériser la transcription de l’ARNr spécifique au locus dans d’autres systèmes peut servir d’outil utile pour évaluer l’intégrité de la fonction de l’ADNr et du ribosome. De plus, cette méthode a été modifiée pour être utilisée avec un variant de délétion chez la drosophile, et ce variant structurel unique de l’ARNr était adapté à la détection (peut-être en raison d’une plus grande affinité de liaison à la cible sans masque de sonde)17. L’utilisation de variants structurels de l’ARNr potentiellement combinés avec des variants SNP élargit le potentiel d’application dans d’autres systèmes. Étant donné que les mécanismes qui régulent la transcription du locus de l’ADNr restent largement incertains, la flexibilité et l’adaptabilité potentielle de l’ARNr SNP-FISH à d’autres systèmes en font un outil puissant pour les études futures visant à étudier la transcription de l’ARNr.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Nous remercions le Bloomington Drosophila Stock Center, le Kyoto Drosophila Stock Center et FlyBase pour leurs ressources. Ces travaux ont été soutenus par des fonds de démarrage fournis par le Département de biochimie et de biologie cellulaire de l’Université Stony Brook et la Renaissance School of Medicine (JON).
matériels
| Name | Company | Catalog Number | Comments |
| PTC Tempo 96 Thermal Cycler | Bio Rad | 12015382 | Any thermal cycler can be used |
| 0.2 mL 8 strip flatcap PCR tubes | VWR | 89133-912 | Any compatable tubes can be used |
| 1 kb DNA ladder | NEB | N3232S | To use when checking sequencing PCR amplicon size by gel electrophoresis |
| 1.5 mL graduated microcentrifuge tubes | USA Scientific | 1615-5510 | Any certified RNase free tube will do |
| 2 mM dNTP Mix | ThermoFisher Scientific | R0241 | |
| 50x TAE Buffer | Bio Rad | 1610743 | Used for gel electrophoresis. Any TAE buffer can be used. |
| 5M NaCl | Any NaCl can be used or prepared from any source | ||
| Agarose | VWR | 97064-250 | Any Agarose can be used |
| ApE - A plasmid Editor software | N/A | N/A | https://jorgensen.biology.utah.edu/wayned/ape/ |
| Clear nail polish | Any nail polish from any retailer can be used | ||
| Cover glass, No. 1 Thickness | Thomas Scientific | 6672A38 | |
| Deionized Formamide | Fisher Scientific | NC9569627 | |
| Dextran Sulfate Sodium | Sigma Aldrich | D8906-10G | |
| DreamTaq Green PCR Master Mix | ThermoFisher Scientific | K1081 | |
| Dumont #5 forceps | Fine Science Tools | 11252-20 | Used for dissecting samples |
| EDTA (0.5M), Ph 8.0 | ThermoFisher Scientific | R1021 | Any comparable product can be used |
| Ethanol | VWR | 89125-172 | Any 200 proof Ethanol can be used. Used for dilution to 70% ethanol for permeabilization and cleaning for Rnase-free conditions |
| Ethidium Bromide | ThermoFisher Scientific | 15585011 | |
| Horizontal Mini Gel Electrophoresis System | Fisher Scientific | 14-955-170 | Any gel electrophoresis system can be used |
| Kimwipes | Fisher Scientific | 06-666 | |
| Micro-Test Staining Dish | Electron Microscopy Sciences | 71564 | Used for dissecting samples |
| Nutating shaker | Sigma Aldrich | BMSB3D1020 | Any nutating shaker can be used |
| Parafilm | USA Scientific | 3023-4526 | |
| Phosphate-Buffered Saline (10X) pH 7.4, RNase-free | Life Technologies | AM9624 | |
| Pierce 16% Formaldehyde (w/v), Methanol-free | Life Technologies | 28908 | |
| Precision General Purpose Water Bath | Life Technologies | TSGP02 | Any water bath can be used |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | Any gel extraction kit can be used |
| Recombinant Proteinase K Solution (20 mg/mL) | Invitrogen | AM2546 | Any comparable product can be used |
| Rnase-free UltraPure BSA | ThermoFisher Scientific | AM2618 | |
| S. cerevisiae tRNA | Sigma Aldrich | R8759 | |
| SSC (20X), RNase-free | Fisher Scientific | AM9763 | |
| Triton-X 100 | Life Technologies | A16046.AE | |
| UltaPure 1M Tris-HCl Buffer, pH 7.5 | ThermoFisher Scientific | 15567027 | Any comparable product can be used |
| UltraPure DNase/RNase-Free Distilled Water | Life Technologies | 10977023 | |
| Vanadyl ribonucleoside complex | NEB | S1402S | |
| VECTASHIELD mounting media with DAPI | Vector Laboratories | H-1200-10 | |
| VWR Superfrost Microscope Slide | VWR | 48311-601 | |
| y[1]w[1] Drosophila melanogaster line | Bloomington Drosophila Stock Center | 1495 | |
| Zeiss LSM 980 confocal microscope | Zeiss microscopy | Any confocal microscope with compatable emission and detection can be used | |
| Zeiss Stemi 2000-C Stereo Microscope and light source | Microscope Central | 455053 | Any steromicroscope can be used |
Références
- Baßler, J., Hurt, E. Eurokaryotic ribosome assembly. Ann Rev Biochem. 88 (1), 1-26 (2018).
- Hall, A. N., Morton, E., Queitsch, C. First discovered, long out of sight, finally visible: ribosomal DNA. Trend Genet. 38 (6), 587-597 (2022).
- Srivastava, R., Srivastava, R., Ahn, S. H. The epigenetic pathways to ribosomal DNA silencing. Microbiol Mol Biol Rev. 80 (3), 545-563 (2016).
- Kuo, B. A., Gonzalez, I. L., Gillespie, D. A., Sylvester, J. E. Human ribosomal RNA variants from a single individual and their expression in different tissues. Nucl Acid Res. 24 (23), 4817-4824 (1996).
- Tseng, H., et al. Mouse ribosomal RNA genes contain multiple differentially regulated variants. PLoS ONE. 3 (3), e1843(2008).
- Locati, M. D., et al. Expression of distinct maternal and somatic 5.8S, 18S, and 28S rRNA types during zebrafish development. RNA. 23 (8), 1188-1199 (2017).
- Roussel, P., André, C., Comai, L., Hernandez-Verdun, D. The rDNA transcription machinery is assembled during mitosis in active NORs and absent in inactive NORs. J Cell Biol. 133 (2), 235-246 (1996).
- Pontes, O., et al. Postembryonic establishment of megabase-scale gene silencing in nucleolar dominance. PLoS ONE. 2 (11), e1157(2007).
- Earley, K. W., et al. Mechanisms of HDA6-mediated rRNA gene silencing: suppression of intergenic Pol II transcription and differential effects on maintenance versus siRNA-directed cytosine methylation. Gene Dev. 24 (11), 1119-1132 (2010).
- Greil, F., Ahmad, K. Nucleolar dominance of the Y chromosome in Drosophila melanogaster. Genetics. 191 (4), 1119-1128 (2012).
- Preuss, S., Pikaard, C. S. rRNA gene silencing and nucleolar dominance: Insights into a chromosome-scale epigenetic on/off switch. Biochim Biophys Acta. 1769 (5-6), 383-392 (2007).
- Sharifi, S., Bierhoff, H. Regulation of RNA pPolymerase I transcription in development, disease, and aging. Ann Rev Biochem. 87 (1), 1-23 (2018).
- Lu, K. L., Nelson, J. O., Watase, G. J., Warsinger-Pepe, N., Yamashita, Y. M. Transgenerational dynamics of rDNA copy number in Drosophila male germline stem cells. eLife. 7, e32421(2018).
- Nurk, S., et al. The complete sequence of a human genome. Science. 376 (6588), 44-53 (2022).
- Ritossa, F. M. Unstable redundancy of genes for ribosomal RNA. Proc Natl Acad Sci. 60 (2), 509-516 (1968).
- Nelson, J. O., Slicko, A., Yamashita, Y. M. The retrotransposon R2 maintains Drosophila ribosomal DNA repeats. Proc Natl Acad Sci. 120 (23), e2221613120(2023).
- Warsinger-Pepe, N., Li, D., Yamashita, Y. M. Regulation of nucleolar dominance in Drosophila melanogaster. Genetics. 214 (4), (2020).
- Levesque, M. J., Ginart, P., Wei, Y., Raj, A. Visualizing SNVs to quantify allele-specific expression in single cells. Nat Meth. 10 (9), 865-867 (2013).
- Davis, M. W., Jorgensen, E. M. ApE, a plasmid editor: A freely available DNA manipulation and visualization program. Front Bioinfo. 2, 818619(2022).
- Qazi, M. C. B., Heifetz, Y., Wolfner, M. F. The developments between gametogenesis and fertilization: ovulation and female sperm storage in Drosophila melanogaster. Dev Biol. 256 (2), 195-211 (2003).
- Rogers, M. J., et al. Structural features of the large subunit rRNA expressed in Plasmodium falciparum sporozoites that distinguish it from the asexually expressed subunit rRNA. RNA. 2 (2), 134-145 (1996).
- Potapova, T., et al. Epigenetic control and inheritance of rDNA arrays. bioRxiv. , (2024).
- Tang, Y. Z., Gin, K. Y. H., Lim, T. H. High-temperature fluorescent in situ hybridization for detecting Escherichia coli in seawater samples, using rRNA-targeted oligonucleotide probes and flow cytometry. Appl Environ Microbiol. 71 (12), 8157-8164 (2005).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon