Method Article
ウサギ筋肉からの筋小胞体Ca2+-ATPaseの精製
要約
このプロトコルは、最終遠心分離ステップに二糖類トレハロースを含む改良されたSERCA精製法について説明しています。この炭水化物は、過酷な条件下でタンパク質を安定化させます。精製されたSERCAは触媒活性があり、高純度を示したため、構造的および機能的研究に適していました。
要約
サルコ/小胞体Ca2+-ATPase(SERCA)などの一部のP型ATPアーゼは、本質的に不安定な膜タンパク質であり、高純度で構造品質が高く、触媒活性な形態でそれらを得るためには、精製中に特定の物理化学的条件が必要です。二糖類トレハロースは、酵母の細胞質に高濃度で合成および蓄積され、膜とタンパク質を安定化する適合溶質です。原形質膜H+-ATPaseの精製のためのプロトコルにおける添加剤としてのトレハロースの使用は、高品質の調製をもたらし、そのヘキサマー構造は生化学的分析法によって示される。したがって、トレハロースは、膜タンパク質(P-ATPase)の精製のための安定化添加剤として使用できます。このプロトコルは、トレハロース濃度勾配でSERCAを遠心分離することにより、SERCA精製のための古典的なプロトコルの修正を説明しています。この炭水化物の含有は、高純度の触媒活性形態で、そして重要なことに、安定した形態でのSERCAの精製につながった。精製されたSERCAの部分的な生化学的特性評価(SDS-PAGE、酵素速度論、FITC標識、円二色性分光法)により、この酵素が機能的および構造的研究に適していることが示されました。P型ATPaseおよび他の不安定な膜(および細胞質)タンパク質の精製プロトコルにおけるトレハロースの使用が提案されています。
概要
膜タンパク質/酵素は、さまざまなプロセスで重要な役割を果たすため、細胞の必須の生物学的構成要素です1,2,3。機能の一部には、細胞/内部コンパートメント内外へのイオンおよび分子の輸送(能動的/受動的)、細胞間認識、細胞間結合、固定/付着、および正常および過酷な物理的および化学的条件(高塩分、低水、高温、薬剤耐性、 など)3 したがって、膜タンパク質および/または酵素の三次元(3D)構造の決定は、基礎研究と応用研究の両方にとって非常に重要になっています4,7,6。重要なことに、膜タンパク質/酵素は、創薬の標的として広く使用されてきました(天然または設計によるものか(7,8,9〉。つまり、膜タンパク質は健康において固有の重要性を持っています9,10,11。
膜タンパク質/酵素の疎水性は、実験実験室12,13,14にとって最も技術的に困難な物理化学的特性であり、オリゴマーおよび/または非常に不安定な内在性膜タンパク質15,16を扱う場合はなおさらです。機能的な実験的アッセイおよび構造研究のために、可能な限り高い品質で膜タンパク質/酵素の適切な量の単離が非常に望ましい17。膜タンパク質は、本質的に疎水性であるため、精製が非常に困難であり、界面活性剤の選択は通常、考慮すべき最も重要な問題の1つです17,18,19,20。この点に関して、膜タンパク質を単離するために使用される実験室の方法は、典型的には、タンパク質21の3D構造配置にある程度の損傷を引き起こす。これらの方法には、(a)超音波処理(高周波/エネルギー超音波)、(b)タンパク質可溶化剤(過酷、中程度、または穏やか)22、(c)カラムクロマトグラフィーおよび高速超遠心分離における比較的高圧23、(d)沈殿分子、(e)消化酵素、およびその他21の使用が含まれます。これらすべてのプロセスは、精製中のタンパク質不安定化に寄与するか、または主な原因となる可能性があります21。この点で、一部のプロトコルは、特定の膜タンパク質に対して比較的うまく機能するように思われます。しかし、満足のいく結果を得るためにより高品質の膜タンパク質調製を必要とする新しい最新のアッセイや方法を使用する場合、最適化は常に歓迎される24。最適化ステップには、コンストラクト設計の改善、膜タンパク質発現の最適条件の発見、より良い取り扱い条件(すなわち、pH、温度など)の確立、最適な適合性界面活性剤の発見、超音波処理時間などの精製ステップの調整、遠心分離速度、安定化剤の添加による緩衝液の再配合などが含まれるが、これらに限定されない25,26,27。.したがって、精製された膜タンパク質/酵素の品質(純度)と活性の向上につながる精製方法の変更は重要です。
P型ATPアーゼファミリーでは、酵母原形質膜H+-ATPアーゼは最も不安定なメンバーの1つであるようである28,29。H+-ATPアーゼは、脱水(凍結乾燥)やヒートショックなどによりATP加水分解活性を失う28,29。タンパク質安定剤としてのトレハロースの使用は、酵母K.lactisからの原形質膜H + -ATPaseの単離でテストされました30,31;得られたH+-ATPase調製物は高純度で触媒活性がありました。重要なことに、これにより、酵素のオリゴマー状態が生化学的方法によって分解され、六量体であることが明らかになり、後にクライオ電子顕微鏡31,32,33,34,35によって確認されました。したがって、膜タンパク質の繊細な3次元(3D)配列は、精製中に比較的過酷な条件下で失われる可能性があると考えられる36。二相性不活性化速度は、熱媒介性不活化中にH+-ATPaseで観察されます29。酵母細胞では、他の多くの生物と同様に、二糖類トレハロースは環境ストレス条件下で高濃度で蓄積する37,38,39。トレハロースは、タンパク質(膜埋め込み型および細胞質型の両方)および細胞膜を安定化することにより、膜の完全性および輸送機能を維持する40,41。トレハロースの安定化メカニズムは、いくつかのグループと私たちの研究室42,43,44,45によって広く研究されています。H+-ATPaseおよび他の酵素を用いた実験は、トレハロースが単糖類および二糖類28,29の中で最も効果的なタンパク質安定剤であることを示した。これにより、H+-ATPase精製プロトコル30に組み込まれました。最近、トレハロースは、ウサギの速筋から筋小胞体Ca2+-ATPase(SERCA)の精製にも使用されており、タンパク質の純度と活性において良好な結果が得られています46。したがって、トレハロースは、P型ATPアーゼ、およびおそらく他の膜および細胞質タンパク質の精製のための良好で適切な添加剤であると思われます。
P-ATPaseの場合、構造的な細胞質ドメインの存在は、特に基質/リガンド相互作用の研究において実験的利点である47;ATP結合は、高純度の組換えNドメイン47,48,49で研究されており、したがって、全膜酵素47,48,50、とりわけ51の精製に関する技術的な考慮事項を排除している。残念ながら、一部の機能的(触媒/エネルギー変換)および構造的(サブユニットの配置と他のタンパク質との相互作用)研究では、依然として全体のP-ATPaseが必要です52,53。この点に関して、SERCAの精製は、いくつかの研究グループ54,55,56,57,58によって達成されている。しかしながら、改良は依然として実施することができ、例えば、精製されたATPアーゼ59の無傷性を増大させ、タンパク質複合体のタンパク質変性/破壊25を回避し、膜タンパク質の変性を伴わない可溶化を増加させる(すなわち、高分子凝集体の形成を避ける)60、アッセイおよび他の下流の分析方法61とのより良い適合性をもたらすさらに、新しい実験戦略、添加物、酵素阻害剤などが科学文献61,62,63,64,65,66に現れると、それらは時々、全体のP-ATPaseで試験される必要がある。この研究では、SERCAの精製と、タンパク質構造とATPase活性を安定化するための添加剤としてのトレハロースの使用に関するプロトコルについて説明します。すなわち、酵素(構造)の品質を向上させることに加えて、トレハロースは、酵素の単離および貯蔵中の酵素の構造および活性の損失を防ぐのに役立ち、これにより生物学的物質を節約し、酵素精製の数を減らすのに役立ちます。
プロトコル
すべての動物処置は、実験実験室での動物の取り扱いに関する国際的および地域的(NORMA Oficial Mexicana NOM-062-ZOO-1999)ガイドライン51,67,68に従って実施されました。筋肉組織は、地元の動物取り扱いユニット(INE/CITES/DGVS-ZOO-E0055-SLP-98)から野生型Oryctolagus cuniculusから得られた46。実験動物管理の専門知識を持つ獣医師が、最初の筋肉解剖と処理を行いました。本試験で使用した試薬および装置の詳細は、材料表に記載されています。
1. 速筋ウサギ筋からの筋小胞体の単離(タイミング、7〜9時間)
注:手順の詳細については、Champeil et al.69を参照してください。
- 野生型 Oryctolagus cuniculusから速筋組織(動物1匹あたり両後肢から~70g)を取得します。
注:解剖する筋肉の事前の視覚的識別が必要です70,71。 - ブレンダーで速筋を100 mMのKClの3つの容量に懸濁し、1分間オンにし、1分間氷冷した休息で1分間、72回3回繰り返します。
- 遠心分離(1500 × g)により、4°Cで5分間、組織破片を除去します。必要に応じて、2回目の遠心分離(4200 × g)を行います。
- 上清を収集し、ティッシュグラインダー(低速で10ストローク、すなわち500〜1,500rpm)を使用して均質化します72,73。ホモジネート(10,000 × g)を4°Cで15分間遠心分離します。
注:均質化する場合は、比較的高速を使用するとシステムとオペレーターに損傷を与える可能性があるため、注意が必要です。 - 上清(筋小胞体を含む)(33,000 × g)を4°Cで120分間遠心分離します。
- ペレットを0.5 Mショ糖40 mLに懸濁し、4°C(12,000 × g)で15分間遠心分離します。上清(0.6 MのKClと0.15 Mのスクロース)を希釈し、懸濁液(34,000 × g)を4°Cで165分間遠心分離します。
- 筋小胞体小胞(SRV)シートを含むペレットを、0.3 Mのスクロース、0.1 MのKCl、および5 mMのTris-HCl、pH 7.0に懸濁します。Lowry assay74 とヒト血清アルブミンをタンパク質標準試料として使用して、タンパク質濃度を測定します。
- SRV(30-32 mg / mL)を1 mLの容量で分注します。.SRVサンプルは、さらなる処理のために-72°Cで保存します。
注:タンパク質収量は、動物あたり100〜110 mgのSRVです。
2. SERCA精製(タイミング、18〜20時間)
注:手順の詳細については、Rivera-Morán et al.46を参照してください。
- 氷冷した75 mMのTris-HCl、pH 7.2、0.6 MのKCl、6 mMのEDTA、1 mMのEGTAおよび0.1%(w / v)デオキシコール酸(DOC)を使用して、SRV懸濁液を2 mgのタンパク質/mLに希釈します。SRV懸濁液を氷上で10分間インキュベートし、穏やかに攪拌します。SRV懸濁液(100,000 × g)を4°Cで60分間遠心分離します。
注:DOCは一滴ずつ追加する必要があります。 - ペレットを<3 mLの25 mMのTris-HCl、pH 7.5、0.3 MのKCl、45%グリセロール(v/v)、および2 mM EDTAに懸濁します。懸濁液を均質化し、最終容量の10mLに調整します。タンパク質濃度を決定し、6.5 mg/mLに調整します。アゾレクチンを5 mg/mLおよび0.85(w/w)Zwittergent 3-14で添加します。
注:アゾレクチンと双性体3-14は一滴ずつ追加する必要があります。 - 懸濁液を均質化し、(100,000 × g)を4°Cで60分間遠心分離します。上清を回収し、2 mMのEGTAで1:2に希釈します(pH 7.2)。
- 懸濁液を、10 mMのTris-HCl、pH 7.0、1 mMのEDTA、0.1%デオキシコール酸、および1 mg/mLのアゾレクチン中の不連続なトレハロース濃度勾配(45、40、35、および30%w/v)に穏やかに注ぎます。遠心分離機(100,000 × g)を4°Cで14時間遠心分離します。チューブの底に形成された透明でわずかに黄色がかったペレットを収集します。
注:トレハロースグラジエントの形成とサンプル懸濁液の添加は、どちらも適切な技術を用いて行わなければなりません。一晩の遠心分離をお勧めします。 - ペレットを少量(<1.5 mL)の25 mMのTris-HCl、pH 7.5、0.3のM KCl、45%グリセロール、および2 mMのEDTAに穏やかに懸濁します。タンパク質濃度を決定します。タンパク質濃度を2 mg / mLに希釈して調整します。懸濁液のアリコート(~50 μL)を採取し、精製したSERCAを-72°Cで使用まで保存します。
- 精製されたSERCA72のSDS-PAGEと、精製の異なるステップで採取したタンパク質サンプルのSDS-PAGEを実行します。ジェルをクマシーブルー72で染色します。
注:懸濁液緩衝液中のグリセロールの代わりに0.6Mトレハロースを使用すると、保存および凍結融解サイクル28,29中の酵素の安定性が向上する。
3. ATPase活性のアッセイ(タイミング、1〜2時間)
- 酵素共役アッセイ:ATPase-ピルビン酸キナーゼ-乳酸デヒドロゲナーゼ
- 反応混合物(1 mL)を調製します:50 mM MOPS(pH 7.0)、1 mM EGTA、80 mM KCl、5 mM MgCl2、3 mM CaCl2、5 mMホスホエノールピルビン酸、250 μM NADH。可変ATP濃度(0.001〜0.25 mM)を含めます。
- ボルテックスにより、反応アッセイを慎重に均質化します。反応アッセイを37°Cで10分間インキュベートし、0.9 U L-乳酸デヒドロゲナーゼ(LDH)と1.5 Uピルビン酸キナーゼ(PK)を加えます。
- 10 μgのSERCAを添加してATPase反応を開始します。
- NADHの形成を決定します。サーモスタット付きセルホルダー付きの分光光度計を使用して、波長340nmの波長(λ)で600秒間、毎秒吸光度強度の経時変化を追跡します。
注:吸光度測定の時間間隔は異なる場合がありますが、ATPase反応の初期直線を描くために、できるだけ多くのデータポイントを記録するようにしてください。
- ATP加水分解の速度を決定する
注:手順の詳細については、Rivera-Morán et al.46を参照してください。- 形成された各曲線の線形部分の勾配値(Δabs/min)を計算します。NADH(λ = 6,220 M-1・cm-1)のモル吸光係数(ε)を使用して、ATPアーゼ活性(μmoles ATP hydrolyzed/min.mg prot.)を決定します。
- 動力学パラメータKmおよびVmaxを決定します
- データ解析およびグラフ作成ソフトウェアを使用して、速度データとATP濃度をプロットします。
- 非線形回帰によってデータをMichaelis-Menten方程式(式1)46に当てはめます。
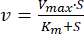
ここで、 v は速度、 Vmax は最大速度、[S] は ATP 濃度、[ Km ] は Michaelis-Menten 定数です。 - Km と Vmax を反復で決定します。
注:DOCを含めると、ATPアーゼ速度の応答が増加する可能性があります。最適な濃度を見つけるために、さまざまな濃度のDOCをテストすることをお勧めします。
4. フルオレセインイソチオシアネート(FITC)によるSERCAの標識アッセイ(タイミング、30〜45分、FITC標識のみ)
注:手順の詳細については、54、75、76を参照してください。
- SETCを1 mMのFITCを含む標識バッファー(100 mMのKCl、5 mM MgCl2、および30 mMのTris-HCl、pH 8.9)の50 μL(最終容量)にSERCA(20 μg)を懸濁します。ボルテックスで混ぜます。
- サンプルを異なる時間(15分、10分、7分、5分、2分)で暗所および室温でインキュベートします。
- ATP(5 mM)を含む1容量の氷冷停止バッファー(480 mMのスクロースと48 mMのMOPS、pH 7.0)を添加して、標識反応を停止します。暗闇の中で氷上で5分間インキュベートします。
- FITCでラベル付けされたSERCAをSDS-PAGE72に提出します。透明ゲルを紫外線(λ = 302 nm)にさらします。結果を写真で記録します。
- ジェルをクマシーブルーで染色します。結果を写真で記録します。
注:暗室でFITC標識を行うと、蛍光シグナルの品質が向上する可能性があります。
5. 円二色性(CD)スペクトル(タイミング、30分)
注意: 手順の詳細については、11,77を参照してください。
- SERCA (3 μM) を 400 μL の 10 mM リン酸バッファー (pH 7.0) に 25 °C で懸濁します。
- 光路長0.1cmのセルにサンプルをロードします。遠紫外スペクトルのλ範囲を190〜260nmに設定します。内部分解能と帯域幅を 1nm に設定します。
- CD信号を50 nm/min、25°Cで記録します。
結果
精製のさまざまな段階におけるSERCAのSDS-PAGE(図1)。Coomassieブルー染色ゲルは、精製プロトコルが進行するにつれて、SERCAタンパク質バンド(見かけの分子量>100 kDa)の濃縮を示しています。SERCAに対応するタンパク質バンドは、トレハロース濃度勾配での遠心分離後、>90%の純度を示します。ATP加水分解速度とATP濃度のプロット(図2A)。Michaelis-Menten ATPase速度論(双曲線パターン)は、精製されたSERCAで観察されます。図2A(図2B)のデータのLineweaver-Burkプロット。プロットでは、SERCAのKm値(10μM)は、x切片(K m-1)から得られており、これは以前の研究25,78,79で見つかったものと同様です。FITCによるSERCAラベリング(図3)。ラベル貼付後、SERCAはSDS-PAGEの対象となります。次に、透明なゲルをUV光にさらして、蛍光SERCA-FITCバンドを露出させます(図3A)。SERCAのFITC標識は、無傷のヌクレオチド結合部位を示しています。これは、SERCAのFITC標識が、リジン(Lys)残基47,54,80とのその後の化学反応に対するFITC結合のためのアミノ酸残基の適切な配置に依存するためです。すなわち、FITCラベリングは、適切に折り畳まれたSERCA47においてのみ行われる。図3Bでは、Coomassie Blue染色されたSERCAタンパク質は、FITCで標識されたものと正確に一致しました(図3B)。SERCAの円二色性(CD)スペクトルを図4Aに示します。SERCAの二次構造含有量は、CDスペクトル27,47,81の解析によって決定される。
特に、SRVには大量のSERCAが含まれています。小胞内のタンパク質の高い割合(73%)はSERCAに対応しています(図1B)。トレハロース濃度勾配での超遠心分離後、SERCAの純度は90%以上に増加しました(図1B)。つまり、このプロセス中に付随するタンパク質のほとんどが除去されました。データ取得には、imageJ (https://imagej.net/ij/)82 と Origin をそれぞれデータ取得と解析 (Gaussian multipeak fit) に使用しました。SERCA調製物は高い構造品質です(図1および 図4)。タンパク質間相互作用アッセイを行うことができ、DCスペクトルの変化によってATP結合部位の滴定をモニターすることができ、ヌクレオチド親和性を計算することができる46。精製されたSERCA(図2)が示す触媒活性は、酵素のATPアーゼ活性(構造)を変化させる可能性のある物理的および化学的条件の試験に使用できます。さらに、小胞内のイオン輸送や、酵素による化学エネルギーからポテンシャルエネルギーへの変換の研究も行うことができます。さらに、天然または合成のいずれであっても、阻害剤試験を実施することができます。これに関連して、ヌクレオチド結合部位の無傷性は、FITC標識によって明確に示されています(図3)。異なる条件下での活性部位の構造および潜在的な相互作用因子の存在に関する実験を行うことができる。

図1:SDS-PAGEおよびデンシトメトリー分析 (A)筋小胞体Ca2+-ATPase(SERCA)のSDS-PAGE。SERCAは、ウサギの速筋から単離された筋小胞(SRV)から精製されました。トレハロース濃度勾配で遠心分離した後、ペレット(純度>90%のSERCAを含む)を回収しました。この図は、Rivera-Morán et al.46から採用されています。(B)(A)の孤立したSRVおよびSERCAレーンの密度測定分析。赤い線は、SRVおよびSERCAレーン解析後にImageJソフトウェアによって生成された実験データ(ベースライン補正付き)セットであり、黒と青の線はマルチピークガウス近似を表しています。結果ははめ込みテーブルとして含まれます。 この図の拡大版を表示するには、ここをクリックしてください。
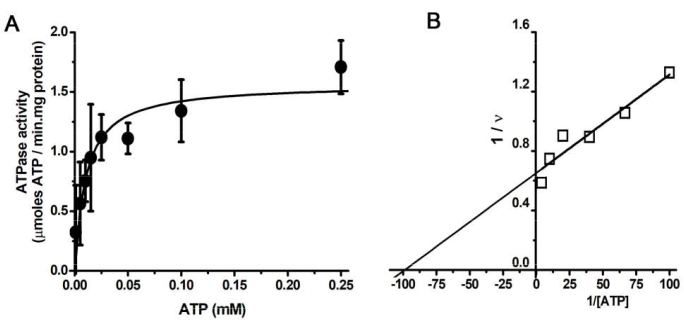
図2:SERCAによるATP加水分解(A)ATPアーゼの速度とATP濃度のプロット。ATP加水分解の速度(波長340nmでのNADH吸光度)を、ピルビン酸キナーゼと乳酸デヒドロゲナーゼからなる酵素共役アッセイで追跡しました。データポイントは、標準偏差±3つの独立した実験の平均です。(B) AのデータのLineweaver-Burkプロット。直線の x 軸切片は、SERCA の K m-1 値です。この図は、Rivera-Morán et al.46から採用されています。この図の拡大版を表示するには、ここをクリックしてください。
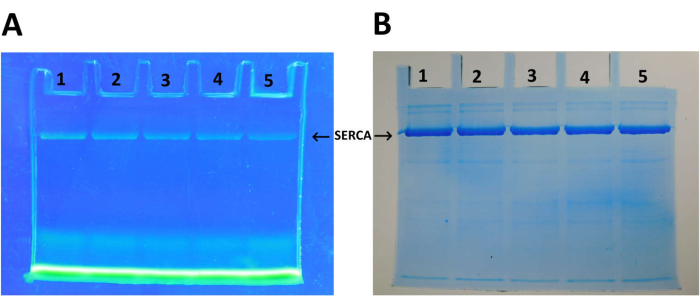
図3:SERCAのFITC標識 (A)インキュベーション時間でFITCで標識したSERCAを、(1)15分、(2)10分、(3)7分、(4)5分、(5)2分でSDS-PAGEに供した。SERCA-FITC蛍光は、UV光曝露後の透明ゲル中で観察されます。(B)(A)のゲルのクーマシーブルーの染み。この図は、Rivera-Morán et al.46から採用されています。 この図の拡大版を表示するには、ここをクリックしてください。
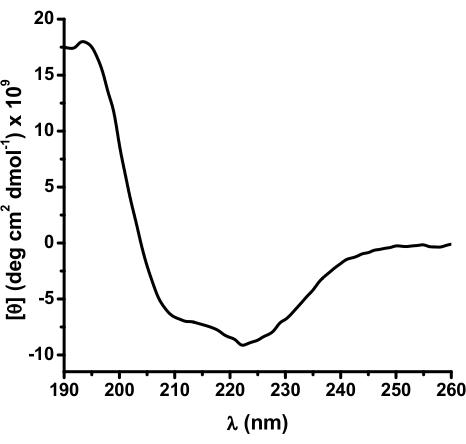
図4:SERCAの環二色性(CD)スペクトル。 SERCA (3 μM) の CD スペクトルを 10 mM のリン酸緩衝液 pH 8.0 に懸濁。この図は、Rivera-Morán et al.46から採用されています。 この図の拡大版を表示するには、ここをクリックしてください。
ディスカッション
ほとんどの分子およびイオンは細胞膜を自由に横切ることができず、例えば、プロトン(H+)は、ミトコンドリア83,84のような様々な生物および細胞小器官の原形質膜における膜トランスポーターを必要とする。細胞膜は選択的であり、細胞膜を通過する分子やイオンは多様であるため、細胞内には(a)ABCトランスポーター、(b)イオンチャネル、(c)膜結合型ATPアーゼ、(d)SLCトランスポーター、(e)水チャネル5など、いくつかの種類の膜タンパク質が見出されます。構造生物学では、機能的(触媒的)サイクルのほとんどのステップで酵素/トランスポーターの3Dコンフォメーションを取得することが大きな問題となっている85,86。すなわち、酵素の機能構造ランドスケープに関する詳細な知識は、触媒メカニズム5,87,88,89を理解するために重要である。高純度で活性な形態の膜タンパク質の単離は、これらの研究の最初のステップである90。疎水性(膜タンパク質の固有の物理化学的特性)は、これらのタンパク質を扱う際の主な問題であるように思われます12。最適でない精製プロトコル、不適切な実験条件、および膜タンパク質が懸濁している水性緩衝液は、通常、膜タンパク質25,27,91を扱おうとするときに遭遇する。保存中の触媒活性の喪失は、精製後の膜酵素でも起こり得る21。
P-ATPアーゼの場合、優れた代替手段は、組換えドメイン(例えば、Nドメイン)47,48、およびタンパク質異種発現システム53を用いて実験的研究を行うことである。しかし、実験では、天然のP-ATPアーゼ53全体を使用する必要がある場合があるため、単離手順中および保存中に安定化される高品質の酵素を得るためには、効率的なプロトコールが必要です。上記は特に生体組織とその生体源の過度の使用を節約するでしょう。
現在、人工知能(AI、進化し続けるツール)の使用は、新しく設計されたタンパク質と既存のタンパク質の両方におけるタンパク質の溶解性、安定性、および機能を改善することを目的とした究極の戦略の1つです36,92,93。ただし、AIは主に可溶性タンパク質に焦点を当てています。膜タンパク質は、少なくともしばらくの間、この新しい技術には型破りであるように思われます。
膜タンパク質/酵素の精製のためのプロトコルの最適化は、私たちの研究室が20年間取り組んできたオプションです。単離されたSRVのSERCA含有量は~73%です。改善のために特定された精製ステップは、最終的な遠心分離ステップであり、現在はトレハロース濃度勾配で行われています。精製されたSERCAの純度と構造品質は高かった(>90%)(図1Bおよび図4)。SERCA Vの最大値は、他の調製物について報告された範囲内であった94,95,96、一方、Km値はSRVのそれと同様であった。トレハロースは、一部の精製ステップで適用されるような環境ストレス条件下でタンパク質の3D構造を安定化することが知られている分子である。これは、酵母原形質膜H+-ATPaseなどのP型ATPaseファミリーのメンバーですでに実証されており、今回のSERCA31,46でも実証されています。他のP型ATPアーゼは、生物物理学的、酵素速度論的(阻害剤試験)、および構造的(四次構造)研究のための本プロトコルを使用することにより精製できる可能性があります。したがって、不安定な膜/細胞質タンパク質の精製プロトコル(および保管)におけるトレハロースの使用が提案されます。
開示事項
著者は、競合する金銭的利益がないことを宣言しています。
謝辞
著者は、ビデオの編集におけるEdmundo Mata-Morales、SRVの浄化におけるVM、SRVの浄化におけるValentín de la Cruz-Torres、SERCAの精製と分析におけるMiguel A. Rivera-Moran、およびビデオ制作におけるJuan C. Gonzalez-Castro、Franco E. Juarez、Alejandra Nevarez、Nicolas Rocha-Vizuet、およびJocelin I. Ramírez-Alonsoの支援に感謝します。この研究を実施するための資金、助成金、またはその他の支援は受け取られませんでした。
資料
| Name | Company | Catalog Number | Comments |
| ATP | Sigma-Aldrich Corp | A2383 | |
| Azolectin from soybean | Sigma-Aldrich Corp | 44924 | |
| Benchtop UV transilluminator | Cole-Parmer | EW-97623-08 | Dual intensity High setting is ideal for analytical documentation. Low setting reduces photonicking or photobleaching of gel samples while doing preparative work. |
| CaCl2 • 2H2O | Sigma-Aldrich Corp | 223506 | |
| Coolpix B500 camera | Nikon Corp | S210 | |
| Coomassie brilliant blue G-250 | Bio-Rad | 1610406 | |
| Dodecyl maltoside | Sigma-Aldrich Corp | D4641 | |
| Ethylene glycol-bis(2-aminoethylether)-N,N,N′,N′-tetraacetic acid | Sigma-Aldrich Corp | E4378 | |
| Ethylenediaminetetraacetic acid tetrasodium salt dihydrate | Sigma-Aldrich Corp | E6511 | |
| Fluorescein isothiocyanate | Sigma-Aldrich Corp | F3651 | |
| KCl | JT Baker | 7447-40-7 | |
| MgCl2 • 6H2O | Sigma-Aldrich Corp | M9272 | |
| MOPS | Sigma-Aldrich Corp | M1254 | |
| NADH | Chem-Impex International Inc | 230 | |
| N-tetradecyl-N,N- dimethyl-3-ammonium-1-propanesulfonate | Sigma-Aldrich Corp | D0431 | |
| Phosphoenolpyruvate (PEP) | Chem-Impex International Inc | 9711 | |
| Rabbit muscle lactate dehydrogenase | Roche | 10003557103 | |
| Rabbit muscle pyruvate kinase | Sigma-Aldrich Corp | P1506 | |
| Sodium deoxycholate | Sigma-Aldrich Corp | D6750 | |
| Sodium dodecyl sulfate | Bio-Rad | 1610302 | |
| Spectropolarimeter | Jasco Corp. | Jasco J1500 | |
| Sucrose | Sigma-Aldrich Corp | 84100 | |
| Trehalose | Sigma-Aldrich Corp | T0167 | Dihydrate |
| Tris(hydroxymethyl)aminomethane hydrochloride | Sigma-Aldrich Corp | 857645 | |
| Ultracentrifuge | Beckman | Optima XPN | |
| UV/VIS spectrophotometer | Agilent Technologies | 8453 | The Agilent 8453 UV-Vis Spectrophotometer uses a photodiode array for simultaneous measurement of the complete ultra-violet to visible light spectrum |
| WiseStir HS- 30E | Daihan Scientific Co. | DH.WOS01010 | Ideal for all disperging and homogenizing applications, designed for tissue grinders. |
参考文献
- Schuberth, C., Wedlich-Söldner, R. Building a patchwork - The yeast plasma membrane as model to study lateral domain formation. Biochim Biophys Acta Mol Cell Res. 1853 (4), 767-774 (2015).
- Douglas, L. M., Konopka, J. B. Fungal membrane organization: The eisosome concept. Annu Rev Microbiol. 68 (1), 377-393 (2014).
- Borrell, J. H., Domènech, &. #. 2. 1. 0. ;., Keough, K. M. W. . Membrane protein - Lipid interactions: Physics and Chemistry in the Bilayer. , (2016).
- Moraes, I., Evans, G., Sanchez-Weatherby, J., Newstead, S., Stewart, P. D. . The next generation in membrane protein structure determination. , (2016).
- White, S. H. Biophysical dissection of membrane proteins. Nature. 459 (7245), 344-346 (2009).
- Lacapère, J. -. J. Membrane protein structure determination. Methods Mol Biol. , 459 (1002).
- Palmgren, M. G. Plant plasma membrane H+-ATPases: Powerhouses for nutrient uptake. Annu Rev Plant Physiol Plant Mol Biol. 52 (1), 817-845 (2001).
- Yatime, L., et al. P-type ATPases as drug targets: Tools for medicine and science. Biochim Biophys Acta Bioenerg. 1787 (4), 207-220 (2009).
- Drews, J. Tetrahydrobiopterin biosynthesis as an off-target of sulfa drugs. Science. 287 (5460), 1960-1964 (2000).
- Xu, Z., Meshcheryakov, V. A., Poce, G., Chng, S. -. S. MmpL3 is the flippase for mycolic acids in mycobacteria. Proc Natl Acad Sci USA. 114 (30), 7993-7998 (2017).
- Siligardi, G., Hussain, R., Patching, S. G., Phillips-Jones, M. K. Ligand- and drug-binding studies of membrane proteins revealed through circular dichroism spectroscopy. Biochim Biophys Acta Biomembr. 1838 (1 PARTA), 34-42 (2014).
- Rawlings, A. E. Membrane proteins: Always an insoluble problem. Biochem Soc Trans. 44 (3), 790-795 (2016).
- Popot, J. -. L. . Membrane proteins in aqueous solutions. , 708 (2018).
- Palmgren, M. G., Sommarin, M., Ulvskov, P., Larsson, C. Effect of detergents on the H+-ATPase activity of inside-out and right-side-out plant plasma membrane vesicles. Biochim Biophys Acta Biomembr. 1021 (2), 133-140 (1990).
- McBride, Z., Chen, D., Reick, C., Xie, J., Szymanski, D. B. Global analysis of membrane-associated protein oligomerization using protein correlation profiling. Mol Cell Proteomics. 16 (11), 1972-1989 (2017).
- Yoneda, J. S., et al. Multimeric species in equilibrium in detergent-solubilized Na, K-ATPase. Int J Biol Macromol. 89, 238-245 (2016).
- Ohlendieck, K. Extraction of membrane proteins. Protein Purif Protocols. 59, 283-294 (2004).
- Champeil, P., et al. A robust method to screen detergents for membrane protein stabilization, revisited. Anal Biochem. 511, 31-35 (2016).
- Gimpl, K., Klement, J., Keller, S. Characterising protein/detergent complexes by triple-detection size-exclusion chromatography. Biol Proced Online. 18, 4 (2016).
- le Maire, M., et al. Gel chromatography and analytical ultracentrifugation to determine the extent of detergent binding and aggregation, and Stokes radius of membrane proteins using sarcoplasmic reticulum Ca2+-ATPase as an example. Nat Protoc. 3 (11), 1782-1795 (2008).
- Du, M., et al. Progress, applications, challenges and prospects of protein purification technology. Front Bioeng Biotechnol. 10, 1-26 (2022).
- Slotboom, D. J., Duurkens, R. H., Olieman, K., Erkens, G. B. Static light scattering to characterize membrane proteins in detergent solution. Methods. 46 (2), 73-82 (2008).
- Harding, S. E. Analytical ultracentrifugation as a matrix-free probe for the study of kinase related cellular and bacterial membrane proteins and glycans. Molecules. 26, 6080 (2021).
- Chen, Y. -. C., et al. Thermal stability, storage and release of proteins with tailored fit in silica. Sci Rep. 7 (1), 46568 (2017).
- Montigny, C. C., Arnou, B., Marchal, E., Champeil, P. Use of glycerol-containing media to study the intrinsic fluorescence properties of detergent-solubilized native or expressed SERCA1a. Biochemistry. 47 (46), 12159-12174 (2008).
- Simongini, M., Puglisi, A., Genovese, F., Hochkoeppler, A. Trehalose counteracts the dissociation of tetrameric rabbit lactate dehydrogenase induced by acidic pH conditions. Arch Biochem Biophys. 740, 109584 (2023).
- Shivanna, B. D., Rowe, E. S. Preservation of the native structure and function of Ca2+-ATPase from sarcoplasmic reticulum: Solubilization and reconstitution by new short-chain phospholipid detergent 1,2-diheptanoyl-sn-phosphatidylcholine. Biochem J. 325 (2), 533-542 (1997).
- Sampedro, J. G., Guerra, G., Pardo, J. P., Uribe, S. Trehalose-mediated protection of the plasma membrane H+-ATPase from Kluyveromyces lactis during freeze-drying and rehydration. Cryobiology. 37 (2), 131-138 (1998).
- Sampedro, J. G., Cortés, P., Muñoz-Clares, R. A., Fernández, A., Uribe, S. Thermal inactivation of the plasma membrane H+-ATPase from Kluyveromyces lactis. Protection by Trehalose. Biochim Biophys Acta Proteins Proteomics. 1544 (1-2), 64-73 (2001).
- Sampedro, J. G., et al. Fluorescence quenching by nucleotides of the plasma membrane H+-ATPase from Kluyveromyces lactis. Biochemistry. 46 (18), 5616-5622 (2007).
- Ruiz-Granados, Y. G., De La Cruz-Torres, V., Sampedro, J. G. The oligomeric state of the plasma membrane H+-ATPase from Kluyveromyces lactis. Molecules. 24 (5), 958 (2019).
- Zhao, P., Zhao, C., Chen, D., Yun, C., Li, H., Bai, L. Structure and activation mechanism of the hexameric plasma membrane H+-ATPase. Nat Commun. 12 (1), 6439 (2021).
- Auer, M., Scarborough, G. A., Kühlbrandt, W. Surface crystallization of the plasma membrane H+-ATPase on a carbon support film for electron crystallography. J Mol Biol. 287 (5), 961-968 (1999).
- Hennessey, J. P., Scarborough, G. A. Secondary structure of the Neurospora CRASSA plasma membrane H+-ATPase as estimated by circular dichroism. J Biol Chem. 263 (7), 3123-3130 (1988).
- Scarborough, G. A. Crystallization, structure and dynamics of the proton-translocating P-type ATPase. J Exp Biol. 203 (Pt 1), 147-154 (2000).
- Sumida, K. H., et al. Improving protein expression, stability, and function with proteinMPNN. J Am Chem Soc. 146 (3), 2054-2061 (2024).
- Eleutherio, E., Panek, A., De Mesquita, J. F., Trevisol, E., Magalhães, R. Revisiting yeast trehalose metabolism. Curr Genet. 61 (3), 263-274 (2015).
- Attfield, P. V. Trehalose accumulates in Saccharomyces cerevisiae during exposure to agents that induce heat shock response. FEBS Lett. 225 (1-2), 259-263 (1987).
- Marunde, M. R., et al. Improved tolerance to salt and water stress in Drosophila melanogaster cells conferred by late embryogenesis abundant protein. J Insect Physiol. 59 (4), 377-386 (2013).
- Crowe, J. H. Anhydrobiosis: An unsolved problem with applications in human welfare. Subcell Biochem. 71, 263-280 (2015).
- Crowe, J. H., Crowe, L. M., Chapman, D. Preservation of membranes in anhydrobiotic organisms: The role of trehalose. Science. 223 (4637), 701-703 (1984).
- Sampedro, J. G., Rivera-Moran, M. A., Uribe-Carvajal, S. Kramers' theory and the dependence of enzyme dynamics on trehalose-mediated viscosity. Catalysts. 10 (6), 1-19 (2020).
- Sampedro, J. G., Uribe, S. Trehalose-enzyme interactions result in structure stabilization and activity inhibition. The role of viscosity. Mol Cell Biochem. 256 - 257 (1-2), 319-327 (2004).
- Crowe, J. H. Trehalose as a "chemical chaperone": Fact and fantasy. Adv Exp Med Biol. 594, 143-158 (2007).
- Crowe, J. H., Carpenter, J. F., Crowe, L. M., Anchordoguy, T. J. Are freezing and dehydration similar stress vectors? A comparison of modes of interaction of stabilizing solutes with biomolecules. Cryobiology. 27 (3), 219-231 (1990).
- Rivera-Morán, M. A., Sampedro, J. G. Isolation of the sarcoplasmic reticulum Ca2+-ATPase from rabbit fast-twitch muscle. Methods Protoc. 6 (5), 102 (2023).
- Páez-Pérez, E. D., De La Cruz-Torres, V., Sampedro, J. G. Nucleotide binding in an engineered recombinant Ca2+-ATPase N-domain. Biochemistry. 55 (49), 6751-6765 (2016).
- Ramírez-Alonso, J. I., Sampedro, J. G. Effect of cations on ATP binding to the N-domain of Na+, K+-ATPase. J Fluoresc. , (2024).
- De la Cruz-Torres, V., Cataño, V., Olivo-Rodríguez, M., Sampedro, J. G. ANS Interacts with the Ca2+-ATPase nucleotide binding site. J Fluoresc. 30 (3), 483-496 (2020).
- Sampedro, J. G., Nájera, H., Uribe-Carvajal, S., Ruiz-Granados, Y. G. Mapping the ATP binding site in the plasma membrane H+-ATPase from Kluyveromyces lactis. J Fluoresc. 24 (6), 1849-1859 (2014).
- Kiani, A. K., et al. Ethical considerations regarding animal experimentation. J Prev Med Hyg. 63 (2 Suppl 3), E255-E266 (2022).
- Ruiz-Granados, Y., De La Cruz-Torres, V., Sampedro, J. The oligomeric state of the plasma membrane H+-ATPase from Kluyveromyces lactis. Molecules. 24 (5), 958 (2019).
- Wang, S., et al. Structural basis for sarcolipin's regulation of muscle thermogenesis by the sarcoplasmic reticulum Ca2+-ATPase. Sci Adv. 7 (48), eabi7154 (2021).
- Champeil, P., et al. ATP regulation of sarcoplasmic reticulum Ca2+-ATPase. Metal-free ATP and 8-bromo-ATP bind with high affinity to the catalytic site of phosphorylated ATPase and accelerate dephosphorylation. J Biol Chem. 263 (25), 12288-12294 (1988).
- Møller, J. V., Olesen, C. Preparation of Ca(2+)-ATPase1a enzyme from rabbit sarcoplasmic reticulum. Methods Mol Biol. 1377, 11-17 (2016).
- Wuytack, F., De Schutter, G., Casteels, R. Partial purification of (Ca2+ + Mg2+)-dependent ATPase from pig smooth muscle and reconstitution of an ATP-dependent Ca2+-transport system. Biochem J. 198 (2), 265-271 (1981).
- Mandal, A., et al. Solubilization, purification and reconstitution of Ca2+-ATPase from bovine pulmonary artery smooth muscle microsomes by different detergents: Preservation of native structure and function of the enzyme by DHPC. Biochim Biophys Acta Gen Subj. 1760 (1), 20-31 (2006).
- Inesi, G., Ma, H., Hua, S., Toyoshima, C. Characterization of Ca2+ ATPase residues involved in substrate and cation binding. Ann NY Acad Sci. 986 (1), 63-71 (2003).
- Van Winkle, W. B., Pitts, B. J., Entman, M. L. Rapid purification of canine cardiac sarcoplasmic reticulum Ca2+-ATPase. J Biol Chem. 253 (24), 8671-8673 (1978).
- Ruiz-Granados, Y. G., De La Cruz-Torres, V., Sampedro, J. G. The oligomeric state of the plasma membrane H+-ATPase from Kluyveromyces lactis. Molecules. 24 (5), 958 (2019).
- Teramoto, N., Sachinvala, N. D., Shibata, M. Trehalose and trehalose-based polymers for environmentally benign, biocompatible, and bioactive materials. Molecules. 13 (8), 1773-1816 (2008).
- Dao, T. T., et al. Demethoxycurcumin is a potent inhibitor of P-type ATPases from diverse kingdoms of life. PLOS ONE. 11 (9), e0163260 (2016).
- Bleeker, N. P., Cornea, R. L., Thomas, D. D., Xing, C. A novel SERCA inhibitor demonstrates synergy with classic SERCA inhibitors and targets multidrug-resistant AML. Mol Pharmaceutics. 10 (11), 4358-4366 (2013).
- Michelangeli, F., East, J. M. A diversity of SERCA Ca2+ pump inhibitors. Biochem Soc Trans. 39 (3), 789-797 (2011).
- Tadini-Buoninsegni, F., Smeazzetto, S., Gualdani, R., Moncelli, M. R. Drug interactions with the Ca2+-ATPase from Sarco(Endo)Plasmic Reticulum (SERCA). Front Mol Biosci. 5, 36 (2018).
- Clausen, J. D., et al. Elucidation of antimicrobial activity and mechanism of action by N-substituted carbazole derivatives. Bioorg Med Chem Lett. 27 (19), 4564-4570 (2017).
- Leary, S., et al. . AVMA guidelines for the euthanasia of animals: 2020 Edition, Members of the Panel on Euthanasia AVMA Staff Consultants. , (2020).
- de Aluja, A. S. Laboratory animals and official Mexican norms (NOM-062-ZOO-1999). Gac Med Mex. 138 (3), 295-298 (2002).
- Champeil, P., Buschlen-Boucly, S., Bastide, F., Gary-Bobo, C. Sarcoplasmic reticulum ATPase. Spin labeling detection of ligand-induced changes in the relative reactivities of certain sulfhydryl groups. J Biol Chem. 253 (4), 1179-1186 (1978).
- Skalec, A., Janeczek, M., Czerski, A. Anatomy and histology of the rabbit common calcanean tendon. J Vet Med Series C: Anat Histol Embryol. 48 (5), 466-475 (2019).
- Janes, L. E., Mioton, L. M., Fracol, M. E., Ko, J. H. An in vivo comparison: Novel mesh suture versus traditional suture-based repair in a rabbit tendon model. J Hand Surg Glob Online. 4 (1), 32-39 (2022).
- Bollag, D. M., Rozycki, M. D., Edelstein, S. J. . Protein Methods. , (1996).
- Gagné, F. Tissue preparation and subcellular fractionation techniques. Biochem Ecotoxicol. , 21-31 (2014).
- Lowry, O. H., Rosebrough, N. J., Farr, A. L., Randall, R. J. Protein measurement with the Folin phenol reagent. J Biol Chem. 193 (1), 265-275 (1951).
- Autry, J. M., Rubin, J. E., Svensson, B., Li, J. L., Thomas, D. D. Nucleotide activation of the Ca-ATPase. J Biol Chem. 287 (46), 39070-39082 (2012).
- Winters, D. L., Autry, J. M., Svensson, B., Thomas, D. D. Interdomain fluorescence resonance energy transfer in SERCA probed by cyan-fluorescent protein fused to the actuator domain. Biochemistry. 47 (14), 4246-4256 (2008).
- Miles, A. J., Wallace, B. A. Circular dichroism spectroscopy of membrane proteins. Chem Soc Rev. 14 (45), 1130-1135 (2016).
- Scofano, H. M., Vieyra, A., de Meis, L. Substrate regulation of the sarcoplasmic reticulum ATPase. Transient kinetic studies. J Biol Chem. 254 (20), 10227-10231 (1979).
- Yamamoto, T., Tonomura, Y. Reaction mechanism of the Ca++-dependent ATPase of sarcoplasmic reticulum from skeletal muscle: I. Kinetic studies. J Biochem. 62 (5), 558-575 (1967).
- Murphy, A. J. Affinity labeling of the active site of the Ca2+-ATPase of sarcoplasmic reticulum. Biochim Biophys Acta - Biomembr. 946 (1), 57-65 (1988).
- Csermely, P., Katopis, C., Wallace, B. A., Martonosi, A. The E1→E2 transition of Ca2+-transporting ATPase in sarcoplasmic reticulum occurs without major changes in secondary structure. A circular-dichroism study. Biochem J. 241 (3), 663-669 (1987).
- Abràmoff, M. D., Magalhães, P. J., Ram, S. J. Image processing with ImageJ. Biophotonics Int. 11 (7), 36-42 (2004).
- Scarborough, G. A. The plasma membrane proton-translocating ATPase. Cell Mol Life Sci. 57 (6), 871-883 (2000).
- Llopis, J., McCaffery, J. M., Miyawaki, A., Farquhar, M. G., Tsien, R. Y. Measurement of cytosolic, mitochondrial, and Golgi pH in single living cells with green fluorescent proteins. Proc Natl Acad Sci. 95 (12), 6803-6808 (1998).
- Kanai, R., Vilsen, B., Cornelius, F., Toyoshima, C. Crystal structures of Na+, K+-ATPase reveal the mechanism that converts the K+-bound form to Na+-bound form and opens and closes the cytoplasmic gate. FEBS Lett. 597 (15), 1957-1976 (2023).
- Palmgren, M. P-type ATPases: Many more enigmas left to solve. J Biol Chem. 299 (11), 105352 (2023).
- Frauenfelder, H. Ask not what physics can do for biology-Ask what biology can do for physics. Phys Biol. 11 (5), 053004 (2014).
- Frauenfelder, H. The physics of proteins. Annu Rev Biophys Biomol Struct. 36, (2010).
- Narhi, L. O. . Biophysics for Therapeutic Protein Development. , (2013).
- East, J. M. Purification of a membrane protein (Ca2+/Mg2+-ATPase) and its reconstitution into lipid vesicles. Methods Mol Biol. 27, 87-94 (1994).
- Lee, E. -. H., et al. Enhancement of enzyme activity and stability by poly(γ-glutamic acid). Polymer J. 42 (10), 818-822 (2010).
- Norrild, R. K., et al. Increasing protein stability by inferring substitution effects from high-throughput experiments. Cell Rep Methods. 2 (11), 100333 (2022).
- Song, Z., Zhang, Q., Wu, W., Pu, Z., Yu, H. Rational design of enzyme activity and enantioselectivity. Front Bioeng Biotechnol. 11, 1-14 (2023).
- Jidenko, M., Lenoir, G., Fuentes, J. M., le Maire, M., Jaxel, C. Expression in yeast and purification of a membrane protein, SERCA1a, using a biotinylated acceptor domain. Protein Expr Purif. 48 (1), 32-42 (2006).
- Hua, S., Ma, H., Lewis, D., Inesi, G., Toyoshima, C. Functional role of "N" (nucleotide) and "P" (phosphorylation) domain interactions in the sarcoplasmic reticulum (SERCA) ATPase. Biochemistry. 41 (7), 2264-2272 (2002).
- Pang, Y. -. H., Chen, J. -. W. Anisodamine causes the changes of structure and function in the transmembrane domain of the Ca2+-ATPase from sarcoplasmic reticulum. Biosci Biotechnol Biochem. 68 (1), 126-131 (2004).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved