Method Article
単一分子FRET-TIRF顕微鏡法のための長鎖RNAの蛍光末端標識とカプセル化
要約
この記事では、末端位置での長鎖RNAの二色標識と、単一分子FRET TIRF顕微鏡アプリケーションのためのリン脂質小胞へのカプセル化によるそれらの表面固定化について説明します。これらの技術を組み合わせることで、RNAの動態を1分子レベルで精密に可視化・解析することが可能になります。
要約
単一分子フェルスター共鳴エネルギー移動(smFRET)は、経時的な立体配座の変化を正確に観察することにより、動的生体分子の研究に優れています。smFRETでRNAの動態をモニタリングするために、FRETペアの蛍光色素を用いてRNAの末端を共有結合的に標識する方法を開発しました。この直接末端標識戦略は、カルボジイミド(EDC)/N-ヒドロキシスクシンイミド(NHS)の活性化による5'-リン酸と過ヨウ素酸酸化による3'-リボースを標的としており、サイズや配列に関係なく他のRNAに適応させ、人工的な修飾とは無関係に研究することができます。さらに、5'-EDC/NHSの活性化は、5'-リン酸を持つすべての核酸にとって一般的な関心事です。市販の化学物質を使用することで、RNA特異的なプローブを合成する必要がなくなります。
全反射蛍光(TIRF)顕微鏡法では、目的の表面固定化分子が照射されるエバネッセントフィールド内にある必要があります。RNA分子をエバネッセント場内に保持する洗練された方法は、RNA分子をリン脂質小胞にカプセル化することです。カプセル化は、分子を表面につなぎとめながら、分子の自由な拡散を可能にするという、両方の長所を生かしています。各小胞に1つのRNA分子のみが含まれていることを確認し、1分子のイメージングを可能にします。目的のRNAのデュアルエンド標識とカプセル化により、smFRET測定により、RNAの挙動を動的かつ詳細に把握することができます。
概要
フェルスター共鳴エネルギー移動(FRET)は、ナノスケールでの生体分子の分子間および分子内相互作用を研究するための強力で高感度な手法です。これは、励起されたドナー分子から近くのアクセプター分子への非放射エネルギー移動に基づいており、通常は1nm〜10nmの距離で発生します。ドナー色素とアクセプター色素の間の距離がこのエネルギー伝達の効率を決定するため、FRETは、分子動力学、コンフォメーション変化、およびRNAを含む幅広い生物学的システム1,2の相互作用を研究するための非常に貴重なツールとなっています。全反射蛍光法(TIRF)は、表面近くの分子のみを選択的に照らし、個々の分子のFRETダイナミクスを高い空間的および時間的分解能で捉えることができるため、smFRET研究の強力な技術であることが証明されています。ただし、smFRET-TIRF実験を行う前に、まず目的の分子を適切なFRETペアで蛍光標識し、次に顕微鏡表面に固定化する必要があります。ここで説明するsmFRET-TIRFプロトコルは、Saccharomyces cerevisiaeミトコンドリア由来の野生型グループIIイントロンSc.ai5γを使用し、その2つのエキソニック配列(915ヌクレオチド)3に隣接して検証されました。蛍光標識グループIIイントロンとsmFRET TIRF顕微鏡法におけるそれらの固定化の詳細については、レビュー4を参照してください。
理想的な部位特異的なRNA標識戦略は、RNAの構造や機能を変えることなく、ドナー色素とアクセプター色素を所定の位置に正確に取り込むことを可能にし、正確で効率的なFRET測定を保証します。これは、4つの核酸塩基間の化学的類似性のために困難であり、選択的標識を複雑にします。末端標識は、5'-リン酸および3'-リボースを標的とすることにより、ドナー色素およびアクセプター色素をRNA末端に結合します。このアプローチは、低侵襲アプローチを提供しながら、構造ダイナミクスと相互作用に関する貴重な洞察を提供します。グループIIイントロンは、Mg2+ の存在下で自己スプライスできるため、金属イオン依存性酵素の使用が制限されます。ここでは、酵素や特殊なプローブの合成の必要性を回避するデュアルエンドラベルの長く触媒活性なRNA(リボザイム)へのアプローチを紹介します。
TIRF顕微鏡法でRNA分子を表面につなぎとめる一般的なアプローチは、ビオチン部分を直接RNAに共有結合するか、ビオチンを運ぶアンチセンスオリゴヌクレオチド(ASO)5,6,7をハイブリダイズすることです。しかし、この直接固定化法は、RNAと表面の相互作用によるアーチファクトを引き起こす可能性があり、その結果、RNAが誤って折り畳まれる可能性があります8。これらの固定化アーチファクトを軽減するためのエレガントな解決策は、表面に付着したナノスケールのリン脂質小胞9,10,11にRNAをカプセル化することである。直径約100nmのこれらの小胞は、ビオチン-ストレプトアビジン結合12,13,14を介して表面に固定されており、RNAが内部で自由に拡散することを可能にすると同時に、脂質膜10を横切るイオンの交換を可能にする。大きな機能的RNA3を共有結合的に標識した後、RNAの機能を保持するように適合した、表面不動態化と小胞カプセル化のための確立されたプロトコルを組み合わせることにより、そのようなRNAをリン脂質小胞にカプセル化するアプローチを提示する10,11,14。このデュアルエンドラベリングおよびカプセル化アプローチにより、smFRET TIRF顕微鏡用の機能性RNAのモノカプセル化が高速に達成されます。
プロトコル
1. RNAデュアルエンドラベリング
注:次のプロトコルは、ドナー色素(sCy3)を5'-リン酸に、アクセプター色素(Cy5)を3'-リボースに共有結合することにより、FRETペアの蛍光色素を用いたRNAの部位特異的標識について説明しています。触媒活性な長鎖RNA、グループIIイントロンリボザイムが目的のRNAとして選択されます。 表1 と 図1 は、このデュアルエンドラベリングプロトコルをまとめたものです。蛍光色素を含むすべてのステップを暗所で行ってください。
| 0日目 | ▪ 50〜75μgのRNAを1.5mLチューブあたり55μLの総量に分注します。 | |||||
| 1日目 | 5′-リン酸活性化 | |||||
| ▪ 調製したばかりのEDC-NHS、pH 6.0溶液45 μLをddH2O中のRNAに45 μL加え、最終容量100 μLまで調製し、よく混合して25°C、500 rpmで4時間インキュベートします。 | ||||||
| ▪ 精製ラウンド1:一晩のEtOH沈殿。 | ||||||
| 2日目 | ▪ 5′活性化RNAを沈殿させ、洗浄し、乾燥させます。 | |||||
| 5 '-染料アタッチメント | ||||||
| ▪ 95 μL の 100 mM MOPS、pH 7.5 に再懸濁します。 | ||||||
| ▪ 2 mMアミン官能基化色素溶液5 μLを添加します。 | ||||||
| ▪ よく混合し、25°C、500rpmで16時間インキュベートします。 | ||||||
| 3日目 | ▪ 精製ラウンド2:EtOH沈殿。 | |||||
| 4日目 | ▪ 5′活性化RNAを沈殿させ、洗浄し、乾燥させます。 | |||||
| ブロッキングステップ | ||||||
| ▪ 100 μL の 100 mM Tris–HCl、pH 7.5 に再懸濁し、25 °C および 500 rpm で 2 時間インキュベートします。 | ||||||
| ▪ 精製ラウンド3:遠心ろ過。 | ||||||
| → 5′標識RNAを溶出します。 | ||||||
| 5日目 | 3'-過ヨウ素酸酸化 | |||||
| ▪ RNAを20 mM NaIO4 in 50 mM NaOAcバッファー、pH 5.5、最終容量100 μL、25°C、500 rpmで2時間インキュベートします。 | ||||||
| ▪ 余分な過ヨウ素酸をクエンチする:50%グリセロール30μLを加えてよく混合し、25°C、500rpmで30分間インキュベートします。 | ||||||
| ▪ 精製ラウンド4:一晩のEtOH沈殿。 | ||||||
| 6日目 | ▪ 3′-酸化RNAを沈殿させ、洗浄し、乾燥させます。 | |||||
| 3 '-染料アタッチメント | ||||||
| ▪ 95 μL の 50 mM NaOAc、pH 6.0 に再懸濁します。 | ||||||
| ▪ 2 mMヒドラジド官能基化色素溶液5 μLを添加します。よく混合し、25°C、500rpmで16時間インキュベートします。 | ||||||
| 7日目 | ▪ 精製ラウンド5:EtOH沈殿。 | |||||
| 8日目 | ▪ 標識RNAを沈殿させ、洗浄し、乾燥させます。 | |||||
| ▪ 遠心ろ過。 | ||||||
| → 二重末端標識RNAを溶出します。 | ||||||
表1:RNAデュアルエンド標識のプロトコル概要。この表をダウンロードするには、ここをクリックしてください。
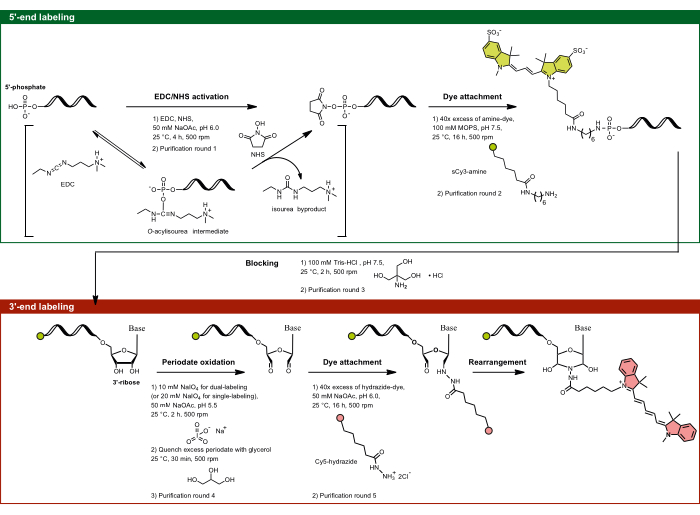
図1:5'-リン酸糖と3'-リボース糖を標的としたデュアルエンド標識の実験的フロー。 5'-リン酸は、NHSの存在下でEDCを使用して活性化され、その後、アミン官能基化色素と結合します。RNAの3'-ジオール部分は、過ヨウ素酸塩の活性によってジアルデヒドに酸化され、ジアルデヒドはさらにヒドラジド官能化色素と反応します。デュアルエンドラベリングでは、クロスラベリングを防ぐために5'ラベリングから始めて、その後に中間ブロッキングステップで3'ラベリングを行うことが重要です。略語:EDC =カルボジイミド;NHS = N-ヒドロキシスクシンイミド;MOPS = 3-モルホリノプロパン-1-スルホン酸;NaOAc = 酢酸ナトリウム。 この図の拡大版を表示するには、ここをクリックしてください。
- 5'末端標識:EDC/NHS活性化による5'-リン酸の標的化
注:この5'標識法は、RNAだけでなく、5'-リン酸を含む任意の一本鎖核酸にも適用されます。反応の特異性はpH依存性によって支配され、RNA骨格に複数のリン酸塩が存在するにもかかわらず、5'-リン酸のみを部位特異的に標的にすることができます。pH 6.0では、5'-リン酸のカルボジイミドに対する独特の反応性は、2つの酸素が脱プロトン化され、1つの酸素がプロトン化したままである特定のプロトン化状態によるものです。これにより、5'-リン酸は反応性になりますが、完全に脱プロトン化された骨格リン酸塩は反応性がないため、EDC/NHSターゲティングによる5'末端の選択的標識が可能になります。- EDC-NHS-NaOAc、pH 6.0溶液を調製します。1.5 mg の EDC と 2.0 mg の NHS を 1 分あたり 35 μL の ddH2O と 10 μL の 0.5 M NaOAc、pH 6.0 に混合します (pH は氷酢酸で調整します)。
- 55 μL の ddH2O にチューブあたり約 50-75 μg の RNA を含むように目的の RNA を分注し、45 μL の EDC-NHS-NaOAc、pH 6.0 混合物を RNA に加え、最終濃度 78 mM EDC、174 mM NHS、および 50 mM NaOAc、pH 6.0 で総容量 100 μL に達します。500 rpmで振とうしながら、25°Cで4時間インキュベートします。
注:ここで、70μgは、 in vitro で転写された915 nt RNAの250 pmolと、最終反応容量100 μLの2.5 μMに相当します。 - 精製ラウンド1:5'-リン酸活性化RNAをEtOH沈殿により精製します。
- 2 mMスルホン化シアニン3アミン(sCy3-アミン)溶液を水溶液に調製します。
- 活性化RNAペレットを100 mM 3-モルフォリノプロパン-1-スルホン酸(MOPS)緩衝液95 μL、pH 7.5(NaOHで調整)に再懸濁します。活性化RNAに5 μLの2 mM sCy3-アミン溶液を添加します。蛍光色素を活性化された5'-リン酸に結合させ、500 rpmで振とうしながら、25°Cで16時間インキュベートします。
注意: MOPSバッファーは、暗闇で4°Cで保管してください。NaOHは、グループIIイントロンリボザイムを不活性に保つためのpH調整に選択されます。蛍光色素は、RNAの少なくとも40倍過剰でなければなりません。 - 精製ラウンド2:分離を改善するために、200 μLのddH2Oを添加して容量を増やします。5'-リン酸標識RNAをEtOH沈殿物で精製し、上清が無色になるまで繰り返します(通常は2ラウンド必要です)。
- ブロッキングステップ:100 μL の 100 mM Tris-HCl、pH 7.5 に RNA を再懸濁し、25 °C および 500 rpm で 2 時間インキュベートします。5'末端シングルラベリングの場合のみ、ブロッキングステップをスキップし、RNAをddH2Oに再懸濁して、ステップ1.1.8に進みます。ただし、簡単にするために、二重ラベルの場合は、手順 1.1.9 の後にのみ 5'end 単一ラベル コントロールを分離します。
注:このステップは、比較的小さな第一級アミン源(トリスなど)と反応することにより、アミン官能化蛍光色素に結合していない活性化5'-リン酸をブロックし、3'末端標識プロトコルに使用されるヒドラジド官能化蛍光色素との交差標識のリスクを最小限に抑えるのに役立ちます。 - 精製ラウンド3:標識RNAを遠心ろ過で洗浄し、少なくとも合計10mLのddH2Oで遊離色素を除去し、次いでddH2Oで溶出させる。
注:フィルターの分子カットオフは、核酸の半分のサイズ未満である必要があります。ddH2Oの代わりに任意のバッファーを使用することができます。溶出はメーカーの指示に従って、サンプルが完全に乾燥するまで回転しないようにする必要があります。 - UV-Vis分光法により、RNAおよびコンジュゲート色素の濃度を測定します。
- 3'末端標識:過ヨウ素酸酸化による3'-リボースの標的化
- ddH2O中の5'末端標識RNAを分注して、90μL中のチューブあたり約50〜75μgのRNAを有するようにします。前ステップの溶出量が高かったため濃度が低かった場合は、ペレットをddH2Oに沈殿させて再懸濁することによりRNAを濃縮し、pH 5.5の1.0 M NaOAc緩衝液を5 μL加えます(100 μLの反応容量に対して50 mM NaOAc、pH 5.5に相当)。
注:5'末端の単一標識RNAと同様に、コントロールとして3'末端の単一標識RNAを日常的に調製します。これを行うには、標識されていないRNAを分注して、86μLのddH2O中にチューブあたり約50〜75μgのRNAを含ませます。 - 調製したばかりの500 mMメタヨウ素酸ナトリウム(NaIO4)ストック溶液(反応容量100 μLで10 mM NaIO4 に相当)を4 μL加えます。3'末端シングルラベリングの場合は、NaIO4 ストック溶液8 μL(反応容量100 μLの20 mM NaIO4 に相当)を添加します。NaIO4 は感光性であるため、暗所で500rpmで振とうしながら、十分に混合し、25°Cで2時間インキュベートします。
注:添加の順序を変更したり、インキュベーション時間をさらに長くしたりすると、付着したドナー色素の光退色を引き起こす可能性があります。デュアルエンドラベリングでは、5'末端に既結合した色素の焼入れを最小限に抑えるために、使用するNaIO4 の濃度を半分に減らします。 - 50%グリセロールを30μL加えて反応を停止します。500 rpmで振とうしながら、25°Cで30分間インキュベートします。
注:グリセロールは、過剰な過ヨウ素酸をクエンチするジオールとして機能します。 - 精製ラウンド4:氷冷したEtOH-NaOAc沈殿混合物400μLを加え、エタノール沈殿を行います。
- 酸化したRNAペレットを95 μLの50 mM NaOAc、pH 6.0に再懸濁します。
- ヒドラジド色素の付着:シアニン5ヒドラジド(Cy5-ヒドラジド)のいくつかの結晶をDMSOに溶解し、次にddH2Oで2 mMの濃度に希釈することにより、蛍光色素溶液を調製します。選択した蛍光色素が水溶性である場合は、ddH2O溶液を調製し、酸化RNAに5 μLの2 mM Cy5-ヒドラジド溶液を加えます。よく混合し、500 rpmで振とうしながら25°Cで16時間インキュベートします。
注:蛍光色素は、RNAの少なくとも40倍過剰である必要があります。 - 精製ラウンド5:ステップ1.2.6および1.2.8と同様に、それぞれEtOH沈殿および遠心ろ過により、二重末端標識RNA(または3'末端単一標識RNA)を精製し、ddH2Oで溶出します。
- UV-Vis分光法により、RNAおよびコンジュゲート色素の濃度を計算します。
- 分析ゲルベースのアッセイ、アンサンブル蛍光分光法(「代表的な結果」のセクションを参照)、および/または他の場所に示すように分析HPLCを使用して、標識されたRNAを特性評価します3。
- ddH2O中の5'末端標識RNAを分注して、90μL中のチューブあたり約50〜75μgのRNAを有するようにします。前ステップの溶出量が高かったため濃度が低かった場合は、ペレットをddH2Oに沈殿させて再懸濁することによりRNAを濃縮し、pH 5.5の1.0 M NaOAc緩衝液を5 μL加えます(100 μLの反応容量に対して50 mM NaOAc、pH 5.5に相当)。
2. マイクロ流体チャンバーの調製
注:一度に6つまたは8つのチャンバーを取り扱うことをお勧めします。超音波処理は、特に指示がない限り室温で行われます。このプロトコルは、微量不純物の可溶化を防ぐために、アセトンなどの有機溶媒の使用を制限します。代替案については、参考文献15,16を参照してください。
- 清掃
- 図2Aに示すスキームに従って、ダイヤモンドドリラーを使用してクォーツスライドに4つの穴を開け、2つのチャネルを形成します。
注:穴あけ加工中にガラスが壊れやすい場合がありますが、カバースリップがイメージング面である目的ベースのTIRF顕微鏡セットアップでは、スライドガラスの費用対効果の高い代替品です(図2B)。これは、バックグラウンド蛍光により顕微鏡スライドがイメージング面となるプリズムベースのTIRFセットアップではオプションではありません。
注:あるいは、使用済みのマイクロ流体チャンバー16をリサイクルします。このためには、使用済みのチャンバーをアセトンに一晩浸し(蒸発を最小限に抑えるためにCoplin染色ジャーをアルミホイルで包む)、ヒュームフードに入れ、その後5分間超音波処理します。チャンバーを分解し、カバースリップとステッカーを廃棄します。分解がすぐに機能しない場合は、チャンバーをアセトンでさらに10分間超音波処理します。手順 2.1.2 に進みます。 - 穴を開けた石英スライドと約2倍のカバースリップ(簡単に壊れる可能性があるため)を、カバー付きのガラスCoplin染色ジャーに入れます。ddH2Oで3回すすぎます。ddH2Oで5分間超音波処理し、次にddH2Oで3回すすぎます。
- 10%実験用ガラス器具洗浄液( 材料表参照)で50°Cで30分間超音波処理します。 洗剤の泡がなくなるまで、ddH2Oで少なくとも3回すすいでください。ddH2Oで5分間超音波処理します。ddH2Oで3回すすぎます。
- 1M KOH溶液で30分間超音波処理し、一晩放置します。ddH2Oで3回すすぎ、ddH2Oで5分間超音波処理します。ddH2Oで3回すすぎます。
注:過度のインキュベーションによる腐食は、Chandradossらによって以前に提起された懸念事項ですが15、ピラニアエッチングを回避できるこの長いインキュベーションをお勧めします。 - スライドとカバースリップをN2(g)で乾かします。
- 乾燥したスライドとカバースリップを酸素プラズマクリーナーで30分間、製造元の指示に従って処理します。
- 図2Aに示すスキームに従って、ダイヤモンドドリラーを使用してクォーツスライドに4つの穴を開け、2つのチャネルを形成します。
- アミノシラン化
- 288.5 mLの絶対EtOH、1.5 mLのddH2O、および9 mLの(3-アミノプロピル)トリエトキシシラン(APTES)を500 mLの三角フラスコで十分に混合することにより、3% APTES-EtOH溶液を調製します。.
- きれいなスライドとカバースリップをCoplin染色ジャーに入れ、3%APTES-EtOH溶液に浸し、1分間超音波処理し、30分間インキュベートします。
- 絶対EtOHで3回すすぎ、次にddH2Oで3回すすぎます。
- スライドとカバースリップをN2(g)の流れで乾かします。
- 表面の不動態化とビオチン化
- 空のピペットチップボックスをddH2Oで半分まで満たして、湿ったボックスを準備します。
- 2 mgのビオチン-ポリエチレングリコール-スクシンイミジル吉草酸5000(ビオチン-PEG-SVA)と80 mgのメトキシポリエチレングリコール-スクシンイミジル吉草酸エスキル(mPEG-SVA)を640 μLの100 mM重炭酸ナトリウム(NaHCO3)緩衝液、pH 8.3に穏やかに混合することにより、1.5 mLの滅菌清浄微遠心チューブでbPEG-mPEG混合物を新たに調製します。bPEG-mPEG混合物を16,000 × g で1分間遠心分離し、気泡を除去します。
- 上清を慎重に取り除き、スライドの中央に30 μLの液滴を追加して、両方のチャネルを覆います。スライドの片側に追加の液滴を置いて、PEG化カバースリップを追加で用意しますが、それらは簡単に壊れるので、予備があると便利です。最後に、液滴を清潔なカバースリップで覆い、湿った箱を閉じ、暗い条件下で一晩PEG化を行います。
- PEG不動態化およびビオチン化スライドとカバースリップをddH2Oで十分にすすぎます。処理された表面の疎水性の変化に注意してください。N2(g)の流れで乾燥させます。
- マイクロ流体チャンバーアセンブリ
- 両面ステッカーをカットしてチャンネルを作成します。ステッカーをスライドに貼り付け、関心のある領域をカバーしていることを確認します。カバーガラスを慎重に上に置き、PEG化表面が互いに向き合うように位置合わせします。
- 組み立てた各チャンバーを50 mLの遠心分離チューブに入れ、チューブにN2(g)を充填し、-20°Cで最大1か月間保存します。

図2:1分子FRET-TIRF顕微鏡 (A)TIRFイメージング用のマイクロ流体チャンバー。(B)FRET標識RNAをビオチン化リン脂質小胞にカプセル化し、ステプタビジン被覆ガラス表面に固定化します。これにより、目的の分子がエバネッセントフィールド(灰色の勾配)内に保たれ、これはTIRF顕微鏡の臨界角度で完全に反射される入射光によって作成されます。ここでは、両方の蛍光色素がその後ALEXスキームで励起されます。略語:FRET=フェルスター共鳴エネルギー移動;TIRF = 全反射蛍光;ALEX = 代替レーザー励起。 この図の拡大版を表示するには、ここをクリックしてください。
3. リン脂質小胞のカプセル化
- 脂質ケーキの準備
注意: クロロホルムは常にドラフトの下で取り扱ってください。- 滅菌針を使用して、 図3Aに示すように、滅菌済みで清潔な2.0 mL微量遠心チューブの蓋に(内側から外側に)穴を開けます。
- 1,2-ジパルミトイル-sn-グリセロ-3-ホスホエタノールアミン-N-キャップビオチニル(bPE)ストック溶液を調製し、少なくとも2 mgのbPEを適切な量のクロロホルムに溶解して、最終濃度1 mg / mLにします。.
- 1,2-ジミリストイル-sn-グリセロ-3-ホスホコリン(DMPC)ストック溶液を調製するには、少なくとも10 mgのDMPCを適切な量のクロロホルムに溶解して、最終濃度を10 mg / mLにします。.
- 100 μLのbPEと990 μLのDMPCストック溶液を混合して、bPE-DMPC混合物を調製します(1:99 w / w比を維持)。2 mLチューブあたり109 μLのbPE-DMPC混合物を分配します。
- マイクロチューブ収納ボックスの段ボール製セルパーティションインサートをシュレンクフラスコに入れ、チューブホルダーとして使用します。長いピンセットを使用して、脂質混合物を含むチューブを500 mLのシュレンクフラスコのチューブホルダーに入れます(図3B)。
注意: それらが直立していて、傾いていないことを確認してください。 - N2(g)の低流量で一晩(または溶媒が完全に蒸発するまで少なくとも2時間)クロロホルムを蒸発させます。
- 穴を覆うためにパラフィルムでチューブを密封します。脂質ケーキは-20°Cで最大1か月間保管してください。
- RNAカプセル化
- 目的の生物学的システムに応じてK+ およびMg2+ 濃度を調整し、以下の緩衝液と溶液を調製します。
- 5x 標準バッファー (5x SB): 2.5 M KCl、400 mM MOPS を調製します。KOHと滅菌フィルターでpHを6.9に調整し、暗所で4°Cで保存します(それぞれ1x SB:500 mM KCl、80 mM MOPS、pH 6.9)。
- まばたき防止バッファー(AB)を調製します:100 mM MgCl2、Trolox、1x SB、滅菌フィルター、および暗所で4°Cで最大1週間保存します。
注:10 mLの最終容量にTroloxのスパチュラチップを使用し、ボルテックスで混合し、pHを再調整します。
- 100 nmのポリカーボネート(PC)メンブレンと10 mmポリエステル(PE)ドレンディスクを使用して押出機を組み立て、シリンジとメンブレンをABで平衡化し、30°C(または基本的にDMPCガラス転移温度の24°C以上)に加熱します。
- 1 μM デュアルエンド標識 RNA 5 μL と AB 45 μL を混合します (総容量 50 μL まで)。
- このRNA溶液で脂質ケーキを水和させます。30°Cで1,400rpmで振とうして5分間混合し、次に700rpmで15分間混合します。13,000 g で2分間遠心分離し、上清を新しいチューブに慎重に移します。
- サンプルを250μLのABで希釈します。
- シリンジにRNA-脂質懸濁液を充填します。加熱ブロック上で30°Cで35倍押出し、直径100 nmのリン脂質小胞に二重末端標識RNAをカプセル化します。
注:押出機使用後の小胞のサイズ分布は、参考文献11に示すように、動的光散乱(DLS)によって検証できます。
- 目的の生物学的システムに応じてK+ およびMg2+ 濃度を調整し、以下の緩衝液と溶液を調製します。

図3:脂質ケーキの調製 (A)チューブキャップを突いて溶媒蒸発を可能にする。(B)脂質混合物を含むチューブをシュレンクフラスコに入れ、クロロホルムを蒸発させて脂質ケーキを得る。 この図の拡大版を表示するには、ここをクリックしてください。
4. smFRET-TIRF顕微鏡
- 表面固定化
- 以下のバッファーと溶液を調製します。
- 糖-点滅防止緩衝液(SAB)を調製します:AB中の1%D-グルコース(w / v)、滅菌フィルター、および暗所で4°Cで最大1週間保存します。
- 100x OSS:100x Oxygen scavenging System(OSS)を調製します:3 mgのグルコースオキシダーゼ(1.7 Uに相当)、10 μLのカタラーゼ(22 Uに対応)を90 μLの1x SBに取り込み、暗所で4°Cで最大1週間保存します。
- 198 μLのSABと2 μLの100x OSS溶液を混合して、イメージングバッファー(IB)を新たに調製します。
- マイクロ流体チャンバーを室温に到達させます。チャンバーを200 μLの1x SBで2回フラッシュします。
- チャンバーに20 μL/mLストレプトアビジン溶液50 μLを充填し、5分間インキュベートします。
- チャンバーを200 μLの1x SBでフラッシュし、次に100 μLのABでフラッシュします。
- 75 μLの小胞懸濁液を添加して、カプセル化されたRNAを表面に固定化し、10分間インキュベートします。
- 調製したばかりのIB200 μLでチャンバーをフラッシュし、5分間インキュベートします。
- これで、チャンバーをデータ取得する準備が整いました(図2B)。長時間の測定で蒸発を防ぐために、 図2Aに示すように穴を塞ぎます。チャネルからチャネルへの漏れが懸念される場合は、表面固定化プロトコルを開始する前に、未使用のチャネルを密閉してください。
注:蛍光汚染に対するマイクロ流体チャンバーとリン脂質小胞の清浄度の品質チェックとして、ブランク測定を行うことをお勧めします。
- 以下のバッファーと溶液を調製します。
結果
私たちは、エキソン配列に隣接する酵母ミトコンドリアSc.ai5γグループIIイントロンである915-nt RNAの部位特異的な単一蛍光および二重蛍光標識を示します。FRETフルオロフォアペアは、EDC/NHSによる5'-リン酸の活性化と3'-リボースの過ヨウ素酸酸化、続いてそれぞれの色素付着を介してRNA末端に位置します。次に、図4Aに示すように、蛍光ゲル電気泳動によりRNA色素コンジュゲートを検証しました。アガロースゲル上のRNAと蛍光色素の共遊走は、標識の成功を裏付けています。次に、図4Bに示すように、アンサンブル蛍光分光法を使用して、二重標識グループIIイントロンの特性評価を行いました。エネルギー移動、すなわちFRETは、ドナー色素の励起時に観察され、RNAの二重標識が証明されました。特に、FRETの距離依存性と一致するように、金属イオンの存在下でのグループIIイントロンRNAのフォールディングは、ドナー放出の減少(緑の矢印)とそれに対応するアクセプター放出の増加(赤矢印)によって証明されるように、FRETの効率の増加をもたらしました。これは、このFRETラベリングがリボザイムのコンフォメーション変化を追跡していることを示しています。
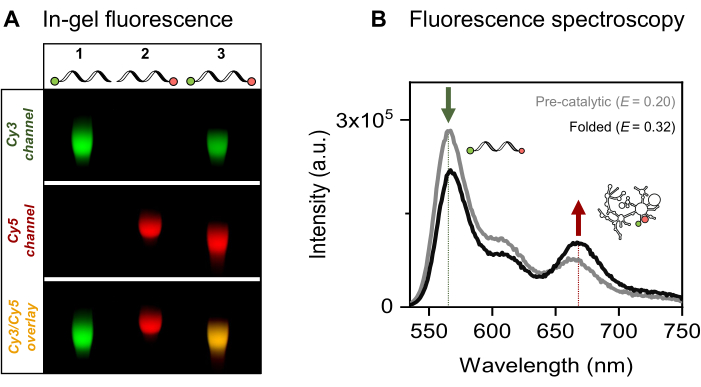
図4:RNA色素コンジュゲートの特性評価 (A)蛍光標識された単一および二重蛍光標識RNAの分析ゲルベースの分析では、2%アガロースゲル上のRNAと色素の共遊走が示されています。二重標識サンプル中の蛍光色素の共局在は、Cy3(上、緑)およびCy5(中央、赤)チャネルのマージ画像(下)の黄色の帯で示され、それぞれ532 nmおよび635 nmの照明下で視覚化されています。レーン1:5'-sCy3のみ標識RNA、レーン2:3'-Cy5のみ標識RNA、レーン3:デュアルエンド(5'-sCy3および3'-Cy5)標識RNA。(B)アンサンブル蛍光分光法により、二重標識が確認されます。ドナー励起時のエネルギー移動(λex = 515 nm、λem = 670 nm)により、両方の色素がRNAに正常に結合していることが確認されます。灰色の曲線は前触媒RNAの発光プロファイルを表し、黒色の曲線はフォールディングされたグループIIイントロンRNA(500 mM KClと70°Cで3分間インキュベートし、42°Cで5分間冷却した後、100 mM MgCl2を添加)のFRET効率の増加を示しています。この図は、Ahunbay et al.3 この図の拡大版を表示するには、ここをクリックしてください。
蛍光二重標識野生型グループIIイントロンを手にしたことで、私たちは現在、そのダイナミクスを単一分子レベルで探索する立場にあります。リン脂質小胞に封入された標識RNAは、非常に低い表面密度で顕微鏡表面に固定化され、smFRET-TIRFの単一分子分解能を達成します。 図5Aに見られるように、いくつかの個々の分子を同時に追跡できます。TIRF顕微鏡は、FRETの効率とその経時変化をリアルタイムでモニタリングすることができます。 図5B は、標識およびカプセル化されたRNAの静的および動的な単一分子FRETトレースを例示しています。一般的なダイナミックトレースは、FRET効率を変動させるドナー信号とアクセプター信号の間に反相関を示します。ドナー励起時のアクセプター放出が増加すると、それに応じてドナー放出も減少し、色素間距離のダイナミックな変化を示します。この反相関は、RNA分子の立体配座変化を示唆しています。

図5:smFRETによって明らかにされたグループIIイントロンRNAの高度に動的な挙動。 二重末端標識グループIIイントロンRNAは、リン脂質小胞にカプセル化され、目的ベースのTIRF顕微鏡でイメージングするために表面に固定化されます。(A)532 nmの励起時にドナー発光(sCy3、緑)とアクセプター発光(Cy5、赤)を示す個々の標識RNA分子の結合画像。(B)典型的なsmFRETの軌跡は、(左)ドナーとアクセプターの強度が経時的に変動しない静的RNA分子、(右)動的RNA分子で、ドナーとアクセプターの強度が逆相関し、FRETの効率は黄色です。単一分子を局在化させ、MASH-FRET17を用いて分析します。直接励起、ブリードスルー、およびγファクター補正が適用されます。略語:smFRET=単一分子フェルスター共鳴エネルギー移動。 この図の拡大版を表示するには、ここをクリックしてください。
ディスカッション
単一分子レベルのFRETは、個々の分子の観察と分析を可能にし、サンプルの不均一性を明らかにし、アンサンブル測定では不明瞭になりがちな過渡状態を捕捉できるという点で独特です1,2。smFRETを使用して個々のRNA分子を観察すると、それらのフォールディング経路とダイナミクスに関する高解像度の洞察が得られます。このプロトコルでは、RNAの化学的デュアルエンド標識とリン脂質小胞カプセル化によるその表面固定化について説明し、これらを組み合わせることでsmFRET-TIRF顕微鏡によるダイナミックなコンフォメーション変化を追跡できます。
RNAダイナミクスの研究は、新しい部位特異的な蛍光標識戦略が必要とされる、常に成長している分野です。RNA末端を標識するには、5'-リン酸をカルボジイミドで、3'-リボース糖を過ヨウ素酸塩で標的にします。これらのアプローチは、以前に説明されています(5'末端18,19および3'末端19,20,21,22)が、野生型Sc.ai5γグループIIイントロンリボザイムと同様のサイズのRNAには以前に適用されておらず、最適化が必要でした。カルボジイミド(EDCなど)による5'-リン酸の活性化は可逆的です。したがって、イミダゾールを使用して、O-アシルイソ尿素中間体と不可逆的に反応して、反応性の高いホスホリミダゾリドを形成しました19,20。しかし、より高いpHでは、カルボジイミドは核酸塩基、特にグアニンとウラシルを修飾できることが現在知られており、最近、構造プローブ剤としての使用につながっています23,24。
交差反応性を避けるためには、色素カップリングステップでpHを7.5に上げる前に、EDCから活性化RNAを精製することが重要です。しかし、活性化と色素付着物25との間に精製工程を導入したところ、非常に低い収率を得た。同様に、タンパク質標識では、表面アクセス可能なリジン残基をカルボジイミドで活性化することができます。ただし、活性化の逆転を防ぐイミダゾールの代わりに、NHSが日常的に使用されています。私たちもこの戦略を採用し、ホスホリミダゾリド中間体をNHSリン酸中間体に置き換えました。このようにして、pH制御だけでなく、低温でインキュベーション時間を短縮した標識密度の増加(37°Cで16時間かかっているのに対し、4時間で25°C)を達成しました。RNA用に開発されたこの5'標識戦略は、5'-リン酸を持つ他の一本鎖核酸に適用できます。
5'-リン酸の活性化と3'-リボースの酸化は、化学的性質が直交していないため、相互に排他的でした。この課題を克服し、交差標識を回避するために、5'末端から開始し、続いて活性化されたが標識されていない部位を阻害するブロッキングステップを経て、3'末端の標識を進めました。3'-ジオールを酸化しながら、過剰なメタ過ヨウ素酸ナトリウム(NaIO4)は、5'末端にすでに付着している蛍光色素をクエンチする可能性があります。そのため、単一標識に用いるNaIO4 の濃度を20 mMから10 mMに下げました。
反応をスケールアップする代わりに、複数のアリコートを並行して使用することをお勧めします。このプロトコルには、複数のエタノール(EtOH)沈殿ステップが必要です。複数のアリコートを並行して使用する場合は、沈殿混合物(100%絶対EtOH30 mLおよび3 M NaOAc1 mL、pH 5.2)を調製します。NaClはEtOHへの溶解性が低いため、使用されていません。この混合物の3.1容量でRNAを-20°Cで一晩インキュベートし、続いて遠心分離します。RNAペレットを氷冷した70%EtOH500μLで2回洗浄し、1回ごとに4°Cでスピンダウンし、真空下で乾燥させます。EtOH沈殿は、RNAの不溶性と遊離色素の70%エタノールへの溶解性を利用します。遠心ろ過は、RNAとのサイズ差が大きいため、遊離色素を効果的に除去し、バッファー交換を促進して塩を排除します。EtOH沈殿法および遠心ろ過法に加えて、遊離色素はゲル抽出および/またはクロマトグラフィー技術(HPLCなど)を使用して除去することもできます。ただし、スケールはそれに応じて調整する必要があります。長いRNAを再懸濁するためにボルテックスしないでください、これは機械的せん断を引き起こす可能性があるためです26。プロトコールを一時停止する最適なタイミングは、RNAがペレット化されるときです。DNA低結合チューブを使用して、核酸回収を改善します。最終反応容量は 100 μL ですが、EtOH 沈殿による精製を改善するには、1.5 mL (低容量ではない) チューブが推奨されます。
未反応の遊離色素を沈殿および遠心ろ過により除去した後、蛍光ゲル電気泳動(図4A)、UV-Vis分光法、分析HPLC3による標識を確認しました。ただし、これらの方法では、両方の蛍光色素を持つRNA分子と、それぞれが単一の色で標識されたRNAの混合物を区別できないことに注意することが重要です。同様に、RNA分子が同じ色の複数の蛍光色素を持っているかどうかを判断するために使用することはできません。質量分析はサイズ制限のため使用できません。アンサンブル3 および単一分子FRET分光法は、図4Bおよび5Bに示すように、二重蛍光標識を裏付けています。smFRET実験における0.5(sCy3とCy5の比率は1:1)の化学量論は、2つの蛍光色素の等しい結合を確認します。1つの懸念は、提案された環化の代わりに両方のアルデヒドを標識することにより、3'末端で二重に標識することでした。smFRET実験では、化学量論が0.25(sCy3とCy5の比率が1:2)の分子種が存在しないことから、色素の付着が立体的に妨げられ、第2の色素の付着が妨げられていることが示唆されています。
この二重蛍光標識により、FRETシグナルの変化は、RNAのフォールディングと触媒作用全体の構造再配列に起因する可能性があります。単一分子TIRFイメージングにおいて、蛍光標識されたRNAをエバネッセントフィールド内に維持するためには、直接的な表面テザリングよりもカプセル化が好ましいとされています。このアプローチでは、個々のRNA分子を小胞の脂質二重層内にトラップし、その動的な挙動を観察するのに役立つ制御された環境を作り出します。記載されたプロトコルは、単一ステップでの光退色が示すように、モノカプセル化を濃縮する11。RNAのフォールディングと機能を理解するためには、in vitroとin vivoのギャップを埋めることが不可欠です27。分子クラウディング剤は、細胞内の条件を模倣して、グループIIイントロンによるRNA触媒作用を増強することができる7,28。あるいは、カプセル化は、RNAの折り畳みを促進する制約のある微小環境を作り出します29、RNAの構造とダイナミクスの理解を現実的な細胞の状況に近づけます。
開示事項
著者は、利益相反を宣言しません。
謝辞
スイス国立科学財団 [200020_192153 to RKOS]、UZH Forschungskredit [FK-20-081 to EA]、UZH Stiftung für wissenschaftliche Forschung [to RKOS and SZP]、Graduate Research Campus (GRC) Short Grant [2024__SG_092 to EA]、チューリッヒ化学分子科学大学院 (CMSZH)、チューリッヒ大学からの財政的支援に感謝しています。
資料
| Name | Company | Catalog Number | Comments |
| RNA labeling | |||
| 1.5 mL DNA LoBind tubes | Eppendorf | 30108035 | |
| 1-ethyl-3-(3-dimethylaminopropyl)carbodiimide hydrochloride (EDC-HCl or EDC) | Thermo Scientific | 11844071 | Pierce EDC, No-Weigh Format. Store at -20 °C |
| 3-morpholinopropane-1-sulfonic acid (MOPS) | Sigma-Aldrich | 69947 | BioUltra, for molecular biology, ≥99.5% (titration) |
| Acetic acid (glacial) | Sigma-Aldrich | 1.00063 | 100%, anhydrous for analysis EMSURE ACS,ISO,Reag. Ph Eur |
| Centrifugal filtration unit | Sartorius | VS0132 | Vivaspin 500, MWCO 50.000, PES, 500 μL |
| Cyanine5 hydrazide (Cy5-hydrazide) | Lumiprobe | 23070 | 5 mg, CAS number 1427705-31-4 |
| Dimethyl sulfoxide (DMSO) | New England Biolabs | 12611P | Molecular biology grade |
| Ethanol | VWR Chemicals | 20821.296P | Absolute ≥99,8% |
| Glycerol | Sigma-Aldrich | G5516 | for molecular biology, ≥99.0% |
| Hydrochloric acid (HCl) | Sigma-Aldrich | 1.00317 | fuming 37%, for analysis EMSURE ACS,ISO,Reag. Ph Eur |
| N-hydroxysuccinimide (NHS) | Sigma-Aldrich | 130672 | 98% |
| Sodium acetate (NaOAc) | Sigma-Aldrich | S8750 | anhydrous, ReagentPlus, ≥99.0% |
| Sodium meta-periodate (NaIO4) | Thermo Fisher Scientific, Life Technologies | 20504 | Pierce product line |
| Sulfo-Cyanine3 amine (sCy3-amine) | Lumiprobe | 213C0 | 5 mg, CAS number 2183440-43-7 |
| Tris(hydroxymethyl)aminomethane (Tris) | Biosolve | 200923 | Molecular biology grade |
| Chamber preparation | |||
| 3-aminopropyl)triethoxysilane (APTES) | Sigma-Aldrich | 440140 | 99% |
| Acetone | Sigma-Aldrich | 1.00014 | for analysis EMSURE ACS,ISO,Reag. Ph Eur |
| Biotin-Polyethylene glycol-Succinimidyl valerate (biotin-PEG-SVA, bPEG) and Methoxy polyethylene glycol-Succinimidyl valerate (mPEG-SVA) | Laysan Bio, Inc. | BIO-PEG-SVA, MW 5,000 and MPEG-SVA, MW 5,000 - Combo Kit | |
| Coverslips | Carl Roth | H876.2 | 24 x 24 mm, 0.13-0.16 mm thickness |
| Deconex 11 universal | Borer Chemie AG | 17416492 | Laboratory glassware cleaning solution |
| Diamond coated core drill bit | Crystalite corporation | 1 mm thickness | |
| Diamond driller | |||
| Ethanol | VWR Chemicals | 20821.296P | Absolute ≥99,8% |
| Glass Coplin staining jar with cover | |||
| Imaging spacer | Grace Bio-Labs | 654008 | SecureSeal, 8 wells, 9 mm × 0.12 mm |
| Oxygen plasma cleaner | Zepto One | Diener | |
| Potassium hydroxide (KOH) | Sigma-Aldrich | P9541 | for molecular biology, ≥99.0% |
| Quartz slides | G. Finkenbeiner, Inc. | 7.5 x 2.5 cm, 1 mm thickness | |
| Sodium bicarbonate (NaHCO3) | Sigma-Aldrich | S6297 | BioXtra, 99.5-100.5% |
| Lipid cake preparation | |||
| 16:0 Biotinyl Cap PE, bPE (1,2-dipalmitoyl-sn-glycero-3-phosphoethanolamine-N-(cap biotinyl) (sodium salt), C53H98N4O11PNaS) | Avanti Polar Lipids | 870277P | Stable for 1 year at -20 °C |
| 14:0 PC, DMPC (1,2-ditetradecanoyl-sn-glycero-3-phosphocholine or 1,2-dimyristoyl-sn-glycero-3-phosphocholine, C36H72NO8P) | Avanti Polar Lipids | 850345P | |
| 2.0 mL microcentrifuge tubes, Safe-Lock | Eppendorf | 0030120094 | Autoclave to sterilize |
| 500 mL Schlenk flask | |||
| Chloroform | Merck | 1.02445 | for analysis EMSURE ACS, ISO, Reag. Ph Eur |
| Parafilm | |||
| Surface immobilization via encapsulation | |||
| (±)-6-Hydroxy-2,5,7,8-tetramethylchromane-2-carboxylic acid (Trolox®) | Thermo Fischer | 53188-07-1 | 97% |
| 3-morpholinopropane-1-sulfonic acid (MOPS) | Sigma-Aldrich | 69947 | BioUltra, for molecular biology, ≥99.5% (titration). Store at +4 °C in the dark. |
| Adhesive seal tabs | Grace Bio-Labs | 629200 | |
| Catalase | Sigma-Aldrich | 9001-05-2, C30 | from bovine liver, aqueous suspension, 10,000-40,000 units/mg protein |
| D-glucose | Sigma-Aldrich | G7528 | ≥99.5% (GC), BioXtra |
| Extruder polycarbonate (PC) membrane | Avanti Polar Lipids | 610005-1EA | 0.1 μm, 19 mm |
| Extruder set with heating block | Avanti Polar Lipids | 610000 | |
| Glucose oxidase | Sigma-Aldrich | 9001-37-0 | from Aspergillus niger, Type VII, lyophilized powder, ≥100,000 units/g solid (without added oxygen) |
| Magnesium chloride (MgCl2) | Sigma-Aldrich | 7786-30-3, M1028 | for molecular biology, 1.00 M ± 0.01 M solution |
| Polyester (PE) drain disc, membrane | Whatman, Cytiva | 230300 | 10 mm |
| Potassium chloride (KCl) | Sigma-Aldrich | 60128 | BioUltra, for molecular biology, ≥99.5% (AT) |
| Potassium hydroxide (KOH) | Sigma-Aldrich | P9541 | for molecular biology, ≥99.0% |
| Streptavidin | Thermo Fischer | 434301 |
参考文献
- Eitan, L., et al. FRET-based dynamic structural biology: Challenges, perspectives and an appeal for open-science practices. Elife. 10, e60416(2021).
- Taekjip, H., et al. Fluorescence resonance energy transfer at the single-molecule level. Nat Rev Methods Primers. 4, 22(2024).
- Ahunbay, E., Steffen, F. D., Zelger-Paulus, S., Sigel, R. K. O. Chemical dual end-labeling of large ribozymes. Methods Mol Biol. 2439, 191-204 (2022).
- Ahunbay, E., Zelger-Paulus, S., Sigel, R. K. O. Group II Introns: Highly structured yet dynamic. Chimia (Aarau). 77 (4), 235-241 (2023).
- Steiner, M., Karunatilaka, K. S., Sigel, R. K. O., Rueda, D. Single-molecule studies of group II intron ribozymes. Proc Natl Acad Sci USA. 105 (37), 13853-13858 (2008).
- Karunatilaka, K. S., Solem, A., Pyle, A. M., Rueda, D. Single-molecule analysis of Mss116-mediated group II intron folding. Nature. 467 (7318), 935-939 (2010).
- Paudel, B. P., Fiorini, E., Börner, R., Sigel, R. K. O., Rueda, D. S. Optimal molecular crowding accelerates group II intron folding and maximizes catalysis. Proc Natl Acad Sci USA. 115 (47), 11917-11922 (2018).
- Saha, R., Verbanic, S., Chen, I. A. Lipid vesicles chaperone an encapsulated RNA aptamer. Nat Commun. 9 (1), 2313(2018).
- Boukobza, E., Sonnenfeld, A., Haran, G. Immobilization in surface-tethered lipid vesicles as a new tool for single biomolecule spectroscopy. J Phys Chem B. 105 (48), 12165-12170 (2001).
- Cisse, I., Okumus, B., Joo, C., Ha, T. Fueling protein DNA interactions inside porous nanocontainers. Proc Natl Acad Sci USA. 104 (31), 12646-12650 (2007).
- Zelger-Paulus, S., Hadzic, M. C. A. S., Sigel, R. K. O., Börner, R. Encapsulation of fluorescently labeled RNAs into surface-tethered vesicles for single-molecule FRET studies in TIRF microscopy. Methods Mol Biol. 2331, 1-16 (2020).
- Okumus, B., Wilson, T. J., Lilley, D. M. J., Ha, T. Vesicle encapsulation studies reveal that single molecule ribozyme heterogeneities are intrinsic. Biophys J. 87 (4), 2798-2806 (2004).
- Ishitsuka, Y., Okumus, B., Arslan, S., Chen, K. H., Ha, T. Temperature-independent porous nanocontainers for single-molecule fluorescence studies. Anal Chem. 82 (23), 9694-9701 (2010).
- Liu, B., Mazouchi, A., Gradinaru, C. C. Trapping single molecules in liposomes: surface interactions and freeze-thaw effects. J Phys Chem B. 114 (46), 15191-15198 (2010).
- Chandradoss, S. D., et al. Surface passivation for single-molecule protein studies. J Vis Exp. (86), e50549(2014).
- Paul, T., Myong, S. Protocol for generation and regeneration of PEG-passivated slides for single-molecule measurements. STAR Protoc. 3 (1), 101152(2022).
- Hadzic, M. C. A. S., Kowerko, D., Börner, R., Zelger-Paulus, S., Sigel, R. K. O. Detailed analysis of complex single molecule FRET data with the software MASH. Imaging, Manipulation, and Analysis of Biomolecules, Cells, and Tissues IX. Farkas, D. L., Nicolau, D. V., Leif, R. C. SPIE 971119, (2016).
- Chu, B. C., Wahl, G. M. Le Orgel. Derivatization of unprotected polynucleotides. Nucleic Acids Res. 11 (18), 6513-6529 (1983).
- Qin, P. Z., Pyle, A. M. Site-specific labeling of RNA with fluorophores and other structural probes. Methods. 18 (1), 60-70 (1999).
- Whitfeld, P. R. A method for the determination of nucleotide sequence in polyribonucleotides. Biochem J. 58 (3), 390-396 (1954).
- Zamecnik, P. C., Stephenson, M. L., Scott, J. F. Partial purification of soluble RNA. Proc Natl Acad Sci USA. 46 (6), 811-822 (1960).
- Proudnikov, D., Mirzabekov, A. Chemical methods of DNA and RNA fluorescent labeling. Nucleic Acids Res. 24 (22), 4535-4542 (1996).
- Wang, P. Y., Sexton, A. N., Culligan, W. J., Simon, M. D. Carbodiimide reagents for the chemical probing of RNA structure in cells. RNA. 25 (1), 135-146 (2019).
- Mitchell, D., et al. In vivo RNA structural probing of uracil and guanine base-pairing by 1-ethyl-3-(3-dimethylaminopropyl)carbodiimide (EDC). RNA. 25 (1), 147-157 (2019).
- Rinaldi, A. J., Suddala, K. C., Walter, N. G. Native purification and labeling of RNA for single molecule fluorescence studies. Methods Mol Biol. 1240, 63-95 (2015).
- Liu, T., Patel, S., Pyle, A. M. Making RNA: Using T7 RNA polymerase to produce high yields of RNA from DNA templates. Methods in Enzymology. , Academic Press. (2023).
- Leamy, K. A., Assmann, S. M., Mathews, D. H., Bevilacqua, P. C. Bridging the gap between in vitro and in vivo RNA folding. Q Rev Biophys. 49, e10(2016).
- Fiorini, E., Börner, R., Sigel, R. K. O. Mimicking the in vivo Environment--The Effect of Crowding on RNA and Biomacromolecular Folding and Activity. Chimia. 69 (4), 207-212 (2015).
- Peng, H., Lelievre, A., Landenfeld, K., Müller, S., Chen, I. A. Vesicle encapsulation stabilizes intermolecular association and structure formation of functional RNA and DNA. Curr Biol. 32 (1), 86-96.e6 (2022).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved