Method Article
תיוג סופי פלואורסצנטי ואנקפסולציה של רנ"א ארוך למיקרוסקופ FRET-TIRF בעל מולקולה אחת
In This Article
Summary
מאמר זה מתאר את התיוג הכפול של רנ"א ארוך בעמדות טרמיני ואת אימוביליזציה של פני השטח שלהם באמצעות אנקפסולציה בשלפוחיות פוספוליפידים עבור יישומי מיקרוסקופיה FRET TIRF של מולקולה יחידה. שילוב טכניקות אלה מאפשר הדמיה מדויקת וניתוח של דינמיקת RNA ברמת המולקולה הבודדת.
Abstract
Single-molecule Förster Resonance Energy Transfer (smFRET) מצטיינת בחקר ביומולקולות דינמיות בכך שהיא מאפשרת תצפית מדויקת על שינויי הקונפורמציה שלהן לאורך זמן. כדי לנטר את דינמיקת הרנ"א באמצעות smFRET, פיתחנו שיטה לסימון קוולנטי של רנ"א בתחנה הסופית שלהם באמצעות זוג פלואורופורים מסוג FET. אסטרטגיית סימון קצה ישירה זו מכוונת להפעלת 5'-פוספט על ידי קרבודימיד (EDC)/N-הידרוקסיסוקסינימיד (NHS) ולהפעלת 3'-ריבוז על ידי חמצון תקופתי, אשר ניתן להתאים לרנ"א אחרים ללא קשר לגודלם ולרצף שלהם כדי לחקור אותם ללא תלות בשינויים מלאכותיים. יתר על כן, הפעלת 5'-EDC / NHS היא בעלת עניין כללי לכל חומצות הגרעין עם פוספט 5'. השימוש בכימיקלים זמינים מסחרית מבטל את הצורך לסנתז בדיקות ספציפיות לרנ"א.
מיקרוסקופ פלואורסצנטי של השתקפות פנימית כוללת (TIRF) דורש שהמולקולות המשותקות על פני השטח המעניינות יהיו בתוך השדה האוונסנטי כדי להיות מוארות. דרך מתוחכמת לשמור על מולקולות הרנ"א בתוך השדה האוונסנטי היא לעטוף אותן בשלפוחיות פוספוליפידים. אנקפסולציה נהנית משני העולמות, קושרת את המולקולה לפני השטח תוך שהיא מאפשרת דיפוזיה חופשית של המולקולה. אנו מוודאים שכל שלפוחית מכילה מולקולת RNA אחת בלבד, מה שמאפשר הדמיה של מולקולה אחת. לאחר תיוג דו-צדדי ואנקפסולציה של הרנ"א המעניין, מדידות smFRET מציעות תצוגה דינמית ומפורטת של התנהגות הרנ"א.
Introduction
Förster Resonance Energy Transfer (FRET) היא טכניקה רבת עוצמה ורגישה לחקר אינטראקציות בין-מולקולריות ותוך-מולקולריות של ביומולקולות בקנה מידה ננומטרי. הוא מבוסס על העברת אנרגיה לא קרינתית ממולקולת תורם מעוררת למולקולת קבלה סמוכה, המתרחשת על פני מרחקים בדרך כלל בין 1 ננומטר ל -10 ננומטר. המרחק בין הצבעים התורמים והמקבלים קובע את היעילות של העברת אנרגיה זו, מה שהופך את FRET לכלי רב ערך לחקר דינמיקה מולקולרית, שינויים קונפורמטיביים ואינטראקציות במגוון רחב של מערכות ביולוגיות 1,2, כולל RNAs. מיקרוסקופ פלואורסצנטי של השתקפות פנימית כוללת (TIRF) הוכח כטכניקה רבת עוצמה לחקירות smFET, מכיוון שהוא מאיר מולקולות באופן סלקטיבי רק קרוב לפני השטח, ומאפשר דינמיקה FRET של מולקולות בודדות עם רזולוציה מרחבית וזמנית גבוהה. עם זאת, לפני ביצוע ניסויי smFRET-TIRF, המולקולה המעניינת חייבת להיות מסומנת תחילה באופן פלואורסצנטי עם זוג FRET מתאים ולאחר מכן משותקת על משטח המיקרוסקופ. פרוטוקול smFRET-TIRF המתואר כאן אומת באמצעות אינטרון מסוג II מסוג פראי Sc.ai5γ מהמיטוכונדריה Saccharomyces cerevisiae, ומשני צדדיו שני הרצפים האקסוניים שלו (915 נוקלאוטידים)3. לקבלת תצוגה מפורטת יותר של אינטרונים מקבוצת תיוג פלואורסצנטי II והאימוביליזציה שלהם עבור מיקרוסקופ smFRET TIRF, עיין בסקירה שלנו4.
אסטרטגיית תיוג RNA אידיאלית ספציפית לאתר תאפשר שילוב מדויק של צבעי תורם ומקבל במיקומים קבועים מראש מבלי לשנות את מבנה הרנ"א או תפקודו, ותבטיח מדידות FRET מדויקות ויעילות. זה מאתגר בגלל הדמיון הכימי בין ארבעת בסיסי הגרעין, מה שמסבך תיוג סלקטיבי. סימון סופי מצמיד את צבעי התורם והמקבל לקצוות הרנ"א על ידי התמקדות ב-5'-פוספט וב-3'-ריבוז. גישה זו מציעה גישה זעיר פולשנית תוך מתן תובנות יקרות ערך לגבי דינמיקה מבנית ואינטראקציות. היכולת של אינטרון מקבוצה II לפצל את עצמו בנוכחות Mg2+ מגבילה את השימוש באנזימים תלויי יוני מתכת. כאן, אנו מציגים גישה ל-RNA (ריבוזימים) בעלי תווית קצה כפולה ארוכה ופעילה קטליטית העוקפים את הצורך באנזימים או בסינתזה של בדיקות מיוחדות.
גישה נפוצה לקשירת מולקולות RNA לפני השטח עבור מיקרוסקופ TIRF היא קישור קוולנטי של ביוטין ישירות לרנ"א או הכלאה של אוליגונוקלאוטיד אנטיסנס נושא ביוטין (ASO)5,6,7. עם זאת, שיטת אימוביליזציה ישירה זו יכולה להציג ממצאים עקב אינטראקציות RNA-פני השטח, מה שעלול לגרום לרנ"א מקופל בצורה שגויה8. פתרון אלגנטי למיתון ממצאי אימוביליזציה אלה הוא לעטוף רנ"א בשלפוחיות פוספוליפידים ננומטריותמחוברות לפני השטח 9,10,11. בועיות אלה, שקוטרם כ-100 ננומטר, מעוגנות לפני השטח באמצעות קישור ביוטין-סטרפטאבידין12,13,14, מה שמאפשר לרנ"א להתפזר בחופשיות פנימה תוך מתן אפשרות לחילופי יונים על פני קרום השומנים10. לאחר תיוג קוולנטי של RNA פונקציונלי גדול3, אנו מציגים גישה לתמצת רנ"א כזה בשלפוחיות פוספוליפידים על ידי שילוב פרוטוקולים מבוססים לפסיבציה על פני השטח ואנקפסולציית שלפוחית, המותאמים לשימור פונקציונליות RNA 10,11,14. גישה דו-סופית זו של תיוג ואנקפסולציה משיגה שיעור גבוה של מונו-אנקפסולציה של RNA פונקציונלי עבור מיקרוסקופ smFRET TIRF.
Protocol
1. תיוג קצה כפול RNA
הערה: הפרוטוקול הבא מתאר את התיוג הספציפי לאתר של רנ"א עם זוג פלואורופורים FRET על ידי חיבור קוולנטי של צבע תורם (sCy3) לפוספט 5', וצבע מקבל (Cy5) לריבוז 3'. רנ"א ארוך פעיל קטליטית, קבוצת II אינטרון ריבוזים, נבחר כ-RNA המעניין. טבלה 1 ואיור 1 מסכמים פרוטוקול תיוג דו-צדדי זה. בצע את כל השלבים הכוללים פלואורופורים בתנאים חשוכים.
| יום 0 | ▪ Aliquot 50-75 מיקרוגרם RNA לנפח כולל של 55 μL לכל צינור 1.5 מ"ל. | |||||
| יום 1 | הפעלת 5′-פוספט | |||||
| ▪ הוסף 45 μL של תמיסת EDC-NHS טרייה, pH 6.0 ל- RNA ב- ddH2O לנפח סופי של 100 μL, ערבב היטב ודגר במשך 4 שעות ב- 25 ° C ו- 500 סל"ד. | ||||||
| ▪ סבב טיהור 1: לילה משקעי EtOH. | ||||||
| יום 2 | ▪ לזרז את הרנ"א המופעל על ידי 5′, לשטוף ולייבש. | |||||
| 5′-צבע מצורף | ||||||
| ▪ השהיה מחדש ב 95 μL של 100 mM MOPS, pH 7.5. | ||||||
| ▪ הוסף 5 μL של 2 mM תמיסת צבע פונקציונלית אמין. | ||||||
| ▪ מערבבים היטב ודגרים במשך 16 שעות ב-25°C וב-500 סל"ד. | ||||||
| יום 3 | ▪ סבב טיהור 2: משקעים EtOH. | |||||
| יום 4 | ▪ לזרז את הרנ"א המופעל על ידי 5′, לשטוף ולייבש. | |||||
| שלב החסימה | ||||||
| ▪ יש להשהות מחדש ב-100 μL של 100 mM Tris-HCl, pH 7.5, ולדגור במשך שעתיים ב-25°C וב-500 סל"ד. | ||||||
| ▪ סבב טיהור 3: סינון צנטריפוגלי. | ||||||
| → Elute את ה-RNA המסומן ב-5′. | ||||||
| יום 5 | 3′-חמצון פריודאט | |||||
| ▪ לדגור על RNA עם 20 mM NaIO4 ב 50 mM NaOAc חיץ , pH 5.5 בנפח סופי של 100 μL במשך 2 שעות ב 25 ° C ו 500 סל"ד. | ||||||
| ▪ להרוות את עודפי הפריודיט: להוסיף 30 מיקרוליטר של 50% גליצרול, לערבב היטב ולדגור במשך 30 דקות ב-25°C וב-500 סל"ד. | ||||||
| ▪ סבב טיהור 4: לילה משקעי EtOH. | ||||||
| יום 6 | ▪ מזרזים את הרנ"א המחומצן 3′, שוטפים ומייבשים. | |||||
| 3′-צבע מצורף | ||||||
| ▪ השהיה מחדש ב 95 μL של 50 mM NaOAc, pH 6.0. | ||||||
| ▪ הוסף 5 μL של 2 mM תמיסת צבע פונקציונלית hydrazide. מערבבים היטב ודגרים במשך 16 שעות ב-25°C וב-500 סל"ד. | ||||||
| יום 7 | ▪ סבב טיהור 5: משקעים EtOH. | |||||
| יום 8 | ▪ לזרז את הרנ"א המסומן, לשטוף ולייבש. | |||||
| ▪ סינון צנטריפוגלי. | ||||||
| → Elute את ה-RNA הדו-צדדי המסומן. | ||||||
טבלה 1: סיכום פרוטוקול לתיוג RNA דו-קצה. אנא לחץ כאן כדי להוריד טבלה זו.
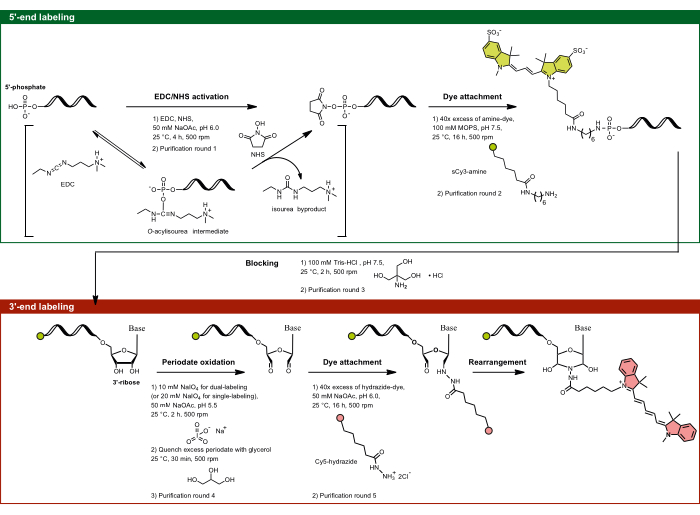
איור 1: הזרימה הניסיונית של תיוג דו-צדדי על-ידי התמקדות ב-5'-פוספט ובסוכר 3'-ריבוז. פוספט 5'מופעל באמצעות EDC בנוכחות NHS ולאחר מכן הוא משולב עם צבע פונקציונלי אמין. ה-3'-diol moiety של RNA מחומצן על ידי פעילות תקופתית לדיאלדהיד, אשר מגיב עוד יותר עם הצבע המתפקד על ידי הידרזיד. עבור תיוג כפול, חשוב להתחיל עם תיוג 5' כדי למנוע תיוג צולב, ואחריו תיוג 3' עם שלב חסימת ביניים. קיצורים: EDC = carbodiimide; NHS = N-hydroxysuccinimide; MOPS = 3-morpholinopropane-1-חומצה סולפונית; NaOAc = נתרן אצטט. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
- תיוג 5'-end: מיקוד 5'-פוספט על ידי הפעלת EDC/NHS
הערה: שיטת תיוג 5' זו חלה לא רק על RNA אלא על כל חומצת גרעין חד-גדילית המכילה פוספט 5'. הספציפיות של התגובה נשלטת על ידי תלות pH, המאפשרת רק את 5'-פוספט להיות ממוקד ספציפית לאתר, למרות נוכחותם של פוספטים מרובים בעמוד השדרה של RNA. ב-pH 6.0, התגובה הייחודית של ה-5'-פוספט כלפי קרבודימידים נובעת ממצב הפרוטונציה הספציפי שלו, שבו שני חמצן מפורקים, ואחד נשאר פרוטוני. זה הופך את פוספט 5'-פוספט לתגובתי, בעוד שהפוספטים בעמוד השדרה המפורקים במלואם נשארים לא תגובתיים, מה שמאפשר תיוג סלקטיבי של קצה 5' באמצעות מיקוד EDC/NHS.- הכן את הפתרון EDC-NHS-NaOAc, pH 6.0. יש לערבב 1.5 מ"ג של EDC ו-2.0 מ"ג של NHS לכל aliquot ב-35 μL של ddH2O ו-10 μL של 0.5 M NaOAc, pH 6.0 (pH מותאם עם חומצה אצטית קרחונית).
- Aliquot RNA עניין יש כ 50-75 מיקרוגרם RNA לכל צינור ב 55 μL של ddH2O. להוסיף 45 μL של EDC-NHS-NaOAc, pH 6.0 תערובת RNA, להגיע נפח כולל של 100 μL עם הריכוזים הסופיים של 78 mM EDC, 174 mM NHS, ו 50 mM NaOAc, pH 6.0. יש לדגור במשך 4 שעות ב-25°C תוך כדי רעידות ב-500 סל"ד.
הערה: כאן, 70 מיקרוגרם מתאים ל- 250 pmol של RNA במבחנה 915 nt ו- 2.5 μM בנפח התגובה הסופית של 100 μL. - סבב טיהור 1: לטהר את הרנ"א המופעל על ידי 5'-פוספט על ידי משקעי EtOH.
- הכינו תמיסת אמין Cyanine3 (sCy3-amine) בנפח 2 מ"מ במים.
- השהה מחדש את גלולת הרנ"א הפעילה במאגר של 95 מיקרוליטר של 100 mM 3-morpholinopropane-1-sulfonic acid (MOPS), pH 7.5 (pH מותאם עם NaOH). הוסף 5 μL של 2 mM sCy3-amine פתרון RNA מופעל. הצמידו את הפלואורופור לפוספט 5' המופעל על ידי דגירה במשך 16 שעות ב-25°C, תוך כדי רעידות ב-500 סל"ד.
הערה: יש לאחסן את מאגר המגבים בטמפרטורה של 4°C בחושך. NaOH נבחר להתאמת pH כדי לשמור על ריבוזימי אינטרון מקבוצה II לא פעיל. הפלואורופור צריך להיות לפחות פי 40 עודף של RNA. - סבב טיהור 2: הגדל את עוצמת הקול על ידי הוספת 200 μL של ddH2O כדי לשפר את ההפרדה. מטהרים את ה-RNA המסומן ב-5'-פוספט על ידי משקעי EtOH וחוזרים על הפעולה עד שהסופרנאטנט חסר צבע (בדרך כלל נדרשים שני סיבובים).
- שלב החסימה: השהה מחדש את הרנ"א ב-100 מיקרוליטר של 100 מילימטר Tris-HCl, pH 7.5, ודגר במשך שעתיים ב-25°C וב-500 סל"ד. עבור תיוג יחיד של 5 אינץ' בלבד, דלג על שלב החסימה, השעה מחדש את הרנ"א ב- ddH2O והמשך לשלב 1.1.8. עם זאת, לשם הפשטות, אם יש תיוג כפול, הפרד את פקד התווית היחידה של 5'end רק לאחר שלב 1.1.9.
הערה: שלב זה משמש לחסימת 5'-פוספטים מופעלים שלא הוצמדו לפלואורופור המתפקד על ידי אמין על ידי תגובה עם מקור אמין ראשוני קטן יחסית (למשל, Tris) כדי למזער את הסיכון של תיוג צולב עם פלואורופור מתפקד הידרזיד המשמש לפרוטוקול התיוג של 3'-end. - סבב טיהור 3: הסר את הצבעים החופשיים על ידי שטיפת ה- RNA המסומן על ידי סינון צנטריפוגלי עם סך של לפחות 10 מ"ל של ddH2O, ולאחר מכן ללוט ב- ddH2O.
הערה: החיתוך המולקולרי של המסנן צריך להיות פחות ממחצית הגודל של חומצת הגרעין. ניתן להשתמש במאגר לפי בחירה במקום ddH2O. Elution צריך לעקוב אחר הוראות היצרן, ולהבטיח שהדגימה לא תסובב ליובש מוחלט. - לקבוע את הרנ"א ולהצמיד את ריכוזי הצבע על ידי ספקטרוסקופיית UV-Vis.
- תיוג 3'-end: מיקוד הריבוז 3'-על ידי חמצון תקופתי
- Aliquot את ה-RNA המסומן ב-ddH2O כ-50-75 מיקרוגרם RNA לכל צינור ב-90 מיקרוליטר. אם נפח האלוציה של השלב הקודם היה גבוה, וכתוצאה מכך ריכוז נמוך, רכז את הרנ"א על ידי זירוז והשעיה מחדש של הגלולה ב- ddH2O. הוסף 5 μL של 1.0 M NaOAc buffer, pH 5.5 (מקביל ל- 50 mM NaOAc, pH 5.5 עבור נפח תגובה של 100 μL).
הערה: בדומה לרנ"א בעל תווית יחידה של 5 אינץ', אנו מכינים באופן שגרתי רנ"א חד-מסומן בקצה 3 כבקרה. כדי לעשות זאת, aliquot RNA unlabeled יש כ 50-75 מיקרוגרם של RNA לכל צינור ב 86 μL של ddH2O. - הוסף 4 μL של 500 mM נתרן meta-periodate טרי מוכן (NaIO4) תמיסת מלאי (מקביל 10 mM NaIO4 עבור נפח תגובה 100 μL). לתיוג יחיד של 3 אינץ', הוסף 8 μL של תמיסת מלאי NaIO4 (המקבילה ל- 20 mM NaIO4 עבור נפח תגובה של 100 μL). יש לערבב היטב ולדגור במשך שעתיים ב-25°C תוך כדי ניעור ב-500 סל"ד בתנאי חושך מכיוון ש-NaIO4 רגיש לאור.
הערה: אין לשנות את סדר ההוספה או להאריך עוד יותר את זמן הדגירה, מכיוון שהדבר עלול לגרום להלבנה של הצבע התורם המצורף. הריכוז של NaIO4 בשימוש יורד בחצי עבור תיוג דו-קצה כדי למזער את המרווה של הצבע שכבר מחובר בקצה 5'. - עצור את התגובה על ידי הוספת 30 μL של 50% גליצרול. יש לדגור במשך 30 דקות ב-25°C תוך כדי רעידות ב-500 סל"ד.
הערה: גליצרול משמש כדיול כדי להרוות את עודף המחזור. - סבב טיהור 4: הוסף 400 μL של תערובת משקעים EtOH-NaOAc קרה כקרח ובצע משקעים אתנול.
- להשהות מחדש את גלולת RNA מחומצן ב 95 μL של 50 mM NaOAc, pH 6.0.
- חיבור לצבע הידרזיד: הכינו את תמיסת הפלואורופור על ידי המסת כמה גבישים של ציאנין5 הידרזיד (Cy5-hydrazide) ב-DMSO ולאחר מכן דילול עם ddH2O לריכוז של 2 מילימול. אם הפלואורופור המועדף הוא מסיס במים, הכינו את התמיסה ב- ddH2O. הוסף 5 μL של תמיסת Cy5-hydrazide 2 mM לרנ"א המחומצן. מערבבים היטב ודגרים במשך 16 שעות ב-25°C תוך כדי ניעור ב-500 סל"ד.
הערה: הפלואורופור צריך להיות לפחות פי 40 עודף של RNA. - סבב טיהור 5: לטהר את ה-RNA בעל התווית הכפולה (או RNA המסומן ב-3') על ידי משקעים EtOH וסינון צנטריפוגלי, בדומה לשלבים 1.2.6 ו-1.2.8, בהתאמה, ו-elute ב-ddH2O.
- חישוב הרנ"א והצמדת ריכוזי הצבע על ידי ספקטרוסקופיית UV-Vis.
- אפיין את הרנ"א המסומן באמצעות בדיקות אנליטיות מבוססות ג'ל, ספקטרוסקופיה פלואורסצנטית אנסמבל (ראה סעיף תוצאות מייצגות ) ו/או HPLC אנליטי כפי שמוצג במקום אחר3.
- Aliquot את ה-RNA המסומן ב-ddH2O כ-50-75 מיקרוגרם RNA לכל צינור ב-90 מיקרוליטר. אם נפח האלוציה של השלב הקודם היה גבוה, וכתוצאה מכך ריכוז נמוך, רכז את הרנ"א על ידי זירוז והשעיה מחדש של הגלולה ב- ddH2O. הוסף 5 μL של 1.0 M NaOAc buffer, pH 5.5 (מקביל ל- 50 mM NaOAc, pH 5.5 עבור נפח תגובה של 100 μL).
2. הכנת תא מיקרופלואידי
הערה: אנו ממליצים לטפל בשישה או שמונה תאים בכל פעם. סוניקציה מבוצעת בטמפרטורת החדר, אלא אם צוין אחרת. פרוטוקול זה מגביל את השימוש בממיסים אורגניים, כגון אצטון, כדי למנוע מסיסות של זיהומים עקבות. לקבלת חלופות, עיין בהפניות 15,16.
- ניקוי
- קדחו ארבעה חורים במגלשות הקוורץ בעזרת מקדח יהלום בהתאם לסכמה שניתנה באיור 2A ליצירת שני ערוצים.
הערה: אף על פי שהן עשויות להישבר בקלות רבה יותר במהלך הקידוח, שקופיות זכוכית הן חלופה חסכונית לשקופיות קוורץ עבור מיקרוסקופ TIRF מבוסס מטרה שבו משטח הכיסוי הוא משטח ההדמיה (איור 2B). זו אינה אפשרות עבור הגדרות TIRF מבוססות מנסרה, כאשר שקופית המיקרוסקופ היא משטח ההדמיה עקב פלואורסצנטיות רקע.
הערה: לחלופין, מחזר את התאים המיקרופלואידים המשומשים16. לשם כך, טובלים את התאים המשומשים למשך הלילה באצטון (עוטפים את צנצנת הכתמים של קופלין ברדיד אלומיניום כדי למזער את האידוי) במכסה האדים, ולאחר מכן סוניק למשך 5 דקות. לפרק את החדר, ולהשליך את הכיסויים ואת המדבקות. אם הפירוק אינו פועל מיד, יש להסוניק את התא באצטון למשך 10 דקות נוספות. המשך לשלב 2.1.2. - הניחו את מגלשות הקוורץ הקדוחות ובערך פי שניים כיסויים (מכיוון שהם עלולים להישבר בקלות) בצנצנת זכוכית עם כיסוי. יש לשטוף 3x עם ddH2O. Sonicate ב-ddH2O במשך 5 דקות, ולאחר מכן לשטוף 3x עם ddH2O.
- יש לבצע סוניקציה בתמיסת ניקוי כלי זכוכית במעבדה 10% (ראו טבלת חומרים) למשך 30 דקות ב-50°C. יש לשטוף לפחות 3 פעמים עם ddH2O עד שבועות חומרי הניקוי נעלמות. סוניק ב ddH2O במשך 5 דקות. יש לשטוף 3 פעמים עם ddH2O.
- יש להסוניק בתמיסת 1 M KOH למשך 30 דקות, ואז להשאיר למשך הלילה. יש לשטוף 3 פעמים עם ddH2O. Sonicate ב-ddH2O במשך 5 דקות. יש לשטוף 3 פעמים עם ddH2O.
הערה: למרות שקורוזיה עקב דגירה מוגזמת היא דאגה שהועלתה בעבר על ידי Chandradoss et al.15, אנו ממליצים על דגירה ארוכה זו המאפשרת להימנע מתחריט פיראנה. - יבשו את המגלשות והכיסויים עם N2(g).
- יש לטפל במגלשות ובכיסויים המיובשים עם שואב פלזמה חמצן למשך 30 דקות בהתאם להוראות היצרן.
- קדחו ארבעה חורים במגלשות הקוורץ בעזרת מקדח יהלום בהתאם לסכמה שניתנה באיור 2A ליצירת שני ערוצים.
- אמינוסילניזציה
- הכן את תמיסת 3% APTES-EtOH על ידי ערבוב יסודי של 288.5 מ"ל של EtOH מוחלט, 1.5 מ"ל של ddH2O ו-9 מ"ל של (3-aminopropyl)triethoxysilane (APTES) בצלוחית Erlenmeyer של 500 מ"ל.
- הניחו את המגלשות והכיסויים הנקיים בצנצנת מכתימה של קופלין, טבלו בתמיסת APTES-EtOH 3%, עברו סוניקציה למשך דקה אחת ודגרו במשך 30 דקות.
- יש לשטוף 3x עם EtOH מוחלט, ולאחר מכן 3x עם ddH2O.
- יבשו את המגלשות והכיסויים תחת זרימת N2(g).
- פסיבציה של פני השטח וביוטינילציה
- הכינו קופסה לחה על ידי מילוי קופסת פיפטה ריקה עד חצי עם ddH2O. הניחו את המגלשות בתוך הקופסה, כשהצד לטיפול פונה כלפי מעלה.
- הכינו את תערובת bPEG-mPEG בצינור מיקרוצנטריפוגה סטרילי-נקי בנפח 1.5 מ"ל על ידי ערבוב עדין של 2 מ"ג ביוטין-פוליאתילן גליקול-סוקסינימידיל ואלרט 5000 (ביוטין-PEG-SVA) ו-80 מ"ג של מתוקסי פוליאתילן גליקול-סוקסינימידיל ואלרט (mPEG-SVA) ב-640 מיקרוליטר של 100 מילימטר נתרן ביקרבונט (NaHCO3) חוצץ, pH 8.3. צנטריפוגו את תערובת bPEG-mPEG במהירות של 16,000 × גרם למשך דקה אחת כדי להסיר בועות אוויר.
- הסר בזהירות את הסופרנאטנט והוסף טיפה של 30 μL למרכז המגלשה כדי לכסות את שני הערוצים. הניחו טיפה נוספת בצד אחד של המגלשה כדי לקבל כיסוי PEGylated נוסף, מכיוון שהם נשברים בקלות, ושימוש ברזרבי הוא שימושי. לבסוף, כסו את הטיפה בכיסוי נקי, סגרו את הקופסה הלחה ובצעו את ה-PEGylation למשך הלילה בתנאים חשוכים.
- יש לשטוף ביסודיות את השקופיות והכיסויים הפסיביים והביוטיניליים עם ddH2O. שימו לב לשינוי בהידרופוביות של המשטחים המטופלים. יבש תחת זרימת N2(g).
- הרכבת תא מיקרופלואידית
- גזור מדבקה דו-צדדית כדי ליצור את הערוצים. הצמידו את המדבקה לשקופית, וודאו שהיא מכסה את תחום העניין. הניחו בזהירות את הכיסוי למעלה, יישרו אותו כך שמשטחי ה-PEGylated יפנו זה לזה.
- הניחו כל תא מורכב בצינור צנטריפוגה של 50 מ"ל ומלאו את הצינורות ב-N2(g), ואחסנו בטמפרטורה של -20°C למשך עד חודש אחד.

איור 2: מיקרוסקופ FRET-TIRF בעל מולקולה אחת. (A) תא מיקרופלואידי להדמיית TIRF. (B) RNA המסומן בתווית FRET עטוף בשלפוחית פוספוליפידית ביוטינילית ומושתק על משטח זכוכית מצופה סטפטאווידין. זה שומר על המולקולה המעניינת בתוך השדה האוונסנטי (גרדיאנט אפור), שנוצר על ידי אור האירוע שמשתקף לחלוטין בזווית הקריטית במיקרוסקופ TIRF. כאן, שני fluorophores נרגשים לאחר מכן עם ערכת ALEX. קיצורים: FRET = Förster Resonance Energy Transfer; TIRF = פלואורסצנטיות השתקפות פנימית כוללת; ALEX = עירור לייזר חלופי. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
3. אנקפסולציית שלפוחית פוספוליפידית
- הכנת עוגת שומנים
הערה: יש לטפל תמיד בכלורופורם מתחת למכסה אדים.- באמצעות מחט סטרילית, חוררים חורים במכסה (מבפנים החוצה) של צינורות מיקרוצנטריפוגות סטריליות נקיות בנפח 2.0 מ"ל, כפי שמוצג באיור 3A.
- הכינו את תמיסת המניות 1,2-dipalmitoyl-sn-glycero-3-phosphoethanolamine-N-cap biotinyl (bPE) על ידי המסת לפחות 2 מ"ג bPE בכמות המתאימה של כלורופורם לריכוז סופי של 1 מ"ג/מ"ל.
- הכינו את תמיסת המניות 1,2-dimyristoyl-sn-glycero-3-phosphocholine (DMPC) על ידי המסת לפחות 10 מ"ג DMPC בכמות המתאימה של כלורופורם לריכוז סופי של 10 מ"ג/מ"ל.
- הכן את תערובת bPE-DMPC (תוך שמירה על יחס של 1:99 w/w) על ידי ערבוב של 100 μL של bPE ו- 990 μL של פתרונות מלאי DMPC. יש להפיץ 109 μL של תערובת bPE-DMPC לכל צינור של 2 מ"ל.
- מניחים מחיצת תא קרטון של קופסת אחסון מיקרו-צינורית בצלוחית שלנק, המשמשת כמחזיק צינור. בעזרת פינצטה ארוכה, הניחו את הצינורות המכילים את תערובת השומנים לתוך מחזיק הצינור בבקבוק שלנק בנפח 500 מ"ל (איור 3B).
הערה: ודא שהם זקופים ולא מוטים. - יש לאדות את הכלורופורם תחת זרימה נמוכה של N2(g) למשך הלילה (או למשך שעתיים לפחות עד שהממס מתאדה לחלוטין).
- אטמו את הצינורות עם Parafilm כדי לכסות את החורים. אחסנו את עוגות השומנים ב -20 °C למשך עד חודש אחד.
- אנקפסולציית RNA
- הכינו את המאגרים והתמיסות הבאים על ידי התאמת ריכוזי K+ ו- Mg2+ בהתאם למערכת הביולוגית המעניינת.
- הכן את המאגר הסטנדרטי 5x (5x SB): 2.5 M KCl, 400 mM MOPS; כוונן את ה- pH ל- 6.9 עם KOH, מסנן סטרילי, ואחסן ב- 4 ° C בחושך (בהתאמה 1x SB: 500 mM KCl, 80 mM MOPS, pH 6.9).
- הכינו את המאגר נגד מצמוץ (AB): 100 mM MgCl2, Trolox, 1x SB, מסנן סטרילי, ואחסנו ב-4°C בחושך למשך עד שבוע.
הערה: השתמש בקצה מרית של Trolox עבור נפח סופי של 10 מ"ל, מערבולת כדי לערבב, ולהתאים מחדש את ה- pH.
- הרכיבו את המכבש באמצעות קרום פוליקרבונט 100 ננומטר (PC) ודיסק ניקוז 10 מ"מ פוליאסטר (PE), אזנו את המזרק ואת הממברנות עם AB, וחממו ל-30°C (או למעשה מעל טמפרטורת המעבר של זכוכית DMPC של 24°C).
- יש לערבב 5 μL של 1 μM dual-end labeled RNA ו-45 μL של AB (לנפח כולל של 50 μL).
- לחות את עוגת השומנים עם פתרון RNA זה. ב-30°C, ערבבו במשך 5 דקות על ידי ניעור ב-1,400 סל"ד, ולאחר מכן במשך 15 דקות ב-700 סל"ד. צנטריפוגה למשך 2 דקות ב 13,000 גרם ובזהירות להעביר את supernatant לתוך צינור חדש.
- לדלל את הדגימה עם 250 μL של AB.
- ממלאים את המזרק בהשעיית השומנים ב-RNA. הבליטו פי 35 בטמפרטורה של 30°C על בלוק חימום כדי לתמצת את ה-RNA בעל התווית הכפולה בשלפוחיות פוספוליפידים בקוטר 100 ננומטר.
הערה: ניתן לאמת את התפלגות הגודל של שלפוחיות לאחר השימוש במכבש באמצעות פיזור אור דינמי (DLS), כפי שמוצג בהפניה11.
- הכינו את המאגרים והתמיסות הבאים על ידי התאמת ריכוזי K+ ו- Mg2+ בהתאם למערכת הביולוגית המעניינת.

איור 3: הכנת עוגת שומנים. (A) מכסי הצינוריות מחוררים כדי לאפשר אידוי ממס. (B) צינורות המכילים את תערובת השומנים מונחים בצלוחית שלנק, והכלורופורם מתאדה לקבלת עוגת שומנים. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
4. מיקרוסקופ smFRET-TIRF
- אימוביליזציה של פני השטח
- הכן את המאגרים והפתרונות הבאים.
- הכינו את מאגר נוגדי המצמוץ לסוכר (SAB): 1% D-גלוקוז (w/v) במסנן AB, סטרילי, ואחסנו ב-4°C בחושך למשך עד שבוע.
- הכן את 100x OSS: 100x מערכת ניקוי חמצן (OSS): 3 מ"ג של אוקסידאז גלוקוז (מקביל 1.7 U), 10 μL של קטלאז (מקביל 22 U) ב 90 μL של 1x SB, לאחסן ב 4 ° C בחושך עד 1 שבוע.
- הכן מחדש את מאגר ההדמיה (IB) על ידי ערבוב 198 μL של SAB ו- 2 μL של תמיסת OSS 100x.
- אפשרו לתא המיקרופלואידי להגיע לטמפרטורת החדר. יש לשטוף את התא פי 2 עם 200 μL של 1x SB.
- ממלאים את התא ב-50 מיקרוליטר של תמיסת סטרפטאבידין 20 מיקרוליטר/מ"ל ודגרים למשך 5 דקות.
- שטפו את התא עם 200 μL של 1x SB ולאחר מכן עם 100 μL של AB.
- לשתק את הרנ"א העטוף על פני השטח על ידי הוספת 75 μL של תרחיף שלפוחית ולדגור במשך 10 דקות.
- שטפו את התא ב-200 מיקרוליטר של IB טרי והוכן ודגרו במשך 5 דקות.
- התא מוכן כעת לאיסוף נתונים (איור 2B). כדי למנוע אידוי במהלך מדידות ארוכות, אטמו את החורים כפי שמוצג באיור 2A. אם יש חשש לדליפות מערוץ לתעלה, אטמו את התעלה שאינה בשימוש לפני תחילת פרוטוקול השתקת פני השטח.
הערה: אנו ממליצים לבצע מדידות ריקות כבדיקת איכות לניקיון התאים המיקרופלואידים ובועיות הפוספוליפידים מפני זיהומים פלואורסצנטיים.
- הכן את המאגרים והפתרונות הבאים.
תוצאות
אנו מציגים את התיוג הפלואורסצנטי היחיד והכפול הספציפי לאתר של הרנ"א 915-nt המעניין, אינטרון השמרים המיטוכונדריאלי Sc.ai5γ Group II, ומשני צדדיו רצפים אקסוניים. זוג הפלואורופורים FRET ממוקם בקצות הרנ"א באמצעות הפעלת EDC/NHS של 5'-פוספט וחמצון פריודאט של הריבוז 3', ואחריו חיבורי צבע מתאימים. לאחר מכן אימתנו את הצמידות של צבע הרנ"א באמצעות אלקטרופורזה של ג'ל פלואורסצנטי, כפי שמוצג באיור 4A. הנדידה המשותפת של RNA והפלואורופורים על ג'ל האגרוז מאשרת את התיוג המוצלח. לאחר מכן, כפי שניתן לראות באיור 4B, נעשה שימוש בספקטרוסקופיה פלואורסצנטית של אנסמבל כדי לאפיין את האינטרון מקבוצה II בעל התווית הכפולה. העברת אנרגיה, כלומר, FRET, נצפתה עם עירור של צבע התורם המוכיח את הסימון הכפול של RNA. יש לציין כי בהתאם לאופי תלוי המרחק של FRET, קיפול האינטרון RNA מקבוצה II בנוכחות יוני מתכת הוביל לעלייה ביעילות FRET, כפי שמעידה הירידה בפליטת התורם (חץ ירוק) ועלייה מקבילה בפליטת המקבל (חץ אדום). זה מצביע על כך שתיוג FRET זה עוקב אחר השינויים הקונפורמטיביים של הריבוזים.
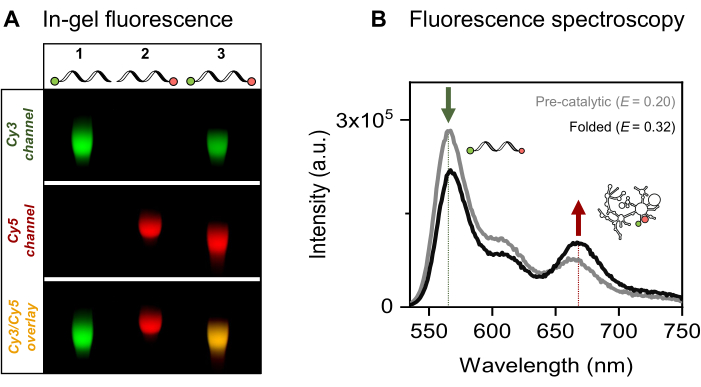
איור 4: אפיון הצמידות של צבע הרנ"א. (A) ניתוח אנליטי מבוסס ג'ל של RNA יחיד וכפול המסומן פלואורסצנטית מראה את הנדידה המשותפת של הצבעים עם הרנ"א על ג'ל אגרוז 2%. לוקליזציה משותפת של הפלואורופורים במדגם המסומן פעמיים מסומנת על ידי הפס הצהוב בתמונה הממוזגת (למטה) של ערוצי Cy3 (למעלה, ירוק) ו- Cy5 (אמצעי, אדום), המתוארים תחת תאורה של 532 ננומטר ו- 635 ננומטר, בהתאמה. נתיב 1: 5'-sCy3 מסומן רק RNA, נתיב 2: 3'-Cy5 מסומן רק RNA, ונתיב 3: קצה כפול (5'-sCy3 ו 3'-Cy5) מסומן RNA. (B) ספקטרוסקופיה פלואורסצנטית של אנסמבל מאשרת תיוג כפול. העברת אנרגיה בעירור התורם (λex = 515 ננומטר, λem = 670 ננומטר) מוודאת ששני הצבעים מצומדים בהצלחה לרנ"א. העקומה האפורה מייצגת את פרופיל הפליטה של הרנ"א הקדם-קטליטי, בעוד שהעקומה השחורה מדגימה יעילות FRET מוגברת ב-RNA אינטרון מקבוצה II המקופלת (מודגר עם 500 mM KCl ב-70°C למשך 3 דקות, מקורר ל-42°C למשך 5 דקות, ואחריו תוספת של 100 mM MgCl2). נתון זה נלקח מ Ahunbay et al.3 לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
עם האינטרון מקבוצה II בעל תווית כפולה פלואורסצנטית ביד, אנו ממוקמים כעת לחקור את הדינמיקה שלו ברמת המולקולה היחידה. לאחר שהוא עטוף בשלפוחיות פוספוליפידים, הרנ"א המסומן משותק על משטח מיקרוסקופיה בצפיפות פני שטח נמוכה מאוד כדי להשיג רזולוציה של מולקולה יחידה עבור smFRET-TIRF. כפי שניתן לראות באיור 5A, ניתן לעקוב אחר כמה מולקולות בודדות בו-זמנית. מיקרוסקופ TIRF מאפשר ניטור בזמן אמת של יעילות FRET והשינויים בה לאורך זמן. איור 5B מדגים את עקבות FRET הסטטיים והדינמיים של מולקולה אחת של הרנ"א המסומן והעטוף. מעקב דינמי טיפוסי מציג אנטי-קורלציה בין אותות התורם והמקבל המשתנים ביעילות ה- FET. כאשר פליטת המקבל בעירור התורם גדלה, פליטת התורם פוחתת בהתאם, מה שמצביע על שינוי דינמי במרחק בין הצבעים. אנטי-קורלציה זו מצביעה על שינויים קונפורמטיביים במולקולת הרנ"א.

איור 5: התנהגות דינמית מאוד של רנ"א אינטרון מקבוצה II שנחשפה על-ידי smFRET. ה-RNA אינטרון מקבוצה II המסומן בדו-קצה עטוף בשלפוחית פוספוליפידית ומושתק על פני השטח לצורך הדמיה במיקרוסקופ TIRF מבוסס מטרה. (A) תמונה ממוזגת של מולקולות RNA בודדות המסומנות כמציגות פליטת תורם (sCy3, ירוק) ופליטת מקבל (Cy5, אדום) בעירור של 532 ננומטר. (B) מסלולי smFRET טיפוסיים של (משמאל) מולקולת רנ"א סטטית שבה עוצמות התורם והמקבל אינן משתנות לאורך זמן, ומולקולת רנ"א דינמית, שבה עוצמות התורם והמקבל נוגדות קורלציה, עם נצילות FRET בצהוב. מולקולות בודדות עוברות לוקליזציה וניתוח באמצעות MASH-FRET17. עירור ישיר, דימום דרך ותיקוני גורם γ מוחלים. קיצורים: smFRET = העברת אנרגיית תהודה של מולקולה אחת Förster. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
Discussion
FRET ברמת המולקולה הבודדת הוא ייחודי משום שהוא מאפשר תצפית וניתוח של מולקולות בודדות, חושף הטרוגניות מדגם ולכידת מצבים חולפים שניתן לטשטש במדידות אנסמבל 1,2. התבוננות במולקולות RNA בודדות באמצעות smFRET מספקת תובנות ברזולוציה גבוהה לגבי מסלולי הקיפול והדינמיקה שלהן. פרוטוקול זה מתאר את הסימון הכימי הדו-צדדי של RNA ואת אימוביליזציה פני השטח שלו באמצעות אנקפסולציית שלפוחית פוספוליפידית, אשר יחד מאפשרים לעקוב אחר שינויים קונפורמטיביים דינמיים באמצעות מיקרוסקופ smFRET-TIRF.
חקר דינמיקת RNA הוא תחום הולך וגדל עם הצורך באסטרטגיות תיוג פלואורסצנטיות חדשות ספציפיות לאתר. אנו מסמנים את קצוות הרנ"א על ידי התמקדות ב-5'-פוספט עם קרבודימידים ובסוכר 3'-ריבוז עם פריודאט. גישות אלה תוארו קודם לכן (5'-end18,19 ו-3'-end 19,20,21,22) אך לא יושמו קודם לכן על RNA בגודל דומה ל-Sc.ai5γ group II intron ribozyme, שדרש אופטימיזציה. הפעלת Carbodiimide (למשל, EDC) של 5'-פוספט היא הפיכה. לכן, imidazole שימש להגיב באופן בלתי הפיך עם O-acylisourea ביניים כדי ליצור phosphorimidazolide תגובתי מאוד 19,20. עם זאת, כיום ידוע כי ב- pH גבוה יותר, carbodiimides יכולים לשנות nucleobases, במיוחד guanines ו uracils, אשר הוביל לאחרונה לשימוש בהם כמו סוכני חיטוט מבני23,24.
כדי למנוע תגובתיות צולבת, טיהור הרנ"א המופעל מה-EDC לפני העלאת ה-pH ל-7.5 בשלב צימוד הצבע הוא חיוני. עם זאת, כאשר הצגנו שלב טיהור בין ההפעלה לבין חיבור הצבע25, השגנו תפוקות נמוכות מאוד. באופן אנלוגי, בתיוג חלבונים, שאריות ליזין נגישות בפני השטח יכולות להיות מופעלות באמצעות קרבודימידים. עם זאת, במקום imidazole, אשר מונע את היפוך ההפעלה, NHS משמש באופן שגרתי. אימצנו גם אסטרטגיה זו, ובכך החלפנו את הביניים phosphorimidazolide עם מתווך NHS-פוספט. בדרך זו השגנו בקרת pH כמו גם צפיפות תיוג מוגברת בטמפרטורות נמוכות יותר וזמני דגירה קצרים יותר, כלומר, 25 ° C במשך 4 שעות לעומת 37 ° C במשך 16 שעות. אסטרטגיית תיוג 5', שפותחה עבור RNA, יכולה להיות מיושמת על כל חומצת גרעין חד-גדילית אחרת עם פוספט 5'.
הפעלת 5'-פוספט וחמצון 3'-ריבוז היו בלעדיים זה לזה, מכיוון שהכימיות אינן אורתוגונליות. כדי להתגבר על אתגר זה ולהימנע מתיוג צולב, התחלנו עם קצה 5', ואחריו שלב חסימה כדי לעכב את האתרים המופעלים אך לא מסומנים לפני שנמשיך עם התיוג של 3'-end. תוך כדי חמצון המטא-פריודט של 3'-דיול, עודף נתרן (NaIO4) יכול להרוות את הפלואורופור שכבר מחובר בקצה ה-5'. לכן, הפחתנו את הריכוז של NaIO4 המשמש לתיוג יחיד מ-20 מילימטר ל-10 מילימול.
אנו ממליצים לעבוד עם aliquots מרובים במקביל במקום להגדיל את התגובות. פרוטוקול זה דורש מספר שלבי משקעים של אתנול (EtOH). כאשר עובדים עם מספר aliquots במקביל, להכין תערובת משקעים (30 מ"ל של 100% EtOH מוחלט ו 1 מ"ל של 3 M NaOAc, pH 5.2). NaCl אינו בשימוש בשל מסיסותו הנמוכה ב- EtOH. לזרז את RNA עם 3.1 vol. של תערובת זו על ידי דגירה לילה ב -20 ° C, ואחריו צנטריפוגה. שטפו את כדורית הרנ"א פעמיים עם 500 μL קר כקרח 70% EtOH, סחררו מטה ב-4°C לאחר כל פעם, וייבשו תחת ואקום. משקעי EtOH מנצלים את חוסר המסיסות של הרנ"א ואת מסיסות הצבעים החופשיים באתנול 70%. סינון צנטריפוגלי מסיר ביעילות צבעים חופשיים בשל הבדלי הגודל המשמעותיים שלהם מהרנ"א ומקל על חילופי חיץ, תוך סילוק מלחים. בנוסף למשקעים EtOH ושיטות סינון צנטריפוגליות, ניתן להסיר צבעים חופשיים גם באמצעות מיצוי ג'ל ו / או טכניקות כרומטוגרפיה (למשל, HPLC); עם זאת, יש להתאים את קנה המידה בהתאם. אין לערבל רנ"א ארוך כדי להשהות מחדש, שכן הדבר עלול לגרום לגזירה מכנית26. הזמן האופטימלי להשהות את הפרוטוקול הוא כאשר הרנ"א הוא כדור. אנו משתמשים בצינורות DNA בעלי קישור נמוך כדי לשפר את התאוששות חומצות הגרעין. למרות שנפח התגובה הסופי הוא 100 μL, צינורות 1.5 מ"ל (ולא נפח נמוך יותר) מועדפים לטיהור טוב יותר על ידי משקעי EtOH.
לאחר שהצבעים החופשיים שלא הגיבו הוסרו על-ידי משקעים וסינון צנטריפוגלי, אישרנו את הסימון על-ידי אלקטרופורזה של ג'ל פלואורסצנטי (איור 4A), ספקטרוסקופיית UV-Vis, HPLC3 אנליטי. עם זאת, חשוב לציין כי שיטות אלה אינן יכולות להבחין בין מולקולת רנ"א הנושאת פלואורופורים ותערובת של רנ"א, שכל אחד מהם מסומן בצבע אחד. באופן דומה, לא ניתן להשתמש בהם כדי לקבוע אם מולקולת רנ"א נושאת פלואורופורים מרובים מאותו צבע. לא ניתן להשתמש בספקטרומטריית מסות עקב מגבלות גודל. ספקטרוסקופיית אנסמבל3 וספקטרוסקופיית FRET של מולקולה בודדת מאששות תיוג פלואורסצנטי כפול, כפי שמוצג באיורים 4B ו-5B. הסטויכיומטריה של 0.5 (יחס sCy3 ל-Cy5 של 1:1) בניסויי smFRET מאשרת את הצמידות השווה של שני הפלואורופורים. אחד החששות היה הסימון הכפול בקצה ה-3 על ידי סימון שני האלדהידים במקום המחזוריות המוצעת. היעדר מינים עם סטויכיומטריה של 0.25 (יחס sCy3 ל-Cy5 של 1:2) בניסויי smFRET מצביע על כך שחיבור הצבע מעכב באופן סטרילי ומונע התקשרות של צבע שני.
עם תיוג פלואורסצנטי כפול זה, ניתן לייחס שינויים באות FRET לסידורים מבניים מחדש במהלך קיפול RNA וקטליזה. כדי לשמור על RNA מסומן פלואורסצנטית בשדה האוונסנטי עבור הדמיית TIRF של מולקולה יחידה, אנקפסולציה עדיפה על פני קשירה ישירה של פני השטח. גישה זו כוללת לכידת מולקולות RNA בודדות בתוך דו-שכבות שומנים של שלפוחיות, יצירת סביבה מבוקרת התורמת להתבוננות בהתנהגותן הדינמית. הפרוטוקול המתואר מעשיר מונו-אנקפסולציה, כפי שמדגים פוטו-הלבנה בשלב אחד11. כדי להבין את קיפול הרנ"א ואת תפקודו, גישור על הפער במבחנה ו-in vivo הוא חיוני27. חומרי צפיפות מולקולרית יכולים לחקות את התנאים בתוך התאים כדי לשפר את קטליזה RNA על ידי אינטרונים מקבוצה II 7,28. לחלופין, אנקפסולציה יוצרת מיקרו-סביבות מוגבלות המקדמות קיפול RNA29, ומקרבות את הבנתנו את מבנה הרנ"א ואת הדינמיקה שלו להקשר תאי מציאותי.
Disclosures
המחברים מצהירים כי אין ניגודי עניינים.
Acknowledgements
תמיכה כספית מהקרן הלאומית השוויצרית למדע [200020_192153 ל- RKOS], UZH Forschungskredit [FK-20-081 ל- EA], קרן UZH für wissenschaftliche Forschung [ל- RKOS ו- SZP], מענק קצר של קמפוס המחקר לתארים מתקדמים (GRC) [2024__SG_092 ל- EA], בית הספר למוסמכים במדעי הכימיה והמולקולרית ציריך (CMSZH) ואוניברסיטת ציריך מוכרים בהכרת תודה.
Materials
| Name | Company | Catalog Number | Comments |
| RNA labeling | |||
| 1.5 mL DNA LoBind tubes | Eppendorf | 30108035 | |
| 1-ethyl-3-(3-dimethylaminopropyl)carbodiimide hydrochloride (EDC-HCl or EDC) | Thermo Scientific | 11844071 | Pierce EDC, No-Weigh Format. Store at -20 °C |
| 3-morpholinopropane-1-sulfonic acid (MOPS) | Sigma-Aldrich | 69947 | BioUltra, for molecular biology, ≥99.5% (titration) |
| Acetic acid (glacial) | Sigma-Aldrich | 1.00063 | 100%, anhydrous for analysis EMSURE ACS,ISO,Reag. Ph Eur |
| Centrifugal filtration unit | Sartorius | VS0132 | Vivaspin 500, MWCO 50.000, PES, 500 μL |
| Cyanine5 hydrazide (Cy5-hydrazide) | Lumiprobe | 23070 | 5 mg, CAS number 1427705-31-4 |
| Dimethyl sulfoxide (DMSO) | New England Biolabs | 12611P | Molecular biology grade |
| Ethanol | VWR Chemicals | 20821.296P | Absolute ≥99,8% |
| Glycerol | Sigma-Aldrich | G5516 | for molecular biology, ≥99.0% |
| Hydrochloric acid (HCl) | Sigma-Aldrich | 1.00317 | fuming 37%, for analysis EMSURE ACS,ISO,Reag. Ph Eur |
| N-hydroxysuccinimide (NHS) | Sigma-Aldrich | 130672 | 98% |
| Sodium acetate (NaOAc) | Sigma-Aldrich | S8750 | anhydrous, ReagentPlus, ≥99.0% |
| Sodium meta-periodate (NaIO4) | Thermo Fisher Scientific, Life Technologies | 20504 | Pierce product line |
| Sulfo-Cyanine3 amine (sCy3-amine) | Lumiprobe | 213C0 | 5 mg, CAS number 2183440-43-7 |
| Tris(hydroxymethyl)aminomethane (Tris) | Biosolve | 200923 | Molecular biology grade |
| Chamber preparation | |||
| 3-aminopropyl)triethoxysilane (APTES) | Sigma-Aldrich | 440140 | 99% |
| Acetone | Sigma-Aldrich | 1.00014 | for analysis EMSURE ACS,ISO,Reag. Ph Eur |
| Biotin-Polyethylene glycol-Succinimidyl valerate (biotin-PEG-SVA, bPEG) and Methoxy polyethylene glycol-Succinimidyl valerate (mPEG-SVA) | Laysan Bio, Inc. | BIO-PEG-SVA, MW 5,000 and MPEG-SVA, MW 5,000 - Combo Kit | |
| Coverslips | Carl Roth | H876.2 | 24 x 24 mm, 0.13-0.16 mm thickness |
| Deconex 11 universal | Borer Chemie AG | 17416492 | Laboratory glassware cleaning solution |
| Diamond coated core drill bit | Crystalite corporation | 1 mm thickness | |
| Diamond driller | |||
| Ethanol | VWR Chemicals | 20821.296P | Absolute ≥99,8% |
| Glass Coplin staining jar with cover | |||
| Imaging spacer | Grace Bio-Labs | 654008 | SecureSeal, 8 wells, 9 mm × 0.12 mm |
| Oxygen plasma cleaner | Zepto One | Diener | |
| Potassium hydroxide (KOH) | Sigma-Aldrich | P9541 | for molecular biology, ≥99.0% |
| Quartz slides | G. Finkenbeiner, Inc. | 7.5 x 2.5 cm, 1 mm thickness | |
| Sodium bicarbonate (NaHCO3) | Sigma-Aldrich | S6297 | BioXtra, 99.5-100.5% |
| Lipid cake preparation | |||
| 16:0 Biotinyl Cap PE, bPE (1,2-dipalmitoyl-sn-glycero-3-phosphoethanolamine-N-(cap biotinyl) (sodium salt), C53H98N4O11PNaS) | Avanti Polar Lipids | 870277P | Stable for 1 year at -20 °C |
| 14:0 PC, DMPC (1,2-ditetradecanoyl-sn-glycero-3-phosphocholine or 1,2-dimyristoyl-sn-glycero-3-phosphocholine, C36H72NO8P) | Avanti Polar Lipids | 850345P | |
| 2.0 mL microcentrifuge tubes, Safe-Lock | Eppendorf | 0030120094 | Autoclave to sterilize |
| 500 mL Schlenk flask | |||
| Chloroform | Merck | 1.02445 | for analysis EMSURE ACS, ISO, Reag. Ph Eur |
| Parafilm | |||
| Surface immobilization via encapsulation | |||
| (±)-6-Hydroxy-2,5,7,8-tetramethylchromane-2-carboxylic acid (Trolox®) | Thermo Fischer | 53188-07-1 | 97% |
| 3-morpholinopropane-1-sulfonic acid (MOPS) | Sigma-Aldrich | 69947 | BioUltra, for molecular biology, ≥99.5% (titration). Store at +4 °C in the dark. |
| Adhesive seal tabs | Grace Bio-Labs | 629200 | |
| Catalase | Sigma-Aldrich | 9001-05-2, C30 | from bovine liver, aqueous suspension, 10,000-40,000 units/mg protein |
| D-glucose | Sigma-Aldrich | G7528 | ≥99.5% (GC), BioXtra |
| Extruder polycarbonate (PC) membrane | Avanti Polar Lipids | 610005-1EA | 0.1 μm, 19 mm |
| Extruder set with heating block | Avanti Polar Lipids | 610000 | |
| Glucose oxidase | Sigma-Aldrich | 9001-37-0 | from Aspergillus niger, Type VII, lyophilized powder, ≥100,000 units/g solid (without added oxygen) |
| Magnesium chloride (MgCl2) | Sigma-Aldrich | 7786-30-3, M1028 | for molecular biology, 1.00 M ± 0.01 M solution |
| Polyester (PE) drain disc, membrane | Whatman, Cytiva | 230300 | 10 mm |
| Potassium chloride (KCl) | Sigma-Aldrich | 60128 | BioUltra, for molecular biology, ≥99.5% (AT) |
| Potassium hydroxide (KOH) | Sigma-Aldrich | P9541 | for molecular biology, ≥99.0% |
| Streptavidin | Thermo Fischer | 434301 |
References
- Eitan, L., et al. FRET-based dynamic structural biology: Challenges, perspectives and an appeal for open-science practices. Elife. 10, e60416 (2021).
- Taekjip, H., et al. Fluorescence resonance energy transfer at the single-molecule level. Nat Rev Methods Primers. 4, 22 (2024).
- Ahunbay, E., Steffen, F. D., Zelger-Paulus, S., Sigel, R. K. O. Chemical dual end-labeling of large ribozymes. Methods Mol Biol. 2439, 191-204 (2022).
- Ahunbay, E., Zelger-Paulus, S., Sigel, R. K. O. Group II Introns: Highly structured yet dynamic. Chimia (Aarau). 77 (4), 235-241 (2023).
- Steiner, M., Karunatilaka, K. S., Sigel, R. K. O., Rueda, D. Single-molecule studies of group II intron ribozymes. Proc Natl Acad Sci USA. 105 (37), 13853-13858 (2008).
- Karunatilaka, K. S., Solem, A., Pyle, A. M., Rueda, D. Single-molecule analysis of Mss116-mediated group II intron folding. Nature. 467 (7318), 935-939 (2010).
- Paudel, B. P., Fiorini, E., Börner, R., Sigel, R. K. O., Rueda, D. S. Optimal molecular crowding accelerates group II intron folding and maximizes catalysis. Proc Natl Acad Sci USA. 115 (47), 11917-11922 (2018).
- Saha, R., Verbanic, S., Chen, I. A. Lipid vesicles chaperone an encapsulated RNA aptamer. Nat Commun. 9 (1), 2313 (2018).
- Boukobza, E., Sonnenfeld, A., Haran, G. Immobilization in surface-tethered lipid vesicles as a new tool for single biomolecule spectroscopy. J Phys Chem B. 105 (48), 12165-12170 (2001).
- Cisse, I., Okumus, B., Joo, C., Ha, T. Fueling protein DNA interactions inside porous nanocontainers. Proc Natl Acad Sci USA. 104 (31), 12646-12650 (2007).
- Zelger-Paulus, S., Hadzic, M. C. A. S., Sigel, R. K. O., Börner, R. Encapsulation of fluorescently labeled RNAs into surface-tethered vesicles for single-molecule FRET studies in TIRF microscopy. Methods Mol Biol. 2331, 1-16 (2020).
- Okumus, B., Wilson, T. J., Lilley, D. M. J., Ha, T. Vesicle encapsulation studies reveal that single molecule ribozyme heterogeneities are intrinsic. Biophys J. 87 (4), 2798-2806 (2004).
- Ishitsuka, Y., Okumus, B., Arslan, S., Chen, K. H., Ha, T. Temperature-independent porous nanocontainers for single-molecule fluorescence studies. Anal Chem. 82 (23), 9694-9701 (2010).
- Liu, B., Mazouchi, A., Gradinaru, C. C. Trapping single molecules in liposomes: surface interactions and freeze-thaw effects. J Phys Chem B. 114 (46), 15191-15198 (2010).
- Chandradoss, S. D., et al. Surface passivation for single-molecule protein studies. J Vis Exp. (86), e50549 (2014).
- Paul, T., Myong, S. Protocol for generation and regeneration of PEG-passivated slides for single-molecule measurements. STAR Protoc. 3 (1), 101152 (2022).
- Hadzic, M. C. A. S., Kowerko, D., Börner, R., Zelger-Paulus, S., Sigel, R. K. O., Farkas, D. L., Nicolau, D. V., Leif, R. C. Detailed analysis of complex single molecule FRET data with the software MASH. Imaging, Manipulation, and Analysis of Biomolecules, Cells, and Tissues IX. SPIE 971119, (2016).
- Chu, B. C., Wahl, G. M. Le Orgel. Derivatization of unprotected polynucleotides. Nucleic Acids Res. 11 (18), 6513-6529 (1983).
- Qin, P. Z., Pyle, A. M. Site-specific labeling of RNA with fluorophores and other structural probes. Methods. 18 (1), 60-70 (1999).
- Whitfeld, P. R. A method for the determination of nucleotide sequence in polyribonucleotides. Biochem J. 58 (3), 390-396 (1954).
- Zamecnik, P. C., Stephenson, M. L., Scott, J. F. Partial purification of soluble RNA. Proc Natl Acad Sci USA. 46 (6), 811-822 (1960).
- Proudnikov, D., Mirzabekov, A. Chemical methods of DNA and RNA fluorescent labeling. Nucleic Acids Res. 24 (22), 4535-4542 (1996).
- Wang, P. Y., Sexton, A. N., Culligan, W. J., Simon, M. D. Carbodiimide reagents for the chemical probing of RNA structure in cells. RNA. 25 (1), 135-146 (2019).
- Mitchell, D., et al. In vivo RNA structural probing of uracil and guanine base-pairing by 1-ethyl-3-(3-dimethylaminopropyl)carbodiimide (EDC). RNA. 25 (1), 147-157 (2019).
- Rinaldi, A. J., Suddala, K. C., Walter, N. G. Native purification and labeling of RNA for single molecule fluorescence studies. Methods Mol Biol. 1240, 63-95 (2015).
- Liu, T., Patel, S., Pyle, A. M. Making RNA: Using T7 RNA polymerase to produce high yields of RNA from DNA templates. Methods in Enzymology. , (2023).
- Leamy, K. A., Assmann, S. M., Mathews, D. H., Bevilacqua, P. C. Bridging the gap between in vitro and in vivo RNA folding. Q Rev Biophys. 49, e10 (2016).
- Fiorini, E., Börner, R., Sigel, R. K. O. Mimicking the in vivo Environment--The Effect of Crowding on RNA and Biomacromolecular Folding and Activity. Chimia. 69 (4), 207-212 (2015).
- Peng, H., Lelievre, A., Landenfeld, K., Müller, S., Chen, I. A. Vesicle encapsulation stabilizes intermolecular association and structure formation of functional RNA and DNA. Curr Biol. 32 (1), 86-96.e6 (2022).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved