Method Article
In vitroユビキチン鎖切断アッセイによる神経発達障害関連脱ユビキチン化酵素の酵素活性の測定
要約
このプロトコルでは、組換えタンパク質精製とユビキチン鎖切断アッセイを組み合わせることにより、神経発達障害に関連する遺伝的変異が脱ユビキチン化酵素活性に及ぼす影響を測定する方法を説明しています。
要約
神経発達障害(NDD)は、神経系機能の障害と関連していますが、分子レベルではよく理解されていません。単一の遺伝子によって引き起こされる離散的な障害は、非定型神経発達を促進するメカニズムを調査するためのモデルを提供します。脱ユビキチン化酵素(DUB)ファミリータンパク質をコードする遺伝子の変異体は、いくつかのNDDと関連していますが、これらの遺伝子変異体によって引き起こされる障害の病原性メカニズムを決定する必要があります。DUB活性に対する遺伝子変異体の影響は、基質に依存しない in vitro ユビキチン切断アッセイを用いて実験的に決定することができます。このアッセイでは、触媒活性を直接測定するために下流の基質に関する知識は必要ありません。ここでは、遺伝子変異体が酵素活性に及ぼす影響を決定するためのプロトコルは、X連鎖性知的障害105(XLID105)で変異したDUB Ubiquitin Specific Protease 27, X-linked(USP27X)を用いてモデル化される。この実験パイプラインは、DUB遺伝子の変異によって引き起こされる神経発達障害の根底にあるメカニズムを明らかにするために使用できます。
概要
神経発達障害(NDD)は、非定型神経系の発達を促進する環境的または遺伝的決定要因を持つさまざまな病因から発生します1。次世代シーケンシング遺伝子検査により、ユビキチン系関連遺伝子の変異体が遺伝的NDDと関連して増加していることが明らかになりました2。ユビキチンシステムは、低分子タンパク質修飾因子ユビキチンが主にタンパク質基質のリジン残基にライゲーションするのを触媒し、局在化、安定性、タンパク質間相互作用、活性などの細胞挙動の変化を促進します3。ユビキチン化は、E1活性化酵素、E2結合酵素、およびE3リガーゼ酵素4 によって媒介され、タンパク質基質5からのユビキチンの切断と除去を触媒する脱ユビキチン化酵素(DUB)の活性によって可逆的である。ユビキチンは、7つのリジン残基(K6、K11、K27、K29、K33、K48、K63)またはユビキチンのM1残基のいずれかに形成されるモノマー(モノユビキチン化)またはポリマー鎖(ポリユビキチン化)として基質にライゲーションすることができます。これらの異なるユビキチン鎖トポロジーとその組み合わせは、シグナル伝達の鍵となる細胞コードを作り出す6。
USP27X、USP7、USP9X、USP48、STAMBP、OTUD4、OTUD6B、OTUD5 などの DUB は、NDD2、7、8、9、10、11 に関連付けられています。ほとんどのNDDでは、病因を引き起こす分子メカニズムは実験的に未定義のままです。最近報告された疾患を引き起こすDUBの一部は、十分に理解されておらず、タンパク質機能に対する遺伝的変異の影響を評価するために使用できる既知の細胞測定値が不足しています。 In vitroでは、ユビキチン鎖切断アッセイは、酵素活性に対する遺伝子変異体の影響を測定できる基質に依存しないDUB活性の読み出しとして、この制限を克服します12。
In vitroユビキチン切断アッセイは、1980年代から使用されてきました。放射性標識基質を用いたこれらのアッセイにより、ヒストンH2Aを脱ユビキチン化する能力が同定されたイソペプチダーゼ13や、さまざまな化学コンジュゲートからユビキチンを加水分解する能力によって同定されたユビキチンカルボキシル末端加水分解酵素(UCH)を含む最初のDUBの発見が可能になった14,15,16.さらに、放射性標識されたポリユビキチン化完全長タンパク質またはペプチドを使用して、赤血球および骨格筋からイソペプチダーゼTおよびいくつかのUCHおよびユビキチン特異的プロテアーゼ(UBP)をそれぞれ同定した17,18,19,20。定義された長さと結合タイプのユビキチン鎖(K48結合テトラユビキチン)を最初に使用して、イソペプチダーゼT21のDUB活性を測定しました。それ以来、このアッセイは、突然変異解析におけるDUB活性を測定するためのゴールドスタンダードとなっています22,23。このアッセイの改良により、現在、電気泳動および従来のゲル染色(Coomassie blue、SYPRO Orange、Ruby、Silver染色、または蛍光または免疫ブロッティングベースの検出)によるユビキチン切断の可視化が可能となっている12,24。最小鎖長や連鎖特異性25,26,27,28などのDUB活性の分子的側面は、異なる長さのユビキチン鎖(例えば、ジユビキチン、トリユビキチン、トリユビキチン、テトラユビキチンなど)および連鎖(K6、K11、K27、K29、K33、K48、K63、直鎖)を機能アッセイで使用することで明らかにすることができます。NDD関連変異体は、ユビキチン鎖結合タイプ特異的なDUB活性欠損を引き起こす可能性がある11。
精製された組換えDUBタンパク質を用いたジユビキチン切断アッセイは、NDDバリアントがDUB活性に及ぼす影響を直接測定することができます。NDD X関連知的障害障害105(XLID105)7,28で変異するUSP27Xは、ここで提示したプロセスをモデル化しています。このアプローチにより、既存および未知のDUB関連NDDの遺伝子変異によってDUB活性がどのように破壊されるかを決定できます。
プロトコル
以下のプロトコルは、コンピテントセルの異なる株で発現する種々のアフィニティータグを用いた組換えタンパク質に適合させることができます。発現するタンパク質によっては、培養条件および一晩の発現条件で、発現誘導、発現時間、発現温度、およびIPTG濃度でのOD600 の最適化が必要になる場合があります。プロトコルの概要を 図 1 に示します。本試験で使用した試薬および装置の詳細は、 材料表に記載されています。
1. コンピテントRosetta 2 E. coli細胞の組換えGST-USP27X発現プラスミドによる形質転換
注:細菌培養物の無菌性を維持するために、培地容器がブンゼンバーナー炎の下で開いている場所で手順を実行してください。最適な酸素移動を可能にするために、軌道19mmから50mm、速度200rpm29のベンチトップ温度制御シェーカーで細菌培養振とうを行います。
- 使用直前に、化学的にコンピテントな Rosetta 2 E. coli 細胞20 μLを氷上で解凍します。pGEX6P1-USP27X発現プラスミド(N末端グルタチオン-S-トランスフェラーゼ(GST)30 タグ付きUSP27Xをコードし、アンピシリン耐性を含む)を1-10 ng加え、氷上で5分間インキュベートします。
- ドライバスで42°Cで30秒間の熱衝撃。氷上で2分間インキュベートします。
- 80 μLの室温(RT)SOC培地を加えます。19 mm軌道ベンチトップ温度制御シェーカーで、200 rpm回転、37°Cで60分間インキュベートします。
- 25 μg/L のクロラムフェニコールと 50 μg/L のアンピシリンを添加した LB 寒天プレートで 50 μL の培養物をプレートします。温度制御されたインキュベーターで、蓋を下にして37°Cで20時間インキュベートします。
2. 発現プラスミドからの組換えタンパク質の一晩の細菌発現
注:細菌培養物の無菌性を維持するために、培地容器がブンゼンバーナー炎の下で開いている場所で手順を実行します。最適な酸素移動を可能にするために、軌道19mmから50mm、速度200rpm29のベンチトップ温度制御シェーカーで細菌培養振とうを行います。分光光度計を使用して培養OD600 を測定します。
- 2 Lのバッフル培養フラスコに47.6 gのTBパウダーを0.4%グリセロールを含む超純水1 Lに加えて、1 Lのテリフィックブロス(TB)培地を準備します。オートクレーブ培地を30分間放置し、室温まで冷却します。
- 形質転換細菌のコロニーを1つ選び、25 μg/Lのクロラムフェニコールと50 μg/Lのアンピシリンを添加した滅菌LB培地10 mLに加えます。19 mm軌道ベンチトップ温度制御シェーカーで、200 rpm回転、37°Cで20時間インキュベートします。
- 25 μg/L クロラムフェニコールと 50 μg/L アンピシリンを添加した結核培地 1 L に 10 mL を一晩培養します (開始培養と発現培養の比率は 1:100)。19 mm軌道ベンチトップ温度制御シェーカーで、培養OD600 が0.5〜0.6になるまで、200 rpm回転で37°Cでインキュベートします。
- 50 μMのIPTGを培養物に加えて発現を誘導し、16°Cまで冷却し、200 rpm回転させて16°Cで20時間インキュベートします。
- 発現培養液の細胞を≥3,000 x g 、4°Cで20分間遠心分離してペレット化します。 ペレットを-80°Cで少なくとも1時間保存します。
注:この時点で、実験を一時停止し、後で(できれば同じ週に)再開できます。ペレットは-80°Cで長期間保存できます。
3. 重力流アフィニティーカラムによるタンパク質精製
注:各精製に適したレジン、結合、洗浄、溶出、および保存バッファーは、精製する組換えタンパク質によって異なります。細胞ペレット、上清、フロースルー、洗浄画分、および溶出画分からSDS-PAGEバッファーにサンプルを採取します。サンプルのSDS-PAGEおよびCoomassie染色を実施して、精製の成功を評価します。4°Cで精製を行い、氷上でフラクションを処理します。タンパク質タグの切断は、適切なプロテアーゼを使用してカラム内外で実施し、関連するプロテアーゼ特異的切断部位を標的とすることができます。
- 空の重力フローカラムをレトルトスタンドに固定し、グルタチオンアガロース樹脂を充填します。2〜3mLの樹脂を使用して、1Lの発現培養物から採取した細胞ペレットを精製します。
- カラムを 20% エタノールの 1 つのレジンベッド容量で洗浄します。1つのレジンベッド容量のMS500バッファー(20 mMのTris pH 7.5、500 mMのNaCl、0.5 mMのTCEP)でカラムを3回洗浄します。セルペレットの調製中に乾燥を防ぐために、レジンがバッファーで覆われたままになるようにカラムを停止します。
- 細胞ペレットを4°Cで解凍します。 融解したペレットにMS500バッファー30mL(20 mMのTris pH 7.5、500 mMのNaCl、0.5 mMのTCEP、60 mgのリゾチーム、および1つのプロテアーゼ阻害剤錠剤)を加えます。4°Cで30分間、端から端まで穏やかに回転させながら溶解します。
- 50 mLの遠心分離チューブまたは氷上の金属ビーカーで溶解した細胞を、ピペットチップから分注したときにライセートが自由に流れるまで超音波処理します。4°Cで30分間遠心分離し、≥20,000 x g で上清を除去します。
注:ソニケーターの設定を経験的に決定します。粘性溶解物を自由流動性で透明な液体に減少させるには合計120秒で十分になるように超音波発生器を設定します。 - 上清をビーカーにデカントし、透明化したライセートをカラムにロードします。ライセートを重力流でカラムに流し、流れを回収します。収集されたフローが通過する状態でカラムをロードし、カラムに通します。
- 少なくとも 2 つのレジンベッド容量の MS500 洗浄バッファー(20 mM の Tris pH 7.5、500 mM の NaCl、0.5 mM の TCEP)でカラムを 5 回洗浄します。洗浄フローを5 mLのフラクションで収集します。各画分1 μLを100 μLのBradford試薬に加えて、タンパク質の存在を視覚的に確認します。結合していないタンパク質が最後の洗浄で存在しなくなるまで洗浄し、必要に応じて追加の洗浄ステップを追加します。
- MS500溶出バッファー(MS500バッファーに10 mMのグルタチオンと10 mMのNaOHを添加)をカラムに通し、5 mLの溶出画分を回収してタンパク質を回収します。100 μLのBradford試薬に1 μLの溶出液を添加して、タンパク質の存在を確認します。Bradford試薬がタンパク質の存在を示さなくなったら、フラクションの収集を停止します。
- 沈殿および遠心分離により緩衝液交換を行います。溶出液に2容量の4 M硫酸アンモニウムを添加し、曇るまで穏やかに反転させた後、4°Cで30分間、≥20,000 x g で遠心分離し、さらに5分間遠心分離してタンパク質を沈殿させます。各遠心分離後は、タンパク質ペレットを乱さずに上清を除去してください。
- タンパク質を25%グリセロールを添加したMS500に再溶解して保存します。タンパク質ペレットまたはタンパク質を-80°Cで保存バッファーに保存します。
4. in vitro ユビキチン鎖切断アッセイ
注:ユビキチン鎖の長さと結合タイプは、以前のレポート31 に記載されている、または経験的に決定されたDUB特異性に基づいて選択してください。必要であれば、このプロトコルを使用して、定義された長さおよび連鎖型の市販のユビキチン鎖のパネル上で、目的の野生型DUBの活性を試験することができる。375〜750ngのジ−ユビキチン鎖量および1〜2μMのDUB濃度は、アッセイ27の開始点として使用することができる。
- 10x DUB活性化バッファー(500 mMのTris-HCl pH 7.5、500 mMのNaCl、100 mMのTCEP)を調製します。
- 各DUBの各時点について、1x DUB活性化バッファー(DUBミックス)に合計2μMの精製GST-USP27Xを10μL調製します。マスターミックスを準備し、それらを時点に分割します。
- DUBミックスをRTで10分間インキュベートします。
- ユビキチン鎖を添加する前に、7 μLのSDS-PAGEローディングバッファーを時間0に添加して、脱ユビキチン化反応の開始を防ぎます。
- 各DUBの各時点に、10 μL 1x DUB活性化バッファーで希釈した375 ngのK-63ジユビキチン鎖を添加します。総容量は反応あたり20μLです。
- チューブを30°Cでインキュベートし、7 μLのSDS-PAGEローディングバッファーで各点を停止します。
- 4%-12%グラジエントゲルとユビキチンおよびUSP27Xのイムノブロット7,32を用いてSDS-PAGEを実施し、選択した時点におけるモノユビキチンの存在の変化を解析します。

図1:研究デザインの概略図(A)組換えタンパク質発現プラスミドによるコンピテント大腸菌細胞の形質転換。(B)組換えデユビキチル化酵素タンパク質の一晩の細菌発現。(C)重力流アフィニティーカラムを用いた組換えデユビキチル化酵素のタンパク質精製。(D)脱ユビキチン化活性を評価するためのin vitroユビキチン鎖切断アッセイ。この図の拡大版を表示するには、ここをクリックしてください。
結果
XLID105随変異体がUSP27X触媒活性に及ぼす影響を調べるため、GSTタグ付き野生型USP27XとXLID105随伴変異体F313V、Y381H、およびS404N USP27Xタンパク質を細菌から精製しました。これらの変異体は、USP27XのUSP触媒ドメイン内に位置しています(図2A)。USP27Xは以前にK63ユビキチン鎖を切断することが報告されていたため31、野生型USP27XおよびXLID105関連変異体であるF313V、Y381H、およびS404N USP27Xタンパク質をK63結合ジユビキチン鎖と1時間インキュベートした。これらのサンプルをSDS-PAGE32で分離し、ユビキチンタンパク質とUSP27Xタンパク質をイムノブロッティングで分析しました。野生型USP27Xは、ジユビキチン切断を誘導し、モノユビキチンを生成します(図2Bおよび補足図1)。XLID105関連F313V、Y381H、およびS404N USP27X変異体タンパク質は、これらの鎖を切断しませんでした。F313V、Y381H、およびS404Nの変異体はUSP27Xの触媒活性を阻害するため、USP27Xの機能破壊がXLID1057の根底にある主要なメカニズムであると考えられる。追加の定量化および補完的な実験は以前に報告されています7。
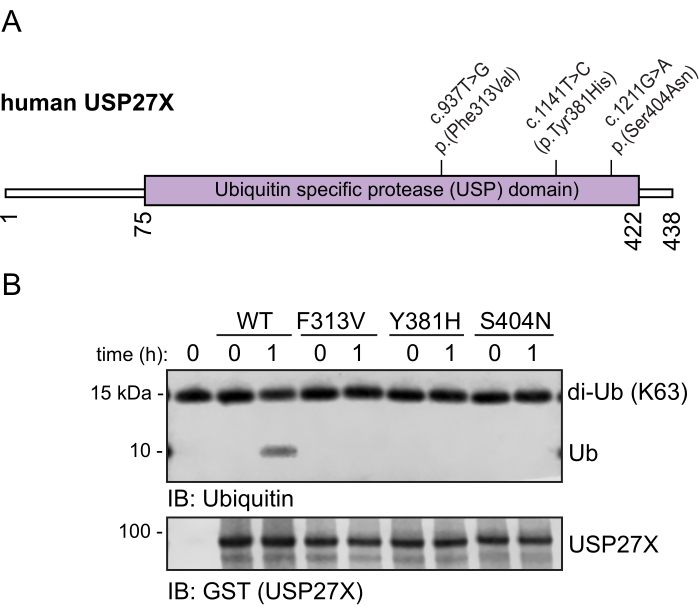
図2:XLID105変異体がUSP27Xの脱ユビキチン化活性に及ぼす影響(A)ヒトUSP27Xタンパク質構造の図で、USPドメイン(紫色)で評価されたXLID105変異体の残基数と位置を示す図。(B)GSTタグ付き野生型USP27XおよびXLID105変異体(F313V、Y381H、およびS404N)を、K63ジユビキチン鎖と示された時間インキュベートした。イムノブロット分析は、抗GST(USP27X)およびユビキチン抗体を用いて行いました。この図の拡大版を表示するには、ここをクリックしてください。
補足図1:図2Bのトリミングされていないブロット。このファイルをダウンロードするには、ここをクリックしてください。
ディスカッション
この記事では、組換えUSP27X DUBの発現と精製のためのプロトコールと、野生型USP27XとNDD関連バリアントタンパク質の脱ユビキチン化活性を比較するための in vitro ユビキチン鎖切断アッセイについて紹介します。このアッセイにより、XLID105関連変異体がUSP27X触媒活性を破壊することが決定されました7。このメカニズムの洞察は、XLID105をUSP27X機能破壊障害として定義するのに役立ちました。
このプロトコールは、分子メカニズムが十分に理解されていない遺伝病に関連する他のDUBにも適用できます。目的の特定のDUBに対する発現および精製プロトコルの最適化は非常に重要です。対象の特定のDUBについて特定のプロトコルが記述されていない場合、ここで説明する方法は出発点として機能できます。タンパク質の発現および精製プロトコルを最適化する際の重要なステップには、(1)発現系(細菌株、昆虫細胞、または哺乳動物細胞など)、(2)発現ベクター、(3)使用するタグおよびアフィニティー樹脂、(4)増殖条件、(5)培養量、および(6)さらなる精製の必要性(例えば、 サイズ排除またはイオン交換クロマトグラフィー による )。この方法のこれらの側面および他の側面に関する具体的な推奨事項が議論されている33。
in vitroユビキチン鎖切断アッセイは、野生型または変異型DUBの脱ユビキチン化活性を測定する簡単な方法です。ユビキチン切断の時間依存性解析は、KmやVmaxなどのパラメータの決定を可能にすることにより、遺伝的変異がDUB酵素動態に及ぼす影響についての洞察を提供することができます。これにより、触媒作用を損なうDUB活性部位の欠陥(Vmaxの減少)と基板認識を損なう欠陥(Kmの増加)を区別することができます。このアッセイの汎用性は、関心のあるDUBに関する明確なメカニズム情報を提供するために使用できるさまざまなユビキチン鎖に由来します。さまざまな長さと結合トポロジーのユビキチン鎖が市販されているため、このアッセイは専門家でなくても利用できます。特定のDUBは、酵素動態、連鎖認識、および切断のための独自の特性を持っています。このアッセイの重要なステップには、(1)DUBの使用濃度の定義、(2)最適な反応時間の決定、(3)ユビキチン鎖結合の種類と長さの選択、(4)検出方法の選択が含まれます。
ユビキチン鎖切断アッセイは、NDDバリアントがDUB活性に及ぼす影響を測定し、7,11、DUB活性を阻害し、非定型的な開発を促進するバリアントの特定に役立ちます。このアッセイは、がんや神経変性など、遺伝的変異が病理を左右するさまざまな状況で使用できます。基質に依存しないアッセイであるため、特定のユビキチン化タンパク質基質を事前に同定する必要はありません。しかし、このアッセイはユビキチン鎖切断活性の変化の測定に限定されており、タンパク質間相互作用、細胞内局在、翻訳後修飾などのDUB機能に対する変異体の影響を評価することはできません。これらのバリアントが個別のNDDをどのように駆動するかに対処するための特定のアッセイを開発するには、目的のDUBが機能する広範なシグナル伝達経路の同定が必要です。
開示事項
著者は、利益相反を宣言しません。
謝辞
この研究は、Sanford ResearchのスタートアップファンドからFBに、NIHの助成金R01CA233700 MJSから資金提供を受けました。アートワークはフェリペ・G・セラーノ(www.illustrative-science.com)が担当しました。GST-USP27Xプラスミドを提供してくださったGreg Findlay博士(ダンディー大学)に感謝いたします。
資料
| Name | Company | Catalog Number | Comments |
| Amersham Protran 0.45 NC 200 mm × 200 mm 25/PK | Cytiva | 10600041 | |
| Ammonium sulfate | Fisher Scientific | AC205872500 | |
| Ampicillin | Fisher Scientific | BP1760 25 | |
| Anti- Ubiquitin (Mouse monoclonal) | Biolegend | Cat# 646302, RRID:AB_1659269 | (WB: 1:1000) |
| Anti-GST (Sheep polyclonal) | MRC-PPU Reagents and Services | Cat# S902A Third bleed | (WB: 1:1000) https://mrcppureagents.dundee.ac.uk/ |
| Baffled Culture Flasks 2 L | Fisher Scientific | 10-042-5N | |
| Bradford Reagent | Millipore Sigma | B6916-500ML | |
| Chloramphenicol | Gold Biotechnology | C-105-25 | |
| Complete, Protease Inhibitor tablets | Millipore Sigma | 5056489001 | |
| Econo-Column 1.5 × 5 cm | Bio-Rad | 7371507 | |
| Eppendorf ThermoMixer F1.5 | Eppendorf | 5384000020 | |
| Excel | Microsoft | https://www.microsoft.com/en-us/microsoft-365/excel | |
| Glycerol | Genesee Scientific | 18-205 | |
| Illustrator | Adobe | https://www.adobe.com/products/illustrator.html | |
| Image Studio | LI-COR Biosciences | https://www.licor.com/bio/image-studio/ | |
| Inkscape | Inkscape | https://inkscape.org/ | |
| Invitrogen 4-12% NuPAGE 1mm 12 well gel | Thermo Fisher Scientific | NP0322BOX | |
| IPTG (Isopropyl-b-D-Thiogalactopyranoside) | Genesee Scientific | 20-109 | |
| IRDye 800CW Donkey anti-Goat IgG Secondary Antibody | LI-COR Biosciences | Cat# 926-32214 | (WB: 1:10000) |
| IRDye 800CW Donkey anti-Mouse IgG Secondary Antibody | LI-COR Biosciences | Cat# 926-32212 | (WB: 1:10000) |
| Isotemp Digital Dry Bath | Fisher Scientific | 88860022 | |
| K63 Di-Ubiquitin | South Bay Bio LLC | SBB-UP0072 | |
| LB Agar | Genesee Scientific | 11-119 | |
| LB Broth | Genesee Scientific | 11-118 | |
| Lysozyme | Gold Biotechnology | L-040-100 | |
| MaxQ 4000 Benchtop Orbital Shaker | Thermo Fisher Scientific | SHKE4000-7 | |
| MES-SDS Running Buffer | Boston Bioproducts Inc | BP-177 | |
| Mini Tube Rotator | Fisher Scientific | 88-861-051 | |
| NuPage LDS Sample buffer 4x | Thermo Fisher Scientific | NP0007 | |
| Odyssey Fc Imager | LI-COR Biosciences | 43214 | |
| PageRuler Plus Ladder | Thermo Fisher Scientific | 26620 | |
| pGEX6P1 human USP27X | MRC-PPU Reagents and Services | DU21193 | https://mrcppureagents.dundee.ac.uk/ |
| pGEX6P1 human USP27X F313V | Addgene | 225715 | Koch et at 2024 (PMID: 38182161) |
| pGEX6P1 human USP27X S404N | Addgene | 225717 | Koch et at 2024 (PMID: 38182161) |
| pGEX6P1 human USP27X Y381H | Addgene | 225716 | Koch et at 2024 (PMID: 38182161) |
| Pierce Glutathione Agarose | Thermo Fisher Scientific | 16100 | |
| PMSF (Phenylmethylsulfonyl fluoride) | Gold Biotechnology | P-470-10 | |
| Polysorbate 20 (Tween 20) | Fisher Scientific | AC233360010 | |
| Rosetta 2 Competent Cells | Millipore Sigma | 71402-M | |
| SimplyBlue SafeStain | Thermo Fisher Scientific | LC6060 | |
| SmartSpec 3000 | Bio-Rad | 170-2501 | |
| SOC medium | Thermo Fisher Scientific | 15544034 | |
| Sodium chloride | Genesee Scientific | 18-216 | |
| Sonifier 250 | Branson | 100-132-135 | |
| Sorvall RC 6 Plus Centrifuge | Thermo Fisher Scientific | 46910 | |
| TCEP (Tris-(carboxyethyl) phosphine hydrochloride) | Gold Biotechnology | TCEP10 | |
| Terrific Broth Powder | Genesee Scientific | 18-225 | |
| Tris Base | Genesee Scientific | 18-146 | |
| XCell SureLock Mini-Cell and XCell II Blot Module | Thermo Fisher Scientific | EI0002 |
参考文献
- Morris-Rosendahl, D. J., Crocq, M. -. A. Neurodevelopmental disorders: the history and future of a diagnostic concept. Dialogues Clin Neurosci. 22 (1), 65-72 (2020).
- Ebstein, F., Küry, S., Papendorf, J. J., Krüger, E. Neurodevelopmental disorders (NDD) caused by genomic alterations of the ubiquitin-proteasome system (UPS): the possible contribution of immune dysregulation to disease pathogenesis. Front Mol Neurosci. 14, 733012 (2021).
- Hershko, A., Ciechanover, A. The ubiquitin system. Annu Rev Biochem. 67 (10), 425-479 (1998).
- Pickart, C. M. Mechanisms underlying ubiquitination. Annu Rev Biochem. 70 (1), 503-533 (2001).
- Komander, D., Clague, M. J., Urbé, S. Breaking the chains: structure and function of the deubiquitinases. Nat Rev Mol Cell Biol. 10 (8), 550-563 (2009).
- Yau, R., Rape, M. The increasing complexity of the ubiquitin code. Nat Cell Biol. 18 (6), 579-586 (2016).
- Koch, I., et al. USP27X variants underlying X-linked intellectual disability disrupt protein function via distinct mechanisms. Life Sci Alliance. 7 (3), e202302258 (2024).
- Hao, Y. -. H., et al. USP7 acts as a molecular rheostat to promote WASH-dependent endosomal protein recycling and is mutated in a human neurodevelopmental disorder. Mol Cell. 59 (6), 956-969 (2015).
- Fountain, M. D., et al. Pathogenic variants in USP7 cause a neurodevelopmental disorder with speech delays, altered behavior, and neurologic anomalies. Genet Med. 21 (8), 1797-1807 (2019).
- Santiago-Sim, T., et al. Biallelic variants in OTUD6B cause an intellectual disability syndrome associated with seizures and dysmorphic features. Am J Hum Genet. 100 (4), 676-688 (2017).
- Beck, D. B., et al. Linkage-specific deubiquitylation by OTUD5 defines an embryonic pathway intolerant to genomic variation. Sci Adv. 7 (4), eabe2116 (2021).
- Cho, J., Park, J., Kim, E. E., Song, E. J. Assay systems for profiling deubiquitinating activity. Int J Mol Sci. 21 (16), 1-16 (2020).
- Matsui, S., Sandberg, A. A., Negoro, S., Seon, B. K., Goldstein, G. Isopeptidase: A novel eukaryotic enzyme that cleaves isopeptide bonds. Proc Natl Acad Sci USA. 79 (5), 1535-1539 (1982).
- Rose, I. A., Warms, J. V. B. An enzyme with ubiquitin carboxy-terminal esterase activity from reticulocytes. Biochemistry. 22 (18), 4234-4237 (1983).
- Wilkinson, K. D., Cox, M. J., Mayer, A. N., Frey, T. Synthesis and characterization of ubiquitin ethyl ester, a new substrate for ubiquitin carboxyl-terminal hydrolase. Biochemistry. 25 (21), 6644-6649 (1986).
- Pickart, C. M., Rose, I. A. Ubiquitin carboxyl-terminal hydrolase acts on ubiquitin carboxyl-terminal amides. J Biol Chem. 260 (13), 7903-7910 (1985).
- Hadari, T., Warms, J. V. B., Rose, I. A., Hershko, A. A ubiquitin C-terminal isopeptidase that acts on polyubiquitin chains: Role in protein degradation. J Biol Chem. 267 (2), 719-727 (1992).
- Woo, S. K., et al. Multiple ubiquitin C-terminal hydrolases from chick skeletal muscle. J Biol Chem. 270 (32), 18766-18773 (1995).
- Woo, S. K., et al. Purification and characterization of a new ubiquitin C-terminal hydrolase (UCH-1) with isopeptidase activity from chick skeletal muscle. J Biochem. 121 (4), 684-689 (1997).
- Baek, S. H., et al. Molecular cloning of a novel ubiquitin-specific protease, UBP41, with isopeptidase activity in chick skeletal muscle. J Biol Chem. 272 (41), 25560-25565 (1997).
- Wilkinson, K. D., et al. Metabolism of the polyubiquitin degradation signal: Structure, mechanism, and role of isopeptidase T. Biochemistry. 34 (44), 14535-14546 (1995).
- Hu, M., et al. Crystal structure of a UBP-family deubiquitinating enzyme in isolation and in complex with ubiquitin aldehyde. Cell. 111 (7), 1041-1054 (2002).
- Hu, M., et al. Structure and mechanisms of the proteasome-associated deubiquitinating enzyme USP14. EMBO J. 24 (21), 3747-3756 (2005).
- Gorka, M., Magnussen, H. M., Kulathu, Y. Chemical biology tools to study deubiquitinases and UBL proteases. Semin Cell Dev Biol. 132, 86-96 (2022).
- Faesen, A. C., et al. The differential modulation of USP activity by internal regulatory domains, interactors, and eight ubiquitin chain types. Chem Biol. 18 (12), 1550-1561 (2011).
- Mevissen, T. E. T., et al. OTU deubiquitinases reveal mechanisms of linkage specificity and enable ubiquitin chain restriction analysis. Cell. 154 (1), 169-184 (2013).
- Kwasna, D., et al. Discovery and characterization of ZUFSP/ZUP1, a distinct deubiquitinase class important for genome stability. Mol Cell. 70 (1), 150-164.e6 (2018).
- Hu, H., et al. X-exome sequencing of 405 unresolved families identifies seven novel intellectual disability genes. Mol Psychiatry. 21 (1), 133-148 (2016).
- Shaker Orbit - Revolving in Space Around the Samples? Eppendorf Lab Academy. Eppendorf Lab Academy Available from: https://www.eppendorf.com/us-en/lab-academy/lab-solutions/others/shaker-orbit-revolving-in-space-around-the-samples (2020)
- Smith, D. B., Johnson, K. S. Single-step purification of polypeptides expressed in Escherichia coli as fusions with glutathione S-transferase. Gene. 67 (1), 31-40 (1988).
- Ritorto, M. S., et al. Screening of DUB activity and specificity by MALDI-TOF mass spectrometry. Nat Commun. 5, 4763 (2014).
- Wingfield, P. T. Overview of the purification of recombinant proteins. Curr Protoc Protein Sci. 80, 6.1.1-6.1.35 (2015).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved