Method Article
Messung der enzymatischen Aktivität von Enzymen, die mit neurologischen Entwicklungsstörungen assoziiert sind, mittels eines in vitro Ubiquitin-Kettenspaltungsassays
In diesem Artikel
Zusammenfassung
In diesem Protokoll wird erläutert, wie die Wirkung genetischer Variation, die mit neurologischen Entwicklungsstörungen verbunden ist, auf die deubiquitylierende Enzymaktivität gemessen werden kann, indem die rekombinante Proteinreinigung mit Ubiquitin-Kettenspaltungsassays kombiniert wird.
Zusammenfassung
Neurologische Entwicklungsstörungen (NDDs) sind mit Beeinträchtigungen der Funktion des Nervensystems verbunden, bleiben aber auf molekularer Ebene oft wenig verstanden. Diskrete Störungen, die durch einzelne Gene verursacht werden, liefern Modelle, um Mechanismen zu untersuchen, die die atypische Neuroentwicklung antreiben. Varianten von Genen, die für Proteine der Familie der deubiquitylierenden Enzyme (DUB) kodieren, sind mit mehreren NDDs assoziiert, aber es besteht die Notwendigkeit, die pathogenen Mechanismen von Erkrankungen zu bestimmen, die durch diese Genvarianten ausgelöst werden. Der Einfluss von Genvarianten auf die DUB-Aktivität kann experimentell mit einem substratunabhängigen in vitro Ubiquitin-Spaltungsassay bestimmt werden. Dieser Assay erfordert keine Kenntnisse der nachgeschalteten Substrate, um die katalytische Aktivität direkt zu messen. Hier wird das Protokoll zur Bestimmung des Einflusses von Genvarianten auf die enzymatische Aktivität unter Verwendung der DUB Ubiquitin Specific Protease 27, X-linked (USP27X) modelliert, die bei X-linked intellectual disability 105 (XLID105) mutiert ist. Diese experimentelle Pipeline kann verwendet werden, um die Mechanismen zu klären, die neurologischen Entwicklungsstörungen zugrunde liegen, die durch Varianten in DUB-Genen ausgelöst werden.
Einleitung
Neurologische Entwicklungsstörungen (NDDs) entstehen durch verschiedene Ätiologien mit umweltbedingten oder genetischen Determinanten, die die atypische Entwicklung des Nervensystems vorantreiben1. Gentests der Next-Generation-Sequenzierung haben eine zunehmende Anzahl von Varianten in Genen, die mit dem Ubiquitin-System in Verbindung stehen, mit genetischen NDDs in Verbindung gebracht2. Das Ubiquitin-System katalysiert die Ligation des kleinen Proteinmodifikators Ubiquitin hauptsächlich an Lysinreste in Proteinsubstraten, um Veränderungen im zellulären Verhalten zu bewirken, einschließlich Lokalisierung, Stabilität, Protein-Protein-Wechselwirkungen oder Aktivität3. Die Ubiquitylierung wird durch E1-aktivierende, E2-konjugierende und E3-Ligase-Enzymevermittelt 4 und ist reversibel durch die Aktivität von deubiquitylierenden Enzymen (DUBs), die die Spaltung und Entfernung von Ubiquitin aus Proteinsubstraten katalysieren5. Ubiquitin kann als Monomer (Monoubiquitylierung) oder polymere Ketten (Polyubiquitylierung) an das Substrat ligiert werden, die an einem der sieben Lysinreste (K6, K11, K27, K29, K33, K48, K63) oder dem M1-Rest von Ubiquitin gebildet werden. Diese verschiedenen Ubiquitin-Kettentopologien und ihre Kombinationen erzeugen einen zellulären Code, der für die Signaltransduktionentscheidend ist 6.
DUBs wie USP27X, USP7, USP9X, USP48, STAMBP, OTUD4, OTUD6B und OTUD5 wurden mit den NDDs 2,7,8,9,10,11 in Verbindung gebracht. Bei den meisten NDDs sind die molekularen Mechanismen, die die Pathogenese antreiben, experimentell noch nicht definiert. Einige der DUBs, die zu den kürzlich beschriebenen Erkrankungen führen, sind schlecht verstanden und es fehlen bekannte zelluläre Messwerte, die zur Beurteilung des Einflusses genetischer Variation auf die Proteinfunktion verwendet werden können. In vitro überwinden Ubiquitin-Kettenspaltungsassays diese Einschränkung, da substratunabhängige DUB-Aktivitätsauslesungen den Einfluss von Genvarianten auf die enzymatische Aktivität messen können12.
In-vitro-Ubiquitin-Spaltungsassays werden seit den 1980er Jahren eingesetzt. Diese Assays mit radioaktiv markierten Substraten ermöglichten die Entdeckung der ersten DUBs, einschließlich der Isopeptidase, die für ihre Fähigkeit zur Deubiquitylierung von Histon H2A13 identifiziert wurde, und der Ubiquitin-Carboxyl-terminalen Hydrolase (UCH), die durch ihre Fähigkeit identifiziert wurde, Ubiquitin aus einer Vielzahl von chemischen Konjugaten zu hydrolysieren 14,15,16 . Des Weiteren wurden radioaktiv markierte polyubiquitylierte Proteine oder Peptide in voller Länge verwendet, um die Isopeptidase T und mehrere UCHs und Ubiquitin-spezifische Proteasen (UBPs) aus Erythrozyten bzw. Skelettmuskulatur zu identifizieren 17,18,19,20. Ubiquitinketten einer definierten Länge und eines definierten Bindungstyps (K48-verknüpftes Tetra-Ubiquitin) wurden zunächst verwendet, um die DUB-Aktivität der Isopeptidase T21 zu messen. Seitdem hat sich dieser Assay zum Goldstandard für die Messung der DUB-Aktivität in Mutationsanalysen entwickelt22,23. Die Verfeinerung dieses Assays ermöglicht derzeit die Visualisierung der Ubiquitin-Spaltung mittels Elektrophorese und konventioneller Gelfärbungen wie Coomassie-Blau-, SYPRO-Orange-, Rubin- und Silberfärbung oder Fluoreszenz- oder Immunblotting-basierter Detektion12,24. Molekulare Aspekte der DUB-Aktivität, wie z.B. die minimale Kettenlänge und die Bindungsspezifität 25,26,27,28, können durch die Verwendung von Ubiquitinketten unterschiedlicher Länge (z.B. Di-, Tri-, Tetra-Ubiquitin) und Bindungen (K6, K11, K27, K29, K33, K48, K63, linear) in funktionellen Assays geklärt werden. NDD-assoziierte Varianten können DUB-Aktivitätsdefekte verursachen, die spezifisch für den Ubiquitin-Kettenverbindungstyp sind11.
Ein Di-Ubiquitin-Spaltungsassay mit aufgereinigten rekombinanten DUB-Proteinen kann den Einfluss von NDD-Varianten auf die DUB-Aktivität direkt messen. USP27X, das in der NDD X-linked intellectual disability disorder 105 (XLID105)7,28 mutiert ist, modelliert den hier vorgestellten Prozess. Dieser Ansatz ermöglicht die Bestimmung, wie die DUB-Aktivität durch Genvarianten in bestehenden und unbekannten DUB-assoziierten NDDs gestört wird.
Protokoll
Das folgende Protokoll kann für rekombinante Proteine angepasst werden, indem verschiedene Affinitätsmarkierungen verwendet werden, die in verschiedenen Stämmen kompetenter Zellen exprimiert werden. Abhängig vom zu exprimierenden Protein können die Kultivierungs- und Übernacht-Expressionsbedingungen eine Optimierung des OD600 bei der Expressionsinduktion, der Expressionszeit, der Expressionstemperatur und der IPTG-Konzentration erfordern. Eine Übersicht über das Protokoll ist in Abbildung 1 dargestellt. Die Einzelheiten zu den Reagenzien und der Ausrüstung, die in dieser Studie verwendet wurden, sind in der Materialtabelle aufgeführt.
1. Transformation kompetenter Rosetta 2 E. coli Zellen mit dem rekombinanten GST-USP27X-Expressionsplasmid
HINWEIS: Um die Sterilität der Bakterienkultur zu erhalten, führen Sie Schritte durch, bei denen die Medienbehälter unter einer Bunsenbrennerflamme geöffnet sind. Um einen optimalen Sauerstofftransfer zu ermöglichen, führen Sie das Schütteln der Bakterienkultur in einem temperaturgesteuerten Tischschüttler mit einer Umlaufbahn von 19 mm bis 50 mm und einer Drehzahl von 200 U/mindurch 29.
- 20 μl chemisch kompetente Rosetta 2 E. coli-Zellen unmittelbar vor der Verwendung auf Eis auftauen. 1-10 ng des pGEX6P1-USP27X-Expressionsplasmids (kodiert für N-terminale Glutathion-S-Transferase (GST)30 -markiertes USP27X und mit Ampicillin-Resistenz) und 5 min auf Eis inkubieren.
- Hitzeschock für 30 s bei 42 °C in einem trockenen Bad. 2 Min. auf Eis inkubieren.
- Fügen Sie 80 μl SOC-Medium bei Raumtemperatur (RT) hinzu. Inkubieren Sie 60 Minuten lang bei 37 °C und einer Drehung von 200 U/min in einem temperaturgesteuerten Tischschüttler mit 19 mm Orbit.
- Platte: 50 μl Kultur auf einer LB-Agarplatte, ergänzt mit 25 μg/l Chloramphenicol und 50 μg/l Ampicillin. 20 h bei 37 °C mit dem Deckel nach unten in einem temperierten Inkubator inkubieren.
2. Bakterielle Expression des rekombinanten Proteins aus dem Expressionsplasmid über Nacht
HINWEISE: Um die Sterilität der Bakterienkultur zu erhalten, führen Sie Schritte durch, bei denen die Medienbehälter unter einer Bunsenbrennerflamme geöffnet sind. Um einen optimalen Sauerstofftransfer zu ermöglichen, führen Sie das Schütteln der Bakterienkultur in einem temperaturgesteuerten Tischschüttler mit einer Umlaufbahn von 19 mm bis 50 mm und einer Drehzahl von 200 U/mindurch 29. Messen Sie die Kultur OD600 mit einem Spektralphotometer.
- Bereiten Sie 1 l Terrific Broth (TB)-Medium vor, indem Sie 47,6 g TB-Pulver zu 1 l Reinstwasser mit 0,4 % Glycerin in einem 2-l-Kulturkolben mit Blende geben. Medium 30 min lang autoklavieren und auf RT abkühlen lassen.
- Wählen Sie eine einzelne Kolonie transformierter Bakterien und fügen Sie 10 ml steriles LB-Medium hinzu, das mit 25 μg/l Chloramphenicol und 50 μg/l Ampicillin ergänzt wird. Inkubieren Sie 20 Stunden lang bei 37 °C und einer Drehung von 200 U/min in einem temperaturgesteuerten Tischschüttler mit 19 mm Orbit.
- Geben Sie 10 ml Übernachtkultur zu 1 l TB-Medium, ergänzt mit 25 μg/l Chloramphenicol und 50 μg/l Ampicillin (Verhältnis von 1:100 zwischen Ausgangskultur und Expressionskultur). Inkubieren Sie bei 37 °C mit einer Drehung von 200 U/min in einem temperaturgesteuerten Tischschüttler mit einer Umlaufbahn von 19 mm, bis der Kultur-OD600 zwischen 0,5 und 0,6 liegt.
- Geben Sie 50 μM IPTG in die Kultur, um die Expression zu induzieren, kühlen Sie auf 16 °C ab und inkubieren Sie 20 Stunden lang bei 16 °C mit einer Drehung von 200 U/min.
- Pelletzellen aus Expressionskultur durch Zentrifugation für 20 min bei ≥3.000 x g und 4 °C. Lagern Sie das Pellet bei -80 °C für mindestens 1 h.
HINWEIS: Zu diesem Zeitpunkt kann das Experiment pausiert und später (vorzugsweise in derselben Woche) erneut gestartet werden. Das Pellet kann langfristig bei -80 °C gelagert werden.
3. Proteinaufreinigung durch Schwerkraft-Flow-Affinitätssäule
HINWEIS: Die für jede Aufreinigung geeigneten Harz-, Bindungs-, Wasch-, Elutions- und Lagerpuffer hängen von dem zu reinigenden rekombinanten Protein ab. Sammeln Sie Proben aus dem Zellpellet, dem Überstand, den Durchflussfraktionen, den Waschfraktionen und den Elutionsfraktionen im SDS-PAGE-Puffer. Führen Sie SDS-PAGE- und Coomassie-Färbungen für die Proben durch, um den Erfolg der Aufreinigung zu bewerten. Führen Sie die Reinigung bei 4 °C durch und behandeln Sie Fraktionen auf Eis. Die Spaltung von Protein-Tags kann an oder außerhalb der Säule unter Verwendung der geeigneten Protease durchgeführt werden, um die relevante Protease-spezifische Spaltstelle zu erreichen.
- Befestigen Sie die leere Schwerkraft-Flow-Säule im Retortenständer und füllen Sie sie mit Glutathion-Agarose-Harz. Verwenden Sie 2-3 ml Harz, um ein Zellpellet zu reinigen, das aus 1 l Expressionskultur gewonnen wurde.
- Waschen Sie die Säule mit einem Harzbettvolumen von 20 % Ethanol. Waschen Sie die Säule 3 Mal mit einem Harzbettvolumen MS500 Puffer (20 mM Tris pH 7,5, 500 mM NaCl, 0,5 mM TCEP). Stoppen Sie die Säule, damit das Harz mit Puffer bedeckt bleibt, um ein Austrocknen während der Vorbereitung des Zellpellets zu verhindern.
- Tauen Sie das Küvettenpellet bei 4 °C auf. Geben Sie 30 ml MS500-Puffer (20 mM Tris pH 7,5, 500 mM NaCl, 0,5 mM TCEP, 60 mg Lysozym und eine Proteasehemmer-Tablette) zum aufgetauten Pellet. 30 min bei 4 °C mit sanfter End-over-End-Drehung lysieren.
- Beschallen Sie die lysierten Zellen entweder in einem 50-ml-Zentrifugenröhrchen oder einem Metallbecherglas auf Eis, bis das Lysat frei fließt, wenn es aus einer Pipettenspitze abgegeben wird. 30 min bei 4 °C und ≥20.000 x g zentrifugieren, um den Überstand zu reinigen.
HINWEIS: Bestimmen Sie die Einstellungen des Ultraschallgeräts empirisch. Stellen Sie den Ultraschallator so ein, dass eine Gesamtzeit von 120 s ausreicht, um das viskose Lysat zu einer rieselfähigen und transparenten Flüssigkeit zu reduzieren. - Dekantieren Sie den Überstand in ein Becherglas und laden Sie das gereinigte Lysat auf die Säule. Lassen Sie das Lysat durch die Schwerkraft durch die Säule laufen, während Sie den Durchfluss auffangen. Laden Sie die Spalte mit dem gesammelten Flow-Through und führen Sie ihn durch die Spalte.
- Waschen Sie die Säule 5 Mal mit mindestens zwei Harzbettvolumina MS500 Waschpuffer (20 mM Tris pH 7,5, 500 mM NaCl, 0,5 mM TCEP). Sammeln Sie den Waschfluss in 5-ml-Fraktionen. Fügen Sie 1 μl jeder Fraktion zu 100 μl Bradford-Reagenz hinzu, um das Vorhandensein von Proteinen visuell zu überprüfen. Waschen Sie, bis bei der letzten Wäsche kein ungebundenes Protein mehr vorhanden ist, und fügen Sie bei Bedarf zusätzliche Waschschritte hinzu.
- Das Protein wird zurückgewonnen, indem MS500-Elutionspuffer (MS500-Puffer, ergänzt mit 10 mM Glutathion und 10 mM NaOH) durch die Säule geführt und 5 ml-Elutionsfraktionen gesammelt werden. Überprüfen Sie das Vorhandensein von Proteinen, indem Sie 1 μl Eluat zu 100 μl Bradford-Reagenz hinzufügen. Hören Sie auf, Fraktionen zu sammeln, wenn das Bradford-Reagenz nicht mehr anzeigt, dass Proteine vorhanden sind.
- Führen Sie den Pufferaustausch durch Fällung und Zentrifugation durch. Fältitieren Sie das Protein, indem Sie dem Eluat 2 Volumen von 4 M Ammoniumsulfat zufügen, es sanft invertieren, bis es trüb ist, und dann 30 Minuten lang bei 4 °C und ≥20.000 x g zentrifugieren und erneut weitere 5 Minuten zentrifugieren. Entfernen Sie den Überstand nach jeder Zentrifugation, ohne das Proteinpellet zu stören.
- Lösen Sie das Protein in MS500, ergänzt mit 25% Glycerin, wieder auf und speichern Sie es. Lagern Sie Proteinpellets oder Protein in Lagerpuffer bei -80 °C.
4. In-vitro-Assay für die Ubiquitin-Kettenspaltung
HINWEIS: Wählen Sie die Länge der Ubiquitinkette und die Verknüpfungstypen basierend auf der DUB-Spezifität aus, die in früheren Berichten31 beschrieben oder empirisch bestimmt wurde. Falls erforderlich, könnte dieses Protokoll verwendet werden, um die Aktivität des interessierenden Wildtyp-DUB an einem Panel kommerziell erhältlicher Ubiquitinketten definierter Länge und Kopplungsart zu testen. Eine Di-Ubiquitin-Kettenmenge von 375-750 ng und eine DUB-Konzentration von 1-2 μM können als Ausgangspunkte für den Assayverwendet werden 27.
- Bereiten Sie 10x DUB-Aktivierungspuffer vor (500 mM Tris-HCl pH 7,5, 500 mM NaCl und 100 mM TCEP).
- Bereiten Sie für jeden Zeitpunkt für jeden DUB insgesamt 10 μl 2 μM gereinigtes GST-USP27X in 1x DUB-Aktivierungspuffer (DUB-Mix) vor. Bereiten Sie Master-Mixe vor und teilen Sie sie in Zeitpunkte auf.
- Inkubieren Sie den DUB-Mix 10 Minuten lang bei RT.
- Fügen Sie 7 μl SDS-PAGE-Ladepuffer zum Zeitpunkt 0 hinzu, bevor Sie Ubiquitinketten hinzufügen, um den Start der Deubiquitylierungsreaktion zu verhindern.
- Zu jedem Zeitpunkt für jeden DUB werden 375 ng K-63-Diubiquitinketten hinzugefügt, die in 10 μl 1x DUB-Aktivierungspuffer verdünnt sind. Das Gesamtvolumen beträgt 20 μL pro Reaktion.
- Inkubieren Sie die Röhrchen bei 30 °C und stoppen Sie jeden Zeitpunkt mit 7 μl SDS-PAGE-Ladepuffer.
- Führen Sie die SDS-PAGE mit einem 4%-12%-Gradientengel und Immunoblot 7,32 für Ubiquitin und USP27X durch, um die Veränderung des Mono-Ubiquitin-Vorhandenseins über ausgewählte Zeitpunkte zu analysieren.

Abbildung 1: Schematische Darstellung des Studiendesigns. (A) Transformation von kompetenten E. coli-Zellen mit rekombinantem Proteinexpressionsplasmid. (B) Bakterielle Expression des rekombinanten Deubiquitylase-Proteins über Nacht. (C) Proteinaufreinigung der rekombinanten Deubiquitylase unter Verwendung einer Schwerkraft-Strömungs-Affinitätssäule. (D) In-vitro-Experiment zur Teilung der Ubiquitinkette zur Bewertung der Deubiquitylierungsaktivität. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergebnisse
Um den Einfluss von XLID105-assoziierten Varianten auf die katalytische Aktivität von USP27X zu bestimmen, wurden GST-markierte Wildtyp-Proteine USP27X und die XLID105-assoziierten Varianten F313V, Y381H und S404N USP27X-Proteine aus Bakterien aufgereinigt. Diese Varianten befinden sich innerhalb der USP-katalytischen Domäne von USP27X (Abbildung 2A). Da zuvor berichtet wurde, dass USP27X die K63-Ubiquitinketten31 spaltet, wurden Wildtyp-USP27X und die XLID105-assoziierten Varianten F313V, Y381H und S404N USP27X-Proteine 1 h lang mit K63-verknüpften Di-Ubiquitin-Ketten inkubiert. Diese Proben wurden mittels SDS-PAGE32 getrennt, und Ubiquitin und USP27X-Proteine wurden mittels Immunblotting analysiert. Der Wildtyp USP27X induziert die Di-Ubiquitin-Spaltung und erzeugt Mono-Ubiquitin (Abbildung 2B und ergänzende Abbildung 1). XLID105-assoziierte Proteine der F313V-, Y381H- und S404N-USP27X-Varianten spalteten diese Ketten nicht. Da die Varianten F313V, Y381H und S404N die katalytische Aktivität von USP27X stören, scheint die funktionelle Störung von USP27X der Hauptmechanismus zu sein, der XLID1057 zugrunde liegt. Zusätzliche Quantifizierungen und ergänzende Experimente wurden bereits berichtet7.
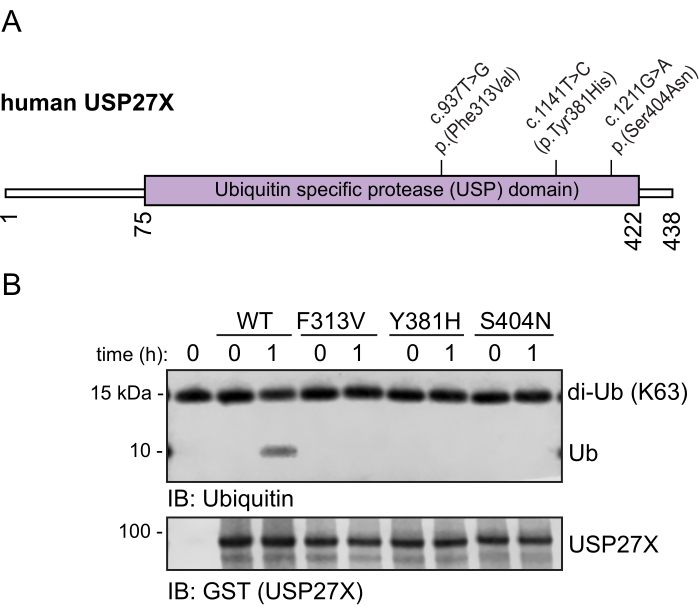
Abbildung 2: Einfluss XLID105 Varianten auf die Deubiquitylierungsaktivität von USP27X. (A) Diagramm der humanen USP27X-Proteinstruktur mit den Restzahlen und der Position der XLID105 Varianten, die in der USP-Domäne ausgewertet wurden (violett). (B) GST-markierte Wildtyp-Varianten USP27X und XLID105 (F313V, Y381H und S404N) wurden für die angegebenen Zeiten mit K63-Di-Ubiquitin-Ketten inkubiert. Die Immunoblot-Analyse wurde mit Anti-GST (USP27X) und Ubiquitin-Antikörpern durchgeführt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Abbildung 1: Ungeschnittene Kleckse von Abbildung 2B. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
In diesem Artikel wird ein Protokoll für die Expression und Aufreinigung von rekombinanten USP27X-DUBs und ein in vitro Ubiquitin-Kettenspaltungsassay vorgestellt, um die deubiquitylierende Aktivität von Wildtyp-USP27X- und NDD-assoziierten Variantenproteinen zu vergleichen. Dieser Assay ergab, dass XLID105-assoziierte Varianten die katalytische Aktivität von USP27X stören7. Diese mechanistische Erkenntnis half uns, XLID105 als USP27X-Funktionsstörung zu definieren.
Dieses Protokoll kann an andere DUBs angepasst werden, die mit genetischen Krankheiten mit wenig verstandenen molekularen Mechanismen in Verbindung gebracht werden. Die Optimierung des Expressions- und Aufreinigungsprotokolls für einen bestimmten DUB von Interesse ist von entscheidender Bedeutung. Wenn für eine bestimmte DUB von Interesse keine spezifischen Protokolle beschrieben wurden, kann die hier beschriebene Methode als Ausgangspunkt dienen. Bei der Optimierung eines Proteinexpressions- und -reinigungsprotokolls umfassen die kritischen Schritte die Auswahl der geeigneten Parameter für (1) das Expressionssystem (z. B. Bakterienstämme, Insektenzellen oder Säugetierzellen), (2) die Expressionsvektoren, (3) das zu verwendende Markierungs- und Affinitätsharz, (4) die Wachstumsbedingungen, (5) die Kulturmengen und (6) die Notwendigkeit einer weiteren Reinigung (z. B. B. Größenausschluss oder Ionenaustauschchromatographie). Spezifische Empfehlungen für diese und andere Aspekte der Methode wurden diskutiert33.
Der in vitro Ubiquitin-Kettenspaltungsassay ist eine einfache Möglichkeit, die deubiquitylierende Aktivität von Wildtyp- oder mutierten DUBs zu messen. Eine zeitabhängige Analyse der Ubiquitin-Spaltung kann Einblicke in die Auswirkungen genetischer Variation auf die enzymatische Kinetik von DUB liefern, indem sie die Bestimmung von Parametern wie Km und Vmax ermöglicht. Dies würde es ermöglichen, zwischen Defekten im aktiven Zentrum von DUB zu unterscheiden, die die Katalyse beeinträchtigen (reduziertes Vmax) und solchen, die die Substraterkennung beeinträchtigen (erhöhtes Km). Die Vielseitigkeit dieses Assays beruht auf der Vielfalt der Ubiquitinketten, die verwendet werden können, um eindeutige mechanistische Informationen über einen DUB von Interesse zu liefern. Ubiquitinketten unterschiedlicher Länge und Kopplungstopologien sind kommerziell erhältlich, so dass dieser Assay auch für Laien zugänglich ist. Spezifische DUBs haben einzigartige Eigenschaften für die enzymatische Kinetik, die Kopplungserkennung und die Spaltung. Zu den kritischen Schritten für diesen Assay gehören (1) die Definition einer Arbeitskonzentration des DUB, (2) die Bestimmung der optimalen Reaktionszeit, (3) die Auswahl des Ubiquitin-Kettengliedertyps und der Länge und (4) die Auswahl einer Nachweismethode.
Ubiquitin-Kettenspaltungsassays messen den Einfluss von NDD-Varianten auf die DUB-Aktivität 7,11 und helfen dabei, Varianten zu identifizieren, die die DUB-Aktivität stören und eine atypische Entwicklung vorantreiben. Dieser Assay kann in einer Reihe von Situationen eingesetzt werden, in denen genetische Variationen die Pathologie bestimmen, einschließlich Krebs und Neurodegeneration. Da es sich um einen substratunabhängigen Assay handelt, ist eine vorherige Identifizierung eines spezifischen ubiquitylierten Proteinsubstrats nicht erforderlich. Der Assay ist jedoch auf die Messung von Veränderungen der Spaltaktivität der Ubiquitinkette beschränkt und kann die Auswirkungen von Varianten auf DUB-Funktionen wie Protein-Protein-Interaktionen, subzelluläre Lokalisation und posttranslationale Modifikationen nicht bewerten. Die Identifizierung der breiten Signalwege, auf denen ein DUB of Interest tätig ist, ist erforderlich, um spezifische Assays zu entwickeln, die untersuchen, wie diese Varianten diskrete NDDs antreiben.
Offenlegungen
Die Autoren erklären, dass keine Interessenkonflikte bestehen.
Danksagungen
Diese Arbeit wurde durch Sanford Research Startkapital für FB und den NIH-Zuschuss R01CA233700 an MJS finanziert. Das Artwork stammt von Felipe G. Serrano (www.illustrative-science.com). Wir danken Dr. Greg Findlay (University of Dundee) für das GST-USP27X-Plasmid.
Materialien
| Name | Company | Catalog Number | Comments |
| Amersham Protran 0.45 NC 200 mm × 200 mm 25/PK | Cytiva | 10600041 | |
| Ammonium sulfate | Fisher Scientific | AC205872500 | |
| Ampicillin | Fisher Scientific | BP1760 25 | |
| Anti- Ubiquitin (Mouse monoclonal) | Biolegend | Cat# 646302, RRID:AB_1659269 | (WB: 1:1000) |
| Anti-GST (Sheep polyclonal) | MRC-PPU Reagents and Services | Cat# S902A Third bleed | (WB: 1:1000) https://mrcppureagents.dundee.ac.uk/ |
| Baffled Culture Flasks 2 L | Fisher Scientific | 10-042-5N | |
| Bradford Reagent | Millipore Sigma | B6916-500ML | |
| Chloramphenicol | Gold Biotechnology | C-105-25 | |
| Complete, Protease Inhibitor tablets | Millipore Sigma | 5056489001 | |
| Econo-Column 1.5 × 5 cm | Bio-Rad | 7371507 | |
| Eppendorf ThermoMixer F1.5 | Eppendorf | 5384000020 | |
| Excel | Microsoft | https://www.microsoft.com/en-us/microsoft-365/excel | |
| Glycerol | Genesee Scientific | 18-205 | |
| Illustrator | Adobe | https://www.adobe.com/products/illustrator.html | |
| Image Studio | LI-COR Biosciences | https://www.licor.com/bio/image-studio/ | |
| Inkscape | Inkscape | https://inkscape.org/ | |
| Invitrogen 4-12% NuPAGE 1mm 12 well gel | Thermo Fisher Scientific | NP0322BOX | |
| IPTG (Isopropyl-b-D-Thiogalactopyranoside) | Genesee Scientific | 20-109 | |
| IRDye 800CW Donkey anti-Goat IgG Secondary Antibody | LI-COR Biosciences | Cat# 926-32214 | (WB: 1:10000) |
| IRDye 800CW Donkey anti-Mouse IgG Secondary Antibody | LI-COR Biosciences | Cat# 926-32212 | (WB: 1:10000) |
| Isotemp Digital Dry Bath | Fisher Scientific | 88860022 | |
| K63 Di-Ubiquitin | South Bay Bio LLC | SBB-UP0072 | |
| LB Agar | Genesee Scientific | 11-119 | |
| LB Broth | Genesee Scientific | 11-118 | |
| Lysozyme | Gold Biotechnology | L-040-100 | |
| MaxQ 4000 Benchtop Orbital Shaker | Thermo Fisher Scientific | SHKE4000-7 | |
| MES-SDS Running Buffer | Boston Bioproducts Inc | BP-177 | |
| Mini Tube Rotator | Fisher Scientific | 88-861-051 | |
| NuPage LDS Sample buffer 4x | Thermo Fisher Scientific | NP0007 | |
| Odyssey Fc Imager | LI-COR Biosciences | 43214 | |
| PageRuler Plus Ladder | Thermo Fisher Scientific | 26620 | |
| pGEX6P1 human USP27X | MRC-PPU Reagents and Services | DU21193 | https://mrcppureagents.dundee.ac.uk/ |
| pGEX6P1 human USP27X F313V | Addgene | 225715 | Koch et at 2024 (PMID: 38182161) |
| pGEX6P1 human USP27X S404N | Addgene | 225717 | Koch et at 2024 (PMID: 38182161) |
| pGEX6P1 human USP27X Y381H | Addgene | 225716 | Koch et at 2024 (PMID: 38182161) |
| Pierce Glutathione Agarose | Thermo Fisher Scientific | 16100 | |
| PMSF (Phenylmethylsulfonyl fluoride) | Gold Biotechnology | P-470-10 | |
| Polysorbate 20 (Tween 20) | Fisher Scientific | AC233360010 | |
| Rosetta 2 Competent Cells | Millipore Sigma | 71402-M | |
| SimplyBlue SafeStain | Thermo Fisher Scientific | LC6060 | |
| SmartSpec 3000 | Bio-Rad | 170-2501 | |
| SOC medium | Thermo Fisher Scientific | 15544034 | |
| Sodium chloride | Genesee Scientific | 18-216 | |
| Sonifier 250 | Branson | 100-132-135 | |
| Sorvall RC 6 Plus Centrifuge | Thermo Fisher Scientific | 46910 | |
| TCEP (Tris-(carboxyethyl) phosphine hydrochloride) | Gold Biotechnology | TCEP10 | |
| Terrific Broth Powder | Genesee Scientific | 18-225 | |
| Tris Base | Genesee Scientific | 18-146 | |
| XCell SureLock Mini-Cell and XCell II Blot Module | Thermo Fisher Scientific | EI0002 |
Referenzen
- Morris-Rosendahl, D. J., Crocq, M. -. A. Neurodevelopmental disorders: the history and future of a diagnostic concept. Dialogues Clin Neurosci. 22 (1), 65-72 (2020).
- Ebstein, F., Küry, S., Papendorf, J. J., Krüger, E. Neurodevelopmental disorders (NDD) caused by genomic alterations of the ubiquitin-proteasome system (UPS): the possible contribution of immune dysregulation to disease pathogenesis. Front Mol Neurosci. 14, 733012 (2021).
- Hershko, A., Ciechanover, A. The ubiquitin system. Annu Rev Biochem. 67 (10), 425-479 (1998).
- Pickart, C. M. Mechanisms underlying ubiquitination. Annu Rev Biochem. 70 (1), 503-533 (2001).
- Komander, D., Clague, M. J., Urbé, S. Breaking the chains: structure and function of the deubiquitinases. Nat Rev Mol Cell Biol. 10 (8), 550-563 (2009).
- Yau, R., Rape, M. The increasing complexity of the ubiquitin code. Nat Cell Biol. 18 (6), 579-586 (2016).
- Koch, I., et al. USP27X variants underlying X-linked intellectual disability disrupt protein function via distinct mechanisms. Life Sci Alliance. 7 (3), e202302258 (2024).
- Hao, Y. -. H., et al. USP7 acts as a molecular rheostat to promote WASH-dependent endosomal protein recycling and is mutated in a human neurodevelopmental disorder. Mol Cell. 59 (6), 956-969 (2015).
- Fountain, M. D., et al. Pathogenic variants in USP7 cause a neurodevelopmental disorder with speech delays, altered behavior, and neurologic anomalies. Genet Med. 21 (8), 1797-1807 (2019).
- Santiago-Sim, T., et al. Biallelic variants in OTUD6B cause an intellectual disability syndrome associated with seizures and dysmorphic features. Am J Hum Genet. 100 (4), 676-688 (2017).
- Beck, D. B., et al. Linkage-specific deubiquitylation by OTUD5 defines an embryonic pathway intolerant to genomic variation. Sci Adv. 7 (4), eabe2116 (2021).
- Cho, J., Park, J., Kim, E. E., Song, E. J. Assay systems for profiling deubiquitinating activity. Int J Mol Sci. 21 (16), 1-16 (2020).
- Matsui, S., Sandberg, A. A., Negoro, S., Seon, B. K., Goldstein, G. Isopeptidase: A novel eukaryotic enzyme that cleaves isopeptide bonds. Proc Natl Acad Sci USA. 79 (5), 1535-1539 (1982).
- Rose, I. A., Warms, J. V. B. An enzyme with ubiquitin carboxy-terminal esterase activity from reticulocytes. Biochemistry. 22 (18), 4234-4237 (1983).
- Wilkinson, K. D., Cox, M. J., Mayer, A. N., Frey, T. Synthesis and characterization of ubiquitin ethyl ester, a new substrate for ubiquitin carboxyl-terminal hydrolase. Biochemistry. 25 (21), 6644-6649 (1986).
- Pickart, C. M., Rose, I. A. Ubiquitin carboxyl-terminal hydrolase acts on ubiquitin carboxyl-terminal amides. J Biol Chem. 260 (13), 7903-7910 (1985).
- Hadari, T., Warms, J. V. B., Rose, I. A., Hershko, A. A ubiquitin C-terminal isopeptidase that acts on polyubiquitin chains: Role in protein degradation. J Biol Chem. 267 (2), 719-727 (1992).
- Woo, S. K., et al. Multiple ubiquitin C-terminal hydrolases from chick skeletal muscle. J Biol Chem. 270 (32), 18766-18773 (1995).
- Woo, S. K., et al. Purification and characterization of a new ubiquitin C-terminal hydrolase (UCH-1) with isopeptidase activity from chick skeletal muscle. J Biochem. 121 (4), 684-689 (1997).
- Baek, S. H., et al. Molecular cloning of a novel ubiquitin-specific protease, UBP41, with isopeptidase activity in chick skeletal muscle. J Biol Chem. 272 (41), 25560-25565 (1997).
- Wilkinson, K. D., et al. Metabolism of the polyubiquitin degradation signal: Structure, mechanism, and role of isopeptidase T. Biochemistry. 34 (44), 14535-14546 (1995).
- Hu, M., et al. Crystal structure of a UBP-family deubiquitinating enzyme in isolation and in complex with ubiquitin aldehyde. Cell. 111 (7), 1041-1054 (2002).
- Hu, M., et al. Structure and mechanisms of the proteasome-associated deubiquitinating enzyme USP14. EMBO J. 24 (21), 3747-3756 (2005).
- Gorka, M., Magnussen, H. M., Kulathu, Y. Chemical biology tools to study deubiquitinases and UBL proteases. Semin Cell Dev Biol. 132, 86-96 (2022).
- Faesen, A. C., et al. The differential modulation of USP activity by internal regulatory domains, interactors, and eight ubiquitin chain types. Chem Biol. 18 (12), 1550-1561 (2011).
- Mevissen, T. E. T., et al. OTU deubiquitinases reveal mechanisms of linkage specificity and enable ubiquitin chain restriction analysis. Cell. 154 (1), 169-184 (2013).
- Kwasna, D., et al. Discovery and characterization of ZUFSP/ZUP1, a distinct deubiquitinase class important for genome stability. Mol Cell. 70 (1), 150-164.e6 (2018).
- Hu, H., et al. X-exome sequencing of 405 unresolved families identifies seven novel intellectual disability genes. Mol Psychiatry. 21 (1), 133-148 (2016).
- Shaker Orbit - Revolving in Space Around the Samples? Eppendorf Lab Academy. Eppendorf Lab Academy Available from: https://www.eppendorf.com/us-en/lab-academy/lab-solutions/others/shaker-orbit-revolving-in-space-around-the-samples (2020)
- Smith, D. B., Johnson, K. S. Single-step purification of polypeptides expressed in Escherichia coli as fusions with glutathione S-transferase. Gene. 67 (1), 31-40 (1988).
- Ritorto, M. S., et al. Screening of DUB activity and specificity by MALDI-TOF mass spectrometry. Nat Commun. 5, 4763 (2014).
- Wingfield, P. T. Overview of the purification of recombinant proteins. Curr Protoc Protein Sci. 80, 6.1.1-6.1.35 (2015).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten