Method Article
ポリトランスフェクションによる細胞状態同定回路の急速開発
要約
複雑な遺伝子回路は、設計、テスト、最適化に時間がかかります。このプロセスを容易にするために、哺乳類細胞は、単一のウェル内の回路コンポーネントの複数の化学量論の試験を可能にする方法でトランスフェクトされます。このプロトコルは、実験計画、トランスフェクション、およびデータ解析の手順を概説しています。
要約
哺乳類の遺伝子回路は、さまざまな病状を感知して治療する可能性を実証していますが、回路コンポーネントのレベルの最適化は依然として困難で労働集約的です。このプロセスを加速するために、私たちの研究室は、従来の哺乳類トランスフェクションのハイスループット拡張であるポリトランスフェクションを開発しました。ポリトランスフェクションでは、トランスフェクションされた集団の各細胞が本質的に異なる実験を行い、異なるDNAコピー数で回路の挙動をテストし、ユーザーがシングルポット反応で多数の化学量論を分析できるようにします。これまでに、細胞の単一ウェルにおける3成分回路の比率を最適化するポリトランスフェクションが実証されています。原則として、同じ方法をさらに大きな回路の開発に使用できます。ポリトランスフェクションの結果は、一過性回路のコトランスフェクションに対するDNAの最適な比率を見つけたり、安定した細胞株を生成するための回路コンポーネントの発現レベルを選択したりするために簡単に適用できます。
ここでは、ポリトランスフェクションを使用して3成分回路を最適化する方法を示します。このプロトコルは、実験デザインの原則から始まり、ポリトランスフェクションが従来のコトランスフェクション法に基づいてどのように構築されるかを説明しています。次に、細胞のポリトランスフェクションを行い、数日後にフローサイトメトリーを行います。最後に、特定の成分比を持つ細胞のサブセットに対応するシングルセルフローサイトメトリーデータのスライスを調べることによって、データを分析します。ラボでは、ポリトランスフェクションを使用して、細胞分類器、フィードバックおよびフィードフォワードコントローラー、双安定モチーフなどを最適化しています。このシンプルでありながら強力な方法は、哺乳類細胞の複雑な遺伝子回路の設計サイクルをスピードアップします。
概要
哺乳類合成生物学の分野は、培養細胞株における単純な感覚応答部分の開発から、診断および治療における現実世界の課題に対処するための遺伝子の複雑なネットワークの最適化まで、急速に進歩しています1。これらの高度な回路は、マイクロRNAプロファイルからサイトカイン、低分子薬物までの生物学的入力を検出し、トランジスタ、バンドパスフィルタ、トグルスイッチ、発振器などのロジック処理回路を実装することができます。彼らはまた、癌、関節炎、糖尿病、その他多くの疾患の動物モデルで有望な結果を示しています1,2,3,4,5。ただし、回路の複雑さが増すにつれて、各コンポーネントのレベルを最適化することはますます困難になります。
特に有用なタイプの遺伝子回路の1つは、細胞状態を感知して応答するようにプログラムすることができる細胞分類器である。特定の細胞状態でのタンパク質またはRNA出力の選択的産生は、細胞およびオルガノイドの分化をガイドおよびプログラムし、疾患細胞および/または望ましくない細胞型を特定および破壊し、治療細胞の機能を調節するための強力なツールです1,2,3,4,5 .しかし、複数の細胞RNAおよび/またはタンパク質種から細胞状態を正確に分類できる回路を哺乳類細胞で作成することは非常に困難でした。
細胞分類回路を開発する最も時間のかかるステップの1つは、回路内のセンサーやプロセシングファクターなどの個々の構成遺伝子の相対的な発現レベルを最適化することです。回路の最適化をスピードアップし、より洗練された回路の構築を可能にするために、最近の研究では、セル分類器回路とそのコンポーネントの数学的モデリングを使用して、最適な構成とトポロジーを予測しています6,7。これはこれまでのところ強力な結果を示していますが、数学的解析は、回路内の構成要素遺伝子の入出力挙動を体系的に特徴付ける必要があるため、時間がかかります。さらに、複雑な遺伝子回路では無数の文脈依存の問題が出現する可能性があり、フル回路の動作が個々の部分の特性評価に基づく予測に反する原因となります8,9。
細胞状態分類器などの複雑な哺乳類回路をより迅速に開発およびテストするために、私たちの研究室は、プラスミドコトランスフェクションプロトコルの進化形であるポリトランスフェクション10と呼ばれる技術を開発しました。コトランスフェクションでは、複数のプラスミドDNA種を正に帯電した脂質または高分子試薬と複合体化し、相関的に細胞に送達します(図1A)。ポリトランスフェクションでは、各トランスフェクション複合体からのDNAが非相関的に細胞に送達されるように、プラスミドを試薬と別々に複合体化します(図1B)。この方法を使用すると、トランスフェクトされた集団内の細胞は、異なる回路コンポーネントを運ぶ2つ以上のDNAペイロードの比率の多数の組み合わせにさらされます。
各細胞に送達される回路成分の比率を測定するために、ポリトランスフェクション内の各トランスフェクション複合体には、複合体の細胞取り込みの代理として機能する構成的に発現された蛍光レポーターが含まれています。哺乳類細胞内で活性な要素を含まないフィラーDNAは、単一のトランスフェクション複合体で細胞に送達される蛍光レポーターと回路成分の相対量を調整するために使用され、議論でより詳細に議論されます。Weissラボで使用されるフィラーDNAの例は、ターミネーター配列を含むプラスミドですが、プロモーター、コード配列などは含まれていません。次に、回路成分の比率が異なる細胞を比較して、遺伝子回路機能の最適な比率を見つけることができます。これにより、回路コンポーネントを遺伝子統合用の単一のベクター(レンチウイルス、トランスポゾン、ランディングパッドなど)に組み合わせるときに最適な遺伝子発現レベルを達成するために、プロモーターやその他の回路要素を選択するための有用な予測が得られます。したがって、ポリトランスフェクションは、直感に基づいて、または時間のかかる試行錯誤プロセス を介して 回路コンポーネント間の比率を選択する代わりに、シングルポット反応で遺伝子部分間の幅広い化学量論を評価します。
私たちの研究室では、ポリトランスフェクションにより、細胞分類器、フィードバックおよびフィードフォワードコントローラー、双安定モチーフなど、多くの遺伝子回路の最適化が可能になりました。このシンプルでありながら強力な方法は、哺乳類細胞の複雑な遺伝子回路の設計サイクルを大幅にスピードアップします。それ以来、ポリトランスフェクションは、いくつかの遺伝子回路の特性評価に使用され、高解像度で多次元入出力伝達関数を明らかにし10、細胞状態分類11の代替回路トポロジーを最適化し、さまざまな公開された12、13 および進行中のプロジェクトを加速しています。
ここでは、ポリトランスフェクションを使用して遺伝子回路を迅速に最適化するためのワークフローについて説明し、説明します(図2)。このプロトコルは、高品質のポリトランスフェクションデータを生成し、ポリトランスフェクションプロトコルおよびデータ解析におけるいくつかの一般的なエラーを回避する方法を示しています(図3)。次に、ポリトランスフェクションを使用して単純な回路コンポーネントの特性評価を行い、その過程でポリトランスフェクションの結果をコトランスフェクションに対してベンチマークする方法を示します(図4)。最後に、ポリトランスフェクションの結果は、がん分類回路の最適化を示しています(図5)。
プロトコル
メモ: 表 1 および 表 2 は、このプロトコルの重要なリファレンスです。表 1 は反応の試薬スケーリングを示し、 表2 はプロトコルに記載されているポリトランスフェクションの例(上半分)と可能なフォローアップ実験(下半分)のDNA比算術を示しています。
1. トランスフェクションのための細胞の準備
- プロトコルを開始する前に、ヒト胚性腎臓(HEK293)細胞の培養が60%〜80%コンフルエントであることを確認してください。これを行うには、2日前に100 mm x 15 mmの組織培養ペトリ皿に1 x 106 細胞を播種し、5%CO2で37°Cでインキュベートします。
注:私たちのプロトコルはHEK293細胞に焦点を当てていますが、他の細胞タイプで代用することもできます。 - 以下に説明するように、トランスフェクション用の培地と細胞を準備します。
- 10%ウシ胎児血清(FBS)および1%非必須アミノ酸(NEAA; 材料の表を参照)を含むダルベッコ改変イーグル培地(DMEM)の溶液の少なくとも20 mLを37°Cに予熱します。 少なくとも2.4 mLのトリプシンと2.4 mLのリン酸緩衝生理食塩水(PBS)も37°Cに予熱します。血清培地を~16°Cに予温します。
注意: すべての組織培養作業は、バイオセーフティキャビネットで注意して実行する必要があります。
- 10%ウシ胎児血清(FBS)および1%非必須アミノ酸(NEAA; 材料の表を参照)を含むダルベッコ改変イーグル培地(DMEM)の溶液の少なくとも20 mLを37°Cに予熱します。 少なくとも2.4 mLのトリプシンと2.4 mLのリン酸緩衝生理食塩水(PBS)も37°Cに予熱します。血清培地を~16°Cに予温します。
- 以下に説明するように、細胞をDMEM溶液に再懸濁します。
- 現在のメディアを吸引して廃棄します。HEK293細胞培養液に2 mLのPBSを分注し、細胞を洗浄します。PBSを吸引して廃棄します。
- 2 mLのトリプシンをHEK293細胞培養液に分注します。ペトリ皿を37°Cのインキュベーターに3分間、または細胞が皿に接着しなくなるまで入れます。ディッシュをバイオセーフティキャビネットに戻し、8 mLのDMEM溶液をプレートに分注して細胞溶液を希釈します。
- 数回穏やかに上下にピペッティングして溶液を混合します。すべての培地を吸引し、15 mLのコニカルチューブに入れます。
- 細胞を300 x g で3分間遠心分離し、ペレット化します。培地を吸引し(細胞を吸引しないように注意して)、廃棄します。細胞を5 mLのDMEM溶液に再懸濁し、穏やかに上下にピペッティングして混合します。
- 自動セルカウンター( 材料表に記載)を使用して現在の細胞濃度を推定します。6 ウェル (この例では) を 1 x 105 セル (播種密度 50,000 セル/cm2 の場合) の 24 ウェルプレートにシードします。
- DMEM溶液を最大500 μLまで添加し(最初にDMEM溶液をウェルに加えます)、次に処理ごとに1つのウェルにカラーコントロールなし、mKO2コントロール、TagBFPコントロール、NeonGreenコントロール、オールカラーコントロール、ポリトランスフェクション1のラベルを付けます。TagBFP、mKO2、およびNeonGreenコントロールウェルは、ポリトランスフェクションに含まれるすべての蛍光タンパク質の単色コントロールです。
2. トランスフェクションの実施
- 各DNA凝集体用のチューブを準備します。1.5 mLの微量遠心チューブを取っておき、チューブにカラーコントロールなし、mKO2コントロール、TagBFPコントロール、NeonGreenコントロール、オールカラーコントロール、ポリトランスフェクションミックス1、ポリトランスフェクションミックス2のラベルを付けます。
- 36 μLの還元血清培地を、カラーコントロールなし、mKO2コントロール、TagBFPコントロール、ネオングリーンコントロール、およびすべてのカラーコントロールチューブに追加します。18 μLの還元血清培地をポリトランスフェクションミックス1およびポリトランスフェクションミックス2のチューブのそれぞれに加えます。
注:プラスミド濃度は150 ng/μLを想定しています。 - 600 ngのフィラープラスミドをカラーコントロールチューブなしに追加します。300 ngのmKO2と300 ngのフィラープラスミドをmKO2カラーコントロールチューブに追加します。300 ngのTagBFPと300 ngのフィラープラスミドをTagBFPカラーコントロールチューブに追加します。
- 300 ngの構成的ネオングリーンプラスミドと300 ngのフィラープラスミドをネオングリーンカラーコントロールチューブに追加します。mKO2、TagBFP、構成ネオングリーンをそれぞれ100 ng、およびフィラープラスミド300 ngをオールカラーコントロールチューブに追加します。
- 150 ngのmKO2をポリトランスフェクションミックス1チューブに加えます。75 ngのレポーターNeonGreenプラスミドと75 ngのフィラープラスミドをポリトランスフェクションミックス1チューブに加えます。
- 150 ngのTagBFPをポリトランスフェクションミックス2チューブに加えます。75 ngのL7aeプラスミドと75 ngのフィラープラスミドをポリトランスフェクションミックス2チューブに加えます。
- 36 μLの還元血清培地を、カラーコントロールなし、mKO2コントロール、TagBFPコントロール、ネオングリーンコントロール、およびすべてのカラーコントロールチューブに追加します。18 μLの還元血清培地をポリトランスフェクションミックス1およびポリトランスフェクションミックス2のチューブのそれぞれに加えます。
- 216 μLの還元血清培地と9.48 μLのトランスフェクション試薬を組み合わせて、1.5 mLの微量遠心チューブでトランスフェクションマスターミックスを作成します(試薬比と反応スケーリングについては 表1 を参照)。上下にピペッティングしてよく混ぜ、取っておきます。
- 1.58 μLのエンハンサー試薬を、カラーコントロールなし、シングルカラーコントロール、およびすべてのカラーコントロールチューブのそれぞれに追加します。79 μLのエンハンサー試薬を各ポリトランスフェクションミックスチューブに加えます。激しくピペッティングして各チューブを個別に混合します。
- DNAを含むトランスフェクションマスターミックスを各チューブに追加します。
- 37.58 μLのトランスフェクションマスターミックスを、カラーコントロールなし、シングルカラーコントロール、およびすべてのカラーコントロールチューブのそれぞれに追加します。激しくピペッティングして各チューブを個別に混合します。
- 18.79 μLのトランスフェクションマスターミックスを各ポリトランスフェクションミックスチューブに加えます。激しくピペッティングして各チューブを個別に混合します。
- トランスフェクションミックスをウェルに分注します。
- 各トランスフェクション 65.97 μL をピペットで、無色、単色、およびすべてのカラーコントロール用に、対応するウェルに入れます。
- 32.98 μLのポリトランスフェクションミックス1をポリトランスフェクションウェルにピペットで入れ、プレートを平坦な表面に沿って8の字型パターンで素早く穏やかに旋回させ、複合体を効果的に分配します。次に、32.98 μLのポリトランスフェクションミックス2を同じポリトランスフェクションウェルにピペットで入れ、同じ方法でプレートを旋回させます。
- プレートを37°Cのインキュベーターに入れ、5%CO2 と共に振とうせずに48時間放置する。
注:細胞の生存率を高めるために、トランスフェクション後6時間ごとに細胞培地を交換することができます(ただし、これは必ずしも必要ではなく、HEK293細胞とその誘導体では、培地を交換するときに細胞をプレートから剥離しないように注意する必要があります)。
| 試薬 | 量 | スケーリング | ||||
| DNA混合用の還元血清培地 | コントロールチューブあたり36 μL、ポリトランスフェクションチューブあたり18 μL | チューブあたり0.05 μLの血清培地/ ng DNAを減少させ、ピペッティングを考慮して10〜20%余分な容量を使用 | ||||
| デオキシリボ核酸 | チューブあたり300-600 ng | |||||
| P3000型 | コントロールチューブあたり1.58 μL、ポリトランスフェクションチューブあたり0.79 μL | チューブあたり0.0022 μL P3000/ng DNA、10-20%追加 | ||||
| リポマスターミックス用の還元血清培地 | コントロールチューブあたり36 μL、ポリトランスフェクションチューブあたり18 μL | 0.05 μLの還元血清培地/ ng総DNA、ピペッティングを考慮して10〜20%の追加容量 | ||||
| トランスフェクションおよびエンハンサー試薬 | コントロールチューブあたり1.58 μL、ポリトランスフェクションチューブあたり0.79 μL | 0.0022 μLリポフェクタミン3000/ng DNA、10-20%追加 | ||||
表1:トランスフェクションの試薬スケーリング。 この表は、単一のウェルに含まれるDNA量に含める試薬の正しい比率を示しています。これは、反応を効果的にスケーリングし、マスターミックスを形成するために使用できます。試薬の量は、20%の過剰を含むようにスケーリングされています。
3. フローサイトメトリー用の細胞調製
- 少なくとも4.2 mLのDMEM溶液を37°Cに予熱します。 少なくとも4.2 mLのトリプシンと4.2 mLのPBSを37°Cに予熱します。 蛍光活性化セルソーティング(FACS)緩衝液は、使用できるようになるまで4°Cに保ちます。
- 細胞をFACS緩衝液(1%BSA、5 mMエチレンジアミン四酢酸[EDTA]、および0.1%アジ化ナトリウム[NaN3]を添加したPBS)に再懸濁して、凝集を減らします; 材料の表を参照)。
- 各ウェルに現在の培地を吸引して廃棄します。5 mLのPBSを各ウェルに分注し、細胞を洗浄します。PBSを吸引して廃棄します。
- 5 mLのトリプシンを各ウェルに分注します。プレートを37°Cのインキュベーターに3分間、または細胞が皿に接着しなくなるまで置きます。プレートをバイオセーフティキャビネットに戻し、5 mLのDMEM溶液を各ウェルに分注して細胞溶液を希釈します。
- 各ウェルについて、数回穏やかに上下にピペッティングして溶液を混合します。各ウェルについて、すべての培地を吸引し、15 mLのコニカルチューブに入れます。
- 細胞を300 x g で3分間遠心分離し、ペレット化します。細胞を吸引して廃棄しないように注意しながら、培地を吸引します。各チューブで、細胞を5mLのFACS緩衝溶液に再懸濁し、上下に穏やかにピペッティングして混合する。
- 各細胞懸濁液をストレーナーに通し(凝集塊を除去するため)、別々のフローサイトメトリーコニカルチューブに入れます。これらのチューブを氷上に1時間以内保持し、できるだけ早くフローサイトメトリーを実行してください。
4. フローサイトメトリーの実施
注:フローサイトメーターの操作には、適切なトレーニングと必要なタスクの知識が必要です。ソフトウェアと機器は異なる場合があり、ユーザーは一般的にトレーニングを受ける必要があるため、このセクションでは、実行に役立つ特定の操作について説明します。
- まず、フィラープラスミドコントロール(カラーコントロールなし)を導入した細胞を調べて、細胞特性を選択し、異常(凝集体、破片などを含む)を回避します。セルを区別するためのパラメーターの組み合わせは多数ありますが、特徴を区別するための良い方法として、次の 3 つの一般的なオプションを使用します。
- 側方散乱領域 (好み/セルの種類ごとの対数または線形スケール) と前方散乱領域 (線形スケール) を確認します。
- 側面散乱高さ (対数スケール) と側面散乱幅 (線形スケール) を確認します。
- 前方散乱幅 (線形スケール) と前方散乱高さ (線形スケール) を確認します。
- 次に、単色コントロールを見てください。オールカラーコントロールを使用して、各蛍光タンパク質からのシグナルが同等の任意の蛍光単位(a.u.)に正規化されるように、機器の電圧を調整します。次に、補正マトリックスの設定に使用される各蛍光タンパク質の単一カラーコントロールを実行し、ブリードスルー補正を可能にします。
注:理想的には、蛍光値の全ダイナミックレンジが見える必要があります。蛍光タンパク質シグナルのさらなる正規化は、標準化された単位(例えば、等価フルオレセイン[MEFLs]の分子;Beal et al.15を参照)への変換を介して行うことができる。分析中にMEFL変換を有効にするには、レインボーキャリブレーションビーズを実行します。このような較正は、機器間および日々の信号変動を低減するためにも有用である16。 - ポリトランスフェクションサンプルチューブを実行します。
注:分析中に高次元のポリトランスフェクションをより多くのビンに細分化する必要があり、各ビンには統計的に有意な比較を行うのに十分な細胞(理想的には>10)が必要であるため、可能であれば、1,000 x 10^(^=ミックス)の細胞を実行することをお勧めします。
5. 実験後解析の実行
- 最初に、コントロール(および該当する場合はビーズ)からのデータを使用して、正確な結果を確保します。利用可能なソフトウェアツールの1つを使用して、生細胞ゲーティング(上記のゲートを使用)、補償、および自己蛍光補正を実行します。
注:通常、グラフィカルユーザーインターフェイスと、前処理段階およびポリトランスフェクション分析に適したPythonライブラリの両方を備えたカスタムMATLABコード(https://github.com/jonesr18/MATLAB_Flow_Analysis またはCytoflow17など)を使用します。
| 方式 | コンプレックス | ng蛍光マーカー | ng L7ae (分数) | ngレポーター(分数) | ngフィラーDNA(フラクション) | 合計 (ng) |
| ポリトランスフェクション1 | 600 | |||||
| → | 1 | 150 | 75 (½) | 75 (½) | 300 | |
| → | 2 | 150 | 75 (½) | 75 (½) | 300 | |
| ポリトランスフェクション2 | 600 | |||||
| → | 1 | 150 | 25 (1/6) | 125 (5/6) | 300 | |
| → | 2 | 150 | 125 (5/6) | 25(1/6) | 300 |
表2:プロトコルで実証されたポリトランスフェクションのDNA量、および調整されたプラスミド比を使用したフォローアップ実験の例。 表の上半分は、単純なポリトランスフェクション実験で使用したプラスミドの組成を示しています。下半分は、プラスミド比を調整して仮想の濃度空間をより適切にサブサンプリングする最新の実験の構成を示しており、遺伝子発現モジュレーターはレポーターに対してより最適な1:5の比率にあり、この比率付近でより多くのトランスフェクト細胞をサンプリングします。
結果
図1では、コトランスフェクションとポリトランスフェクションを比較しています。コトランスフェクションでは、すべてのプラスミドが同じトランスフェクションミックスで送達されるため、単一細胞が受け取る各プラスミドの量に高い相関が生じます(図1A)。各細胞に送達されるプラスミドの総数は大きく異なりますが、集団全体の個々の細胞における2つのレポータータンパク質の蛍光はよく相関しており、2つのプラスミドがかなり一定の比率で同時送達されていることを示しています。トランスフェクションされた細胞は、ほとんどまたは多くの複合体を取り込むことができますが、各複合体は各プラスミドの相関量を持っているため、コトランスフェクションは2つのプラスミド間の濃縮空間の小さな対角領域のみを探索します。対照的に、ポリトランスフェクションでは、プラスミドは複数のトランスフェクション複合体で送達されるため、異なるトランスフェクションミックスでのプラスミドの非相関送達が得られます(図1B)。トランスフェクトされた細胞は、複合体の異なる組み合わせを取り込み、その結果、どちらでもない、一方または両方の複合体からのプラスミドの異なる投与量を含む細胞が得られます。
図2は、代表的なポリトランスフェクションワークフローを示しています。一般に、ポリトランスフェクション実験は、問題の定義、トランスフェクションミックスの作成、細胞のインキュベート、フローサイトメトリー、および分析のステップで構成されます。また、最初のポリトランスフェクションの結果で最適な部品比を正確に絞り込むことができない場合は、最適化された部品比でポリトランスフェクションを繰り返すオプションのステップもあります。これらの手順は、プロトコルで概説されています。
図3では、良好に実行されたコトランスフェクションおよびポリトランスフェクションと一般的なエラーの例をいくつか示します。図3Aは、TagBFPと共送達されたeYFP発現プラスミドとの間に密接な相関がある、十分に実行された同時トランスフェクションを示しています。対照的に、図3Bのコトランスフェクションは、これら2つのプラスミド間の相関が低いことを示しています。図示において、相関不良は、プラスミドが添加される前の還元血清培地にエンハンサー試薬を添加したことによるものである。実験におけるこのような相関の悪さは、蛍光タンパク質の発現を促進する異なるプロモーター、トランスフェクションミックス作成中の混合不良、または複合体のインキュベート間隔が短すぎたり長すぎたりすることが原因である可能性もあります。
図3C は、2次元空間の良好なカバレッジと蛍光タンパク質間のスペクトルブリードスルーの良好な補償を備えた、十分に実行されたポリトランスフェクションを示しています。 図3D は、生細胞数が少ないポリトランスフェクションデータを示していますが、解析のために各ビンに十分な数の細胞がある十分なビンに細分化することは困難です。この問題を改善するには、開始細胞集団が健康で過大化していないこと、および別のトランスフェクション試薬を使用したり、総DNA数を減らしてトランスフェクションしたりすることで実験毒性が低下する必要があります。 図3E は、トランスフェクション効率が悪く、分析用の2次元空間のカバレッジがまばらになったポリトランスフェクションを示しています。この場合、形態ゲーティングを通過する細胞の数は多いが、その細胞は蛍光タンパク質を発現しない。効率を向上させるには、トランスフェクション試薬の選択を最適化し、メーカーのプロトコルに厳密に従う必要があります。各トランスフェクションミックスには等量のDNA質量が含まれている必要があり、細胞が各タイプの複合体を受け取る可能性がほぼ等しくなるようにし、各トランスフェクションミックスはキットの指示に従って作成する必要があります。異なるトランスフェクションキットは、異なる細胞タイプに最適です。目的の細胞タイプで最高の効率を与えるキットを使用する必要があります。最後に、一部の細胞タイプは、使用するキットに関係なく、トランスフェクションが困難です。このような場合は、より多くの細胞をトランスフェクトし、フローサイトメトリー中にできるだけ多くの細胞を収集して、分析するのに十分な数の細胞を確保する必要があります。 図3F は、蛍光タンパク質マーカーの1つが別の蛍光タンパク質へのスペクトルブリードスルーを明確に示しているポリトランスフェクションデータを示しています。システム内のすべての蛍光タンパク質に対して常に単色コントロールを実行し、分析前にすべてのポリトランスフェクションに適用する必要がある補償マトリックスを生成するために使用する必要があります。
ポリトランスフェクション実験を初めて(全体的または新しい実験システムに対して)実施する場合は、標準的なコトランスフェクションに対してベンチマークを行う必要があります。1つのアプローチは、コトランスフェクション(DNA投与量の調整による )とポリトランスフェクションの両方を使用して、主要なシステム部品の入出力伝達関数を個別に測定することです。
図4では、元のポリトランスフェクション出版物10からここに適応されたトランスレーショナルリプレッサーL7Aeのベンチマークを示しています。L7Aeは、RNAキンクターン(KT)モチーフを認識するRNA結合タンパク質である20;2つのKT(2xKT)がmRNAの5'非翻訳領域(UTR)に配置されている場合、下流のオープンリーディングフレーム(ORF)翻訳はL7Ae21によって効果的に抑制できます。L7Aeは、RNAベースの細胞型分類器21および他のRNAベースの回路22を構築するために以前に使用されている。標準的なコトランスフェクションでL7Aeを試験するために、異なるトランスフェクションミックス内でL7Aeをコードするプラスミドとその標的レポーターの比率を調整することができます(図4Aおよび表3)。ポリトランスフェクションでは、構成的L7Aeと対応する2xKTレポーターを別々のトランスフェクションミックスで別々に送達し、広範囲のプラスミド比が細胞に送達されるようにする必要があります(図4B)。トランスフェクションおよびフローサイトメトリーを実行した後、データ解析を行うことができます。データを比較可能にするには、個々のコトランスフェクション結果を単一のデータセットに照合し、ポリトランスフェクションデータに対して同様の多次元ビニングを実行します(図4C、D)。出力レポーターを抑制するL7Aeの用量反応曲線を比較すると、ビンあたりの出力レベルの中央値は、コトランスフェクションデータとポリトランスフェクションデータの間で非常に類似していることがわかります(図4E-G)。
一般に、ポリトランスフェクションデータは、幅広いDNA比にわたってコトランスフェクションデータとよく相関し、非常に歪んだDNA投与比では精度が低くなることがわかっています(ポリトランスフェクション実験では、特に低プラスミド投与量で非常に強く活性な部分では、ビン内の細胞被覆率が低いことが一因です)。このような敏感な部分の測定精度を向上させるために、より弱いプロモーター、上流のオープンリーディングフレーム18、またはマイクロRNAサイレンシング媒介ファインチューナー(miSFIT)19を使用して発現レベルを下げることができます。
図5は、細胞型分類器の最適化のためのポリトランスフェクションの適用の成功を示しており、これもGamら10から適応されている。この分類器は、miR−21−5pの発現に応答して出力を生じる比較的単純な設計であり、多くの腫瘍細胞23において過剰発現しているmiRNAである。miR-21の非存在下では、分類器の出力は細菌由来の転写抑制因子BM3R124によって抑制され、その適応は哺乳類細胞で機能することが以前に示された25。miR-21が存在する場合、BM3R1の3'UTRと5'UTRの両方に配置された4つの標的部位に結合し、その発現をノックダウンして出力転写を可能にします(図5A)。このシステムを最適化するために、(1)BM3R1(miR-21標的部位を含む)、(2)アウトプットレポーター(mKO2)、および(3)出力の転写を活性化するGal4-VP16の3つの回路コンポーネントを別々のトランスフェクションミックスで送達しました(表4)。なお、出力促進剤はBM3R1ではなくロジック(Gal4-VP16)で動作し、Gal4-VP1625の存在下でも出力を抑制する。各部分は、各複合体の相対的なDNA投与量を示すために、同じプラスミド上にトランスフェクションマーカーであるTagBFP、mNeonGreen、およびiRFP720をそれぞれコードしています。一般に、Gal4-VP16発現が増加するとレポーターmKO2出力が増加し、BM3R1発現が増加するとmKO2出力が減少するはずです。BM3R1はmiR-21-5pによってノックダウンされるため、出力発現は、miR-21-5pのレベルが高く、HEK細胞よりもBM3R1の発現が少ないHeLa細胞で高くなるはずです。
HEK293細胞とHeLa細胞の両方にポリトランスフェクションした後、異なるプラスミド比の3D分布を得ました(図5B-HEK細胞)。Gam et al.10 は、関心のある比率から特定のユークリッド距離内の細胞を考慮して、この分布をさまざまな比率でサブサンプリングしました。この距離は、解析から統計的に有意な結果が得られるのに十分なセルを含むのに十分な幅である必要がありますが、3 つの部分の成分比のセットが広すぎることによる不要なノイズを回避するのに十分な幅である必要があります。次に、Gam et al.10 は最適化アルゴリズムを使用して、コトランスフェクションの分類精度を最大化する部分の比率を特定しました(つまり、トランスフェクトされたHeLa細胞は出力発現に対して陽性ですが、トランスフェクションされたHEK細胞は陽性ではありません)。部品の最適な比率は10.9:1.5:1であることがわかりました:Gal4-VP16:出力:BM3R1;ポリトランスフェクション空間全体における最適な比率付近でサブサンプリングした細胞を 図5Cに示します。このサブサンプリングでは、この比率で、HEK293とHeLa細胞を分類する際に、共トランスフェクトされた回路の特異度は91%、感度は62%、精度は77%であると予測されました(図5D)。プラスミド比をこの最適に設定した同時トランスフェクションでは、99%の特異性、68%の感度、84%の精度というさらに優れた結果が得られました10。さらに、この比率は、異なる切断プロモーターおよび上流ORF(uORF)を用いて相対発現を調整した単一プラスミドバージョンの回路の実装を導き、91%の特異性、90%の感度、および90%の精度を有する回路をもたらした10。したがって、ポリトランスフェクションは、細胞分類器および遺伝子回路の設計をより広く導くための強力なツールです。

図1:コトランスフェクションとポリトランスフェクションの比較 。 (A,B)2つのプラスミドのコトランスフェクションおよび2つのプラスミドのポリトランスフェクションによるプラスミドデリバリーの概要と比較。各トランスフェクション法について、左端の図は、負に帯電したDNAと正に帯電した脂質との間のトランスフェクション複合体の形成を示しています。これらの実施例において、各着色プラスミド(青および赤)は、異なる蛍光タンパク質の発現をコードする。中央の図は、細胞へのプラスミド送達の例と、ヒストグラムまたは散布図で予想される分布の概略図を示しています。ヒストグラム上の色強度は、対応するプラスミド色からの蛍光に対応する。右端の図は、与えられた各方法を使用してトランスフェクトされた細胞からの実際のデータを示しています。(A)2つの異なるプラスミドを使用した同時トランスフェクションでは、トランスフェクション試薬を添加する前に両方のプラスミドを混合し、2つのプラスミド種のパッケージングに高い相関性を持たせます。実際のコトランスフェクションデータでは、細胞は両方のプラスミドの相関送達を示します(右)。(B)ポリトランスフェクションでは、回路部分とトランスフェクションマーカーに対応する共送達プラスミドの各セットをトランスフェクション試薬と別々に混合し、それらのプラスミドのみを含む複合体を生成します(左)。実際のポリトランスフェクションデータでは、細胞は広範囲の濃度空間を探索し、多くの異なるプラスミド化学量論を同時に探索します(右)。この数値は10 から変更されました。 この図の拡大版を表示するには、ここをクリックしてください。

図2:ポリトランスフェクションワークフロー。 ステップ1:問題を定義します。部品間の理想的な比率が不明な複数の構成部品を含むシステムから始めて、構成部品間の開始比を選択します。ステップ2:トランスフェクションミックスを作成します。各トランスフェクションミックスには、回路コンポーネントと蛍光トランスフェクションマーカーが含まれています。細胞に送達される回路部分の量は、マーカーの蛍光と相関します。ステップ3:細胞をインキュベートします。一般的なワークフローでは、トランスフェクションとフローサイトメトリーの間に48時間細胞をインキュベートします。ステップ4:フローサイトメトリー。プロトコルで説明されているように、適切なコントロールを実行し、サンプルを実行します。ステップ5:分析:トランスフェクションマーカーを使用して、細胞が受け取った部品の量または比率に応じて細胞をビンビン化します。出力タンパク質を使用して回路性能を測定します。回路性能を最適化する部品のビン/比率を見つけます。ステップ6:最適化された部品比率で繰り返します(オプション)。最適なビン/比の比率が非常に歪んでいる場合、パイロットポリトランスフェクションは最適な部品比を正確に絞り込めない可能性があります。各トランスフェクションミックスを同量投与した細胞が、前のラウンドで決定された最適に近い部品比率を受け取るように、調整された部品比でポリトランスフェクションを繰り返します。この数値は10 から変更されました。インキュベーターの画像はセルヴィエメディカルアートから、サイトメーターの画像はBioRenderから使用されました。 この図の拡大版を表示するには、ここをクリックしてください。
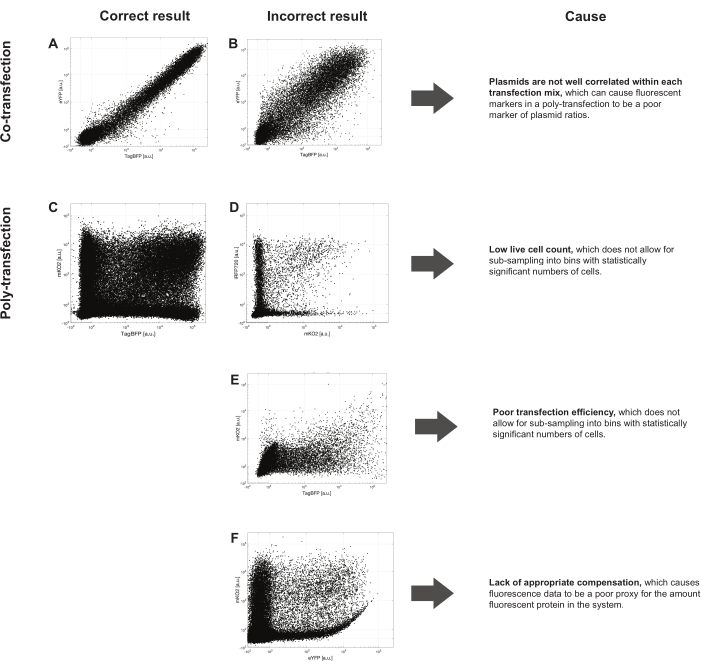
図3:代表的なポジティブおよびネガティブなポリトランスフェクション結果 。 (A)2つの蛍光レポーターの良好な同時トランスフェクションは、密接な相関を示しています。合計20,000個のセルが、比較のためにここと(B)の両方にプロットされています。ポリトランスフェクション実験を開始する前に、2つの蛍光レポーターの同様の小規模試験コトランスフェクションを実行して、プラスミドがトランスフェクションミックス内で十分に相関していることを確認することをお勧めします。(B)ノイズの多いコトランスフェクション:プラスミドは各トランスフェクションミックス内で十分に相関していないため、ポリトランスフェクションの蛍光マーカーがプラスミド比のマーカーとして不十分になる可能性があります。(C)良好な細胞数、トランスフェクション効率、および補償を示す良好なポリトランスフェクション結果。(D)生細胞数が少ないため、統計的に有意な細胞数を持つビンへのサブサンプリングはできません。(E)トランスフェクション効率が低く、統計的に有意な細胞数を持つビンへのサブサンプリングができない。(F)適切な補償がないため、蛍光データがシステム内の蛍光タンパク質量の代用として不十分になります。単色コントロールを使用して理想的な線形補正行列を決定し、さらに処理する前にデータに適用します。ソフトウェアの選択に応じて、すべてのカラーコントロールも推奨されます。 この図の拡大版を表示するには、ここをクリックしてください。

図4:ポリトランスフェクションとコトランスフェクションのベンチマーク。 (A)コトランスフェクションでは、各実験ウェルで蛍光レポータープラスミドに対する翻訳リプレッサーL7Aeの比率を1つずつ試験できます。(B)ポリトランスフェクションでは、L7Aeとレポーターの多くの比率を同じウェルで試験できます。ポリトランスフェクションプラスミドミックスの詳細については、表3を参照してください。(C,D)複数のコトランスフェクションに対するポリトランスフェクションのベンチマークを行うためのビニングワークフロー。トランスフェクションマーカーの寸法ごとに合計10個の双指数間隔のビンを割り当て、2つのプラスミドそれぞれのレベルを近似しました。ここでは、異なるレベルのプラスミド#2を示すビンが示されている。ビニングは、さまざまなプラスミド比(C)と、それぞれ約500,000細胞を含む単一のポリトランスフェクションからのデータ(D)にまたがる11のコトランスフェクションサンプルの照合セットの両方に対して実行されました。色は、遺伝子2(TagBFP)レベルによって定義されるビンのセットに対応します。(E-G)代表的な回路のコトランスフェクションに対するポリトランスフェクションのベンチマーク。中央値出力蛍光を各ビン内の細胞について評価し、代表的な系であるL7Ae翻訳抑制の方法間で比較した。(e)L7Ae活性を測定するための構築物。mKO2蛍光はL7Ae(遺伝子#1)の送達の推定値として機能し、TagBFP蛍光は調節されたmNeonGreen出力(遺伝子#2)の送達の推定値として機能します。(F)L7Aeの多次元滴定曲線。各行は、(C) および (D) に示すように、TagBFP の 1 つのレベルにあるビンのセットを表します。実線はポリトランスフェクションデータを示し、破線はコトランスフェクションデータを示します。レポータープラスミドの各ビン化レベルでは、L7Aeが増加するにつれてレポーター出力が減少します。(G)L7Aeシステムのコトランスフェクションとポリトランスフェクションの視覚的比較。各ポイントは、ポリトランスフェクションおよびコトランスフェクション測定の対応するビンで測定された出力を表し、同等の値は赤い1:1の線に近いほど大きくなります。全体として、ポリトランスフェクション由来とコトランスフェクションの間に観察される差は低く、ポリトランスフェクション法の信頼性に自信があります。この数値は10 から変更されました。この図の拡大版を表示するには、ここをクリックしてください。

図5:ポリトランスフェクションのデータ解析。 ポリトランスフェクションでは、細胞をビンまたはスライスにグループ化して分析することができます。 図4 は、レポータープラスミドレベルなどのシステムコンポーネントのレベルに対応するスライスに細胞をビン化する分析形式を示しており、モデルのフィッティングと投与量反応の理解に役立ちます。別の有用な戦略は、回路コンポーネントのさまざまな比率で回路性能を分析することです。(A)最適化のための分級回路の図。TagBFP、NeonGreen、およびiRFP720のレベルは、それぞれBM3R1、出力mKO2、およびGal4-VP16のレベルに対応します。ポリトランスフェクションプラスミドミックスの詳細については 、表4 を参照してください。(B)ポリトランスフェクションミックスの実験セットアップ。(C)(A)の回路を用いたポリトランスフェクションから収集したフローサイトメトリーデータ。HEK細胞への回路ポリトランスフェクションの結果としての3つのレポーター蛍光タンパク質のそれぞれのレベルは、データに存在する広範囲の回路成分比を示しています。同様の結果は、HEK細胞とHeLa細胞の両方で回路トランスフェクションからも得られました。(D)特定の比率でのポリトランスフェクションのサブサンプリング。データを分析するために、回路コンポーネント間の多数の比率をスキャンし、各比率での分類器の性能を決定しました。例として、良好な性能を示した1つの特定の比率(Gal4-VP16 = 435 ngのDNA、レポーター= 60 ng、およびBM3R1 = 40 ng)を示します。青色でプロットされているのは、3つの異なる回路成分に対する蛍光マーカーの対応する比率です。次に、蛍光軌道から特定のユークリッド距離内の点のみを考慮してデータをサブサンプリングしました。HEKトランスフェクションとHeLaトランスフェクションの両方から同じ軌道の細胞をサブサンプリングしました。(E)HEKセルとHeLaセルの同一軌道での回路性能の比較。感度、特異度、分類精度などの統計を多くの比率で比較することにより、成分比を最適化して理想的な遺伝子回路を生成することができます。この数値は10 から変更されました。 この図の拡大版を表示するには、ここをクリックしてください。
| コンプレックス | ng L7Aeプラスミド | ng レポータープラスミド | オプティメム (μL) | P3000 (μL) | リポ 3000 (μL) |
| 1 | 250 | 75 | 1.5 | 1.5 | |
| 2 | 250 | 75 | 1.5 | 1.5 |
表3:図4に対応するトランスフェクションミックス。 HEK293細胞を、これらのトランスフェクションミックスとともに、24ウェルプレートの1ウェルにポリトランスフェクトしました。
| コンプレックス | ng BM3R1プラスミド | ng Gal4-VP16プラスミド | ng レポータープラスミド | オプティメム (μL) | P3000 (μL) | リポ 3000 (μL) |
| 1 | 900 | 75 | 1.5 | 1.5 | ||
| 2 | 900 | 75 | 1.5 | 1.5 | ||
| 3 | 900 | 75 | 1.5 | 1.5 |
表4: 図5に対応するトランスフェクションミックス。 HEK293およびHeLa細胞をそれぞれ、これらのトランスフェクションミックスを1ウェルそれぞれ6ウェルプレートにポリトランスフェクトした。
ディスカッション
コンピューター支援設計(CAD)、ブレッドボード、3D印刷などのラピッドプロトタイピング手法は、機械、電気、土木工学の分野に革命をもたらしました。特定の課題に対して考えられる多くのソリューションをすばやく検索する機能は、この分野の進歩を大幅に加速します。ポリトランスフェクションは生物工学の類似技術であり、遺伝子回路のラピッドプロトタイピングを可能にすると考えています。さらに、他のラピッドプロトタイピング技術では、複数の可能な溶液をハンズオンで順次反復する必要がありますが、ポリトランスフェクションは多くのソリューションを同時に探索することができます。ポリトランスフェクションは、遺伝子回路成分比の多数の組み合わせを単一のウェルで試験することを可能にし、いくつかの公開された遺伝子回路を最適化するために使用されてきた10、11、13。実験プロトコルは、標準的なコトランスフェクションの単純な拡張であり、多くの哺乳類細胞研究者による直接的な採用を可能にします。一般に、ポリトランスフェクションとコトランスフェクションの結果の間には非常に近い一致が見られます10(例は図4です)。したがって、ポリトランスフェクションは、プラスミド比がコトランスフェクションミックス内で滴定され、出力がシングルセルレベルで測定されるほとんどの場合に使用できます。
ポリトランスフェクションの複雑さと規模は、最適化する遺伝子回路の複雑さとともに増加します。分析するディメンションの数が増えると、さまざまなパーツの組み合わせ比と、それらの分析に必要なセルの数が指数関数的に増加します。例えば、 図5の分類器を最適化するために、細胞を6ウェルプレートフォーマットでトランスフェクトし、少なくとも150万個の生細胞についてデータを収集した。また、回路コンポーネント10を追加して分級器を最適化し、10 cmプレートスケールでのトランスフェクションと数百万の細胞の収集を必要としました。その規模以上では、DNAの生産には時間がかかり、トランスフェクション試薬は高価です。とはいえ、ポリトランスフェクションでは、個々のウェルで同等の数の回路成分比の組み合わせをテストするよりも、細胞、DNA、トランスフェクション試薬の使用量が桁違いに少なくなります。さらに、トランスフェクションミックスを桁違いに少なくすることで、時間を大幅に節約できます。
ポリトランスフェクションは、フローサイトメトリーデータの高度な解析法を可能にします。2つの主なアプローチは、(1)各トランスフェクションマーカーの発現に従って細胞をビニングすること、および(2)トランスフェクションマーカーの定義された比率で細胞を抽出し、それによってコトランスフェクションをシミュレートすることです。前者は、各複合体(したがって各部分)のDNA投与量の特定の組み合わせを持つ細胞を選択し、入出力伝達関数を生成します。後者は、各部分のDNA投与量の特定の比率で細胞を選択し、プラスミドDNA質量のそのような比率での共トランスフェクションをシミュレートします。サブサンプリングされた選択に回路出力レポーターの発現レベルをプロットすると、定義されたレベルまたは入力の比率での出力の測定値が得られます。回路の最適化のために、回路の目的によって定義される最高のパフォーマンスのビン/比率を特定することができます-細胞分類器の場合、これらは回路が標的細胞型でオンで、非標的細胞型でオフになっているビン/比です。ビンサイズを選択するときは、必要な精度のレベルとビンあたりのセル数を考慮する必要があります。ビンが小さいほど、回路部品の理想的な組み合わせにより正確に焦点を当てますが、ビンのセルが少なすぎると、測定に望ましくないノイズが発生する可能性があります。
ポリトランスフェクションデータの計算解析における将来の改善は、ビン/比あたりの細胞カバレッジが低い場合でも最適化を容易にする可能性があります。現在、過度にノイズの多い測定値を避けるために、設定されたセルのしきい値(10など)未満のビンからデータを除外しています。繰り返し測定と組み合わせることで、線量反応曲線、分類器の精度、およびビンごとに定義されたその他のメトリックのより正確で正確な計算が可能になります。しかし、ポリトランスフェクションの各細胞は独立した実験であり、正確な入力レベルで回路出力を細かい分解能で測定すると考えることができます。これを念頭に置いて、用量反応および回路最適化の機構的および表現型モデルは、それらのビン化された要約統計量ではなく、各マーカーおよびレポーターからの遺伝子発現の分布に直接適合することができる10。これにより、より堅牢な測定が可能になり、実験ごとに収集された多くの個々のデータポイントを利用できます。このようなモデリングアプローチにより、サンプリングされた高次元ポリトランスフェクションがまばらであっても、予測回路の特性評価と最適化が可能になります。さらに、応答曲面法およびランダムフォレスト回帰などの機械学習手法を用いて、比較的疎で高次元のデータ10を分析してもよい。
ますます大きくなるポリトランスフェクションに対する別のアプローチは、ポリトランスフェクションの階層を使用して回路を順次最適化することです。このアプローチでは、まず、ポリトランスフェクションを使用して、より大きな回路内の遺伝的コンポーネントのサブセットを含むモジュールを最適化します。次に、これらの最適化されたモジュールは、個別のポリトランスフェクションミックスとして提供され、各回路モジュールの最適な比率/投与量を見つけることができます。このアプローチでは、使用する細胞、DNA、トランスフェクション試薬の数が大幅に少なくなります。しかしながら、コンポーネントのグループは、他のコンポーネントに関して必ずしもモジュール化されているとは限らないため、単独で測定されたモジュール内のコンポーネント部品の最適な比率は、より大きな回路コンテキスト内では最適ではない可能性があります8。
ポリトランスフェクション法は、データ収集に使用されるフローサイトメーターのレーザー/フィルター構成によっても制限されます。測定された蛍光出力に加えて、各トランスフェクションミックスには蛍光タンパク質マーカーが含まれています。したがって、サイトメーターで十分に補償できる蛍光タンパク質を選択することが重要です。我々は以前、5レーザーフローサイトメーター(BD LSRFortessa)で22個の蛍光タンパク質のパネルのブリードスルーを系統的に解析し、シリウス、TagBFP、mNeonGreen、mKO2、およびiRFP720のセットを一緒に使用し、大きな問題なしに十分に補正できることを発見しました10。しかし、より大きな回路の最適化には、スペクトルサイトメトリーなどの高度なサイトメトリー法が必要になる可能性があり、スペクトルの重複が多い蛍光タンパク質を分離し、現在、当研究室では最大8つの蛍光タンパク質で最適化しています。蛍光タンパク質データベース(https://www.fpbase.org)などのデータベースは、特定のスペクトルオーバーラップを有する蛍光タンパク質を選択するのに有用である。ただし、このプロセスはさまざまな要因によって複雑になる可能性があり、そのうちのいくつかは特定のサイトメーターに特異的です。たとえば、tdTomatoのような特定の赤色タンパク質を使用すると、特定のレーザー/フィルター構成でのみ青色チャネルへの望ましくないブリードスルーが発生する可能性があります10。さらに、いくつかの遠赤色タンパク質は、DNA投与量と蛍光出力の間に非線形関係を示しており10、DNA投与量の有効なマーカーとしての使用を減らしています。
さまざまな数の複合体(1つ、2つ、3つなど)を使用して実施される実験間で一貫性を保つために、トランスフェクションミックスごとに同じ量のプラスミドをフラクショナル量使用することが有用です。例えば、3つのプラスミド(A、B、C)のいずれかにコードされた各遺伝子を持つ3つの遺伝子系をテストする場合、トランスフェクションマーカーをコードするプラスミドを同じ比率で、回路プラスミドを1:1:1の比率で同時トランスフェクションすることができます。これにより、各プラスミドはミックスの総質量の4分の1になり、形成される各トランスフェクション複合体の質量の約4分の1になります。A、B、Cを別々の複合体にポリトランスフェクトし、独自のレポーターとプラスミドを1:1で混合したり、総DNA質量を維持したりする代わりに、各プラスミドを各複合体の質量の4分の1に保ち、フィラーDNAが残りの質量を占める方が一貫性があります(例については 表5 を参照)。これは、トランスフェクトされた細胞間での遺伝子発現の分布が、複合体中に送達されるDNAの総質量に依存するのではなく、各複合体中のDNAの分数量に依存するためです10。1:1:1以外の比率が必要な場合は、各部分のトランスフェクションミックス中の総DNA質量の対応する画分を計算します。たとえば、 表 5 [下] は 1:1:4 の比率を示しています。
| 方式 | コンプレックス | ng A (分数) | ng B (分数) | ng C (分数) | ngレポーター(分数) | ngフィラーDNA(フラクション) | 合計 (ng) |
| 同時トランスフェクション | 1 | 150 (¼) | 150 (¼) | 150 (¼) | 150 (¼) | 0 (0) | 600 |
| ポリトランスフェクション | 600 | ||||||
| → | 1 | 50 (¼) | 50 (¼) | 100 (½) | 200 | ||
| → | 2 | 50 (¼) | 50 (¼) | 100 (½) | 200 | ||
| → | 3 | 50 (¼) | 50 (¼) | 100 (½) | 200 | ||
| 同時トランスフェクション | 1 | 75 (⅛) | 75 (⅛) | 300 (½) | 150 (¼) | 0 (0) | 600 |
| ポリトランスフェクション | 600 | ||||||
| → | 1 | 25 (⅛) | 50 (¼) | 125 (⅝) | 200 | ||
| → | 2 | 25 (⅛) | 50 (¼) | 125 (⅝) | 200 | ||
| → | 3 | 100 (½) | 50 (¼) | 50 (¼) | 200 |
表5:コトランスフェクション実験およびポリトランスフェクション実験間で一貫性を保つためにフィラーDNAを使用した例。 ここでは、3つのプラスミドを同量のフィラーDNAで同時トランスフェクトする2つの一般的な例が示されています。上の例では、プラスミドはすべて等しい質量です。下の例では、1つのプラスミドが他のプラスミドと比較して高い質量で送達されます。コトランスフェクション複合体で使用される全DNAの一部はポリトランスフェクション複合体にトランスミュートされ、残りの量のDNA(複合体間で固定され、等しい各複合体の総量まで)はフィラーDNAで構成されます。
フィラーDNAの全体的な目的は、固有のトランスフェクションミックスの数に関係なく、細胞あたりのプラスミドの投与量をほぼ同じに維持することです。この原理の大まかな近似として、3つのプラスミドを3つのミックスに分離し、それぞれのDNA質量(およびトランスフェクション試薬の適切な比率)を維持すると、3つのプラスミドすべてを含む単一のミックスと比較して、特定の複合体を投与される細胞の割合が3分の1減少します。しかし、トランスフェクトされた各細胞は、その後、~3倍高い遺伝子投与量を受け取ります。したがって、フィラーDNAを含まないプラスミド単独の相対量を減少させることは、トランスフェクトされた細胞間の発現の根底にある分布を変化させることなくトランスフェクション効率を低下させるだけなので、一貫したプラスミド投与量を維持するには不十分である10。一方、フィラーDNAは、全体的なトランスフェクション効率に影響を与えることなくDNA投与量を調整することができます(ただし、検出可能なトランスフェクション細胞はシグナルの低下により減少する可能性があります)10。上記の大まかな近似に従って、ポリトランスフェクションミックス中のDNAの画分を3分の1に減らし、フィラーDNAを添加すると、元の同時トランスフェクションと比較して相対的な遺伝子投与量が維持されます。したがって、フィラーDNAは効率的で正確なトランスフェクション複合体形成を保証します。
レポーターがカバーする目的の遺伝子の発現範囲を変えるために、そこで遺伝子を駆動するために使用される各試験プラスミドおよび/またはプロモーターの画分をレポーターに対して調整することができる。低DNA投与量で部分が強/高活性である場合、より低いDNA画分を使用すると、トランスフェクトされた細胞におけるレポーターの蛍光分布の中央に部品のダイナミックレンジを集中させるのに役立ちます。同様に、弱い/高DNA投与量でのみ活性な部分の場合、より高い画分を使用すると便利です。しかしながら、DNA画分を減少させすぎると、レポーター10と比較して細胞への送達の確率性が増加し、高い遺伝子発現レベルが細胞の遺伝子発現機構に過負荷をかける可能性があるため、このような調整には注意が必要である12,14。確率性を回避するために、および各遺伝子が一定の比率で細胞に送達される場合(例えば、回路が単一のレンチ、PiggyBac、またはランディングパッドベクターとして生成される場合)、各遺伝子の相対的発現は、より強い/より弱いプロモーター、小さなuORF18、および/またはmiSFIT19を使用して調整することができる。
プロトコルではリバーストランスフェクション技術について説明していますが、フォワードトランスフェクションではポリトランスフェクションも可能です。逆トランスフェクションでは、細胞は同時に播種され、トランスフェクションされます。フォワードトランスフェクションでは、細胞は最初のプレーティング後~24時間で、リバーストランスフェクションで使用される密度の約半分でトランスフェクションされます(トランスフェクション時までに細胞分裂を可能にするため)。一般に、逆トランスフェクションはより効率的ですが、毒性も高く、試薬、トランスフェクション時間、および細胞株に基づくトランスフェクション分布の形状にいくつかの違いがあることに気づきました。したがって、選択するトランスフェクション法は、トランスフェクション細胞の数と細胞あたりのプラスミド投与量の多次元濃縮空間のカバレッジを最大化するために、細胞株ごとに最適化する必要があります。
全体として、ポリトランスフェクションは哺乳類の遺伝子回路の迅速な最適化を可能にします。回路部品の多くの可能なレシオメトリックの組み合わせは、単一のウェルで簡単にテストできます。さらに、ポリトランスフェクションには、従来のトランスフェクションよりもシステム内の各部分のレベルに関するより多くの情報が含まれているため、さまざまな遺伝子部分の挙動を特徴付けるために非常に価値があることがわかっています10、11、12、13。ポリトランスフェクションの採用により、哺乳類細胞で使用するための新しく改良された遺伝子回路の開発ペースが加速すると予想されます。
開示事項
R.W.はStrand TherapeuticsとReplay Bioの共同創設者です。R.W.とR.J.は、細胞型分類器に関連する仮特許を出願しました。
謝辞
ポリトランスフェクション法の開発とその細胞分類器への応用を主導または貢献した元Weiss Labメンバー、Jeremy Gam、Bre DiAndreth、およびJin Huhに感謝します。さらなるメソッド開発/最適化に貢献した他のWeissラボメンバー:Wenlong Xu、Lei Wang、Christian Cuba-Samaniego。ジョシュ・レオナルド教授とパトリック・ドナヒューとヘイリー・エーデルスタインを含むグループメンバー、ポリトランスフェクションのテストとフィードバックの提供。ニカ・シャキバ教授がこの原稿を招待し、フィードバックを提供してくれました。また、国立衛生研究所[R01CA173712、R01CA207029、P50GM098792]にも感謝します。国立科学財団[1745645];NCIからのがんセンター支援(コア)助成金[P30CCA14051、一部]、および国立衛生研究所[P50GM098792]がこの作業に資金を提供してくれました。
資料
| Name | Company | Catalog Number | Comments |
| 15mL Corning Falcon conical tubes | ThermoFisher Scientific | 14-959-53A | |
| 24-well petri dish | Any company of choice | (Non-pyrogenic, Sterile, RNase, DNase, DNA and Pyrogen Free) | |
| Bovine serum albumin | NEB | B9000S | |
| Centrifuge | Any company of choice | Capable of exposing 15mL Falcon tubes to 300 rcf | |
| Countess 3 Automated Cell Counter | ThermoFisher Scientific | AMQAX2000 | |
| Countess Cell Counting Chamber Slides | ThermoFisher Scientific | C10228 | |
| Cytoflow | Non-commercial software package | https://cytoflow.readthedocs.io/en/stable/# | |
| DMEM | VWR | 10-013-CV | Use the correct media for your cell type |
| EDTA | ThermoFisher Scientific | 03690-100ML | |
| Fetal bovine serum | Sigma Aldrich | F4135 | |
| HEK cells | ATCC | CRL-1573 | Use the relevant cell type for your experiments. HEK cells tend to transfect very efficiently. |
| HeLa cells | ATCC | CRL-12401 | Use the relevant cell type for your experiments. |
| Lipofectamine 3000 and P3000 enhancer | ThermoFisher Scientific | L3000001 | Use the correct reagent for your cell type; transfection and enhancer reagent |
| LSRFortessa flow cytometer | BD Biosciences | N/A | |
| MEM Non-Essential Amino Acids Solution | Gibco | 11140050 | |
| Microcentrifuge Tubes, 1.5 mL | Any company of choice | ||
| Opti-MEM | ThermoFisher Scientific | 31985070 | reduced serum medium |
| Phosphate buffered saline | ThermoFisher Scientific | 70011044 | |
| Rainbow calibration beads | Spherotech | URCP-100-2H | |
| Sodium azide | Sigma Aldrich | S2002 | |
| Trypsin | VWR | 25-053-CI |
参考文献
- Prochazka, L., Benenson, Y., Zandstra, P. W. Synthetic gene circuits and cellular decision-making in human pluripotent stem cells. Current Opinion in Systems Biology. 5, 93-103 (2017).
- Sayeg, M. K., et al. Rationally designed microRNA-based genetic classifiers target specific neurons in the brain. ACS Synthetic Biology. 4 (7), 788-795 (2015).
- Zhen, X., Wroblewska, L., Prochazka, L., Weiss, R., Benenson, Y. Multi-Input RNAi-based logic circuit for identification of specific cancer cells. Science. 333 (6047), 1307-1311 (2011).
- Nissim, L., et al. Synthetic RNA-based immunomodulatory gene circuits for cancer immunotherapy. Cell. 171 (5), 1138-1150 (2017).
- Nissim, L., Bar-Ziv, R. H. A tunable dual-promoter integrator for targeting of cancer cells. Molecular Systems Biology. 6, 444(2010).
- Mohammadi, P., Castel, S. E., Brown, A. A., Lappalainen, T. Quantifying the regulatory effect size of cis-acting genetic variation using allelic fold change. Genome Research. 27 (11), 1872-1884 (2017).
- Prochazka, L., et al. Discrete-to-analog signal pluripotent stem cells conversion in human. BioRxiv. , (2021).
- Del Vecchio, D. Modularity, context-dependence, and insulation in engineered biological circuits. Trends in Biotechnology. 33 (2), 111-119 (2015).
- Shakiba, N., Jones, R. D., Weiss, R., Del Vecchio, D. Context-aware synthetic biology by controller design: Engineering the mammalian cell. Cell Systems. 12 (6), 561-592 (2021).
- Gam, J. J., DiAndreth, B., Jones, R. D., Huh, J., Weiss, R. A 'poly-transfection' method for rapid, one-pot characterization and optimization of genetic systems. Nucleic Acids Research. 47 (18), 106(2019).
- Jones, R. D., et al. Robust and tunable signal processing in mammalian cells via engineered covalent modification cycles. Nature Communications. 13 (1), 1720(2022).
- Jones, R. D., et al. An endoribonuclease-based feedforward controller for decoupling resource-limited genetic modules in mammalian cells. Nature Communications. 11 (1), 5690(2020).
- DiAndreth, B., Wauford, N., Hu, E., Palacios, S., Weiss, R. PERSIST platform provides programmable RNA regulation using CRISPR endoRNases. Nature Communications. 13 (1), 2582(2022).
- Frei, T., et al. Characterization and mitigation of gene expression burden in mammalian cells. Nature Communications. 11 (1), 4641(2020).
- Beal, J., Weiss, R., Yaman, F., Adler, A., Davidsohn, N. A method for fast, high-precision characterization of synthetic biology devices. Computer Science and Artificial Intelligence Laboratory Technical Report. Massachusetts institute of technology. , Cambridge. MIT-CSAIL-TR-2012-008 (2012).
- Beal, J., et al. Meeting measurement precision requirements for effective engineering of genetic regulatory networks. ACS Synthetic Biology. 11 (3), 1196-1207 (2022).
- Teague, B. Cytoflow: A Python toolbox for flow cytometry. bioRxiv. , (2022).
- Ferreira, J. P., Overton, K. W., Wang, C. L. Tuning gene expression with synthetic upstream open reading frames). Proceedings of the National Academy of Sciences. 110 (28), 11284-11289 (2013).
- Michaels, Y. S., et al. Precise tuning of gene expression levels in mammalian cells. Nature Communications. 10 (1), 818(2019).
- Saito, H., et al. Synthetic translational regulation by an L7Ae-kink-turn RNP switch. Nature Chemical Biology. 6 (1), 71-78 (2010).
- Wroblewska, L., et al. Mammalian synthetic circuits with RNA binding proteins for RNA-only delivery. Nature Biotechnology. 33 (8), 839-841 (2015).
- Wagner, T. E., et al. Small-molecule-based regulation of RNA-delivered circuits in mammalian cells. Nature Chemical Biology. 14 (11), 1043-1050 (2018).
- Sekuklu, S. D., Donoghue, M. T. A., Spillane, C. miR-21 as a key regulator of oncogenic processes. Biochemical Society Transactions. 37, 918-925 (2009).
- Stanton, B. C., et al. Genomic mining of prokaryotic repressors for orthogonal logic gates. Nature Chemical Biology. 10 (2), 99-105 (2014).
- Stanton, B. C., et al. Systematic transfer of prokaryotic sensors and circuits to mammalian cells. ACS Synthetic Biology. 3 (12), 880-891 (2014).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved