Method Article
インシリコで 宿主-病原体相互作用におけるcircRNAの同定と特性評価
要約
ここで提出されたプロトコルは、宿主と病原体の相互作用を研究するRNAシーケンシングトランスクリプトームデータからcircRNAを予測し、機能的に特徴付けるために必要な完全な インシリコ パイプラインを説明しています。
要約
環状RNA(circRNA)は、バックスプライシング によって 形成されるノンコーディングRNAの一種です。これらのcircRNAは、主に様々な生物学的プロセスの調節因子としての役割について研究されています。特に、病原体(インフルエンザやコロナウイルスなど)に感染すると宿主のcircRNAが差次的に発現(DE)できることが新たな証拠で示されており、宿主の自然免疫応答の調節におけるcircRNAの役割が示唆されています。しかし、病原性感染におけるcircRNAの役割に関する研究は、RNAシーケンシング(RNA-seq)データからDE circRNAを同定するために必要なバイオインフォマティクス解析を実行するために必要な知識とスキルによって制限されています。バイオインフォマティクスの予測とcircRNAの同定は、検証、および費用と時間のかかるウェットラボ技術を使用した機能研究の前に重要です。この問題を解決するために、RNA-seqデータを使用したcircRNAの インシリコ 予測と特性評価の段階的なプロトコルが本稿で提供されています。プロトコルは4つのステップに分けることができます:1)CIRIquantパイプライン を介した DE循環RNAの予測と定量。2)circBase を介した アノテーションとDEサーキットRNAの特性評価。3)Circrパイプラインを介したCircRNA-miRNA相互作用予測。4)遺伝子オントロジー(GO)および京都遺伝子ゲノム百科事典(KEGG)を用いたcircRNA親遺伝子の機能強化解析。このパイプラインは、宿主と病原体の相互作用におけるcircRNAの役割をさらに解明するための将来のin vitro および in vivo 研究を推進するのに役立ちます。
概要
宿主と病原体の相互作用は、病原体と宿主生物の間の複雑な相互作用を表し、宿主の自然免疫応答を引き起こし、最終的には侵入病原体の除去をもたらします1,2。病原性感染の間、多数の宿主免疫遺伝子が病原体の複製および放出を阻害するように調節される。例えば、病原性感染時に調節される一般的なインターフェロン刺激遺伝子(ISG)には、ADAR1、IFIT1、IFIT2、IFIT3、ISG20、RIG-I、およびOASL 3,4が含まれる。タンパク質をコードする遺伝子に加えて、長鎖ノンコーディングRNA(lncRNA)、マイクロRNA(miRNA)、環状RNA(circRNA)などのノンコーディングRNAも役割を果たし、病原性感染中に同時に制御されることも報告されています5,6,7。主にタンパク質を機能分子としてコードするタンパク質コード遺伝子とは対照的に、ノンコーディングRNA(ncRNA)は転写レベルおよび転写後レベルで遺伝子の調節因子として機能することが知られています。しかし、宿主の免疫遺伝子の制御にノンコーディングRNA、特にcircRNAが関与する研究は、タンパク質をコードする遺伝子と比較して十分に報告されていません。
CircRNAは、バックスプライシング8と呼ばれる非標準的なスプライシングプロセスによって生成される共有結合的に閉じた連続ループ構造によって広く特徴付けられます。バックスプライシングのプロセスは、同族の直鎖RNAのスプライシングプロセスとは異なり、下流のドナー部位を上流のアクセプター部位にライゲーションし、円形の構造を形成します。現在、circRNAの生合成のための3つの異なるバックスプライシングメカニズムが提案されている。これらは、RNA結合タンパク質(RBP)を介した環状化9,10、イントロン対駆動環状化11、およびラリアット駆動環状化12,13,14である。circRNAが円形構造でエンドツーエンドで連結されていることを考えると、それらは正常なエキソヌクレアーゼ消化に対して自然に耐性がある傾向があり、したがって、それらの線形対応物よりも安定であると考えられる15。circRNAによって示される別の共通の特徴は、宿主16における細胞または組織タイプ特異的発現を含む。
その独特な構造と細胞または組織特異的な発現が示すように、circRNAは細胞内で重要な生物学的機能を果たすことが発見されています。今日まで、circRNAの顕著な機能の1つは、マイクロRNA(miRNA)スポンジとしての役割です17,18。circRNAのこの調節的役割は、circRNAヌクレオチドとmiRNAのシード領域との相補的結合を介して生じる。このようなcircRNA-miRNA相互作用は、標的mRNA上のmiRNAの正常な調節機能を阻害し、したがって遺伝子の発現を調節する19,20。さらに、circRNAは、RNA結合タンパク質(RBP)と相互作用し、RNAタンパク質複合体を形成することによって遺伝子発現を調節することも知られています21。circRNAはノンコーディングRNAに分類されますが、circRNAがタンパク質翻訳のテンプレートとして作用できるという証拠もあります22,23,24。
最近、circRNAは、特に宿主とウイルスの間の宿主と病原体の相互作用を制御する上で極めて重要な役割を果たすことが実証されています。一般に、宿主のcircRNAは、侵入する病原体を排除するために宿主の免疫応答を調節するのに役立つと考えられています。宿主免疫応答を促進するcircRNAの例は、Guoらによって報告されたcircRNA_0082633である25。このcircRNAは、A549細胞内のI型インターフェロン(IFN)シグナル伝達を増強し、インフルエンザウイルスの複製を抑制するのに役立ちます25。さらに、Quらは、IFN-βのシグナル伝達因子であるCREB結合タンパク質(CREBBP)の発現を調節することによって免疫を促進する、circRNA AIVRと呼ばれるヒトイントロニックcircRNAも報告しました26,27。しかし、感染時に疾患の病態形成を促進することが知られているcircRNAも存在する。例えば、Yuらは最近、宿主細胞オートファジーの阻害を通じてH1N1ウイルス複製を促進する上で、2A遺伝子を含むGATAジンクフィンガードメイン(circGATAD2A)からスプライシングされたcircRNAが果たす役割を報告した28。
circRNAを効果的に研究するために、通常、ゲノムワイドなcircRNA予測アルゴリズムが実装され、その後、機能研究を実施する前に、予測されたcircRNA候補の インシリコ 特性評価が行われます。circRNAを予測および特性評価するためのこのようなバイオインフォマティクスアプローチは、コストが低く、時間効率が高くなります。これは、機能的に研究される候補の数を絞り込むのに役立ち、新しい発見につながる可能性があります。ここでは、宿主と病原体の相互作用中のcircRNAのイン シリコ 同定、特性評価、および機能アノテーションのための詳細なバイオインフォマティクスベースのプロトコルを提供します。このプロトコルには、RNAシーケンシングデータセットからのcircRNAの同定と定量、circBase を介した アノテーション、およびcircRNAの種類、重複する遺伝子の数、および予測されるcircRNA-miRNA相互作用の観点からのcircRNA候補の特性評価が含まれます。この研究はまた、遺伝子オントロジー(GO)および京都遺伝子とゲノムの百科事典(KEGG)エンリッチメント分析を通じて、circRNA親遺伝子の機能アノテーションを提供します。
プロトコル
このプロトコルでは、インフルエンザAウイルスに感染したヒトマクロファージ細胞から調製した匿名化されたリボソームRNA(rRNA)枯渇RNA-seqライブラリデータセットをダウンロードし、遺伝子発現オムニバス(GEO)データベースから使用しました。circRNAの予測から機能特性評価までのバイオインフォマティクスパイプライン全体を 図1にまとめます。パイプラインの各部分については、以下のセクションで詳しく説明します。
1. データ分析前の準備・ダウンロード・セットアップ
注:この調査で使用されたすべてのソフトウェアパッケージは無料でオープンソースです。
- Linux プラットフォームで必要なツールをダウンロードする
- 開発者が提供する手順を使用して、 材料表 に記載されている必要なソフトウェアとツールをLinux高性能コンピューターにダウンロードしてインストールします。
注:ほとんどのツールとソフトウェアには、ツールのインストールと使用の手順を含む独自のオンラインGitHubページまたはドキュメントがあります(資料表を参照)。 - 配列アーカイブのウェブサイト(欧州ヌクレオチドアーカイブや遺伝子発現オムニバスなど)から、circRNAの検出と分析に必要なRNA-seqデータセットをダウンロードしてください。
- RNA-seqデータセットの調製元となった宿主と互換性のあるリファレンスゲノム(FASTA形式)およびアノテーションファイル(GTF/GFF3形式)をダウンロードします。宿主参照ゲノムとアノテーションファイルは通常、国立バイオテクノロジー情報センター(NCBI)、カリフォルニア大学サンタクルーズ校(UCSC)、アンサンブルのウェブサイトなどのオンラインゲノムブラウザにあります。
- 開発者が提供する手順を使用して、 材料表 に記載されている必要なソフトウェアとツールをLinux高性能コンピューターにダウンロードしてインストールします。
- RNA-seqの品質チェック
- FASTQファイルをFASTQCプログラムに入力して、RNA配列の品質を判断します。FASTQファイルの品質が低い場合(29,30などのツールを使用してさらにトリミングする必要があります。
2. CIRIquantを用いたcircRNAの予測と発現差解析
注:差分発現解析のインストールと実行に関するより詳細なマニュアルは、CIRIquant論文31のコードの可用性のセクションにあります。補足データには、このプロトコルで使用される基本的なコマンドの一部も含まれています。
- サークRNA予測
- 最初にBWAおよびHISAT2アライナーを使用して、宿主のリファレンスゲノムにインデックスを作成します。次に、Linux ターミナルで、ホストの参照ゲノムのディレクトリでコマンド bwa index 32 および hisat2-build33 を実行して、インデックスを作成します。
- 次に、ファイルの名前、ツールのパス(BWA、HISAT2、ストリングタイ34、samtools35)、ダウンロードした参照ファイルへのパス(ホストの参照ゲノムFASTAファイル、アノテーションファイル)、および手順2.1.1のインデックスファイルへのパスを含むYML設定ファイルを準備します。
- CIRIquantツールは、デフォルトまたは手動パラメータを使用してターミナルから実行します。ユーザーは、CIRIquantツールの実行時にRNA-seqデータのライブラリタイプ(鎖状または非鎖状)を指定できます。
注:RNA-seqデータのライブラリタイプは、使用するライブラリ調製キットのタイプを知ることで決定できます。ライブラリ調製キットの同定が不明な場合は、RSeQC36 と呼ばれるRNA-seqコントロールバイオインフォマティクスパッケージを使用して、RNA-seqデータの鎖状を決定できます。
- 発現差次解析
注: CIRIquant パッケージには、 prep_CIRIquant、 prepDE.py、および CIRI_DE_replicateが含まれています。したがって、これら 3 つのツールに追加のダウンロードは必要ありません。- 以下を含むデータのリストを含むテキスト ファイル (.lst) を準備します。
1列目 :ステップ2.1.3で使用したRNA-seqデータのID
2番目の 列: CIRIquant によって出力された GTF ファイルへのパス
3列 目:RNA-seqデータのグループ化(対照群か治療群か)。 - 例については、以下の 表 1 を参照してください。
注:ヘッダーは参照用であるため、ヘッダーを入れる必要はありません。 - Linux ターミナルで、手順 2.2.1 で用意したテキスト ファイル (.lst) を入力として prep_CIRIquant を実行します。実行すると、 library_info.csv、 circRNA_info.csv、 circRNA_bsj.csv、 circRNA_ratio.csvのファイルの一覧が生成されます。
- RNA-seq IDとそれぞれのStringTie出力へのパスを含むデータのリストを含む2番目のテキストファイルを準備します。ファイルレイアウトは、グループ化列のない手順2.2.1のテキストファイルと同様である必要があります。
- 手順2.2.4で用意したテキストファイルを入力として prepDE.py を実行し、遺伝子数マトリックスファイルを生成します。
- 手順 2.2.3 の library_info.csv ファイルと circRNA_bsj.csv ファイル、手順 2.2.5 の gene_count_matrix.csv ファイルを入力として CIRI_DE_replicate を実行し、最終的な circRNA_de.tsv ファイルを出力します。
- 以下を含むデータのリストを含むテキスト ファイル (.lst) を準備します。
- DEサークRNAのフィルタリング
- R(コンピュータターミナルまたはRStudio)または任意のスプレッドシートソフトウェア(Microsoft Excelなど)を使用して、手順2.2.6で生成された circRNA_de.tsv ファイルを開き、差次的に発現(DE)されたcircRNAの数をフィルタリングして決定します。
- 基準LogFC > |2| に従って DE circRNA をフィルタリングします。FDR<0.05です。
- DE circRNAの情報を格納する ためにDE_circRNAs.txt という名前のファイルを作成します。
3. 予測されるDE circRNAの特性評価とアノテーション
- DE周回RNAのアノテーション状態
- 手順 2.3.3 でフィルタリングされた DE circRNA のリストで構成される RStudio に DE_circRNAs.txt という名前のファイルを読み込みます。ゲノム位置(Chr、開始、終了)、鎖の向き(+または-)、遺伝子名、circRNAタイプなどの他の情報を含めます。先に進む前に、1塩基対を引くことにより、circRNAゲノム開始座標をCIRIquantから0ベースに変換します。
注:上記のその他の情報は、CIRIquantによって出力されたGTFファイル(補足ファイル1)から取得できます。 - circRNAデータベース(例えば、circBase)寄託されたcircRNAのゲノム位置を含むライブラリをダウンロードして、予測されたDE circRNAのアノテーションステータスを決定します。
注:比較を行う前に、circRNAの予測に使用されるゲノムバージョンがcircRNAデータベースライブラリと同一であることを確認してください。ここで使用するcircBaseデータファイルは、Github(https://github.com/bicciatolab/Circr)37で提供されているドライブフォルダで自由に入手できます。 - 手順 3.1.1 と手順 3.1.2 の両方のファイルが準備できたら、 補足ファイル 1 に記載されている R スクリプトを実行します。DE circRNAの染色体位置は、アノテーション付きまたはアノテーションなしのステータスを割り当てる前にライブラリに照会されます。
- 手順 2.3.3 でフィルタリングされた DE circRNA のリストで構成される RStudio に DE_circRNAs.txt という名前のファイルを読み込みます。ゲノム位置(Chr、開始、終了)、鎖の向き(+または-)、遺伝子名、circRNAタイプなどの他の情報を含めます。先に進む前に、1塩基対を引くことにより、circRNAゲノム開始座標をCIRIquantから0ベースに変換します。
- DEサーキットRNAのキャラクタリゼーション
- Rおよびその他の表計算ソフトウェアを使用して、circRNAの種類(エクソン、イントロン、遺伝子間、アンチセンス)に応じてcircRNAの数と、circRNAがまたがる遺伝子の数(1または>1)を要約します(補足ファイル1)。注:CIRIquantは、4種類のcircRNA(エクソン、イントロン、遺伝子間、アンチセンス)のみを検出できます。ElciRNAとしても知られるエクソンイントロンcircRNAは、CIRIquantでは検出できません。
4. Circrを用いたサークRNA-miRNA相互作用の予測
注:circRNA-miRNA相互作用解析のためのCircrのインストール方法と使用方法に関するより詳細なマニュアルは、https://github.com/bicciatolab/Circr37にあります。
- ファイルの準備
- Circr.zipファイルを「WinRar」や「7-zip」などの関連ソフトウェアを使用してCircr GitHubページからダウンロードした後、分析を行う新しいディレクトリに解凍して抽出します。
- circRNA-miRNA分析を行う前に、前提条件となるソフトウェアアプリケーション(miRanda、RNAhybrid、Pybedtools、およびsamtools)をインストールしてください。
- 関心のあるいくつかの生物の参照ゲノムとアノテーションファイル、rRNA座標ファイル、検証済みのmiRNA相互作用ファイル、およびcircBaseのcircRNAファイルは、Githubページ(https://github.com/bicciatolab/Circr)37でCircrの著者によって提供されています。ドライブフォルダ内のサポートファイルをクリックしたら、目的の生物のフォルダ、miRNAフォルダ、circBaseテキストファイルを選択してダウンロードします。
- 手順4.1.3で必要なファイルをダウンロードした後、手順4.1.1で説明したディレクトリに support_files という名前の新しいディレクトリを作成します。次に、コンテンツを解凍して support_files ディレクトリに抽出します。
- samtools faidx コマンド (補足ファイル 1) を使用して、目的の生物の参照ゲノムファイルにインデックスを付けます。
- 表2に示すように、目的のDE circRNAの座標からなる入力ファイルをタブ区切りのBEDファイルに用意します。
注: CIRIquant によって予測される circRNA は 0 ベースではないため、BED 形式に変換する前に、開始座標で 1 bp をマイナスする必要があります (手順 3.1.1 で説明)。 表 2 に示すヘッダーは参照用であり、BED ファイルには必要ありません。 - この時点で、Circr 分析で予想されるフォルダー・ツリー構造が 図 2 のようになることを確認します。
- ランニング Circr.py
- Python 3 を使用して Circr.py を実行し、引数として、コマンドラインで circRNA 入力ファイル、目的の生物の FASTA ゲノム、選択した生物のゲノム バージョン、スレッド数、および出力ファイルの名前を指定します。
- 目的の生物が手順4.1.3にリストされているドライブフォルダに提供されていない場合、またはユーザーが分析を実行するためのファイルのカスタムセットを持つことを好む場合は、Circr.py を実行するときにこれらのファイルの場所を指定する追加のコマンドを含める必要があります。
- Circr解析が完了すると、プログラムはcsv形式でcircRNA-miRNA相互作用ファイルを出力します。
- ユーザー固有の好みに応じて、circRNA-miRNA相互作用結果をフィルタリングします。この調査では、以下の基準に従ってRstudioを使用して予測をフィルタリングします。
-3つのソフトウェアツールすべてで検出
-ターゲットスキャンとミランダの両方によって報告された2つ以上の結合部位
-"AGO" 列または "検証済み" 列のいずれかで識別されます。
-シード領域の相互作用を除外しない - 手順 4.2.3 でフィルタリングされた条件を渡す circRNA を circRNA_miRNA.txt という名前の新しいテキスト ファイルに書き込み ます。このようなフィルタリングにより、予測された交互作用の信頼度を高めることができます。
5. ceRNAネットワークの構築
注:Cytoscapeの使用方法に関する詳細なマニュアルは、次の場所にあります:http://manual.cytoscape.org/en/stable/ および https://github.com/cytoscape/cytoscape-tutorials/wiki#introduction
- ダウンロードと準備
- Cytoscape38 の最新バージョンを以下からダウンロードしてください: https://cytoscape.org/download.html.
- 手順5.1.1でダウンロードしたインストーラウィザードを実行し、Cytoscapeソフトウェアのファイルの場所を選択します。
- 目的のcircRNAとその標的miRNAを含むタブ区切りファイルを準備します。最初の列は circRNA 名で構成されています。2番目の列は、最初の列のRNAの種類を指定します。3番目の列は標的miRNAです。4番目の列は3番目の列のRNAの種類を指定します。ファイルの例を 表 3 に示します。
- ceRNAネットワークマップの構築
- 手順5.1.2でインストールしたCytoscapeソフトウェアを開きます。
- Cytoscapeで、[ ファイルからネットワーク>インポート]に移動し>。手順 5.1.3 で準備したファイルを選択します。
- 新しいタブで、1番目と2番目の列を「ソースノード」と「ソースノード属性」として選択し、3番目と4番目の列をそれぞれ「ターゲットノード」と「ターゲットノード属性」として選択します。 OKをクリックすると、ネットワークがCytoscapeの右上に表示されます。
- ネットワークの表示スタイルを変更するには、Cytoscapeの左側にある スタイル ボタンを押します。
- [塗りつぶしの色] の右側にある矢印を押します。列には [タイプ] を選択し、マッピング タイプには [離散マッピング] を選択します。次に、各RNAタイプに必要な色を選択します。
- 色を変更した後、[ 形状 ] に移動し、手順 5.2.5 に従ってノードの形状を変更します。
6. 機能エンリッチメント解析
- 遺伝子オントロジー(GO)および京都遺伝子ゲノム百科事典(KEGG)によるcircRNAの親遺伝子の解析
- クラスタプロファイラ39、40 および組織を確認します。Hs.eg.db41 個のパッケージが Rstudio にインストールされました。組織。Hs.eg.db41パッケージは、ヒト専用のゲノムワイドアノテーションパッケージです。 関心のある生物が別の種である場合は、以下を参照してください。 https://bioconductor.org/packages/release/BiocViews.html#OrgDb
- 手順 2.3.1 のDE_circRNA情報を Rstudio ワークスペースにインポートします。
- このファイルで提供されているcircRNAの親遺伝子を使用して、以降のステップで濃縮解析を行います。ただし、ユーザーが遺伝子シンボルをEntrez IDなどの他の形式に変換したい場合は、「bitr」などの関数を使用します。
- 遺伝子 ID を入力として使用して、デフォルトのパラメーターを使用して、clusterProfiler 39,40 パッケージ内の enrichGO 関数を使用して GO エンリッチメント分析を実行します。
- 遺伝子 ID を入力として使用して、デフォルトのパラメーターを使用して、clusterProfiler39,40 パッケージ内の enrichKEGG 関数を使用して KEGG エンリッチメント分析を実行します。
結果
前のセクションで参加したプロトコルは、Linux OS システムに合わせて変更および構成されました。主な理由は、circRNAの分析に関与するほとんどのモジュールライブラリとパッケージは、Linuxプラットフォームでしか動作しないことです。この分析では、インフルエンザAウイルスに感染したヒトマクロファージ細胞から調製された非同定リボソームRNA(rRNA)枯渇RNA-seqライブラリデータセットをGEOデータベース42 からダウンロードし、代表的な結果を生成するために使用した。
CircRNAの予測と定量
この解析では、インフルエンザAウイルスに感染したヒトマクロファージ細胞から調製したリボソームRNA(rRNA)枯渇RNA-seqライブラリデータセットを使用して、circRNA検出と機能解析を実施しました。プロトコルセクションで指定されているように、CIRIquantを使用して、RNA-seqライブラリデータセットを入力として使用して、同定されたcircRNAのDE分析を実行しました。使用される参照ファイルは、最新のヒトゲノムバージョン(hg38)に基づいています。 表 4 に、CIRIquant 分析の最終出力の例を示します。CIRIquant出力からのDE周回RNAの同定とフィルタリングは、単純なRStudioスクリプト(補足ファイル1)を介して実行されました。CircRNAは、偽発見率(FDR)値が<0.05および対数フォールド変化(LogFC)>|2|の場合にのみDEとして分類されます。 表5 は、検出されたcircRNAおよびDEのcircRNAの総数を示す。合計35,846個のcircRNAが検出され、そのうち306個がDEでした。この出力で検出されたDE circRNAは完全にアップレギュレーションされ(LogFC > 2)、ダウンレギュレーションされるものはありません(LogFC < 2)。
DE circRNAのアノテーションとキャラクタリゼーション
DE周回RNAのアノテーション状態
同定されたDEのcircRNAは、確立されたcircRNAデータベースcircBaseとクロスチェックされました。ただし、circBaseに寄託されたcircRNA座標は以前のヒトゲノムバージョン(hg19)に基づいているため、この研究でクロスチェックするには、circBaseからのcircRNA座標を現在のヒトゲノムバージョン(hg38)に変換する必要があります。さらに、開始座標は、CIRIquant の 1 から始まる出力から 0 から始まるものに変換する必要があります。hg38バージョン変換されたcircRNA座標は、Github(https://github.com/bicciatolab/Circr)37のドライブフォルダで提供されています。次に、Rstudioスクリプト(補足ファイル1)を使用して、新しいデータフレーム列にcircRNAの注釈ステータスを割り当てました。 表6 に、アノテーションステータスのcircRNAの例を示す。
DEサーキットRNAのキャラクタリゼーション
この部分は、RStudio ソフトウェアの R スクリプトを使用して完全に実行されました。R スクリプトは分析プロセスを容易にし、基本的な知識のみが必要です。
サークRNAの種類
このステップでは、DE circRNAは、ゲノム位置に基づいて、それらのcircRNAタイプ(アンチセンス、エキソニック、遺伝子間、およびイントロニック)によって特徴付けられました。以下の 表7 は、同定されたDE circRNAに含まれる異なるcircRNAタイプのパーセンテージ内訳を示しています。合計306個のDE circRNAのうち、263個のcircRNA(85.95%)がエキソン型であるが同定された。イントロニックcircRNAは、17個のDEサーキットRNAで構成される2番目に同定されたcircRNAタイプとして登場し、DEサーキットRNA全体の最大5.56%を占めています。これに続いて、遺伝子間ractRNA(16個のDE回回RNA~5.23%)とアンチセンス系circRNA(10個のDE回回RNA~3.27%)が続きます。
circRNAあたりの遺伝子数
CIRIquantによって同定されたCircRNAは、多くの遺伝子にわたって重複する可能性があります。今日まで、ほとんどの研究は1つの遺伝子にまたがるcircRNAに焦点を当てています。したがって、このプロトコルでは、複数の遺伝子にまたがるcircRNA候補はダウンストリーム分析から除外されます。以下の 表8 は、1つ以上の遺伝子にまたがるDE circRNAの数および割合を記載する。この表では、遺伝子間racRNA(16 DE circRNA)は宿主遺伝子と重複しないため除外されていますが、残りのcircRNAタイプ(290 DE circRNA)はこの分析にかけられています。290個のDE circRNAのうち、DE circRNAの大部分(261個のcircRNA~90%)は1つの遺伝子のみにまたがっていますが、残りの29個のcircRNA(~10%)は複数の遺伝子にまたがっています。
ceRNAネットワークの構築
ceRNAネットワークは通常、予測された後にcircRNA-miRNA相互作用を視覚化するために描画されます。下の 図3 では、代表的な結果として1つのDE circRNAのみが選択された、これがhsa_DE_58 circRNAである。Circrの予測に基づいて、hsa_DE_58は最大9つの異なるmiRNAをスポンジ化することができます。これらの9つのmiRNAは、厳しい基準でフィルタリングした後に同定されます。
機能エンリッチメント解析
サークRNA親遺伝子のGOおよびKEGG解析
下の図4は、GO解析によるDE circRNA親遺伝子の機能的濃縮のバブルプロットを示す。基本的に、GO分析は、研究された条件(この場合はウイルスに感染したサンプル)で濃縮または影響を受ける生物学的プロセス、細胞位置、および分子機能を解明することを目的としています。情報付加は統計的に有意であると見なされ、p値が0.01<場合にのみバブルプロットにプロットされます。図4に示すように、生物学的プロセス(BP)の上位3つの濃縮には、リボ核タンパク質複合体の生合成、ウイルスへの応答、および生物刺激に対する応答の制御が含まれますが、分子機能(MF)では、RNAに作用する触媒活性と一本鎖RNA結合のみが統計的に濃縮されています。一方、レトロマー複合体のみが細胞成分(CC)について統計的に濃縮されている。
図5 は、DE circRNA親遺伝子のKEGG濃縮分析をバブルプロットで示す。GOエンリッチメント分析と同様に、KEGGエンリッチメントは統計的に有意であると見なされ、p値が0.01<場合にのみバブルプロットにプロットされます。この場合、KEGGの用語が強化されたのは、インフルエンザA経路とウイルスライフサイクル(HIV-1)経路の2つだけです。
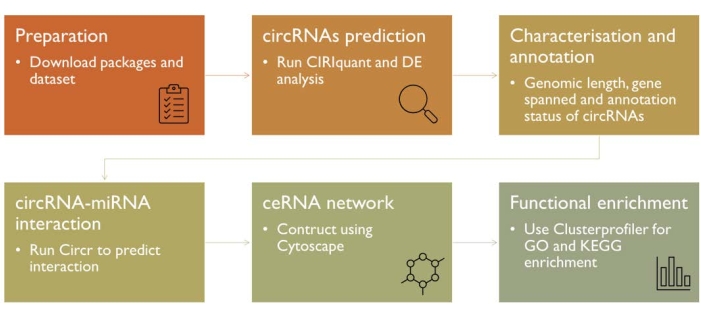
図1:circRNAの予測と機能特性評価のためのパイプライン。 パイプラインは、必要なソフトウェアパッケージのインストール、circRNA発現の予測と定量化、ceRNAネットワークの構築、circRNA親遺伝子機能強化の実行など、最初から最後までの主要なステップの簡単な概要を示しています。 この図の拡大版を表示するには、ここをクリックしてください。

図2:Circrのフォルダツリー構造。 このフォルダツリー構造は、分析に必要なファイルを検出するために、Circrソフトウェアを実行する前に確立する必要があります。 この図の拡大版を表示するには、ここをクリックしてください。

図3:circRNA-miRNA相互作用からなるceRNAネットワーク。 青い楕円形はcircRNAを表し、オレンジ色の三角形はmiRNAを表します。circRNAとmiRNAをつなぐ実線は、hsa_DE_58 circRNAの潜在的なmiRNAスポンジ機能を示しています。 この図の拡大版を表示するには、ここをクリックしてください。
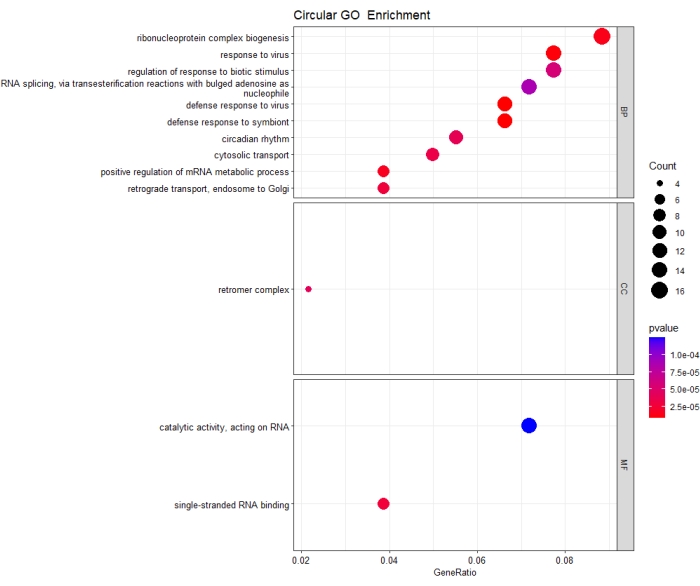
図4:DE circRNA親遺伝子のGO濃縮分析のバブルプロット。 x軸のGeneRatioは、与えられたGO項に関連付けられた入力リスト内の遺伝子の数を、入力遺伝子の総数で割ったものです。プロットのドットサイズは、指定されたGO項に関連付けられた入力リスト内の遺伝子の数であるカウント値で表されます。ドットのサイズが大きいほど、用語に関連する入力遺伝子の数が多くなります。さらに、プロット内のドットはp値に基づいて色分けされています。p値は、アノテーション項の観測頻度と偶然に予想される頻度を比較することによって計算されます。個々の項は、カットオフ値(p値<0.01)を超えてエンリッチされていると見なされます。青から赤の範囲のp値の色のグラデーションは、項のエンリッチメントが増加していることを示します。 この図の拡大版を表示するには、ここをクリックしてください。

図5:DE circRNA親遺伝子のKEGG濃縮解析。 x軸のGeneRatioは、指定されたKEGG項に関連付けられた入力リスト内の遺伝子の数を、入力遺伝子の総数で割ったものです。プロットのドットサイズは、指定されたKEGG項に関連付けられた入力リスト内の遺伝子の数であるカウント値で表されます。ドットのサイズが大きいほど、用語に関連する入力遺伝子の数が多くなります。さらに、プロット内のドットはp値に基づいて色分けされています。p値は、アノテーション項の観測頻度と偶然に予想される頻度を比較することによって計算されます。個々の項は、カットオフ値(p値<0.01)を超えてエンリッチされていると見なされます。青から赤の範囲のp値の色のグラデーションは、項のエンリッチメントの増加を示します。 この図の拡大版を表示するには、ここをクリックしてください。
| サンプル名 | CIRIquant 出力 GTF ファイルへのパス | グルーピング |
| コントロール 1 | /path/to/CIRIquant/ctrl1.gtf | C |
| コントロール 2 | /path/to/CIRIquant/ctrl2.gtf | C |
| 感染者 1 | /path/to/CIRIquant/infect1.gtf | T |
| 感染者2 | /path/to/CIRIquant/infect2.gtf | T |
表 1: CIRIquant の .lst ファイルの準備。 CIRIquant出力からのコントロールサンプルと処理サンプルの宛先パスは、2種類のサンプル間のcircRNAの発現を比較するためにテキストファイルに書き込まれます。
| ティッカー | 始める | 終わり | 名前 | . | ストランド |
| CHR2 | 137428930 | 137433876 | hsa_circ_000076 | . | - |
| CHR2 | 154705868 | 154706632 | hsa_circ_000105 | . | - |
| CHR2 | 159104273 | 159106793 | hsa_circ_000118 | . | - |
| CHR2 | 159215701 | 159226125 | hsa_circ_000119 | . | - |
| CHR4 | 39980067 | 39980129 | hsa_circ_002584 | . | - |
表 2: Circr の BED ファイルの例を示します。 BED ファイルを生成するには、circRNA に関連付けられた 6 つのカラム (Chr、開始、終了、名前、遺伝子、および鎖) が必要です。
| circRNA_name | 種類 | miRNA_name | 種類 |
| DE_circRNA_1 | サーキットRNA | miR-001 | マイクロナ |
| DE_circRNA_1 | サーキットRNA | miR-002 | マイクロナ |
| DE_circRNA_2 | サーキットRNA | miR-003 | マイクロナ |
| DE_circRNA_2 | サーキットRNA | miR-004 | マイクロナ |
表3:サイトスケープ入力ファイル。 テキスト ファイルに書き込むには、4 つの列 (circRNA_name、型、miRNA_name、および型) が必要です。
| サークRNA細胞 | ログFC | ログインプレッション | ロイズ船級協会 | p値 | デ | ティッカー |
| CHR4:17595410|17598558 | 8.167934481 | -0.039318634 | 185.5341965 | 3.00E-42 | 1 | 1.08E-37 |
| CHR16:18834892|18850467 | -3.955083482 | -4.397235736 | 2.982607619 | 0.08416358 | 0 | 0.282478158 |
| CHR14:73198031|73211942 | 2.493964729 | -4.448176684 | 2.736442046 | 0.09808293 | 0 | 0.282478158 |
表 4: CIRIquant の最終出力 (.csv) ファイルの一部。 CIRIquantは、LogFC、100万あたりのログ数(LogCPM)、ロジスティック回帰(LR)、p値、微分式、FDRなどの情報を提供します。
| CIRIquant の結果 | |||
| トータル | デ | 上 | 下 |
| 35846 | 306 | 306 | 0 |
表5:同定された総および差次的発現(DE)circRNAの数の要約。 合計35,846個のcircRNAが検出され、そのうち306個がDEのcircRNAです。306 DE circRNAはすべて、対照サンプルと比較した場合、処理されたサンプルでアップレギュレーションされています(ダウンレギュレーションされているものはありません)。
| Custom_Name | Annotation_Status |
| hsa_DE_22 | 注釈なし |
| hsa_DE_2 | 注釈 付き |
| hsa_DE_58 | 注釈なし |
| hsa_DE_3 | 注釈 付き |
表6:アノテーションステータス付きのカスタムcircRNA名の表。 CircRNAは、既知の寄託されたcircRNA(circBase)のデータベースで照会されます。circRNAがデータベース内に存在する場合、アノテーションされるようにラベル付けされ、circRNAが存在しない場合はアノテーションなしとしてラベル付けされます。
| サークRNAタイプ | 周波数 | 百分率 |
| アンチセンス | 10 | 3.27% |
| エクソン | 263 | 85.95% |
| インタージェニック | 16 | 5.23% |
| イントロン | 17 | 5.56% |
表7:同定されたcircRNAの種類。 CircRNAはさらに、その配列領域、すなわちエキソニック、イントロニック、アンチセンス、および遺伝子間性に基づいて、異なるタイプのcircRNAに分類することができる。
| 親遺伝子の数 | 周波数 | 百分率 |
| 1 | 261 | 90% |
| > 1 | 29 | 10% |
表8:異なる数の遺伝子を持つcircRNAの割合。 CircRNAは一般に1つの遺伝子のエクソンからコードされていますが、複数の遺伝子にまたがるcircRNAもCIRIquantによって検出できます。
補足ファイル 1: プロトコルで使用されるスクリプト。このファイルをダウンロードするには、ここをクリックしてください。
ディスカッション
このプロトコルの有用性を説明するために、インフルエンザAウイルス感染ヒトマクロファージ細胞由来のRNA−seqを例として使用した。宿主と病原体の相互作用において潜在的なmiRNAスポンジとして機能するCircRNAと、宿主内でのGOおよびKEGGの機能強化を調べました。オンラインで入手できるさまざまなcircRNAツールがありますが、それぞれが互いに相互作用しないスタンドアロンパッケージです。ここでは、circRNAの予測と定量、circRNAの機能強化、circRNA-miRNA相互作用の予測、およびceRNAネットワークの構築に必要なツールをいくつかまとめました。この合理化されたプロトコルは時間を節約し、臨床サンプルに適用して、診断値と予後値を持つcircRNA候補を検出することができます。
基本的には、CIRI2をあらかじめパッケージ化したcircRNA定量ツールであるCIRIquant31を採用し、circRNAの検出とDE解析を実施しました。DE circRNA は、LogFC > |2| のカットオフ値に基づいてフィルタリングされます。FDR < 0.05は、ダウンストリーム分析での潜在的な誤検知を排除するのに役立ちます。アノテーションの状態、circRNAの種類、および遺伝子の数に関するDE circRNAの特性評価は、circRNA候補の分類とさらなるフィルタリングに役立ちます。続いて、circRNA-miRNA予測ツールであるCircr37を使用して、潜在的なmiRNAスポンジ候補を予測します。circRNAの標的となる可能性のあるmiRNAを予測した後、ceRNAネットワークを描画します。最後に、circRNAの親遺伝子に基づいて、Rクラスタプロファイラパッケージ39 は、GOおよびKEGG経路エンリッチメント解析 を介した 機能アノテーションに使用される。GOとKEGGの結果は、circRNAの影響を受ける生物学的メカニズムの解明に役立つ可能性があります。
現在までに、CIRI243、CIRCexplorer2 44、find_circ 45、MapSplice46、UROBORUS 47など、いくつかの異なるcircRNA予測ツールが開発されています。Hansenらが実施した研究では、CIRI2は全体的な性能が高いと報告されています。これは、de novo予測と偽陽性識別の減少の点でうまく機能できる数少ないcircRNA検出ツールの1つです48。したがって、この研究では、circRNAの検出と定量にCIRI2を利用するCIRIquantを使用しました。CIRIquantを使用してバックスプライスジャンクション(BSJ)リードをカウントし、カウントデータを同じ遺伝子座から転写された同族の線形RNAにマッピングされたリードに正規化しました。これにより、サンプル中のcircRNAの定量が可能になります。実験条件全体でのcircRNAの発現差を決定するために、CIRIquantはDE分析のためにedgeR49に一般化線形モデルを実装し、正確な速度比検定を統計的検定として使用して、circRNA接合比比比の差の有意性を決定しました。CIRCexplorer3-CLEAR50などの他のcircRNA定量ツールを使用してcircRNAの発現量を定量することもできますが、このツールは、サンプル中のBSJリードをカウントし、カウントデータを同じサンプルからの同族の直鎖RNAカウントに対して正規化するため、サンプル中のcircRNA定量のみが可能です。サークエクスプローラー3-CLEARは、実験条件間でサーキットRNA発現を比較することはできません。さらに、CIRCexplorer3-CLEARには、定量化された発現レベルをサポートするための統計解析ツールは実装されていません。CIRIquant内に実装されるデフォルトのcircRNA予測ツールはCIRI2であるが、find_circおよびCIRCexplorer2などの他のツールからの予測結果も、定量化およびDE分析31に利用することができる。このプロトコルでは、予測に使用されたcircRNA予測ツール(CIRI2)は1つだけであり、それでも偽陽性のcircRNA候補が得られる可能性があります。偽陽性を減少させるために、分析のために他のcircRNA予測ツールを組み合わせ、異なるcircRNA予測ツール48,51の中から検出された共通のcircRNAを選択することができる。circRNA検出をさらに改善するには、rRNAが枯渇し、RNase Rの前処理を受けたRNAシーケンシングデータセットを使用することが理想的です。
研究の目的に応じて、de novoおよび注釈付きDE circRNAは、circBaseデータベース52に基づいて別々に同定することができる。ただし、複数の遺伝子にまたがるcircRNAは、circRNAの信憑性を判断し、偽陽性を排除するために、UCSCまたはその他のゲノムブラウザーで手動で検査する必要があることがよくあります。それにもかかわらず、融合遺伝子に由来するcircRNAなど、複数の遺伝子にまたがるcircRNAも最近報告されています53,54。
Circrは、3つの異なるmiRNA-mRNA予測アルゴリズム、すなわちターゲットスキャン55、miRanda 56、およびRNAハイブリッド57を組み合わせて、circRNA-miRNA結合部位を予測することによって機能します。さらに、このアルゴリズムには、circRNA-miRNA分析におけるAGOピークと以前に検証された相互作用の情報も組み込まれています。ここでは、より信頼性の高いcircRNA-miRNA予測が得られるように厳しいフィルタリング基準が適用され、偽陽性がさらに減少しました。ただし、このフィルタリングステップの厳密性は、ユーザーの好みに応じて高くまたは低く設定できます。
ClusterProfiler は、さまざまな生物の遺伝子セットに機能的に注釈を付けることができる、十分に文書化された R パッケージです。このプロトコルで言及されている R clusterProfiler パッケージ内の関数 (enrichGO および enrichKEGG ) の他に、過剰表現分析を利用する関数もあります。 clusterProfilerがワークフローに適していない場合は、"AllEnricher"58や"Metascape"59などのWebサイトベースのツールなど、遺伝子のセットに機能的に注釈を付けることができる他のツールやパッケージもあります。最後に、上記のパイプラインは潜在的なcircRNAとその機能アノテーションを予測するのに役立ちますが、確かな証拠を提供するにはウェットラボでの検証が必要になります。
開示事項
著者は開示するものは何もありません。
謝辞
著者は、この原稿の批判的なレビューについて、Tan KeEnとCameron Bracken博士に感謝したいと思います。この研究は、基礎研究助成スキーム(FRGS/1/2020/SKK0/UM/02/15)およびマラヤ大学ハイインパクト研究助成金(UM)からの助成金によってサポートされました。C/625/1/HIR/MOE/CHAN/02/07)。
資料
| Name | Company | Catalog Number | Comments |
| Bedtools | GitHub | https://github.com/arq5x/bedtools2/ | Referring to section 4.1.2. Needed for Circr. |
| BWA | Burrows-Wheeler Aligner | http://bio-bwa.sourceforge.net/ | Referring to section 2.1.1 and 2.1.2. Needed to run CIRIquant, and to index the genome |
| Circr | GitHub | https://github.com/bicciatolab/Circr | Referring to section 4. Use to predict the miRNA binding sites |
| CIRIquant | GitHub | https://github.com/bioinfo-biols/CIRIquant | Referring to section 2.1.3. To predict circRNAs |
| Clusterprofiler | GitHub | https://github.com/YuLab-SMU/clusterProfiler | Referring to section 7. For GO and KEGG functional enrichment |
| CPU | Intel | Intel(R) Xeon(R) CPU E5-2620 V2 @ 2.10 GHz Cores: 6-core CPU Memory: 65 GB Graphics card: NVIDIA GK107GL (QUADRO K2000) | Specifications used to run this entire protocol. |
| Cytoscape | Cytoscape | https://cytoscape.org/download.html | Referring to section 5.2. Needed to plot ceRNA network |
| FastQC | Babraham Bioinformatics | https://www.bioinformatics.babraham.ac.uk/projects/fastqc/ | Referring to section 1.2.1. Quality checking on Fastq files |
| HISAT2 | http://daehwankimlab.github.io/hisat2/ | Referring to section 2.1.1 and 2.1.2. Needed to run CIRIquant, and to index the genome | |
| Linux | Ubuntu 20.04.5 LTS (Focal Fossa) | https://releases.ubuntu.com/focal/ | Needed to run the entire protocol. Other Ubuntu versions may still be valid to carry out the protocol. |
| miRanda | http://www.microrna.org/microrna/getDownloads.do | Referring to section 4.1.2. Needed for Circr | |
| Pybedtools | pybedtools 0.8.2 | https://pypi.org/project/pybedtools/ | Needed for BED file genomic manipulation |
| Python | Python 2.7 and 3.6 or abover | https://www.python.org/downloads/ | To run necessary library modules |
| R | The Comprehensive R Archive Network | https://cran.r-project.org/ | To manipulate dataframes |
| RNAhybrid | BiBiServ | https://bibiserv.cebitec.uni-bielefeld.de/rnahybrid | Referring to section 4.1.2. Needed for Circr |
| RStudio | RStudio | https://www.rstudio.com/ | A workspace to run R |
| samtools | SAMtools | http://www.htslib.org/ | Referring to section 2.1.2. Needed to run CIRIquant |
| StringTie | Johns Hopkins University: Center for Computational Biology | http://ccb.jhu.edu/software/stringtie/index.shtml | Referring to section 2.1.2. Needed to run CIRIquant |
| TargetScan | GitHub | https://github.com/nsoranzo/targetscan | Referring to section 4.1.2. Needed for Circr |
参考文献
- Raman, K., Bhat, A. G., Chandra, N. A systems perspective of host-pathogen interactions: predicting disease outcome in tuberculosis. Molecular BioSystems. 6 (3), 516-530 (2010).
- Casadevall, A., Pirofski, L. A. Host-pathogen interactions: basic concepts of microbial commensalism, colonization, infection, and disease. Infection and Immunity. 68 (12), 6511-6518 (2000).
- Yang, E., Li, M. M. H. All About the RNA: Interferon-stimulated genes that interfere with viral RNA processes. Frontiers in Immunology. 11, 605024 (2020).
- Schneider, W. M., Chevillotte, M. D., Rice, C. M. Interferon-stimulated genes: A complex web of host defenses. Annual Review of Immunology. 32 (1), 513-545 (2014).
- Shirahama, S., Miki, A., Kaburaki, T., Akimitsu, N. Long non-coding RNAs involved in pathogenic infection. Frontiers in Genetics. 11, 454 (2020).
- Chandan, K., Gupta, M., Sarwat, M. Role of host and pathogen-derived microRNAs in immune regulation during infectious and inflammatory diseases. Frontiers in Immunology. 10, 3081 (2019).
- Chen, X., et al. Circular RNAs in immune responses and immune diseases. Theranostics. 9 (2), 588-607 (2019).
- Kristensen, L. S., et al. The biogenesis, biology and characterization of circular RNAs. Nature Reviews Genetics. 20 (11), 675-691 (2019).
- Ashwal-Fluss, R., et al. circRNA biogenesis competes with pre-mRNA splicing. Molecular Cell. 56 (1), 55-66 (2014).
- Conn, S. J., et al. The RNA binding protein quaking regulates formation of circRNAs. Cell. 160 (6), 1125-1134 (2015).
- Zhang, X. O., et al. Complementary sequence-mediated exon circularization. Cell. 159 (1), 134-147 (2014).
- Robic, A., Demars, J., Kuhn, C. In-depth analysis reveals production of circular RNAs from non-coding sequences. Cells. 9 (8), 1806 (2020).
- Eger, N., Schoppe, L., Schuster, S., Laufs, U., Boeckel, J. N. Circular RNA splicing. Advances in Experimental Medicine and Biology. 1087, 41-52 (2018).
- Barrett, S. P., Wang, P. L., Salzman, J. Circular RNA biogenesis can proceed through an exon-containing lariat precursor. eLife. 4, 07540 (2015).
- Memczak, S., et al. Circular RNAs are a large class of animal RNAs with regulatory potency. Nature. 495 (7441), 333-338 (2013).
- Misir, S., Wu, N., Yang, B. B. Specific expression and functions of circular RNAs. Cell Death and Differentiation. 29 (3), 481-491 (2022).
- Bai, S., et al. Construct a circRNA/miRNA/mRNA regulatory network to explore potential pathogenesis and therapy options of clear cell renal cell carcinoma. Scientific Reports. 10 (1), 13659 (2020).
- Sakshi, S., Jayasuriya, R., Ganesan, K., Xu, B., Ramkumar, K. M. Role of circRNA-miRNA-mRNA interaction network in diabetes and its associated complications. Molecular Therapy - Nucleic Acids. 26, 1291-1302 (2021).
- Hansen, T. B., et al. miRNA-dependent gene silencing involving Ago2-mediated cleavage of a circular antisense RNA. The EMBO Journal. 30 (21), 4414-4422 (2011).
- Lu, M. Circular RNA: functions, applications, and prospects. ExRNA. 2 (1), 15 (2020).
- Liu, K. S., Pan, F., Mao, X. D., Liu, C., Chen, Y. J. Biological functions of circular RNAs and their roles in occurrence of reproduction and gynecological diseases. American Journal of Translational Research. 11 (1), 1-15 (2019).
- Pamudurti, N. R., et al. Translation of CircRNAs. Molecular Cell. 66 (1), 9-21 (2017).
- Legnini, I., et al. Circ-ZNF609 Is a circular RNA that can be translated and functions in myogenesis. Molecular Cell. 66 (1), 22-37 (2017).
- Weigelt, C. M., et al. An insulin-sensitive circular RNA that regulates lifespan in Drosophila. Molecular Cell. 79 (2), 268-279 (2020).
- Guo, Y., et al. Identification and characterization of circular RNAs in the A549 cells following Influenza A virus infection. Veterinary Microbiology. 267, 109390 (2022).
- Qu, Z., et al. A novel intronic circular RNA antagonizes influenza virus by absorbing a microRNA that degrades CREBBP and accelerating IFN-β production. mBio. 12 (4), 0101721 (2021).
- Kawarada, Y., et al. TGF-β induces p53/Smads complex formation in the PAI-1 promoter to activate transcription. Scientific Reports. 6 (1), 35483 (2016).
- Yu, T., et al. Circular RNA GATAD2A promotes H1N1 replication through inhibiting autophagy. Veterinary Microbiology. 231, 238-245 (2019).
- FastQC: A quality control tool for high throughput sequence data. Available from: https://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (2010)
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Zhang, J., Chen, S., Yang, J., Zhao, F. Accurate quantification of circular RNAs identifies extensive circular isoform switching events. Nature Communications. 11 (1), 90 (2020).
- Li, H., Durbin, R. Fast and accurate long-read alignment with Burrows-Wheeler transform. Bioinformatics. 26 (5), 589-595 (2010).
- Kim, D., Paggi, J. M., Park, C., Bennett, C., Salzberg, S. L. Graph-based genome alignment and genotyping with HISAT2 and HISAT-genotype. Nature Biotechnology. 37 (8), 907-915 (2019).
- Pertea, M., et al. StringTie enables improved reconstruction of a transcriptome from RNA-seq reads. Nature Biotechnology. 33 (3), 290-295 (2015).
- Li, H., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), 2078-2079 (2009).
- Wang, L., Wang, S., Li, W. RSeQC: quality control of RNA-seq experiments. Bioinformatics. 28 (16), 2184-2185 (2012).
- Dori, M., Caroli, J., Forcato, M. Circr, a computational tool to identify miRNA:circRNA associations. Frontiers in Bioinformatics. 2, 852834 (2022).
- Shannon, P., et al. Cytoscape: a software environment for integrated models of biomolecular interaction networks. Genome Research. 13 (11), 2498-2504 (2003).
- Wu, T., et al. clusterProfiler 4.0: A universal enrichment tool for interpreting omics data. The Innovation. 2 (3), 100141 (2021).
- Yu, G., Wang, L. G., Han, Y., He, Q. Y. clusterProfiler: an R package for comparing biological themes among gene clusters. OMICS: A Journal of Integrative Biology. 16 (5), 284-287 (2012).
- . org.Hs.eg.db: Genome wide annotation for human. 2022. R package version 3.15.0 Available from: https://bioconductor.org/packages/release/data/annotation/html/org.Hs.eg.db.html (2022)
- Barrett, T., et al. NCBI GEO: archive for functional genomics data sets-update. Nucleic Acids Research. 41, 991-995 (2012).
- Gao, Y., Zhang, J., Zhao, F. Circular RNA identification based on multiple seed matching. Briefings in Bioinformatics. 19 (5), 803-810 (2018).
- Zhang, X. O., et al. Diverse alternative back-splicing and alternative splicing landscape of circular RNAs. Genome Research. 26 (9), 1277-1287 (2016).
- Memczak, S., et al. Circular RNAs are a large class of animal RNAs with regulatory potency. Nature. 495 (7441), 333-338 (2013).
- Wang, K., et al. MapSplice: Accurate mapping of RNA-seq reads for splice junction discovery. Nucleic Acids Research. 38 (18), 178 (2010).
- Song, X., et al. Circular RNA profile in gliomas revealed by identification tool UROBORUS. Nucleic Acids Research. 44 (9), 87 (2016).
- Hansen, T. B. Improved circRNA identification by combining prediction algorithms. Frontiers in Cell and Developmental Biology. 6, 20 (2018).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: A bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Ma, X. K., et al. CIRCexplorer3: A CLEAR pipeline for direct comparison of circular and linear RNA expression. Genomics Proteomics Bioinformatics. 17 (5), 511-521 (2019).
- Gaffo, E., Buratin, A., Dal Molin, A., Bortoluzzi, S. Sensitive, reliable and robust circRNA detection from RNA-seq with CirComPara2. Briefings in Bioinformatics. 23 (1), (2022).
- Glažar, P., Papavasileiou, P., Rajewsky, N. circBase: a database for circular RNAs. RNA. 20 (11), 1666-1670 (2014).
- Tan, S., et al. Circular RNA F-circEA-2a derived from EML4-ALK fusion gene promotes cell migration and invasion in non-small cell lung cancer. Molecular Cancer. 17 (1), 138 (2018).
- Guarnerio, J., et al. Oncogenic role of Fusion-circRNAs Derived from cancer-associated chromosomal translocations. Cell. 165 (2), 289-302 (2016).
- McGeary, S. E., et al. The biochemical basis of microRNA targeting efficacy. Science. 366 (6472), (2019).
- Enright, A. J., et al. MicroRNA targets in Drosophila. Genome Biology. 5 (1), 1 (2003).
- Rehmsmeier, M., Steffen, P., Hochsmann, M., Giegerich, R. Fast and effective prediction of microRNA/target duplexes. RNA. 10 (10), 1507-1517 (2004).
- Zhang, D., et al. AllEnricher: a comprehensive gene set function enrichment tool for both model and non-model species. BMC Bioinformatics. 21 (1), 106 (2020).
- Zhou, Y., et al. Metascape provides a biologist-oriented resource for the analysis of systems-level datasets. Nature Communications. 10 (1), 1523 (2019).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved