Method Article
In Silico Identifizierung und Charakterisierung von circRNAs während Wirt-Pathogen-Interaktionen
In diesem Artikel
Zusammenfassung
Das hier eingereichte Protokoll erläutert die vollständige In-silico-Pipeline , die für die Vorhersage und funktionelle Charakterisierung von circRNAs aus RNA-Sequenzierungs-Transkriptomdaten zur Untersuchung von Wirt-Pathogen-Interaktionen erforderlich ist.
Zusammenfassung
Zirkuläre RNAs (circRNAs) sind eine Klasse nicht-kodierender RNAs, die durch Back-Sppleißen gebildet werden. Diese circRNAs werden vor allem auf ihre Rolle als Regulatoren verschiedener biologischer Prozesse untersucht. Bemerkenswert ist, dass neue Erkenntnisse zeigen, dass Wirts-circRNAs bei einer Infektion mit Krankheitserregern (z. B. Influenza und Coronaviren) differentiell exprimiert werden können, was auf eine Rolle von circRNAs bei der Regulierung der angeborenen Immunantwort des Wirts hindeutet. Untersuchungen zur Rolle von circRNAs bei pathogenen Infektionen sind jedoch begrenzt durch die Kenntnisse und Fähigkeiten, die erforderlich sind, um die notwendigen bioinformatischen Analysen durchzuführen, um DE-circRNAs aus RNA-Sequenzierungsdaten (RNA-seq) zu identifizieren. Die bioinformatische Vorhersage und Identifizierung von circRNAs ist von entscheidender Bedeutung vor jeder Verifizierung und funktionellen Studien mit kostspieligen und zeitaufwändigen Nasslabortechniken. Um dieses Problem zu lösen, wird in diesem Manuskript ein Schritt-für-Schritt-Protokoll zur In-silico-Vorhersage und Charakterisierung von circRNAs unter Verwendung von RNA-seq-Daten bereitgestellt. Das Protokoll kann in vier Schritte unterteilt werden: 1) Vorhersage und Quantifizierung von DE-circRNAs über die CIRIquant-Pipeline; 2) Annotation mittels circBase und Charakterisierung von DE circRNAs; 3) Vorhersage der CircRNA-miRNA-Interaktion durch die Circr-Pipeline; 4) Analyse der funktionellen Anreicherung von circRNA-Elterngenen unter Verwendung von Gene Ontology (GO) und Kyoto Encyclopedia of Genes and Genomes (KEGG). Diese Pipeline wird nützlich sein, um die zukünftige In-vitro - und In-vivo-Forschung voranzutreiben, um die Rolle von circRNAs bei Wirt-Pathogen-Interaktionen weiter zu entschlüsseln.
Einleitung
Wirt-Pathogen-Interaktionen stellen ein komplexes Zusammenspiel zwischen den Krankheitserregern und den Wirtsorganismen dar, das die angeborenen Immunantworten des Wirts auslöst, die schließlich zur Entfernung eindringender Krankheitserreger führen 1,2. Bei pathogenen Infektionen wird eine Vielzahl der Immungene des Wirts reguliert, um die Vermehrung und Freisetzung von Krankheitserregern zu hemmen. Zu den häufigen Interferon-stimulierten Genen (ISGs), die bei pathogenen Infektionen reguliert werden, gehören ADAR1, IFIT1, IFIT2, IFIT3, ISG20, RIG-I und OASL 3,4. Studien haben gezeigt, dass neben proteinkodierenden Genen auch nicht-kodierende RNAs wie lange nicht-kodierende RNAs (lncRNAs), microRNAs (miRNAs) und zirkuläre RNAs (circRNAs) eine Rolle spielen und gleichzeitig bei pathogenen Infektionen reguliert werden 5,6,7. Im Gegensatz zu proteinkodierenden Genen, die Proteine hauptsächlich als funktionelle Moleküle kodieren, sind nicht-kodierende RNAs (ncRNAs) als Regulatoren von Genen auf transkriptioneller und posttranskriptioneller Ebene bekannt. Allerdings sind Studien, in denen nicht-kodierende RNAs, insbesondere circRNAs, an der Regulation der Immungene des Wirts beteiligt sind, im Vergleich zu den proteinkodierenden Genen nicht gut berichtet.
CircRNAs zeichnen sich weitgehend durch ihre kovalent geschlossene Endlosschleifenstruktur aus, die durch einen nicht-kanonischen Spleißprozess namens Back-Spleißen8 erzeugt wird. Im Gegensatz zum Spleißen verwandter linearer RNAs wird beim Back-Splicing die nachgeschaltete Donorstelle an die stromaufwärts gelegene Akzeptorstelle ligiert, wodurch eine kreisförmige Struktur entsteht. Derzeit werden drei verschiedene Back-Spleiß-Mechanismen für die Biogenese von circRNAs vorgeschlagen. Dabei handelt es sich um RNA-bindende Proteine (RBP)-vermittelte Zirkularisierung 9,10, Intron-Paarungs-getriebene Zirkularisierung 11 und Lariat-getriebene Zirkularisierung12,13,14. Da circRNAs in einer kreisförmigen Struktur Ende-zu-Ende verbunden sind, neigen sie dazu, von Natur aus resistent gegen normale Exonuklease-Verdauungen zu sein und gelten daher als stabiler als ihre linearen Gegenstücke15. Ein weiteres gemeinsames Merkmal von circRNAs ist die zell- oder gewebetypspezifische Expression in Wirten16.
Wie ihre einzigartige Struktur und ihre zell- oder gewebespezifische Expression vermuten lassen, haben circRNAs wichtige biologische Funktionen in Zellen übernommen. Bis heute ist eine der herausragenden Funktionen von circRNAs ihre Rolle als microRNA (miRNA)-Schwämme17,18. Diese regulatorische Rolle von circRNAs erfolgt durch die komplementäre Bindung von circRNA-Nukleotiden an die Seed-Region von miRNAs. Eine solche circRNA-miRNA-Interaktion hemmt die normalen regulatorischen Funktionen der miRNAs auf den Ziel-mRNAs und reguliert so die Expression der Gene19,20. Darüber hinaus ist bekannt, dass circRNAs die Genexpression regulieren, indem sie mit RNA-bindenden Proteinen (RBPs) interagieren und RNA-Protein-Komplexe bilden21. Obwohl circRNAs als nicht-kodierende RNAs klassifiziert werden, gibt es auch Hinweise darauf, dass circRNAs als Vorlagen für die Proteintranslation fungieren können22,23,24.
In jüngster Zeit wurde gezeigt, dass circRNAs eine zentrale Rolle bei der Regulierung der Wirt-Pathogen-Interaktionen spielen, insbesondere zwischen Wirt und Viren. Im Allgemeinen wird angenommen, dass Wirts-circRNAs bei der Regulierung der Immunantwort des Wirts helfen, um die eindringenden Krankheitserreger zu eliminieren. Ein Beispiel für circRNA, die die Immunantwort des Wirts fördert, ist circRNA_0082633, berichtet von Guo et al.25. Diese circRNA verstärkt die Signalübertragung von Typ-I-Interferon (IFN) in A549-Zellen, was dazu beiträgt, die Replikation von Influenzaviren zu unterdrücken25. Darüber hinaus berichteten Qu et al. auch über eine humane intronische circRNA, genannt circRNA AIVR, die die Immunität fördert, indem sie die Expression des CREB-bindenden Proteins (CREBBP), einem Signalwandler von IFN-βreguliert 26,27. Es gibt jedoch auch circRNAs, von denen bekannt ist, dass sie die Pathogenese von Krankheiten bei einer Infektion fördern. So berichteten Yu et al. kürzlich über die Rolle einer circRNA, die aus der GATA-Zinkfingerdomäne gespleißt wird, die das 2A-Gen (circGATAD2A) enthält, bei der Förderung der Replikation des H1N1-Virus durch die Hemmung der Autophagie der Wirtszelle28.
Um circRNAs effektiv untersuchen zu können, wird in der Regel ein genomweiter circRNA-Vorhersagealgorithmus implementiert, gefolgt von einer In-silico-Charakterisierung der vorhergesagten circRNA-Kandidaten, bevor funktionelle Studien durchgeführt werden können. Ein solcher bioinformatischer Ansatz zur Vorhersage und Charakterisierung von circRNAs ist kostengünstiger und zeiteffizienter. Es hilft, die Anzahl der Kandidaten zu verfeinern, die funktionell untersucht werden sollen, und könnte möglicherweise zu neuen Erkenntnissen führen. In dieser Arbeit stellen wir ein detailliertes bioinformatisches Protokoll für die in silico Identifizierung, Charakterisierung und funktionelle Annotation von circRNAs während der Wirt-Pathogen-Interaktionen zur Verfügung. Das Protokoll umfasst die Identifizierung und Quantifizierung von circRNAs aus RNA-Sequenzierungsdatensätzen, die Annotation über circBase und die Charakterisierung der circRNA-Kandidaten in Bezug auf circRNA-Typen, Anzahl überlappender Gene und vorhergesagte circRNA-miRNA-Interaktionen. Diese Studie liefert auch die funktionelle Annotation der circRNA-Elterngene durch Genontologie (GO) und die Anreicherungsanalyse der Kyoto Encyclopedia of Genes and Genomes (KEGG).
Protokoll
In diesem Protokoll wurden de-identifizierte ribosomale RNA (rRNA)-depletierte RNA-seq-Bibliotheksdatensätze, die aus den mit dem Influenza-A-Virus infizierten menschlichen Makrophagenzellen erstellt wurden, aus der Gene Expression Omnibus (GEO)-Datenbank heruntergeladen und verwendet. Die gesamte bioinformatische Pipeline von der Vorhersage bis zur funktionellen Charakterisierung von circRNAs ist in Abbildung 1 zusammengefasst. Jeder Teil der Pipeline wird in den folgenden Abschnitten näher erläutert.
1. Vorbereitung, Download und Einrichtung vor der Datenanalyse
HINWEIS: Alle in dieser Studie verwendeten Softwarepakete sind kostenlos und quelloffen.
- Herunterladen der erforderlichen Tools auf der Linux-Plattform
- Laden Sie die erforderliche Software und die Tools, die im Materialverzeichnis aufgeführt sind, herunter und installieren Sie sie auf einem Linux-Hochleistungscomputer, indem Sie die Anweisungen des Entwicklers befolgen.
HINWEIS: Die meisten Tools und Software verfügen über eigene Online-GitHub-Seiten oder Dokumentationen mit Anweisungen zur Installation und Verwendung ihrer Tools (siehe Materialtabelle). - Laden Sie die gewünschten RNA-seq-Datensätze für die circRNA-Detektion und -Analyse von Sequenzarchiv-Websites herunter (z. B. European Nucleotides Archive und Gene Expression Omnibus).
- Laden Sie die Referenzgenome (FASTA-Format) und Annotationsdateien (GTF/GFF3-Format) herunter, die mit dem Host kompatibel sind, von dem der RNA-seq-Datensatz erstellt wurde. Referenzgenome und Annotationsdateien des Wirts sind in der Regel in Online-Genombrowsern wie dem National Center for Biotechnology Information (NCBI), der University of California Santa Cruz (UCSC) und den Ensembl-Websites zu finden.
- Laden Sie die erforderliche Software und die Tools, die im Materialverzeichnis aufgeführt sind, herunter und installieren Sie sie auf einem Linux-Hochleistungscomputer, indem Sie die Anweisungen des Entwicklers befolgen.
- Qualitätsprüfung von RNA-seq
- Geben Sie die FASTQ-Dateien in das FASTQC-Programm ein, um die Qualität der RNA-Sequenzen zu bestimmen. Wenn die Qualität der FASTQ-Dateien niedrig ist (z. B. 29,30 erforderlich sein.
2. Vorhersage und differentielle Expressionsanalyse von circRNAs mit CIRIquant
HINWEIS: Ein ausführlicheres Handbuch zur Installation und Durchführung der Differentialexpressionsanalyse finden Sie im Abschnitt Codeverfügbarkeit des CIRIquant-Papiers31. Die ergänzenden Daten enthalten auch einige der grundlegenden Befehle, die in diesem Protokoll verwendet werden.
- CircRNA-Vorhersagen
- Indizieren Sie zuerst das Referenzgenom des Wirts mit BWA- und HISAT2-Alignern. Führen Sie dann auf einem Linux-Terminal die Befehle bwa index 32 und hisat2-build33 im Verzeichnis des Referenzgenoms des Hosts aus, um es zu indizieren.
- Bereiten Sie als Nächstes eine YML-Konfigurationsdatei vor, die den Namen der Datei, den Pfad der Tools (BWA, HISAT2, stringtie34, samtools35), den Pfad zu den heruntergeladenen Referenzdateien (FASTA-Dateien des Referenzgenoms des Hosts, Annotationsdateien) und den Pfad zu den Indexdateien aus Schritt 2.1.1 enthält.
- Führen Sie das CIRIquant-Tool vom Terminal aus mit den Standard- oder manuellen Parametern aus. Der Benutzer kann den Bibliothekstyp (entweder stranged oder non-stranged) der RNA-seq-Daten angeben, wenn er das CIRIquant-Tool ausführt.
HINWEIS: Der Bibliothekstyp der RNA-seq-Daten kann bestimmt werden, indem der Typ des verwendeten Bibliotheksvorbereitungskits bekannt ist. Wenn die Identität des Bibliothekspräparationskits unbekannt ist, kann ein bioinformatisches RNA-seq-Kontrollpaket namens RSeQC36 verwendet werden, um die Strandedness von RNA-seq-Daten zu bestimmen.
- Differentielle Expressionsanalyse
HINWEIS: Das CIRIquant-Paket enthält prep_CIRIquant, prepDE.py und CIRI_DE_replicate; Daher sind für diese drei Tools keine zusätzlichen Downloads erforderlich.- Bereiten Sie eine Textdatei (.lst) mit einer Liste von Daten vor, die Folgendes enthält:
1. Spalte : IDs der in Schritt 2.1.3 verwendeten RNA-seq-Daten
2. Spalte: Pfad zu den GTF-Dateien, die von CIRIquant ausgegeben werden
3. Spalte: Gruppierung der RNA-seq-Daten, unabhängig davon, ob es sich um eine Kontroll- oder eine behandelte Gruppe handelt. - Ein Beispiel finden Sie in Tabelle 1 unten.
HINWEIS: Es ist nicht notwendig, die Kopfzeilen einzufügen, da sie nur als Referenz dienen. - Führen Sie auf dem Linux-Terminal prep_CIRIquant mit der in Schritt 2.2.1 vorbereiteten Textdatei (.lst) als Eingabe aus. Bei der Ausführung wird eine Liste von Dateien generiert: library_info.csv, circRNA_info.csv, circRNA_bsj.csv und circRNA_ratio.csv.
- Bereiten Sie eine zweite Textdatei mit einer Liste von Daten vor, die die RNA-seq-IDs und den Pfad zu ihrer jeweiligen StringTie-Ausgabe enthält. Das Dateilayout muss der Textdatei in Schritt 2.2.1 ohne die Gruppierungsspalte ähneln.
- Führen Sie prepDE.py mit der in Schritt 2.2.4 vorbereiteten Textdatei als Eingabe aus, um die Genzählmatrixdateien zu generieren.
- Führen Sie CIRI_DE_replicate mit den library_info.csv - und circRNA_bsj.csv Dateien aus Schritt 2.2.3 und der gene_count_matrix.csv Datei aus Schritt 2.2.5 als Eingaben aus, um die endgültige circRNA_de.tsv-Datei auszugeben.
- Bereiten Sie eine Textdatei (.lst) mit einer Liste von Daten vor, die Folgendes enthält:
- Filterung von DE-circRNAs
- Verwenden Sie R (im Computerterminal oder RStudio) oder eine beliebige Tabellenkalkulationssoftware (z. B. Microsoft Excel), um die aus Schritt 2.2.6 generierte Datei circRNA_de.tsv zu öffnen, um die Anzahl der differentiell exprimierten (DE) circRNAs zu filtern und zu bestimmen.
- Filtern Sie die DE-circRNAs nach den Kriterien LogFC > |2| und FDR < 0,05.
- Erstellen Sie eine Datei mit dem Namen DE_circRNAs.txt , um die Informationen von DE-circRNAs zu speichern.
3. Charakterisierung und Annotation von vorhergesagten DE-circRNAs
- Annotationsstatus von DE-circRNAs
- Laden Sie die Datei mit dem Namen DE_circRNAs.txt in RStudio , die aus der Liste der DE-circRNAs besteht, die aus Schritt 2.3.3 gefiltert wurden. Fügen Sie weitere Informationen hinzu, wie z. B. die genomischen Positionen (Chr, Start, Ende), die Strangorientierung (+ oder -), den Gennamen und den circRNA-Typ. Bevor Sie fortfahren, konvertieren Sie die genomischen Startkoordinaten der circRNA von CIRIquant in 0-basiert, indem Sie 1 Basenpaar subtrahieren.
HINWEIS: Die anderen oben genannten Informationen können den GTF-Dateien entnommen werden, die von CIRIquant (Supplementary File 1) ausgegeben werden. - Bestimmen Sie den Annotationsstatus der vorhergesagten DE-circRNAs, indem Sie eine Bibliothek herunterladen, die die genomischen Positionen der in der circRNA-Datenbank (z.B. circBase) hinterlegten circRNAs enthält.
HINWEIS: Stellen Sie sicher, dass die Genomversion, die zur Vorhersage der circRNAs verwendet wird, mit der circRNA-Datenbankbibliothek identisch ist, bevor Sie den Vergleich durchführen. Die hier verwendete circBase-Datendatei ist in dem in Github (https://github.com/bicciatolab/Circr)37 bereitgestellten Laufwerksordner frei verfügbar. - Sobald die beiden Dateien aus Schritt 3.1.1 und Schritt 3.1.2 vorbereitet sind, führen Sie das in Ergänzende Datei 1 angegebene R-Skript aus. Chromosomale Positionen von DE-circRNAs werden an die Bibliothek abgefragt, bevor ihnen der Status Annotated oder Unannotated zugewiesen wird.
- Laden Sie die Datei mit dem Namen DE_circRNAs.txt in RStudio , die aus der Liste der DE-circRNAs besteht, die aus Schritt 2.3.3 gefiltert wurden. Fügen Sie weitere Informationen hinzu, wie z. B. die genomischen Positionen (Chr, Start, Ende), die Strangorientierung (+ oder -), den Gennamen und den circRNA-Typ. Bevor Sie fortfahren, konvertieren Sie die genomischen Startkoordinaten der circRNA von CIRIquant in 0-basiert, indem Sie 1 Basenpaar subtrahieren.
- Charakterisierung von DE-circRNAs
- Verwenden Sie R und andere Tabellenkalkulationsprogramme, um die Anzahl der circRNAs entsprechend den circRNA-Typen (d. h. Exon, Intron, intergen und Antisense) und die Anzahl der Gene, über die sich die circRNAs erstrecken (1 oder >1), zusammenzufassen (Ergänzungsdatei 1).HINWEIS: CIRIquant kann nur vier Arten von circRNAs nachweisen (Exon, Intron, intergen und Antisense). Exon-Intron-circRNAs, auch ElciRNAs genannt, können von CIRIquant nicht nachgewiesen werden.
4. Vorhersage der circRNA-miRNA-Interaktion mit Circr
HINWEIS: Eine ausführlichere Anleitung zur Installation und Verwendung von Circr für die circRNA-miRNA-Interaktionsanalyse finden Sie unter: https://github.com/bicciatolab/Circr37.
- Vorbereitung von Dateien
- Entpacken und extrahieren Sie den Inhalt der Circr.zip Datei, nachdem Sie sie von der Circr GitHub-Seite mit der entsprechenden Software wie "WinRar" oder "7-zip" heruntergeladen haben, in ein neues Verzeichnis, in dem die Analyse durchgeführt wird.
- Installieren Sie die erforderlichen Softwareanwendungen (miRanda, RNAhybrid, Pybedtools und samtools), bevor Sie die circRNA-miRNA-Analyse durchführen.
- Referenzgenome und Annotationsdateien für verschiedene Organismen von Interesse, eine rRNA-Koordinatendatei, eine validierte miRNA-Interaktionsdatei und circBase-circRNA-Dateien werden vom Circr-Autor auf der Github-Seite (https://github.com/bicciatolab/Circr)37 bereitgestellt. Wenn Sie auf die Unterstützungsdateien im Laufwerksordner klicken, wählen Sie den Ordner für den gewünschten Organismus, den miRNA-Ordner und die circBase-Textdatei aus und laden Sie sie herunter.
- Nachdem Sie die erforderlichen Dateien in Schritt 4.1.3 heruntergeladen haben, erstellen Sie ein neues Verzeichnis mit dem Namen support_files in dem in Schritt 4.1.1 genannten Verzeichnis. Entpacken Sie dann den Inhalt und extrahieren Sie ihn in das support_files Verzeichnis.
- Indizieren Sie die Referenzgenomdatei des interessierenden Organismus mit dem Befehl samtools faidx (Supplementary File 1).
- Bereiten Sie eine Eingabedatei vor, die aus den Koordinaten der gewünschten DE-circRNAs in einer tabulatorgetrennten BED-Datei besteht, wie in Tabelle 2 dargestellt.
HINWEIS: Da circRNAs, die von CIRIquant vorhergesagt werden, nicht 0-basiert sind, ist es notwendig, 1 bp an der Startkoordinate zu minuieren (wie in Schritt 3.1.1 erwähnt), bevor sie in das BED-Format konvertiert werden. Die in Tabelle 2 gezeigten Header dienen nur als Referenz und werden in den BED-Dateien nicht benötigt. - Stellen Sie an dieser Stelle sicher, dass die erwartete Ordnerstruktur für die Circr-Analyse mit der in Abbildung 2 dargestellten Struktur übereinstimmt.
- Ausführen Circr.py
- Führen Sie Circr.py mit Python 3 aus, und geben Sie als Argumente die circRNA-Eingabedatei, das FASTA-Genom des gewünschten Organismus, die Genomversion des ausgewählten Organismus, die Anzahl der Threads und den Namen der Ausgabedatei in der Befehlszeile an.
- Wenn der betreffende Organismus nicht in dem in Schritt 4.1.3 aufgeführten Laufwerksordner bereitgestellt wird oder wenn der Benutzer es vorzieht, einen benutzerdefinierten Satz von Dateien zum Ausführen der Analyse zu haben, müssen beim Ausführen von Circr.py zusätzliche Befehle enthalten sein, die den Speicherort dieser Dateien angeben.
- Nachdem die Circr-Analyse abgeschlossen ist, gibt das Programm eine circRNA-miRNA-Interaktionsdatei im csv-Format aus.
- Filtern Sie die Ergebnisse der circRNA-miRNA-Interaktion entsprechend der benutzerspezifischen Präferenz. Für diese Studie werden die Vorhersagen mit Rstudio nach den folgenden Kriterien gefiltert:
-Wird von allen drei Software-Tools erkannt
-Zwei oder mehr Bindungsstellen, die sowohl von Targetscan als auch von miRanda gemeldet werden
-Wird entweder in den Spalten "AGO" oder "validiert" identifiziert
-Filtern Sie keine Seed-Region-Interaktionen heraus - Schreiben Sie die circRNAs, die die gefilterten Bedingungen aus Schritt 4.2.3 übergeben, in eine neue Textdatei mit dem Namen circRNA_miRNA.txt. Eine solche Filterung kann die Zuverlässigkeit der vorhergesagten Wechselwirkungen erhöhen.
5. Aufbau des ceRNA-Netzwerks
HINWEIS: Eine ausführliche Anleitung zur Verwendung von Cytoscape finden Sie unter: http://manual.cytoscape.org/en/stable/ und https://github.com/cytoscape/cytoscape-tutorials/wiki#introduction
- Download und Vorbereitung
- Laden Sie die neueste Version von Cytoscape38 herunter von: https://cytoscape.org/download.html.
- Führen Sie den in Schritt 5.1.1 heruntergeladenen Installationsassistenten aus und wählen Sie den Speicherort für die Cytoscape-Software aus.
- Bereiten Sie eine tabulatorgetrennte Datei vor, die die interessierenden circRNAs und ihre Ziel-miRNA enthält. Die erste Spalte besteht aus dem circRNA-Namen; die zweite Spalte gibt den RNA-Typ aus der ersten Spalte an. die dritte Spalte ist die Ziel-miRNA; und die vierte Spalte gibt die Art der RNA aus der dritten Spalte an. Ein Beispiel für die Datei ist in Tabelle 3 dargestellt.
- Aufbau der ceRNA-Netzwerkkarte
- Öffnen Sie die in Schritt 5.1.2 installierte Cytoscape-Software.
- Navigieren Sie in Cytoscape zu Datei > importieren Sie > Netzwerk aus Datei. Wählen Sie die Datei aus, die in Schritt 5.1.3 vorbereitet wurde.
- Wählen Sie auf der neuen Registerkarte die erste und zweite Spalte als "Quellknoten" und "Quellknotenattribut" aus, während Sie die dritte und vierte Spalte als "Zielknoten" bzw. "Zielknotenattribut" auswählen. Klicken Sie auf OK und das Netzwerk wird oben rechts in Cytoscape angezeigt.
- Um den visuellen Stil des Netzwerks zu ändern, klicken Sie auf die Schaltfläche Stil auf der linken Seite von Cytoscape.
- Drücken Sie auf den Pfeil auf der rechten Seite von Füllfarbe. Wählen Sie Typ für die Spalte und Diskrete Zuordnung für den Zuordnungstyp. Wählen Sie dann die gewünschte Farbe für jeden der RNA-Typen aus.
- Nachdem Sie die Farbe geändert haben, ändern Sie die Form der Knoten, indem Sie zu Form navigieren und Schritt 5.2.5 ausführen.
6. Analyse der funktionalen Anreicherung
- Genontologie (GO) und Kyoto Encyclopedia of Genes and Genomes (KEGG) Analyse für das Elterngen der circRNAs
- Stellen Sie sicher, dass clusterProfiler39,40 und org. Hs.B.db41 Pakete wurden in Rstudio installiert. Die org. Hs.eg.db41-Paket ist ein genomweites Annotationspaket nur für Menschen. Wenn es sich bei dem interessierenden Organismus um eine andere Art handelt, siehe: https://bioconductor.org/packages/release/BiocViews.html#OrgDb
- Importieren Sie die DE_circRNA Informationen aus Schritt 2.3.1 in den Rstudio-Arbeitsbereich.
- Verwenden Sie das elterliche Gen der circRNAs, die in dieser Datei bereitgestellt werden, für die Anreicherungsanalyse in den nächsten Schritten. Wenn der Benutzer jedoch das Gensymbol in andere Formate, wie z. B. die Entrez-ID, konvertieren möchte, verwenden Sie eine Funktion wie "bitr".
- Wenn Sie die Gen-ID als Eingabe verwenden, führen Sie die GO-Anreicherungsanalyse mit der enrichGO-Funktion innerhalb des clusterProfiler 39,40-Pakets mit Standardparametern aus.
- Wenn Sie die Gen-ID als Eingabe verwenden, führen Sie die KEGG-Anreicherungsanalyse mit der enrichKEGG-Funktion innerhalb des clusterProfiler 39,40-Pakets mit den Standardparametern aus.
Ergebnisse
Das im vorherigen Abschnitt aufgeführte Protokoll wurde modifiziert und konfiguriert, um dem Linux-Betriebssystem gerecht zu werden. Der Hauptgrund dafür ist, dass die meisten Modulbibliotheken und Pakete, die an der Analyse von circRNAs beteiligt sind, nur auf der Linux-Plattform funktionieren können. In dieser Analyse wurden de-identifizierte ribosomale RNA (rRNA)-depletierte RNA-seq-Bibliotheksdatensätze, die aus den mit dem Influenza-A-Virus infizierten menschlichen Makrophagenzellen hergestellt wurden, aus der GEO-Datenbank42 heruntergeladen und zur Generierung der repräsentativen Ergebnisse verwendet.
CircRNA-Vorhersage und -Quantifizierung
In dieser Analyse wurden ribosomale RNA (rRNA)-depletierte RNA-seq-Bibliotheksdatensätze, die aus den mit dem Influenza-A-Virus infizierten humanen Makrophagenzellen hergestellt wurden, verwendet, um circRNA-Detektion und funktionelle Analysen durchzuführen. Wie im Protokollabschnitt spezifiziert, wurde CIRIquant verwendet, um identifizierte circRNAs zu identifizieren und eine DE-Analyse durchzuführen, wobei die Datensätze der RNA-seq-Bibliothek als Eingabe verwendet wurden. Die verwendeten Referenzdateien basieren auf der neuesten Version des menschlichen Genoms (hg38). Tabelle 4 zeigt ein Beispiel für das endgültige Ergebnis der CIRIquant-Analyse. Die Identifizierung und Filterung von DE-circRNAs aus der CIRIquant-Ausgabe erfolgte durch einfache RStudio-Skripte (Supplementary File 1). CircRNAs werden nur dann als DE klassifiziert, wenn der Wert für die False-Discovery-Rate (FDR) <0,05 und die LogFC-Änderung (LogFC) >|2| beträgt. Tabelle 5 zeigt die Gesamtzahl der nachgewiesenen circRNAs und DE-circRNAs. Insgesamt wurden 35.846 circRNAs nachgewiesen, davon 306 DE. Die in diesem Output detektierten DE-circRNAs werden vollständig hochreguliert (LogFC > 2), wobei keine herunterreguliert wird (LogFC < 2).
Annotation und Charakterisierung von DE-circRNAs
Annotationsstatus von DE-circRNAs
Die identifizierten DE-circRNAs wurden mit einer etablierten circRNA-Datenbank, circBase, abgeglichen. Da die in circBase hinterlegten circRNA-Koordinaten jedoch auf einer früheren humanen Genomversion (hg19) basieren, müssen die circRNA-Koordinaten von circBase für den Abgleich in dieser Studie in die aktuelle Humangenomversion (hg38) konvertiert werden. Zusätzlich muss die Startkoordinate aus der 1-basierten Ausgabe von CIRIquant in 0-basiert konvertiert werden. Die in die hg38-Version konvertierten circRNA-Koordinaten von circBase werden in einem Laufwerksordner in Github (https://github.com/bicciatolab/Circr)37 bereitgestellt. Anschließend wurden die Rstudio-Skripte (Supplementary File 1) verwendet, um den Annotationsstatus von circRNAs in einer neuen Datenrahmenspalte zuzuweisen. Tabelle 6 zeigt ein Beispiel für circRNAs mit dem Annotationsstatus.
Charakterisierung von DE-circRNAs
Dieser Teil wurde vollständig über R-Skripte in der RStudio-Software ausgeführt. R-Skripts vereinfachen die Analyseprozesse, und es sind nur grundlegende Kenntnisse erforderlich.
CircRNA-Typen
In diesem Schritt wurden DE-circRNAs anhand ihrer circRNA-Typen (Antisense, Exonic, Intergenic und Intronic) anhand ihrer genomischen Positionen charakterisiert. Tabelle 7 zeigt die prozentuale Aufschlüsselung der verschiedenen circRNA-Typen, die von den identifizierten DE-circRNAs umfasst werden. Von den insgesamt 306 DE-circRNAs wurden 263 circRNAs (85,95%) als exonische circRNAs identifiziert, der am häufigsten identifizierte circRNA-Typ. Intronische circRNAs sind der am zweithäufigsten identifizierte circRNA-Typ, der 17 DE-circRNAs umfasst, was bis zu 5,56 % der gesamten DE-circRNAs ausmacht. Es folgen intergene circRNAs (16 DE circRNAs ~5,23%) und Antisense circRNAs (10 DE circRNAs ~3,27%).
Anzahl der Gene pro circRNA
CircRNAs, die von CIRIquant identifiziert wurden, können sich über eine Reihe von Genen hinweg überlappen. Bisher konzentrieren sich die meisten Studien auf circRNAs, die sich über ein Gen erstrecken. Daher werden in diesem Protokoll die circRNA-Kandidaten, die mehr als ein Gen umfassen, von der nachgelagerten Analyse ausgeschlossen. Tabelle 8 beschreibt die Anzahl und den prozentualen Anteil der DE-circRNAs, die sich über ein und mehrere Gene erstrecken. In dieser Tabelle werden intergene circRNAs (16 DE-circRNAs) ausgeschlossen, da sie keine Wirtsgene überlappen, während die übrigen circRNA-Typen (290 DE-circRNAs) dieser Analyse unterzogen werden. Von den 290 DE-circRNAs umfasst die Mehrheit der DE-circRNAs (261 circRNAs ~90%) nur ein Gen, während die restlichen 29 circRNAs (~10%) mehr als ein Gen umfassen.
Aufbau des ceRNA-Netzwerks
Ein ceRNA-Netzwerk wird in der Regel gezeichnet, um die circRNA-miRNA-Interaktionen zu visualisieren, nachdem sie vorhergesagt wurden. In Abbildung 3 unten wurde nur eine DE-circRNA als repräsentatives Ergebnis ausgewählt, nämlich die hsa_DE_58 circRNA. Basierend auf Circr-Vorhersagen können hsa_DE_58 bis zu neun verschiedene miRNAs mit einem Schwamm versehen. Diese neun miRNAs werden identifiziert, nachdem sie durch strenge Kriterien gefiltert wurden.
Analyse der funktionalen Anreicherung
GO- und KEGG-Analyse der circRNA-Elterngene
Abbildung 4 unten zeigt ein Blasendiagramm der funktionellen Anreicherung von DE circRNA Elterngenen durch die GO-Analyse. Grundsätzlich zielt die GO-Analyse darauf ab, die biologischen Prozesse, zellulären Standorte und molekularen Funktionen zu entschlüsseln, die bei der untersuchten Erkrankung, in diesem Fall der virusinfizierten Probe, angereichert oder beeinflusst werden. Die Anreicherung wird nur dann als statistisch signifikant betrachtet und im Blasendiagramm dargestellt, wenn der p-Wert < 0,01 liegt. Wie in Abbildung 4 gezeigt, umfassen die drei wichtigsten Anreicherungen für die biologischen Prozesse (BP) die Biogenese des Ribonukleoproteinkomplexes, die Reaktion auf das Virus und die Regulation der Reaktion auf einen biotischen Stimulus, während für die molekularen Funktionen (MF) nur die katalytische Aktivität, die auf RNA wirkt, und die Bindung einzelsträngiger RNA statistisch angereichert sind. Dagegen ist nur der Retromer-Komplex statistisch für die zellulären Komponenten (CC) angereichert.
Abbildung 5 zeigt die KEGG-Anreicherungsanalyse der DE circRNA Elterngene in einem Bubble Plot. Ähnlich wie bei der GO-Anreicherungsanalyse wird die KEGG-Anreicherung nur dann als statistisch signifikant betrachtet und in einem Blasendiagramm dargestellt, wenn der p-Wert < 0,01 beträgt. In diesem Fall wurden nur zwei KEGG-Begriffe angereichert, nämlich der Influenza-A- und der virale Lebenszyklus (HIV-1).
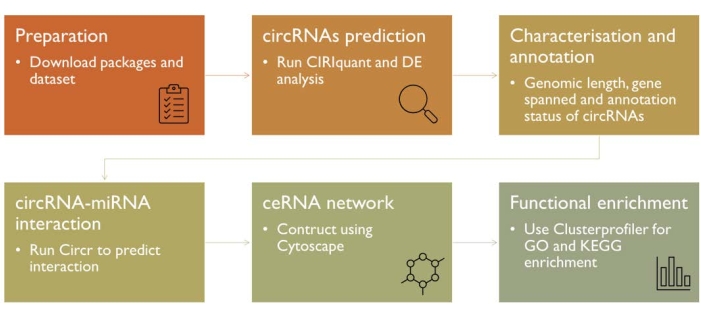
Abbildung 1: Die Pipeline zur Vorhersage und funktionellen Charakterisierung von circRNAs. Die Pipeline zeigt einen einfachen Überblick über die wichtigsten Schritte von Anfang bis Ende, einschließlich der Installation der erforderlichen Softwarepakete, der Vorhersage und Quantifizierung der circRNA-Expression, des Aufbaus des ceRNA-Netzwerks und der Durchführung der funktionellen Anreicherung des circRNA-Elterngens. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 2: Ordnerbaumstruktur für Circr. Diese Ordnerbaumstruktur muss vor dem Ausführen der Circr-Software festgelegt werden, um die erforderlichen Dateien für die Analyse zu erkennen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 3: ceRNA-Netzwerk, bestehend aus der circRNA-miRNA-Interaktion. Die blaue ovale Form stellt die circRNA dar, während die orangefarbenen Dreiecke die miRNAs darstellen. Die durchgezogenen Linien, die die circRNA mit miRNAs verbinden, beschreiben die potentielle miRNA-Schwammfunktion der hsa_DE_58 circRNA. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
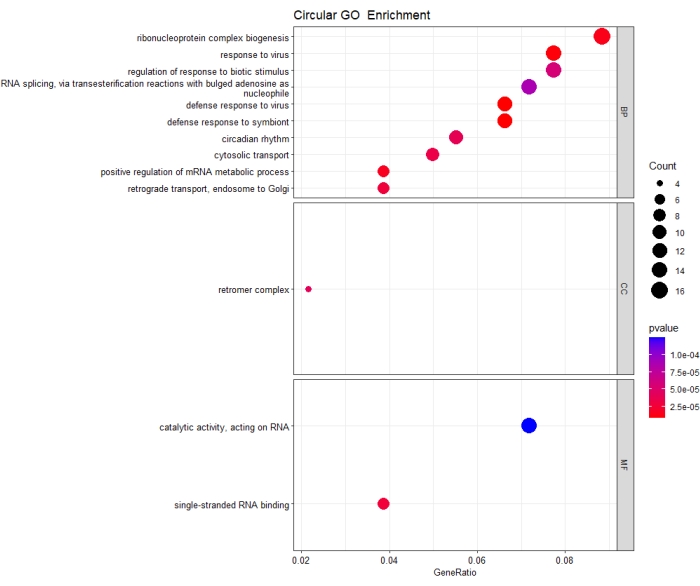
Abbildung 4: Blasendiagramm der GO-Anreicherungsanalyse von DE circRNA-Elterngenen. GeneRatio auf der x-Achse ist die Anzahl der Gene in der Eingabeliste, die dem angegebenen GO-Term zugeordnet sind, und dividiert die Gesamtzahl der Eingabegene. Die Punktgröße im Diagramm wird durch den Zählwert dargestellt, d. h. die Anzahl der Gene in der Eingabeliste, die dem angegebenen GO-Term zugeordnet sind. Je größer die Punkte sind, desto größer ist die Anzahl der Eingabegene, die mit dem Begriff verbunden sind. Außerdem sind die Punkte im Diagramm basierend auf dem p-Wert farbcodiert. Der p-Wert wird berechnet, indem die beobachtete Häufigkeit eines Annotationsterms mit der zufällig erwarteten Häufigkeit verglichen wird. Die einzelnen Terme gelten über einen Cut-off-Wert hinaus als angereichert (p-Wert < 0,01). Der Farbverlauf des p-Wertes von blau bis rot deutet auf eine zunehmende Anreicherung der Terme hin. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 5: KEGG-Anreicherungsanalyse von DE circRNA-Elterngenen. GeneRatio auf der x-Achse ist die Anzahl der Gene in der Eingabeliste, die dem angegebenen KEGG-Term zugeordnet sind, und dividiert die Gesamtzahl der Eingabegene. Die Punktgröße im Diagramm wird durch den Zählwert dargestellt, d. h. die Anzahl der Gene in der Eingabeliste, die dem angegebenen KEGG-Term zugeordnet sind. Je größer die Punkte sind, desto größer ist die Anzahl der Eingabegene, die mit dem Begriff verbunden sind. Außerdem sind die Punkte im Diagramm basierend auf dem p-Wert farbcodiert. Der p-Wert wird berechnet, indem die beobachtete Häufigkeit eines Annotationsterms mit der zufällig erwarteten Häufigkeit verglichen wird. Einzelne Terme gelten als über einen Cut-off-Wert hinaus angereichert (p-Wert < 0,01). Der Farbverlauf des p-Wertes von blau bis rot deutet auf eine zunehmende Anreicherung der Terme hin. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
| Name des Beispiels | Pfad zur CIRIquant-Ausgabe-GTF-Datei | Gruppierung |
| Steuerung 1 | /path/to/CIRIquant/ctrl1.gtf | C |
| Steuerung 2 | /pfad/zu/CIRIquant/ctrl2.gtf | C |
| Infiziert 1 | /pfad/zu/CIRIquant/infect1.gtf | T |
| Infiziert 2 | /pfad/zu/CIRIquant/infect2.gtf | T |
Tabelle 1: Die .lst-Dateivorbereitung von CIRIquant. Die Zielpfade der Kontroll- und behandelten Proben aus der CIRIquant-Ausgabe werden in eine Textdatei geschrieben, um die Expressionen der circRNA zwischen den beiden Probentypen zu vergleichen.
| Chr | Anfangen | Ende | Name | . | Litze |
| Chr2 | 137428930 | 137433876 | hsa_circ_000076 | . | - |
| Chr2 | 154705868 | 154706632 | hsa_circ_000105 | . | - |
| Chr2 | 159104273 | 159106793 | hsa_circ_000118 | . | - |
| Chr2 | 159215701 | 159226125 | hsa_circ_000119 | . | - |
| Chr4 | 39980067 | 39980129 | hsa_circ_002584 | . | - |
Tabelle 2: Beispiel für eine BED-Datei für Circr. Sechs Spalten (Chr, Start, Ende, Name, Gen und Strang), die mit den circRNAs verknüpft sind, sind erforderlich, um die BED-Datei zu generieren.
| circRNA_name | Art | miRNA_name | Art |
| DE_circRNA_1 | circRNA | miR-001 | Mirna |
| DE_circRNA_1 | circRNA | miR-002 | Mirna |
| DE_circRNA_2 | circRNA | miR-003 | Mirna |
| DE_circRNA_2 | circRNA | miR-004 | Mirna |
Tabelle 3: Cytoscape-Eingabedatei. Vier Spalten (circRNA_name, Type, miRNA_name und Type) müssen in eine Textdatei geschrieben werden.
| CircRNA | logFC | logCPM | LR | P-Wert | DE | FDR |
| Chr4:17595410|17598558 | 8.167934481 | -0.039318634 | 185.5341965 | 3.00E-42 | 1 | 1.08E-37 |
| Chr16:18834892|18850467 | -3.955083482 | -4.397235736 | 2.982607619 | 0.08416358 | 0 | 0.282478158 |
| Chr14:73198031|73211942 | 2.493964729 | -4.448176684 | 2.736442046 | 0.09808293 | 0 | 0.282478158 |
Tabelle 4: Teil der finalen Ausgabedatei (.csv) von CIRIquant. CIRIquant liefert Informationen wie LogFC, Log-Counts per Million (LogCPM), logistische Regression (LR), p-Wert, Differentialausdruck und FDR.
| CIRIquant Ergebnisse | |||
| Gesamt | DE | Oben | Herab |
| 35846 | 306 | 306 | 0 |
Tabelle 5: Eine Zusammenfassung der Gesamtzahl der identifizierten und differentiell exprimierten (DE) circRNAs. Insgesamt wurden 35.846 circRNAs nachgewiesen, davon 306 DE-circRNAs. Alle 306 DE-circRNAs sind in behandelten Proben im Vergleich zu Kontrollproben hochreguliert (wobei keine herunterreguliert wird).
| Custom_Name | Annotation_Status |
| hsa_DE_22 | Nicht kommentiert |
| hsa_DE_2 | Kommentierte |
| hsa_DE_58 | Nicht kommentiert |
| hsa_DE_3 | Kommentierte |
Tabelle 6: Tabelle der benutzerdefinierten circRNA-Namen mit Annotationsstatus. CircRNAs werden in einer Datenbank bekannter hinterlegter circRNAs (circBase) abgefragt. Wenn die circRNA in der Datenbank vorhanden ist, wird sie als annotiert markiert, während das Fehlen der circRNA als nicht annotiert markiert wird.
| CircRNA-Typ | Freq | Prozentsatz |
| Antisense | 10 | 3.27% |
| exon | 263 | 85.95% |
| intergen | 16 | 5.23% |
| intron | 17 | 5.56% |
Tabelle 7: Identifizierte circRNA-Typen. CircRNAs können basierend auf ihrer Sequenzregion weiter in verschiedene Arten von circRNAs kategorisiert werden, nämlich exonisch, intronisch, antisense und intergen.
| Anzahl der elterlichen Gene | Freq | Prozentsatz |
| 1 | 261 | 90% |
| > 1 | 29 | 10% |
Tabelle 8: Prozentualer Anteil der circRNAs mit der unterschiedlichen Anzahl von Genen überspannt. CircRNAs werden üblicherweise von Exons eines Gens kodiert, aber auch circRNAs, die mehr als ein Gen umfassen, können von CIRIquant nachgewiesen werden.
Ergänzende Datei 1: Skripte, die im Protokoll verwendet werden. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Um den Nutzen dieses Protokolls zu veranschaulichen, wurde RNA-seq aus mit dem Influenza-A-Virus infizierten humanen Makrophagenzellen als Beispiel verwendet. CircRNAs, die als potentielle miRNA-Schwämme in Wirt-Pathogen-Interaktionen fungieren, und ihre GO- und KEGG-funktionelle Anreicherung innerhalb eines Wirts wurden untersucht. Obwohl es eine Vielzahl von circRNA-Tools gibt, die online verfügbar sind, ist jedes von ihnen ein eigenständiges Paket, das nicht miteinander interagiert. Hier stellen wir einige der Werkzeuge zusammen, die für die circRNA-Vorhersage und -Quantifizierung, die funktionelle Anreicherung von circRNAs, die Vorhersage der circRNA-miRNA-Interaktion und den Aufbau von ceRNA-Netzwerken erforderlich sind. Dieses optimierte Protokoll ist zeitsparend und kann auf klinische Proben angewendet werden, um circRNA-Kandidaten mit diagnostischen und prognostischen Werten nachzuweisen.
Im Wesentlichen haben wir CIRIquant31 verwendet, ein circRNA-Quantifizierungstool, das mit CIRI2 vorverpackt ist und die DE-Analyse von circRNAs nachweisen und durchführen kann. DE circRNAs werden auf der Grundlage eines Cut-off-Wertes von LogFC > |2| und FDR < 0,05, was dazu beiträgt, potenzielle Fehlalarme in nachgelagerten Analysen zu eliminieren. Die Charakterisierung von DE-circRNAs in Bezug auf den Annotationsstatus, die circRNA-Typen und die Anzahl der gestreckten Gene hilft bei der Kategorisierung und weiteren Filterung von circRNA-Kandidaten. Anschließend wird Circr37, ein circRNA-miRNA-Vorhersagewerkzeug, verwendet, um potenzielle miRNA-Schwammkandidaten vorherzusagen. Nach der Vorhersage potenzieller miRNAs als Ziele von circRNAs wird ein ceRNA-Netzwerk gezeichnet. Schließlich wird auf der Grundlage der elterlichen Gene von circRNAs das R clusterProfiler-Paket39 für die funktionelle Annotation über die GO- und KEGG-Signalweg-Anreicherungsanalyse verwendet. Die Ergebnisse von GO und KEGG könnten dazu beitragen, die biologischen Mechanismen zu entschlüsseln, die von circRNAs beeinflusst werden.
Bis heute wurden verschiedene circRNA-Vorhersagetools entwickelt, darunter CIRI2 43, CIRCexplorer2 44, find_circ 45, MapSplice 46 und UROBORUS 47. In einer von Hansen et al. durchgeführten Studie wird berichtet, dass CIRI2 eine hohe Gesamtleistung aufweist. Es gehört zu den wenigen circRNA-Nachweisinstrumenten, die im Hinblick auf die De-novo-Vorhersage und die Reduzierung der Identifizierung falsch positiver Ergebnisse gut funktionierenkönnen 48. In dieser Studie wurde daher CIRIquant verwendet, das CIRI2 für die circRNA-Detektion und -Quantifizierung verwendet. CIRIquant wurde verwendet, um die BSJ-Reads (Back Splice Junction) zu zählen, und die Zähldaten wurden auf die Reads normalisiert, die auf verwandte lineare RNAs abgebildet wurden, die von denselben Genloci transkribiert wurden. Dies ermöglicht die Quantifizierung von circRNAs in einer Probe. Um die differentielle Expression von circRNAs unter experimentellen Bedingungen zu bestimmen, implementierte CIRIquant ein verallgemeinertes lineares Modell in edgeR49 für die DE-Analyse, und der exakte Rate-Ratio-Test wurde als statistischer Test verwendet, um die Signifikanz des Unterschieds im circRNA-Übergangsverhältnis zu bestimmen. Obwohl andere circRNA-Quantifizierungsinstrumente wie CIRCexplorer3-CLEAR50 verwendet werden können, um das Expressionsniveau von circRNAs zu quantifizieren, erlaubt dieses Tool nur die circRNA-Quantifizierung in einer Probe, da es die BSJ-Lesevorgänge in einer Probe zählt und die Zähldaten mit den verwandten linearen RNA-Zählungen derselben Probe vergleicht. CIRCexplorer3-CLEAR kann circRNA-Expressionen unter experimentellen Bedingungen nicht vergleichen. Darüber hinaus ist in CIRCexplorer3-CLEAR kein statistisches Analysewerkzeug implementiert, um die quantifizierte Expressionsebene zu unterstützen. Obwohl das standardmäßige circRNA-Vorhersagewerkzeug, das in CIRIquant implementiert ist, CIRI2 ist, können die Vorhersageergebnisse von anderen Werkzeugen wie find_circ und CIRCexplorer2 auch für die Quantifizierung und DE-Analyse31 verwendet werden. In diesem Protokoll wurde nur ein circRNA-Vorhersagewerkzeug (CIRI2) für die Vorhersage verwendet, das immer noch falsch-positive circRNA-Kandidaten liefern könnte. Um falsch positive Ergebnisse zu reduzieren, kann man andere circRNA-Vorhersageinstrumente für die Analyse kombinieren und gemeinsame circRNAs auswählen, die unter den verschiedenen circRNA-Vorhersagewerkzeugen entdeckt wurden48,51. Um die circRNA-Detektion weiter zu verbessern, ist es ideal, RNA-Sequenzierungsdatensätze zu verwenden, die sowohl rRNA-depletiert als auch einer RNase R-Vorbehandlung unterzogen werden.
Je nach Ziel der Studie können de novo und annotierte DE circRNAs auf Basis der circBase-Datenbank separat identifiziert werden52. CirrcRNAs, die mehr als ein Gen umfassen, müssen jedoch häufig manuell auf UCSC oder einem anderen Genombrowser untersucht werden, um die Authentizität von circRNAs zu bestimmen und falsch positive Ergebnisse zu eliminieren. Nichtsdestotrotz wurden kürzlich auch circRNAs beschrieben, die mehr als ein Gen umfassen, wie z. B. circRNAs, die von Fusionsgenen abgeleitet sind53,54.
Circr kombiniert drei verschiedene miRNA-mRNA-Vorhersagealgorithmen, nämlich TargetScan55, miRanda 56 und RNAhybrid57, um die circRNA-miRNA-Bindungsstellen vorherzusagen. Darüber hinaus bezieht der Algorithmus auch Informationen über AGO-Peaks und zuvor validierte Wechselwirkungen in die circRNA-miRNA-Analyse ein. Hier wurden strenge Filterkriterien angewendet, um eine zuverlässigere circRNA-miRNA-Vorhersage zu erhalten und so die Zahl der falsch positiven Ergebnisse weiter zu reduzieren. Die Stringenz dieses Filterschritts kann jedoch je nach Benutzerpräferenz höher oder niedriger eingestellt werden.
ClusterProfiler ist ein gut dokumentiertes R-Paket, das Gensätze in verschiedenen Organismen funktionell annotieren kann. Neben den Funktionen innerhalb des R clusterProfiler-Pakets, die in diesem Protokoll erwähnt werden (enrichGO und enrichKEGG), die die Überrepräsentationsanalyse verwenden, gibt es auch andere Funktionen wie gseGO und gseKEGG, die verwendet werden können. Wenn clusterProfiler keine geeignete Wahl für den Workflow ist, gibt es auch andere Tools und Pakete wie den "AllEnricher"58 oder die websitebasierten Tools wie "Metascape"59, die eine Reihe von Genen funktional annotieren können. Obwohl die oben beschriebene Pipeline bei der Vorhersage potenzieller circRNAs und ihrer funktionellen Annotationen hilft, ist eine Nasslaborverifizierung erforderlich, um solide Beweise zu liefern.
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Der Autor dankt Tan Ke En und Dr. Cameron Bracken für die kritische Durchsicht dieses Manuskripts. Diese Arbeit wurde durch Zuschüsse aus dem Fundamental Research Grant Scheme (FRGS/1/2020/SKK0/UM/02/15) und dem University of Malaya High Impact Research Grant (UM. C/625/1/HIR/MOE/CHAN/02/07).
Materialien
| Name | Company | Catalog Number | Comments |
| Bedtools | GitHub | https://github.com/arq5x/bedtools2/ | Referring to section 4.1.2. Needed for Circr. |
| BWA | Burrows-Wheeler Aligner | http://bio-bwa.sourceforge.net/ | Referring to section 2.1.1 and 2.1.2. Needed to run CIRIquant, and to index the genome |
| Circr | GitHub | https://github.com/bicciatolab/Circr | Referring to section 4. Use to predict the miRNA binding sites |
| CIRIquant | GitHub | https://github.com/bioinfo-biols/CIRIquant | Referring to section 2.1.3. To predict circRNAs |
| Clusterprofiler | GitHub | https://github.com/YuLab-SMU/clusterProfiler | Referring to section 7. For GO and KEGG functional enrichment |
| CPU | Intel | Intel(R) Xeon(R) CPU E5-2620 V2 @ 2.10 GHz Cores: 6-core CPU Memory: 65 GB Graphics card: NVIDIA GK107GL (QUADRO K2000) | Specifications used to run this entire protocol. |
| Cytoscape | Cytoscape | https://cytoscape.org/download.html | Referring to section 5.2. Needed to plot ceRNA network |
| FastQC | Babraham Bioinformatics | https://www.bioinformatics.babraham.ac.uk/projects/fastqc/ | Referring to section 1.2.1. Quality checking on Fastq files |
| HISAT2 | http://daehwankimlab.github.io/hisat2/ | Referring to section 2.1.1 and 2.1.2. Needed to run CIRIquant, and to index the genome | |
| Linux | Ubuntu 20.04.5 LTS (Focal Fossa) | https://releases.ubuntu.com/focal/ | Needed to run the entire protocol. Other Ubuntu versions may still be valid to carry out the protocol. |
| miRanda | http://www.microrna.org/microrna/getDownloads.do | Referring to section 4.1.2. Needed for Circr | |
| Pybedtools | pybedtools 0.8.2 | https://pypi.org/project/pybedtools/ | Needed for BED file genomic manipulation |
| Python | Python 2.7 and 3.6 or abover | https://www.python.org/downloads/ | To run necessary library modules |
| R | The Comprehensive R Archive Network | https://cran.r-project.org/ | To manipulate dataframes |
| RNAhybrid | BiBiServ | https://bibiserv.cebitec.uni-bielefeld.de/rnahybrid | Referring to section 4.1.2. Needed for Circr |
| RStudio | RStudio | https://www.rstudio.com/ | A workspace to run R |
| samtools | SAMtools | http://www.htslib.org/ | Referring to section 2.1.2. Needed to run CIRIquant |
| StringTie | Johns Hopkins University: Center for Computational Biology | http://ccb.jhu.edu/software/stringtie/index.shtml | Referring to section 2.1.2. Needed to run CIRIquant |
| TargetScan | GitHub | https://github.com/nsoranzo/targetscan | Referring to section 4.1.2. Needed for Circr |
Referenzen
- Raman, K., Bhat, A. G., Chandra, N. A systems perspective of host-pathogen interactions: predicting disease outcome in tuberculosis. Molecular BioSystems. 6 (3), 516-530 (2010).
- Casadevall, A., Pirofski, L. A. Host-pathogen interactions: basic concepts of microbial commensalism, colonization, infection, and disease. Infection and Immunity. 68 (12), 6511-6518 (2000).
- Yang, E., Li, M. M. H. All About the RNA: Interferon-stimulated genes that interfere with viral RNA processes. Frontiers in Immunology. 11, 605024 (2020).
- Schneider, W. M., Chevillotte, M. D., Rice, C. M. Interferon-stimulated genes: A complex web of host defenses. Annual Review of Immunology. 32 (1), 513-545 (2014).
- Shirahama, S., Miki, A., Kaburaki, T., Akimitsu, N. Long non-coding RNAs involved in pathogenic infection. Frontiers in Genetics. 11, 454 (2020).
- Chandan, K., Gupta, M., Sarwat, M. Role of host and pathogen-derived microRNAs in immune regulation during infectious and inflammatory diseases. Frontiers in Immunology. 10, 3081 (2019).
- Chen, X., et al. Circular RNAs in immune responses and immune diseases. Theranostics. 9 (2), 588-607 (2019).
- Kristensen, L. S., et al. The biogenesis, biology and characterization of circular RNAs. Nature Reviews Genetics. 20 (11), 675-691 (2019).
- Ashwal-Fluss, R., et al. circRNA biogenesis competes with pre-mRNA splicing. Molecular Cell. 56 (1), 55-66 (2014).
- Conn, S. J., et al. The RNA binding protein quaking regulates formation of circRNAs. Cell. 160 (6), 1125-1134 (2015).
- Zhang, X. O., et al. Complementary sequence-mediated exon circularization. Cell. 159 (1), 134-147 (2014).
- Robic, A., Demars, J., Kuhn, C. In-depth analysis reveals production of circular RNAs from non-coding sequences. Cells. 9 (8), 1806 (2020).
- Eger, N., Schoppe, L., Schuster, S., Laufs, U., Boeckel, J. N. Circular RNA splicing. Advances in Experimental Medicine and Biology. 1087, 41-52 (2018).
- Barrett, S. P., Wang, P. L., Salzman, J. Circular RNA biogenesis can proceed through an exon-containing lariat precursor. eLife. 4, 07540 (2015).
- Memczak, S., et al. Circular RNAs are a large class of animal RNAs with regulatory potency. Nature. 495 (7441), 333-338 (2013).
- Misir, S., Wu, N., Yang, B. B. Specific expression and functions of circular RNAs. Cell Death and Differentiation. 29 (3), 481-491 (2022).
- Bai, S., et al. Construct a circRNA/miRNA/mRNA regulatory network to explore potential pathogenesis and therapy options of clear cell renal cell carcinoma. Scientific Reports. 10 (1), 13659 (2020).
- Sakshi, S., Jayasuriya, R., Ganesan, K., Xu, B., Ramkumar, K. M. Role of circRNA-miRNA-mRNA interaction network in diabetes and its associated complications. Molecular Therapy - Nucleic Acids. 26, 1291-1302 (2021).
- Hansen, T. B., et al. miRNA-dependent gene silencing involving Ago2-mediated cleavage of a circular antisense RNA. The EMBO Journal. 30 (21), 4414-4422 (2011).
- Lu, M. Circular RNA: functions, applications, and prospects. ExRNA. 2 (1), 15 (2020).
- Liu, K. S., Pan, F., Mao, X. D., Liu, C., Chen, Y. J. Biological functions of circular RNAs and their roles in occurrence of reproduction and gynecological diseases. American Journal of Translational Research. 11 (1), 1-15 (2019).
- Pamudurti, N. R., et al. Translation of CircRNAs. Molecular Cell. 66 (1), 9-21 (2017).
- Legnini, I., et al. Circ-ZNF609 Is a circular RNA that can be translated and functions in myogenesis. Molecular Cell. 66 (1), 22-37 (2017).
- Weigelt, C. M., et al. An insulin-sensitive circular RNA that regulates lifespan in Drosophila. Molecular Cell. 79 (2), 268-279 (2020).
- Guo, Y., et al. Identification and characterization of circular RNAs in the A549 cells following Influenza A virus infection. Veterinary Microbiology. 267, 109390 (2022).
- Qu, Z., et al. A novel intronic circular RNA antagonizes influenza virus by absorbing a microRNA that degrades CREBBP and accelerating IFN-β production. mBio. 12 (4), 0101721 (2021).
- Kawarada, Y., et al. TGF-β induces p53/Smads complex formation in the PAI-1 promoter to activate transcription. Scientific Reports. 6 (1), 35483 (2016).
- Yu, T., et al. Circular RNA GATAD2A promotes H1N1 replication through inhibiting autophagy. Veterinary Microbiology. 231, 238-245 (2019).
- FastQC: A quality control tool for high throughput sequence data. Available from: https://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (2010)
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Zhang, J., Chen, S., Yang, J., Zhao, F. Accurate quantification of circular RNAs identifies extensive circular isoform switching events. Nature Communications. 11 (1), 90 (2020).
- Li, H., Durbin, R. Fast and accurate long-read alignment with Burrows-Wheeler transform. Bioinformatics. 26 (5), 589-595 (2010).
- Kim, D., Paggi, J. M., Park, C., Bennett, C., Salzberg, S. L. Graph-based genome alignment and genotyping with HISAT2 and HISAT-genotype. Nature Biotechnology. 37 (8), 907-915 (2019).
- Pertea, M., et al. StringTie enables improved reconstruction of a transcriptome from RNA-seq reads. Nature Biotechnology. 33 (3), 290-295 (2015).
- Li, H., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), 2078-2079 (2009).
- Wang, L., Wang, S., Li, W. RSeQC: quality control of RNA-seq experiments. Bioinformatics. 28 (16), 2184-2185 (2012).
- Dori, M., Caroli, J., Forcato, M. Circr, a computational tool to identify miRNA:circRNA associations. Frontiers in Bioinformatics. 2, 852834 (2022).
- Shannon, P., et al. Cytoscape: a software environment for integrated models of biomolecular interaction networks. Genome Research. 13 (11), 2498-2504 (2003).
- Wu, T., et al. clusterProfiler 4.0: A universal enrichment tool for interpreting omics data. The Innovation. 2 (3), 100141 (2021).
- Yu, G., Wang, L. G., Han, Y., He, Q. Y. clusterProfiler: an R package for comparing biological themes among gene clusters. OMICS: A Journal of Integrative Biology. 16 (5), 284-287 (2012).
- . org.Hs.eg.db: Genome wide annotation for human. 2022. R package version 3.15.0 Available from: https://bioconductor.org/packages/release/data/annotation/html/org.Hs.eg.db.html (2022)
- Barrett, T., et al. NCBI GEO: archive for functional genomics data sets-update. Nucleic Acids Research. 41, 991-995 (2012).
- Gao, Y., Zhang, J., Zhao, F. Circular RNA identification based on multiple seed matching. Briefings in Bioinformatics. 19 (5), 803-810 (2018).
- Zhang, X. O., et al. Diverse alternative back-splicing and alternative splicing landscape of circular RNAs. Genome Research. 26 (9), 1277-1287 (2016).
- Memczak, S., et al. Circular RNAs are a large class of animal RNAs with regulatory potency. Nature. 495 (7441), 333-338 (2013).
- Wang, K., et al. MapSplice: Accurate mapping of RNA-seq reads for splice junction discovery. Nucleic Acids Research. 38 (18), 178 (2010).
- Song, X., et al. Circular RNA profile in gliomas revealed by identification tool UROBORUS. Nucleic Acids Research. 44 (9), 87 (2016).
- Hansen, T. B. Improved circRNA identification by combining prediction algorithms. Frontiers in Cell and Developmental Biology. 6, 20 (2018).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: A bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Ma, X. K., et al. CIRCexplorer3: A CLEAR pipeline for direct comparison of circular and linear RNA expression. Genomics Proteomics Bioinformatics. 17 (5), 511-521 (2019).
- Gaffo, E., Buratin, A., Dal Molin, A., Bortoluzzi, S. Sensitive, reliable and robust circRNA detection from RNA-seq with CirComPara2. Briefings in Bioinformatics. 23 (1), (2022).
- Glažar, P., Papavasileiou, P., Rajewsky, N. circBase: a database for circular RNAs. RNA. 20 (11), 1666-1670 (2014).
- Tan, S., et al. Circular RNA F-circEA-2a derived from EML4-ALK fusion gene promotes cell migration and invasion in non-small cell lung cancer. Molecular Cancer. 17 (1), 138 (2018).
- Guarnerio, J., et al. Oncogenic role of Fusion-circRNAs Derived from cancer-associated chromosomal translocations. Cell. 165 (2), 289-302 (2016).
- McGeary, S. E., et al. The biochemical basis of microRNA targeting efficacy. Science. 366 (6472), (2019).
- Enright, A. J., et al. MicroRNA targets in Drosophila. Genome Biology. 5 (1), 1 (2003).
- Rehmsmeier, M., Steffen, P., Hochsmann, M., Giegerich, R. Fast and effective prediction of microRNA/target duplexes. RNA. 10 (10), 1507-1517 (2004).
- Zhang, D., et al. AllEnricher: a comprehensive gene set function enrichment tool for both model and non-model species. BMC Bioinformatics. 21 (1), 106 (2020).
- Zhou, Y., et al. Metascape provides a biologist-oriented resource for the analysis of systems-level datasets. Nature Communications. 10 (1), 1523 (2019).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten