Method Article
脂質ナノ粒子を用いた化学修飾mRNAの哺乳動物細胞への導入
要約
このプロトコールは、化学的に修飾されたmRNAのin vitro転写(IVT)、カチオン性リポソーム調製、および哺乳動物細胞におけるリポソーム対応mRNAトランスフェクションの機能分析を示します。
要約
近年、化学修飾メッセンジャーRNA(mRNA)は、新規クラスのワクチン、タンパク質置換療法、免疫療法など、幅広い治療用途を開発するための強力な核酸分子として浮上しています。デリバリーベクターの中で、脂質ナノ粒子はRNA分子(siRNA、miRNA、mRNAなど)の送達においてより安全で効果的であることがわかっており、いくつかの製品はすでに臨床使用されています。脂質ナノ粒子を介したmRNAデリバリーを実証するために、機能的なme1Ψ-UTP修飾eGFP mRNAの合成、カチオン性リポソームの調製、カチオン性リポソームによるmRNAの静電複合体形成、および哺乳類細胞におけるトランスフェクション効率の評価に最適化されたプロトコルを提示します。この結果は、これらの修飾がカチオン性リポソームと共に送達されたmRNAの安定性を効率的に改善し、哺乳類細胞におけるeGFP mRNAの翻訳効率と安定性を増加させることを示しています。このプロトコルは、目的のmRNAを合成し、哺乳類細胞における標的遺伝子発現のためのカチオン性リポソームをトランスフェクションするために使用することができます。
概要
治療用分子としてのmRNAは、その非統合性およびプラスミドDNA(pDNA)1と比較して非有糸分裂細胞をトランスフェクションする能力により、いくつかの利点を提供します。mRNAの送達は1990年代初頭に実証されましたが、その安定性の欠如、免疫活性化の欠如、および翻訳効率の低さのために、治療への応用は限られていました2。最近同定された、mRNA上のシュードウリジン5'-三リン酸(Ψ-UTP)やメチルシュードウリジン5'-三リン酸(me1Ψ-UTP)などの化学修飾は、これらの制限を克服するのに役立ち、mRNA研究に革命をもたらし、ひいてはmRNAを基礎研究と応用研究の両方で有望なツールにしました。その適用範囲は、iPS細胞の作製からワクチン接種および遺伝子治療まで多岐にわたります3,4。
mRNA技術の進歩と並行して、非ウイルス性送達システムの大幅な進歩により、mRNAの送達が効果的になり、この技術は複数の治療用途に実現可能になりました5。非ウイルスベクターの中で、脂質ナノ粒子は核酸の送達に有効であることがわかっています6,7。最近、Alnylamは、遺伝性トランスサイレチン介在性アミロイドーシス(hATTRアミロイドーシス)に対するパティシランや急性肝ポルフィリン症(AHP)8に対するジボシランなど、肝疾患の治療薬として脂質ベースのsiRNA医薬品のFDA承認を取得しました。COVID19のパンデミック時には、ファイザー・ビオンテック社とモデルナ社の脂質カプセル化mRNAベースのワクチンがその有効性を実証し、FDAの承認を受けました9,10。したがって、脂質可能mRNAデリバリーは大きな治療可能性を秘めています。
ここでは、化学修飾されたin vitro転写eGFP mRNA、カチオン性リポソーム調製物、mRNA-脂質複合体の最適化、および哺乳動物細胞へのトランスフェクション(図1)の作製に関する詳細なプロトコールについて説明します。
プロトコル
1. me1 Ψ-UTP修飾mRNAの作製
- In vitro転写(IVT)DNAテンプレートの調製
注:IVT DNAテンプレート(T7プロモーター-遺伝子のオープンリーディングフレーム(ORF))調製のために、目的の遺伝子の遺伝子特異的プライマーセットを設計してください。遺伝子特異的フォワードプライマーの前にT7プロモーター(5'-NNNNNNTAATACGACTCACTATAGGGNNNNNNNN-3')配列を添加します。- 表1に記載されているPCR反応混合物を調製します。
注:IVT DNAテンプレートの濃度とIVTの品質を向上させるために、少なくとも4回のPCR反応を実行します。 - 反応混合物をマイクロピペットで完全に混合し、マイクロフュージを使用してスピンダウンします。
- 表2に示すPCRサイクリングプロトコルをサーモサイクラーで実行します。
- 表1に記載されているPCR反応混合物を調製します。
- 有機抽出/エタノール沈殿によるIVT DNAテンプレートの精製
- 1.5 mLのマイクロチューブ(ヌクレアーゼフリー)でDEPC処理水を使用して、増幅したPCR反応混合物を合計200 μLに調整します。
- 200 μLのTE飽和フェノール/クロロホルム(pH 8.0)を加えます。10秒間激しく渦巻きます。
- 12,000 x gで5分間遠心分離して相を分離し、水性上相(約200 μL)を新しい1.5 mLマイクロチューブに移します。
- 1/10( 20 μL) 容量の 3 M 酢酸ナトリウム (pH 5.5) と 2 容量 (400 μL) の 99-100% エタノールを加えます。よく混合し、-20°Cで少なくとも30分間インキュベートします。
- DNAテンプレートを12,000 x gで4°Cで15分間遠心分離してペレット化します。
- マイクロピペットを使用して、ペレットを乱さずに上清を完全に取り除きます。
- ペレットに0.5mLの75%エタノールを加え、5〜10回反転させます。
- 12,000 x g、4°Cで2分間遠心分離します。 次に、DNAペレットを乱さずにピペットでエタノールを完全に除去します。
- ペレットが少し半透明になるまで、ペレットを室温で乾燥させます。
- ヌクレアーゼフリーの水20μLを加え、数秒間完全に再懸濁します。
- 精製IVT DNAテンプレートの品質管理
- 定量化
- 精製したIVT DNAテンプレートの濃度と品質を、マイクロ分光光度計で測定します。
注:予想されるDNA濃度は約300-600 ng/μLです。IVT DNAテンプレートは-20°Cで長期間保存してください。
- 精製したIVT DNAテンプレートの濃度と品質を、マイクロ分光光度計で測定します。
- DNAアガロースゲル電気泳動
注:この実験は、精製されたIVT DNAテンプレートが正しいサイズであり、非特異的な製品の汚染がないかどうかを確認するためのものです。- 1%アガロースゲルを調製するには、0.5 gのアガロースと50 mLの1x TAEを円錐形フラスコに加えます。アガロースが完全に溶けるまで電子レンジで加熱します。アガロースを室温で5分間冷却します。
- 50 mLのアガロース溶液に1 μLの核酸染色剤(SafeView色素)を加えます。
- アガロース溶液をコーム付きのゲルキャスティングトレイに注ぎ、ゲルが固まるまで放置します。
- ゲルからコームを取り出し、ゲルを1x TAEバッファードタンクに保管します。
- 10 μL の 100-10000 bp DNA ラダーと 100-200 ng の PCR 精製テンプレート製品 2 μL の 6x DNA ローディングバッファーを混合して、総容量 12 μL にします。
- 各サンプルをそれぞれのウェルにロードし、100 Vで少なくとも45〜60分間運転します。
- ゲルドキュメンテーション装置でDNAバンドを可視化します(図2)。
- 定量化
- me1Ψ-UTP修飾RNAの合成
注:この実験を開始する前に、作業領域(層流)をDEPC処理水中の70%エタノールで洗浄する必要があります。滅菌ヌクレアーゼおよびエンドトキシンフリーの低保持チューブとフィルターバリアチップを使用してください。手袋をはめた手に70%エタノールを頻繁に塗布します。- 表3に示すIVT反応混合物を室温で0.2 mLチューブに調製し、マイクロピペットを使用して十分に混合します。
- チューブをマイクロフュージで10秒間回転させます。
- サーモサイクラーで37°Cで3時間インキュベートします。
- DNase 1処理によるIVT DNAテンプレートの分解
- 1 U/μL DNase 1 (RNase free) 1 μL を IVT 反応ミックスに加え、37 °C で 30 分間インキュベートします。
- 有機抽出/酢酸アンモニウム沈殿法によるRNAの精製
- IVT反応ミックスの容量を200 μLに調整し、179 μLのDEPC処理水を使用します。
- 200 μL の TE 飽和フェノール/クロロホルム pH 8.0 を添加します。10秒間渦巻きます。
- 12,000 x gで25°Cで5分間遠心分離し、2つの相を分離します。
- 水性上相(200 μL)を1.5 mLチューブに移し、200 μLの5 M酢酸アンモニウムを加えます。よく混合し、氷上で15分間インキュベートしてRNAを沈殿させます。
- 沈殿したRNAを12,000 x gで4°Cで15分間遠心分離してペレット化し、マイクロピペットで上清を完全に除去します。
- 70%エタノールを使用してRNAペレットを洗浄し、5〜10回反転させます。12,000 x gで4°Cで5分間遠心分離します。
- RNAペレットを乱さずにマイクロピペットで上清を完全に取り除きます。
- ペレットが半透明になるまで、ペレットを室温で乾燥させます。次に、ペレットを60〜75 μLのRNaseフリー水に再懸濁します。
- 精製RNAの品質管理
- 定量化
- 精製されたRNAの濃度と品質をマイクロ分光光度計で測定します。
注:予想されるRNA収量は、目的の遺伝子のサイズとIVT DNAテンプレートの品質に応じて、反応ごとに未修飾RNAで140〜180μg、me1Ψ-UTP修飾RNAで100〜150μgになります。最適な品質は、OD260/OD280 の比率が約 1.9-2.0 で、OD260/OD230 の比率が >2.0 です。RNAを-20°Cで短時間保存します。
- 精製されたRNAの濃度と品質をマイクロ分光光度計で測定します。
- 変性RNAアガロースゲル電気泳動
注:この実験は、合成されたRNAが正しい長さであり、IVT副産物の汚染がないかどうかを確認するために行われます。- 1%アガロースゲルを調製するには、0.5 gのアガロースと50 mLの1x TAEを円錐形フラスコに加えます。アガロースが完全に溶解するまで溶液を電子レンジで加熱します。アガロース溶液を室温で5分間保冷します。
- 1%アガロース溶液50 mLに対して1 μLの核酸染色液を添加します。
- アガロース溶液をコームでゲルキャスティングトレイに注ぎ、ゲルが固まるまで放置します。
- ゲルからコームを取り出し、ゲルを1x TAEバッファードタンクに保管します。
- 表4に記載されているように、RNAローディング色素サンプルを調製します。
- サンプルを65°Cで10分間加熱し、サンプルを氷上に保ちます。
- 各サンプルをそれぞれのウェルにロードし、100 Vで少なくとも45〜60分間運転します。
- ゲルドキュメンテーション装置でRNAバンドを可視化します(図3)。
- 定量化
- 酵素ベースキャッピングとpoly-Aテーリングによるme1Ψ-UTP修飾mRNAの合成
注:酵素法を使用したIVT RNAのキャッピング効率は100%です。したがって、このプロトコルでは、mRNA合成におけるCap-1の酵素キャッピングを使用しました。mRNAの翻訳効率を改善するために、分子あたり>150A塩基の長さでpoly-Aテールを添加しました。- 精製したIVT RNAを55〜60 μg加え、1.5 mLチューブにRNaseフリーウォーターを入れて最大72 μLにします。
- RNAを65°Cでサーモミキサーで10分間変性させた後、すぐにチューブを氷上に5分間置きます。
- その間、 表5に示すようにキャッピング反応混合物を調製する。
- 変性したRNAにキャッピング反応混合物と4μLのキャッピング酵素を加え、マイクロピペットでよく混合します。チューブをマイクロフュージで10秒間回転させます。
- 反応混合物を37°Cで2時間インキュベートします。
- 2時間後、チューブを氷上に置き、 表6に示すようにPoly Aテーリングマスターミックスを調製します。
- キャップ付きRNA溶液にPoly Aテーリングマスターミックスを加え、マイクロピペットでよく混合します。チューブをマイクロフュージで10秒間回転させます。
- 反応混合物を37°Cで2時間インキュベートします。
注:mRNAはさらに精製にかけることも、粗mRNAを-20°Cで一晩保存することもできます。
- IVT mRNA精製
- プロトコールセクション1.6に記載されているように、有機抽出/酢酸アンモニウム沈殿法によりmRNAを精製します。
- mRNAペレットを60 μLのRNaseフリー水で再懸濁します。
- 精製されたmRNAの品質管理
- プロトコルセクション1.7で説明されている品質管理プロトコルに従ってください。
注:mRNAバンドは、変性RNAアガロースゲル電気泳動にPoly-Aテーリングが付加されるため、RNAバンドの上に表示されるはずです(図3)。また、Poly-AテーリングはmRNAの収量を増加させます(RNA濃度>必要があります)。定量後、複数の mRNA アリコートを 1 μg/μL 濃度で入れ、直ちに -80 °C で保存します。 合成されたmRNAの分解を防ぐために、RNAの複数回の凍結融解サイクルを避けてください。
- プロトコルセクション1.7で説明されている品質管理プロトコルに従ってください。
2. カチオン性リポソームの調製とin vitro mRNAトランスフェクション特性の評価
- リポソーム調製
- 1 mMカチオン性リポソームの調製には、カチオン性脂質:DOPE:コレステロールをモル比1:1:0.5で使用します。
- カチオン性脂質、コレステロール、およびDOPE(1,2-ジオレオイル-sn-グリセロール-3-ホスホエタノールアミン)の適切なモル比をガラスバイアル中のクロロホルム(200μL)に溶解します。
- 溶媒除去には、水分を含まない窒素ガスの細い流れを使用してください。
- 乾燥した脂質を高真空下で保存し、さらに2時間乾燥させます。
- 真空乾燥後、乾燥脂質に滅菌脱イオン水1mLを加え、混合物を一晩膨潤させます。
- バイアルを室温でボルテックスして、マルチユニラメラベシクル(MUV)を作成します。
- バス超音波処理とそれに続く25 W電力でのプローブ超音波処理を使用して、マルチユニラメラベシクル(MUV)から小さなユニラメラベシクル(SUV)を作成します。
注:SUVは半透明のリポソーム溶液のように見える必要があります。そうでない場合は、30秒のパルスのオンとオフの数を1分間隔で増やします。流体力学的直径と表面電位は、粒度分布分析装置で測定されます(図4)。
- mRNA/リポソーム複合体形成とゲル遅延アッセイ
- 1:1 から 8:1 までの異なる脂質-RNA 電荷比を調製するには、 表 7 に示すように、mRNA とカチオン性リポソームを脱イオン水で別々に希釈します。
- 表7に示すように、希釈したmRNAをリポソーム溶液に混合し、室温で10分間インキュベートしてリポプレックスを形成します。
- 20 μLの2x RNAローディング色素を複合体に加え、ウェルにロードします。mRNAのみでコントロールとして機能します。
- サンプルを1x TAEバッファー中の1%アガロースゲルにロードし、100 Vで45分間分析します。
- ゲルドキュメンテーション装置でRNAバンドを可視化します(図5)。
- in vitro mRNAトランスフェクション
- 48ウェルプレートのウェルあたり45,000個の哺乳動物細胞を完全培地に播種し、37°Cで16〜20時間のトランスフェクションをインキュベートします。
- 16〜20時間後、細胞密度を確認します。トランスフェクション時には、細胞のコンフルエントは約80%である必要があります。
- 0.5 mLチューブに、カチオン性リポソームとmRNAの電荷比が1:1のGFPタンパク質コードmRNA複合体を、血清を含まないDMEM培地に150 ngを加えます。総容量は最大20μLになります。
- 室温で10分間インキュベートします。
- リポプレックスを細胞に加え、37°Cおよび5%CO2 インキュベーターで4時間インキュベートします。
- 細胞を乱さずにメディアを取り出します。250 μL の完全培地 (10% FBS) (表 8) を各ウェルに加えます。
- トランスフェクションの72時間後、蛍光顕微鏡でGFP発現を観察します(図6、7)。
- GFP発現を定量するには、フローサイトメーター分析のために細胞を解析します。
- メディアを取り出し、1x PBSで2回洗浄します。細胞をトリプシン化し、細胞を処理して、フローサイトメーターでGFP陽性細胞の割合を定量化します。
- Laser 488を使用してフローサイトメーターで細胞を採取します。そこから生きた集団をゲートし、GFP陽性細胞の割合を分析します。平均蛍光強度(MFI)を定量化します(図6、7)。
結果
me1Ψ-UTP修飾mRNA産生、リポソーム調製、およびカチオン性リポソームを用いた複数の哺乳類細胞へのmRNAトランスフェクション実験のプロトコールを最適化しました(図1)。mRNAを合成するために、哺乳類コドン最適化eGFP IVTテンプレートをmEGFP-N1哺乳類発現ベクターから増幅し、有機抽出/エタノール沈殿法で精製しました(図2)。その後、me1Ψ-UTP修飾RNAとmRNAがIVTプロセスによって作製されました。変性RNAアガロースゲル電気泳動データにより、これらの合成RNAは良好な完全性と正しい長さ(RNAラダーに対して750塩基RNA、~1000塩基mRNA)を持つことが示されました(図3)。
カチオン性リポソームを調製するために、薄膜水和法および超音波処理を用いて、小さな単層小胞(SUV)を形成した。リポソームの物理化学的特性評価により、流体力学的直径は約65 nmで観察され、表面電位は約+20 meVであることが明らかになりました(図4)。ゲル遅延アッセイは、脂質/塩基の電荷比が1:1から8:1までのさまざまな電荷比でリポソーム-mRNA複合体を用いて行われました。カチオン性リポソームは、1:1の電荷比でもmRNAに対して高い結合効率を示しました(図5)。したがって、mRNAトランスフェクション実験には1:1の電荷比を使用しました。リポソームを介したeGFP mRNAトランスフェクション実験をHEK-293TおよびNIH/3T3細胞株で行いました。eGFPの発現をフローサイトメトリーで解析しました。ME1Ψ-UTP修飾mRNAは、トランスフェクション後3日目にHEK-293TおよびNIH/3T3細胞において、未修飾mRNAと比較して、優れた安定したeGFPタンパク質発現を示しました(図6、7)。
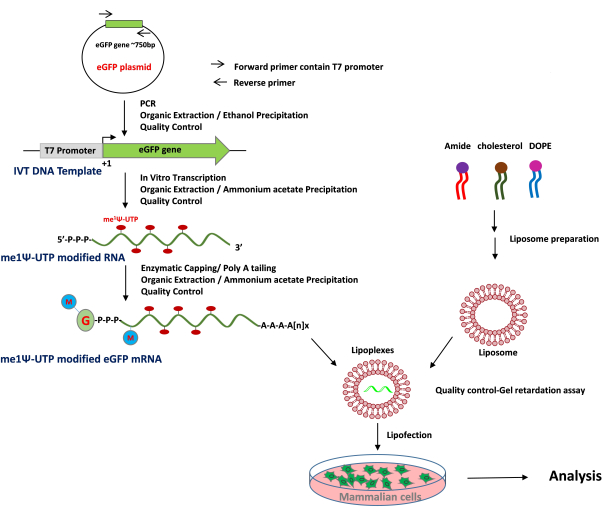
図1:me1Ψ-UTP修飾mRNA産生、リポソーム調製、トランスフェクションプロトコルの概略図。 IVT DNAテンプレート(T7 promoter-Gene ORF)をPCRで増幅し、精製します。me1Ψ-UTP修飾mRNAは、IVT DNAテンプレートを用いてIVTプロセスで作製し、精製します。カチオン性リポソームは調製され、me1Ψ-UTP修飾mRNA(Lipoplex)と複合体を形成し、哺乳動物細胞にトランスフェクションすることができます。 この図の拡大版を表示するには、ここをクリックしてください。

図2:アガロースゲル電気泳動におけるIVT DNAテンプレートの品質測定。 精製したeGFP IVT DNAテンプレートを1%アガロースゲル上で実行し、ゲルで可視化しました。 この図の拡大版を表示するには、ここをクリックしてください。

図3:アガロースゲル中での変性RNA電気泳動によるme1Ψ-UTP修飾IVT RNAのサイズと品質の解析。 精製したme1Ψ-UTP修飾RNAを変性させて1%TAEアガロースゲルにロードし、RNAのサイズと品質をゲルで決定しました。レーン1:ssRNAラダー、レーン2:me1Ψ-UTP修飾IVT RNA、レーン3:Cap1およびPoly Aテールを持つme1Ψ-UTP修飾IVT RNA。この図の拡大版を表示するには、ここをクリックしてください。
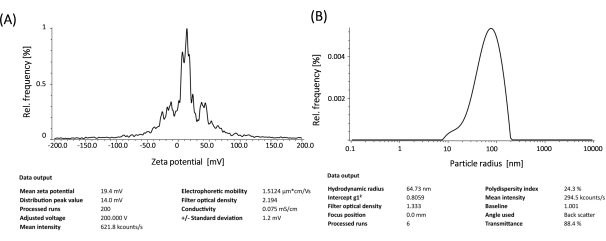
図4:リポソームの物理化学的特性評価:表面電位(A)と流体力学的直径(B)この図の拡大版を表示するには、ここをクリックしてください。
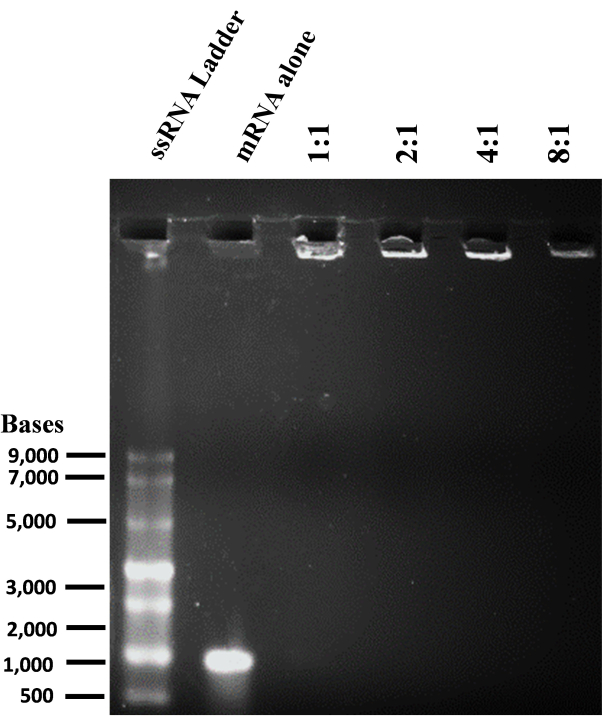
図5:リポソームのmRNA結合能は、変性アガロースゲル遅延アッセイによって決定されました。 リポソーム-mRNA複合体(Lipoplexes)を異なる脂質/塩基電荷比で調製し、1%アガロースゲルおよび文書化されたゲルにロードしました。 この図の拡大版を表示するには、ここをクリックしてください。

図6:ヒトHEK-293T細胞におけるme1Ψ-UTP改変eGFP mRNAのタンパク質発現効率。 (A)トランスフェクション後3日目(倍率100倍)に得られた、未修飾eGFP mRNAおよびカチオン性リポソームをHEK-293T細胞にトランスフェクションしたme1Ψ-UTP修飾eGFP mRNAの蛍光画像。eGFPタンパク質発現率(B)および平均蛍光強度(MFI)の割合。(C)トランスフェクションした細胞をフローサイトメトリーを用いて解析した。(N=3)この図の拡大版を表示するには、ここをクリックしてください。
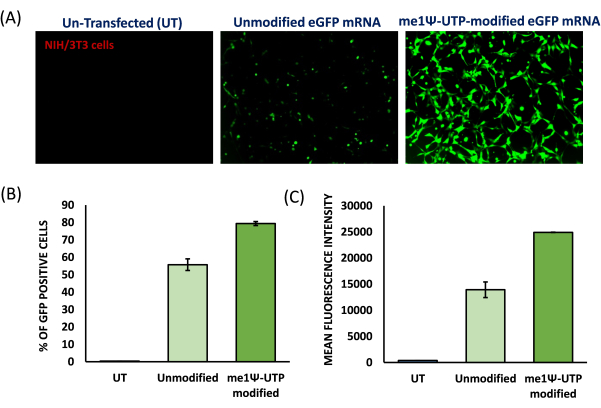
図7:マウスNIH/3T3細胞におけるme1Ψ-UTP修飾eGFP mRNAの翻訳効率。 (A)トランスフェクション後3日目(100倍倍)に得られたNIH/3T3細胞にトランスフェクションした未修飾eGFP mRNAおよびme1Ψ-UTP修飾eGFP mRNAの蛍光画像。eGFPタンパク質発現率(B)および平均蛍光強度(MFI)の割合。(C)トランスフェクションした細胞をフローサイトメトリーを用いて解析した。(N=3)この図の拡大版を表示するには、ここをクリックしてください。
| コンポーネント | 25 μLの反応 | 最終濃度 |
| 5x Q5バッファ | 5 μL | 1倍 |
| 10 mM dNTP | 0.5μL | 200 μM |
| 10 μM フォワードプライマー | 1.25μL | 0.5 μM |
| 10 μM リバースプライマー | 1.25μL | 0.5 μM |
| Q5ポリメラーゼ | 0.25 μL | 0.02 U/μL |
| プラスミドの目的遺伝子(テンプレート) | 1-5 ng | 変数 |
| ヌクレアーゼフリー水 | 25μLまで |
表1:PCR反応混合物の調製
| ステップス | 期間 | 温度 | サイクル番号 |
| 初期変性 | 30秒 | 98°C | 1 |
| 変性 | 10秒 | 98°C | |
| アニーリング | 20秒 | 変数 | 18-25 |
| 延長 | 変数 | 72°C | |
| 最終的な延長 | 2分間 | 72°C | 1 |
| 持つ | ∞ | 1 5°C |
表2:PCRサイクリング条件
| コンポーネント | 非修飾RNA | me1Ψ-UTP修飾RNA |
| RNaseフリーウォーター | 変数 | 変数 |
| T7 RNAPプロモーターによる直鎖状テンプレートDNA | 変数 (1 μg) | 変数 (1 μg) |
| 10x T7 転写バッファー | 2 μL | 2 μL |
| 100 mM N1-メチルシュードウリジン | - | 1.5 μL |
| 100 mM ATP | 1.8μL | 1.8μL |
| 100 mM UTP | 1.8μL | - |
| 100 mM CTP | 1.8μL | 1.8μL |
| 100 mM GTPの | 1.8μL | 1.8μL |
| 100 mM DTT | 2 μL | 2 μL |
| 40 U/μL RNase阻害剤 | 0.5μL | 0.5μL |
| T7酵素溶液 | 2 μL | 2 μL |
| 総反応量 | 20μL | 20μL |
表3:IVT反応混合物の調製
| コンポーネント | RNAラダー | RNAサンプル |
| 2x RNAローディング色素(NEB) | 6 μL | 6 μL |
| RNA | 2 μL | 1 μL (0.5-1 μg ) |
| DEPC 処理水 | 4 μL | 5 μL |
| 総ボリューム | 12 μL | 12 μL |
表4:RNAローディング色素調製
| コンポーネント | 量 |
| 10xキャッピングバッファ | 10 μL |
| 20 mM GTP | 5 μL |
| 20 mM SAM | 2.5 μL |
| RNase阻害剤 | 2.5 μL |
| 2'-O-メチルトランスフェラーゼ | 4 μL |
| 総ボリューム | 24μL |
表5:酵素Cap-1合成反応混合物
| コンポーネント | 量 |
| 5'キャップIVT RNA | 100μL |
| RNase阻害剤 | 0.5μL |
| 10x Aプラステーリングバッファー | 12 μL |
| 20 mM ATP | 6 μL |
| 4 U/μL A-Plus ポリ(A)ポリメラーゼ | 5 μL |
| 総ボリューム | 123.5μL |
表6:ポリAテーリング反応混合物
| 電荷率 | リポソーム | DI水 | mRNA(500ng) | DI水 |
| 1:1 | 1.5 μL | 8.5 μL | 1 μL | 9 μL |
| 2:1 | 3 μL | 7 μL | 1 μL | 9 μL |
| 4:1 | 6 μL | 4 μL | 1 μL | 9 μL |
| 8:1 | 12 μL | - | 1 μL | 9 μL |
| 総容量 (20 μL) | 10 μL | 10 μL | ||
表7:電荷比に基づくリポプレックスの調製
| 名前 | コンポーネント |
| 50x TAEバッファ | 50 mM EDTAナトリウム塩、2 Mトリス、1 M氷酢酸を1 Lの水に溶解します |
| HEK-293TおよびNIH/3T3細胞培養培地 | 4.5 g/L グルコース、L-グルタミン、1% ペニシリン/ストレプトマイシン、10% FBS を含む DMEM |
表8:バッファーと培地の調製
ディスカッション
未修飾mRNAの治療応用は、半減期が短いことや細胞内自然免疫応答を活性化する能力があるため、トランスフェクションされた細胞でのタンパク質発現の低下につながるため、限定的でした11。Katalinらは、m5C、m6A、ΨU、me1Ψ-UTPなどの修飾ヌクレオシドを含むRNAがTLR活性化を回避できることを実証しました12。さらに重要なことに、IVT mRNAへのΨUまたはme1Ψ-UTPの取り込みは、標的タンパク質の優れた翻訳効率、室温での安定性の向上、およびヌクレアーゼからの分解の防止を示しました13、14。
このビデオでは、脂質対応me1Ψ-UTP修飾mRNAを複数の培養細胞に導入するためのプロトコールを実演しました。このプロトコールには、me1Ψ-UTP修飾mRNAの産生、カチオン性リポソーム調製、細胞へのトランスフェクション、およびタンパク質発現の評価が含まれます。哺乳類のコドン最適化eGFPレポーター遺伝子をトランスフェクション実験に用い、蛍光強度を測定することでタンパク質の発現量を解析しました。カチオン性リポソームはmRNAを複合体化するために調製され、それらの静電錯体化は、1:1から8:1までのさまざまな脂質/塩基電荷比で分析されました。1:1では、カチオン性リポソームはmRNAを完全に複雑にする可能性があるため、トランスフェクションには1:1の電荷比を使用しました。私たちは、カチオン性リポソームを用いたmRNAのトランスフェクションが、HEK-293T細胞では90%のトランスフェクション効率で修飾および非修飾のeGFP mRNAを効率的に送達できることを実証しました。一方、NIH/373細胞では修飾mRNAで80%、非修飾mRNAで60%の効率を示しました。さらに重要なことに、me1Ψ-UTP修飾mRNAは、非修飾mRNAと比較して、哺乳類細胞で3日間優れたeGFPタンパク質発現を示しました(HEK-293Tでは>倍、NIH/3T3細胞では>倍)。これらの研究は、mRNA上のme1Ψ-UTPの修飾が、哺乳類細胞におけるmRNAの翻訳と安定性を向上させる可能性があることを実証しました。
カチオン性リポソームのトランスフェクション効率と合成されたmRNAの翻訳効率は、細胞の種類によって異なります。したがって、異なる細胞タイプごとにmRNA濃度を最適化することが重要です。このプロトコールを使用して、機能的なme1Ψ-UTP修飾mRNAを最大6 kbのサイズで合成しましたが、IVT DNAテンプレートの濃度と時間を最適化して、良好なmRNA収量と正しい長さを得ることができました。
開示事項
開示なし
謝辞
MSは、財政支援(BT/PR25841/GET/119/162/2017)、VelloreのCSCR責任者であるAlok Srivastava博士、イメージングおよびFACS実験のためのCSCRコア施設であるSandhya博士に感謝します。リポソームの物理化学的データの分析に協力してくださった CSIR-Indian Institute of Chemical Technology Uppal Road, Tarnaka, Hyderabad, 500 007, TS, India の応用生物学部門の R. Harikrishna Reddy 氏と Rajkumar Banerjee 氏に感謝します。Vigneshwaran V、およびJoshua A、CSCRは、ビデオ制作の支援に対して。
資料
| Name | Company | Catalog Number | Comments |
| Agarose | Lonza | 50004 | |
| Bath sonicator | DNMANM Industries | USC-100 | |
| Cationic lipid | Synthesized in the lab | ||
| Chlorofrom | MP biomedicals | 67-66-3 | "Caution" |
| Cholesterol | Himedia | GRM335 | |
| DEPC water | SRL BioLit | 66886 | |
| DMEM | Lonza | 12-604F | |
| DNA Ladder | GeneDireX | DM010-R50C | |
| DOPE | TCI | D4251 | |
| EDTA sodium salt | MP biomedicals | 194822 | |
| Ethanol | Hayman | F204325 | "Caution" |
| Fetal bovine serum | Gibco | 10270 | |
| Flow cytometry | BD | FACS Celesta | |
| Fluroscence Microscope | Leica | MI6000B | |
| Gel documentation system | Cell Biosciences | Flurochem E | |
| Glacial acetic acid | Fisher Scientific | 85801 | "Caution" |
| mEGFP-N1, Mammalian expression vector | Addgene | 54767 | |
| N1-Methylpseudo-UTP | Jena Bioscience | NU-890 | |
| Phenol:chloroform:isoamyl alchol (25:24:1), pH 8.0 | SRL BioLit | 136112-00-0 | "Caution" |
| Phosphate Buffer Saline (PBS), pH 7.4 | CellClone | CC3041 | |
| Probe sonicator | Sonics Vibra Cells | VCX130 | |
| RNA ladder | NEB | N0362S | |
| RNase inhibitor | Thermo Scientific | N8080119 | |
| SafeView dye | abm | G108 | |
| Sodium acetate | Sigma | S7545 | |
| Thermocycler | Applied biosystems | 4375786 | |
| Thermomixer | Eppendrof | 22331 | |
| Tris buffer | SRL BioLit | 71033 | |
| Trypsin | Gibco | 25200056 |
参考文献
- Sahin, U., Kariko, K., Tureci, O. mRNA-based therapeutics--developing a new class of drugs. Nature Reviews Drug Discovery. 13 (10), 759-780 (2014).
- Schlake, T., Thess, A., Fotin-Mleczek, M., Kallen, K. J. Developing mRNA-vaccine technologies. RNA Biology. 9 (11), 1319-1330 (2012).
- Carlile, T. M., et al. Pseudouridine profiling reveals regulated mRNA pseudouridylation in yeast and human cells. Nature. 515 (7525), 143-146 (2014).
- Kariko, K., Buckstein, M., Ni, H., Weissman, D. Suppression of RNA recognition by Toll-like receptors: the impact of nucleoside modification and the evolutionary origin of RNA. Immunity. 23 (2), 165-175 (2005).
- Guan, S., Rosenecker, J. Nanotechnologies in delivery of mRNA therapeutics using nonviral vector-based delivery systems. Gene Therapy. 24 (3), 133-143 (2017).
- Srujan, M., et al. The influence of the structural orientation of amide linkers on the serum compatibility and lung transfection properties of cationic amphiphiles. Biomaterials. 32 (22), 5231-5240 (2011).
- Dharmalingam, P., et al. Transfection: Cationic Lipid Nanocarrier System Derivatized from Vegetable Fat, Palmstearin Enhances Nucleic Acid Transfections. ACS Omega. 2 (11), 7892-7903 (2017).
- Hoy, S. M. Patisiran: First Global Approval. Drugs. 78 (15), 1625-1631 (2018).
- Anderson, E. J., et al. Safety and Immunogenicity of SARS-CoV-2 mRNA-1273 Vaccine in Older Adults. New England Journal of Medicine. , (2020).
- Polack, F. P., et al. Safety and Efficacy of the BNT162b2 mRNA Covid-19 Vaccine. New England Journal of Medicine. , (2020).
- Schlee, M., Hartmann, G. Discriminating self from non-self in nucleic acid-sensing. Nature Reviews Immunology. 16 (9), 566-580 (2016).
- Kariko, K., Buckstein, M., Hi, H., Weissman, D. Suppression of RNA recognition by Toll-like receptors: The impact of nucleoside modification and evolutionary origin of RNA. Immunity. 23 (2), 165-175 (2005).
- Mauger, D. M., et al. mRNA structure regulates protein expression through changes in functional half-life. Proceedings of the National Academy of Sciences of the United States of America. 116 (48), 24075-24083 (2019).
- Vaidyanathan, S., et al. Uridine Depletion and Chemical Modification Increase Cas9 mRNA Activity and Reduce Immunogenicity without HPLC Purification. Molecular Therapy - Nucleic Acids. 12, 530-542 (2018).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved