Method Article
腫瘍微小環境における免疫細胞集団の可視化、定量化、およびマッピング
要約
ここでは、ホルマリン固定パラフィン埋め込み腫瘍組織切片における免疫細胞の可視化、定量、およびマッピングのための簡単でアクセス可能な戦略について述べている。この方法論は、既存のイメージングおよびデジタル解析技術と、イメージングアッセイの多重化能力と多パラメータ分析を拡大することを目的と組み合わせたものです。
要約
腫瘍微小環境(TME)の免疫景観は、がんの進行および治療に対する応答の決定因子である。具体的には、TMEにおける免疫細胞の密度と位置は、重要な診断および予後値を有する。TMEの多元的プロファイリングは、腫瘍の開始と進行を調節する多数の細胞および分子ネットワークに対する我々の理解を指数関数的に増加させてきた。ただし、これらの手法では、セルの空間的な構成やセルとの相互作用に関する情報は提供されません。単一細胞ベースのハイスループット技術を補完するために、組織切片内の免疫細胞の空間分解能を可能にする、手頃な価格でアクセスしやすく、簡単に多重化技術が必要です。ここでは、シリアルイメージング、シーケンシャルラベリング、画像アライメントを統合して、組織セクション全体の仮想マルチパラメータスライドを生成する戦略を説明します。仮想スライドは、ユーザー定義のプロトコルを使用して、対象となる細胞集団の識別、定量、マッピングを可能にする、自動化された方法で分析されます。画像解析は、この場合、組織整列、作成者、および HISTOmap 解析モジュールを使用して行われます。この戦略を1つの臨床標本にうまく適用し、限られた組織サンプルから得られる情報を最大化し、組織セクション全体でTMEの公平な見解を提供する例を紹介します。
概要
がんの発症は、悪性細胞とTMEとの間の相互相互作用を含む多段階のプロセスの結果である。腫瘍細胞以外に、TMEは、非悪性細胞、間質細胞、免疫細胞集団、および細胞外マトリックス(ECM)1から構成される。1腫瘍組織の異なる細胞および構造成分の空間的構成と癌と隣接する非癌細胞との動的交換は、最終的に腫瘍の進行と応答を調節する2,3,,4.癌における免疫応答は、時空間的に調節される55,66が示されている。腫瘍性病変および隣接組織に浸透する異なる免疫細胞集団は、独特の空間分布パターンおよび異なる機能に関連する多様な活性化および分化状態(例えば、プロ対抗腫瘍)を示す。これらの異なる免疫集団およびそれらのパラメータは、腫瘍および間質コンパートメントと残業を共存させる。
単一細胞マルチオミクスプロファイリングを可能にする技術の出現は、発がんおよび腫瘍進行を調節する多数の細胞および分子ネットワークに対する我々の理解を指数関数的に増加させてきた。しかし、ほとんどの単一細胞ベースのハイスループット分析ツールは、組織の破壊と単一細胞の単一の分離を必要とし、細胞の空間組織と細胞と細胞の相互作用に関する情報を失う結果 7.TME内の特定の免疫細胞の位置と配置は診断および予後の価値を有するため、空間分解能を可能にする技術は、単一細胞ベースの免疫プロファイリング技術の不可欠な補完である。
従来、免疫体化学(IHC)や多重免疫蛍光(mIF)などのイメージング技術は、同時に可視化できる少数のバイオマーカーに制限されてきました。この制限は、腫瘍浸潤免疫細胞の時空間的ダイナミクスの研究を妨げているが、これは典型的には複数の表向きのマーカーによって定義される。近年、イメージングや解析ツールの進歩により、多重化の可能性が広がっています。ヒストサイトメトリーやイメージング質量サイトメトリーなどの新しい抗体ベースの標識技術は、それぞれ88,99個のバイオマーカーをそれぞれ12個と32個まで空間的に分離するために用いられている。質量分析法イメージングは、標識を必要としない技術であり、1つの組織セクション10、11,11において数千のバイオマーカーを同時に画像化する可能性を有する。これらの技術は、すでに癌の組織免疫景観を解剖するための大きな可能性を示しているが、彼らは非常に洗練された高価な機器やソフトウェアを使用し、研究者の大半に容易にアクセスすることはできません。
あるいは、従来のIHCおよびmIFの多重化能力は、シリアルイメージング、シーケンシャルなラベ,リング、およびスペクトルイメージング7、12、13、14、15、1612,13を使用して拡張された。14,15,167これらの技術は、同じまたはシリアル組織セクションから複数の画像を生成し、画像解析ソフトウェアを使用して仮想マルチパラメータースライドに統合することができます。その結果、同時に視覚化および解析できるマーカーの数が増加します。
ここでは、市販の試薬、手頃な顕微鏡装置、およびユーザーフレンドリーなソフトウェアを用いた組織多重アッセイの合理的設計戦略を提案する(図1)。この方法論は、シリアルイメージング、シーケンシャルマルチプレックスラベリング、組織全体のイメージング、組織アライメントを統合して、組織切片内の免疫細胞の自動定量とマッピングに使用できる仮想マルチパラメータスライドを生成します。この戦略を用いて、11個のバイオマーカーと2つの頻繁に使用される組織学的ステイン(ヘマトキシリンとエオシン(H&E)とピクロシリウスレッド(PSR)からなる1つの仮想スライドを作成しました。複数の免疫細胞集団を同定し、異なる組織区画内で同定し、同定し、定量化し、組織ヒートマップを用いてそれらの空間分布を解決した。この戦略は、限られた臨床検体から得ることができる情報を最大化し、組織全体、コアニードルバイオプシー、および組織マイクロアレイを含むホルマリン固定パラフィン埋め込み(FFPE)アーカイブ組織サンプルに適用可能です。この方法論を、TMEにおける免疫細胞集団の同定、定量、マッピングのためのカスタムアッセイを設計するための有用なガイドとして提案する。
プロトコル
切除されたB型肝炎ウイルス(HBV)関連ヒト肝細胞癌の3つの連続FFPEセクションは、モントリオール大学(CHUM)肝突栓性腫瘍癌臨床データベースおよび生物学的標本から得られたリポジトリ(HBPバイオバンク)。この組織バンクに参加している患者は、インフォームド・コンセントを提供した。この研究は、機関倫理委員会(プロトコル番号09.237)によって承認され、ヘルシンキ宣言に従って行われました。
1. ヘマトキシリンおよびエオシン(H&E)染色プロトコル
注:H&E染色は、以下のプログラムを使用してシャンドンマルチプログラムロボットスライド染色剤を使用して、モントリオール大学(CRCHUM)センター・ド・ル・センター・ホスピタル・ド・ル・レシェルの分子病理学コア施設によって行われました。
- 脱パラフィンの場合、スライド3xを2.5分間それぞれキシレンの代用に浸漬します。
注意:キシレンの代替品は、可燃性、皮膚刺激物、および吸入すると有害である。 - 水分補給のために、100%エタノール3xにそれぞれ2.5分間浸漬スライド。二重蒸留水(ddH2O)で1分間洗浄し、水分補給を行います。
- ヘマトキシリンで1分間インキュベートする。3倍の3倍をddH2Oでそれぞれ1分間洗います。
- 5 sをエオシンでインキュベートする。95%エタノールで30sを洗います。100%エタノールで2倍の1分間洗浄します。
注意:エタノールは可燃性であり、目の刺激性です。エオシンは眼刺激物である。 - 脱水の場合は、3倍の1.5分間、それぞれをキシレンの代用物に浸します。手動でスライドをマウントします。
注: プロトコルのこの部分を実行する推定時間は 30 分です。
2. FFPEセクション用多重免疫蛍光染色プロトコル
注:このプロトコルは、ロバートソンら17から適応されました。
- 脱パラフィンと水分補給
注:IHCまたはmIFによるFFPE切片の抗体媒介標識の前に、パラフィンを除去する必要があります。パラフィンを効率的に除去しないと、最適でない染色が生じる。- 4 μm FFPE組織セクションスライドをガラススライドホルダーに配置します。ヒュームフードの下で、37°Cのプレウォームドキシレンを含むコプリン瓶にスライドを10分間浸します。
注意:キシレンは可燃性、皮膚刺激性、吸入すると有害です。 - スライドを手動で2分ごとに10 s撹拌し、新鮮なキシレンで1倍の間隔でさらに5分間繰り返します。
- 化学フードに、スライドを次の各溶液の5分間連続して浸します: 1) キシレン : エタノール (1:1 v/v);2)100%エタノール;3)70%エタノール;4)50%エタノール;5)30%エタノール;6)リン酸緩衝生理食塩分(PBS)
注:抗原検索を行う準備ができるまで、スライドをPBSに保管してください。脱ワックスされたセクションは常に水分補給してください。乾燥すると、非特異的な抗体結合が生じ、バックグラウンド染色が高くなります。
- 4 μm FFPE組織セクションスライドをガラススライドホルダーに配置します。ヒュームフードの下で、37°Cのプレウォームドキシレンを含むコプリン瓶にスライドを10分間浸します。
- 熱誘導抗原の検索
注:抗原は、フォーマルリン固定の際にマスクすることができ、抗体結合を防止し、結果的に可視化することができます。抗原マスク解除緩衝液および手順の使用は、部分的にエピトープの天然の立体構造を再確立し、それによって抗体認識を回復させる。抗原の取得バッファーの種類と持続時間は、特定のアッセイ条件(例えば、標的、抗体、組織など)に合わせて最適化する必要があります。- 抗原検索溶液を含むコプリン瓶に脱蝋スライドを浸す(材料表のレシピ)。
- 閉じたコプリン瓶を水道水で電気圧力鍋に入れます。水位は、水が抗原の検索溶液と混合しないように、瓶の半分の高さを超えてはならない。
- 調理器具の蓋と圧力弁を閉じます。10分間高圧を選択して開始します。完了したら、調理器具を抜き、圧力を解放し、蓋を開け、瓶を調理器の中に30分間入れておき、スライドを冷却します。
- 非特異的バインディングのブロック
- スライド付きのラックをPBSで満たされたコプリン瓶に移します。PBS 2xで抗原検索バッファーを5分間リンスします。
- PAPペンで組織切片を囲み、疎水性バリアを作ります。スライドをPBSに0.1 Mグリシンを含むコプリン瓶に浸します。室温(RT)で15分間インキュベートします。
注:グリシンは、抗原の取り出し中に生成されたアルデヒド基を飽和します。これらのグループは、一次抗体と二次抗体を非特異的に結合する可能性がある。 - 2xをPBSで5分間洗浄してグリシン溶液を洗い流し、スライドを湿度チャンバーに入れ、すべての組織切片を覆うのに十分なブロッキング溶液を加えます。疎水性バリアのオーバーフローを避けてください。RTで30分間インキュベートします。
注: ブロッキングソリューションのレシピは、資料一覧を参照してください。ブロッキング溶液は、非特異的結合部位をブロックするタンパク質(例えば、BSA)を含むべきである。また、Triton X-100やTween 20のような洗剤を組み込むことで、抗体と組織標的の疎水性相互作用を低減し、抗原認識をより選択的にすることができます。組織が由来する種からの10%の総血清の添加は、Fc受容体をブロックし、したがって、非特異的抗体結合を減少させるであろう。最後に、二次抗体が上げられた種から血清の10%を加えると、組織部への二次抗体の直接非特異的な結合を最小限に抑えるであろう。
- 免疫蛍光標識
- PBS-Tween (0.1% v/v) 2xでそれぞれ5分間リンスし、スライドを湿度チャンバーに戻します。
- ブロッキング溶液中に再懸濁された一次抗体のカクテルを加える。4°Cで一晩インキュベートする。この研究に使用される一次抗体と二次抗体は、材料表にリストされています。
注:一次抗体のカクテルには、異なる種で育てられた抗体、または同じ種から生まれたが、異なるアイソタイプの抗体のいずれかが含まれている必要があります。本研究で用いた一次二次抗体対のリストについては、表2を参照してください。使用されるすべての抗体の詳細は、資料表および表 2にあります。 - PBS-Tween (0.1% v/v) 3x で 5 分間リンスし、スライドを湿度チャンバーに戻します。暗闇の中で、二次抗体のカクテルを加え、RTで1時間インキュベートする。
注:一次抗体が異なる種からのものである場合、二次抗体は、それぞれが一次抗体の1つにだけ結合し、互いに結合しないように選択する必要があります。これは、一次抗体が生成された種とは異なる限り、同じ種で全て上げられた二次抗体を用いて達成される。一次抗体が同じ種で育てられたが、異なるアイソタイプを有する場合、アイソタイプ特異的な二次抗体を使用すべきである。 - PBS-Tween (0.1% v/v) 3x でそれぞれ 5 分間リンスします。ddH2O. 余分な液体を取り除き、DAPIを搭載したメディアに取り付けます。使用されるボリュームは、セクションのサイズによって異なります。通常40 μLは、通常の顕微鏡スライドの表面を覆うのに十分です。
- カバースライドをセクションに置き、気泡の形成を避けて余分な取り付け媒体を静かに絞り出します。スライドを暗闇の中でRTで20分間乾燥させ、取得の準備ができるまで4°Cで保管してください。
- スライド スキャナ全体を使用して、すべてのチャンネルのイメージを取得します (「資料表」を参照)。
注:抗体は、陽性対照としてヒト肝細胞癌組織を用いて検証された。各一次抗体について、3つの連続切片を、染色プロトコルの残りの部分にばらつきを伴わず、それぞれ一次抗体、アイソタイプ制御、またはブロッキング溶液のいずれかで染色した。取得した画像を比較して染色の特異性を確立した。この染色は、一次抗体と共にインキュベートされたセクションのシグナルが期待されるパターンを有し、バックグラウンドと容易に区別できる場合に特異的と考えられた。アイソタイプで高いバックグラウンドシグナルまたはラベリング組織成分を与える一次抗体と、一次抗体切片は非特異的とは考えなかった。プロトコルのこの部分を完了するための推定時間は 2 日です。必要な制御は、(1)一次抗体の非特異的結合の寄与をバックグラウンドシグナルに確立するためのアイソタイプ制御を含む。一方のセクションは、一次抗体の同じアイソタイプおよび起源を有する抗体でインキュベートされるが、組織セクションに存在しない標的に特異的であることを除いて、他のサンプル組織と同じように染色される。適切なアイソタイプ対照抗体が利用できない場合、一次抗体が上げられたのと同じ種の全IgGに置き換えることができます。(2)染色の特異性を確立し、バックグラウンドシグナルに対する二次抗体の非特異的結合の寄与を推定する一次抗体コントロール(すなわち、陰性対照)がない。この場合、対照セクションは、一次抗体が添加されないものを除いて、他のセクションと同じ方法で染色されます。(3)正のコントロールは、染色が機能することを確立する。この場合、染色は、一次抗体によって認識されるマーカーを発現することが知られている組織部に対して行われる。
3. ピクロシリウスレッド(PSR)/高速緑色染色プロトコル
注:この染色の目的は、FFPE組織切片でフィブリラールコラーゲンIおよびIIIを可視化することです。このプロトコルは、セガニら18.すべてのステップは化学フードで行われる。
- FFPEセクションの多重免疫蛍光染色プロトコル(セクション2.1)と同様の組織切片の脱パラフィン化および水分補給を行う。
注:染色するセクションが免疫蛍光標識に使用され、パラフィンがすでに除去されている場合、脱パラフィン化・水分補給ステップは、取り付け培地を除去するのに有用である。DAPI はこの手順を使用して除去されませんが、PSR染色を知覚的に妨げません。 - ピクロシリウスの赤/高速緑色の溶液(材料表のレシピ)を含む瓶にスライドを浸し、RTで30分間インキュベートします(30分以上は肝細胞の核の非特異的染色をもたらします)。
- スライドをddH2O(5ディップ)で素早く洗います。その後、エタノール100%(5ディップ)で素早く洗浄する。キシレン-100%エタノール(1:1 v/v)で30s洗浄してください。キシレンで30s洗います。キシレンが完全に蒸発する前に取り付けメディア(材料表を参照)を取り付けます(これは取り付けに役立ちます)。
注: プロトコルのこの部分を実行するための推定時間は 1 時間です。
4. 組織切片からの抗体の溶出
注:シーケンシャルラベリングアッセイで組織切片を再利用するには、一次抗体と二次抗体の完全な除去が必要です。結合抗体は、前述の13のように除去された。
水浴を56°Cに予熱します。剥離バッファー(材料のテーブルのレシピ)を含む瓶の中にセクションを入れて、蓋を閉め、パラフィンフィルムテープで密封して、揺れ時に漏れるのを防ぎます。
- 水浴の中に瓶を入れ、攪拌で30分間インキュベートします。
- RT. PBS-Tween(0.1%v/v)でddH2Oでそれぞれ15分間4倍洗浄します。
- 第2の一次抗体でセクションを再調査する準備ができるまで、セクションをPBS-Tweenまたは水で水分補給してください。
注: プロトコルのこの部分を実行するための推定時間は 2 時間です。 - 抗体溶出手順の効率を検証します。
注: 逐次標識アッセイで抗体溶出のプロトコルを使用する前に、一次抗体および二次抗体の除去の効率を検証する必要があります。- FFPEセクションの多重免疫蛍光染色プロトコル(セクション2.1-2.4.6)に示されているように、特定の一次二次抗体ペアの対象となるセクションの染色および画像取得を行います。
- 画像取得時に、セクション4.1~4.3に示すように、組織結合型一次二次抗体複合体の溶出を行う。
- ステップ2.4.3で使用した同じ二次抗体と同じ条件でセクションをインキュベートする。
- 2.4.4~2.4.6 に示すように、洗浄、取り付け、および画像取得の手順を実行します。
- 特定の信号が消えたかどうかを確立するために、ストリッピングの前後に取得した画像を並べて比較します。
注:抗体除去前後の画像を比較すると、溶出手順の効率が検証されます。しかし、DAPIの拡散だけでなく、すべてのチャネルでバックグラウンド信号の増加を見ることは正常です。これにより、同じ組織セクションで実行できるストリッピングのラウンド数が制限されます。ストリッピングの3ラウンドが最大のようです。
5. 画像の取得
- スライド スキャナー全体を使用してイメージを生成します。
- 20x 0.75NA対物レンズと0.3225 μm/ピクセルの解像度を使用します。
6. 画像解析
注: ここで説明する方法は、現在の例を示します。表 1と他の特定のサンプルに適合するテキストを参照してください。
- 画像解析ソフトウェアのTissualignモジュールを使用して組織の位置合わせを行います(このプロトコルのVISは、材料表を参照)。
- 画像解析ソフトウェアを開き、[組織整列]タブをクリックします。
- [ファイル] メニューの [ファイル] メニューの[スライド トレイ] に整列するイメージをインポートする |データベースをクリックし、位置合わせする最初のイメージを選択します。[組織揃え] タブに戻り、スライド トレイの[読み込み] ボタンをクリックしてイメージを読み込みます。イメージは、スライド トレイとワークスペースに表示されます。
メモ:目的のスタックのみをスライドトレイにロードする必要があります。 - すべての画像を整列させる順序で、それらを1つずつロードするために、ステップ6.1.2を繰り返します。目的の画像がすべてスライド トレイに読み込まれたら、リボンのワークフロー ステップで[次へ] を押して、イメージをリンクします。
- 次に、最初の画像の上に 2 番目の画像をドラッグ アンド ドロップします。最初と 2 番目の画像がリンクされます。他のイメージを 1 つずつ整然と整列させるために、この手順を繰り返します。最初の画像の名前が変わり、他の画像にリンクされていることを示します。同時に、リンクされた画像はスライドトレイの右側のワークスペースに表示されます。
- この時点で、自動配置、半自動配置、または手動配置を使用して、イメージを配置します。最初に自動配置を試みることが常に好ましいです。自動配置の場合は、リボンのワークフロー ステップ (手順 3) の[次へ] ボタンを押します。
- 組織の異なる位置をナビゲートし、異なる画像内の対応する構造が画像の2次元で同じように配置されていることを視覚的に確認することによって、自動配置を確認します。
- 自動整列の結果が満足できない場合は、リンクされた画像内の相同組織の特徴を示すピン(画像ごとに最低3つのピンを使用)を使用して改善してください。ピンがリンクされた画像の相同な位置に配置されると、半自動配置または手動配置の2つの選択肢があります。半自動整列の場合は、リボンの現在のピンポイントに基づいて[自動整列]ボタンをクリックします。手動で配置するには、リボンの[ピンを適用] ボタンをクリックします。
- 配置に問題がなければ、ワークフローステップの「次へ」ボタンをクリックし、複合画像をデータベースに保存します。
注: 11 個のマーカーにまたがって 6 つのスライドを配置し、H&E と PSR イメージを揃える場合、15 分の分析が行われました。
- ユーザー定義のプロトコル分析プロトコルパッケージ1(APP 1,表1)を使用して組織検出を実行する。
- リボンの[イメージ解析]タブをクリックして、ソフトウェアの[画像解析]モジュールを開きます。
- [ファイル] メニューの [ファイル] に進み、合成 (整列) イメージをインポートします。データベースを使用して目的の画像を選択し、[画像解析] タブをクリックします。
- [APP を開く] アイコンをクリックして[APPの選択] ダイアログを開き、使用する分析プロトコル パッケージ (APP) を選択します。この場合、組織検出用のAPP1を選択する。
- APP 1が開いたら、選択した組織の場所に移動してプレビューボタンをクリックして、APP1が正常に動作していることを確認します。結果が満足のいくものであれば、次のステップに進みます。
- クリックして APP 1 を実行し、選択した APP を使用してイメージを処理します。
- 解析が終了したら、[ファイル/エクスポート]をクリックしてデータ(画像、測定値など)をエクスポートします。
注意:APP 1は、組織(ROI組織)を非道な領域(ROI)を作成し、組織の面積を計算します。 - [ファイル] メニューの [ファイル] に進み、新しく作成した ROI を使用して変更したイメージを保存します。保存します。
注: 上記の例で APP 1 を使用して組織を検出し、ROI を作成する場合は、画像解析ステーションで 5 分かかりました。処理された組織の面積は3.2 cm2であった。
- APP2を用いて、ストロマおよびパレンチマへの組織セグメンテーションを行う(表1)。
注:APP 2は、事前定義されたROI組織で動作します。APP 2は、組織をROIsストロマとパレンチマにセグメント化します。- リボンの [イメージ解析] タブをクリックして、[画像解析] モジュールを開きます。
- ROI 組織を含む画像をインポートするには、[ファイル] |データベースとステップ 6.2.7 で保存されたイメージを選択します。[イメージ分析] タブに戻り、スライド トレイの[読み込み] ボタンをクリックしてイメージを読み込みます。イメージは、スライド トレイとワークスペースに表示されます。
- 6.2.3 のように、APP 選択ダイアログを使用して APP 2 を開きます。
- 選択した視野で処理して APP 2 をプレビューします。結果が満足のいく場合は、[実行] ボタンをクリックして、完全なイメージで APP 2 を実行します。APP 2の出力として、ROI組織はROISストロマとパンキマにセグメント化され、それぞれの領域が決定されます。結果を 6.2.6 のようにエクスポートします。6.2.7 で変更した画像を保存します。
注:APP 2を使用してストロマとパレンチマで組織をセグメント化することは、提示された分析ステーションで4時間かかりました。処理された組織の面積は3.2 cm2であった。
- ユーザー定義プロトコル APP 3 (表 1) を使用して FoxP3hiCD4+ セルを特定し、定量化します。
注意:APP 3は、事前に定義されたROIsストロマとパレンチマで動作します。- 画像解析モジュールを開き、6.3.1 および 6.3.2 のように、ROIs Stroma および Parenchyma を含む画像をインポートします。6.2.3 のように APP 選択ダイアログを使用して APP 3 を開きます。
- FoxP3hiCD4+セルに富んだ選択された視野でのプレビューAPP 3処理。結果が満足のいくものであれば、完全なイメージで APP 3 を実行します。APP 3の出力として、すべての個々のFoxP3hiCD4+オブジェクトにラベルが付き、その組織座標が保存されます。ROIsストロマとパレンチマにおけるFoxP3ハイCD4+オブジェクトの密度が決定されます。結果を 6.2.6 のようにエクスポートします。
- FoxP3hiCD4+ラベル付きオブジェクトの組織ヒートマッピングを実行します。
- 6.2.3 のように、APP 選択ダイアログを使用して、ユーザー定義プロトコル FoxP3hiCD4+ MAP を開きます。
注: FoxP3hiCD4+ MAP は、FoxP3hiCD4+ ラベル付きオブジェクトの座標を使用して、密度ヒートマップを生成します。APP 3を使用してFoxP3hiCD4+ラベル付けされたオブジェクトを識別し、カウントすることは、説明された画像分析ステーションで25分かかりました。処理された組織の面積は3.2 cm2であった。 - [実行] ボタンを押して、FoxP3ハイCD4+ MAP を実行します。[ファイル] メニューの [組織のヒートマップをエクスポート] をクリックして、組織のヒートマップをエクスポートします。エクスポート |作業領域。
注:FoxP3hiCD4+ラベル付きオブジェクトをFoxP3hiCD4 + MAPを使用してマッピングすると、画像解析ステーションで5分かかりました。
- 6.2.3 のように、APP 選択ダイアログを使用して、ユーザー定義プロトコル FoxP3hiCD4+ MAP を開きます。
- ユーザー定義のプロトコル APP 4、APP5、APP6、APP7、および APP 8 を使用して、CD8+、CD68+、MPO+、および CD34+ オブジェクトを識別し、定量化します (表 1) 各ケースで関心のある APP を読み込むセクション 6.4 ~ 6.4.3.2 で行います。
注: AP4 ~ 8 は、定義済みの ROIs Stroma および Parenchyma で動作します。
結果
TMEで関心のある細胞集団を可視化、定量化、マッピングするための戦略の概要
異なる組織区画(TC)内の目的とする細胞集団(COI)を定量化し、その空間的組織を特徴付けるために、手頃な価格で使いやすい技術を統合し、貴重なFFPE臨床検体から得られる位置情報を最大化するワークフローを設計しました(図1)。まず、連続組織全体FFPE切片を、COI(例えば、免疫細胞)およびTC(例えば、間質対パレンチマ)の可視化のために染色した(図1、ステップ1)。染色される連続したセクションの数は、研究の質問に対処するために必要な関心のある細胞または組織の特徴の視覚化を可能にする最小限に抑える必要があります。連続セクションの数が少ないほど、組織アーキテクチャは隣接するセクション間で類似し、一貫性が高くなります。さらに、多重化能力は、ストリッピングおよび再探査技術19を通じて蛍光染色切片の再利用によって拡張することができる。
染色手順が完了すると、スライドスキャナ全体を使用して画像をデジタル化しました。シリアルセクションから取得した画像は、自動で仮想多重スライドに整列および統合されました(図1、セクション2)。次に、組織のROIを、組織関連ピクセル(TAP)を同定するユーザ定義のプロトコルで分線化した(図1、ステップ3)。その後、ROI組織を追加のROIとして定義されたTCにセグメント化した。(図1、ステップ4)。次に、ユーザ定義プロトコルが検出され、異なるTCでCOIを定量化します(図1、ステップ5)。最後に、COIの組織ヒートマップは、それらの密度とそれらの組織座標に基づいて生成された(図1、ステップ6)。

図1:TMEにおける免疫細胞の可視化、定量化、マッピングの戦略の概略図(1)シリアル全体の組織切片をCOIおよびTCの標識のために染色した。(2) シリアルセクションから取得した画像は、組織配置解析モジュールを使用して自動でリンク、整列、および再登録された。個々の画像の高精度な配置から合成画像が生成されました。(3) 複合画像内の組織関連ピクセル (TAP) の自動検出にユーザ定義のプロトコルを使用した。(4)組織を、ROIとして定義されたTC(例えば、間質およびパレンチマ)に分けた。(5) ユーザー定義プロトコルは、異なるTCにおけるCOIの自動検出と定量に使用された6。この図の大きなバージョンを表示するには、ここをクリックしてください。
イメージング COI と TC
HBV関連肝細胞癌を有する被験体から切除された腫瘍の3つの連続FFPE全組織切片は、図2AAのように1回以上の染色で染色した。セクションIは、組織アーキテクチャ、細胞形態を示すためにH&Eで染色され、悪性腫瘍の種類、腫瘍グレード、および免疫浸潤の全体的な評価などの臨床的に関連するパラメータを決定する(図2C)。連続したセクションIIでは、2ラウンドのmIFが肝パレンチマルおよび非パンキマル細胞の標識に使用された(図2A)。第1ラウンドでは、正常および腫瘍の血管を、CD34内皮細胞の染色を用いて可視化した。また、上皮細胞(肝細胞および胆管球)をサイトケラチン8/18を用いて同定し、線維原性活性化肝星状細胞をα平滑筋アクチン陽性(αSMA+)細胞として同定した(図2C)。画像取得後、組織切片を、マクロファージ(CD68)および筋線維芽細胞(desmin)に対する抗体で除去し、再プローブした。より良い腫瘍免疫浸潤を特徴付けるために、隣接するシリアルセクションIIIは、細胞マーカーCD3、CD4、CD8、フォークヘッドボックスP3(FoxP3)、および骨髄ペルオキシダーゼ(MPO)に対して2ラウンドのmIFを使用して染色した。すべての場合において、DAPIは核カウンターステインとして使用されました。最後に、セクションIIIはPSR染色で染色され、カウンターは速い緑色で染色され、線維性コラーゲンを視覚化し、組織を間質およびパレンチマに分ける(図2C)。
20X対物レンズを搭載したスライドスキャナ全体を使用して、染色されたセクションをデジタル化し、仮想スライドを作成しました。図1の回路図に従って、3つのシリアルセクション(図2B)から6つの画像を取得し、仮想スライドをVISソフトウェアを使用して解析した。
画像解析
画像解析は、5つのステップで構成されています: 1) 組織のアライメント;2)組織検出;3)組織のセグメンテーション;4) COIsの自動定量化;5)組織ヒートマッピング。画像解析用のプロトコルはすべて、画像解析ソフトウェアの作者モジュールを使用して開発され、テキストではAPPと呼ばれています。
組織アライメント
3つのシリアルセクションから6つの仮想スライド(11個のマーカーとH&EとPSR染色)を取り出し、画像解析ソフトウェアのTissualignモジュールにロードしました。次に、画像をリンク、整列、および自動登録し、11プレックスとH&EおよびPSR仮想合成画像を生成し、個々の画像のすべての層を含む(図2A-C)。隣接するシリアルセクションから発生する画像の場合はアライメントが正確であり、アライメント時に相同的に配置および配置された対応する組織構造を示す(図2CCおよび図S1A)。さらに、同じセクションから発生した画像の個々のセルレベルでの位置合わせは正確であった(図S1B)。自動配置の時間は、整列する画像の数、サイズ、複雑さ、および類似性によって異なります。上記の6つの仮想スライドのアライメントは、当社のVISステーションで15分かかりました。
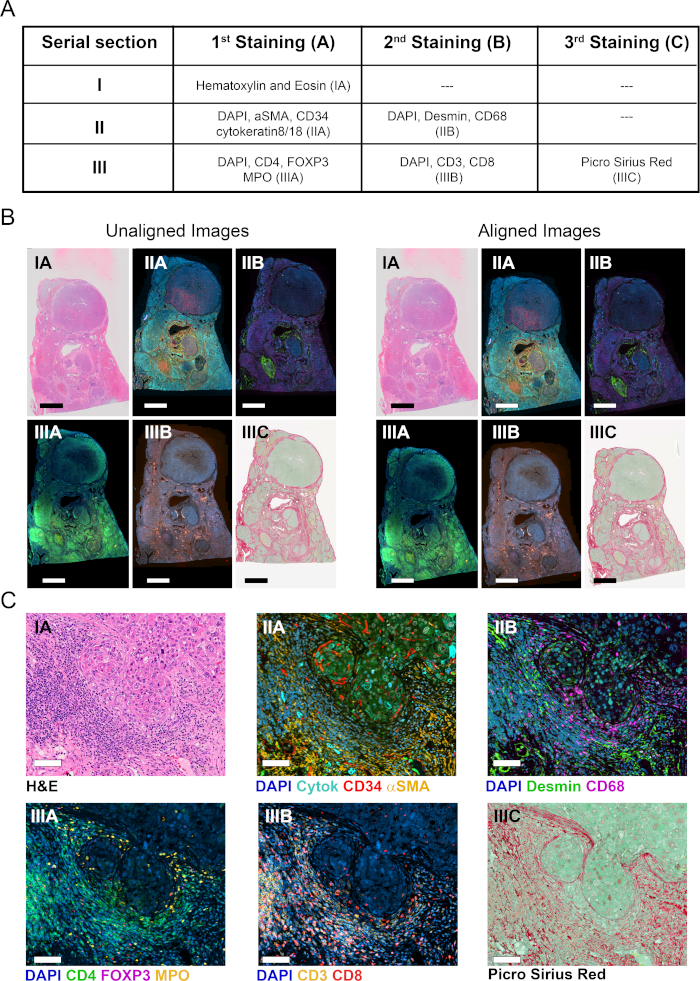
図2:連続組織切片と画像アライメントの染色。(A) COI と TC の視覚化のために 3 つのシリアルセクションで行われる染色の概要。第II条およびIII節については、組織を剥ぎ取り、抗体の第2のカクテルで再調査した。(B)組織の整列前と後の6つの個々の組織全体像の概要(それぞれ左右)。スケールバー = 3,500 μm(C) 整列画像のズームビュー。スケールバー= 80 μm.この図の大きなバージョンを表示するには、ここをクリックしてください。
組織検出
画像がリンクされ、整列されたら、タップを特定しようとしました(図3A)。タップの自動検出用のAPP(APP 1,表1)を設計するために、タップと組織に関連しないピクセルを区別する2つの特性を利用しました。まず、DAPIシグナル(青いバンド)は、組織に排他的に配置されている核に制限され、すべてのDAPI +ピクセルがタップのサブセットであることを意味する。第二に、TAPは、組織に関連しないピクセルと比較して、緑色および黄色のバンドでより高い自己蛍光信号を有する。その結果、簡単な閾値技術を用いて、これらのチャネルのベースライン信号に基づいてタップを検出する組織検出用のAPP 1(表1)を開発しました。青、緑、黄色のバンドのしきい値は、TAP がしきい値を超えるバックグラウンド強度値を持ち、組織に関連付けられていないピクセルが以下の値になるように設定されました。組織検出用のAPP 1を画像IIAに適用し、青色、緑、黄色のチャンネルに層を含む(図3A)。APP 1の出力として、明るい緑色のマスクがタップの上に置かれ、「ティッシュ」と呼ばれるROIが線分化された(出力、図3A)。さらに、組織の面積を定量的な出力変数として決定した。APP 1は組織に関連付けられていない画素をROI組織に組み込んでいないので、このROIに基づく後続の分析から除外された(図3A)。図3Aに、タップの識別における APP 1 の精度を示します。
TCのROIの組織の分割と線引き
次に、組織を間質対パレンチマに分けて、ROI組織内部の異なるコンパートメントを定義しました。PSR染色像(IIIC、図2C)を用いて、この間質はフィブリラーコラーゲンの堆積に関連する領域(赤帯)として定義することができ、フィブリラーコラーゲンが存在しない領域としてパレンチマ、高速緑色の対染色が優勢(緑色のバンド)(図3B)である。私たちは、TCsストロマとパレンチマをデジタル的に区切るためにAPP 2(表1)を作成しました。このアプリは、事前定義されたROI組織(出力、図3A)で動作し、画像解析モジュールに統合された分類器ツールを訓練するために代表的なストロマとパレンチマ領域を使用します。訓練された分類器は、ピクセルを間質またはパンキマラベル(それぞれ、図3B)に割り当てます。画素の分類に際して、APP 2は、ROISストロマとパレンチマの定義を目指して形態学的操作を実行した(図3Bおよび表1)。ピクセルを分類し、それぞれの ROI を生成する際の APP 2 のパフォーマンスを図 3Bに示します。さらに、APP 2は、間質とパンキマの面積を定量化します。最後に、セグメンテーションはPSR染色部を使用して行われますが、輪郭付きのストロマ領域とパレンチマ領域は、PSR画像に整列した任意の画像に転送することができます。

図3:組織検出/セグメンテーションとそれぞれのROIの生成(A) 画像 IIA を使用して、タップを識別しました (左画像、スケールバー = 6,000 μm)。明るい緑色のマスクが APP 1 ( 表1) を使用して、組織 (出力 1) と呼ばれる ROI を生成して、その TAP に割り当てられました。右、インセットは、タップを検出する際のAPP 1の精度を示すズームビューを示しています。スケールバー= 350 μm(B) ROI組織(出力1)はAPP 2を使用して間質とパレンチマにセグメント化されます。左の画像はROIストロマ(サーモン)とROIパレンチマ(緑色)に分けられたROI組織の図です。スケールバー= 4,500 μm右側には、ROI組織のインセット、元のPSR染色(画像IIIC)、およびROIsストロマとパンキマのズームビュー。スケールバー= 250 μm。この図の大きなバージョンを表示するには、ここをクリックしてください。
COIsの自動定量
次に、ROISストロマとパンキマのCOIを特定、特定、定量化しました。APP 3~8(表1)は、それぞれCD4+FoxP3+、CD8+、CD68+、MPO+、αSMA+、CD34+の各セルを見つけてカウントするために作成されました。APP 3は、CD4+FoxP3+細胞(画像IIIA、図2C)を制御T細胞(Tregs)の代理マーカーとして検出してカウントするように設計されました。このプロトコルは、核転写因子FoxP3(赤帯)とDNA標識色素DAPI(ブルーバンド)からのシグナルの共局在化を検出する。最近活性化されたT細胞がFoxP3をアップレガンジしていることを考えると、Tregsのために濃縮するために、我々は唯一の明るいFoxP3 +細胞を事前選択するためのしきい値を設定します(FoxP3hi).次に、選択済みのすべての DAPI+FoxP3ハイセルのうち、明るいリング型の CD4 信号 (緑色のバンド) に囲まれたものだけが、FoxP3hiCD4+ セルとしてラベル付けされ、カウントされた (ピンクのラベル,図 4A)。ROIsストロマおよびパレンチマにおけるFoxP3ハイCD4+細胞の密度をAPP 3の量的出力変数として求めた(図4A)。
同様に、AP4~6はCD8+、CD68+、MPO+細胞の検出用に設計されています。これらの APP は、COI の検出と定量を行う同じベースライン設計を共有しています。具体的には、COIは、特定の細胞集団バイオマーカーからのシグナル強度に基づいて同定され、次に、個々の細胞を線引するためにいくつかの後処理形態学的ステップが実行される(表1)。個々の細胞またはCOIは、ラベル付けされ、カウントされ、それらの組織座標が登録されます。AP4~6は、ROIsストロマおよびパンキマにおけるCOIの密度も決定します(図4B–D)。
DAPI染色の品質は、核セグメンテーションをAP3〜6に統合するのに十分ではなかったので、個々にラベルが付いたすべてのオブジェクトが個々の細胞であることを保証することはできません。このため、ラベル付きオブジェクト/mm2の数でセルの密度を表現しました (図4)。しかし、細胞凝集体は、AP3〜6に組み込まれた後処理ステップで個々の細胞に正常に分離され、広範な目視検査では、ほとんどのラベル付きオブジェクトが単一細胞に対応することが示されました。
αSMA+およびCD34+領域の検出のために、我々は、それぞれAP7および8を開発した(表1)。両方の APP は、しきい値に基づいて特定の信号を検出し、ROIs Stroma および Parenchyma の正の領域のパーセンテージを決定します (図 4E–F)。
仮想多重スライドを生成する最も興味深い可能性の1つは、共局在式の分析です。我々は、αSMAとデスミンの共局在化を検出するAPP10を生成し、肝臓内の筋線維芽細胞によって共発現する2つのマーカーである。APP 10は、αSMA、デスミン、およびαSMAプラスデスミンの正のピクセルを求めるしきい値を使用します(表1)。量的な出力変数として、APP10はαSMA+領域、デスミン+領域、およびこれら2つのマーカーの共局化式の面積を決定する(図S3)。

図4:TCs間質およびパンキマにおけるCOIの同定と定量化(A–F)プロトコル3、4、5、6、7、および8を使用して、ROIsストロマおよびパンキマにおけるCD4+FoxP3+、CD8+、CD8+、CD68+、MPO+、αSMA+、およびCD34+COIの自動検出と定量化(表1)。左側に表示されるのが元の画像、処理された画像の中央、右側の定量です。図 4A–Dの場合、スケールバー = 40 μm。図4EとFの場合、スケールバー= 350 μm。この図の大きなバージョンを表示するには、ここをクリックしてください。
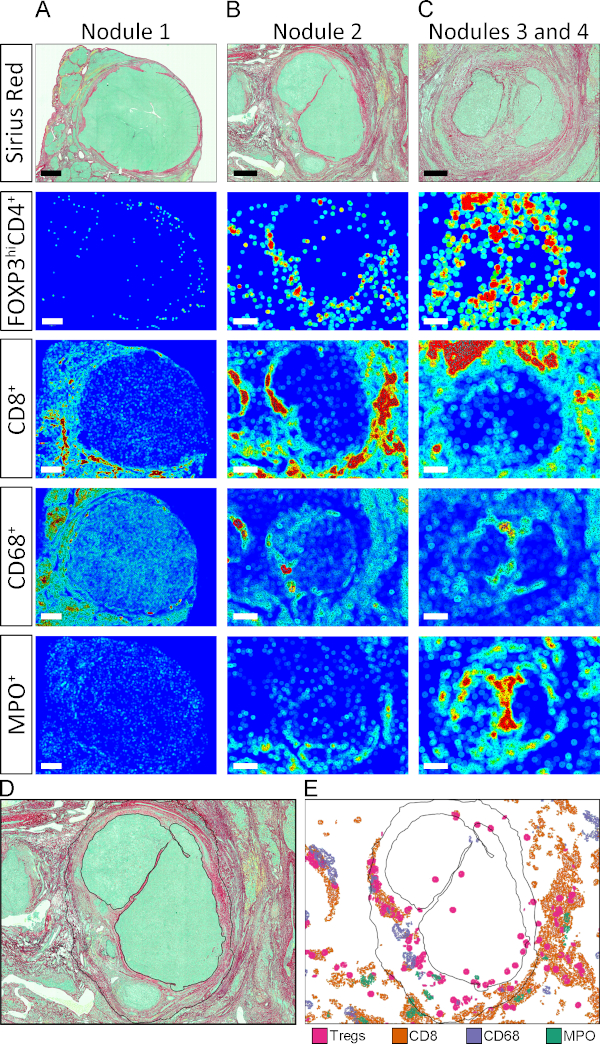
TCsストロマとパンキマのCOIを定量化する代わりに、1~4という異なる悪性結節における免疫細胞の密度を決定しました(図5 A,H,およびAI)。各結節の ROI は、図 5Aに示すように手動で表されます。特徴的な組織免疫シグネチャは各結節を特徴付け、さらにTMEの本質的な不均一性を明らかにした。
組織ヒートマップ
前述のように、APP3〜8は、個々にラベル付けされた各オブジェクトの組織座標を格納する。この機能により、特定の細胞集団の高密度の領域がホットスポット(赤)として表示され、比較的密度の低い領域がコールドスポット(ダークブルー)として表示される組織マップが自動生成されます。中間密度値は、図 5に示すカラー スケールに従って色を割り当てられます。組織ヒートマップは、画像を直径50μmの円に分割し、円内の所定のCOIの相対的な密度に従って色を割り当てたAPによって生成されました。図5B–Gに示すように、TME内の異なるCOIの位置決めパターンと強度分布は非常に多様であった。さらに、個々の結節のレベルでは、組織領域における異なる集団の配置は独特であった(図S2A-C)。この手法の力の例を示し、同じ結節内の異なる集団からのホットスポットの空間的構成を可視化するために、個々の細胞タイプのホットスポットを手動で抽出し、結節2の輪郭にマッピングした(図S2、図D、図E)。

図5:TMEにおけるCOIの組織ヒートマップ(A)ピクロシリウス赤染色は、結節1、2、3、および4の位置を示す。(B–G)CD4+FoxP3+、CD8+、CD68+、MPO+、CD34+、およびαSMA+COIの組織ヒートマップ。濃い青色は相対的に低密度を示し、赤は相対的な高密度を示します。中間密度値には、表示されているカラースケールに従ってカラーが割り当てられます。(HおよびI) 各細胞タイプおよび結節当たり 1、2、および 3 + 4 の COI の定量化。この図の大きなバージョンを表示するには、ここをクリックしてください。

補助図S1:組織の整列の検証。(A)セクション II (入力 1) で行われる CD34 染色 (入力 1) は、緑色 (出力 1) で CD34 マスクを生成するために使用されます。緑色のマスク(出力1)は、位置合わせされたシリアルセクションI(入力2)のH&Eイメージ上に重ねられます。マージ画像は、血管構造の完全な対応を示しています。スケールバー= 50 μm(B) DAPI、CD4、および FoxP3 (入力 1) の結合を示すイメージ IIIA を使用して、CD4+FoxP3+ セルのラベルを生成しました (マゼンタの出力 1)。出力1ラベルは整列した画像IIIB(入力2)に転送され、マージイメージ内のFoxP3/DAPIとCD4 / CD3のペア間の完全な対応を示しています。スケールバー= 15 μm。この図の大きなバージョンを表示するには、ここをクリックしてください。
補助図 S2: 組織ヒートマップの拡大表示(A–C)小結節1~4のCD4+FoxP3+、CD8+、CD68+、MPO+セルの組織ヒートマップ。小節1、2、3+4のスケールバーは、それぞれ1,500 μm、700 μm、500 μmを表します。(D) 黒実線を持つ結節2の輪郭。(E) CD4+FoxP3+、CD8+、CD68+、および MPO+セルのホット スポットを、Dで定義された結節 2 のアウトラインに抽出し、マッピングした。この図の大きなバージョンを表示するには、ここをクリックしてください。

補助図 S3: 共局在分析(A)左と中央にそれぞれ赤で緑色とデスミンラベルのαSMAラベルの画像です。右側は黄色のαSMA/デスミン二重正の領域です。(B)αSMA+面積、デスミン+面積、およびαSMA/デスミン二重正極面積の定量化。スケールバー= 150 μm。この図の大きなバージョンを表示するには、ここをクリックしてください。
| アプリ | 目的 | 分類 | 分類 | 後処理ステップ | 出力変数 |
| メソッド | 機能 | ||||
| (ピクセル値) | |||||
| 1 | 組織検出 | しきい値 | チャネル DAPI (150) | o 3 チャンネルのしきい値を超える共局化されたオブジェクトにラベルを付ける | o ROI組織 |
| チャンネル FITC/A488 (120) | o 正のオブジェクトを 5 ピクセル閉じる | o 組織領域 | |||
| チャンネル・トリットク/A568 (40) | o ROI組織を作成する | ||||
| 2 | 組織セグメンテーション | ディシジョンフォレスト | RGB-R 中央値 | o 穴を埋める | o ROIストロマ |
| RGB-G 中央値 | o ROIストロマを作成する | o ストロマエリア | |||
| RGB-B 中央値 | o ROIパレンチマを作成する | o ROIパレンチマ | |||
| IHS-S中央値 | o パレンチマエリア | ||||
| H&E エオシン中央値 | |||||
| 3 | CD4+ FoxP3+ セルを検索して定量化するには | しきい値 | チャネル DAPI (>600) | DAPI と Cy5/A647 の共存を伴うラベルオブジェクト(FITC/A488 信号に囲まれた) | O ROISストロマおよびパンキマにおけるCD4+FoxP3+細胞の数と密度 |
| チャネル FITC/A488 ポリスムージング (>850) | o 7 μm2より小さいクリアオブジェクト | o 個々の CD4+FoxP3+ セルの座標 | |||
| チャンネルCy5/A647(>800) | |||||
| 4 | CD8+ セルを検索して定量化するには | しきい値 | チャネル DAPI (<1200) | o 15 μm より小さい正の物体をクリア2 | o ROIsストロマおよびパンキマにおけるCD8+細胞の数と密度 |
| チャンネル Cy5/A647 中央値 (>80) | o 正のオブジェクトを閉じる 2 ピクセル | o 個々のセルの座標 | |||
| o オブジェクトを分離する | |||||
| 5 | CD68+ セルを検索して定量化するには | しきい値 | チャンネル FITC/A488 (>200) | o 20 μm より小さい正のオブジェクトをクリアする 2 | O ROIsストロマおよびパンキマにおけるCD68+細胞の数と密度 |
| o 正のオブジェクトを3ピクセルに拡張する | o 個々のCD68+セルの座標 | ||||
| o オブジェクトを分離する | |||||
| 6 | MPO+ セルを検索して定量化するには | しきい値 | チャネル DAPI (>400) | o 5 μm2より小さいクリアオブジェクト | o ROIsストロマおよびパンキマにおけるMPO+細胞の数と密度。 |
| チャンネル・トリットC/A568 (900-4000) | o 3ピクセルの正のオブジェクトを拡張する | o 個々のMPO+セルの座標。 | |||
| o オブジェクトを分離する | |||||
| 7 | αSMA+エリアを検索して定量化するには | しきい値 | チャネル・トリットC/CF568 (>1050) | o 25 μm より小さい正のオブジェクトをクリアする 2 | o ROIsストロマとパレンチマにおけるαSMA+面積の数と密度 |
| o 3ピクセルの正のオブジェクトを拡張する | o αSMA+ ピクセルの座標 | ||||
| 8 | CD34+ 領域を検索して定量化するには | しきい値 | チャネル DAPI (<5000) | o 25 μm より小さい正のオブジェクトをクリアする 2 | O ROIsストロマとパンキマにおけるCD34+面積の数と密度 |
| チャンネル Cy5/A647 中央値 (>120) | o 3ピクセルの正のオブジェクトを拡張する | o CD34+ ピクセルの座標 | |||
| 9 | 特定の細胞集団に対する組織ヒートマップの作成 | オブジェクトヒートマップ | オブジェクトヒートマップ | o ヒートマップ | |
| 描画半径 50 μm | --- | ||||
| 10 | αSMAとデスミンの共局在化の定量 | しきい値 | チャネル・トリットC (CF568) (>1050) | TRITC (CF568) のしきい値を超えるラベル・オブジェクト | αSMAとデスミンの共局的発現を定量化する |
| チャンネルCy5 (A647) (>1000) | O ラベル オブジェクトのしきい値を超える Cy5 (A647) | ||||
| O TRITC (CF568) および Cy5 (A647) のしきい値以上の共局在化を持つラベルオブジェクト | |||||
| o 25 μm より小さい正のオブジェクトをクリアする 2 |
表 1: 画像解析に使用する APP の設計に使用される一般的なパラメータこの表で指定されたパラメータは、この分析で使用される画像の固有の特性(例えば、背景、アーティファクトなど)に調整され、他の画像には適用されない場合があります。この研究で分析した特定の画像に対して、前述の処理後のステップが定義されていたため、意図的には詳細ではありません。ユーザーは、分析対象の画像に対して APP をカスタマイズする必要があります。
| セクション/染色 | 一次抗体 | 二次抗体 |
| セクション II/1st染色 | マウス IgG2a 抗ヒト αSMA マウス IgG1 抗ヒト CD34 ウサギの抗ヒトサイトケラチン 8/18 | ヤギアンチマウスIgG2a CF568 ラット抗マウス IgG1 A647 ロバアンチラビットA488 |
| セクション II/2第二染色 | ウサギの抗ヒトデスミン マウス抗ヒトCD68 | ロバアンチラビットA647 ロバアンチマウスダイライト755 |
| セクション III/1st染色 | マウス抗ヒトCD4 ウサギの反ヒトFoxP3 ヤギ反ヒトMPO | ロバアンチマウスA488 ロバアンチラビットA647 ロバアンチヤギA568 |
| セクション III/2第 2染色 | ウサギの抗ヒトCD3 マウス抗ヒトCD8 | ロバアンチマウスダイライト755 ロバアンチラビットA647 |
表2:mIF用の二次抗体対
ディスカッション
がんやその他の免疫疾患の免疫の風景をマッピングするために、組織切片の免疫細胞の空間分解能を可能にする、簡単でアクセスしやすく、簡単に多重化技術を実行する必要があります。ここでは、イメージングアッ,セイ12、13、17、19,13,17の多重化能力と多次元評価を拡大するために広く利用可能なラベリングおよびデジタル分析技術を統合した戦略を説明する。19異なるマーカーのための3つの連続セクションの染色、およびストリップと再探査の技術によるセクションの再利用により、H&EおよびPSR染色に加えて11個のパラメータを可視化することができました。これらのセクションからの6つの画像はティッシュの位置合わせモジュールを使用して自動化された方法で整列した。同じセクションから発生した画像の個々のセルレベルでの位置合わせは正確で、隣接するセクションから発生する画像に対しては非常に一致していました。仮想多重化により、あるセクションで視覚化されたマーカーが、別の連続したセクションで視覚化されたマーカーと空間的にどのように関連するかを決定することができました。一部の染色にはCOIとラベル付けされたものもあれば、TCとラベル付けされ、異なるTCでCOIを定量化できるものもあります。COIの自動定量化のためのソフトウェアツールの使用は、画像の処理を大幅に簡素化し、加速しました。さらに、デジタル解析は、選択した視野の代わりに組織切片全体に適用され、TMEの公平な表現をもたらした。さらに、COIの組織座標を登録したため、組織ヒートマップを生成することが可能であった。
このプロトコルには、トラブルシューティングが必要な領域がいくつかあります。まず、不十分な抗原検索はmIFの品質に影響を与える可能性があるため、使用される特定のアッセイ/バイオマーカー条件に合わせて抗原検索バッファーの種類と持続時間を最適化する必要があります。第二に、使用されるブロッキング溶液の種類は、一次および二次抗体の組織/抗原/種に適合すべきである。我々の手では、組織がブロックされたFc受容体から来ている種からの10%の総血清の添加、したがって非特異的抗体結合を減少させた。二次抗体が上げられた種から血清の10%を添加することは、組織セクションへの二次抗体の直接非特異的な結合を最小限に抑えるであろう。第3に、適切な陽性および陰性対照を用いた一次および二次抗体の特異性の検証が必須である。第4に、一次抗体ストリッピング時の一部のチャネルにおける自己蛍光の増加およびDAPIの拡散も一般的である。強化された自己蛍光に対処するために、我々は、特定の信号が背景の少なくとも5倍の強度値を有する一次/二次抗体対を使用した。最後に、一部の高親和性抗体は、通常のストリッピング手順では溶出できません。この場合、我々は、標識の最後のラウンドでそのような抗体を使用することをお勧めします。ユーザは、目的の抗体に最適な構成を見つけるために、異なる染色配列を試す必要があります。ストリッピングの効率は、ラベリングの第2または第3ラウンドに進む前に確認する必要があります。
この戦略の主な制限と課題は、目的のマーカーに対する一次蛍光抗体と二次蛍光抗体の適切な組み合わせを見つけることです。異なる種で、または同時に使用することができる異なるアイソタイプで育てられた一次抗体を見つけることは、市販されているものによって制限される。ほとんどのスライドスキャナーには、最大5チャンネルのイメージングを可能にするランプとフィルタが装備されており、適切な種と右のフルオロフォアの二次抗体は常に利用できるとは限りません。シリアル染色とシーケンシャルラベリングを使用して、これらの制限を部分的に克服しました。いくつかの抗体の組み合わせは、目的のマーカーのための最良の組み合わせに到達するためにテストする必要があります。もう一つの制限は、DAPI染色の品質であり、ストリッピングおよびリプロレーションは常に核セグメンテーションを行うことを可能にするわけではないのでである。
ティッシュの整列モジュールは最低の訓練およびユーザーからのプログラミングの技術を要求しない。ソフトウェアは理論的には、画像の無制限の数の整列を可能にします。しかし、正確な整列は、より組織学的に一致する近いセクションがより正確に整列されるセクションの関連度に依存する。我々は、APPsを生成するためにVISの作者モジュールを使用しました。画像解析の基本的な知識は、APPs を作成するために必要ですが、他の画像解析ソフトウェアを使用する場合も同様です。VISの他の画像解析ソフトウェアと比較したユニークな利点は、異なる方法(例えば、IF、ヒストケミストリー、IHC)を使用して調製されたセクションからの画像の自動整列を含む。これにより、仮想多重化を使用して、関心のある複数のマーカーの共同ローカリゼーション研究が可能になります。さらに、柔軟で使いやすい APP の設計により、ユーザー固有のカスタマイズが可能です。自動化された定量化とマッピング、および組織切片全体を処理する可能性は、時間を節約し、目視検査による手動カウントと比較してバイアスを削減します。
この戦略は、がんと自己免疫の文脈で組織免疫学のための非常に有用な研究ツールですが、臨床使用のために検証されていないまま.追加の標準化と検証により、将来的に複数の用途に使用することができます(例えば、がんの免疫の風景をマッピングして免疫療法薬への応答を予測および監視します)。また、異なる炎症性条件(例えば、炎症性腸疾患)に適応して、予後バイオマーカーと病理学的評価を組み合わせることができる。
このプロトコルの主な重要なステップは、ラベルの効率/特異性と、意図された使用またはバイオマーカー用に設計されたApPの堅牢性です。したがって、目視検査による定期的な検証は、特に新しいアプリを設計する際に不可欠です。ストリッピングとリプロービングの複数のラウンドの効率的な使用、または同じセクション上の異なるタイプの汚れは重要なコンポーネントであり、組織またはセクション固有である可能性があります。大規模なバッチ分析を進める前に、このようなプロセスの効率を確認することが重要です。
要約すると、貴重な臨床組織サンプルから得られる定量的・空間的情報を最大限に活用する戦略を提供します。この方法論を実装するために必要なリソース、設備、知識は広くアクセス可能です。この方法論を、TMEにおける免疫細胞集団の同定、定量、マッピングを目的としたアッセイを計画するための有用なガイドとして提案する。
開示事項
著者らは利益相反を宣言しない。
謝辞
研究参加者に感謝します。HBPバイオバンクのコーディネーターであるルイーズ・ルソーは、組織サンプルと関連するすべての臨床情報の回収に感謝します。我々は、優れた技術支援のために、CRCHUMの分子病理学および細胞イメージングコア施設と、Visiopharmのマイケル・パーシュの核施設を認める。資金:この研究は、カナダ肝臓財団、ケベック・サンテ(FRQS)エイズ・感染症ネットワーク(レゾーSIDA-MI)、C型肝炎に関するカナダネットワーク(CanHepC)からの助成金によって支援されました。CanHepCは、カナダ保健研究所(CIHR)(NHC-142832)とカナダ公衆衛生庁の共同イニシアチブによって資金提供されています。M.F.M.はモントリオール大学、ブルス・ガブリエル・マーキス、FRQSからフェローシップを受けました。T.F.はCIHRとCanHepCから博士課程のフェローシップを受けました。S.T.は、モントリオール大学の肝胆管および膵臓の病的手術でロジャー・デ・グロサイユ・チェアを開催しています。
著者の貢献: M.F.M. 設計、実験、および分析データ。T.F.は実験を設計しました。A.C-B.技術指導を受けています。G.S.は、研究対象のすべての病理学的評価を行い、すべての病理学的側面に関するインプットを提供した。L.M.はH&E染色、最適化、画像取得を行いました。M.N.A.はPSR染色を行い、貴重な技術的インプットを提供しました。N.B.は画像解析に貢献しました。S.T.はHBPバイオバンクの主任研究者であり、バイオバンクの全体的な運営を監督する責任があります。彼はまた、プロジェクトのすべての側面とその臨床的影響に関する貴重なインプットを提供しました。M.F.M、T.F.、N.H.S.は、この研究を概念化し、設計しました。N.H.S.は作業を監督し、資金を得ました。M.F.M.、T.F.、A.C-B、N.H.S.が原稿を書いた。すべての著者は原稿をレビューし、承認しました。
資料
| Name | Company | Catalog Number | Comments |
| Antigen Retrieval Solution: Sodium Citrate Buffer (10 mM Sodium Citrate, 0.05% v/v Tween 20, pH 6.0) | |||
| Blocking Solution: 1 % BSA, 10 % filtered human serum, 10 % filtered donkey serum, 0.1 % Tween 20, and 0.3% Triton in PBS | |||
| Bovine serum albumin (BSA) | Multicell | 800-095-EG | |
| Coplin jars (EASYDIP SLIDE STAINING SYSTEM) | Newcomersupply | 5300KIT | |
| Cover slides | Fisherbrand | 12-545E 22*50 | |
| Direct Red 80 | Sigma Aldrich | 365548 | |
| Donkey Serum | Sigma Aldrich | D9663 | |
| Ethanol 100% | |||
| Electric pressure cooker | Salton | ||
| Eosin | Leica Biosystems | 3801600 | CAUTION, eye irritation |
| Fast Green FCF | Sigma Aldrich | F7252 | CAUTION, harmful by inhalation, ingestion and skin absortion |
| FFPE section (4μm) slides | |||
| Glycine 0,1 M in PBS | |||
| Hematoxylin Stain Solution, Gil 1. Formulation, Regular Strength | Ricca Chemical Company | 3535-32 | |
| Holder (EasyDip Staining Jar Holder) | Newcomersupply | 5300RK | |
| Human Serum | Gemini | 22210 | |
| Humidity chamber | Millipore Sigma | Z670138-1EA | |
| Pap pen | abcam | ab2601 | |
| PBS | |||
| PBS-Tween 20 (0.1% v/v) | |||
| Permount Mounting Media | Fisher Chemical | SP15-500 | |
| Picric Acid 1.3 % | Sigma Aldrich | P6744 | CAUTION, skin and eye irritation |
| Picro-Sirius Red/Fast Green solution: Fast Green 0.1 % w/v + Sirius Red 0.2 % w/v in 1,3 % picric acid solution | |||
| Primary Antibody Anti-αSMA | Mouse IgG2a 1A4 | Sigma A2547 | Dilution 1/100 |
| Primary Antibody Anti-CD34 | Mouse IgG1 HPCA1/763 | Novus Biologicals NBP2-44568 | Dilution 1/250 |
| Primary Antibody Anti-Cytokeratin 8/18 | Rabbit EP17/EP30 | Agilent IR09461-2 | Ready to use |
| Primary Antibody Anti-CD68 | Mouse KP1 | Abcam ab955 | Dilution 1/200 |
| Primary Antibody Anti-Desmin | Rabbit Polyclonal | Invitrogen PA5-16705 | Dilution 1/200 |
| Primary Antibody Anti-CD4 | Mouse N1UG0 | Affymetrix 14-2444 | Dilution 1/250 |
| Primary Antibody Anti-FoxP3 | Rabbit 1054C | R & D MAB8214 | Dilution 1/100 |
| Primary Antibody Anti-MPO | Goat Polyclonal | R & D Systems AF3667 | Dilution 1/250 |
| Primary Antibody Anti-CD3 | Rabbit SP7 | Abcam ab16669 | Dilution 1/200 |
| Primary Antibody Anti-CD8 | Mouse C8/144B | Invitrogen 14-0085-80 | Dilution 1/200 |
| Secondary Antibody Donkey anti-mouse A488 | Polyclonal | Invitrogen A-21202 | Dilution 1/500 |
| Secondary Antibody Donkey anti-Rabbit A488 | Polyclonal | Invitrogen A-21206 | Dilution 1/500 |
| Secondary Antibody Donkey anti-goat A568 | Polyclonal | Invitrogen A-11057 | Dilution 1/500 |
| Secondary Antibody Donkey anti-rabbit A647 | Polyclonal | Invitrogen A-31573 | Dilution 1/500 |
| Secondary Antibody Rat anti-mouse IgG1 A647 | RMG1-1 | Biolegend 406618 | Dilution 1/500 |
| Secondary Antibody Goat anti-mouse IgG2a CF568 | Polyclonal | Sigma Aldrich SAB4600315 | Dilution 1/500 |
| Secondary Antibody Donkey anti-mouse DyLight 755 | Polyclonal | Invitrogen SA5-10171 | Dilution 1/500 |
| Secondary Antibody Donkey anti-rabbit DyLight 755 | Polyclonal | Invitrogen SA5-10043 | Dilution 1/500 |
| SDS | BioShop | SDS001,500 | CAUTION, oral skin and eye toxicity |
| Shandon multi-program robotic slide stainer | LabX | 11384903 | |
| Shandon Xylene Substitute, | Thermo Fisher Scientific | CA89413-336 | CAUTION, Flammable, skin and eye irritation, Harmful when inhaled |
| Shaking water bath | |||
| SlowFade Gold antifade reagent with DAPI | Invitrogen | S36938 | |
| Sodium Citrate Dihydrate | Millipore Sigma | 1545801 | CAUTION, eye irritation |
| Stripping Buffer: mix 20 ml 10% w/v SDS with 12.5 ml 0.5 M Tris-HCl (pH 6.8), and 67.5 ml ultra-pure water. Under a fume hood, add 800 uL of 2-mercapto ethanol (114,4 mM final concentration) | |||
| Triton X-100 | Sigma Aldrich | T8787-50ML | |
| Tris-HCl | BioShop | 77-86-1 | |
| Tween 20 | Fisher Scientific | BP337-500 | |
| VIS Software | Visiopharm | ||
| Whole slide scanner Olympus BX61VS | Olympus | Microscope: Olympus Slide Scanner BX61VS, 5 slides scanner, motorized stage, autofocus. Camera: Lightsource: Xcite-120. Filters: BrightLine® Sedat filter set (# LED-DA/FI/TR/Cy5-4X4M-B-000, Semrock) | |
| Xylene | Sigma Aldrich | 214736-4L | CAUTION, Flammable, skin and eye irritation, Harmful when inhaled |
| Xylene : Ethanol solution (1:1 v/v) | |||
| 2-mercaptoethanol | Sigma | M6250 | CAUTION, harmful by ingestion, inhalation, fatal if sking absortion. Eye irritation. Use fume hood |
参考文献
- Greten, F. R., Grivennikov, S. I. Inflammation and Cancer:Triggers, Mechanisms, and Consequences. Immunity. 51 (1), 27-41 (2019).
- Pages, F., et al. International validation of the consensus Immunoscore for the classification of colon cancer: a prognostic and accuracy study. Lancet. 391 (10135), 2128-2139 (2018).
- Binnewies, M., et al. Understanding the tumor immune microenvironment (TIME) for effective therapy. Nature Medicine. 24 (5), 541-550 (2018).
- Taube, J. M., et al. Implications of the tumor immune microenvironment for staging and therapeutics. Modern Pathology: an official journal of the United States and Canadian Academy of Pathology, Inc. 31 (2), 214-234 (2018).
- Bindea, G., et al. Spatiotemporal dynamics of intratumoral immune cells reveal the immune landscape in human cancer. Immunity. 39 (4), 782-795 (2013).
- Galon, J., et al. Towards the introduction of the 'Immunoscore' in the classification of malignant tumours. The Journal of Pathology. 232 (2), 199-209 (2014).
- Finotello, F., Eduati, F. Multi-Omics Profiling of the Tumor Microenvironment: Paving the Way to Precision Immuno-Oncology. Frontiers in Oncology. 8, 430(2018).
- Gerner, M. Y., Kastenmuller, W., Ifrim, I., Kabat, J., Germain, R. N. Histo-cytometry: a method for highly multiplex quantitative tissue imaging analysis applied to dendritic cell subset microanatomy in lymph nodes. Immunity. 37 (2), 364-376 (2012).
- Giesen, C., et al. Highly multiplexed imaging of tumor tissues with subcellular resolution by mass cytometry. Nature Methods. 11 (4), 417-422 (2014).
- Porta Siegel, T., et al. Mass Spectrometry Imaging and Integration with Other Imaging Modalities for Greater Molecular Understanding of Biological Tissues. Molecular Imaging and Biology : MIB: the official publication of the Academy of Molecular Imaging. 20 (6), 888-901 (2018).
- Buchberger, A. R., DeLaney, K., Johnson, J., Li, L. Mass Spectrometry Imaging: A Review of Emerging Advancements and Future Insights. Analytical Chemistry. 90 (1), 240-265 (2018).
- Pirici, D., et al. Antibody elution method for multiple immunohistochemistry on primary antibodies raised in the same species and of the same subtype. Journal of Histochemistry and Cytochemistry. 57 (6), 567-575 (2009).
- Gendusa, R., Scalia, C. R., Buscone, S., Cattoretti, G. Elution of High-affinity (>10-9 KD) Antibodies from Tissue Sections: Clues to the Molecular Mechanism and Use in Sequential Immunostaining. Journal of Histochemistry and Cytochemistry. 62 (7), 519-531 (2014).
- van der Loos, C. M. Multiple immunoenzyme staining: methods and visualizations for the observation with spectral imaging. Journal of Histochemistry and Cytochemistry. 56 (4), 313-328 (2008).
- Stack, E. C., Wang, C., Roman, K. A., Hoyt, C. C. Multiplexed immunohistochemistry, imaging, and quantitation: a review, with an assessment of Tyramide signal amplification, multispectral imaging and multiplex analysis. Methods. 70 (1), 46-58 (2014).
- Toth, Z. E., Mezey, E. Simultaneous visualization of multiple antigens with tyramide signal amplification using antibodies from the same species. Journal of Histochemistry and Cytochemistry. 55 (6), 545-554 (2007).
- Robertson, D., Savage, K., Reis-Filho, J. S., Isacke, C. M. Multiple immunofluorescence labeling of formalin-fixed paraffin-embedded (FFPE) tissue. BMC Cell Biology. 9, 13(2008).
- Segnani, C., et al. Histochemical Detection of Collagen Fibers by Sirius Red/Fast Green Is More Sensitive than van Gieson or Sirius Red Alone in Normal and Inflamed Rat Colon. PloS One. 10 (12), 0144630(2015).
- Bolognesi, M. M., et al. Multiplex Staining by Sequential Immunostaining and Antibody Removal on Routine Tissue Sections. Journal of Histochemistry and Cytochemistry. 65 (8), 431-444 (2017).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved