Method Article
長距離逆PCRによるホットLINE-1のレトロトランスポジション活性の検出
要約
この記事では、活性LINE-1レトロトランスポゾンの活性を監視し、所定のゲノム内のデノボレトロトランスポジションをマッピングするための簡単なPCRベースのアッセイについて概説する。MCF7細胞株を用いて、この方法を22q12.1にあるLINE-1の活動を検出するためにどのように適用できるかを示す。
要約
長く散在する核要素1(LINE-1)は、自律的に移動できるヒトゲノムの移動遺伝元素の唯一のファミリーです。彼らは逆転写と呼ばれるプロセスによって行われ、その結果、逆転転写によってゲノムに挿入されるmRNA中間体を形成するために転写する。正常な細胞では沈黙しているにもかかわらず、LINE-1は異なる上皮腫瘍において非常に活性である。デ・ノボLINE-1挿入は腫瘍形成を引き起こす可能性があるため、がんにおけるLINE-1レトロトランスポジションを体系的に研究することが重要です。ヒトゲノムに存在する約150のレトロトランスポジション能力を持つLINE-1のうち、"ホット"LINE-1とも呼ばれるLINE-1遺伝子座はほんの一握りで、異なるがんタイプにおけるデノボLINE-1挿入の大部分を占めています。これらのホットLINE-1のレトロトランスポジション活性をモニタリングする簡易ポリメラーゼ連鎖反応(PCR)ベースの方法を開発した。この方法は、長距離逆(LDI)-PCRに基づいて、LINE-1が横方向の非反復領域を動員するメカニズムである3~3のトランスダクションを利用し、その後、デノボLINE-1 3 3のトランスダクションイベントを識別するために使用することができます。特定のホットLINE-1に由来します。
概要
デノボLINE-1挿入を検出する既存の方法は、(1)全ゲノムシーケンシングアプローチ4、7、8、異なる計算アルゴリズムを使用してde novo LINE-1挿入を見つけるために使用されますWGSデータ、または(2)若い、潜在的にアクティブなLINE-1s 9、10、11、12、13の3~の終わりをターゲットとする次世代シーケンシング。しかし、これらの方法を用いた数千近い同一コピーの中から新しい挿入を見つけることは些細なものであり、LINE-1挿入4に伴う腫瘍の不均一性とゲノム変化によってさらに悪化する。
これらの既存の方法を用いて行った研究では、腫瘍7、8で観察されたデノボLINE-1挿入の大部分を占めるLINE-1がほんのわずかであることを示した。したがって、特定の腫瘍サンプルがLINE-1活性を示すかどうかを答えるために、この一握りの非常に活発なLINE-1遺伝子座によって引き起こされるレトロトランスポジションイベントをマッピングするだけで十分である。本稿では、大腸癌で高活性である22q12.1でTTC28遺伝子の第1イントロンにおける特定のLINE-1遺伝子座の活性を監視するために使用できる単純なポリメラーゼ連鎖反応(PCR)ベースの方法14について説明する。7,8.このLINE-1遺伝子座は、記事全体を通じてTTC28-LINE-1と呼ばれます。このアッセイは、ソースLINE-1の3~横面領域に非反復配列を動員するデノボLINE-1レトロトランスポジションイベントを、3~トランスダクション15と呼ばれるメカニズムによって具体的に特定する。3 3 3 3 0/4 トランスダクションは、転写機械がそれをスキップし、代わりにより強いPAS下流で転写を終了させる弱いLINE-1ポリアデニル化信号(PAS)のために起こり、したがって、横向きの非反復配列((その後、LINE-1シーケンスと並んでターゲットの位置に挿入される「ユニークタグ」と呼ばれます。Philippe et al.16は最近、異なる細胞型が異なる LINE-1 遺伝子座を発現できることを示した。この知見を踏まえ、がん型の独自のタグを動員する最も高い表現力を持つLINE-1の活動をモニタリングするために、この手法を適用することができる。
LDI-PCRの最初のステップは、アッセイされるLINE-1を含む制限断片を生成する制限酵素を有するゲノムDNAの消化であり(ここでは、TTC28-LINE-1)とそのユニークなタグ(図1)。消化されたDNAは、自己ライゲーションによって円形化され、一意のタグ内にある逆プライマーを用いてPCR増幅される。そうすることで、「ネイティブ」の位置にあるフルレングスソースLINE-1が常に増幅され、それと一緒に、一意のタグを含む異なるターゲット遺伝子座での子孫LINE-1挿入も増幅されます(図1)。問題のLINE-1の遡及活動。
プロトコル
この研究は、ヘルシンキ大学病院の機関審査委員会と倫理委員会によって承認されました。署名されたインフォームドコンセントは、このプロトコルを実証するために使用される血液サンプルの被験者から得られた。
1. インバースプライマーの設計と制限酵素の選択(バイオインフォマティクス)
- LINE-1 関連の一意のタグの判別
- L1Base17などの LINE-1 データベースから FASTA 形式でTTC28-LINE-1 シーケンスをダウンロードします。TTC28-LINE-1 の L1 ベース ID は 135 です。
- LINE-1シーケンスの5~3/4の両端を横切る5 kbシーケンスを含め、ワードプロセッサでアニテートします。
注: ここでは、LINE-1 の横面シーケンスに茶色のフォントで注がれ、LINE-1 シーケンスは灰色のフォント (補足ファイル)で表記されています。 - 選択した LINE-1 の認識 PAS の下流の 1 kb シーケンスをポリアドック18やドラゴン ポリA スポッター19などの PAS 予測ツールに入力し、この 1 kb ウィンドウのすべてのポリアデニル化信号に注釈を付けます。
注: 1 kb ウィンドウに PAS がない場合は、次の 1 kb ウィンドウダウンストリームで PAS を検索します。TTC28-LINE-1自身の弱いPASはピンクでハイライトされ、下流の1 kbウィンドウの他のすべてのPASは赤で強調表示されます(補足ファイル)。 - LINE-1の認識PASの終わりと最強のPASダウンストリームの間のシーケンスに「ユニークなタグ」としてアニュテーします。
注:TTC28-LINE-1の「一意のタグ」は黄色(補足ファイル)で強調表示されます。
- 逆プライマーの設計
- プライマー3(http://bioinfo.ut.ee/primer3-0.4.0/)やNCBIのプライマー-BLAST(https://www.ncbi.nlm.nih.gov/tools/primer-blast/)のようなウェブベースのプライマー設計ツールに「ユニークなタグ」シーケンスを入力して逆PCRプライマーを設計します。これらのツールによって設計されたプライマーペアは、従来のPCRを容易にする互いに向き合っているので、プライマーペアの逆補数機能を使用して逆PCRを実行します。
注:LINE-1トランスダクションは5~00分の1の端部で大幅に切り捨てられ、変換領域の大きさは非常に変動しているので、NCBIプライマー-BLASTの「PCR製品長」パラメータを設定することで、2つの逆プライマー間の距離を最小限に抑えることを目指します。最小。一意のタグ内に複数の PAS がある場合は、異なる PAS に対応する複数のプライマー ペアを設計します。RNA中間体の3~3~の端からLINE-1挿入が開始され、5~の端が可変的に切り捨てられるように、PASに近い設計プライマー。ここでは、TTC28 LINE-1(補足ファイル)のユニークなタグで3つの強いポリアデニル化信号に対応し、ティールとグリーンで強調表示された3つのプライマーペアを設計しました。
- プライマー3(http://bioinfo.ut.ee/primer3-0.4.0/)やNCBIのプライマー-BLAST(https://www.ncbi.nlm.nih.gov/tools/primer-blast/)のようなウェブベースのプライマー設計ツールに「ユニークなタグ」シーケンスを入力して逆PCRプライマーを設計します。これらのツールによって設計されたプライマーペアは、従来のPCRを容易にする互いに向き合っているので、プライマーペアの逆補数機能を使用して逆PCRを実行します。
- 制限酵素の選択
- 制限Mapperなどのウェブベースのツールを使用して、シリコの5 kbの上流および下流の側面と一緒にLINE-1シーケンスをダイジェストします。これにより、この領域を消化する制限酵素の包括的なリストが提供され、異なる制限断片が生成されます。
- LINE-1のネイティブ軌跡をカットする制限酵素を選択します:5~5の端で、LINE-1自体の上流または遠い5~5の端、そしてLINE-1のユニークなタグの下流の3~終わりです。
注:選択された制限酵素は、DNAメチル化に対して無感覚であるべきであり、熱不活性化であるべきであり、互いに相補的であるずらりと「粘着性」端を生成する必要があります。LDI-PCRを実証するために、GAGCTC部位でDNAを切断するSacI制限酵素が、ここで明るい緑色で強調表示され、使用される(補足ファイル)。 - 選択した制限酵素によって作られた制限断片サイズに注意してください。これは、PCR によって効率的に増幅されない可能性があるため、12 kb より長くしないでください。
2. 長距離逆PCR用の円形DNAテンプレートの作成
- DNA抽出と品質評価
- 市販のDNA抽出キットを用いてサンプル(腫瘍または血液)からゲノムDNAを抽出し、製造元の指示に従ってLDI-PCRに必要な高品質、高分子量DNAを抽出することができます。あるいは、高分子量DNAは、フェノール:クロロホルム20によって抽出することもできる。
- メーカーの指示に従ってフッ素計を用いてDNA濃度を測定し、1xトリスアセテートEDTA(TAE)バッファーで1%(w/v)のアガロースゲルを1%(w/v)で100ngのDNAを実行し、λ-HindIIIと並んで4.5V/cm1のバッファーを使用します。DNAの質と量をチェックするDNA分子量マーカー。
- ゲノムDNAの消化
- 消化反応ミックス(50μLの最終容積)を20単位のSacI制限酵素、10倍の反応バッファーの5μL(材料表)、100ngのDNA(最大44μL)を氷上の0.2 mL PCRチューブ(ほとんどのメーカーから制限酵素の1μL)を加えることで、消化反応ミックスを作ります。ゲノムDNAの100ngを完全に消化するのに十分です)。チューブをフリックして溶液を混ぜ、遠心分離機を短時間混ぜます。
- サーマルサイクラーを使用して、反応ミックスを1時間37°Cでインキュベートし、その後65°Cで5分間熱不活性化を行います。
- 消化されたゲノムDNAを自己合振
- 50 μL消化ミックス(ステップ2.2.2の後)に、10x T4 DNAリゲスバッファーの8 μL、T4 DNAリゲスの1μL(5単位)、および21 μLの超純水を加え、80 μLの最終反応量を作り、チューブをフリックして溶液を混合します。、遠心分離機を簡単に。
- 22 °Cで熱サイクラーで10分間インキュベートし、65°Cで10分間熱不活性化工程で終端する。
3. 長距離逆PCR
- グラデーション PCR によるプライマー アニーリング温度の決定
- (A-4) °C, (A-2)°C, A, (A+2)°C, (A+4) °C のアニーリング温度の勾配を設定します。
注: 一部のメーカーのサーマルサイクラーでは、温度勾配を手動で設定できない場合があります。その場合、自動勾配設定は、(A-4) °C から (A+4) °C までの温度範囲で使用できます。 - 1.5 mLマイクロ遠心分離管で次のコンポーネントを組み合わせて混合することにより、PCRのマスターミックスを準備する:5x反応バッファーの4 μL、10 mM dNTPの0.4 μL、2 μM PCRプライマーの5 μL(ステップ1.3.1で設計された前方および逆)。、反応あたりのDNAポリメラーゼの0.2 μL(0.1 U)。グラデーション内のアニーリング温度ごとに1つの反応を設定します。
- 各反応について、マスターミックスのアリコート19 μLを0.2 mL PCRチューブに加え、セクション2で作られた円形DNAテンプレートの1 μL(1.25 ng)を添加する。
注:この最適化ステップで潜在的に貴重な腫瘍DNAを消費しないように、通常の血液DNAから生成された円形の自己ライゲーションDNAをテンプレートとして使用してください。 - 以下に説明するように、サーマルサイクラー上でグラデーションPCRプログラムを実行します: (i) 98 °C(脱彩)で3分の1サイクル。(ii) 35サイクル(98°C[変性]で10秒)、[A-4]~[A+4]の温度勾配で20秒、72°Cで1−6分[予想PCR産物の1キロベース当たり30秒]。(iii)72°C(最終延長)で10分の1サイクル。
- 4.5V/cmで1x TAEバッファーで調製した1%アガロースゲル21でPCR生成物の6μLを実行し、異なるアニーリング温度で得られたPCR製品を分析します。
- 期待サイズに対応するPCR製品を生成するアニーリング温度を選択します。
- (A-4) °C, (A-2)°C, A, (A+2)°C, (A+4) °C のアニーリング温度の勾配を設定します。
- 腫瘍ゲノムにおけるデノボLINE-1レトロトランスポジション活性の検出
- 腫瘍サンプルから生成された円形DNAテンプレート上の逆PCRプライマーペアを使用してLDI-PCRを実行します(セクション2)。グラデーションPCR(セクション3.1)と同じ手順に従いますが、今回は温度勾配を最適なアニーリング温度に置き換えます。
- ステップ 3.1.5 で行われたように、アガロース ゲル電気泳動によって PCR 製品を分析します。既知のサイズのPCR産物またはそのネイティブ遺伝子座でLINE-1に対応する「ネイティブ」PCR産物は、各反応について可視化されるべきである。デ・ノボアッセイされた腫瘍試料中のLINE-1 3333/4の経電は、アガロースゲル中の天然PCR産物と共に、異なるサイズのPCR産物として検出可能である。
4. LDI-PCR製品のシーケンシングを行い、LINE-1 3 3~の対象サイトのアイデンティティを明らかにする
- 各LDI-PCR反応で発生したすべてのPCRアンプリコンの単分子長読シーケンシングを実行し、これらのLINE-1 33のトランスダクションイベントの標的積分部を特定する。
注: LDI-PCR 製品のクローニングとサンガー シーケンスも可能ですが、面倒なアプローチです。 - 標準配列アライメントパイプラインを使用して、単分子の長い読み取りシーケンスプラットフォームによって生成された読み取りを参照ゲノムに合わせます。LDI-PCRソフトウェア14を用いて整列読み取りを分析し、デノボLINE-1挿入とそのターゲットサイトを特定する。
結果
TTC28-LINE-1 の場合、認識 PAS の下流 1 kb ウィンドウ内に複数の PASがあるため、TTC28-LINE-1 PAS と 811 bp 下流の最も強い PAS の間の領域は、TTC28-LINE-1 の固有のタグと見なされました。3つの逆PCRプライマーペアは、存在する異なるPASに対応するこのユニークなタグで設計されました14.TTC28-LINE-1 の上流 5 をカットするNsiI と、(ii) SacI および (iii) PstI の 3 つの制限酵素を選択しました。それぞれ 10,288 bp、5,699 bp、および 6,305 bp の制限フラグメントが生成されます。
この方法を実証するために、MCF7細胞株から抽出したDNAに対してLDI-PCRを行った。この乳癌細胞株は、TTC28-LINE-1活性16を表示することが以前に報告されている。 簡略化のため、3つのうち1つの制限酵素であるSacIを用いて円形DNAテンプレートを作成し、3つのうち1つのプライマーペア(表1)を使用してLDI-PCRを行い、TTC28に由来するデノボLINE-1挿入物を検出した-ライン-1。
MCF7細胞株から抽出されたDNAの良好な品質は、アガロースゲル電気泳動によって確保された(図2)。無傷の高分子量DNAは、ゲノムDNAがこのアッセイに最適な品質であることを示す。スミアが代わりに見える場合、これは抽出されたDNAの品質が悪いことを示し、下流の手順を妨げる。
図3は、TTC28-LINE-1逆プライマーペアの最適なアニーリング温度を決定することを目的とした勾配PCR実験の代表的な結果を示す。SacIで消化された血液ゲノムDNAは、続いて自己ライゲーションを経て円形DNAテンプレートを形成し、この反応に用いた。62、64および66 °Cの期待されるサイズ(5,649 bp)の非常に特異的なPCRプロダクトは、このプライマー対の最適なアニーリング温度が62−66 °Cの範囲内にあることを示す。
MCF7ゲノムDNAをSacI制限酵素で消化し、その後自己ライゲーションを行い、循環DNAテンプレートを生成した。図4は、TTC28-LINE-1 3 3 33°CがMCF7細胞株で起こることを示しています:デノボ挿入は、既知のサイズ(5,649 bp)のネイティブPCR産物と共に、様々なサイズのLDI-PCR産物として検出することができる。 デノボ標的部位のゲノム座標を同定するために、PCRアンプリコンを配列することができる(プロトコルセクション4参照)。

図 1: LINE-1 3 3の経度を検出するLDI-PCRの概要円形DNAテンプレートは、最初に(I)制限酵素および自己合流(II)を伴って消化することによって生成される。このステップの後に、インタレストのLINE-1のユニークなタグ(LINE-1自身の弱いPAS、ピンク、より強いPASの下流、赤)をターゲットにした逆PCRプライマーを持つ逆PCR(III)が続きます。この図のより大きなバージョンを表示するには、ここをクリックしてください。

図 2: 抽出されたDNAの品質評価コントロールサンプルとして使用される正常な個体からMCF7細胞株と血液から抽出された100ngのDNAを、M.としてラベル付けされたλ DNA/HindIIIマーカーの1μLと2μLと一緒に実行したのを見るには、こちらをクリックしてこの図のより大きなバージョンを表示してください。

図 3: 逆PCRプライマーに最適なアニーリング温度を決定する勾配PCR(表1)。56~66°Cのアニーリング温度で逆プライマーペアを用いたLDI-PCRは、62−66°Cで異なるPCR産物を示しています。緑色の矢印は、将来の実験のために選択されたアニーリング温度を示します。この最適化工程には、SacIを用いて正常な個体から血液ゲノムDNAを消化して生成した円形DNAテンプレートを使用し、その後自己ライゲーションを行った。M、マーカー(1kbプラスDNAはしご)。この図のより大きなバージョンを表示するには、ここをクリックしてください。
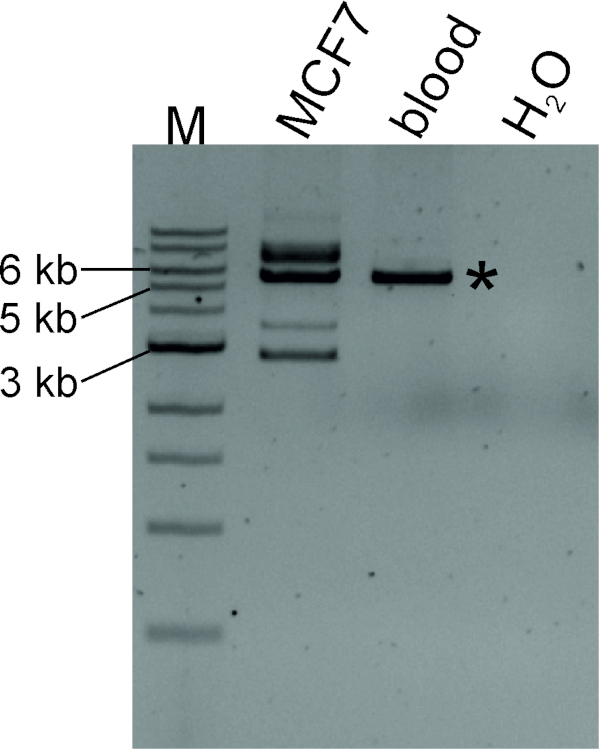
図 4: LDI-PCRを同定するLINE-1 3 3 3 3に起因するトランスダクションTTC28-ライン-1。MCF7と血液(正常個体由来)をSacIで消化して生成した円形DNAテンプレートを、自己ライゲーションに続いて、最適なアニーリング温度で逆プライマーにより増幅した。期待サイズ(5,649bp)のアスタリスクを付いた「ネイティブ」PCR製品は、MCF7 DNAと正常な血液DNAの両方で検出され、MCF7はまた、様々なサイズの追加のPCR産物を生産し、de novo LINE-1レトロトランスポジションを示します。M、マーカー(1kb DNAはしご)。この図のより大きなバージョンを表示するには、ここをクリックしてください。
| プライマー名 | シーケンス (5/ → 3' |
| L1_001 (改訂版) | TTCACTAAGGTGTGAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA |
| L1_002 (fwd) | CCCAAAATATATCAATTATGGCA |
表 1: 固有のタグ用に設計された逆 PCR プライマー ペアTTC28-ライン-114歳.
ディスカッション
ここでは、関心のある任意のアクティブなLINE-1に起因するデノボLINE-1挿入を識別するために使用できる方法を説明します。我々は、22q12.1に位置する非常に活性なLINE-1のためにこの方法を最適化し、以前に大腸癌14におけるサブクローン挿入を検出する上で高感度であることを実証した。
LDI-PCRの成功はゲノムDNAの質に依存する。したがって、プロトコルの開始時に高分子量DNAが存在することを保証するための追加の品質管理ステップが含まれています(ステップ2.1.2)。長期保存のために-20°CでゲノムDNAを保存し、凍結解凍のサイクルを避けるためにアリコートを調製することをお勧めします。血液または患者と一致した正常組織からのゲノムDNAを使用して、検出されたLINE-1レトロトランスポジションが生殖細胞系または体細胞イベントであるかどうかを区別することを強くお勧めします。制限酵素の切断部位はゲノム中で確率的であるため、特定のde novo LINE-1挿入部位は、その近傍で使用されている制限酵素の切断部位を収容しない可能性があります。したがって、腫瘍DNA中のデノボLINE-1挿入物の大部分を検出する可能性を高めるために、複数の制限酵素を別々の反応で使用して、円形DNAテンプレートの異なるライブラリーを生成する必要があります。さらに、対象のLINE-1のユニークなタグが複数のPASを持っている場合、各PASに隣接するプライマーペアを使用すると、重く切り捨てられたトランスダクションを検出する可能性が向上します。
デノボLINE-1挿入物のゲノム全体検出のためのエレガントな方法は存在するが、特定の細胞コンテキストにおける特定のLINE-1のレトロトランスポジション能力をプローブすることを目的とすれば、それらは圧倒的である可能性がある。このため、LDI-PCRは、LINE-1レトロトランスポジションイベントを可視化するための安価でシンプルで堅牢なアプローチです。この方法で使用されるターゲティングアプローチは、TS-ATLAS22に似ています。しかし、LDI-PCRはリンカーオリゴヌクレオチドの使用を回避し、デノボLINE-1挿入の5~3~のジャンクションを同時に増幅することができます。LINE-1挿入の5~3/4ジャンクション、統合の対象部位、ポリAテールおよびターゲットサイトの変更に関する情報は、いずれもLINE-1レトロトランスポジションの特徴であり、LDI-PCRを単一分子と結合することで得ることができる。長読みシーケンシング技術。このように生成された長い読み取りには、挿入されたLINE-1シーケンス、その一意のタグ、およびターゲットシーケンスが1回の読み取りで含まれ、繰り返し領域での短い読み取りのマッピングの難しさを回避します。
LINE-1のアクティビティを検出するためにLDI-PCRを使用することには、2つの大きな制限があります。1 つ目は PCR に固有のものです: 最大 10 kb のフラグメントを確実に増幅できます。ネイティブフラグメントがこの制限を超えてはならないため、制限酵素を選択する際に考慮する必要があります。第二に、この方法は、LINE-1の33の側面領域を3/4のトランスダクションによって動員するレトロトランスポジションイベントのみを検出することができます。したがって、この方法では、3~3分の1のトランスダクションを示さないLINE-1の活動は検出されません。さらに、LDI-PCRによって増幅されているにもかかわらず、(a)「ネイティブ」位置または他のレトロトランスポジションと同様のサイズのPCRターゲットを生成する一部のLINE-1レトロトランスポジションイベント、または(b)まれまたはサブクローナルであり、アガロースゲルによって検出されない場合があります。電気 泳 動。このようなLINE-1レトロトランスポジションイベントは、単分子長読シーケンシング技術14を用いてLDI-PCR産物をシーケンシングすることによって捕捉することができる。
ここで説明するワークフローは、適切な制限酵素を使用し、これらのLINE-1をターゲットとする逆プライマーを設計することにより、他の「ホット」LINE-1の活性を検出するために簡単に変更することができます。この方法は、LINE-1媒介3~3/4トランスダクションの検出に加えて、より頻度の低いLINE-1媒介5~3/4トランスダクション23を検出するように適合させることができる。同様の方法は、細胞ベースのアッセイ24および癌25におけるプロウイルス統合部位におけるLINE-1レポーターの統合部位を同定するために用いられている。この方法は、LINE-1挿入に加えて、DNA再配置などの他のゲノム収差を検出するためにも利用することができ、再配置が起こりやすい領域に関する情報が存在する26。
開示事項
著者は何も開示していない。
謝辞
この方法が最初に説明された記事の共同執筆者全員、特にタチアナ・カジュソ、キムモ・パリン、ウティ・キルピヴァーラ、エサ・ピトケネンの共著者の皆さんに感謝します。L.K.は、フィンランドアカデミー(助成番号25996、292789、306026および314394)、シグリッド・ジュセリウス財団、フィンランド癌協会によって資金提供されています。B.P.は、ヘルシンキ大学研究財団博士課程、フィンランド癌学会論文交付金、およびイダ・モンティニン・セーティオ博士研究助成金を受けています。また、L.K.の研究グループのテム・マサリン(ヘルシンキ大学)とクル・シュレスタ氏に、映像制作を支援してくださったことに感謝します。
資料
| Name | Company | Catalog Number | Comments |
| 1 Kb DNA Ladder | New England Biolabs | N3232L | |
| 10 mM dNTP | ThermoFisher Scientific | 18427013 | |
| Acetic acid | ThermoFisher Scientific | 64-19-7 | Used to make TAE buffer |
| Agarose | BioNordika | BN-50004 | |
| Blood sample (frozen) | Blood sample from a healthy individual for control PCR | ||
| ChemiDoc XRS+ System | Bio-rad | 1708265 | |
| DNA Gel Loading Dye (6X) | ThermoFisher Scientific | R0611 | |
| DNeasy Blood & Tissue Kits | Qiagen | 69504 | |
| Ethidium Bromide | Bio-rad | 161-0433 | |
| Ethylenediaminetetraacetic acid (EDTA) | ThermoFisher Scientific | 25102-12-9 | Used to make TAE buffer |
| FastDigest buffer | ThermoFisher Scientific | B64 | |
| FastDigest SacI | ThermoFisher Scientific | FD1133 | |
| Generuler 1 Kb plus DNA Ladder | ThermoFisher Scientific | SM1331 | |
| Generuler Lambda DNA/HindIII Marker, 2 | ThermoFisher Scientific | SM0103 | |
| Mini-Sub Cell GT Cell | Bio-rad | 1704406 | |
| Phusion Green Hot Start II High-Fidelity DNA Polymerase | ThermoFisher Scientific | F537L | |
| PowerPac Basic Power Supply | Bio-rad | 1645050 | |
| Quantus Fluorometer | Promega | E6150 | |
| T4 DNA Ligase | ThermoFisher Scientific | EL0011 | |
| Tear-A-Way 96/8, 96 Well PCR Plate | 4titude | 4ti-0750/TA | |
| Tris(hydroxymethyl)aminomethane | ThermoFisher Scientific | 77-86-1 | Used to make TAE buffer |
| Veriti Thermal Cycler | Applied Bioscience | 4375786 |
参考文献
- Lander, E. S., et al. Initial sequencing and analysis of the human genome. Nature. 409 (6822), 860-921 (2001).
- Brouha, B., et al. Hot LINE-1s account for the bulk of retrotransposition in the human population. Proceedings of the National Academy of Sciences of the United States of America. 100 (9), 5280-5285 (2003).
- Goodier, J. L. Restricting retrotransposons: a review. Mobile DNA. 7, 16 (2016).
- Lee, E., et al. Landscape of somatic retrotransposition in human cancers. Science. 337 (6097), 967-971 (2012).
- Miki, Y., et al. Disruption of the APC gene by a retrotransposal insertion of L1 sequence in a colon cancer. Cancer Research. 52 (3), 643-645 (1992).
- Scott, E. C., et al. A hot L1 retrotransposon evades somatic repression and initiates human colorectal cancer. Genome Research. 26 (6), 745-755 (2016).
- Pitkanen, E., et al. Frequent L1 retrotranspositions originating from TTC28 in colorectal cancer. Oncotarget. 5 (3), 853-859 (2014).
- Tubio, J. M., et al. Mobile DNA in cancer. Extensive transduction of nonrepetitive DNA mediated by L1 retrotransposition in cancer genomes. Science. 345 (6196), 1251343 (2014).
- Badge, R. M., Alisch, R. S., Moran, J. V. ATLAS: a system to selectively identify human-specific L1 insertions. American Journal of Human Genetics. 72 (4), 823-838 (2003).
- Rodic, N., et al. Retrotransposon insertions in the clonal evolution of pancreatic ductal adenocarcinoma. Nature Medicine. 21 (9), 1060-1064 (2015).
- Ewing, A. D., Kazazian, H. H. High-throughput sequencing reveals extensive variation in human-specific L1 content in individual human genomes. Genome Research. 20 (9), 1262-1270 (2010).
- Sanchez-Luque, F. J., Richardson, S. R., Faulkner, G. J. Retrotransposon Capture Sequencing (RC-Seq): A Targeted, High-Throughput Approach to Resolve Somatic L1 Retrotransposition in Humans. Methods in Molecular Biology. 1400, 47-77 (2016).
- Zhao, B., et al. Somatic LINE-1 retrotransposition in cortical neurons and non-brain tissues of Rett patients and healthy individuals. PLOS Genetics. 15 (4), e1008043 (2019).
- Pradhan, B., et al. Detection of subclonal L1 transductions in colorectal cancer by long-distance inverse-PCR and Nanopore sequencing. Scientific Reports. 7 (1), 14521 (2017).
- Moran, J. V., DeBerardinis, R. J., Kazazian, H. H. Exon shuffling by L1 retrotransposition. Science. 283 (5407), 1530-1534 (1999).
- Philippe, C., et al. Activation of individual L1 retrotransposon instances is restricted to cell-type dependent permissive loci. Elife. 5, (2016).
- Penzkofer, T., et al. L1Base 2: more retrotransposition-active LINE-1s, more mammalian genomes. Nucleic Acids Research. 45 (D1), D68-D73 (2017).
- Tabaska, J. E., Zhang, M. Q. Detection of polyadenylation signals in human DNA sequences. Gene. 231 (1-2), 77-86 (1999).
- Kalkatawi, M., et al. Dragon PolyA Spotter: predictor of poly(A) motifs within human genomic DNA sequences. Bioinformatics. 29 (11), 1484 (2013).
- Sambrook, J., Russell, D. W. Purification of nucleic acids by extraction with phenol:chloroform. Cold Spring Harbor Protocols. 2006 (1), (2006).
- Green, M. R., Sambrook, J. Agarose Gel Electrophoresis. Cold Spring Harbor Protocols. 2019 (1), (2019).
- Macfarlane, C. M., et al. Transduction-specific ATLAS reveals a cohort of highly active L1 retrotransposons in human populations. Human Mutation. 34 (7), 974-985 (2013).
- Beck, C. R., Garcia-Perez, J. L., Badge, R. M., Moran, J. V. LINE-1 elements in structural variation and disease. Annual Review of Genomics and Human Genetics. 12, 187-215 (2011).
- Morrish, T. A., et al. DNA repair mediated by endonuclease-independent LINE-1 retrotransposition. Nature Genetics. 31 (2), 159-165 (2002).
- Li, J., et al. Leukaemia disease genes: large-scale cloning and pathway predictions. Nature Genetics. 23 (3), 348-353 (1999).
- Pradhan, B., et al. Detection and screening of chromosomal rearrangements in uterine leiomyomas by long-distance inverse PCR. Genes, Chromosomes and Cancer. 55 (3), 215-226 (2016).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved