Method Article
キイロタマホコリカビの選択に基づいて細胞と細菌の成長の遺伝子工学
要約
キイロタマホコリカビは細胞遊走、エンドサイトーシス、開発など複雑な細胞プロセスを勉強する人気モデル生物です。生物のユーティリティは、遺伝子操作の可能性に依存しています。液体培地における培養細胞の既存の制限を克服するキイロタマホコリカビの細胞を使っての方法を紹介します。
要約
キイロタマホコリカビは開発、細胞のシグナル伝達、および他の重要な細胞生物学の質問中に細胞分化プロセスの調査のための魅力的なモデル生物です。細胞性粘菌を遺伝子操作するための技術はよく発達しました。Transfections は、別の選択可能なマーカーとマーカーの再循環、相同組換えと挿入変異を含むを使用して実行できます。これはよく注釈ゲノムによってサポートされます。ただし、これらのアプローチは液体文化で育つ無菌細胞は最適化されており、細菌にのみフィード非無菌の野生型細胞に適用しにくい。無菌系統に存在する突然変異は、Ras シグナリング、授乳に必要な過剰マクロピノサイトーシスの原因を邪魔し、シグナル伝達とそれらの系統の化学走性実験の解釈を混同する細胞の移動を低下させます。非無菌細胞を遺伝子操作する以前の試み効率と複雑な実験手続きを欠いていた。これらの制限を克服して初めて、簡単なトランスフェクション プロトコルを開発しました。それら一連の細胞性粘菌の分子遺伝学に大きな改善を簡単に標準的な実験室の緊張として操作できる野生型細胞を許可します。破損していないシグナル伝達と運動性のプロセスを研究するための利点に加えてマクロピノサイトーシス ベースの成長を混乱させる突然変異体今容易に分離することができます。さらに、週間ではなく、日を生成する遺伝子組換え細胞と、全体のトランスフェクション ワークフローは大きく加速します。別の利点は、分子遺伝学を環境から新鮮単離の野生型細胞性粘菌のサンプルをさらに実行できます。これはこれらの研究分野で使用されているアプローチの範囲を拡張することができます。
概要
細胞性粘菌属は、主に細菌の餌土生活社会アメーバです。門アメーボゾアに置かれて、種の数が多いは、分離されている 4 つの異なるクレード1をグループ化できます。種キイロタマホコリカビ(D. キイロタマホコリカビ) 細胞遊走、貪食など複雑な細胞プロセスを勉強する人気モデル生物となっています。制御し、細菌2の不在で複雑なまたは定義された液体培地で成長することができる考案されている無菌の細胞実験条件を標準化します。特に重要な Ax3、ax2 横型、すべては 1970 年代に生成され、最終的に単一の野生から派生された Ax4 系統分離 NC43。19874,5で最初公開されたノックアウトの結果、これらの無菌系統の遺伝子工学のためのツールが開発されました。プロトコルさらに開発され無菌条件6、7の下での使用に最適します。
適応プロトコル野生型D. キイロタマホコリカビを液体スープで育つことができない系統は、いくつかの研究所で試行されています。しかし、トランスフェクション プロトコルは複雑であり、選択的試薬8,9のシンクとして機能する細菌の能力による部分で効率性に欠けるので完全成功したこのないなっています。その結果、 D. キイロタマホコリカビの本質的にすべて分子データは単一の野生型分離株の子孫から来ています。この制限を克服し、遺伝的液体媒体の成長する能力の独立したD. キイロタマホコリカビセルを変更する方法を開発したいです。そのような方法の必要性は、無菌の成長を許可する突然変異は主に中立的だった過去に就任したし、細胞生理学を損なわないか観察によって説明できます。この仮定は部分的に正しいのみです。一般に、2 つの顕著な違いがあります。最初、違い無菌系統と第二に、これらの無菌系統は非無菌野生菌株8,9と比較した時に分離されました。
おそらく最も重要な因子は、キー無菌遺伝子、斧B、RasGAP NF1 として最近識別されました。NF1、RasGAP としての主要な機能は、Ras アクティビティ3を抑制することです。すべて無菌系統で酵素の削除は、明らかに大規模なアクティブな Ras パッチ形成として過度の Ras の活性につながります。これらの拡大の Ras のパッチは、細胞膜で PIP3 の蓄積に します。PIP3 およびアクティブな Ras のそれらの偶然現れるパッチは、最終的に終了し、macropinosomes10の形成につながる円形フリルの形成のためのテンプレートです。結果は、macropinocytic の活動の過度な増加です。マクロピノサイトーシスはアクチン ドリブン プロセスです。Macropinosomes または仮足の細胞骨格分子の形成のための競争は、結果です。細胞挙動への影響は、葉酸11栄養細胞の走化性のほぼ完全な予防に反映されます。大きく拡大された PIP3 パッチが非常にしつこいです。でも飢餓状態の細胞、PIP3 パッチが残る、仮のキャンプに走化性に関する研究の解釈の問題を引き起こすことができると誤解されることができます。
場合によっては、NF1 変異実験に便利です。これは細菌によって成長したD. キイロタマホコリカビのトランスフェクション法を開発するための 2 番目の動機につながる細胞、マクロピノサイトーシス率の増加は、無菌細胞がこのプロセス12 の基本的な側面を調査するため貴重なので.ただし、マクロピノサイトーシス Ras と PI3 キナーゼ10などに必要な遺伝子の突然変異はほとんど細菌の成長を介してこれらの細胞を操作する必要がある、無菌の成長を廃止しています。貴重な細菌ベースの transfections をレンダリングする別の理由は多細胞型13,14、血縁認識15,16進化の質問を探検する細胞性粘菌の使用の増加新鮮単離野生型の使用に主に依存する利他的な細胞行動され17。すべては、研究分野が非無菌と液体スープで、成長しない野生株の遺伝的操作のための効率的な方法で促進することを述べた。
説明の制限を克服するため私達のプロトコルを許可します。まとめると、高速ために選択プロセスの高速化だけだ場合でも、細菌の細胞性粘菌研究者のd. キイロタマホコリカビ細胞保持している利点を成長と遺伝子操作を実行の可能性無菌メディア (倍加時間 10 h) の成長と比較して菌のアメーバ (4 h 倍加時間) の成長。
プロトコル
1. セルと材料の準備
-
SorMC バッファーの準備
- 20.36 g KH2PO4 (15 mM) と 5.47 g ナ2HPO4·7 H2O (2 mM) ddH2O 水 100 mL に溶解して SorMC (ソレンセン MgCl2 CaCl2など) バッファー x 100 の 100 mL を準備します。常温 (RT) ソリューションをかき混ぜるし、dH2o. と 100 mL にボリュームをもたらす
注:結果バッファー 6 の pH がありさらに必要はありませんを調整します。 - DdH2O. 追加 50 μ L 各 MgCl2および CaCl2各 50 μ M の最終的な濃度を達成するのに作業ソリューション x 1 の 1000 mL を生成します。フィルターは、0.22 μ m フィルターを使用して、ソリューションを殺菌します。
注:常に 100 x 貯蔵液の塩類の析出を避けるため 1 x バッファーに MgCl2および CaCl2を追加します。 - また、50 μ M MgCl2を添加した KK2バッファー (KH2PO4 2.2 g と K2HPO4バッファーの 1 L 用の 0.7 g) と 50 μ M CaCl2 (ここ KK2MC と呼ばれる) を準備します。SorMC ではなく全体でこのバッファーを使用します。
- 20.36 g KH2PO4 (15 mM) と 5.47 g ナ2HPO4·7 H2O (2 mM) ddH2O 水 100 mL に溶解して SorMC (ソレンセン MgCl2 CaCl2など) バッファー x 100 の 100 mL を準備します。常温 (RT) ソリューションをかき混ぜるし、dH2o. と 100 mL にボリュームをもたらす
-
D. キイロタマホコリカビの食料源として細菌の準備
- 健食の単一コロニーを使用し、-LB 培地 (ホストゲノム スープ) の 1 L を接種します。2 L フラスコを使用します。220 rpm で振とうしながら 37 ° C で一晩成長細菌をしましょう。
注:大量の細菌が必要な場合は、2xTY のような豊かなメディアを使用して (酵母エキス トリプトン媒体) またはポンドではなくすすり泣き (超最適スープ)。に備えて安全性の制限のためは、健食菌は使用できません、BL21エシェリヒア属大腸菌を代わりに使用することができます。 - 〜 6,600 × g 20 分洗浄細菌 500 mL の SorMC バッファーに一度で 2 つの 500 mL 遠沈管でスピンダウン: それらによって次の日セルを収穫します。
- SorMC 20 mL にペレットを再懸濁します。外径600をチェック (光学密度 600 nm) 光度計を使用して。約 100 の OD600を同じバッファーで希釈します。
注:健食菌は、細菌をスピンダウンの比較的高速食品の細菌の損失を避けるために必要があるので、ペレットが困難です。結果細菌原液は冷蔵庫 4 ° C で最大 4 ヶ月間保存でき、 D. キイロタマホコリカビの食料源としてその有用性を維持します。一晩通常 LB 培地で栽培されたk. 水素生成菌懸濁液 1 L には、約 100 の OD600の 20 mL が得られます。
注意:準備細菌が健食のモノであることを確認するには、フードの下のすべての手順を実行します。
- 健食の単一コロニーを使用し、-LB 培地 (ホストゲノム スープ) の 1 L を接種します。2 L フラスコを使用します。220 rpm で振とうしながら 37 ° C で一晩成長細菌をしましょう。
-
H40 エレクトロポレーション バッファーの準備
- 緩衝液 100 mL を準備、ddH2O の水、HEPES の 0.952 g を溶解し、1 M 原液から MgCl2の 100 μ L を追加します。7 の pH を調整する KOH を用いた滴定。0.22 μ m のフィルターまたはオートクレーブを使用してバッファーを滅菌します。酸フリー HEPES とナトリウム塩ではない使用します。
- 次の製造元のプロトコルと材料表にまとめたキットを使用してプラスミッドの準備を実行します。表 1に要約プラスミドを使用します。
注:遺伝子導入用 DNA の品質は重要です。D. 菌の形質転換細胞の細菌の成長の選択に選択と式カセットを運転するプロモーターの特定の要件は、(議論を参照)。
| プラスミドの名称 | 細菌の抵抗/選択 | 細胞性粘菌で抵抗値/選択 | タグ |
| 染色体外発現プラスミド | |||
| pDM1203 | アンピシリン | G418 | 違います |
| pDM1207 | アンピシリン | G418 | N 末端 GFP |
| pDM1208 | アンピシリン | G418 | N 末端 mCherry |
| pPI159 | アンピシリン | G418 | N 末端 mNeon |
| pPI437 | アンピシリン | G418 | N 末端 mScarlet |
| pPI54 | アンピシリン | G418 | N 末端 mTurquoise2 |
| pDM1209 | アンピシリン | G418 | GFP の C 末端 |
| pDM1210 | アンピシリン | G418 | C 末端 mCherry |
| pPI143 | アンピシリン | G418 | C 末端 mNeon |
| pPI459 | アンピシリン | G418 | C 末端 mScarlet |
| pPI142 | アンピシリン | G418 | C 末端 mTurquoise2 |
| シャトル プラスミド | |||
| pDM344 | アンピシリン | 違います | 違います |
| pDM1019 | アンピシリン | 違います | N 末端 GFP |
| pDM1018 | アンピシリン | 違います | N 末端 mCherry |
| pPI152 | アンピシリン | 違います | N 末端 mNeon |
| pPI418 | アンピシリン | 違います | N 末端 mScarlet |
| pPI150 | アンピシリン | 違います | N 末端 mTurquoise2 |
| pDM1021 | アンピシリン | 違います | GFP の C 末端 |
| pDM1020 | アンピシリン | 違います | C 末端 mCherry |
| pPI153 | アンピシリン | 違います | C 末端 mNeon |
| pPI457 | アンピシリン | 違います | C 末端 mScarlet |
| pPI151 | アンピシリン | 違います | C 末端 mTurquoise2 |
| 染色体外発現プラスミド | |||
| pDM1038 | アンピシリン | Hygromycin | 違います |
| pDM1047 | アンピシリン | Hygromycin | N 末端 GFP |
| pDM1046 | アンピシリン | Hygromycin | N 末端 mCherry |
| pPI450 | アンピシリン | Hygromycin | N 末端 mNeon |
| pPI452 | アンピシリン | Hygromycin | N 末端 mScarlet |
| pPI449 | アンピシリン | Hygromycin | N 末端 mTurquoise2 |
| pDM1049 | アンピシリン | Hygromycin | GFP の C 末端 |
| pDM1048 | アンピシリン | Hygromycin | C 末端 mCherry |
| pPI470 | アンピシリン | Hygromycin | C 末端 mNeon |
| pPI460 | アンピシリン | Hygromycin | C 末端 mScarlet |
| pPI469 | アンピシリン | Hygromycin | C 末端 mTurquoise2 |
| プラスミドをターゲットact5安全な避難所 | |||
| pDM1501 | アンピシリン | Hygromycin | 違います |
| pDM1513 | アンピシリン | Hygromycin | N 末端 GFP |
| pDM1514 | アンピシリン | Hygromycin | N 末端 mCherry |
| pPI231 | アンピシリン | Hygromycin | N 末端 mNeon |
| pPI419 | アンピシリン | Hygromycin | N 末端 mScarlet |
| pPI228 | アンピシリン | Hygromycin | N 末端 mTurquoise2 |
| pDM1515 | アンピシリン | Hygromycin | GFP の C 末端 |
| pDM1516 | アンピシリン | Hygromycin | C 末端 mCherry |
| pPI230 | アンピシリン | Hygromycin | C 末端 mNeon |
| pPI458 | アンピシリン | Hygromycin | C 末端 mScarlet |
| pPI229 | アンピシリン | Hygromycin | C 末端 mTurquoise2 |
| レミ発現プラスミド | |||
| pDM1220 | アンピシリン | Hygromycin | 違います |
| pDM1351 | アンピシリン | Hygromycin | N 末端 GFP |
| pDM1259 | アンピシリン | Hygromycin | N 末端 mCherry |
| pPI465 | アンピシリン | Hygromycin | N 末端 mNeon |
| pPI468 | アンピシリン | Hygromycin | N 末端 mScarlet |
| pPI466 | アンピシリン | Hygromycin | N 末端 mTurquoise2 |
| pDM1352 | アンピシリン | Hygromycin | GFP の C 末端 |
| pDM1305 | アンピシリン | Hygromycin | C 末端 mCherry |
| pPI471 | アンピシリン | Hygromycin | C 末端 mNeon |
| pPI467 | アンピシリン | Hygromycin | C 末端 mScarlet |
| pPI472 | アンピシリン | Hygromycin | C 末端 mTurquoise2 |
| フレームのプラスミッドの対象と | |||
| pDM1355 | アンピシリン | Hygromycin | GFP の C 末端 |
| pPI461 | アンピシリン | Hygromycin | C 末端 mCherry |
| pPI462 | アンピシリン | Hygromycin | C 末端 mNeon |
| pPI464 | アンピシリン | Hygromycin | C 末端 mScarlet |
| pPI463 | アンピシリン | Hygromycin | C 末端 mTurquoise2 |
| ノックアウト プラスミド | |||
| pDM1079 | アンピシリン | ブラストサイジン | 違います |
| pDM1080 | アンピシリン | Nourseothricin | 違います |
| pDM1081 | アンピシリン | Hygromycin | 違います |
| pDM1082 | アンピシリン | G418 | 違います |
| CRE 発現プラスミド | |||
| pDM1483 | アンピシリン | Nourseothricin | 違います |
| pDM1489 | アンピシリン | Hygromycin | 違います |
| pDM1488 | アンピシリン | G418 | 違います |
表 1: 非無菌 transfections プラスミド リストです。
-
トランスフェクションの細胞性粘菌を設定
- SM 培地 (栄養分豊富な媒体) で合流する健食を成長した文化で一晩は 4 ° C で最大 2 週間に保存できます。
- SM 寒天プレート上にこの細菌懸濁液の約 400 μ L を追加 (ペプトン 10 g/L; 酵母エキス 1 g/L; ブドウ糖 10 g/L;KH2PO4 1.9 g/L;K2HPO4 × 3 H2O、1.3 g/L;MgSO4無水 0.49 g/L;1.7% 寒天) と均等に 。滅菌ループを取るし、細胞性粘菌を接種します。プレートの 1 つの端にあるセルを広めます。
- Transfection のための十分に大きい成長ゾーンを確保するため 2 日間の 22 ° C でプレートを孵化させなさい。
注:大きな成長ゾーン (例えばAx3、DH1、または JH10) を作成しない、または経験の浅い実験者用細胞性粘菌の菌株の代わりにプレートをクリアします。このため、1.5.4 に 1.5.6 の手順の指示に従います。 - 滅菌ループを取るし、細胞性粘菌(およそ 2-4 105セル x) で接種します。密度の高い健食の 800 μ L にセルを転送、上下ピペッティングによる SM. ミックス細胞懸濁液。
- 新鮮な SM 寒天上 400 μ L、200 μ L、100 μ L、50 μ L を転送します。追加 SM ・ k ・水素生成菌懸濁液のすべてのプレート 400 μ L を加える、均等に乾燥します。
- プレートは半透明になるまで約 2 日間の 22 ° C でプレートを孵化させなさい。
注:その成長速度により当初合流芝生を生産する細菌と、アメーバその後「クリア」細菌のプレート。このプロセスに必要な時間ひずみ背景と細菌に成長する突然変異体の細胞の機能によって異なります。
2. 細菌の選択に基づく細胞性粘菌細胞のトランスフェクション

図 1: 細胞性粘菌の細菌成長の transfection のためワークフロー 。Transfection のための手順は次のとおりです。D. キイロタマホコリカビ細胞を健食菌でシード SM 板 (赤) になります。(ダーク グリーン) をすでに開発している細胞を回避する (緑) の供給の前面からのみセルを収穫します。H40 のセルを洗浄します。2-4 107セル/mL の x の最終密度に細胞を再懸濁します。1-2 μ g の DNA と細胞懸濁液を混ぜます。エレクトロポレーション キュベットとパルス セルに混合物を転送します。エレクトロポレーション後直接セルを SorMC とバクテリアで皿に転送します。5 h の選択可能なマーカーを追加する前に回復する細胞を許可します。染色のプラスミッドのためには、皿に直接選択を追加します。形質転換細胞は ~ 2 日後通常表示されます。ゲノムに単一の統合を目指して一直線に並べられた構成、示される 3 つの希釈を設定し、選択範囲を追加します。細菌は外径600から取られて 100 原液を =。チューブをよく混合し、96 ウェル平底組織培養プレートにセルを転送します。希釈あたり 2 枚の板を使用します。各ウェルに、細胞懸濁液を 150 μ l 添加をピペットします。タイトな植民地が表示されるまで約 5 日かかります。赤の井戸は、正常に変換された細胞 (上部パネル前の文書22から変更) の通常の量の例を示します。この図の拡大版を表示するのにはここをクリックしてください。
- K. 水素生成菌懸濁液を板を準備するには、10 mL の OD600の密度に健食菌を含む SorMC バッファーを追加 = 2 (準備外径600を 200 μ l 添加目的のため 100 k. 食原液を =細菌濃度) 10 cm に組織培養シャーレを扱われます。
注:このプレートは、トランスフェクションの育成に必要な後D. キイロタマホコリカビ細胞。また、6 ウェル培養プレートが使用できます。染色のプラスミドのトランスフェクションの場合 6 ウェル培養プレートは、リソース効率的です。健食SorMC 2 mL を使用して (外径600 = 2) ウェルあたり懸濁液。 -
細胞性粘菌の準備
- 10 μ L 使い捨て接種ループを使用して、培養プレート (クリアされた領域の端) またはプレートをオフの成長ゾーン (約 3 cm) から細胞をこすり取る。1 mL の冷たい H40 バッファーを含む 1.5 mL チューブに細胞を転送します。
注: プレートをクリアからセルを収穫のタイミングは重要です。後半増加する収穫しながら細胞の量も少し早い収穫を収穫部分的降伏のリスクは、細胞を開発しました。 - 1,000 x gまたは 2 フラッシュ回転で 2 分間を回してセルを洗浄して 10,000 x gで s。上澄みを廃棄し、2-4 107セル/mL の x の最終密度 H40 バッファー内の細胞を再懸濁します。全体トランスフェクション プロシージャ中にセルの寒さを維持します。直接チューブと氷の氷-水スラリーを使用します。
- 10 μ L 使い捨て接種ループを使用して、培養プレート (クリアされた領域の端) またはプレートをオフの成長ゾーン (約 3 cm) から細胞をこすり取る。1 mL の冷たい H40 バッファーを含む 1.5 mL チューブに細胞を転送します。
-
エレクトロポレーション
- 1-2 μ g の DNA とチューブに細胞の 100 μ L を追加します。上下にピペッティングして慎重に混ぜます。
- 中古冷蔵エレクトロポレーション キュベット (2 mm ギャップ) に細胞の DNA の混合物を転送します。
- 矩形波の次の設定を使用してセルをパルス: 350 V、8 ms、2 パルス、1 s パルス間隔。
注:以上 2 μ g の DNA を追加しないでください。高い金額は細胞に有毒、トランスフェクション効率を減らします。DNA 量は 5 μ L を超えないようにしてください。 合計が追加されます。 - 以前準備 10 cm シャーレ健食SorMC ですぐにセルを転送でき、5 h を回復する細胞。
注:倒立顕微鏡下でシャーレ内のセルを確認します。セルはエレクトロポレーション後は直接ラウンドが表示されますが、彼らは表面に接続するための十分な時間があったが、約 30 分後、アメーバの形に戻る。
-
形質転換細胞の選択: かどうかまたは生成するノックアウト、ノックイン、 act5ノックで、または染色体外プラスミッドから蛍光レポーター蛋白質を表現するため、トランスフェクションが目指している、に応じてのいずれかを選択プロセスを実行方法を次に説明します。
注:Axenically 成長の細胞とは異なり、細菌によって成長した細胞、ブラストサイジンに対する優れた耐性。選択、したがって、常に用いて G418 または hygromycin (ディスカッションを参照してください)。- ノックアウト ノック アドイン、 act5 ノック アドイン
- 繰り返し表面上にピペットから液を強制的にシャーレから慎重に細胞をデタッチします。
- SorMC k. 水素生成菌懸濁液の 3 つの希釈を設定 (外径600 = 2) し、抵抗使用される (図 1参照) によると選択のエージェント追加。
- 低希釈: SorMC の 20.4 mL と健食原液の 600 μ L 細胞懸濁液 9 mL を混ぜます。
- 培地の希釈: SorMC 28.5 mL と健食原液の 600 μ L 900 μ L の細胞懸濁液をミックスします。
- 高希釈: 29.3 SorMC mL と健食原液の 600 μ L 細胞懸濁液の 90 μ L をミックスします。
- 選択可能なマーカー (100 x 貯蔵液) の 30 μ L を追加します。
- 各ウェルに、細胞懸濁液の 150 μ L 分注で 96 ウェル平底組織培養プレートに準備された希釈を配布します。
注:この手順は、画面の単一クローンではなく、集団を目指しています。選択範囲は、使用する構文に応じて、約 5-7 日かかります。
- 染色体外プラスミド
- 10 mL の皿に直接選択可能なマーカーを追加 (図 1参照)。
注: クローン個体群のための欲求がないので希釈を設定する必要はありません。染色のプラスミッドのための選択プロセスは、高速高コピー数D. キイロタマホコリカビの細胞内に存在する予定です。2 日間に 32 時間後形質転換細胞が期待できます。
注意: 選択可能なマーカーとして抗生物質、有害です。手袋を着用します。
- 10 mL の皿に直接選択可能なマーカーを追加 (図 1参照)。
- ノックアウト ノック アドイン、 act5 ノック アドイン
-
肯定的な tranfectants のための得られたクローンをスクリーンします。ノックアウトまたはノックでの試行を 2.5.1 のステップの指示に従います。2.5.2 の手順で指示を使用して染色のプラスミッドのための遺伝子導入成功を確認します。
- ノックアウト、ノック アドイン、 act5ノック アドイン、正しいゲノム遺伝子座に、構造の統合を確認する PCR によって最初の画面を実行します。
注:クローンの集団の可能性を最大限、最高希釈可能選択後形質転換細胞を生成するを使用します。占められる井戸の最大 1/3 にあるプレートを目指してください。- クローン個体群を展開するには、ゲノム DNA の隔離のための十分な細胞を成長する 12 ウェル培養プレートに 96 ウェル培養プレートからの選択の後で育ってきたクローンを転送します。SorMC健食の 1 mL の各ウェルを供給 (外径600 = 2) と新鮮な選択可能なマーカー。
注: 1 日は通常 DNA の隔離のためによく適して合流を得るのに十分です。 - ミニ ゲノム DNA の隔離を実行するための合流も細胞を収穫し、製造元の指示に従ってミニ DNA 抽出キットを使用してゲノムの DNA を隔離します。
- 適切なプライマーとゲノム DNA の分離およびTaqを使用-ポリメラーゼ (材料表参照) 肯定的な積分の PCR 画面に。
注:肯定的な統合正しい genomic 位置の確認後、南しみ分析を実行してください。これにより大きな自信を持って不特定のゲノム領域に追加の挿入イベントが発生していないことです。
- クローン個体群を展開するには、ゲノム DNA の隔離のための十分な細胞を成長する 12 ウェル培養プレートに 96 ウェル培養プレートからの選択の後で育ってきたクローンを転送します。SorMC健食の 1 mL の各ウェルを供給 (外径600 = 2) と新鮮な選択可能なマーカー。
- 視覚的に蛍光レポーター蛋白質、蛍光陽性を特定して蛍光顕微鏡でチェックします。また、生化学的同定の適切な抗体で西部のしみを実行します。
- ノックアウト、ノック アドイン、 act5ノック アドイン、正しいゲノム遺伝子座に、構造の統合を確認する PCR によって最初の画面を実行します。
| PCR プログラム | ||
| ステップ | 温度 | 時間 |
| 初期変性 | 94 ° C | 30 s |
| 30 サイクル | 94 ° C | 15-30 秒 |
| 42 ° C | 15-60 s | |
| 68 ° C | 1 分/kb | |
| 最終的な拡張 | 68 ° C | 5 分 |
| ホールド | 4-10 ° C | |
| 反応成分 | ||
| コンポーネント | 25 μ L 反応 | 最終濃度 |
| 10 μ M 前方プライマー | 0.5 Μ L | 0.2 Μ M |
| 10 μ M 逆プライマー | 0.5 Μ L | 0.2 Μ M |
| テンプレート DNA | 変数 (ca. 5 μ L) | < 1,000 ng |
| ポリメラーゼを含む標準的なバッファーを持つマスター ミックス × 2 (材料の表参照) | 12.5 Μ L | 1 x |
| ヌクレアーゼ フリー水 | 25 μ L を | < 1,000 ng |
表 2: 増幅 PCR プログラムとサンプル組成D. キイロタマホコリカビゲノム DNA 。
結果
染色体外プラスミドはレポーター研究セルまたは変異細胞の細胞構造の変化内のある特定の蛋白質のローカリゼーションを識別する目的に使用されます。細胞周期のモニタリングなど、多くのアプローチ記者 2 人を同時に表現することが重要です。これはデュアル レポーター染色体外プラスミド システム (表 1) を使ってられるようになりました。1 日目、細胞は後 5 時間 (図 1) G418 の選択のマーカーを追加する前に導入させた。独立して派生の野生菌株 V12M2 と WS2162、ax2 横型、DdB NC4 例 (補足表 1) GFP-TubulinA、微小管とはタンパク質 mCherry PCNA のマーカーを符号化するプラスミド pPI289 を導入させた細胞周期 (図 2A) を監視するために使用されます。32 時間後細胞は顕微鏡の下で観察されました。セルの大半は両方の蛍光融合タンパク質を表明した一貫性のある前の表現を報告その同じプラスミドを示しています同じような発現量からの 2 人のレポーターの異なる 2 種のプラスミドを使用する場合、ほぼ不可能であります。18,19。代表的なセル (NC4、DdB、ax2 横型、V12M2、および WS2162) 各セルライン図 2Bに示す目的デュアル レポーターを表現します。トランスフェクションの効率は、図2 Cにまとめます。NC4 由来細胞株は、最高のトランスフェクション効率を示します。ただし、V12M2 と WS2162 の細胞、形質転換細胞の数がかなり多いが得られました。

図 2: 染色体外プラスミドの式。(A) 染色体外プラスミドは、円形のフォームのトランスフェクション直接。たとえば、デュアル レポーター pPI289 が表示されます。NgoMIV サイトを示す染色体外発現プラスミドに 2 番目の記者を挿入。(B) GFP TubulinA (細胞質) と 5 別の野生型細胞ラインの使用 (NC4、DdB、ax2 横型、V12M2、WS2162)、mCherry-PCNA (主核) を表現する代表的なセルの Z 投影。写真 (B) の 5 つのセルラインの (C)、トランスフェクション効率を算出しました。2 つの実験の平均値は、示されています。誤差範囲を示す ± SD.この図の拡大版を表示するのにはここをクリックしてください。
ターゲット ベクトルの指定された genomic 位置への統合は難しく、生成された細胞ラインのより慎重な分析が必要です。図 3、 act5- mCherry NC4 で氣が試みられます。まず、プラスミッドをトランスフェクション再結合事象の頻度を増やす線形補間する必要があります。このため、 NgoMIV でプラスミド pDM1514 を切ります。Agarose のゲルのダイジェストを実行した後は、2 つのバンドが得られます。4127 bp のバンドには、目的の構成 (図 3) が含まれています。トランスフェクション、消化の DNA ゲルから抽出する必要があり、製造元の指示、次ゲル抽出キットを使用して浄化します。

図 3: トランスフェクションのact5のノックで、DNA の準備。プラスミド pDM1514行動5 ノックの使用の例を示します。準備のための手順は次のとおりです。(A) 穿孔前に示されたNgoMIV サイトを使用してプラスミッドをリニア化します。(B、C)2 つの予想されるバンドは正しく区切らまで agarose のゲルのカットのプラスミドを実行します。4127 bp のバンドが再結合腕、mCherry、および耐性カセットを含むを切り取って、ゲルの DNA を抽出します。DNA は、transfection のため準備が整いました。この図の拡大版を表示するのにはここをクリックしてください。
精製した DNA は NC4 セルの transfection のために使用されました。選択の 5-6 日後のクローンが得られました。代表的なトランスフェクションの効率は、いくつかの試みのノックでact5の肯定的な識別されたクローンの量は、表 3にまとめます。両方示した予測バンド パターンはノックでの予想し、南しみ分析を単一の統合と検証されてさらに、NC4 トランスフェクションの 2 つのクローンがランダムに選択され (図 4A) pcr 法による分析ゲノム20に構成のイベントです。しみは、望ましいact5軌跡 (図 4B) の明確な単一の統合を示しています。生成されたNC4::act5 mCherryセルの行は、実験で今すぐ使用できます。

図 4: Act5mCherry NC4 で KIs の検証します。 (A) 方式と制御 PCRs act5軌跡に肯定的な統合の検証。指定されたプライマーは、2 つの独立したクローンと親を分析する使用されました。両方のクローンは、親株には存在しない抵抗カセットと下流 (P1) または上流のプライマー (P2) の期待のバンドを示します。プライマーの組み合わせ (P1 ・ P2) は、 act5の軌跡への mCherry と抵抗のカセットの正しい統合を確認します。両方 KI クローンこのバンドがない代わりに大きい 1400 塩基対について PCR の製品を表示し、野生型 NC4 は予想 2800 bp のバンドを示しています。使用制限のダイジェストと南しみ (B) 方式です。両方のノックでクローン表示小さい 3400 bp のバンド 5 を行動の軌跡への構築の統合の結果である、私はサイトの具体的追加Bclhygromycin 抵抗カセット。私のサイト、野生型のコントロールに表示される 2 つの下流に位置するBclの結果予想される 5.8 kb。しみは、トリミングされ、わかりやすくするためにスプライシングします。この図の拡大版を表示するのにはここをクリックしてください。
| act5 の軌跡にターゲットを絞った構築 | プラスミドの名称 | 占められた井戸の数 | チェックのクローン数 | 肯定的なクローン | 正しいクローン (%) | 細胞性粘菌のひずみを使用 | トランスフェクション効率 (形質転換細胞/2 x 10 ^6/1 μ g DNA) |
| LifeAct mCherry | pPI226 | 7 | 7 | 1 | 14.2 | AX2 横型 | 3.5 x 10 ^-6 |
| LifeAct GFP | pPI227 | 12 | 12 | 5 | 41.6 | AX2 横型 | 6 x 10 ^-6 |
| GFP | pDM1513 | 3 | 3 | 2 | 66.6 | AX2 横型 | 1.5 x 10 ^-6 |
| mCherry | pDM1514 | 3 | 3 | 1 | 33.3 | AX2 横型 | 1.5 x 10 ^-6 |
| GFP | pDM1513 | 66 | 9 | 5 | 55.5 | AX2 横型 | 3.3 x 10 ^-5 |
| mCherry | pDM1514 | 221 | 12 | 10 | 83.3 | AX2 横型 | 1.1 × 10 ^-4 |
| H2B mCherry | pPI420 | 3 | 3 | 1 | 33.3 | AX2 横型 | 1.5 x 10 ^-6 |
| H2B mCherry | pPI420 | 7 | 7 | 6 | 85.7 | AX2 横型 | 3.5 x 10 ^-6 |
| mCherry | pDM1514 | 10 | 10 | 7 | 70 | DdB | 5 x 10 ^-6 |
| mCherry | pDM1514 | 240 | 12 | 11 | 91.6 | DdB | 1.2 × 10 ^-4 |
| mCherry | pDM1514 | 320 | 12 | 12 | 100 | NC4 | 1.6 x 10 ^-4 |
テーブル 3: トランスフェクションの効率は、数世代の肯定的な形質転換細胞を得られるact5別の系統の背景にキシュ22.
行動5 軌跡統合記者21の比較的均質な表現を提供しています。生成されたNC4::act5 mCherryセルには、GFP など異なる蛍光タンパク質を用いた他のact5ノックと同様実行ミックス実験が可能します。このシステムの大きな利点を強調するには、 Ax2::act5 GFPの混合実験のとおりです。非無菌の野生型細胞を使ってできないのためこのタイプのアプローチをする前に実行できませんでした。ミックス実験は、彼らは同一の実験条件が発生して細胞間の直接比較を許可するセルの動作を分析するための重要なツールです。NC4 細胞成長速く、ax2 横型細胞 (図 5A) よりも細菌の芝生の上に。細菌や移動、食料源に向けて走化性を表示する機能の強化を貪食能力が高い可能性があります。下の寒天葉酸走化性の試金を使用して、 NC4::act5 mCherryが葉酸のセンシングではるかに効率的であることを示すNC4::act5 mCherry Ax2::act5 GFPの人口の直接比較は実行されました。4 時間後よりNC4::act5 mCherry細胞Ax2::act5 GFPの細胞 (図 5B) よりも agarose の下にクロールすることができた。NC4::act5 mCherry細胞が速くなったし、 Ax2::act5 GFPの細胞 (図 5C E よりも強力な走化性反応を示したことを明らかにした agarose 下移行細胞の走化性の標準的な指標を分析).
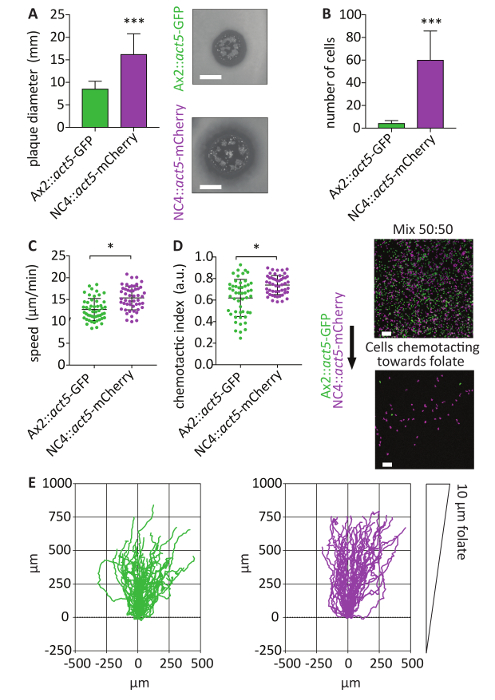
図 5: 走化性のイメージ ベース ミックス実験による行動5 キシュ。(A) NC4::act5 mCherryとAx2::act5 GFP表現行動5 軌跡から指定された蛍光タンパク質が細菌の芝生の成長する能力の分析しました。4 日後メッキ細胞性粘菌細胞から生じたプラーク直径を測定しました。非無菌act5:: NC4 細胞作る無菌Ax2::act5 GFPの細胞よりもかなり大きな斑 (± SD を意味 * * * p < 0.0001、n = 3、棒 5 mm スケール)。(B) agarose の下で葉酸走化性の使用は、22 NC4::act5 mCherryとAx2::act5 GFP (a) 使用の走化性の能力を直接比較するアッセイの細菌 - 両系統のアメーバを成長した混合されました。50: 50 の比率。セルは agarose の下にクロールを許可されました。4 h 後、葉酸グラデーションを移行した細胞は、共焦点顕微鏡を用いたをイメージしました。NC4::act5 mCherryとAx2::act5 GFPの細胞の数は、定められました。NC4::act5 mCherry細胞が効率よく葉酸をセンシングします。Ax2::act5 GFPの細胞と比較して 10 倍以上NC4::act5 mCherryについて細胞が見いだされた (± SD を意味 * * * p < 0.0001、n = 6、スケール 100 μ m バー)。(C, D)60 分間撮影されたセルとの速度と走化性指数を算出しました。走最も応答性細胞 (agarose 下移行ものだけ) の事前選択後Ax2::act5 GFPの細胞セル速度と走化性の低い値を示した。細胞あたり 50 セルを行った。(平均 ± SD、* p < 0.01, n = 3)。(E) NC4::act5 mCherryとAx2::act5 GFPact5 ノック ins の軌跡プロットNC4::act5 mCherry細胞の chemoattractent ソースに向かってもっと監督の動きを示す 60 分以上 (平均 ± SD、* ** p < 0.0001、n = 6)。この図の拡大版を表示するのにはここをクリックしてください。
フローサイトメトリー用act5ノックイン細胞株を使用もできます。細菌の成長と同様、無菌系統と非無菌野生型の開発の主要な違いがあります。後の発展を NC4 細胞の子実体が約 2 倍ものと大 ax2 横型細胞から派生しました。セルが混在している場合は、中間の大きさで分類された子実体が取得されます。両方の細胞の寄与をより定量的に解析、フローサイトメトリーを使用しました。これらの解析は明らか、両方の細胞から異なるレベルの貢献のため中間サイズの子実体であります。NC4::act5 mCherry細胞測定の胞子の約 75% 間、 Ax2::act5 GFPは非無菌系統 (図 6) のための潜在的なフィットネスの利点を明らかに 25% だけを貢献しました。分析は、茎セル人口を監視しません、ax2 横型 NC4 と開発の不均衡を説明する 2 つの可能性があります。1 つの可能性は、ax2 横型細胞が胞子細胞集団に入るのではなく、茎セル人口に主に寄与することです。また、NC4 セルは比較的遅延、ax2 横型と発達のサイクルを入力可能性があります、彼らは従って子実体のアセンブリに貢献することができません。非無菌の野生型細胞を使って可能性はさらに他のアプローチを開発し、実験手順を大幅に簡略化します。

図 6:法5 キシュ フローサイトメトリーを用いたミックス実験の分析が可能です。(A) NC4::act5 mCherryとAx2::act5 GFP個別にまたは非栄養寒天版で 50: 50 の混合物を開発しました。NC4::act5 mCherry細胞Ax2::act5 GFPより大型の子実体を形成します。ミックスは代わりに中間サイズを示します (棒 5 mm スケール)。共焦点レーザー蛍光顕微鏡では、ミックスから派生した胞子ヘッドNC4::act5 mCherry胞子の高い量を示唆しています。(B) この観察は、(A) の混合の実験から頭いたフローサイトメトリーで分析した収穫の胞子の胞子の量を定量化します。胞子の約 75% に由来NC4::act5 mCherry細胞Ax2::act5 GFPの細胞から 25% だけ。(C) 代表的な分布流れの cytometry の散布図に示すように両方の細胞からの胞子。約 0.05% を示す正 mCherry と GFP の信号、有性融合プロセスが発生したことまたは互いに胞子を固執していることを示唆しています。この図の拡大版を表示するのにはここをクリックしてください。
| セルライン | 遺伝的背景 | 参照番号 | 前に公開されました。 | タイプ |
| Ax2 横型 (Ka) | DBS0235521 | ブルーム フィールド et al., 2008 | 野生型 | |
| NC4 (S) | ブルーム フィールド et al., 2008 | 野生型 | ||
| act5::mCherry クローン 5 | NC4 | HM1912 | Paschke et al., 2018 | act5 のノックで |
| act5::GFP クローン 2 | AX2 横型 | HM1930 | Paschke et al., 2018 | act5 のノックで |
| V12M2 | ブルーム フィールド et al., 2008 | 野生型 | ||
| WS2162 | ブルーム フィールド et al., 2008 | 野生型 |
補足表 1: 異なる系統細胞性粘菌検討します。
ディスカッション
非無菌、野生型の細胞性粘菌の使用は、分子生物学の研究にところ非常に制限されています。信頼性と効率23、彼らの一般的な養子縁組を防止するこれらの系統の遺伝子工学の使用可能なメソッドを欠いていた。生成された材料およびここに示すプロトコルは、液体媒体の成長する能力の独立したD. キイロタマホコリカビ緊張に使用できます。特筆すべきはこのプロトコルは NC4 由来細胞株が最適です。トランスフェクションの効率は野生から新鮮単離株の異なる NC4、我々 は前に観察するいるし、V12M2 と WS21628,9を次に示します。エレクトロポレーションの条件は、特にトランスフェクションの効率に大きな影響を持っているようだし、いくつかの系統のさらなる最適化を必要があります。一般に、これらのメソッドが実行可能なことを示すこれまでのところ、テストすべての系統の形質転換細胞の十分な量が観測されています。NC4 から派生した系統を使用して得られた肯定的な形質転換細胞の数はくらべると他の非無菌の野生型株がすべてのケースで、さらに実験を許可する形質転換細胞の十分な数字が得られます。これに加えて、我々 のプロトコルのシンプルさは、以前の試み8,9と比較して大きな改善です。
これらの新しい非無菌プロトコルは、すべての標準的な遺伝的手続きをカバーし、transfections 迅速かつ効率的に並列で実行できるので、それ以上の利点を追加します。肯定的なクローンは、細菌半分成長部門の時間以来、週間ではなく、日で取得できます。新しく設立されたプラスミドも無菌条件の下で働くし、日常的に使用できる両方の成長条件の下でこの方法22のもう一つの進歩であります。プロトコルの導入、細菌の選択に使用されるプラスミド必要特別な遺伝子導入の成功のために重要であります。特に、式および抵抗のカセットを運転プロモーター、成功の transfection のため重要です。抵抗または式カセットを駆動するため頻繁に使用される, act6 (アクチン 6) プロモーター24は、細菌のセルは育つとき効率を欠いています。プラスミド体制で高活性act15 (アクチン 15) プロモーター ドライブすべて式カセットのcoA (coactosin A) プロモーター、無菌条件下で、アクティブであるの両方の制御の下で抵抗のカセットが細菌の成長のセル。記者の表現のための要件をノックで、構築し、ノックアウト構造が私たちのプラスミッドのレパートリーを使用する必要が、残念なことに使用する非効率的な, act6プロモーターを既に作成ベクトルの使用は制限されます。
プロモーターの効率は正しい genomic 位置に単一の統合イベントに依存している transfections 特に重要です。プロモーターが耐性遺伝子発現を運転の改善のためのため十分な抵抗性蛋白質は単一軌跡インテグレーション環境から生成されます。主要な関心事だった hygromycin または G418 に従って使用する場合、ゲノムに複数の統合の支持します。複数の統合の支持は観察されていないところ、G418 の選択で古いプロモーター システム25を使用して以前に報告します。つまり、両方 hygromycin G418、マーカーが選択可能な世代のきれいなノックアウトおよびノック保険の残念なことに、選択可能なマーカー ブラストサイジンは非無菌条件の下では動作しません。ブラストサイジン抵抗カセットを日常的に使ってD. キイロタマホコリカビでノックアウトの構成要素を生成するので、これは私たちの方法の主な欠点です。ブラストサイジン耐性カセットを既に生成された構造体は、実行可能な選択可能なマーカーの 1 つを使用して rederived する必要があります。この限定を克服するもう一つの可能性は、最近設立された無菌D. キイロタマホコリカビCRISPR を結合するこのトランスフェクション プロトコル26技術。適切な単一ガイド Rna (sgRNAs) の生成は、簡単かつ完全なノックアウト構造の復元よりも高速です。今後の方向性、可能性の複数のノックアウトを生成する CRISPR/Cas9 このトランスフェクション プロトコルと組み合わせてを使用して魅力的で、細胞性粘菌のコミュニティで多くの研究者のための道を開く可能性があります。ただし、無菌栽培セルに CRISPR/Cas9 に使用される確立された一過性発現システムの施工性を慎重に検討する必要があります。
紹介システム ノックでact5 D. キイロタマホコリカビで安定したセルラインの他生物22年同様のサイトの利点との世代のため信頼性の高い、安全なシステムを提供しています。また、ゲノムの単一の統合イベントに依存し、多くの異なる研究の方向性の可能性を提供しています。Act5のプロモーターが強くアクティブで、栽培条件の独立したほぼ同種式27を保証します。蛍光レポーター蛋白質は、設計されたターゲット プラスミドを使用してこの安全な避難所の軌跡に簡単に統合できます。これことができます、たとえば、細胞追跡用ミックス実験で示すよう。セル自動細胞を追跡を支援することができます最小限のセルに式の可変性を示します。重要なは、行為5 遺伝子座に必要なシーケンスの挿入表現型中性28,29が表示されます。ランダムな配合成分または染色体外ベクトル軸受け細胞に見られるように、式は蛋白質の表現を維持するために選択可能なマーカーに依存しない、 act5システムはレスキュー実験できます。細胞は均一なので、すべてのセルが分析できます。これは染色体外プラスミドを用いた式でかなりの不均一性が表現可能ではありません。
すべての緊張のためのこれらの一般的な改良に加えて分子研究に非無菌の野生型細胞を使用する能力は、現在の実験室の緊張の蓄積された突然変異の効果の評価をことができます。複数遺伝子の再配列は、以前に公開されたマイクロ アレイ解析26に示すよう 1970 年に ax2 横型ひずみの採用以来発見されています。これらの突然変異の存在のため、合成の表現型が観察されます。たとえば、報告されたphg2 変異は 1 研究室ひずみのバック グラウンドで別の3で粘着性が向上減少接着を示しています。このような競合しているデータは、(この例では、DdB) で共通の祖先ひずみの実験を繰り返すことによって今解決できます。このアプローチの強さは、小さい gtp アーゼ RasS30,31の最近示されています。
D. キイロタマホコリカビ緊張で遺伝子実験を実行する機能は、社会の進化、血縁認識、および性的サイクル22を含む特にそれら野生株の使用に依存する新たな研究方向性の可能性を展開します。
開示事項
著者はある利益相反を開示します。
謝辞
著者は、このメソッドへの入力のケイ ラボと LMB 顕微鏡と流れ cytometry 施設優れた科学的、技術的なサポートをありがとうございます。この仕事医学研究評議会 MC_U105115237 r. r. ケイ、BBSRC (バイオ テクノロジー ・生物科学研究会議) に助成 BB/K009699/1 r. r. ケイによって資金が供給された、がん研究英国 r. h. 窶、Wellcome の信頼シニア親睦 A15672 を付与します。202867/Z/16/Z はジョナサン ・ R チャブと MRC UCL で MRC の LMCB 大学の単位 (MC_U12266B) の資金。
資料
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| Eppendorf Microcentrifuges 5424/5424R | ThermoScientific | 05-400-002 | |
| Eppendorf 5702R Centrifuges with A-4-38 Model Rotor | ThermoScientific | 12823252 | |
| Eppendorf Mastercycler Nexus Thermal Cyclers | Sigma Aldrich | EP6331000025 | |
| Gene Pulser X Cell, including CE & PC modules | BioRad | 1652660 | |
| Safety Cabinet Foruna - SCANLAF | Labogene | ||
| BioPhotometer Plus | Eppendorf | ||
| CLSM 710 | Zeiss | ||
| Media & Agaroses | |||
| LoFlo Medium | Formedium | LF0501 | |
| Seakem GTG Agarose | Lonza | 50071 | |
| Low EEO Agarose | BioGene | 300-200 | |
| Purified Agar | Oxoid | LP0028 | |
| SM broth | Formedium | SMB0102 | |
| 2x LB broth | Formedium | LBD0102 | |
| Chemicals | |||
| SYBR Safe DNA Gel Stain | ThermoScientific | S33102 | |
| 1 Kb Plus DNA Ladder | ThermoScientific | 10787018 | |
| 1 Kb DNA Ladder | NEB | N3232L | |
| HEPES free acid | Sigma Aldrich/Merck | 391340 EMD | |
| KH2PO4 | VWR | P/4800/53 | |
| Na2HPO4 * 2 H2O | VWR | 10028-24-7 | |
| MgCl2 * 6 H2O | Sigma Aldrich/Merck | 5982 EMD | |
| CaCl2 * 2 H2O | Sigma Aldrich | 442909 | |
| Folic Acid | Sigma Aldrich | F7876 | |
| KOH | VWR | 26668.263 | |
| Cultur Dishes | |||
| 96 Well Cell Culture Cluster, Flat Bottom with Low Evaporation Lid, Tissue Culture Treated | Corning Incorporated | 3595 | |
| 6 Well Cell Culture Cluster, Flat Bottom with Lid, Tissue Culture Treated | Corning Incorporated | 3516 | |
| 100 x 20 mm style Tissue Culture Dish, Tissue Culture Treated | Corning Incorporated | 353003 | |
| 50 mm Glass Bottom Microwell Dishes No1.5 | MatTek Corporation | P50G-1.5-30-F | |
| Antibiotics | |||
| Geneticin G418-sulphate | Gibco by Life Technologies | 11811-023 | |
| Hygromycin B Gold | InvivoGen | ant-hg-1 | |
| Additional Consumables | |||
| 2 mm gap electroporation cuvettes, long electrode | Geneflow Limited | E6-0062 | |
| 10 µL Inoculating Loop, Blue 10 Micro L | ThermoScientific | 129399 | |
| Spreader, L-shaped, sterile | greiner bio-one | 730190 | |
| Combitips advanced 5 mL | Eppendorf BIOPUR | 30089669 | |
| Kits | |||
| ZR Plasmid Miniprep Classic | Zymoresearch | D4016 | |
| Quick DNA Miniprep Kit | Zymoresearch | D3025 | |
| Zymoclean DNA Recovery Kit | Zymoresearch | D40002 | |
| Enzymes | |||
| Restriction enzymes | NEB | ||
| OneTaq 2X Master Mix with Standard Buffer | NEB | M0482L | |
| T4 DNA Ligase | NEB | M0202S | |
| PrimeSTAR Max DNA Polymerase | TakaraBio | R045A |
参考文献
- Schaap, P. Evolutionary crossroads in developmental biology: Dictyostelium discoideum. Development. 138 (3), 387-396 (2011).
- Sussman, R., Sussman, M. Cultivation of Dictyostelium discoideum in axenic culture. Biochemical and Biophysical Research Communications. 29, 53-55 (1967).
- Bloomfield, G., Tanaka, Y., Skelton, J., Ivens, A., Kay, R. R. Widespread duplications in the genomes of laboratory stocks of Dictyostelium discoideum. Genome Biology. 9 (4), 75(2008).
- Witke, W., Nellen, W., Noegel, A. Homologous recombination in the Dictyostelium alpha-actinin gene leads to an altered mRNA and lack of the protein. The EMBO Journal. 6, 4143-4148 (1987).
- De Lozanne, A., Spudich, J. A. Disruption of the Dictyostelium myosin heavy chain gene by homologous recombination. Science. 236, 1086-1091 (1987).
- Howard, P. K., Ahern, K. G., Firtel, R. A. Establishment of a transient expression system for Dictyostelium discoideum. Nucleic Acids Research. 16, 2613-2623 (1988).
- Knecht, D., Pang, K. M. Electroporation of Dictyostelium discoideum. Methods in Molecular Biology. 47, 321-330 (1995).
- Wetterauer, B., et al. Wild-type strains of Dictyostelium discoideum can be transformed using a novel selection cassette driven by the promoter of the ribosomal V18 gene. Plasmid. 36, 169-181 (1996).
- Lloyd, M. M., Ceccarelli, A., Williams, J. G. Establishment of conditions for the transformation of nonaxenic Dictyostelium strains. Developmental Genetics. 11, 391-395 (1990).
- Bloomfield, G., et al. Neurofibromin controls macropinocytosis and phagocytosis in Dictyostelium. eLife. 4, (2015).
- Veltman, D. M., et al. A plasma membrane template for macropinocytic cups. eLife. 5, (2016).
- Veltman, D. M., Lemieux, M. G., Knecht, D. A., Insall, R. H. PIP(3)-dependent macropinocytosis is incompatible with chemotaxis. The Journal of Cell Biology. 204 (4), 497-505 (2014).
- Hoeller, O., Kay, R. R. Chemotaxis in the absence of PIP3 gradients. Current Biology. 17, 813-817 (2007).
- Chubb, J. R., Wilkins, A., Thomas, G. M., Insall, R. H. The Dictyostelium RasS protein is required for macropinocytosis, phagocytosis and the control of cell movement. Journal of Cell Science. 113, 709-719 (2000).
- Schaap, P., et al. Molecular phylogeny and evolution of morphology in the social amoebas. Science. 314, 661-663 (2006).
- Du, Q., Kawabe, Y., Schilde, C., Chen, Z. H., Schaap, P. The Evolution of Aggregative Multicellularity and Cell-Cell Communication in the Dictyostelia. Journal of Molecular Biology. 427 (23), 3722-3733 (2015).
- Hirose, S., Benabentos, R., Ho, H. I., Kuspa, A., Shaulsky, G. Self-recognition in social amoebae is mediated by allelic pairs of tiger genes. Science. 333 (6041), 467-470 (2011).
- Strassmann, J. E., Queller, D. C. Evolution of cooperation and control of cheating in a social microbe. Proceedings of the National Academy of Sciences of the United States of America. 108 (2), 10855-10862 (2011).
- Wolf, J. B., et al. Fitness Trade-offs Result in the Illusion of Social Success. Current Biology. 25 (8), 1086-1090 (2015).
- Veltman, D. M., Akar, G., Bosgraaf, L., Van Haastert, P. J. A new set of small, extrachromosomal expression vectors for Dictyostelium discoideum. Plasmid. 61 (2), 110-118 (2009).
- Southern, E. M. Detection of specific sequences among DNA fragments separated by gel electrophoresis. Journal of Molecular Biology. 98 (3), 503-517 (1975).
- Paschke, P., et al. Rapid and efficient genetic engineering of both wild type and axenic strains of Dictyostelium discoideum. PLoS One. 13 (5), 0196809(2018).
- Woznica, D., Knecht, D. A. Under-agarose chemotaxis of Dictyostelium discoideum. Methods in Molecular Biology. 346, 311-325 (2006).
- Dubin, M., Nellen, W. A versatile set of tagged expression vectors to monitor protein localisation and function in Dictyostelium. Gene. 465 (1-2), 1-8 (2010).
- de Hostos, E. L., et al. Dictyostelium mutants lacking the cytoskeletal protein coronin are defective in cytokinesis and cell motility. Journal of Cell Biology. 120, 163-173 (1993).
- Sekine, R., Kawata, T., Muramoto, T. CRISPR/Cas9 mediated targeting of multiple genes in Dictyostelium. Scientific Reports. 8 (1), 8471(2018).
- Sadelain, M., Papapetrou, E. P., Bushman, F. D. Safe harbours for the integration of new DNA in the human genome. Nature Reviews Cancer. 12 (1), 51-58 (2011).
- Rosengarten, R. D., et al. Leaps and lulls in the developmental transcriptome of Dictyostelium discoideum. BMC Genomics. 16, 294(2015).
- Tunnacliffe, E., Corrigan, A. M., Chubb, J. R. Promoter-mediated diversification of transcriptional bursting dynamics following gene duplication. Proc Natl Acad Sci USA. 115 (33), 8364-8369 (2018).
- Gebbie, L., et al. Phg2, a kinase involved in adhesion and focal site modeling in Dictyostelium. Molecular Biology of the Cell. 15, 3915-3925 (2004).
- Jeon, T. J., Lee, D. J., Merlot, S., Weeks, G., Firtel, R. A. Rap1 controls cell adhesion and cell motility through the regulation of myosin II. Journal of Cell Biology. 176, 1021-1033 (2007).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved