Method Article
定量的リアルタイム逆転写 PCR を用いたカニクイザルにおける血中マイクロ Rna の絶対定量
要約
このレポートでは、中古増幅の有無、定量的リアルタイム逆転写 PCR を用いたプラズマ miRNA の絶対レベルを測定するためのプロトコルについて説明します。このプロトコル プラズマ miRNAs の量のより良い理解とさまざまな研究や研究室から対応するデータの質的評価をことができます。
要約
RT qPCR は個々 のターゲットの Mirna を評価する最も一般的な方法の 1 つです。Mirna のレベルは一般に参照サンプルを基準にして測定されます。このアプローチは、ターゲット遺伝子の表現のレベルの生理学的変化を調べることに適しています。しかしより良い統計解析を用いた絶対定量遺伝子発現レベルの包括的な評価をお勧めします。絶対定量はまだ共通の使用します。このレポートでは、中古増幅の有無 Rt-qpcr を使用してプラズマ miRNA の絶対レベルを測定するためのプロトコルについて説明します。
EDTA 血漿の固定ボリューム (200 μ L) を意識したカニクイザルの大腿静脈から集められた血から調製した (n = 50)。総 RNA は、市販のシステムを使用して抽出しました。プラズマ miRNAs は miRNA 特定フォワード/リバースの PCR プライマーとプローブを含むプローブ ベースの RT qPCR アッセイによって定量化されました。絶対的な定量化のための標準的な曲線は、市販の合成 RNA オリゴヌクレオチドを使用して生成されました。合成 cel-ミール-238 は、正規化と品質評価のための外部コントロールとして使用されました。35 上記サイクル (Cq) 値の定量化を示した miRNAs は qPCR ステップ前にあらかじめ増幅.
検査 8 Mirna の中では、ミール-122、ミール-133a、ミール 192 ミール 1 とミール-206、ミール 499a の低発現量のため中古増幅に必要なに対しあった中古増幅なし検出。ミール 208a とミール 208b 検出されなかった中古増幅後でさえも。サンプル処理効率は、スパイクの cel ミール 238 の Cq 値で評価しました。この測定法の技術的な変化が 3 倍未満と推定され、定量化 (LLOQ) の下限が 102コピー/μ L、検討の miRNAs のほとんどのため。
このプロトコル プラズマ miRNAs の量のよりよい見積もりを提供でき、さまざまな研究から対応するデータの品質評価。体液、中古増幅 miRNAs の低い数字は悪いの検出を向上に役立ちます考慮した Mirna を表明しました。
概要
研究の増加する数の診断と癌の予後のバイオ マーカーとしてマイクロ Rna (Mirna) を探索または監視と非臨床および臨床的研究1,2,3 で他の疾患を検出.定量的リアルタイム逆転写 PCR (RT qPCR) は個々 のターゲットの Mirna を評価するため、この手法はより敏感なマイクロ アレイ4と RNA シーケンス ベースのプラットフォーム5よりも、最も一般的な方法の 1 つであります。一般に、miRNA の表現は、ΔCq 方法6を使用して参照サンプルを基準にして測定されます。このアプローチは、ターゲット遺伝子発現レベルでの生理的変化を調査に適しています。ただし、循環する Mirna の相対的な定量化は、少量のためユーティリティを制限されています。また、技術の変化、異なる所カスタマイズ RT qPCR 実験プロトコル、異なる矛盾につながるかも矛盾に起因するため、さまざまな研究からの結果を比較することは困難異なった調査7。
上記懸念の観点から絶対定量が体液の miRNAs の少量の評価に適してあります。絶対定量法は、対応するターゲット miRNA8と同じシーケンス合成の RNA オリゴヌクレオチドの既知濃度から生成される標準曲線を使用します。保健環境科学研究所 (HESI) ゲノミクス研究会は最近複数のテスト サイトのプラズマ Mirna 絶対測定結果を比較するための包括的な研究を実施しました。結果はことの Mirna 絶対 quantitation のための標準プロトコルを使用して複数のテスト サイト9間で同等の結果をもたらした。本研究では説明されている RT qPCR 測定法は HESI の標準のプロトコルは、複数の miRNA ターゲット、および低式 miRNAs の検出を支援するために中古の増幅の多重分析が含まれていますとほぼ同じです。
本研究では一定量 (200 μ L) EDTA 血漿、血液から調製したが意識したカニクイザルの大腿静脈から収集 (n = 50) 使用10だった。次のプロトコルでは、抽出、miRNA と Rt-qpcr、中古増幅を含む血漿検体の準備のための手順について説明します。もっと重要なは、よく修飾プロセスと組み合わせてターゲット miRNAs サンプルの数量を検証できるように、プロトコルに関する追加の技術情報が、含まれていた。まず、各 miRNA の標準曲線は、生体試料中の定量前にその個々 の検出範囲で検証されました。第二に、現在の方法論の質は、外部コントロール (cel-ミール-238) の Cq 値による総合的に行った。したがって、このプラットフォームでは、さまざまな研究や研究室からの結果を比較するためより有益で信頼性の高いデータが得られます。
8 miRNAs のプロファイルは、代表的な結果としてこのレポートに含まれているここで説明した測定法から。潜在的な安全性バイオ マーカーは、心 (ミール-1、ミール 208a、ミール-208b およびミール 499a) と、齧歯動物および人間3筋 (ミール 133a とミール 206) (ミール 122 とミール 192)、肝臓組織の損傷に関連付けられているこれらの Mirna が提案されています。 11,12,13。
プロトコル
すべての実験は動物介護制度と株式会社第一三共使用委員会によって承認されました。
1. サンプル準備
- EDTA の 2 K を含むチューブにカニクイザルの大腿静脈から血 (少なくとも 0.5 mL) を収集します。
注: クエン酸とヘパリンです・・・・これらの凝固を抑制するその後 PCR14,15 - 氷とコレクションの 2 時間以内に血漿分離のプロセスに収集されたサンプルをすぐに配置します。
- 4 ° C、5 分で 10,000 x g でサンプルを遠心します。
- 残留血小板細胞の残骸を削除する 5 分の 4 ° C で 16,000 × g で遠心分離後、2 mL のマイクロ チューブに上清を移します。
注: miRNAs の定量化は、血小板汚染16大きく影響することができます。 - 新鮮な 2 mL のマイクロ チューブの上清 200 μ L 因数を置き、使用するまで-80 ° C で保存します。
注: 各サンプルの固定量は RNA 抽出用します。したがって、因数のボリュームは正確でなければなりません。
2. RNA の抽出
- (手順 1.5) から氷の上凍結試料を解凍します。
- RNA 抽出時に氷の冷たいサンプルをしてください。溶解試薬を冷やすし、使用する前に氷のクロロホルムします。
- 溶解試薬、サンプル (200 μ L) のフェノールとグアニジン イソチオシアネートの単相性溶液の 5 冊 (1000 μ L) を加え、1分間ボルテックスによって精力的に混ぜます。
- 5 nM の合成の 5 μ L を追加線虫miRNA (Syn-cel-ミール-238-3 p)。
- クロロホルム、1 ボリューム (200 μ L) を追加し、1 分間ボルテックスによって精力的に混ぜます。
- 2 〜 3 分の氷の上を維持し、4 ° C 15 分で 12,000 × g でサンプルを遠心分離します。
- 新しいチューブに水相を慎重に転送します。
注: 任意の有機相 (赤) または中間期 (白) は転送されません。収集水様段階のボリュームは、高められた技術的な変化の結果の矛盾を処理するため均一なはずです。このプロトコルで転送水相の通常の量は 650 μ L です。 - エタノールの 1.5 ボリューム (975 μ L) を追加し、上下にピペッティングでよく混ぜます。
- 対応する列とアダプター、真空乾燥真空マニホールドを使用して 3 分後にサンプルを転送します。試料の量が以上 700 μ L の場合は、残りの溶液を処理するこの手順を繰り返します。
注: 列に基づく RNA の隔離は真空遠心分離法と互換性があります。 - エタノールの 200 μ L を 1 分の真空乾燥が続く列に追加します。
- RWT バッファーの 800 μ L を 2 分の真空乾燥が続く列に追加します。
- RPE バッファーの 800 μ L を 2 分の真空乾燥が続く列に追加します。
- 2.11 手順を繰り返します。
- エタノールの 300 μ L を 1 分の真空乾燥が続く列に追加します。
- 新しいチューブに列を置き、室温 (15 ~ 25 ° C) 1 分で 12,000 × g で遠心分離します。
- 列を新しいチューブに転送し、ヌクレアーゼ フリー水の 50 μ L を追加します。
- 3 分、室温 (15 ~ 25 ° C) で 8,000 × g で 1 分間遠心するため部屋の温度 (15 ~ 25 ° C) に立ちます。
- 再溶出液を列に適用します。
- 2.16、手順を繰り返し、使用するまで-80 ° c の溶出液を格納します。
3. cDNA 合成
- (ステップ 2.18) から凍結試料を解凍します。
-
Mirna ターゲットに対応する総合的な RNA オリゴヌクレオチドの既知濃度を準備します。
- QPCR で標準的な曲線を生成する RNA の合成オリゴヌクレオチドを使用します。1 x 108コピー/μ L 濃度のストック溶液を調製して、ストレージ目的のため。
- 原液を 10 倍希釈標準曲線の最高濃度として 1 x 107コピー/μ L (ない中古増幅サンプル) または 1 × 105コピー/μ L (中古増幅サンプル) 実用的なソリューションを取得します。一般に、標準曲線の濃度の推奨範囲は 1 x 107 1 x 102コピー/μ L (ない中古増幅サンプル) 1 × 105 1 × 100コピー/μ L (中古増幅サンプル)。
- ターゲットの miRNAs の 20 x RT プライマーの平等なボリュームを混合することによって、多重 RT プライマー プールを準備します。
注:図 2のように、最大 4 ターゲット Mirna を含むプールはプールの miRNAs のそれぞれの 20 x RT プライマーを混合することによって行うことが。Cel-ミール-238 (外部コントロール) は、各チューブのターゲット miRNAs の 1 つとして含める必要があります。少ない 4 ターゲット miRNAs の場合 20 x RT プライマーの代わりに 1/10 TE バッファーの平等なボリュームを追加します。 - RT 反応混合物の準備: RT プライマー (ステップ 3.3) からプールの dTTP の 100 mM dNTPs の 0.15 μ、1 μ L の逆転写酵素 (50 U/μ L)、RT バッファー x 1.5 μ 10、RNase 阻害剤 (20 U/μ L)、0.19 μ 3 μ と 4.16 μ L のヌクレアーゼ フリー水します。
- RT の反作用のミックス 10 μ L ピペットで 5 μ L の RNA のサンプル (ステップ 3.1) からまたはオリゴヌクレオチド (ステップ 3.2) から混ぜてし、氷上で 5 分間インキュベートします。
- サーマルサイクラー装置で逆のトランスクリプションを実行: 16 ° C、30 分、30 分、42 ° C の続きが 85 ° C で 5 分間で最終的な逆転写酵素不活性化ステップの-80 ° c ストア逆転写サンプルに使用されるまで。
4. プリアンプ (オプション)
注: Cq 以降 qPCR で 35 以上上記値を示す miRNAs はあらかじめ増幅です。
- (ステップ 3.6) から凍結試料を解凍します。
- 総合的な RNA オリゴヌクレオチド; から RT 製品の 10 倍のシリアル希薄を作る1 x 105 1 × 100コピー/μ L 標準曲線を生成するため。
- 200-fold 希釈されて各アッセイ プライマーの最終的な集中とターゲットの miRNAs のアッセイ プライマー x 20 の等しいボリューム (5 μ L) を混合することによって多重アッセイ プライマー プールを準備 1,000 μ L の最終巻で TE バッファーによって。
注: がターゲットの miRNAs は後続の qPCR 中古増幅と cel-ミール-238 (外部制御) のためのそれらなしで検出することができます分析プライマーはプライマー プールには含まれません。 - 中古増幅反応混合物の準備: 2 に使える preamplification 試薬、アッセイ プライマー プール (手順 4.3) からの 3.75 μ L、ヌクレアーゼ フリー水の 6.25 μ L × 12.5 μ L。
- 中古増幅反応のミックス 22.5 μ L ピペット (手順 4.1) から逆転写サンプルまたはオリゴヌクレオチド (ステップ 4.2) からの 2.5 μ L でミックスし、氷上で 5 分間加温します。
- サーマルサイクラー装置で反応を実行: 95 ° C、10 分;15 s と 60 ° C で 4 分間使用するまで-80 ° C で増幅された事前サンプルを店のため 95 ° c 12 サイクルが続きます。
5. 量的なリアルタイム PCR (qPCR)
- (ステップ 3.6 またはステップ 4.6) から凍結試料を解凍します。
- サンプルを 5 倍希釈滅菌水で。
- 総合的な RNA オリゴヌクレオチド; から派生した RT 製品の 10 倍のシリアル希薄を準備します。1 x 107 1 x 102コピー/μ L (中古増幅サンプルない) または 1 × 105 1 × 100コピー/μ L (中古増幅サンプル) 標準的な曲線を生成するために。
- QPCR 反応混合物の準備: すぐ使える増幅検出試薬、試金の試薬、前進/後進 PCR プライマーおよびプローブ ターゲット miRNA に対応するを含む x 20 の 1 μ L、ヌクレアーゼ フリー水の 7 μ L × 2 の 10 μ L。
- QPCR 反応混合物から高速光 96 ウェル反応板に 18 μ L を転送し、井戸に (ステップ 5.2 または 5.3 ステップ) から希釈サンプルの 2 μ L を追加します。
注: サンプルと qPCR の基準の重複設定されます。 - 接着フィルム、プレートを密封し、遠心分離機の簡潔に。
- リアルタイムのサーマルサイクラーで反応を実行: 95 ° C、20 s、95 ° c 45 サイクルが続く 1 s の 60 ° C、20 s。
6. データの分析
- 対応するリアルタイム サーマルサイクラーと連動するデータ解析ソフトウェアを使用して各サンプルのプレーン テキストのコピー数を計算します。
注: しきい値を表す線は、その Cq 値との比較による再現性を確認するため、研究では, すべてのプレートに「1.0」に手動で設定。カットオフ レベル Cq に設定 > 40 サイクル。 - 重複するコンテンツの各サンプルのプレーン テキストのコピー数の平均値を計算します。
- Cel-ミール-238 対応する管のすべてのサンプルからのコピー数の平均 cel-ミール-238 各サンプルでのコピー数で割って補正係数を計算します。
注:図 2のように、各チューブを cel-ミール-238 (外部コントロール) を含む 4 ターゲット miRNAs まで含まれています。したがって、cel-ミール-238 の対応する値を使用して各チューブの補正係数が計算されます。 - 各サンプルの (ステップ 6.3) から補正係数を (ステップ 6.2) から各サンプルの平均生コピー数を割ることによって調整されたコピー数を計算します。
- (ステップ 6.4) から各サンプルの調整済み raw コピー数を乗じてによって絶対コピー数を計算、各サンプルの希釈倍率。
注: 希釈倍率はステップ 5.2 から派生したこのプロトコルでは「5」です。 - ログ cDNA 濃度に対する各シリアル希釈の Cq 値のプロットの斜面からの PCR 増幅の効率を計算します。
注: 効率の計算式E = (10(-1 斜面/) -1) x 100%。 - Cel-ミール-238 式 E を使用してすべてのサンプルからの Cq 値を使用して技術的な変化の計算 = (2 x 増幅効果)ΔCt(Max(Cq)-Min(Cq))。
注: 手順 6.5 と 6.7、プロシージャの品質評価に使用されます。
結果
RT qPCR による miRNA アッセイのワークフロー、品質 assessment
図 1は、qPCR10を使用して血液サンプルからミルナ アッセイのワークフローを示しています。実験の品質は、cel-ミール-238 として外部からの制御を含む、検証できます。これは RNA の抽出の技術の変化を明らかにし、その後 RT qPCR 処理します。本研究では、50 サンプルから計算された Cq 値の平均 ± SD は 21.0 ± 0.4 (表 1) をだった。Cel ミール 238 中古増幅ステップのため希釈し、Cq の価値は 24.2 ± 0.4 (表 1) をだった。ここで説明する測定法、技術的な変化の範囲 Cq 価値観の違いに基づく 3 倍未満であると推定されました。変化の程度は、中古増幅する追加の手順が含まれているべき持っていた場合でも一貫性のある推移。これらの値は、本研究室 (データは示されていない) で実行されるマウス, ラットおよび人間など動物の種から他のサンプルのためのそれらと互換性があります。
ターゲットの miRNA の標準曲線から検出範囲
サンプルのない中古増幅
1 x 107コピー/μ L の濃度で総合的な RNA オリゴヌクレオチドから逆転写 (RT) 製品は、後続 qPCR の前に連続希釈。これらのシリーズは、中古増幅ステップなし検出されたすべての生物試料の一貫した取材を保障した標準曲線を生成するために使用されました。1 x 102コピー/μ L 濃度の標準試料の Cq 値に各ターゲットの miRNA のレベルをオフにカット一般的に近かった。したがって、検出限界は、1 × 102コピー/μ L (図 3) と推定されました。
中古増幅サンプル
中古増幅を必要とするサンプルの検出の範囲を決定するには、は、図 4に示すよう、希釈サンプルの 2 つの異なるシリーズが比較しました。標準カーブ A の生成、中古増幅ステップ前に RT 製品のシリアル希薄を調製しました。あらかじめ増幅基準のシリーズは後続の qPCR の使用されました。標準曲線 B を生成する最初の希釈にはあらかじめ増幅されたサンプルの 1 x 105コピー/μ L の濃度させられました。これらの標準的な曲線は明らかに異なる検出限界 (図 5) を示した。標準曲線は標準曲線 B は、1 コピー/μ L まで増加される線形の範囲を示したが 10 未満の濃度で特定の産物のため使用できませんでした2または 103コピー/μ L (図 5)。この結果は、miRNAs の非常に少量を中古増幅ステップとも評価できないことを示唆されました。Cq の値があらかじめ増幅されたサンプルを慎重に解釈する必要がありますさらに、> 30、あらかじめ増幅試料の検出限界があったのでこの値に近い。したがって、原則として標準曲線 B は、標準的なプロットの数が少ないを使用を避けるためにだけ検出範囲を決定した後、ここで説明する方法で使用されました。
したがって、定量化 (LLOQ) の下限 102コピー/μ L (図 3および図 5) miRNAs のほとんどのためであった。ミール-1 より高い LLOQ を示しただけ (103コピー/μ L) (図 5)。平均増幅効率 1.000 0.998 に至る標準曲線の相関係数 (R2) 約 90% であった。正確な RT qPCR 法の特徴は、線形 R2 0.98 と 90 ~ 11017の PCR 増幅効率よりも以上になると見なされます。したがって、アッセイの標準曲線の質を検証しました。
Mirna の少量のため中古増幅の効果
後続 qPCR で 35 以上上記 Cq 値を示した miRNAs はあらかじめ増幅.この手順には、miRNAs の少量の検出が強化されています。たとえば、非プレ増幅ミール 206 の Cq 値 (平均 ± SD) あった 37.9 ± 1.9、事前増幅ミール 206 の 27.0 ± 2.2 に減少しました。高 Cq 値 (図 6) でのサンプルの前のアンプを内蔵で重複した測定のペア間に有意差を認めた。対照的に、事前に増幅されたサンプルは重複 (図 6) でほぼ等しい値を示した。その中古増幅はサンプルの少量の場合のより正確で信頼性の高いデータを提供することに効果的であることが示唆されました。
カニクイザルにおけるプラズマ miRNAs のプロファイリング
確立された分析プラットフォームを使用して、猿 50 カニクイザルから 8 の miRNAs (ミール-1、ミール 122、ミール 133a、ミール 192、ミール 206、ミール 208a、ミール-208b およびミール 499a) の血漿中濃度分析 (図 7)10です。このアッセイでは、ミール-122、ミール-133a とミール 192 ミール 1 とミール-206、ミール 499a の低発現量のため中古増幅に必要なに対しだった中古増幅することがなく検出。しかし、ミール 208a もミール 208b 中古増幅によっても検出可能でした。データが正規分布していません。したがって、平均と標準偏差を計算する対数変換を行った。検討、ミール 122 miRNAs の間で小さいダイナミック レンジ (20 倍) 最高平均血漿中濃度 (104コピー/μ L × 5.71) を示した。対照的に、広いダイナミック レンジがミール - 1 (581-fold)、観察されたミール 133a (971-fold) とミール 206 (426-fold)。
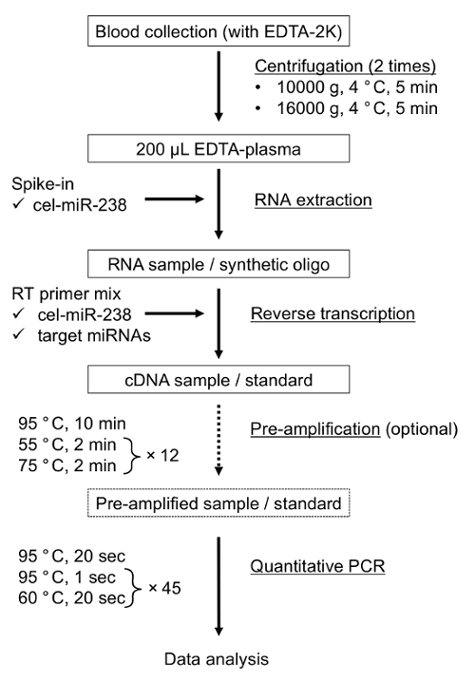
図 1: RT qPCR による miRNA アッセイのスケマティック ワークフロー 。この図の拡大版を表示するのにはここをクリックしてください。

図 2: MiRNA アッセイの逆のトランスクリプションのスケマティック ワークフロー 。最大 4 ターゲット Mirna を含むプールは、各プールの miRNAs の 20 x RT プライマーを混合することによって作成できます。Cel-ミール-238 (外部コントロール) は、各チューブのターゲット miRNAs の 1 つとして含める必要があります。この図の拡大版を表示するのにはここをクリックしてください。
| ミール 238 データ分析 | ||
| なし 中古増幅 | と 中古増幅 | |
| N | 50 | 50 |
| 意味 | 21.0 | 24.2 |
| SD | 0.4 | 0.4 |
| 最大 Cq | 21.8 | 25.3 |
| 分 Cq | 20.3 | 23.5 |
| 中央値 | 21.0 | 24.2 |
| 最大値最小値 | 1.3 | 1.8 |
| 増幅効果 | 89 | 89 |
| バリエーション | 2.4 | 2.8 |
表 1: cel-ミール-238 の定量化サイクル (Cq) 値を使用して評価します。技術の変化は、ミール-238 各アッセイでの Cq の値から評価しました。50 サンプルが (右) に対応する 2 つのグループに分けられたまたは中古増幅のステップ (左) なし。変化は、式 E を使用して算出した = (2 x 増幅効果)ΔCt(Max(Cq)-Min(Cq))。増幅効果は、標準曲線の傾きから算出しました。中古増幅の段階希釈された中古増幅ステップを必要とする試料 (ミール 238 自体は、あらかじめ増幅でした)。この図は、私たちのレポート10から変更されています。
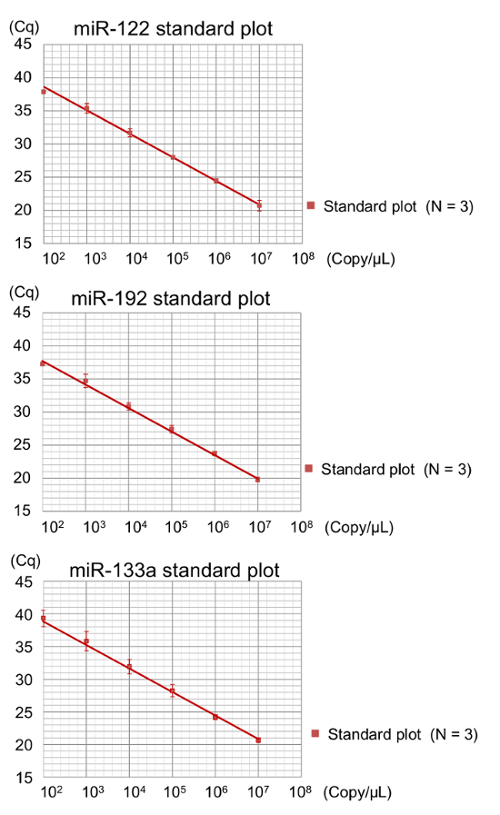
図 3:サンプルの前のアンプを内蔵の標準曲線のプロットします。標準曲線 (N = 3、重複) Cq。 線形回帰分析は、増幅効率に対応するスロープを決定する計算された対ログ濃度をプロットすることによって生成されました。ミール-122 (上側) の標準曲線にミール-192 (中央) とミール 133a (下) は試験範囲で Cq と濃度の線形関係を示した。標準曲線の誤差範囲は 3 つの独立した試金から 1 つの標準偏差を表します。この図の拡大版を表示するのにはここをクリックしてください。
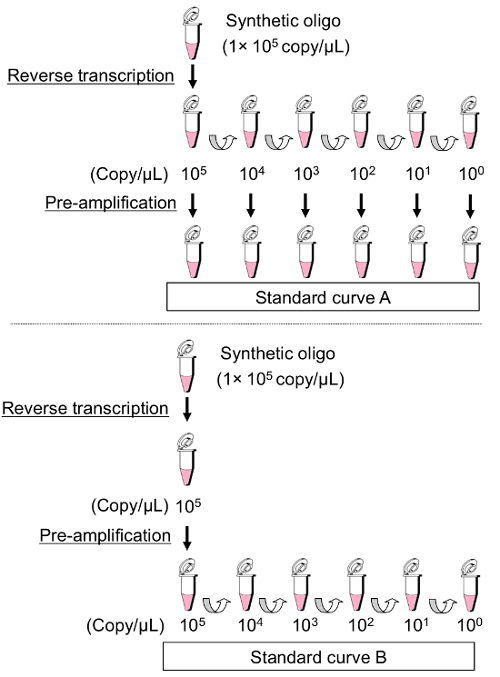
図 4: あらかじめ増幅されたサンプルの標準的な曲線を生成する手順です。(標準曲線 A)合成オリゴ (1 x 105コピー/μ L) の代表的な濃度は逆転写、標準試料の 10 倍連続希釈系列を作成し続きます。これらの事前増幅の基準は、後続の qPCR を使用しました。(標準曲線 B)転写し、あらかじめ増幅合成オリゴ (1 x 10 の5コピー/μ L) の代表的な濃度が逆だった。その後、後続の qPCR の前に標準試料の 10 倍連続希釈系列を調製しました。この図の拡大版を表示するのにはここをクリックしてください。
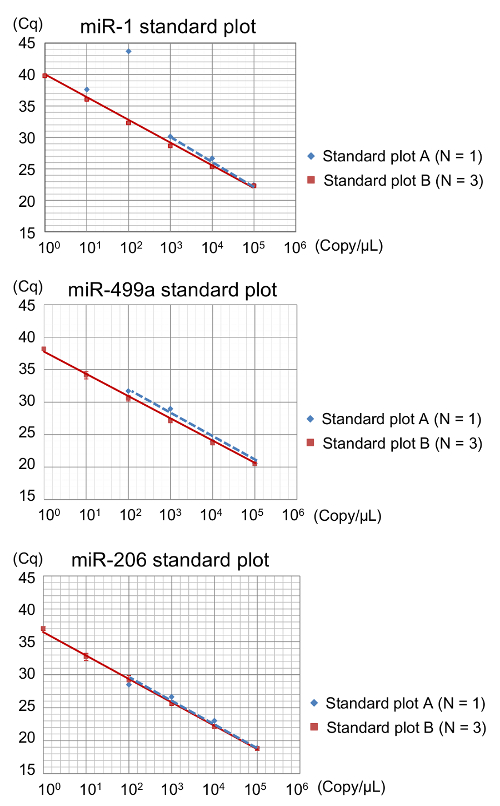
図 5: あらかじめ増幅されたサンプルの標準曲線 A と B のプロットします。標準曲線 A (n = 1、重複) と標準曲線 B (n = 3、重複) Cq。 線形回帰分析は、増幅効率に対応するスロープを決定する計算された対ログ濃度をプロットすることによって生成されました。(上部)標準曲線 B (実線) は、ミール-1 の試験範囲で Cq と濃度の線形関係を示したに対し、標準曲線 (点線) は 1 × 102コピー/μ L でどうやら非特異的増幅を示した。(中央と下)標準曲線 (点線) 1 x 102コピー/μ L またはそれ以下の濃度で私アンプリコンを示さなかった標準曲線 B に対し (実線) Cq と濃度の線形関係で示したミール 499a のテスト範囲とミール-206。標準曲線 B の誤差範囲は 3 つの独立した試金から 1 つの標準偏差を表します。この図の拡大版を表示するのにはここをクリックしてください。

図 6:非プレ増幅し、増幅前のサンプルの増幅プロット。(上部)ミール-206 (上側)、なしの代表的なサンプル (a、B、C) での増幅プロットまたは中古増幅 (下)。測定は、重複して設置されました。高い Cq サンプル (A および B) を中心に、中古のアンプを内蔵のサンプル内の重複としてセットアップ 2 つのサンプル間の相違点があった。この図の拡大版を表示するのにはここをクリックしてください。

図 7: カニクイザルにおけるプラズマ マイクロ Rna (Mirna) の絶対値を返します。Mirna の発現レベルは、ドット プロットによって表されます。対数変換後、平均と上記の意味 (グレーのボックスで示されている)、以下 2 つの標準偏差の値を求めた。この図は、私たちのレポート10から変更されています。この図の拡大版を表示するのにはここをクリックしてください。
ディスカッション
私たちの包括的な評価には、個々 のサンプルのばらつきの大きさだったテスト miRNAs の間で非常に異なることを明示するダイナミック レンジの範囲のより厳密な統計的分析が用意されています。これらのバリエーションは、その少量の体液中に起因するかもしれないが、ことこれらのデータ反映生物学的変化だけでなく、技術的な変化に留意。技術的な変化のほとんどは、他の分析プラットフォーム18で使用されている外部コントロール (cel-ミール-238) の Cq 値による評価できます。以来、miRNA 解析に標準化された内部統制をされているはありません、技術的な変化については試金19の品質を決定するための分析に含める必要があります。技術的な変化が 3 倍未満と推定されたこのレポートで説明した方法で。他の研究に匹敵する技術情報の不足のためこの手順の品質レベルを決定することはないが、このような情報を含む、信頼性の向上に貢献しての間の互換性を保証さまざまな研究。
最近の論文数は、検体の取扱い、保管条件、処理する前に記憶域の期間などさまざまな事前分析変数は、信頼性とマイクロ Rna 測定20を循環の再現性に影響を与えることができますを報告しています。,21します。 次の手順が変化を最小限に抑えるためにサンプル準備の間慎重に続いていたが、本研究では、事前分析変数の効果は考慮しなかった。まず、プラズマは、プラズマ8,22miRNAs の安定性の影響を最小限に抑える (内 2 時間後コレクション) できるだけ早く処理でした。Mirna は室温 (15 ~ 25 ° C) で安定するが、まだわかりにくい採血血漿または血清の処理間隔が miRNA レベルに影響するかどうか。第二に、各血漿サンプルは、miRNAs23の循環のレベルに影響を与える可能性があります交絡要因の 1 つを表す溶血赤血球混入に起因解析バイアスを除去するために視覚的に調べられました。
中古増幅検出バイアス24を導入することがなく生体の miRNAs の非常に小さい量の検出を改善するために役に立ちます。本研究では事前増幅合成オリゴヌクレオチドを使用して構築された標準曲線の Cq 値は法で高い再現性を示した。これは、miRNAs の少量の検出を有効にする事前増幅試料を用いた絶対定量可能。その一方で、低コピー数の基準 (1 x 102 103コピー/μ L) は、中古増幅との組み合わせで特定産物には表示されません。Mestdaghら。24報告 miRNA の結果であることができる逆のトランスクリプション、効率が低いため、番号の miRNAs 中程度または高いコピーと比較して低コピー数の miRNAs の測定が中古増幅のプロシージャの後の高い変動を示したシーケンスの特性。したがって、各 miRNA の標準曲線の検出範囲は、偽陽性を排除するために、あらかじめ増幅の miRNAs の特に試料の定量化される前に検証する必要があります。もっと重要なは、あらかじめ増幅と非プレ増幅サンプルなど異なる加工の miRNAs の絶対レベル比較できないこと直接各 miRNA であらかじめ増幅されたサンプルの数量が指定されていないので注意しなければなりません。
このレポートでは、Rt-qpcr を用いるプラズマの miRNA 絶対レベルの決定のための手順について説明します。中古増幅後もいくつかの miRNAs は検出されなかった。このような低式 Mirna を検出するには、別のアプローチを使用する必要があります。より多くのサンプル量を使用して単純なソリューションとしてこの問題を回避可能性があります、大量サンプルの常にご利用いただけません。日には、技術の進歩は、miRNA プロファイリング25,26のさまざまなプラットフォームの開発を有効にしています。液滴デジタル PCR (ddPCR) 最近開発された従来 Rt-qpcr 絶対定量化のために別の方法を提供しています。この革新的な技術は、1 コピー/μ L27,28のレベルでターゲットの miRNA を検出することができますの精度と再現性のある、敏感な方法であると報告されています。DdPCR は、miRNAs 中古増幅ステップなしの低濃度を検出できる場合、複数の miRNAs のレベルを比較することができますのでそれ大きな利点となります。使用する試薬分子プラットフォームとその後 RT qPCR 抽出手順の変化必然的に結果をもたらすしない変数。外部コントロールと個々 の miRNAs の絶対レベルの透明な結果が提供される場合、これらの変化は解決できます。メソッドの適切な品質評価は、miRNA プロファイリング研究の生物学的変化の洞察力を改善するための鍵です。
開示事項
著者には、利益相反を開示するはありません。
謝辞
本研究は、公共、商業、または非営利セクターの資金調達機関から任意の特定の助成金を受信しませんでした。
資料
| Name | Company | Catalog Number | Comments |
| BD Microtainer tube (K2EDTA) | Becton, Dickinson and Company | 365974 | For blood collection |
| Eppendorf PCR Tubes, 0.2 mL | Eppendorf | 0030124359 | |
| Eppendorf Safe-Lock micro test tubes 1.5 mL | Eppendorf | 0030120086 | |
| Eppendorf Safe-Lock micro test tubes 2.0 mL | Eppendorf | 0030120094 | |
| Synthetic oligonucleotide | Hokkaido System Science | - | Individual miRNA (0.2 μmol,HPLC grade) |
| Tris-EDTA Buffer (pH 8.0) | Nippon Gene | 314-90021 | TE buffer |
| Buffer RPE | QIAGEN | - | Contents in miRNeasy mini kit |
| Buffer RWT | QIAGEN | - | Contents in miRNeasy mini kit |
| miRNeasy Mini Kit | QIAGEN | 217004 | |
| Nuclease-Free Water | QIAGEN | 129114 | |
| QIAzol Lysis Reagent | QIAGEN | - | Contents in miRNeasy mini kit |
| Syn-cel-miR-238-3p miScript miRNA Mimic | QIAGEN | 219600 | ID:MSY0000293, 5 nmol |
| SC Adapters | TAIGEN Bioscience Corporation | S0120 | For RNA extraction |
| VacEZor 36 Complete System | TAIGEN Bioscience Corporation | M3610 | For RNA extraction |
| 7900HT Fast Real-Time PCR System | Thermo Fisher Scientific Inc. | 4351405 | Fast 96-Well Block |
| GeneAmp PCR System 9700 | Thermo Fisher Scientific Inc. | 9700 | |
| MicroAmp Fast Optical 96-Well Reaction Plate | Thermo Fisher Scientific Inc. | 4346907 | |
| MicroAmp Optical Adhesive Film | Thermo Fisher Scientific Inc. | 4311971 | |
| TaqMan Fast Advanced Master Mix | Thermo Fisher Scientific Inc. | 4444557 | |
| TaqMan MicroRNA Assays (cel-miR-238-3p) | Thermo Fisher Scientific Inc. | 4427975 | Assay ID: 000248 |
| TaqMan MicroRNA Assays (hsa-miR-122-5p) | Thermo Fisher Scientific Inc. | 4427975 | Assay ID: 002245 |
| TaqMan MicroRNA Assays (hsa-miR-133a-3p) | Thermo Fisher Scientific Inc. | 4427975 | Assay ID: 002246 |
| TaqMan MicroRNA Assays (hsa-miR-1-3p) | Thermo Fisher Scientific Inc. | 4427975 | Assay ID: 002222 |
| TaqMan MicroRNA Assays (hsa-miR-192-5p) | Thermo Fisher Scientific Inc. | 4427975 | Assay ID: 000491 |
| TaqMan MicroRNA Assays (hsa-miR-206) | Thermo Fisher Scientific Inc. | 4427975 | Assay ID: 000510 |
| TaqMan MicroRNA Assays (hsa-miR-208a-3p) | Thermo Fisher Scientific Inc. | 4427975 | Assay ID: 000511 |
| TaqMan MicroRNA Assays (hsa-miR-208b-3p) | Thermo Fisher Scientific Inc. | 4427975 | Assay ID: 002290 |
| TaqMan MicroRNA Assays (hsa-miR-499a-5p) | Thermo Fisher Scientific Inc. | 4427975 | Assay ID: 001352 |
| TaqMan MicroRNA Reverse Transcription Kit | Thermo Fisher Scientific Inc. | 4366597 | |
| TaqMan PreAmp Master Mix (2×) | Thermo Fisher Scientific Inc. | 4391128 | |
| Chloroform | Wako Pure Chemicals | 035-02616 | |
| Ethanol (99.5) | Wako Pure Chemicals | 057-00456 |
参考文献
- Schöler, N., Langer, C., Döhner, H., Buske, C., Kuchenbauer, F. Serum microRNAs as a novel class of biomarkers: a comprehensive review of the literature. Exp. Hematol. 38, 1126-1130 (2010).
- Viereck, J., Thum, T. Circulating Noncoding RNAs as Biomarkers of Cardiovascular Disease and Injury. Circ. Res. 120 (2), 381-399 (2017).
- D'Alessandra, Y., et al. Circulating microRNAs are new and sensitive biomarkers of myocardial infarction. Eur Heart J. 31 (22), 2765-2773 (2010).
- Chen, Y., Gelfond, J. A., McManus, L. M., Shireman, P. K. Reproducibility of quantitative RT-PCR array in miRNA expression profiling and comparison with microarray analysis. BMC Genomics. 10, 407 (2009).
- Nassirpour, R., et al. Identification of tubular injury microRNA biomarkers in urine: comparison of next-generation sequencing and qPCR-based profiling platforms. BMC Genomics. 15, 485 (2014).
- Derveaux, S., Vandesompele, J., Hellemans, J. How to do successful gene expression analysis using real-time PCR. Methods. 50 (4), 227-230 (2010).
- Bustin, S. A. Why the need for qPCR publication guidelines?--The case for MIQE. Methods. 50 (4), 217-226 (2010).
- Kroh, E. M., Parkin, R. K., Mitchell, P. S., Tewari, M. Analysis of circulating microRNA biomarkers in plasma and serum using quantitative reverse transcription-PCR (qRT-PCR). Methods. 50 (4), 298-301 (2010).
- Thompson, K. L., et al. Absolute Measurement of Cardiac Injury-Induced microRNAs in Biofluids across Multiple Test Sites. Toxicol Sci. 154 (1), 115-125 (2016).
- Iguchi, T., Niino, N., Tamai, S., Sakurai, K., Mori, K. Comprehensive Analysis of Circulating microRNA Specific to the Liver, Heart, and Skeletal Muscle of Cynomolgus Monkeys. Int. J. Toxicol. 36 (3), 220-228 (2017).
- Matsuzaka, Y., et al. Three novel serum biomarkers, miR-1, miR-133a, and miR-206 for Limb-girdle muscular dystrophy, Facioscapulohumeral muscular dystrophy, and Becker muscular dystrophy. Environ. Health. Prev. Med. 19 (6), 452-458 (2014).
- Starkey Lewis, P. J., et al. Circulating microRNAs as potential markers of human drug-induced liver injury. Hepatology. 54 (5), 1767-1776 (2011).
- Wang, K., et al. Circulating microRNAs, potential biomarkers for drug-induced liver injury. Proc. Natl. Acad. Sci. U. S. A. 106 (11), 4402-4407 (2009).
- Moldovan, L., Batte, K., Wang, Y., Wisler, J., Piper, M. Analyzing the Circulating MicroRNAs in Exosomes/Extracellular Vesicles from Serum or Plasma by qRT-PCR. Methods Mol. Biol. 1024, 129-145 (2013).
- Li, S., Chen, H., Song, J., Lee, C., Geng, Q. Avoiding heparin inhibition in circulating MicroRNAs amplification. Int. J. Cardiol. 207, 92-93 (2016).
- Cheng, H. H., et al. Plasma processing conditions substantially influence circulating microRNA biomarker levels. PLoS One. 8 (6), e64795 (2013).
- Serafin, A., et al. Identification of a set of endogenous reference genes for miRNA expression studies in Parkinson's disease blood samples. BMC Res. Notes. 7, 715 (2014).
- Akiyama, H., et al. A set of external reference controls/probes that enable quality assurance between different microarray platforms. Anal. Biochem. 472, 75-83 (2015).
- Kirschner, M. B., van Zandwijk, N., Reid, G. Cell-free microRNAs: potential biomarkers in need of standardized reporting. Front Genet. 4, 56 (2013).
- Malentacchi, F., et al. SPIDIA-RNA: second external quality assessment for the pre-analytical phase of blood samples used for RNA based analyses. PLoS One. 9 (11), e112293 (2014).
- Sourvinou, I. S., Markou, A., Lianidou, E. S. Quantification of circulating miRNAs in plasma: effect of preanalytical and analytical parameters on their isolation and stability. J Mol Diagn. 15 (6), 827-834 (2013).
- Yamaura, Y., Nakajima, M., Takagi, S., Fukami, T., Tsuneyama, K., Yokoi, T. Plasma microRNA profiles in rat models of hepatocellular injury, cholestasis, and steatosis. PLoS One. 7 (2), e30250 (2012).
- Kirschner, M. B., Edelman, J. J., Kao, S. C., Vallely, M. P., van Zandwijk, N., Reid, G. The Impact of Hemolysis on Cell-Free microRNA Biomarkers. Front Genet. 4, 94 (2013).
- Mestdagh, P., et al. High-throughput stem-loop RT-qPCR miRNA expression profiling using minute amounts of input RNA. Nucleic Acids Res. 36 (21), e143 (2008).
- Pritchard, C. C., Cheng, H. H., Tewari, M. MicroRNA profiling: approaches and considerations. Nat. Rev. Genet. 13 (5), 358-369 (2012).
- Moldovan, L., Batte, K. E., Trgovcich, J., Wisler, J., Marsh, C. B., Piper, M. Methodological challenges in utilizing miRNAs as circulating biomarkers. J. Cell. Mol. Med. 18 (3), 371-390 (2014).
- Mangolini, A., et al. Diagnostic and prognostic microRNAs in the serum of breast cancer patients measured by droplet digital PCR. Biomark. Res. 3, 12 (2015).
- Miotto, E., Saccenti, E., Lupini, L., Callegari, E., Negrini, M., Ferracin, M. Quantification of circulating miRNAs by droplet digital PCR: comparison of EvaGreen- and TaqMan-based chemistries. Cancer Epidemiol. Biomarkers Prev. 23 (12), 2638-2642 (2014).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved