Method Article
個々の腎細胞がんの中のタンパク質発現変動を探るために逆相タンパク質アレイ(RPPA)の使用
要約
RPPAは、蛍光標識抗体を用いて、同時に尋問するニトロセルローススライド上に印刷されたサンプルの何百ものタンパク質の発現を可能にします。このテクニックは、明細胞腎癌内薬物治療の異質の効果を研究するために適用されています。
要約
現在、転移性明細胞腎細胞癌に対する根治的治療法、病気の最も一般的な変形はありません。この治療抵抗性の重要な要因は、病気の1の分子複雑であると考えられている。例えば、チロシンキナーゼ阻害剤(TKI)スニチニブとして標的療法が利用されてきたが、患者のわずか40%は1年以内に2回再発、これらの患者の圧倒的多数で、対応させていただきます。腎細胞癌患者における内因性および獲得耐性のような質問のように、関連性の高い3です。
効果的な、パーソナライズされた治療法の開発を最終目標として、TKIsに対する抵抗性を研究するために、標的療法の具体的な期間の後、順次組織は、慢性骨髄性白血病4で成功した証明したアプローチが必要になります。しかし腎細胞癌におけるそのような戦略のアプリケーションは、bの高レベルによって複雑になる腎細胞癌5,6だけでなく、他の固形腫瘍7の特徴であるOTH間および腫瘍内不均一性。トランスクリプトームとの遺伝的差異に起因Intertumoral異質性は十分にあっても同様のプレゼンテーション、ステージと腫瘍の悪性度の患者では確立されています。それに加えて、さらに大きな分子多様性を表現する可能性がある、RCCに大きな形態学(腫瘍)異質性が、そこにあることは明らかである。結合された形態素解析とFuhrmanグレーディングによってRCC腫瘍の詳細なマッピングや分類は、プロテオーム解析のための代表的な領域を選択することができます。
RCC 8のタンパク質ベースの分析では、原因病理研究室でその広範な可用性に魅力的ですが、そのアプリケーションは、特異的抗体9の限られた有用性のために問題になることがあります。逆相タンパク質アレイのドットブロット性質(RPPA)、抗体の特異性がなければなりませんbに起因電子事前検証、使用する抗体のような厳密な品質管理としては極めて重要である。この制限にもかかわらず、ドットブロット形式は、単一のニトロセルローススライド上にサンプルの数の印刷が可能になり、アッセイの小型化が許可されています。印刷されたスライドは、その後、多重化を可能にする、標的特異的一次抗体と蛍光標識二次抗体を用いてウエスタン分析と同様の方法で分析することができます。スライド上のすべてのサンプル間の差動タンパク質の発現は、その後、より費用対効果の高い、高スループットの方法で蛍光の相対的なレベルを比較することによって、同時に解析することができます。
プロトコル
1。形態学的および分子腫瘍の不均一性の識別
- 腫瘍は、-80℃の冷凍庫から取り出し、ドライアイス上に保持。
- 約1cm 3の部分に腫瘍を分割します。各腫瘍互いに相対セクションとユニークな名前のラベルの元の位置をマップします。ストアサンプル-80個クライオバイアル℃·使用するための準備が整うまで。
- コートのサンプル10月と-22℃のクライオスタットでカット
- サンプルは、ヘマトキシリンおよびエオシン対比染色法を用いて染色した。
- 彼らはccRCC自然、生存腫瘍及び格付のための(凍結切片上Fuhrmanグレード1から4を区別するために挑戦低悪性度、高悪性度または混合高/低グレード)があることを確認するように凍結切片の顕微鏡分析図1&B(H&E染色高い低いと混合グレード)。
- タンパク質抽出のために各々の腫瘍内の各形態学的に異なる領域から最大で4つのサンプルを選択します。
2。腫瘍サンプルからのタンパク質抽出
- 10月からの腫瘍サンプルをカットします。
- 溶解バッファー990μLで2 mlチューブにティッシュの代わりに50から75ミリグラム[50mMトリス - 塩酸(pH7.5)、5mMのEGTA液(pH8.5)、150mMのNaCl]アプロチニン(シグマA6279)(10ミリグラムを補充/ ml)を、ホスファターゼ阻害剤カクテル2(シグマP5716)、ホスファターゼ阻害剤カクテル3(シグマP0044)およびプロテアーゼ阻害剤カクテル(Roche、11836153001)。
- 各チューブに単5ミリメートルの鋼球を追加し、二回各5分間経過後、TissueLyserを用いた均質化のレベルを確認する5分間50Hzでホモジナイズする。
- 後ろに鋼球を残して、ピペットを用いて新しい2 mlチューブに均質化したサンプルを転送します。
- 4℃で30分間13000×gで遠心分離する前に、各サンプルにTriton X-100を10μlを追加
- 新しいマイクロチューブに上清を移してください。
- BCAアッセイ( 図2)を用いてタンパク質濃度を決定します。
- 1 mg / mlとなるようにタンパク質濃度を正常化する。
3。抗体の検証
- ウェスタンブロットのために、タンパク質サンプルを(適切な細胞株や組織から抽出)を準備し、10%SDS-PAGEゲル上で実行されます。
- 4℃で一晩、ニトロセルロース膜上にサンプルを転送℃に
- 室温で1時間LI-CORのオデッセイブロッキングバッファー(PBSに50:50に希釈)で膜をブロックします。
- メーカーでLI-CORのオデッセイブロッキングバッファーで一次抗体(PBS中に50:50に希釈)希釈する希釈は1,000で、典型的には1をお勧めします。
- 4℃で一晩、一次抗体で膜をインキュベート
- 0.1パーセントのPBS-Tween20にて(; 1ミリリットルツイーン20 / 1L PBS PBS-T)を作る。
- 5分(×3)室温でPBS-Tで洗浄する。
- 1:10,000希釈(1.5μl/15ml)でSDS(PBS中で50:50に希釈)を0.01%ブロッキングバッファーオデッセイに蛍光標識二次抗体を希釈します。
- 二次で膜をインキュベート穏やかに振盪しながら45分間室温で抗体 - それが最終的にスキャンされているような時間まで光から膜を保護することが重要です。
- 暗闇の中で膜を保つ5分(×3)、室温でPBS-Tで洗浄する。
- 再び暗闇の中で膜を維持し、残留Tween20を削除するには、5分(×3)室温でPBS中で洗浄する。
- 暗闇の中で濾紙上に平坦な膜を嘘と空気乾燥することができます - メンブレンを乾燥させると、信号を強化し、バックグラウンドを低減するが、ストリッピング、再プロービングするため、それが役に立たなくなる可能性があります。
- LiCorオデッセイスキャナー上に膜をスキャンします。それをスキャンしたようになるまで、暗闇の中で膜を保つ。
- 唯一の正しい分子量の単一支配的なバンドを生成する抗体を選択する。 図3は RPPAで使用するために許容され、受け入れられない抗体の例を示します。
4。 RPPAぷりnting
- タンパク質可溶化液をマイクログリッドIIロボットスポッターを用いてニトロセルロースでコーティングされたガラススライド(Fastslides-Whatman社製)上にスポットした。使用されるスライドは、試料をスポットし、その上に2つのパッドを含んでいた。各パッドは同一のサンプル、この場合は100サンプルで発見された。他の利用可能なフォーマットは、1、8、16パッドのスライドが含まれています。パッドの数字が大きいほど小さく、それぞれにスポットできるサンプル数。
- 5 2倍希釈系列を、各サンプルから作られた、それぞれがその後(サンプルあたり15箇所の合計になります)三重で発見された。
5。 RPPAタンパク質検出
- 過剰LiCorブロッキングバッファー(PBSで希釈50:50)でぬれたスライド。ロッキングプラットフォーム上で室温で1時間インキュベートします。
- 所望の濃度のLiCorブロッキングバッファー(PBSに50:50に希釈)で一次抗体800μlのを準備し、氷上で保存する。
- は、(i)単一のフレーム·チップ·クリップでスライドをマウントタイトなシールがスライドとインキュベーションチャンバーとの間に形成されるように、または(ii) 'FastFrame' 4ベイのスライドホルダー。
- 井戸から残った液を取り除き、それぞれのウェルに600μlの一次抗体を加える。
- 一晩4プラットフォームを揺らしに密封されたウェットボックス、インキュベートにスライドとチャンバーを置き℃に
- 0.1パーセントのPBS-Tween20にて(;100μlのツイーン100分の20 mlのPBS PBS-T)を作る。
- 寒い部屋からスライドを取り外し、慎重に各ウェルからの一次抗体を除去します。
- PBS-Tを600μlを加え、5分間(X3)をRTでプラットフォームを揺らしでスライドを洗浄してください。
- オデッセイブロッキングバッファー(PBSに50:50に希釈)0.01%第一審の1:2,000希釈(1μL/ 2ミリリットル)のSDSで希釈することにより、蛍光標識二次抗体を準備します。
- 井戸からバッファを削除し、各ウェルに600μlの蛍光標識二次抗体を加える。穏やかに振とうしながら45分間室温で二次抗体をインキュベートする - それをそれが最終的にスキャンされているようになるまで、光から膜を保護することが重要です。
- 室温で600μlのPBS-Tでよく洗浄し、短時間(X3)からの二次抗体を除去する。キャリアからスライドを外し、適当な容器に移し、暗闇の中で膜を維持し、15分間超過PBS-Tで洗浄してください。
- 再び暗闇の中で膜を維持し、残留Tween-20を除去するために、PBS-T、15分間室温でPBSでさらに洗浄膜を取り除きます。
- 10分間、50℃のオーブンでFastslideを乾燥してから、LI-CORオデッセイのスキャナでスキャンしてください。それがスキャンされるまで、暗闇の中でスライドを保つ。
- 二次抗体/使用する抗体に応じて680 nmおよび/または800 nmでスライドをスキャンします。 2色の検出のために常に交差反応性を最小限にするために高度に架橋吸着した二次抗体を使用しています。一次抗体および二次抗体を慎重に選択するには、2つの色の検出のために必要である。第一に重要での選択である2次抗体に異なるホスト種( 例えば、ウサギ、マウス)。これは容易に区別できる発光スペクトルと色素で標識された抗ウサギ抗マウス二次抗体による差別ができます。
- 画像ファイルは、。TIFFファイルとして保存されます。 図4(スキャンしたファイルの画像)。
6。データ解析
- MicroVigeneソフトウェア(VigeneTech、カーライル、マサチューセッツ州、米国)を起動します。
- RPPAスライドのスキャンを含む。TIFFの画像ファイルを開きます。
- RPPAスライドの画像をオーバーレイするグリッドを持って事前に定義されたテンプレートファイルを選択します。
- クリックしてグリッドを起動するために、 関心領域 (ROI)ボタンの領域を定義します 。
- RPPAスポット上にグ リッドを配置します。 図6a(画像上にグ リッドの画像)。
- すべてのROIを強調表示するには、[ すべて選択]ボタンをクリックします。
- すべて検索]をクリックします。 MicroVigeneはAUTOMますatically ROIを見つけ、スポットを見つける、バックグラウンドを差し引き、ほこりを除去し、スポットを定量化する。
- RPPAスライド上のすべてのサンプルの結果を表示するために表示希釈曲線]ボタンをクリックします。
- 希釈のデータを保存]をクリックします 。
- として各サンプルはエラーのリスクを削減し、カーブフィッティングの質を向上させ、分析する15点が存在する三重に各5希釈ポイントにプリントされています。 MicroVigeneは、抗原抗体結合反応速度のS字曲線を生成するために全てのスポットを組み込んだ4パラメータロジスティックログモデル"Supercurve"アルゴリズム( 図6b)を生産しています。前提は同じ抗体-抗原結合反応速度は、一般的な応答曲線に合わせてアレイ上のすべてのスポットを取ることによって、このようにしても、別のサンプルでは、各試料スポットで行われていると、カーブフィッティング10,11の自信を高めることができるということです
Y = A +((BA)/(1 + E(C * D-LN(X)))
ここで、xはdilutioですnの係数とYは信号強度である。
サンプルは比較的我々の分析でsupercurveへそれらをマッピングした後のxの値の中間点でyの値に対応したY0値を用いて解析することができます。 - 図7のように、Microsoft ExcelとプロットY0でデータをエクスポートします。
- 腫瘍内タンパク質変動をANOVAの枠組みの中で未処理と処理治療を受けた患者のために別々に計算された。分析されたすべてのタンパク質からのデータを組み合わせる分散分布は、Mann-Whitney検定(MWT)に比較した。個々のタンパク質の腫瘍内分散はLillefoursとFlignerテストを用いて評価し、それぞれ正常と等分散性の仮定が開催されたF検定により比較した、偽の発見率(FDR)の補正が12を適用した。未処理および処理された患者のサンプル間の差動タンパク質発現は、スチューデントのt検定を用いて、各タンパク質について試験したところ正常と等分散性の仮定sがそうでなければ、MWTを行ったところ、満たされた、ルーズベルトは、結合されたt-検定とMWT値12の上に塗布した。タンパク質のタンパク質発現と分散ピアソン相関の有意性は、[R参照]とFDR応用12の標準的なアプローチを用いて推定された。
結果
スキャンRPPAスライドの例が示されて680と800nm の両方のチャネルを図4(i)で見ることができます。波長は、 図4(ii)で画像を分離することにより、解析対象とするRPPAスライドや図4(iii)を決定し、個々のタンパク質の発現に各パッドを有効にします。 図4に見られるように、(iii)のサンプル間の個々のタンパク質の発現がゲルゾリンははるかに低いタンパク質の発現を持っCMYCに比べてスライドを越え高レベルの発現を備えた一意のですが、それでも普遍的にすべてのサンプルを横切っ表明テストされています。これとは対照的に、CD10は、特定のサンプルがほとんど、あるいは全く発現が検出持つ他の試料にもかかわらず、高レベルの発現を与えると、変数式を行いました。最後pJAK2はバックグラウンドレベルより上の任意の検出可能なタンパク質発現を生み出すことができなかった。品質管理の観点からpJAK2の結果は、それらのに対し、容認できないでしょうCD10は、スライド上のサンプル数の検出されたタンパク質の発現レベルに起因する許容できるでしょう。 図5は、二次抗体とは別の種の一次抗体の間に交差反応性の欠如を強調しています。個々のスライドを横切ってサンプルを計算するために使用Supercurveの例を図6に見ることができます。これらの曲線は、 図7(AC)のように、相対的なタンパク質ベースの発現データを生成するために使用されています。 図7aは RPPAは、処理および未処理サンプル間の差異を検出することができますどのように強調表示3 RPPAのスライドにわたって代表タンパク質の発現レベルを、 図7bは焦点図7c 7aからのサンプルは前処理であることが判明しているより高いタンパク質発現を用いた治療、に基づいてグループ化された後、差動タンパク質発現レベル上図は、より高い腫瘍内に焦点を当ててサンプルは、ポスト処理サンプルで発見されて、より高い分散で、治療に基づいてグループ化された後のポスト処理サンプルの分散。
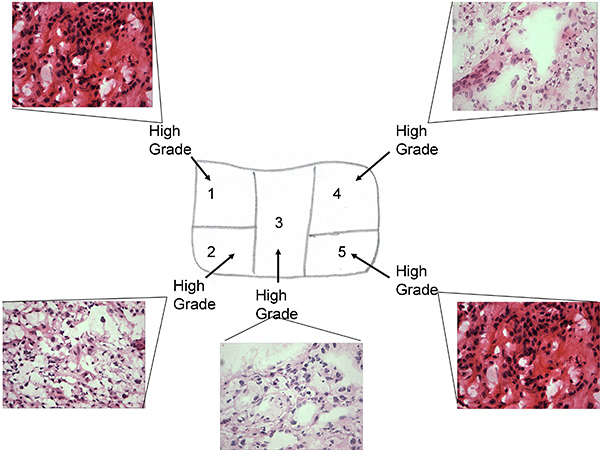
図1a。組織凍結切片の関連H&E染色による前治療組織のサンプルマップ
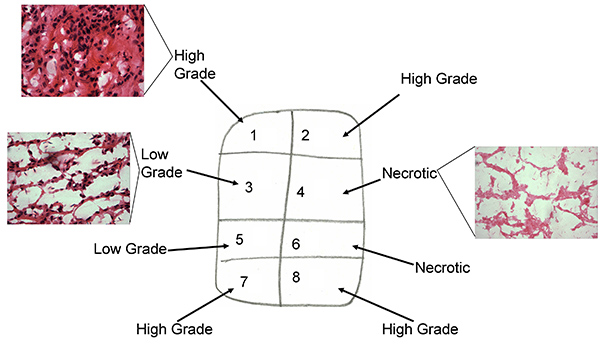
凍結切片の関連H&E染色による後処理の組織図1b。組織サンプルマップ
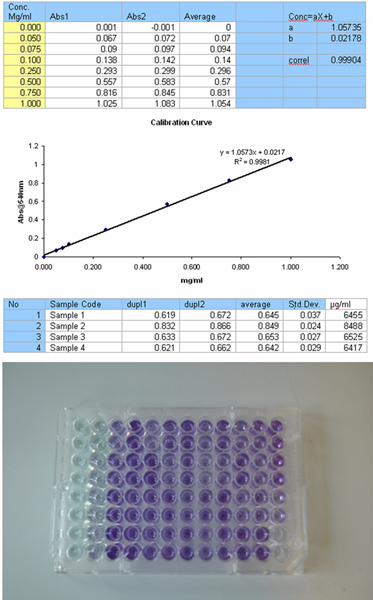
図2。タンパク質決定のためBradfordアッセイのための設定は、including検量線、結果表、96ウェルプレートフォーマット。 拡大図を表示するには、ここをクリックしてください 。
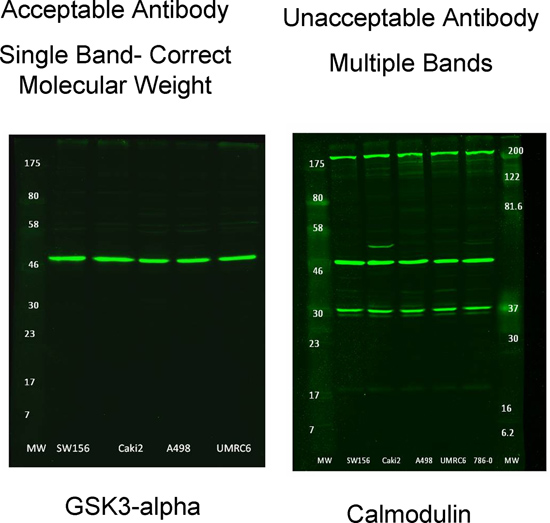
図3 RPPAでの使用に適したと不向き一次抗体の例

図4 RPPAスキャン処理のフロー図は、。 拡大図を表示するにはここをクリック 。
図5二次抗体および他の種の一次抗体の間で交差反応性の欠如の例。 680 nmと800 nmの波長の正しい正しくない波長でスキャンした二次抗体を用いたタンパク質発現の左画像検出。

図6aは。RPPAスライドスポットにグリッドをフィッティング。 拡大図を表示するには、ここをクリックしてください 。

図6b。RPPAスライド上のすべてのサンプルの比較分析のために使用さSuperCurveの例。グリーンスポットは "受け入れを表すできる解析ソフトウェアで事前定義された制限に基づいて外れ値を ""シミや赤を表す "。

図6c。目撃サンプルの再現性の例は、図示の例ではブランクに加え、単一試料の希釈液5の三乗のスポットが含まれています。
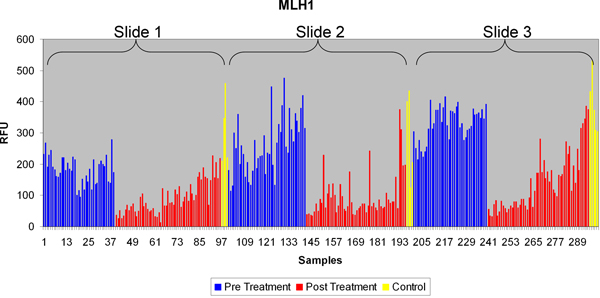
図7a。代表RPPA タンパク質のためのデータ(MLH1)は、複数のスライド間で比較。各サンプルは、腫瘍のサブ領域に対応する。

図7b。差別事前と事後処理サンプル用MLH1用Alタンパク質発現。
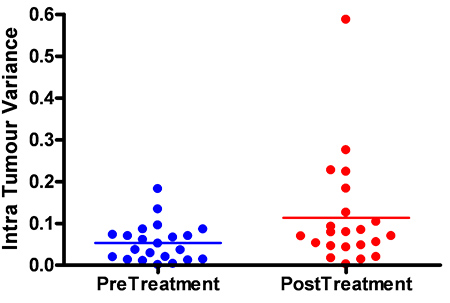
事前と事後処理サンプル間で発現のMLH1 7C。差異を示しています。
ディスカッション
ここで紹介するRPPA法はタンパク質解析の広く使われているが、比較的低いスループットウエスタンブロット法に高スループットの代替を表しています。この方法は、サンプルの数は半定量的に解析し、細胞株や組織サンプルの幅広い選択を越えて重要なタンパク質の直接比較を可能にし同時に比較することができます。異なる抗体種と多重化はさらに複数の抗体を同時に使用することができるようにする技術の電力を増加させる。ここで紹介する例では、腫瘍の不均一性と異質性についての標的療法の効果の研究でRPPAの能力を強調しています。
手法としてRPPAは、抗体の選択が非常に重要となっている重要なステップの数を持っています。技術の抗体特異性のドットブロットの性質のため、分析中に確認することができませんし、使用前に確認しておく必要があり。現在の研究では83抗体が有効であった唯一の58(〜30%の割合に失敗)に合格しているのウエスタンブロットにated。 RPPA(95%の成功率)の分析。その他の重要なステップはRPPAでサンプルを発見する濃度の選択にスライドだけでなく、スポッティングの均一性が含まれている場合に、これらの58のうち、55抗体は、許容可能な結果を与えた。高い濃度は、より敏感な結果が得られますが、存在している不十分な蛋白質による目撃されているサンプル数になることがあります。 RPPAスポッティングのための十分なタンパク質を提供する組織の最小量は、依存性腫瘍である。 RCCに50mgの最小はまだ1 mg / mlのスポッティング濃度を維持しながら腫瘍の複数のサブセクションでは、分析することを許可されて使用されていました。分析は、これがスライドはスポッティングした後の総タンパク質の抗体を使用して検証することができますが、全てのサンプルは、単一濃度で発見されていることを前提としていますようにスポッティングの均一性も重要です。最後の検討事項は、サンプル数が非常に高いときに使用するコントロールサンプルの使用であることここで例の事件があったように、それらは単一のスライド上にスポットすることができません。ここで使用した例では、腎細胞ラインからだけでなく、コントロールとしてHUVEC細胞ラインからタンパク質を組み込んだ。細胞系タンパク質の使用は、大量のタンパク質を抽出し、複数のスライドに使用することができます。コントロールは、スポッティングまたは抗体インキュベーションに起因するいかなる固有誤差を補正し、別の日に使用されている別のユーザーやスライドのための偏差を補正します。
技術としてRPPAは、使用するサンプルの数につきましては柔軟性があります。ここに示す例では、各スライド上の2つのパッドと、3つのスライドに広がった45の腫瘍試料から〜300のサンプルを使用し提出した。あるいは、現在のフォーマットが1パッドを備えた単一のスライド上にスポットされた可能性があります。そのような形式は、実験の個々のニーズに合わせて調整することができ、スライド上の複数のパッドはそれ以上の抗体を同時に使用することができますが、SAMの数を制限するよう公正妥当。
開示事項
特別な利害関係は宣言されません。
謝辞
ETM37と癌研究英国の実験的がん医療センターでサポートされている:チーフサイエンティストオフィス、承認番号によって運営されている上記の作家FCO、DF西澤、DJHとGDSの仕事は言及した。 ALの仕事はエディンバラロバート·トラスト、ケア、がんや医療研究評議会の治療のためのメルヴィル·トラストの外科医のロイヤルカレッジによって賄われています。 IOはマリー·キュリー·アクションと英国医学研究評議会によってcofundedエジンバラスコットランド政府フェローシップの王立協会によってサポートされています。著者らは、この論文に記載された内容の一部に彼らの有益な議論のためにSCOTRRCC共同申請者と協力者に感謝したいと思います。
資料
| Name | Company | Catalog Number | Comments |
| 試薬の名称 | 会社 | カタログ番号 | |
| アプロチニン | シグマ | A6279 | |
| ホスファターゼ阻害剤カクテル2 | シグマ | P5726 | |
| ホスファターゼ阻害剤カクテル3 | シグマ | P0044 | |
| プロテアーゼ阻害剤カクテル | ロッシュ | 11836153001 | |
| トリトンX-100 | トリトン-X | T8787 | |
| ブロッキングバッファーLI-CORのオデッセイ | LI-COR | 927から40000 | |
| TissueLyserを | キアゲン | 85600 | |
| マイクログリッドIIロボットスポッター | Biorobotics | ||
| FastFrame '4ベイのスライドホルダー | ワットマン | 10486001 | |
| FASTスライド - 2 - パッド | ワットマン | 10485317 | |
| IRDye 680LTヤギ抗マウスIgG | Licor | 926から68020 | |
| IRDye 800CWヤギ抗ウサギIgG | Licor | 926から32211 |
参考文献
- Stewart, G. D., O'Mahony, F. C., Powles, T., Riddick, A. C., Harrison, D. J., et al. What can molecular pathology contribute to the management of renal cell carcinoma. Nat. Rev. Urol. 8, 255-265 (2011).
- Rini, B. I., Michaelson, M. D., Rosenberg, J. E., Bukowski, R. M., Sosman, J. A., et al. Antitumor activity and biomarker analysis of sunitinib in patients with bevacizumab-refractory metastatic renal cell carcinoma. J. Clin. Oncol. 26, 3743-3748 (2008).
- Swanton, C., Larkin, J. M., Gerlinger, M., Eklund, A. C., Howell, M., et al. Predictive biomarker discovery through the parallel integration of clinical trial and functional genomics datasets. Genome Med. 2, 52 (2010).
- Cortes, J., Jabbour, E., Kantarjian, H., Yin, C. C., Shan, J., et al. Dynamics of BCR-ABL kinase domain mutations in chronic myeloid leukemia after sequential treatment with multiple tyrosine kinase inhibitors. Blood. 110, 4005-4011 (2007).
- Gerlinger, M., Rowan, A. J., Horswell, S., Larkin, J., Endesfelder, D., et al. Intratumor heterogeneity and branched evolution revealed by multiregion sequencing. N. Engl. J. Med. 366, 883-892 (2012).
- Fisher, R., Larkin, J., Swanton, C. Inter and intratumour heterogeneity: a barrier to individualized medical therapy in renal cell carcinoma. Front. Oncol. 2, 49 (2012).
- Yachida, S., Jones, S., Bozic, I., Antal, T., Leary, R., et al. Distant metastasis occurs late during the genetic evolution of pancreatic cancer. Nature. 467, 1114-1117 (2010).
- O'Mahony, F. C., Faratian, D., Varley, J., Nanda, J., Theodoulou, M., et al. The use of automated quantitative analysis to evaluate epithelial-to-mesenchymal transition associated proteins in clear cell renal cell carcinoma. PLoS One. 7, e31557 (2012).
- Spurrier, B., Ramalingam, S., Nishizuka, S. Reverse-phase protein lysate microarrays for cell signaling analysis. Nat. Protoc. 3, 1796-1808 (2008).
- Hu, J., He, X., Baggerly, K. A., Coombes, K. R., Hennessy, B. T., et al. Non-parametric quantification of protein lysate arrays. Bioinformatics. 23, 1986-1994 (2007).
- Wang, X., Dong, Y., Jiwani, A. J., Zou, Y., Pastor, J., et al. Improved protein arrays for quantitative systems analysis of the dynamics of signaling pathway interactions. Proteome Sci. 9, 53 (2011).
- Benjamini, Y., Hochberg, Y. Controlling the False Discovery Rate: A Practical and Powerful Approach to Multiple Testing. Journal of the Royal Statistical Society Series B (Methodological). 57, 289-300 (1995).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved