Method Article
Un metodo scalabile basato su celle per la valutazione funzionale delle varianti Ube3a
In questo articolo
Riepilogo
È stato sviluppato un metodo semplice e scalabile per valutare il significato funzionale delle varianti missenso in Ube3a, un gene la cui perdita e guadagno di funzione sono legati sia alla sindrome di Angelman che al disturbo dello spettro autistico.
Abstract
L'aumento dell'uso del sequenziamento in medicina ha identificato milioni di varianti codificanti nel genoma umano. Molte di queste varianti si verificano in geni associati a disturbi dello sviluppo neurologico, ma il significato funzionale della stragrande maggioranza delle varianti rimane sconosciuto. Il presente protocollo descrive lo studio di varianti per Ube3a, un gene che codifica per una ubiquitina ligasi E3 legata sia all'autismo che alla sindrome di Angelman. La duplicazione o triplicazione di Ube3a è fortemente legata all'autismo, mentre la sua delezione causa la sindrome di Angelman. Pertanto, comprendere la valenza dei cambiamenti nell'attività della proteina UBE3A è importante per i risultati clinici. Qui viene descritto un metodo rapido basato su cellule che accoppia le varianti di Ube3a con un reporter di percorso Wnt. Questo semplice test è scalabile e può essere utilizzato per determinare la valenza e l'entità dei cambiamenti di attività in qualsiasi variante di Ube3a . Inoltre, la facilità di questo metodo consente la generazione di una ricchezza di informazioni struttura-funzione, che possono essere utilizzate per ottenere informazioni approfondite sui meccanismi enzimatici di UBE3A.
Introduzione
I recenti progressi tecnologici hanno reso il sequenziamento di esomi e genomi di routine in ambito clinico 1,2. Ciò ha portato alla scoperta di un gran numero di varianti genetiche, tra cui milioni di varianti missenso che tipicamente cambiano un amminoacido in una proteina. Comprendere il significato funzionale di queste varianti rimane una sfida, e solo una piccola frazione (~2%) delle varianti missenso conosciute ha un'interpretazione clinica 1,3.
Un esempio importante di questo problema è Ube3a, un gene che codifica per una ubiquitina ligasi E3 che prende di mira le proteine del substrato per la degradazione4. Ube3a risiede all'interno del cromosoma 15q11-13 ed è espresso esclusivamente dall'allele 5,6,7 ereditato per via materna. Le osservazioni della genetica delle malattie suggeriscono fortemente che l'attività insufficiente o eccessiva dell'enzima UBE3A causa distinti disturbi dello sviluppo neurologico. La delezione del cromosoma materno 15q11-13 causa la sindrome di Angelman (AS)8, un disturbo caratterizzato da grave disabilità intellettiva, menomazioni motorie, convulsioni, un contegno felice con frequenti sorrisi e caratteristiche facciali dismorfiche 8,9,10. Al contrario, la duplicazione o triplicazione del cromosoma materno 15q11-13 causa la sindrome di Dup15q, una condizione eterogenea riconosciuta come una delle forme sindromiche più diffuse di autismo11,12,13. Inoltre, ci sono centinaia di varianti missenso identificate in Ube3a, la maggior parte delle quali sono considerate varianti di significato incerto (VUS) in quanto il loro significato funzionale e clinico è sconosciuto. Pertanto, vi è un notevole interesse nello sviluppo di metodi in grado di valutare empiricamente le varianti di Ube3a per determinare se contribuiscono alla malattia dello sviluppo neurologico.
L'enzima UBE3A appartiene alla famiglia di domini HECT (omologa a E6-AP C-terminale) delle ubiquitina ligasi E3 che possiedono tutte l'omonimo dominio HECT, che contiene il macchinario biochimico necessario per accettare l'ubiquitina attivata dagli enzimi E2 e trasferirla alle proteine del substrato14. Storicamente, la caratterizzazione degli enzimi E3 si è basata su sistemi in vitro ricostituiti che richiedono un insieme di proteine purificate 4,15,16. Tali metodi sono lenti e laboriosi e non suscettibili di valutare l'attività di un gran numero di varianti. In lavori precedenti, UBE3A è stato identificato per attivare la via Wnt nelle cellule HEK293T modulando la funzione del proteasoma per rallentare la degradazione della β-catenina17. Questa intuizione consente l'uso di Wnt pathway reporter come metodo efficiente e rapido per identificare sia le varianti di perdita che di guadagno di funzione di Ube3a18. Il protocollo seguente descrive in dettaglio un metodo per generare varianti di Ube3a e un reporter basato sulla luciferasi per valutare i cambiamenti nell'attività delle varianti di Ube3a.
Protocollo
1. Clonazione mutagenetica per generare varianti di Ube3a
- Clonare tutte le varianti di Ube3a nel plasmide pCIG2 (Figura 1A), un vettore bicistronico che contiene un promotore di pollo-β-actina e un sito di ingresso interno del ribosoma (IRES) per l'espressione di EGFP19. I costrutti Ube3a a lunghezza intera contengono una sequenza di tag Myc-terminali N-terminali e sono clonati in pCIG2 usando un sito SacI di 5' e un sito XmaI di 3'. Inoltre, i siti EcoRI, EcoRV e PstI presenti in natura all'interno della sequenza di codifica Ube3a vengono utilizzati anche per subclonare i frammenti.
NOTA: Le varianti de-identificate per Ube3a possono essere ottenute attraverso la letteratura e in archivi accessibili al pubblico come il database ClinVar1. Ulteriori varianti non segnalate possono essere ottenute attraverso la comunicazione personale con i centri medici. - Utilizzare un metodo di mutagenesi megaprimer adattato in due fasi20 per introdurre mutazioni nel frame di lettura Ube3a . Nella prima fase, progettare l'oligonucleotide mutageno che contiene la mutazione desiderata (l'esempio mostrato in questo protocollo è quello di generare una variante UBE3A Q588E) affiancato da almeno 30 coppie di basi (bp) di omologia 5′ e 3′ alla mutazione (Figura 1B e Tabella 1).
- Utilizzare l'oligonucleotide mutageno insieme a un oligonucleotide complementare per la PCR del primo ciclo per generare un megaprimer da 200-400 bp contenente la mutazione (Figura 1C; Tabella 2 e tabella 3). Utilizzare l'intero volume di reazione PCR di 50 μL per l'elettroforesi su gel di agarosio utilizzando un gel all'1% e successiva purificazione del gel (Tabella dei materiali). Eluire il prodotto PCR in 30 μL di H 2 O deionizzato(diH2O) per l'uso nella fase del secondo ciclo di PCR riportata di seguito.
- Impostare il secondo ciclo di PCR come indicato (Figura 1C; Tabella 2 e tabella 3). Questa fase genererà frammenti di DNA più grandi (tipicamente ~1-1,5 kb di lunghezza) contenenti i siti di mutazione e restrizione terminale adatti per la sub-clonazione (Figura 1C).
- Dopo il completamento della reazione PCR, purificare il prodotto risultante utilizzando un kit di purificazione PCR (Table of Materials). Eluire in 30 μL e digerire tutto il prodotto purificato e il costrutto pCIG2 Ube3a WT con gli enzimi di restrizione appropriati (l'esempio mostra la digestione con EcoRI e XmaI).
- Dopo 2 ore di digestione a 37 °C, risolvere i prodotti della digestione mediante elettroforesi su gel di agarosio (gel all'1%) e gelificare i prodotti appropriati. Impostare le legature secondo il protocollo del produttore (Table of Materials), trasformare i prodotti di legatura in un ceppo batterico DH10B (Table of Materials) e quindi placcare con ampicillina (100 μg / mL) selezione.
- Il giorno successivo, scegliere da due a quattro colonie per inoculare 3 mL di colture contenenti Luria Brodo (LB) e 100 μg/mL di ampicillina. Lasciare crescere le colture durante la notte a 37 °C in un incubatore orbitale con agitazione (225 rpm).
- Il giorno seguente, utilizzare 1-1,5 ml di coltura batterica per miniprep il DNA plasmidico (Table of Materials). Salvare la coltura rimanente posizionandola a 4 °C. Schermare i plasmidi minipreparati mediante sequenziamento Sanger utilizzando un oligonucleotide che lega ~ 200 bp dalla mutazione desiderata.
- Utilizzare le colture batteriche salvate a 4 °C per reinoculare colture da 50 ml per midiprep il plasmide corretto (Table of Materials). Sequenziare completamente la sequenza codificante Ube3a per convalidare la sequenza corretta del DNA.
- Creare stock di plasmidi della variante Ube3a, così come il reporter attivato da β-catenina (BAR)21, TK-Renilla luciferasi, Ube3a morto in ligasi (mutazione UBE3A C820A) e il vettore vuoto pCIG2 a una concentrazione di 100 ng / μL utilizzando H2O sterile per le successive fasi di trasfezione.
2. Preparazione e trasfezione di cellule 293T (HEK293T) del rene embrionale umano (HEK293T)
- Eseguire i test in cellule HEK293T coltivate nel mezzo Eagle modificato (DMEM) di Dulbecco pre-integrato con glutammina, siero bovino fetale al 10% (FBS) e agente antibiotico-antimicotico come descritto in precedenza17. Far crescere le cellule in un incubatore sterile e umidificato a 37 °C con il 5% di CO2.
- Utilizzando un armadio di biosicurezza, prima placcare le cellule HEK293T sospese su piastre a fondo piatto trattate con colture tissutali a 96 pozzetti a una densità di 2,2 × 104 cellule per pozzetto in 100 μL di terreno di coltura e lasciarle crescere durante la notte.
- Il secondo giorno, trasfettare le cellule in un armadio di biosicurezza con plasmidi reporter Firefly e Renilla luciferasi (Tabella 4) e plasmidi Ube3a in triplice copia. Eseguire le trasfezioni in 10 μL di volume totale per pozzetto utilizzando un rapporto 10:1:8 di plasmidi (50 ng di BAR, 5 ng di TK-Renilla e 40 ng di plasmide Ube3a per pozzetto, rispettivamente), come descritto in precedenza18, e 0,4 μL di reagente di trasfezione (Tabella 4).
NOTA: Per ogni esperimento, un vettore vuoto (solo GFP) e un plasmide che codifica Ube3a morto di ligasi sono anche trasfettati per fungere da controlli negativi. - Creare un master mix per le trasfezioni per ogni variante in una provetta da microcentrifuga da 1,5 ml (Tabella 4) e incubare a temperatura ambiente (RT) per 15 minuti. Dopo l'incubazione, agitare delicatamente la miscela di trasfezione toccando il tubo, quindi aggiungere 10 μL della miscela di trasfezione direttamente al terreno di crescita esistente nei pozzetti.
- Successivamente, riportare le cellule all'incubatore e consentire ai plasmidi di esprimersi per 48 ore. La sostituzione dei substrati di crescita non è necessaria.
- Monitorare l'efficienza di trasfezione mediante fluorescenza GFP utilizzando un microscopio a fluorescenza. Le cellule HEK293T sono trasfettate in modo efficiente con questo metodo e >80% delle cellule dovrebbe esprimere GFP dopo 48 ore.
3. Misurazione dell'attività della luciferasi
NOTA: L'attività della luciferasi viene valutata utilizzando un sistema disponibile in commercio che analizza sia Firefly che Renilla luciferasi (Table of Materials) secondo il protocollo del produttore.
- Aspirare accuratamente i terreni di coltura dalle cellule HEK293T trasfettate, quindi lavare le cellule con soluzione salina tamponata con fosfato freddo (PBS). Aspirare il PBS e lisare le cellule aggiungendo 25 μL di tampone di lisi non denaturante e incubare per 15 minuti con un leggero dondolio sul ghiaccio.
- Successivamente, aggiungere 20 μL del lisato risultante (non è necessaria alcuna centrifugazione) in una nuova piastra di dosaggio a 96 pozzetti contenente 100 μL di un reagente substrato della luciferasi Firefly. Mescolare delicatamente il piatto picchiettando.
- Caricare la piastra su un lettore di piastre e misurare la luminescenza utilizzando un rilevatore superiore con un'altezza di lettura di 1 mm e un tempo di integrazione di 1 s.
NOTA: Potrebbe essere necessario regolare il guadagno del rilevatore in base alla potenza del segnale. - Subito dopo aver letto la luminescenza della Luciferasi Firefly, aggiungere 100 μL di substrato di Renilla luciferasi ai pozzetti. Questo passaggio spegne simultaneamente l'attività della luciferasi Firefly mentre valuta l'attività della renilla luciferasi. Mescolare i campioni agitando delicatamente la piastra e misurare nuovamente la luminescenza per valutare l'attività della Renilla luciferasi.
NOTA: Mentre altre piastre possono essere utilizzate per questo test, l'uso di piastre con cornici nere e pozzetti bianchi fornirà i risultati più sensibili in quanto ciò amplifica il segnale della luciferasi riducendo al minimo il rumore. - Quantificare le risposte del reporter come rapporto Luciferasi di Firefly / Renilla luciferasi (Firefly / Renilla) e calcolare la media dei valori delle misurazioni triplice. Successivamente, normalizzare il valore Firefly/Renilla per ciascuna variante rispetto ai valori per le cellule che esprimono Ube3a wild-type (WT) per confrontare l'attività relativa delle proteine varianti.
Risultati
Lo screening funzionale su larga scala delle varianti missenso di Ube3a identifica un ampio panorama di mutazioni con perdita e guadagno di funzione
Precedenti lavori con mutanti Ube3a hanno suggerito che la risposta Wnt può servire come reporter dell'attività cellulare della proteina UBE3A. Queste osservazioni sono state ampliate e sono stati eseguiti ulteriori esperimenti di convalida per indagare se il test BAR è adatto a segnalare una serie di attività UBE3A nella cellula. In primo luogo, le cellule HEK293T sono state trasfettate con quantità variabili di DNA plasmidico che codifica WT Ube3a umano. Questo esperimento ha dimostrato che la risposta BAR cambia linearmente con la quantità di Ube3a trasfettata nelle cellule (Figura 2A). In secondo luogo, il test BAR è stato testato utilizzando DNA plasmidico che codifica varianti legate alla malattia precedentemente caratterizzate in letteratura22. Tra questi c'erano varianti benigne (R39H e A178T), una variante di guadagno di funzione legata all'autismo (T458A) e una raccolta di varianti confermate legate alla sindrome di Angelman che causano una perdita della funzione UBE3A attraverso vari meccanismi15,22. Il saggio BAR ha identificato correttamente ciascuna variante (Figura 2B), comprese tutte le varianti con perdita di funzione, dimostrando l'accuratezza e la generalità di questo metodo18.
Il test BAR è stato utilizzato per esaminare 152 varianti missenso, la maggior parte delle quali sono state identificate dal database ClinVar. Analogamente alle prove genetiche riguardanti le variazioni del numero di copie nei disturbi dipendenti da Ube3a, l'impatto funzionale delle varianti missenso era ampio e includeva varianti benigne, nonché varianti con perdita di funzione e guadagno di funzione (Figura 3)18. Di particolare interesse, le varianti di guadagno di funzione identificate utilizzando questo metodo erano tutte nuove, e la successiva convalida ha fortemente suggerito che definissero una sottoclasse di disturbi dipendenti da Ube3a con fenotipi che differiscono dalla classica sindrome di Angelman18.
L'effetto delle varianti missenso è spesso imprevedibile, sottolineando la necessità di una valutazione funzionale
Un'osservazione interessante dallo screening funzionale delle varianti di Ube3a è che i cambiamenti in alcune posizioni degli amminoacidi producono effetti drasticamente diversi, sottolineando così l'importanza della valutazione empirica delle varianti18. Ad esempio, tre individui sono stati identificati con varianti nella posizione glutammina 588 (Q588) in UBE3A che includevano una sostituzione del glutammato con guadagno di funzione (Q588E), una sostituzione dell'arginina con perdita di funzione (Q588R) e un'altra sostituzione della prolina con perdita di funzione (Q588P; Figura 4A). È stato utilizzato un approccio mutazionale completo per mutare la posizione del Q588 in ogni possibile amminoacido. Quando l'attività di queste varianti è stata misurata utilizzando il saggio BAR, ha mostrato che qualsiasi amminoacido caricato negativamente nella posizione 588 produce un enzima iperattivo (Figura 4B). Questa intuizione ha fornito importanti indizi funzionali che hanno permesso la scoperta di un nuovo sito all'interno del dominio HECT di UBE3A che facilita l'allungamento della catena dell'ubiquitina18.

Figura 1: Mutagenesi PCR-dipendente di Ube3a. (A) Rappresentazione del vettore di espressione di Ube3a che indica i siti di restrizione rilevanti. (B) La sequenza di DNA che codifica UBE3A è mostrata in alto. Il codone Q588 è indicato da caratteri in grassetto. Il primer di mutagenesi Q588E (Mut. primer) è mostrato allineato alla sequenza UBE3A. La scritta rossa indica il sito mutagenizzato. (C) Viene mostrato uno schema della PCR per generare il megaprimer e il frammento di DNA utilizzato per subclonare il frammento Q588E. La posizione della mutazione Q588E è indicata dall'asterisco rosso. Fare clic qui per visualizzare una versione ingrandita di questa figura.

Figura 2: Validazione del test BAR come reporter dell'attività dell'ubiquitina ligasi UBE3A. (A) Le cellule HEK293T trasfettate con quantità crescenti di plasmide codificante WT UBE3A mostrano un aumento lineare della risposta BAR. I valori medi sono mostrati per i rapporti Firefly/Renilla luciferasi ± errore standard (SE). N= 3 esperimenti indipendenti. (B) Le varianti Ube3a precedentemente caratterizzate sono state testate nel test BAR. Le risposte sono state normalizzate a WT UBE3A e i valori sono mostrati come media ± SE. Il numero di esperimenti (n) e valori p calcolati utilizzando un t-test a un campione (a due code) con correzione dei confronti multipli di Benjamini-Hochberg (FDR = 0,05) sono i seguenti: GFP, n = 19, ***p = 1,733 × 10−31; WT UBE3A, n = 19; UBE3A LD, n = 19, ***p = 1,519 × 10−31; R39H, n = 3, p = 0,567; A178T, n = 4, p = 0,828; T485A, n = 5, *p = 0,019; C21Y, n = 3, ***p = 8,725 × 10−6; C117R, n = 3, ***p = 3,419 × 10−4; N243K, n = 3, **p = 0,0072; E269G, n = 3, **p = 7,787 × 10−4; L273F, n = 3, **p = 0,0052; S349P, n = 3, **p = 0,0016; L502P, n = 3, **p = 0,0033; R506C, n = 3, **p = 0,0033; T106K, n = 6, **p = 0.0.0078; T106P, n = 3, **p = 0,0013; I130T, n = 6, *p = 0,016; R477P, n = 3, ***p = 4,24 × 10−4; M478I, n = 3, ***p = 3,39 × 10−4; R482P, n = 3, **p = 5,82 × 10−4; I329T, n = 5, ***p = 1,22 × 10−5; E550L, n = 3, *p = 0,0013. Dati ristampati con il permesso di Weston et al.18. Fare clic qui per visualizzare una versione ingrandita di questa figura.
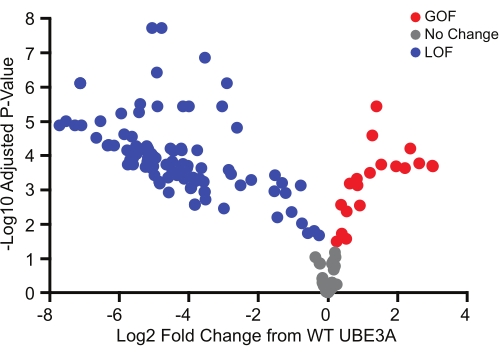
Figura 3: Varianti UBE3A che comprendono un'ampia gamma di effetti funzionali. Screening del test BAR di 152 varianti di Ube3a che mostrano mutazioni benigne (grigio), con perdita di funzione (blu) e guadagno di funzione (rosso) rispetto a WT UBE3A . La significatività è stata determinata utilizzando un t-test a un campione (a due code) con correzione dei confronti multipli di Benjamini-Hochberg (FDR = 0,05). Rosso, guadagno di funzione; Grigio, nessuna variazione rispetto a WT UBE3A; Blu, perdita di funzione. Dati ristampati con il permesso di Weston et al.18. Fare clic qui per visualizzare una versione ingrandita di questa figura.

Figura 4: Varianti UBE3A che comprendono un'ampia gamma di effetti funzionali. (A) Risultati del test BAR delle varianti alla posizione 588 di UBE3A che mostrano sia sostituzioni di perdita che di guadagno di funzione. Q588E, n = 6; Q588P, n = 3; Q588R, n = 4; p < 0,0005, test t a un campione (a due code) con correzione del confronto multiplo di Benjamini-Hochberg (FDR = 0,05). (B) Grafico termico che mostra i risultati normalizzati di BAR per un'analisi mutazionale completa alla posizione 588 in UBE3A. L'ombreggiatura bianca rappresenta i livelli di attività WT UBE3A, l'ombreggiatura blu indica la perdita di funzione e l'ombreggiatura rossa indica il guadagno di funzione. La barra della scala mostra la variazione percentuale rispetto a WT UBE3A. N = 3 esperimenti indipendenti per Q588S, Q588T, Q588N, Q588K, Q588P, Q588M, Q588G, Q588A, Q588F, Q588Y, Q588W, Q588L, Q588V, Q588I, Q588C; n = 8 per Q588E, n = 6 per Q588D, n = 5 per Q588R, n = 4 per Q588H, *p < 0,05, **p < 0,005, ***p < 0,0005. T-test a un campione (a due code) con correzione dei confronti multipli di Benjamini-Hochberg (FDR = 0,05). Dati ristampati con il permesso di Weston et al.18. Fare clic qui per visualizzare una versione ingrandita di questa figura.
| Nome | Sequenza | Utilizzato per | |
| Primer 1 | GATTATATTATGACAATAGAA TTCGCATGTACAGTGAAC | Ube3a Senso EcoRI | |
| Mut. Primer | CCAGACCCAGTACTATGCCAAT CAGAGTAAACTCACCCTCAGTT TCAAAAGAAGATGGATTAAACC | UBE3A Q588E mutagenesi antisenso | |
| Primer 2 | ATTATATTCCCGGGTTACAGCAT GCCAAATCCTTTGG | Ube3a Antisenso di XmaI | |
Tabella 1: Primer utilizzati per la mutagenesi di UBE3A Q588E.
| 1° turno PCR | |
| Reagente | Quantità di reazione |
| Buffer ad alta fedeltà 5x | 10 μL |
| dNTP (10 mM stock) | 1 μL |
| Primer 1 (10 mM di stock) | 1 μL |
| Primer 2 (10 mM stock) | 1 μL |
| WT-Ube3a DNA (150 ng/μL stock) | 1 μL |
| H2O | 35 μL |
| DNA polimerasi ad alta fedeltà | 1 μL |
| Volume totale | 50 μL |
| 2° turno PCR | |
| Reagente | Quantità di reazione |
| Buffer ad alta fedeltà 5x | 10 μL |
| dNTP (10 mM stock) | 1 μL |
| Primer 3 (10 mM stock) | 1 μL |
| Prodotto PCR purificato (Megaprimer) | 30 μL |
| WT-Ube3a DNA (150 ng/μL stock) | 1 μL |
| H2O | 6 μL |
| DNA polimerasi ad alta fedeltà | 1 μL |
| Volume totale | 50 μL |
Tabella 2: Parametri PCR per mutagenesi.
| 1° turno PCR | ||
| Passo | Temperatura | Ore |
| Denaturazione iniziale | 95 °C | 2 minuti |
| (1 ciclo) | ||
| Denaturazione | 95 °C | 30 secondi |
| Ricottura | 50 °C | 20 secondi |
| Estensione | 72 °C | 15 secondi |
| (30 cicli) | ||
| Proroga finale | 72 °C | 1 min |
| Tenere | 4 °C | Infinito |
| 2° turno PCR | ||
| Passo | Temperatura | Ore |
| Denaturazione iniziale | 95 °C | 2 minuti |
| (1 ciclo) | ||
| Denaturazione | 95 °C | 30 secondi |
| Ricottura | 50 °C | 20 secondi |
| Estensione | 72 °C | 35 secondi |
| (30 cicli) | ||
| Proroga finale | 72 °C | 1 min |
| Tenere | 4 °C | Infinito |
Tabella 3: Parametri del programma PCR per la mutagenesi.
| Reagente | Concentrazione di lavoro | 1x trasfezione | Master mix 3,5x |
| pGL3 BAR plasmide | 100 ng/μL | 0,5 μL | 1,75 μL |
| pTK Renilla plasmide | 100 ng/μL | 0,05 μL | 0,175 μL |
| Plasmide Ube3a | 100 ng/μL | 0,4 μL | 1,4 μL |
| DMEM integrato con glutammina | 8,65 μL | 30,275 μL | |
| Reagente di trasfezione | 0,4 μL | 1,4 μL |
Tabella 4: Miscele di trasfezione.
Discussione
Il protocollo qui descritto fornisce un metodo efficiente e scalabile per valutare l'attività enzimatica delle varianti di Ube3a. Ci sono diversi dettagli tecnici che meritano un'attenta considerazione quando si utilizza questo test. Una considerazione è la scelta dei plasmidi reporter Wnt utilizzati in questo test. Il protocollo qui descritto utilizza specificamente il reporter attivato da β-catenina (BAR)21, un reporter che contiene un concatenatore di 12 elementi di risposta del fattore delle cellule T (TCF) separati da sequenze di linker appositamente progettate per ridurre al minimo la ricombinazione e la perdita dei siti di legame TCF. Ci sono ulteriori plasmidi reporter Wnt basati sulla luciferasi descritti nella letteratura23, e sebbene studi precedenti indichino che alcuni possono essere utilizzati per valutare l'attività di UBE3A, il plasmide BAR è stato caratterizzato in modo più approfondito per questo scopo17,18. In secondo luogo, la scelta del plasmide Renilla luciferasi è fondamentale. Un plasmide che codifica Renilla luciferasi è incluso durante la fase di trasfezione per normalizzare l'efficienza della trasfezione. Il plasmide utilizzato in questo protocollo esprime costitutivamente Renilla luciferasi sotto il controllo di un promotore della timidina chinasi (TK) (TK-Renilla). L'uso di plasmidi contenenti altri promotori, come il promotore del citomegalovirus (CMV) ampiamente utilizzato, può portare a esiti variabili nell'espressione della Renilla luciferasi.
Una seconda considerazione è il comportamento delle cellule a seconda del tipo e del lotto di FBS utilizzato per la coltura cellulare. Ad esempio, FBS può influenzare in modo variabile il tasso di crescita delle cellule HEK293T. Il protocollo attuale è stato ottimizzato con cellule che mostrano un tempo di raddoppio tipico di 18-24 ore, rendendo la densità cellulare al momento della trasfezione di circa ~ 4 × 104 cellule per pozzetto. Poiché la densità cellulare può influenzare l'efficienza della trasfezione, i tassi di crescita cellulare devono essere attentamente valutati dall'utente e il numero di cellule regolato di conseguenza al momento della placcatura. Inoltre, la risposta BAR dipendente da UBE3A richiede la presenza di una certa quantità di ligando Wnt durante l'esperimento. Nella maggior parte delle condizioni standard, c'è abbastanza Wnt nell'FBS per guidare questa risposta; Tuttavia, questa risposta può anche variare. Si raccomanda la titolazione dei rapporti plasmidici utilizzati per la trasfezione per garantire che la risposta BAR sia misurata entro l'intervallo lineare appropriato. Un approccio alternativo consiste nello stimolare la segnalazione Wnt utilizzando un ligando Wnt ricombinante o un mezzo di crescita integrato con Wnt17.
Un vantaggio del test BAR è che può riportare l'attività dell'ubiquitina ligasi di tutte le isoforme di Ube3a . Gli esseri umani esprimono tre isoforme di Ube3a che differiscono al loro estremo N-termini a causa dell'uso di siti di inizio trascrizionali alternativi24. Il test BAR fornisce la stessa lettura agnostica alle isoforme e, quindi, questo test può essere utilizzato per valutare tutte le varianti di Ube3a . Inoltre, la caratterizzazione su larga scala delle varianti di Ube3a fornisce dati profondi sulla funzione strutturale che possono scoprire nuovi domini e meccanismi critici per la funzione enzimatica. Uno studio precedente ha utilizzato dati di variante per scoprire un dominio di legame con l'ubiquitina all'interno di UBE3A che facilita l'allungamento della catena dell'ubiquitina18. Ulteriori studi struttura-funzione forniranno probabilmente nuove informazioni sui meccanismi biochimici di UBE3A che possono essere sfruttati per lo sviluppo terapeutico.
Ci sono alcune limitazioni al test BAR che giustificano un'attenta considerazione quando si determina la patogenicità di una variante. A causa dell'imprinting epigenetico di Ube3a nei neuroni, questo test non può discriminare gli alleli materni e paterni e ulteriori prove genetiche devono essere valutate insieme ai dati funzionali per determinare la patogenicità di una variante. Inoltre, ulteriori variabili oltre all'attività ubiquitina ligasi di UBE3A devono essere considerate nel contesto della malattia. Ad esempio, lavori precedenti hanno dimostrato che UBE3A localizzato nucleare contribuisce alla patologia della sindrome di Angelman25 e che alcune varianti perturbano questo modello di localizzazione dell'enzima26. Pertanto, devono essere eseguite ulteriori caratterizzazioni a valle per ottenere una comprensione completa del contributo delle varianti di Ube3a alla patologia dello sviluppo neurologico.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato sostenuto da un Simons Foundation Bridge to Independence Award (SFARI Award #387972; J.J.Y.), un NARSAD Young Investigator Award dalla Brain and Behavior Research Foundation (J.J.Y.), una borsa di ricerca dalla Alfred P. Sloan Foundation (J.J.Y.), e borse di ricerca dalla Angelman Syndrome Foundation (J.J.Y.), dalla Whitehall Foundation (J.J.Y.) e dal NIMH (R01MH122786; J.J.Y.).
Materiali
| Name | Company | Catalog Number | Comments |
| 0.05% Trypsin-EDTA (1x), phenol red | Gibco | 25300-054 | |
| 1 Kb DNA ladder | Lambda Biotech | M108-S | |
| 100 bp DNA Ladder | Lambda Biotech | M107 | |
| 10x Buffer for T4 DNA Ligase with 10 mM ATP | New England BioLabs | B0202A | |
| 5x Phusion HF Reaction Buffer | New England BioLabs | B0518S | |
| Antibiotic-Antimycotic Solution | Corning | 30004CI | |
| Black/White Isoplate-96 Black Frame White Well plate | PerkinElmer | 6005030 | |
| Carbenicillin Disodium Salt | Midwest Scientific | KCC46000-5 | |
| Countess cell counting chamber slides | Invitrogen by Thermo Fisher Scientific | C10283 | |
| Countess II Automated Cell Counter | life technologies | Cell counting machine | |
| Custom DNA oligos | Integrated DNA Technologies (IDT) | ||
| Deoxynucleotide (dNTP) Solution Mix | New England BioLabs | N0447S | |
| DMEM, high glucose, GlutaMAX Supplement, pyruvate | Gibco | 10569044 | Basal medium for supporting the growth of HEK293T cell line |
| DPBS (1x) | Gibco | 14190-136 | |
| Dual-Luciferase Reporter Assay System | Promega | E1910 | |
| EcoRI-HF | New England BioLabs | R3101S | Restriction enzyme |
| Fetal Bovine Serum, qualified, heat inactivated | Gibco | 16140071 | Fetal bovine serum |
| Fisherbrand Surface Treated Tissue Culture Dishes | Fisherbrand | FB012924 | |
| FuGENE 6 Transfection Reagent | Promega | E2691 | |
| Gel Loading Dye Purple (6x) | New England BioLabs | B7024A | |
| HEK293T cells | ATCC | CRL-3216 | |
| High Efficiency ig 10B Chemically Competent Cells | Intact Genomics | 1011-12 | E. coli DH10B cells |
| HiSpeed Plasmid Midi Kit | Qiagen | 12643 | Midi prep |
| pCIG2 plasmid | |||
| pGL3 BAR plasmid | |||
| Phusion HF DNA Polymerase | New England BioLabs | M0530L | DNA polymerase |
| ProFlex 3 x 32 well PCR System | Applied biosystems by life technologies | Thermocycler | |
| pTK Renilla plasmid | |||
| QIAprep Spin Miniprep Kit (250) | Qiagen | 27106 | Mini prep |
| QIAquick Gel Extraction Kit (250) | Qiagen | 28706 | Gel purification |
| QIAquick PCR Purification Kit (250) | Qiagen | 28106 | PCR purification |
| rCutSmart Buffer | New England BioLabs | B6004S | |
| SacI-HF | New England BioLabs | R3156S | Restriction enzyme |
| Synergy HTX Multi-Mode Reader | BioTek | Plate reader runs Gen5 software v3.08 (BioTek) | |
| T4 DNA Ligase | New England BioLabs | M0202L | Ligase |
| TAE Buffer, Tris-Acetate-EDTA, 50x Solution, Electrophoresis | Fisher Scientific | BP13324 | |
| Tissue Culture Plate 96 wells, Flat Bottom | Fisherbrand | FB012931 | |
| UltraPure Ethidium Bromide Solution | Invitrogen by Thermo Fisher Scientific | 15585011 | |
| XmaI | New England BioLabs | R0180S | Restriction enzyme |
Riferimenti
- Landrum, M. J., et al. ClinVar: Public archive of relationships among sequence variation and human phenotype. Nucleic Acids Research. 42, 980-985 (2014).
- Lek, M., et al. Analysis of protein-coding genetic variation in 60,706 humans. Nature. 536 (7616), 285-291 (2016).
- Starita, L. M., et al. Variant interpretation: Functional assays to the rescue. American Journal of Human Genetics. 101 (3), 315-325 (2017).
- Scheffner, M., Huibregtse, J. M., Vierstra, R. D., Howley, P. M. The HPV-16 E6 and E6-AP complex functions as a ubiquitin-protein ligase in the ubiquitination of p53. Cell. 75 (3), 495-505 (1993).
- Albrecht, U., et al. Imprinted expression of the murine Angelman syndrome gene, Ube3a, in hippocampal and Purkinje neurons. Nature Genetics. 17 (1), 75-78 (1997).
- Rougeulle, C., Glatt, H., Lalande, M. The Angelman syndrome candidate gene, UBE3A/E6-AP, is imprinted in brain. Nature Genetics. 17 (1), 14-15 (1997).
- Vu, T. H., Hoffman, A. R. Imprinting of the Angelman syndrome gene, UBE3A, is restricted to brain. Nature Genetics. 17 (1), 12-13 (1997).
- Kishino, T., Lalande, M., Wagstaff, J. UBE3A/E6-AP mutations cause Angelman syndrome. Nature Genetics. 15 (1), 70-73 (1997).
- Jiang, Y. H., et al. Mutation of the Angelman ubiquitin ligase in mice causes increased cytoplasmic p53 and deficits of contextual learning and long-term potentiation. Neuron. 21 (4), 799-811 (1998).
- Mabb, A. M., Judson, M. C., Zylka, M. J., Philpot, B. D. Angelman syndrome: Insights into genomic imprinting and neurodevelopmental phenotypes. Trends in Neuroscience. 34 (6), 293-303 (2011).
- Hogart, A., Wu, D., LaSalle, J. M., Schanen, N. C. The comorbidity of autism with the genomic disorders of chromosome 15q11.2-q13. Neurobiology of Disease. 38 (2), 181-191 (2010).
- Urraca, N., et al. The interstitial duplication 15q11.2-q13 syndrome includes autism, mild facial anomalies and a characteristic EEG signature. Autism Research. 6 (4), 268-279 (2013).
- de la Torre-Ubieta, L., Won, H., Stein, J. L., Geschwind, D. H. Advancing the understanding of autism disease mechanisms through genetics. Nature Medicine. 22 (4), 345-361 (2016).
- Scheffner, M., Staub, O. HECT E3s and human disease. BMC Biochemistry. 8, Suppl 1 (2007).
- Cooper, E. M., Hudson, A. W., Amos, J., Wagstaff, J., Howley, P. M. Biochemical analysis of Angelman syndrome-associated mutations in the E3 ubiquitin ligase E6-associated protein. Journal of Biological Chemistry. 279 (39), 41208-41217 (2004).
- Yi, J. J., Barnes, A. P., Hand, R., Polleux, F., Ehlers, M. D. TGF-beta signaling specifies axons during brain development. Cell. 142 (1), 144-157 (2010).
- Yi, J. J., et al. The autism-linked UBE3A T485A mutant E3 ubiquitin ligase activates the Wnt/beta-catenin pathway by inhibiting the proteasome. Journal of Biological Chemistry. 292 (30), 12503-12515 (2017).
- Weston, K. P., et al. Identification of disease-linked hyperactivating mutations in UBE3A through large-scale functional variant analysis. Nature Communications. 12 (1), 6809(2021).
- Hand, R., Polleux, F. Neurogenin2 regulates the initial axon guidance of cortical pyramidal neurons projecting medially to the corpus callosum. Neural Development. 6, 30(2011).
- Karginov, A. V., Ding, F., Kota, P., Dokholyan, N. V., Hahn, K. M. Engineered allosteric activation of kinases in living cells. Nature Biotechnology. 28 (7), 743-747 (2010).
- Biechele, T. L., Moon, R. T. Assaying beta-catenin/TCF transcription with beta-catenin/TCF transcription-based reporter constructs. Methods in Molecular Biology. , 99-110 (2008).
- Yi, J. J., et al. An Autism-linked mutation disables phosphorylation control of UBE3A. Cell. 162 (4), 795-807 (2015).
- Kuhnle, S., et al. Angelman syndrome-associated point mutations in the Zn(2+)-binding N-terminal (AZUL) domain of UBE3A ubiquitin ligase inhibit binding to the proteasome. Journal of Biological Chemistry. 293 (47), 18387-18399 (2018).
- Yamamoto, Y., Huibregtse, J. M., Howley, P. M. The human E6-AP gene (UBE3A) encodes three potential protein isoforms generated by differential splicing. Genomics. 41 (2), 263-266 (1997).
- Avagliano Trezza, R., et al. Loss of nuclear UBE3A causes electrophysiological and behavioral deficits in mice and is associated with Angelman syndrome. Nature Neuroscience. 22 (8), 1235-1247 (2019).
- Bossuyt, S. N. V., et al. Loss of nuclear UBE3A activity is the predominant cause of Angelman syndrome in individuals carrying UBE3A missense mutations. Human Molecular Genetics. 30 (6), 430-442 (2021).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon