Method Article
Fissazione del tessuto embrionale di topo per l'analisi del citonemo
In questo articolo
Riepilogo
Qui, viene fornito un protocollo passo-passo ottimizzato per la fissazione, l'immunocolorazione e il sezionamento degli embrioni per rilevare filopodi di segnalazione specializzati chiamati citonemi nello sviluppo di tessuti di topo.
Abstract
Il pattern tissutale dello sviluppo e l'omeostasi tissutale post-sviluppo dipendono dalla consegna controllata di segnali cellulari chiamati morfogeni. I morfogeni agiscono in modo dipendente dalla concentrazione e dal tempo per specificare programmi trascrizionali distinti che istruiscono e rafforzano il destino cellulare. Un meccanismo attraverso il quale vengono assicurate adeguate soglie di segnalazione morfogeno è attraverso la consegna delle proteine di segnalazione da parte di filopodi specializzati chiamati citonemi. I citonemi sono molto sottili (≤200 nm di diametro) e possono raggiungere lunghezze di diverse centinaia di micron, il che rende difficile la loro conservazione per l'analisi di immagini fisse. Questo articolo descrive un metodo raffinato per la manipolazione delicata degli embrioni di topo per la fissazione, l'immunocolorazione e la sezione spessa per consentire la visualizzazione dei citonemi utilizzando la microscopia confocale standard. Questo protocollo è stato utilizzato con successo per visualizzare citonemi che collegano distinti compartimenti di segnalazione cellulare durante lo sviluppo del tubo neurale del topo. La tecnica può anche essere adattata per rilevare citonemi tra i tipi di tessuto per facilitare l'interrogazione della segnalazione dello sviluppo a una risoluzione senza precedenti.
Introduzione
Lo sviluppo embrionale è orchestrato attraverso l'attivazione coordinata di vie di segnalazione morfogene. I morfogeni sono piccole proteine secrete che sono classificate in Sonic Hedgehog (SHH), trasformando il fattore di crescita β (TGF-β) / proteina morfogenica ossea (BMP), sito di integrazione correlato ad Ala (WNT) e fibroblasti e fattori di crescita epidermici (FGF / EGF). I morfogeni vengono prodotti e rilasciati dai centri di organizzazione cellulare durante lo sviluppo dei tessuti e stabiliscono gradienti di segnalazione attraverso i campi organizzativi delle cellule per informare la morfogenesi tissutale 1,2,3,4,5. Una rappresentazione dei gradienti morfogeni si trova nel sistema nervoso in via di sviluppo, dove il presunto sistema nervoso centrale è modellato attraverso l'attivazione della via morfogena. Questo tessuto, indicato come tubo neurale, è composto da gradienti opposti di SHH secreti dalla notocorda ventrale e dalla piastra del pavimento, e NNT / BMP secreti dalla piastra del tetto dorsale, per modellare distinte regioni progenitrici neurali6. Il tubo neurale è comunemente usato per interrogare l'integrità del gradiente morfogeno nella ricerca sullo sviluppo.
La formazione del gradiente morfogeno si basa su una stretta regolazione della dispersione del segnale7. Un meccanismo cellulare attraverso il quale ciò si verifica è attraverso la formazione di filopodi di segnalazione lunga chiamati citonemi che facilitano la consegna diretta di morfogeni dalle cellule produttrici di segnale a specifiche popolazioni di cellule bersaglio. I citonemi sono stati osservati per estendere centinaia di micrometri per depositare morfogeni sulle membrane cellulari che ricevono il segnale 8,9. L'interruzione del trasporto di morfogeno mediato dal citonema porta ad anomalie dello sviluppo sia nelle mosche che nei vertebrati, evidenziando la loro importanza durante il pattern tissutale 10,11,12,13,14.
Ad oggi, i citonemi sono stati documentati in modelli di Drosophila, pulcino e zebrafish, ma l'imaging delle strutture nello sviluppo di embrioni di mammiferi rimane impegnativo 8,9,15. Un ostacolo all'imaging efficace dei citonemi nei tessuti complessi dei mammiferi in situ è la loro natura sottile e fragile, che li rende suscettibili ai danni con i metodi di fissazione convenzionali8. In precedenza avevamo sviluppato e ottimizzato protocolli per un fissativo al microscopio elettronico modificato (MEM-fix) per preservare i citonemi nelle cellule coltivate e consentire il loro studio utilizzando la microscopia confocale16,17.
L'uso della tecnica MEM-fix ha permesso l'identificazione di alcune delle molecole coinvolte nella formazione e nella funzione 11,16,17 del citonemo indotto da SHH. Tuttavia, la conferma di questi risultati nel contesto fisiologicamente rilevante del pattern del tubo neurale ha richiesto lo sviluppo di nuove tecniche per fissare e visualizzare il tessuto embrionale del topo. Un protocollo per fissare gli embrioni di topo in modo da mantenere l'integrità del citonemo e consentire l'immunocolorazione e il successivo sezionamento del tessuto embrionale per l'analisi confocale è delineato qui. Questo protocollo è stato sviluppato utilizzando una proteina fluorescente verde legata alla membrana (GFP) per etichettare le estensioni di membrana dalle cellule produttrici di SHH nel tubo neurale in via di sviluppo. L'implementazione di questo protocollo affronterà le domande senza risposta relative alla prevalenza e al significato dei citonemi nello sviluppo di sistemi di mammiferi.
Protocollo
Questo protocollo segue le linee guida approvate per la cura degli animali dell'Institutional Animal Care and Use Committee (IACUC) e del St. Jude Children's Research Hospital. Tutti i ceppi sono stati incrociati di cinque generazioni con il ceppo C57BL/6J.
1. Isolamento embrionale e colorazione dell'intero monte
- Alleva femmine di 6 settimane e monitora la presenza di tappo vaginale.
- Eutanasia della diga gravida mediante inalazione di CO2 in una camera di CO2 seguita da lussazione cervicale secondo le linee guida AVMA18. Fai un'incisione a Y nella cavità peritoneale usando forbici e pinze di dissezione. Asportare l'utero contenente gli embrioni E9.5 secondo le linee guida istituzionali approvate.
- Sezionare gli embrioni in mezzo di crescita completo (Dulbecco's Modified Eagle Medium, integrato con aminoacidi non essenziali, Na-piruvato, L-glutammina e siero bovino fetale al 10%). Utilizzare una pinza per rimuovere il sacco vitellino, la placenta e le membrane circostanti.
NOTA: Quando necessario, salvare ogni sacco vitellino per la genotipizzazione dell'embrione. - Risciacquare gli embrioni isolati nella soluzione salina bilanciata di Hank (HBSS) per rimuovere qualsiasi residuo di tessuto amniotico e sangue.
- Preparare il fissativo. Aggiungere paraformaldeide (PFA) a HBSS per una concentrazione finale di lavoro del 4% di PFA.
ATTENZIONE: il PFA è una sostanza chimica tossica, quindi l'inalazione o l'esposizione diretta della pelle devono essere evitate. La preparazione della soluzione fissativa viene eseguita sotto una cappa aspirante con l'adeguato dispositivo di protezione individuale di guanti e un cappotto da laboratorio. - Aggiungere 1 mL di fissativo a ciascun pozzetto di una piastra a 24 pozzetti e posizionare ogni embrione in un pozzetto individuale. Incubare gli embrioni in fissativo per 45 minuti con una delicata agitazione su un bilanciere.
NOTA: Critico: Tutti i lavaggi e le incubazioni devono essere eseguiti con agitazione delicata (massimo 20 RPM) su un bilanciere o uno shaker circolare, poiché la manipolazione o il movimento brusco degli embrioni distruggerà i citonemi fissi. Utilizzare una pipetta per rimuovere delicatamente tutte le soluzioni. - Rimuovere il fissativo e lavare gli embrioni 3 x 30 min in Soluzione Salina Tamponata con Fosfato (PBS) con Ca2+ e Mg2+ con aggiunta dello 0,1% di Tritone.
- Dopo il lavaggio, incubare gli embrioni in soluzione bloccante (PBS con Ca2+ e Mg2+, 0,1% Tritone e 5% siero di capra) con delicata agitazione. Blocco 2 x 1 h. Dopo la seconda incubazione bloccante, eseguire un rapido risciacquo degli embrioni utilizzando una soluzione bloccante fresca.
- Durante la seconda fase di blocco, preparare la soluzione anticorpale primaria11. Diluire gli anticorpi alla concentrazione ottimizzata in PBS con Ca2+ e Mg2+, 0,1% Tween-20 e 5% siero di capra.
NOTA: per una migliore visualizzazione della GFP a membrana, è possibile utilizzare l'anti-GFP di pollo (1:250). - Rimuovere la soluzione bloccante e aggiungere 1 mL di soluzione anticorpale primaria a ciascun pozzetto. Incubare a 4 °C con rotazione delicata per 3 giorni.
- Dopo l'incubazione dell'anticorpo primario, lavare gli embrioni 5 x 1 h a 20 RPM su un bilanciere in PBS con Ca2+ e Mg2+, 0,1% Tween-20 e siero di capra al 5%.
- Preparare la soluzione anticorpale secondaria utilizzando frammenti secondari F(ab')2 a una diluizione 1:1.000 in PBS con Ca2+ e Mg2+, 0,1% Tween-20 e siero di capra al 5%.
NOTA: L'uso di frammenti F(ab')2 migliora notevolmente la penetrazione degli anticorpi nel campione. - Aggiungere 1 mL di soluzione anticorpale secondaria a ciascun pozzetto. Incubare con dolce dondolo a 4 °C al buio per 3 giorni.
NOTA: Importante: da questo momento in poi, ridurre al minimo l'esposizione dell'embrione alla luce diretta. Facoltativo: per prevenire la crescita batterica, aggiungere lo 0,2% di azide di sodio alla soluzione anticorpale secondaria. - Rimuovere la soluzione anticorpale secondaria e lavare gli embrioni 3 x 30 minuti in PBS con Ca2+ e Mg2+, 0,1% Tween-20 e siero di capra al 5%. Se non si esegue la colorazione dell'actina con falloidina o altri coloranti a base di actina, dopo il primo lavaggio, aggiungere 4′,6-diamidino-2-fenilindolo (DAPI) a PBS con Ca2+ e Mg2+, 0,1% Tween-20 e 5% siero di capra e incubare per 1 ora, seguito da 3 lavaggi di 3 minuti come descritto sopra.
NOTA: A questo punto, gli embrioni possono essere conservati durante la notte a 4 °C in PBS o HBSS al buio fino alla fase di incorporamento il giorno successivo. Tuttavia, si raccomanda che gli embrioni siano incorporati e sezionati immediatamente.
2. Incorporamento, sezionamento e montaggio di embrioni
- Preparare una soluzione di agarosio a basso punto di fusione (LMP) al 4% p/v disciolta in HBSS o PBS con Ca2+ e Mg2+ fino ad un volume finale di ~3 mL per embrione. Aggiungere il peso appropriato di agarosio LMP a HBSS o PBS con Ca2+ e Mg2+ e microonde fino a quando l'agarosio LMP non è sciolto.
NOTA: Una volta sciolto l'agarosio LMP, conservare la soluzione in un bagno di perline, incubatore o bagno d'acqua impostato a 55 °C per evitare che la soluzione di agarosio LMP si solidifichi. - Usa una piastra a 12 pozzetti come stampo di incorporamento per gli embrioni. Posizionare la piastra a 12 pozzetti nel bagno di perline a 55 °C. Aggiungere 2,5-3 ml di agarosio LMP al 4% a ciascun pozzetto che conterrà un embrione.
NOTA: Sebbene possano essere utilizzati altri stampi, i volumi di piastre a 12 pozzetti sono ottimali per preparare il blocco di agarosio nelle fasi successive per il sezionamento. - Utilizzando un cucchiaio perforato, trasferire gli embrioni in singoli pozzetti contenenti una soluzione di agarosio LMP al 4% (Figura 1A).
- Trasferire la piastra a 12 pozzetti dal bagno di perline a un banco. Utilizzare le punte delle pipette per incorporare e orientare delicatamente l'embrione in modo che sia centrato all'interno della soluzione. Una volta orientati gli embrioni, posizionare la piastra a -20 °C per 10 minuti per consentire una rapida solidificazione del blocco.
CRITICO: Assicurarsi che l'embrione sia posizionato al centro del blocco. Gli embrioni che affondano sul fondo o sono troppo vicini al bordo dello stampo saranno probabilmente rimossi dal blocco di agarosio durante il sezionamento.
- Trasferire la piastra a 12 pozzetti dal bagno di perline a un banco. Utilizzare le punte delle pipette per incorporare e orientare delicatamente l'embrione in modo che sia centrato all'interno della soluzione. Una volta orientati gli embrioni, posizionare la piastra a -20 °C per 10 minuti per consentire una rapida solidificazione del blocco.
- Rimuovere l'intero blocco di agarosio dal pozzo usando un bisturi e tagliare un blocco rettangolare attorno all'embrione lasciando ~ 0,3 cm di blocco su ciascun lato. Lasciare una lunghezza extra lungo l'estremità caudale dell'embrione. Se montato sul vibratoma, orientare l'embrione in posizione verticale nella sezione superiore del blocco (Figura 1B).
- Applicare una striscia di nastro adesivo al portacampione del vibratoma e sovracollare il blocco di agarosio al nastro, orientato in modo che la lama generi sezioni assiali dell'embrione in una sequenza anteriore (cranica) a posteriore.
- Riempire la camera del vibratome con HBSS freddo per assicurarsi che il campione sia completamente immerso, quindi circondare la camera con ghiaccio.
- Impostare la velocità del vibrato su 0,2 mm/s e la frequenza tra 5 e 7 (50-70 Hz) con spessore di taglio impostato su 100 μm. Eseguire il sezionamento assiale seriale dell'embrione.
NOTA: utilizzare una velocità lenta per il sezionamento. Se la velocità di taglio viene aumentata al di sopra di 0,25 mm/s, il sezionamento può strappare l'embrione o rimuovere l'embrione dal blocco.- Durante la serie di sezionamento, utilizzare una pinza per trasferire delicatamente le singole sezioni su un piatto separato da 60 mm riempito con HBSS. Utilizzare una pinza per afferrare il blocco, non il tessuto, per evitare danni ai tessuti e la distruzione dei citonemi.
NOTA: le sezioni di tessuto devono rimanere all'interno del blocco di agarosio. Se il tessuto cade dal blocco nella camera del vibrato, trasferire delicatamente sollevando la sezione. Non afferrare o pizzicare il tessuto. CRITICO: Qualsiasi ripiegamento di sezioni di tessuto o manipolazione improvvisa distruggerà i citonemi fissi nelle sezioni tissutali.
- Durante la serie di sezionamento, utilizzare una pinza per trasferire delicatamente le singole sezioni su un piatto separato da 60 mm riempito con HBSS. Utilizzare una pinza per afferrare il blocco, non il tessuto, per evitare danni ai tessuti e la distruzione dei citonemi.
- Se non si esegue la colorazione F-actina, procedere al passaggio 2.10.
- Per eseguire la colorazione F-actina, rimuovere HBSS e incubare sezioni per 40 minuti con soluzione di Actin-Red e DAPI diluita in PBS con Ca2+ e Mg2+, 0,1% Tween-20 e siero di capra al 5% a temperatura ambiente.
- Sezioni di lavaggio 3 x 20 min in PBS con Ca2+ e Mg2+ e 0,1% Tween-20.
- Utilizzando un pennarello idrofobo, disegnare una barriera idrofobica attorno ai bordi di un vetrino per microscopio carico e aggiungere un piccolo volume di HBSS per riempire l'area.
- Utilizzare una pinza o un cucchiaio perforato per trasferire le sezioni alla diapositiva.
NOTA: Tutte le sezioni di tessuto che non sono racchiuse in agarosio possono essere trasferite tramite una pipetta di trasferimento. - Rimuovere il blocco di agarosio in eccesso usando una pinza.
- Una volta che tutte le sezioni sono state trasferite sul vetrino, rimuovere il liquido in eccesso tramite pipettaggio e l'angolo di un towelette assorbente, quindi aggiungere diverse gocce di mezzo di montaggio al vetrino. Utilizzare un supporto di montaggio sufficiente per coprire l'intera area di copertura quando è indurito. Montare il coperchio posizionandolo delicatamente sulla diapositiva.
NOTA: Evitare di applicare pressione o disturbare il coverslip fino a quando il mezzo di montaggio non si è indurito.
3. Imaging
- Eseguire l'imaging di sezioni di tessuto su qualsiasi microscopio confocale o ad alta risoluzione11. Analizzare un minimo di tre embrioni per genotipo.
NOTA: Le immagini delle sezioni di tessuto sono state acquisite utilizzando un microscopio confocale TCS SP8 STED 3x, seguito dalla deconvoluzione LIGHTNING.
Risultati
L'uso di uno stampo più grande di una piastra a 12 pozzetti con 2,5-3 ml di soluzione di agarosio per pozzetto era ideale per incorporare e sospendere diversi embrioni in un breve periodo di tempo (Figura 1A). L'area in eccesso consente un orientamento corretto durante il taglio del blocco di agarosio per il sezionamento. Quando si taglia il blocco di agarosio, è importante mantenere l'agarosio in eccesso lungo il fondo del blocco dove sarà incollato al nastro sul supporto dell'oggetto. L'embrione dovrebbe trovarsi nella metà superiore del blocco (Figura 1B). Tuttavia, la sezione inferiore non deve essere troppo grande poiché il blocco in eccesso aumenta le possibilità di alterare l'angolo di taglio mentre la lama spinge nel blocco durante la sezionamento. Vengono mostrati esempi di sezioni orientate correttamente (Figura 1C,D).
Durante lo sviluppo di questo protocollo, il sezionamento del vibratoma è stato confrontato con il sezionamento del criostato. Sezionamento criostato delle estensioni cellulari raramente conservate (Figura 2 criostato e Figura 3A,B vibratoma). Le sezioni di criostato hanno permesso il rilevamento di alcuni frammenti di membrana GFP-positivi tra cellule della notocorda e del tubo neurale (Figura 2A punta di freccia) e tra cellule del tubo neurale adiacenti (Figura 2B, punte di freccia B). Tuttavia, la colorazione F-actina delle estensioni cellulari nelle cellule mesenchimali altamente filamentose che circondano il tubo neurale è stata compromessa nelle sezioni criostatali (Figura 2C, freccia C, vs Figura 3C, D). Questi risultati criostati indicano che solo alcune tracce di estensioni cellulari rotte rimangono seguendo questo metodo. Pertanto, il sezionamento del vibratomo è preferito per preservare in modo efficiente queste delicate estensioni per l'analisi successiva.
L'interruzione minima dell'intero embrione e delle singole sezioni di tessuto è essenziale per l'imaging di alta qualità delle estensioni cellulari. Le sezioni di tessuto che hanno subito qualsiasi piegatura o deformazione saranno evidenti dall'assenza della notocorda o da una grande separazione (>30 μm) tra la notocorda e la piastra del pavimento ventrale del tubo neurale (Figura 3B). Questo di solito è accompagnato dalla perdita di cellule mesenchimali che normalmente circondano il tubo neurale (Figura 3A, freccia). Le sezioni danneggiate comporteranno anche una perdita di estensioni visibili della membrana cellulare che migrano tra le cellule epiteliali (Figura 3B rispetto alla Figura 3A, punte di freccia). Anche piccole distorsioni alle sezioni possono causare la frammentazione delle estensioni a base di actina e la formazione di grandi spazi tra le celle (Figura 3D, punte di freccia, rispetto alla Figura 3C ben conservata), sottolineando la necessità di una manipolazione delicata in tutti i passaggi.
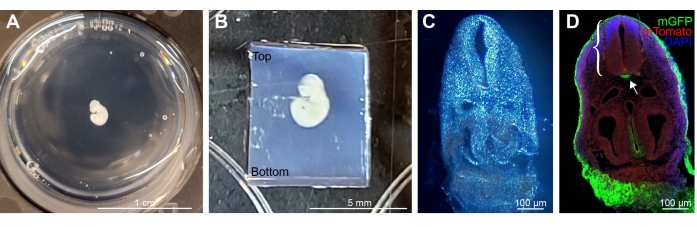
Figura 1: Esempio di E9.5 Shh-Cre; Rosa26 mTmG embrione macchiato, incorporato e sezionato. (A) Un singolo embrione in una piastra a 12 pozzetti, incorporato in agarosio LMP al 4%. (B) Esempio di embrione correttamente orientato all'interno del blocco di agarosio ridotto a misura per il montaggio del vibratoma. Il blocco di agarosio in eccesso è presente lungo il fondo. (C) Immagine in campo luminoso di una sezione embrionale spessa 100 μm incorporata nell'agarosio LMP. (D) Imaging a immunofluorescenza di un paragrafo dopo la rimozione dell'agarosio. L'mGFP anti-GFP (verde) rappresenta le cellule che esprimono Shh nel tessuto, con altre linee cellulari che esprimono la membrana Tomato (rosso), DAPI in blu. Il tubo neurale (staffa) e la notocorda mGFP-positiva (freccia) sono chiaramente visibili. Barre di scala = 1 cm (A), 5 mm (B) e 100 μm (C,D). Abbreviazioni: LMP = basso punto di fusione; mGFP = proteina fluorescente verde membrana; Shh = Riccio sonico; DAPI = 4',6-diamidino-2-fenilindolo. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 2: Esempio di piastra del pavimento del tubo neurale a sezione criostata e notocorda con estensioni di membrana frammentate. (A) 20 μm di spessore E9.5 Shh-Cre; Rosa26 mTmG 19,20,21 sezione colorata per GFP (verde), F-actina (rosso) e DAPI (blu). I puncta mGFP sono visibili tra la notocorda e la piastra del pavimento (punta di freccia) e (B,B') che migrano tra le cellule adiacenti del tubo neurale. (C,C') La colorazione F-actina non riesce a rilevare alcun citonemo chiaro sulle cellule mesenchimali che circondano il tubo neurale (freccia). Barre di scala = 20 μm. Abbreviazioni: mGFP = proteina fluorescente verde membrana; Shh = Riccio sonico; DAPI = 4',6-diamidino-2-fenilindolo. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 3: Esempi di sezioni di tessuto vibratome ottimali e subottimali e colorazione della piastra del pavimento del tubo neurale, della notocorda e delle cellule circostanti. Esempi di sezioni delicatamente maneggiate (A, C), rispetto a (B) una sezione di tessuto piegato e (D) una sezione mal gestita da un Shh-Cre; Rosa26 mTmG e un embrione ShhGFP/+ 19,20,21. Le sezioni gestite in modo ottimale consentono il rilevamento di citonemi tra cellule epiteliali neurali della piastra del pavimento localizzate adiacente (A, punte di freccia). Il tubo neurale adiacente notocorda (A, mGFP-expressing) e le cellule mesenchimali (A, freccia) dovrebbero essere visibili. (C,D) Le sezioni colorate di F-actina e DAPI dovrebbero avere una spaziatura coerente delle cellule mesenchimali e dei citonemi a base di F-actina (frecce) che circondano il tubo neurale e la notocorda (C). Qualsiasi piccola piegatura o interruzione delle sezioni può causare lacune e frammenti di F-actina rotti (D, punte di freccia). Barre della scala = 10 μm. Abbreviazioni: mGFP = proteina fluorescente verde membrana; Shh = Riccio sonico; DAPI = 4',6-diamidino-2-fenilindolo. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
Questo protocollo è stato sviluppato per la conservazione di citonemi delicati in sezioni di tessuto embrionale per la microscopia a fluorescenza ad alta risoluzione. Ad oggi, la visualizzazione dei citonemi nei tessuti attraverso vari organismi modello si è basata in gran parte sull'espressione ectopica di proteine marcate fluorescentemente utilizzando l'imaging di tessuti vivi. Sfortunatamente, l'uso di live-imaging marcato fluorescentemente è raramente favorevole all'analisi di proteine espresse endogenamente, può introdurre vincoli di tempo e richiede fasi di imaging e microscopi specializzati. Il metodo di colorazione e sezionamento qui descritto preserva i citonemi e altre estensioni cellulari per consentire l'immunoistochimica per l'eventuale rilevazione di proteine espresse endogenamente. Questa tecnologia faciliterà nuove intuizioni su come i morfogeni vengono trasportati attraverso diversi tessuti in vivo e amplia la portata dei sistemi modello in cui i citonemi possono essere studiati.
Questo protocollo utilizza la colorazione a tutto monte e la sezione spessa del vibratoma, che evita l'uso di soluzioni di equilibrio come il glicerolo o soluzioni crioprotettive come il saccarosio. Ha lo scopo di ridurre al minimo la manipolazione dei tessuti sezionati per consentire la massima conservazione delle estensioni cellulari. La ridotta manipolazione del tessuto dopo il sezionamento ha fornito risultati costantemente migliori nella conservazione complessiva dei citonemi. Pertanto, il sezionamento dell'embrione è uno dei passaggi finali prima dell'imaging.
MEM-fix, una tecnica di fissazione sviluppata per la conservazione dei citonemi delle cellule in coltura, consiste in una combinazione di glutaraldeide e PFA che consente una migliore conservazione 3D dell'architettura cellulare innata paragonabile alle loro dimensioni vive16,17. Sfortunatamente, MEM-fix non è stato utile per la fissazione dell'embrione perché la glutaraldeide può auto-fluorescersi e limita gravemente la penetrazione degli anticorpi e il legame dell'epitopo nelle cellule a causa dell'elevata attività di reticolazione proteica22,23. Pertanto, i protocolli di sezionamento post le condizioni di fissazione del PFA sono stati ottimizzati per consentire la conservazione delle estensioni cellulari nel tessuto embrionale. Sebbene il PFA possa preservare le estensioni citonemiche nel tessuto embrionale, l'analisi delle immagini deve essere eseguita con cautela. Il PFA può causare parziale disidratazione di singole cellule e tessuti, portando a una minore riduzione del volume e alla formazione di piccoli spazi extracellulari all'interno del tessuto fisso.
Questo protocollo evita le fasi extra di ricaturazione del saccarosio per la crioprotezione e la pulizia dei tessuti perché le soluzioni di saccarosio o glicerolo possono causare una piccola ricatturazione del tessuto24. Il conseguente gonfiore minore può distruggere le estensioni cellulari fisse e i citonemi che migrano tra le cellule e attraverso i tessuti. Ciò è stato evidente quando si confronta il vibratoma a sezione spessa con le sezioni criostatali. Sebbene l'aumento dello spessore dei tessuti abbia migliorato la conservazione dei citonemi intatti, le sezioni di criostato reseturate di saccarosio hanno costantemente avuto una minore incidenza di citonemi rilevabili.
L'ottimizzazione di questo protocollo ha rivelato che le sezioni spesse 100 μm erano le migliori per garantire la conservazione dei citonemi intatti. Le sezioni più sottili sono tipicamente utilizzate per l'imaging perché la maggior parte dei microscopi confocali sono in grado di eseguire l'imaging solo a una risoluzione sufficiente per visualizzare le estensioni cellulari a una profondità di ~ 20-40 μm senza cancellare25. Tuttavia, le forze meccaniche e la distorsione applicate alle cellule embrionali dalla lama durante il taglio di sezioni sottili distruggono i citonemi. Le sezioni più spesse consentono una maggiore area di profondità all'interno della sezione preservando le estensioni intatte all'interno del tessuto. Inoltre, l'uso di sezioni più spesse consente una facile manipolazione per evitare un eccessivo ripiegamento o instabilità delle sezioni di tessuto.
Questo protocollo è stato ottimizzato per la massima conservazione dei citonemi nei tessuti di embrioni di topo E8-10,5. La manipolazione minima del tessuto dopo il sezionamento ha fornito una conservazione complessiva costantemente migliorata dei citonemi. È probabile che sarà necessaria un'ulteriore ottimizzazione per adattare la tecnica per le fasi di sviluppo successive e le sezioni di tessuto adulto a causa delle maggiori dimensioni e complessità dei tessuti. Ciò richiederà il sezionamento seguito da colorazione immunofluorescenza. Tale tessuto può richiedere ulteriori passaggi di compensazione e adattamento della manipolazione dei tessuti durante il sezionamento per preservare le estensioni simili a citonemi. Questi passaggi aggiuntivi dovranno essere considerati e affrontati per le future applicazioni di questo protocollo.
Divulgazioni
Gli autori non hanno interessi concorrenti da dichiarare.
Riconoscimenti
Le immagini sono state acquisite utilizzando microscopi gestiti dal Cell and Molecular Biology Imaging Core presso il St. Jude Children's Research Hospital. I ceppi di topo sono stati ottenuti da JAX. Questo lavoro è stato sostenuto dalla sovvenzione del National Institutes of Health R35GM122546 (SKO) e dall'ALSAC del St. Jude Children's Research Hospital.
Materiali
| Name | Company | Catalog Number | Comments |
| 12-well plate; Nunc Cell-Culture Treated | Thermo Scientific | 150628 (Fisher Scientific 12-565-321) | |
| 24-Well Plate, Round Nunc | Thermo Fisher Scientific | 142475 | |
| 60 mm dish | Corning, Falcon | 353004 (Fisher Scientific 08772F) | |
| Absorbant towels (Professional Kimtech Science Kimwipes) | Kimberly-Clark KIMTECH | 34155 (Fisher Scientific 06666A) | |
| Actin Red 555 ReadyProbes Reagent | Invitrogen | R37112 | |
| Alexa Fluor 488 AffiniPure F(ab')2 Fragment Donkey Anti-Chicken IgY (IgG) (H+L) | Jackson Immunoresearch Lab Inc | 703-546-155 | 1:1,000 working concentration |
| Bead Bath 6L 230V | Lab Armor | Item #12L048 (Mfr. Model #74220-706) | set to 55 °C |
| chicken anti-GFP | Aves Labs | SKU GFP-1020 | 1:250 working concentration |
| CO2 chamber | plexiglass chamber connected to a CO2 emitting line. | ||
| Confocal laser-scanning microscope | Leica Microsystems | model: Leica TCS SP8 | |
| DAPI Solution (1 mg/mL) | Thermo Fisher Scientific | 62248 | |
| Dissecting scissors (Vannas Scissors) | World Precision Instruments | 14003 | |
| Dulbecco's Modified Eagle Medium, high glucose, no glutamine | Thermo Fisher Scientific, Gibco | 11960044 | |
| Eppendorf Research Plus single channel pipette, 0.1-2.5 µL | Eppendorf | 3123000012 | |
| Eppendorf Research Plus single channel pipette, 0.5-10 µL | Eppendorf | 3123000020 | |
| Eppendorf Research Plus single channel pipette, 100-1,000 µL | Eppendorf | 3123000063 | |
| Eppendorf Research Plus single channel pipette, 20-200 µL | Eppendorf | 3123000055 | |
| Feather Disposable Scalpel #10 | Fisher Scientific | Catalog No.NC9999403 | |
| Fetal Bovine Serum, certified, United States | Thermo Fisher Scientific, Gibco | 16000044 | |
| Fisherbrand Fine Point High Precision Forceps | Fisher Scientific | 22-327379 | |
| Fisherbrand Labeling Tape | Fisher Scientific | 15-954 | |
| Formaldehyde, 16%, methanol free, Ultra Pure EM Grade | Polysciences | 18814-10 | |
| Hank's Balanced Salt Solution (HBSS) | Thermo Fisher Scientific, Gibco | 14025 | |
| ImmEdge Hydrophobic Barrier Pap Pen | Vector laboratories | H-4000 | |
| Leica Application Suite X (LAS X) with LIGHTNING | Leica Microsystems | Microscope software | |
| L-Glutamine (200 mM) | Thermo Fisher Scientific, Gibco | 25030081 | |
| Low Melting Point Agarose- UltraPure | Invitrogen | 16520 | |
| MEM Non-Essential Amino Acids Solution (100x) | Thermo Fisher Scientific, Gibco | 11140050 | |
| Moria MC 17 BIS Perforated Spoon | Fine Science Tools | 1037018 | |
| Mounting media (ProLong Diamond Antifade Mountant) | Thermo Fisher Scientific, Invitrogen | P36961 | |
| Mouse: B6.129X1(Cg)-Shhtm6Amc/J (ShhGFP/+) | The Jackson Laboratory | Strain #:008466 | |
| Mouse: B6.Cg-Shhtm1(EGFP/cre)Cjt/J (Shh-Cre) | The Jackson Laboratory | Strain #:005622 | |
| Mouse: C57BL/6J (B6) | The Jackson Laboratory | Strain #:000664 | |
| Mouse: Gt(ROSA)26Sortm4(ACTB-tdTomato,-EGFP)Luo/J (Rosa26 mTmG) | The Jackson Laboratory | Strain #:007576 | |
| Normal Goat Serum | Jackson ImmunoResearch | 005-000-121 (Fisher Scientific NC9660079) | |
| PBS + Ca2+ + Mg2+ | Thermo Fisher Scientific, Gibco | 14040133 | |
| Premium Microcentrifuge Tubes: 1.5 mL | Fisher Scientific | 05-408-129 | |
| SHARP Precision Barrier Tips, Extra Long for P-10 and Eppendorf 10 µL | Denville Scientific | P1096-FR | |
| SHARP Precision Barrier Tips, For P-1000 and Eppendorf 1,000, 1,250 µL | Denville Scientific | P1126 | |
| SHARP Precision Barrier Tips, For P-20 and Eppendorf 20 µL | Denville Scientific | P1121 | |
| SHARP Precision Barrier Tips, For P-200 and Eppendorf 200 µL | Denville Scientific | P1122 | |
| Sodium Azide | Sigma-Aldrich | S8032 | |
| Sodium Pyruvate (100 mM) (100x) | Thermo Fisher Scientific, Gibco | 11360070 | |
| Super Glue | Gorilla | ||
| SuperFrost Plus microscope slides | Fisher Scientific | 12-550-15 | |
| TPP centrifuge tubes, volume 15 mL, polypropylene | TPP Techno Plastic Products | 91015 | |
| TPP centrifuge tubes, volume 50 mL, polypropylene | TPP Techno Plastic Products | 91050 | |
| Transfer pipette, polyethylene | Millipore Sigma | Z135003 | |
| Triton X-100 | Sigma-Aldrich | T9284 | |
| Tween-20 | Sigma-Aldrich | P1379 | |
| Vari-Mix Platform Rocker | Thermo Fisher Scientific | 09-047-113Q | set to 20 RPM |
| Vibratome | Leica Biosytems | VT1000 S |
Riferimenti
- Crick, F. Diffusion in embryogenesis. Nature. 225 (5231), 420-422 (1970).
- Zeng, X., et al. A freely diffusible form of Sonic hedgehog mediates long-range signalling. Nature. 411 (6838), 716-720 (2001).
- Yu, S. R., et al. Fgf8 morphogen gradient forms by a source-sink mechanism with freely diffusing molecules. Nature. 461 (7263), 533-536 (2009).
- Zhou, S., et al. Free extracellular diffusion creates the Dpp morphogen gradient of the Drosophila wing disc. Current Biology. 22 (8), 668-675 (2012).
- Ribes, V., Briscoe, J. Establishing and interpreting graded Sonic Hedgehog signaling during vertebrate neural tube patterning: the role of negative feedback. Cold Spring Harbor Perspectives in Biology. 1 (2), 002014 (2009).
- Le Dréau, G., Martí, E. Dorsal-ventral patterning of the neural tube: a tale of three signals. Developmental Neurobiology. 72 (12), 1471-1481 (2012).
- Kornberg, T. B., Guha, A. Understanding morphogen gradients: a problem of dispersion and containment. Current Opinion in Genetics & Development. 17 (4), 264-271 (2007).
- Ramirez-Weber, F. A., Kornberg, T. B. Cytonemes: cellular processes that project to the principal signaling center in Drosophila imaginal discs. Cell. 97 (5), 599-607 (1999).
- Sanders, T. A., Llagostera, E., Barna, M. Specialized filopodia direct long-range transport of SHH during vertebrate tissue patterning. Nature. 497 (7451), 628-632 (2013).
- Bischoff, M., et al. Cytonemes are required for the establishment of a normal Hedgehog morphogen gradient in Drosophila epithelia. Nature Cell Biology. 15 (11), 1269-1281 (2013).
- Hall, E. T., et al. Cytoneme delivery of sonic hedgehog from ligand-producing cells requires Myosin 10 and a dispatched-BOC/CDON co-receptor complex. eLife. 10, 61432 (2021).
- Huang, H., Kornberg, T. B. Cells must express components of the planar cell polarity system and extracellular matrix to support cytonemes. eLife. 5, 18979 (2016).
- Brunt, L., et al. Vangl2 promotes the formation of long cytonemes to enable distant Wnt/β-catenin signaling. Nature Communications. 12 (1), 2058 (2021).
- Roy, S., Huang, H., Liu, S., Kornberg, T. B. Cytoneme-mediated contact-dependent transport of the Drosophila decapentaplegic signaling protein. Science. 343 (6173), 1244624 (2014).
- Mattes, B., et al. Wnt/PCP controls spreading of Wnt/β-catenin signals by cytonemes in vertebrates. eLife. 7, 36953 (2018).
- Bodeen, W. J., Marada, S., Truong, A., Ogden, S. K. A fixation method to preserve cultured cell cytonemes facilitates mechanistic interrogation of morphogen transport. Development. 144 (19), 3612-3624 (2017).
- Hall, E. T., Ogden, S. K. Preserve cultured cell cytonemes through a modified electron microscopy fixation. Bio-protocol. 8 (13), (2018).
- American Veterinary Medical Association. AVMA guidelines for the euthanasia of animals. American Veterinary Medical Association. , (2020).
- Harfe, B. D., et al. Evidence for an expansion-based temporal Shh gradient in specifying vertebrate digit identities. Cell. 118 (4), 517-528 (2004).
- Chamberlain, C. E., Jeong, J., Guo, C., Allen, B. L., McMahon, A. P. Notochord-derived Shh concentrates in close association with the apically positioned basal body in neural target cells and forms a dynamic gradient during neural patterning. Development. 135 (6), 1097-1106 (2008).
- Muzumdar, M. D., Tasic, B., Miyamichi, K., Li, L., Luo, L. A global double-fluorescent Cre reporter mouse. Genesis. 45 (9), 593-605 (2007).
- Bacallao, R., Sohrab, S., Phillips, C., Pawley, J. B. . Handbook Of Biological Confocal Microscopy. , 368-380 (2006).
- Tagliaferro, P., Tandler, C. J., Ramos, A. J., Pecci Saavedra, J., Brusco, A. Immunofluorescence and glutaraldehyde fixation. A new procedure based on the Schiff-quenching method. Journal of Neuroscience Methods. 77 (2), 191-197 (1997).
- Neckel, P. H., Mattheus, U., Hirt, B., Just, L., Mack, A. F. Large-scale tissue clearing (PACT): Technical evaluation and new perspectives in immunofluorescence, histology, and ultrastructure. Scientific Reports. 6 (1), 1-13 (2016).
- Clendenon, S. G., Young, P. A., Ferkowicz, M., Phillips, C., Dunn, K. W. Deep tissue fluorescent imaging in scattering specimens using confocal microscopy. Microscopy and Microanalysis. 17 (4), 614-617 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon