Method Article
Isolamento dei nuclei dal rene di topo adulto per il sequenziamento dell'RNA a singolo nucleo
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Qui, presentiamo un protocollo per isolare nuclei di alta qualità da reni di topo congelati che migliorano la rappresentazione dei tipi di cellule renali midollari ed evitano gli artefatti di espressione genica dalla dissociazione enzimatica del tessuto.
Abstract
I reni regolano diversi processi biologici come l'acqua, l'elettrolita e l'omeostasi acido-base. Le funzioni fisiologiche del rene sono eseguite da più tipi di cellule disposte in un'architettura complessa attraverso l'asse corticomemidollare dell'organo. I recenti progressi nella trascrittomica monocellulare hanno accelerato la comprensione dell'espressione genica specifica del tipo cellulare nella fisiologia e nella malattia renale. Tuttavia, i protocolli di dissociazione tissutale basati su enzimi, che sono frequentemente utilizzati per il sequenziamento dell'RNA a singola cellula (scRNA-seq), richiedono per lo più tessuto fresco (non archiviato), introducono risposte allo stress trascrizionale e favoriscono la selezione di abbondanti tipi di cellule della corteccia renale con conseguente sottorappresentazione delle cellule del midollo.
Qui, presentiamo un protocollo che evita questi problemi. Il protocollo si basa sull'isolamento dei nuclei a 4 °C dal tessuto renale congelato. I nuclei sono isolati da un pezzo centrale del rene di topo composto dalla corteccia, dal midollo esterno e dal midollo interno. Ciò riduce la sovrarappresentazione delle cellule corticali tipica dei campioni di rene intero a beneficio delle cellule midollari in modo tale che i dati rappresentino l'intero asse corticomemidollare in abbondanza sufficiente. Il protocollo è semplice, rapido e adattabile e fornisce un passo avanti verso la standardizzazione della trascrittomica a singolo nucleo nella ricerca sui reni.
Introduzione
I reni mostrano un'architettura tissutale altamente complessa. Sono costituiti da segmenti funzionalmente e anatomicamente distinti lungo un asse corticomemidollare e mediano funzioni biologiche, come la regolazione del volume del fluido extracellulare, l'equilibrio elettrolitico o l'omeostasi acido-base1.
I progressi nella trascrittomica monocellulare hanno permesso la caratterizzazione approfondita di tessuti complessi e accelerato la comprensione dell'espressione genica specifica del segmento e del tipo di cellula nella fisiologia, nello sviluppo e nella malattia renale 2,3,4.
Tuttavia, i protocolli di dissociazione basati sugli enzimi che sono frequentemente utilizzati per scRNA-seq mostrano diversi inconvenienti e vincoli. A seconda del protocollo, generano risposte allo stress trascrizionale e dissociazione tissutale verso tipi di cellule corticali più facili da dissociare 5,6. Sebbene i protocolli che utilizzano proteasi attive a freddo per i reni embrionali siano in grado di mitigare le alterazioni trascrizionali legate allo stress, non riescono a superare il pregiudizio di dissociazione verso le cellule corticali e potrebbero non essere facilmente adattabili a diversi tipi di tessuti renali malati7. Inoltre, gli approcci unicellulari non sono facilmente compatibili con campioni di tessuto congelato, limitando la loro applicazione principalmente a tessuti freschi non archiviati, rendendo così la raccolta di tessuti un fattore restrittivo6.
Il sequenziamento dell'RNA a singolo nucleo (snRNA-seq) può aggirare queste limitazioni 8,9. Qui, presentiamo un protocollo per l'isolamento dei nuclei da una fetta centrale di tessuto renale di topo adulto congelato (Figura 1) 10. Il nostro protocollo è semplice e fornisce un approccio standardizzato per ottenere librerie di sequenziamento dell'RNA con una rappresentazione bilanciata di diversi tipi di cellule renali per modelli sperimentali che non comportano forti cambiamenti tissutali regionali. In quest'ultimo caso, il nostro protocollo può essere eseguito anche con reni interi.
Protocollo
Tutti gli esperimenti sugli animali sono stati condotti in conformità con l'Animal Welfare Act (TierSchG) e l'Animal Welfare Experimental Animal Regulation (TierSchVersV) e sono stati autorizzati dalle autorità locali e dagli Animal Welfare Officers presso la nostra istituzione (MDC).
1. Preparazione dei tessuti
- Preparare una piastra a 6 pozzetti contenente 2 ml di 1x soluzione salina tamponata fosfato (PBS) per pozzetto per ciascun rene che si otterrà. Preparare una piastra a 6 pozzetti contenente 2 ml di soluzione di stabilizzazione dell'RNA per pozzetto e rene. Pre-raffreddare entrambi i piatti sul ghiaccio.
- Eutanasia di un topo maschio C57BL/6 di 3-6 mesi. Posizionare il mouse su un vassoio da dissezione, fissare le estremità e sterilizzare l'addome con etanolo al 70%.
- Apri l'addome fino alla gabbia toracica usando una pinza e delle forbici. Sollevare l'intestino e altri organi di lato e rimuovere i reni tagliando con cura l'uretere, l'arteria renale e la vena con una forbice. Lavare il rene nel PBS 1x ghiacciato precedentemente preparato e rimuovere la fascia renale e qualsiasi grasso residuo dal rene fino a quando tutto il tessuto bianco non viene rimosso (Figura 2A).
- Posizionare il rene su una piastra di dissezione fredda e utilizzare un bisturi affilato o una lama di rasoio per ottenere una fetta centrale di 1-2 mm. Assicurarsi che il pezzo di tessuto contenga l'intero asse corticomemidollare (Figura 2B). Utilizzare forbici e pinze per microdissezione per tagliare con cura la corteccia dai lati del pezzo centrale. All'interno del pezzo di tessuto sezionato, i tre segmenti corteccia, midollo esterno e midollo interno dovrebbero essere chiaramente visibili (Figura 2C).
NOTA: La fetta non deve superare uno spessore di 2 mm o un peso di 20 mg per garantire quantità tampone sufficienti per un'efficace lisi tissutale e al fine di ridurre al minimo l'RNA di fondo ambientale nelle librerie di cDNA. L'RNA ambientale spreca la capacità di sequenza, in quanto non è associato a singoli nuclei. - Trasferire il pezzo di rene nella soluzione di stabilizzazione dell'RNA precedentemente preparata e incubare per 24 ore a 4 °C per evitare la degradazione dell'RNA. Dopo 24 ore, rimuovere la soluzione di stabilizzazione dell'RNA e conservare il tessuto a -80 °C fino a nuovo utilizzo. Rimuovere con cautela la soluzione in eccesso con carta velina.
2. Isolamento dei nuclei
- Fasi di pulizia e preparazione
- Pulire i piani di lavoro e le pipette con etanolo al 70% e soluzione di decontaminazione della RNasi.
- Pulire un tubo di macina tissue a fondo rotondo da 2 ml e il pestello A e B corrispondenti con soluzione di decontaminazione della RNasi, seguiti da etanolo al 70% e acqua priva di RNasi (1 tubo di tritacarne e set di pestello per campione). Lasciate asciugare completamente.
- Preraffreddare la centrifuga a 4 °C.
- Etichettare e pre-raffreddare tre provette di raccolta da 15 ml, una provetta di raccolta da 1,5 ml, una provetta di raccolta FACS (fluorescence-activated cell sorting) da 5 mL e una provetta per macinatrice a secco per ogni campione su ghiaccio.
- Preparazione del buffer
- Riscaldare la soluzione madre del complesso ribonucleoside-vanadile a 65 °C fino a quando non viene ricostituita in una soluzione limpida verde-nera secondo le istruzioni del produttore. 11
- Preparare 1x PBS contenente il 4% di albumina sierica bovina (BSA) come descritto nella Tabella 1A. Inoltre, preparare 1x PBS con 0,04% BSA (Tabella 1B). Filtrare entrambe le soluzioni utilizzando un filtro a siringa a membrana SFCA (SurioCession) privo di tensioattivi (SFCA) da 0,2 μm e mantenere in ghiaccio fino a ulteriore utilizzo.
- Preparare il tampone di lisi dei nuclei 1 (NLB1, Tabella 1C). Aggiungere 4 mL di tampone di lisi EZ per il tampone di lisi dei nuclei 2 (NLB2, Tabella 1D) e 2 ml di BSA / PBS allo 0,04% per il tampone di sospensione dei nuclei (NSB, Tabella 1E) a tubi da 15 ml. Aggiungere la soluzione di inibitore della RNasi a NLB2 e NSB direttamente prima dell'uso, come indicato di seguito nel protocollo. Conservare in ghiaccio fino a nuovo utilizzo.
- Preparare il tampone di lisi EZ con il 10% di saccarosio (Sucrose Gradient Buffer, Tabella 1F). Mescolare bene e filtrare il tampone in un tubo fresco da 15 ml utilizzando un filtro a siringa a membrana SFCA da 0,2 μm. Conservare in ghiaccio fino a nuovo utilizzo.
- Omogeneizzazione dei tessuti e lisi cellulare
NOTA: Al fine di ridurre al minimo la degradazione dell'RNA, tutte le fasi vengono eseguite su ghiaccio. Il tubo della smerigliatrice, la piastra di Petri e tutti i buffer devono essere pre-raffreddati. Tutte le fasi di risospensione vengono eseguite mediante pipettaggio accurato della sospensione dei nuclei. Non vortice il campione per evitare forze di taglio e danni ai nuclei.- Prendere il pezzo di rene congelato e trasferirlo in una capsula di Petri di polistirene da 60 mm su ghiaccio contenente 1 mL di NLB1.
- Tritare accuratamente il tessuto usando una lametta o un bisturi (Figura 3A).
- Tagliare la punta di una punta di pipetta da 1 mL e trasferire il tessuto tritato e il tampone nel tubo della smerigliatrice. Assicurati di trasferire tutti i pezzi di tessuto. Lavare la capsula di Petri 5-10 volte con il tampone, se necessario.
- Omogeneizzare la sospensione sul ghiaccio spostando lentamente il pestello A, 25x su e giù nel tubo della smerigliatrice. Evitare bolle d'aria causate da movimenti rapidi (Figura 3B).
- Far passare l'omogenato attraverso un filtro da 100 μm in un tubo di raccolta preraffreddato da 15 ml e lavare il filtro con un altro 1 mL di NLB1.
- Lavare il tubo della smerigliatrice con tampone di lisi a freddo per nuclei EZ e scartare il tampone.
- Trasferire nuovamente l'omogenato nel tubo della smerigliatrice e omogeneizzare la sospensione sul ghiaccio spostando lentamente il pestello B, 15x su e giù nel tubo della smerigliatrice. Evitare bolle d'aria causate da movimenti rapidi (Figura 3C).
- Trasferire l'omogenato in un tubo di raccolta preraffreddato da 15 ml. Lavare il tubo della smerigliatrice con altri 2 ml di NLB1 e assicurarsi di trasferire tutti i frammenti di tessuto nel tubo di raccolta. Incubare l'omogenato (volume totale di 4 ml) per 5 minuti su ghiaccio per lisare le cellule.
- Depurazione dei nuclei
- Far passare l'omogenato attraverso un filtro da 40 μm in un tubo di raccolta pre-raffreddato da 15 ml. Centrifugare il tubo di raccolta per 5 minuti a 500 x g a 4 °C in una centrifuga con rotore a benna oscillante. Nel frattempo, aggiungere la soluzione di inibitore della RNasi a NLB2 (Tabella 1D).
- Rimuovere il surnatante senza disturbare il pellet. Risospendere con cautela il pellet in 4 mL di NLB2.
- Coprire accuratamente la sospensione con un cuscino da 1 mL di Sucrose Gradient Buffer. Centrifugare a 500 x g per 5 minuti a 4 °C in una centrifuga con rotore a benna oscillante. Nel frattempo, aggiungere la soluzione di inibitore della RNasi a NSB (Tabella 1E).
- Dopo la centrifugazione, rimuovere delicatamente il tubo di raccolta dalla centrifuga e fare attenzione a non disturbare i due strati durante la manipolazione del tubo di raccolta. I detriti cellulari sono visibili tra i due strati. Rimuovere il surnatante con attenzione iniziando dai detriti. Rimuovere il surnatante rimanente senza disturbare il pellet nucleico e risospendere con attenzione il pellet in 1 mL di NSB.
NOTA: Il volume di risospensione dipende dalla quantità di tessuto utilizzato per l'isolamento e dalle dimensioni del pellet acquisite dopo l'ultima fase di centrifugazione. Potrebbe essere necessario adattare il volume al numero previsto di nuclei. - Far passare l'omogenato attraverso un filtro da 20 μm nel tubo di raccolta FACS da 5 mL preraffreddato.
3. Ordinamento dei nuclei
- Aggiungere 20 μL di 4′,6-diamidino-2-fenilindolo (DAPI) per mL di NSB ad una concentrazione finale di 2 μM nell'omogenato nella provetta di raccolta FACS e mescolare accuratamente. Incubare per 5 minuti su ghiaccio.
- Preparare la provetta di raccolta preraffreddata da 1,5 mL con 20 μL di BSA al 4% /1x PBS e aggiungere la soluzione di inibitore della RNasi 0,5 μL ad una concentrazione finale di 1 U/μL. Procedere immediatamente alla selezione.
- Ordinare i nuclei usando un selezionatore di cellule.
- Mescolare brevemente la sospensione dei nuclei prima di inserire il tubo di raccolta FACS nella selezionatrice.
- Impostare un primo gate P1 basato sulla dispersione diretta (FSC) e sulla dispersione laterale (SSC) per escludere detriti e aggregati (Figura 4A).
- Per escludere nuclei vuoti o danneggiati e multipletti, impostare un gate successivo basato sull'area DAPI rispetto all'altezza DAPI (DAPI-A vs DAPI-H) (Figura 4B).
- Ordinare singoli nuclei nella provetta di raccolta da 1,5 mL contenente il 4% di BSA/1x PBS con 1 soluzione di inibitore della RNasi U/μL preparata in 3.2.
4. Controllo qualità
- Misurare la concentrazione finale dei nuclei al microscopio a fluorescenza o in una camera di conteggio automatizzata in almeno due conteggi indipendenti e valutare la qualità della sospensione (Figura 5).
NOTA: Le concentrazioni ottimali sono comprese tra 700 - 1.200 nuclei / μL. Concentrazioni cellulari più basse come 700 nuclei / μL possono essere preferibili in quanto le librerie di cDNA risultanti contenevano meno RNA di fondo ambientale (trascritti non associati a singoli nuclei). - Calcolare il volume richiesto di sospensione di nuclei per il recupero desiderato di singoli nuclei sequenziati. Al fine di evitare l'aggregazione dei nuclei e la degradazione dell'RNA, procedere immediatamente alla preparazione della libreria.
Risultati
Per determinare le prestazioni del nostro protocollo, abbiamo utilizzato il 10x Genomics Chromium Single Cell 3' Gene Expression Kit v3.1 per la preparazione della libreria e analizzato i dati snRNA-seq con il pacchetto Seurat12,13.
La Figura 6 mostra i risultati di una libreria snRNA-seq rappresentativa. Per valutare la qualità dei nostri nuclei, abbiamo tracciato il numero di geni rispetto al numero di trascritti (definiti da identificatori molecolari univoci (UMI)) colorati dalla frazione di letture mitocondriali (Figura 6A). I nuclei di buona qualità mostrano generalmente un numero più elevato di letture, correlando UMI e numeri di geni e basse frazioni di lettura mitocondriale.
Per l'analisi successiva, sono stati esclusi nuclei con meno di 500 o più di 4000 geni contati, o più del 5% di RNA mitocondriale (n = 828). Sono stati inclusi solo i geni espressi in un minimo di tre nuclei. Abbiamo rilevato circa 20.000 geni in totale nei restanti 6.000 nuclei con 1.600 geni mediani e 2.800 UMI mediani per nucleo (Figura 6B).
Il clustering era basato su geni altamente variabili. Abbiamo identificato un totale di 18 cluster. Le identità cellulari sono state annotate sulla base di geni marcatori noti (non mostrati). I sottogruppi di un tipo di cellula sono stati riassunti in un cluster risultando in un totale di 11 tipi di cellule distinte: podociti, tubulo prossimale (PT), arto sottile (tL), arto ascendente spesso (TAL), tubulo contorto distale (DCT), tubulo di collegamento (CNT), cellule principali del dotto collettore e intercalate (CD-PC, A-IC, B-IC), epitelio midollare profondo del bacino (DMEP) ed endotelio. I modelli di espressione genica dei marcatori arricchiti di cluster sono stati visualizzati in un dot plot (Figura 6C) e cluster di tipi cellulari in un grafico t-distributed stochastic neighbor embedding (t-SNE) (Figura 6D).
Per valutare le distribuzioni dei tipi di cellule nel nostro campione, è stata calcolata la percentuale di ciascun tipo di cellula (Figura 6E) e utilizzata per determinare il rapporto tra PT e TAL. Il PT si trova principalmente nella corteccia renale e spesso sovrarappresentato nei set di dati renali a singola cellula poiché le cellule del PT sono facili da dissociare e altamente abbondanti nei campioni di rene intero. Il TAL d'altra parte si estende su tutto il midollo esterno14. Pertanto, il rapporto tra le frazioni PT e TAL rappresenta una buona misura per l'arricchimento dei tipi di cellule midollari in un set di dati di cellule singole renali. In generale, il rapporto PT/TAL nei set di dati di rene intero a singola cellula variava da 8 (dati non pubblicati da tessuto renale intero trattato con proteasi fredda) a 45 per tessuto dissociato enzimaticamente10,14,15. Nel set di dati snRNA-seq qui presentato siamo stati in grado di raggiungere un rapporto PT / TAL di 2. Questo risultato illustra che la rimozione della corteccia in eccesso durante la dissezione tissutale combinata con snRNA-seq si traduce in una rappresentazione del tipo di cellule renali notevolmente migliorata.
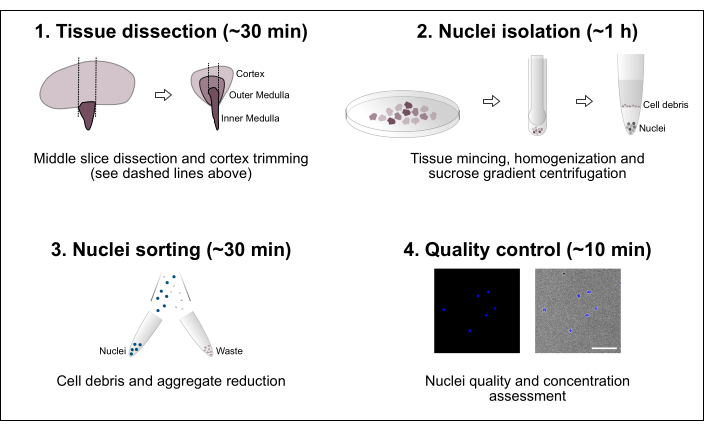
Figura 1: Panoramica schematica del flusso di lavoro. Il protocollo consiste in quattro fasi principali che includono la dissezione dei tessuti seguita dall'isolamento dei nuclei, dalla selezione dei nuclei e da una valutazione finale della purezza e della concentrazione. Barra di scala = 100 μm. Fare clic qui per visualizzare una versione ingrandita di questa figura.
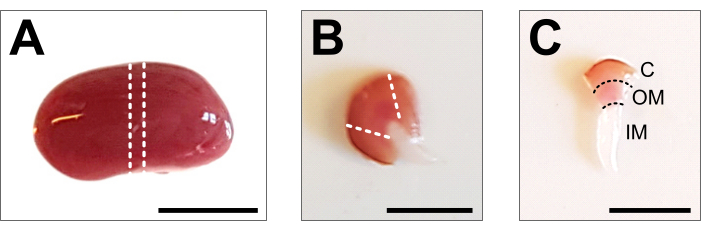
Figura 2: Dissezione renale e preparazione dei tessuti . (A) Immagine rappresentativa del rene intero sezionato. Le linee tratteggiate indicano i tagli necessari per ottenere una fetta centrale di 1-2 mm con una rappresentazione di tutti i tipi di cellule renali. (B) Immagine rappresentativa della fetta centrale ottenuta. Le linee tratteggiate indicano i tagli per il taglio della corteccia dal lato. (C) Immagine rappresentativa del pezzo di rene centrale con corteccia tagliata. La corteccia (C), il midollo esterno (OM) e il midollo interno (IM) sono chiaramente visibili. Barra di scala = 500 μm. Fare clic qui per visualizzare una versione ingrandita di questa figura.
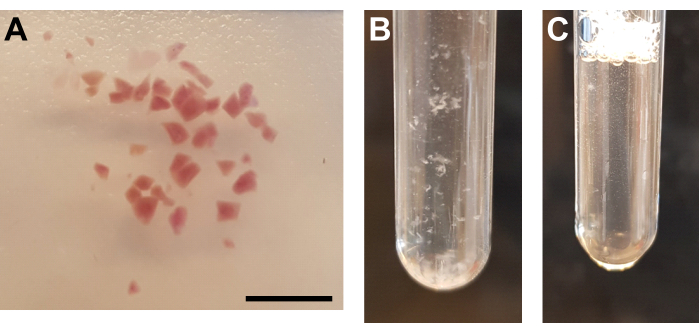
Figura 3: Omogeneizzazione dei tessuti e purificazione dei nuclei . (A) Immagine rappresentativa che mostra tessuto renale sufficientemente tritato. Barra della scala = 500 μm. (B) Omogenato dopo la prima fase di omogeneizzazione (25 colpi con pestello A, tubo di macinazione da 2 ml). (C) Omogenato dopo seconda fase di omogeneizzazione (15 colpi con pestello B, tubo di macinazione da 2 ml). Fare clic qui per visualizzare una versione ingrandita di questa figura.

Figura 4: Strategia di gating per l'ordinamento dei nuclei. (A ) Un primo gate P1 è stato impostato in base allo scatter in avanti (FSC) rispetto allo scatter laterale (SSC) per escludere detriti e aggregati. (B ) Un gate successivo basato su DAPI-Area (DAPI-A) vs DAPI-Height (DAPI-H) escludeva nuclei vuoti o danneggiati e multipletti. Fare clic qui per visualizzare una versione ingrandita di questa figura.
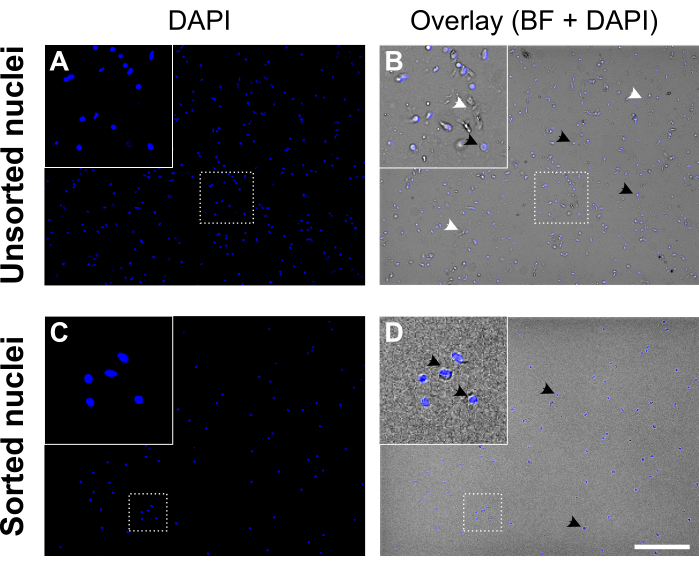
Figura 5: Sospensione dei nuclei prima e dopo l'ordinamento dei nuclei. (A, C) Nuclei colorati con DAPI (blu). (B, D) Sovrapposizione di DAPI e canale brightfield (BF). Prima dello smistamento (pannello superiore) la sospensione dei nuclei contiene detriti cellulari e aggregati (etichettati con punte di freccia bianche). Dopo l'ordinamento (pannello inferiore) la sospensione dei nuclei appare molto più pulita. Esempi di nuclei colorati con DAPI sono etichettati con punte di freccia nere. I nuclei di buona qualità appaiono rotondi e lisci con una membrana intatta e sono ben separati, mentre i nuclei di scarsa qualità appaiono rugosi e mostrano la perdita della membrana nucleare. Barra di scala = 250 μm. Fare clic qui per visualizzare una versione ingrandita di questa figura.
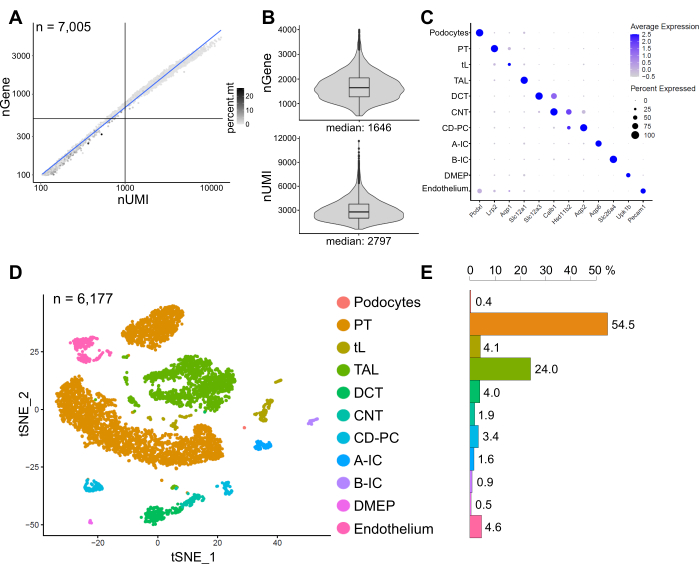
Figura 6: Controllo di qualità e analisi di un set di dati rappresentativo di snRNA-seq. (A) Numero di geni (nGene) tracciati rispetto al numero di identificatori molecolari univoci (nUMI) colorati dalla frazione di letture mitocondriali (percent.mt). I nuclei di bassa qualità corrispondono al quadrante in basso a sinistra del grafico (n = 828) e sono stati esclusi dall'analisi successiva. (B) Distribuzione e mediana di nGene e nUMI rilevati per nucleo nel set di dati snRNA-seq, che rappresentano 6.177 nuclei (> 500 geni). Le librerie sono state sequenziate a una profondità mediana di ~ 8.200 letture mappate per nucleo. (C) Dot plot che mostra i modelli di espressione genica di marcatori arricchiti di cluster (asse x) per singoli tipi di cellule (asse y). La dimensione del punto corrisponde alla proporzione di cellule che esprimono il gene indicato. Il colore corrisponde all'espressione media. (D) T-distributed stochastic neighbor embedding (t-SNE) plot di tipi cellulari identificati. (E) Distribuzione del tipo di cellula nel set di dati snRNA-seq. PT, tubulo prossimale; tL, arto sottile; TAL, arto ascendente spesso, DCT, tubulo contorto distale; CNT, tubulo di collegamento; CD-PC, raccolta delle celle principali del condotto; A-IC, cellule intercalate di tipo A; B-IC, cellule intercalate di tipo B; DMEP, epitelio midollare profondo del bacino. Fare clic qui per visualizzare una versione ingrandita di questa figura.
| Reagente | Concentrazione finale | Volume (ml) | ||
| (A) 4 % BSA / PBS | ||||
| Soluzione salina tamponata con fosfato (PBS) con albumina bovina al 10% | 4% | 2 | ||
| PBS (soluzione salina tamponata con fosfato) 1X senza calcio o magnesio | - | 3 | ||
| (B) 0,04 % BSA / PBS | ||||
| 4 % BSA / PBS | 0.04 % | 0.5 | ||
| PBS (soluzione salina tamponata con fosfato) 1X senza calcio o magnesio | - | 49.5 | ||
| c) Tampone di lisi nucleica 1 (NLB1) | ||||
| Tampone di lisi nucleare EZ | - | 4 | ||
| Inibitore della RNasi RiboLock (40 U/μL) | 1 U/μL | 0.1 | ||
| Complesso ribonucleoside-vanadile (200 mM) | 10 mM | 0.2 | ||
| d) Tampone di lisi nucleica 2 (NLB2) | ||||
| Tampone di lisi nucleare EZ | - | 4 | ||
| Inibitore della RNasi RiboLock (40 U/μL) | 1 U/μL | 0.1 | ||
| e) Tampone di sospensione dei nuclei (NSB) | ||||
| 0,04 % BSA / PBS | - | 2 | ||
| Inibitore della RNasi RiboLock (40 U/μL) | 1 U/μL | 0.05 | ||
| f) tampone sfumato di saccarosio (10 % di saccarosio) | ||||
| Peso 1 g di saccarosio | ||||
| Sciogliere in 6 ml di tampone di lisi nucleare EZ | ||||
| Riempire fino a 10 mL con Nuclear EZ Lysis Buffer | ||||
| Filtrare attraverso un filtro a siringa da 0,2 μm in un tubo nuovo | ||||
Tabella 1: Ricette di soluzione: (A) Preparazione di BSA/1x PBS al 4%. Filtrare con un filtro a siringa a membrana SFCA da 0,2 μm e conservare in ghiaccio fino all'uso. (B) Preparazione dello 0,04% BSA/1 x PBS. Filtrare con un filtro a siringa a membrana SFCA da 0,2 μm e conservare in ghiaccio fino all'uso. c) preparazione del tampone di lisi dei nuclei 1 (NLB1). I volumi indicati sono forniti per campione. Conservare in ghiaccio fino all'uso. d) Preparazione del tampone di lisi nucleica 2 (NLB2). I volumi indicati sono forniti per campione. Aggiungere RiboLock RNasi Inhibitor a NLB2 direttamente prima dell'uso, come indicato nel protocollo. Conservare in ghiaccio fino all'uso. e) Preparazione del tampone di sospensione dei nuclei (NSB). I volumi indicati sono forniti per campione. Aggiungere RiboLock RNase Inhibitor a NSB direttamente prima dell'uso come indicato nel protocollo. Conservare in ghiaccio fino all'uso. f) preparazione del tampone sfumato di saccarosio. Filtrare con un filtro a siringa a membrana SFCA da 0,2 μm e conservare in ghiaccio fino all'uso.
Discussione
La trascrittomica a singola cellula fa progredire la comprensione dell'espressione genica specifica del tipo di cellula nella fisiologia e nella malattia renale. Qui, abbiamo fornito un metodo semplice e riproducibile per isolare singoli nuclei di alta qualità dal tessuto renale di topo congelato per snRNA-seq in modo standardizzato.
Per snRNA-seq, è fondamentale utilizzare nuclei di alta qualità come input per la generazione di librerie ed evitare la degradazione dell'RNA durante l'elaborazione dei tessuti. Pertanto, l'incubazione di pezzi di tessuto in soluzione di stabilizzazione dell'RNA immediatamente dopo la dissezione è essenziale per proteggere e stabilizzare l'RNA cellulare e consente di conservare campioni a - 80 °C indefinitamente. Quando si applica questo protocollo a tessuti congelati senza trattamento con soluzione di stabilizzazione dell'RNA, come materiale d'archivio, è necessario un test e la qualità dell'RNA deve essere valutata poiché abbiamo osservato una significativa perdita di integrità dell'RNA nel tessuto congelato senza previa incubazione nella soluzione di stabilizzazione dell'RNA.
In generale, un'adeguata gestione dei campioni è fondamentale per massimizzare il recupero di singoli nuclei intatti. Tutte le fasi di risospensione devono essere eseguite mediante pipettaggio con attenzione per evitare sollecitazioni da taglio e danni fisici. I buffer per la risospensione finale dei nuclei e lo smistamento dei nuclei dovrebbero contenere BSA per evitare la perdita e l'aggregazione dei nuclei.
I volumi tampone in questo protocollo sono ottimizzati per campioni di tessuto molto piccoli (~15 mg). È fondamentale garantire la lisi cellulare completa e un lavaggio sufficiente per generare sospensioni di alta qualità. Blocchi di tessuto più grandi o campioni di rene interi si tradurranno in concentrazioni eccessive di nuclei che portano a aggregazione e aggregazione, elevata abbondanza di RNA ambientale e scarsa qualità complessiva della sospensione. Se vengono elaborati campioni più grandi o altri tessuti, si consiglia vivamente di eseguire prove per determinare i volumi tampone ottimali per livelli minimi di RNA ambientale. La qualità e le concentrazioni dei nuclei e dell'RNA devono essere esaminate attentamente poiché il sovraccarico si traduce in prestazioni complessive scadenti.
Inoltre, grandi quantità di detriti cellulari, che causano alti livelli di RNA ambientale non associati a singoli nuclei, influenzano negativamente i risultati del sequenziamento. La chiarificazione della sospensione dei nuclei mediante centrifugazione attraverso un cuscino di saccarosio mitiga questo problema in una certa misura, ma può anche portare a distorsioni nella rappresentazione del tipo cellulare mediante controselezione contro nuclei densi e piccoli presenti, ad esempio, nelle cellule immunitarie16. Se questo è preoccupante, il gradiente di saccarosio dovrebbe essere omesso. Al contrario, abbiamo scoperto che la citometria a flusso basata sulla colorazione DAPI era fondamentale per ridurre la quantità di detriti cellulari al fine di produrre una sospensione di nuclei singoli di alta qualità.
L'isolamento di singoli nuclei presenta notevoli vantaggi rispetto agli approcci unicellulari8. È compatibile con tessuti correttamente congelati, rendendo la raccolta dei tessuti più flessibile e aggira la necessità di dissociazione tissutale basata sugli enzimi, che può introdurre risposte di stress trascrizionale 6,17. Inoltre, supera il bias di dissociazione che favorisce la selezione di tipi cellulari facilmente dissociabili della corteccia renale, che può portare ad una sottorappresentazione dei tipi di cellule midollari in alcuni approcci basati sugli enzimi 5,6,10.
L'uso di un pezzo di rene centrale invece dell'intero tessuto renale consente di risparmiare risorse e corregge ulteriormente la sovrarappresentazione di abbondanti tipi di cellule come descritto in precedenza10. Tuttavia, a seconda del modello murino o del fenotipo studiato, può essere utile utilizzare campioni di rene intero invece di una singola fetta centrale. I campioni di rene intero possono essere più rappresentativi delle proporzioni cellulari reali o dei cambiamenti che si verificano nell'intero rene, mentre una fetta centrale tagliata si è dimostrata vantaggiosa per i fenotipi midollari o quando il materiale del campione era limitato. Questa decisione, quindi, è altamente specifica per l'utente e dovrebbe essere considerata attentamente.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Ringraziamo la piattaforma di genomica scientifica presso il Centro Max Delbrück per la medicina molecolare dell'Associazione Helmholtz, Berlino per il supporto tecnico.
JL e KMSO sono stati sostenuti dal gruppo di formazione alla ricerca della Fondazione tedesca per la ricerca (DFG) GRK 2318 e dall'unità di ricerca FOR 2841. KMSO è stato supportato dal Collaborative Research Grant 1365. AB è stata sostenuta dal finanziamento del Premio Gottfried Wilhelm Leibniz del DFG assegnato a NR.
Materiali
| Name | Company | Catalog Number | Comments |
| Cell sorter | - | - | For fluorescence-activated cell sorting (FACS); e.g. BD FACSAria Cell Sorter. |
| Centrifuge 5810 R | Eppendorf | 5811000015 | |
| Countess Cell Counting Chamber Slides | Invitrogen | C10228 | |
| Countess II FL Automated Cell Counter | Invitrogen | AMQAF1000 | Needs to contain DAPI light cube to count nuclei. Alternatively, nuclei can be counted manually under fluorescence microscope. |
| 4′,6-Diamidino-2-phenyl-indol-dihydrochlorid (DAPI) | Biotrend | 40043/b | Stock solution prepared with a concentration of 100 µM. Used for nuclei staining in a final concentration of 2 µM. |
| D(+)-Sucrose ≥99.9%, ultrapure DNAse-, RNAse-free | VWR | 0335-500G | |
| DNA LoBind Microcentrifuge Tubes (1.5 mL) | Eppendorf | 22431021 | |
| Ethanol, 70 % | - | - | |
| FACS tubes | pluriSelect | 43-10100-46 | |
| KIMBLE Dounce tissue grinder set 2 mL complete | Sigma-Aldrich | D8938-1SET | |
| Minisart Syringe Filters 0.2 µm | Sartorius | 16534-GUK | |
| Nuclease-free Water | Invitrogen | AM9937 | |
| Nuclei EZ Prep Nuclei Isolation Kit | Sigma-Aldrich | NUC-101 | Nuclei EZ Lysis Buffer (Product No. N3408) needed for buffer preparation. |
| PBS (Phosphate-Buffered Saline) 1X without calcium or magnesium | Corning | 21-040-CV | |
| Petri dishes, polystyrene 60 mm | Sigma-Aldrich | P5481 | |
| Phosphate-Buffered Saline (PBS) with 10% Bovine Albumin | Sigma-Aldrich | SRE0036 | |
| pluriStrainer Mini 100 µm | pluriSelect | 43-10100-46 | |
| pluriStrainer Mini 20 µm | pluriSelect | 43-10020-40 | |
| pluriStrainer Mini 40 µm | pluriSelect | 43-10040-40 | |
| Polystyrene Centrifuge Tube (15 mL) | Falcon | 352099 | |
| Razor blades | - | - | |
| RiboLock RNase Inhibitor (40 U/µL) | Thermo Fisher | EO0384 | |
| Ribonucleoside-vanadyl complex | New England Biolabs | S1402S | Follow manufacturer's instructions (https://international.neb.com/products/s1402-ribonucleoside-vanadyl-complex#Product%20Information). Upon use the 200 mM stock solution is reconstituted to a green-black clear solution by incubating at 65 °C. |
| RNAlater Stabilization Solution | Invitrogen | AM7020 | |
| RNase AWAY | Fisher Scientific | 11952385 |
Riferimenti
- Thomas, R. S. Kidney modeling and systems physiology. Wiley Interdisciplinary Reviews: Systems Biology and Medicine. 1 (2), 172-190 (2009).
- Potter, S. S. Single-cell RNA sequencing for the study of development, physiology and disease. Nature Reviews Nephrology. 14 (8), 479-492 (2018).
- Park, J., Liu, C. L., Kim, J., Susztak, K. Understanding the kidney one cell at a time. Kidney International. 96 (4), 862-870 (2019).
- Clark, A. R., Greka, A. The power of one: advances in single-cell genomics in the kidney. Nature Reviews Nephrology. 16 (2), 73-74 (2020).
- Lake, B. B., et al. A single-nucleus RNA-sequencing pipeline to decipher the molecular anatomy and pathophysiology of human kidneys. Nature Communications. 10 (1), 2832(2019).
- Wu, H., Kirita, Y., Donnelly, E. L., Humphreys, B. D. Advantages of single-nucleus over single-cell RNA sequencing of adult kidney: Rare cell types and novel cell states revealed in fibrosis. Journal of the American Society of Nephrology. 30 (1), 23-32 (2019).
- Adam, M., Potter, A. S., Potter, S. S. Psychrophilic proteases dramatically reduce single-cell RNA-seq artifacts: a molecular atlas of kidney development. Development. 144 (19), 3625-3632 (2017).
- Habib, N., et al. Massively parallel single-nucleus RNA-seq with DroNc-seq. Nature Methods. 14 (10), 955-958 (2017).
- Muto, Y., et al. Single cell transcriptional and chromatin accessibility profiling redefine cellular heterogeneity in the adult human kidney. Nature Communications. 12 (1), 2190(2021).
- Hinze, C., et al. Kidney single-cell transcriptomes predict spatial corticomedullary gene expression and tissue osmolality gradients. Journal of the American Society of Nephrology. 32 (2), 291(2021).
- Berger, S. L. Isolation of cytoplasmic RNA: ribonucleoside-vanadyl complexes. Methods in Enzymology. 152, 227-234 (1987).
- Butler, A., Hoffman, P., Smibert, P., Papalexi, E., Satija, R. Integrating single-cell transcriptomic data across different conditions, technologies, and species. Nature Biotechnology. 36 (5), 411-420 (2018).
- Stuart, T., et al. Comprehensive Integration of single-cell data. Cell. 177 (7), 1888-1902 (2019).
- Park, J., et al. Single-cell transcriptomics of the mouse kidney reveals potential cellular targets of kidney disease. Science. 360 (6390), 758-763 (2018).
- Kirita, Y., Wu, H., Uchimura, K., Wilson, P. C., Humphreys, B. D. Cell profiling of mouse acute kidney injury reveals conserved cellular responses to injury. Proceedings of the National Academy of Sciences of the United States of America. 117 (27), 15874-15883 (2020).
- Schneeberger, S., et al. The neuroinflammatory interleukin-12 signaling pathway drives Alzheimer's disease-like pathology by perturbing oligodendrocyte survival and neuronal homeostasis. bioRxiv. , 441313(2021).
- Nguyen, Q. H., Pervolarakis, N., Nee, K., Kessenbrock, K. experimental considerations for single-cell RNA sequencing approaches. Frontiers in Cell and Developmental Biology. 6, 108(2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon