Method Article
Conversione di cellule staminali pluripotenti indotte dall'uomo (IPSC) in neuroni motore spinale e cranico funzionale utilizzando vettori PiggyBac
In questo articolo
Riepilogo
Questo protocollo consente la conversione rapida ed efficiente delle cellule staminali pluripotenti indotte in neuroni motori con un'identità spinale o cranica, mediante l'espressione ectopica dei fattori di trascrizione dai vettori inducibili piggyBac.
Abstract
Descriviamo qui un metodo per ottenere funzionali neuroni motori spinale e craniale da cellule staminali pluripotenti indotte dall'uomo (IPSC). La conversione diretta in motore neurone è ottenuta per espressione ectopica di moduli alternativi di fattori di trascrizione, vale a dire Ngn2, Isl1 e Lhx3 (NIL) o Ngn2, Isl1 e Phox2a (NIP). NIL e NIP specificano, rispettivamente, l'identità del motore neuronale spinale e craniale. Il nostro protocollo inizia con la generazione di linee iPSC modificate in cui NIL o NIP sono stabilmente integrati nel genoma tramite un vettore di trasposizione piggyBac. L'espressione dei transgeni viene quindi indotta dalla doxiciclina e conduce, in 5 giorni, alla conversione degli iPSCs in progenitori MN. La successiva maturazione, per 7 giorni, conduce a popolazioni omogenee di MNs spinale o craniale. Il nostro metodo contiene diversi vantaggi rispetto ai protocolli precedenti: è estremamente rapido e semplificato; non richiede infezione virale o ulteriore isolamento MN; permette di generare diverse sottopopolazioni MN (spinali e craniche) con un notevole grado di maturazione, come dimostrato dalla capacità di sparare i treni dei potenziali d'azione. Inoltre, un gran numero di motoneuroni può essere ottenuto senza purificazione da popolazioni miste. i neuroni del motore spinale e craniale derivati da iPSC possono essere utilizzati per la modellazione in vitro della sclerosi laterale amiotrofico e di altre malattie neurodegenerative del Neuron motorio. Le popolazioni di neuroni omogenei del motore potrebbero rappresentare una risorsa importante per proiezioni di farmaci specifici per tipo di cellula.
Introduzione
La degenerazione del motoneurico (MN) svolge un ruolo causale nelle malattie umane come la sclerosi laterale amiotrofico (ALS) e l'atrofia muscolare spinale (SMA). La creazione di sistemi di modelli cellulari idonei in vitro che ricapitolano la complessità del MN umano è un passo importante verso lo sviluppo di nuovi approcci terapeutici. Le cellule staminali pluripotenti indotte (iPSCs), che sono dotate di notevoli proprietà di differenziazione plurilineage, sono state ora derivate da un numero di pazienti affetti da malattie motoneuriche1,2. Ulteriori linee iPSC che trasportano mutazioni patogene associate a malattie MN sono state generate dall'editing genico, partendo dal controllo delle cellule staminali pluripotenti "sane"3. Queste linee rappresentano strumenti utili per la modellazione delle malattie in vitro e lo screening dei farmaci, purché siano disponibili metodi appropriati per la differenziazione iPSC in MNs. La logica alla base dello sviluppo di questo metodo è quella di fornire alla comunità scientifica interessata alle malattie MN un protocollo di differenziazione veloce ed efficiente che dà origine a MNs funzionali maturi. Il primo vantaggio di questo metodo è il suo lasso di tempo di esecuzione. Un altro punto di forza rilevante deriva dall'eliminazione di qualsiasi fase di purificazione. Infine, il protocollo può essere utilizzato per generare due distinte popolazioni di motoneuroni.
La possibilità di generare diversi sottotipi di MNs è particolarmente rilevante per la modellazione delle malattie MN. Non tutti i sottotipi MN sono ugualmente vulnerabili in ALS e SMA e l'insorgenza dei sintomi in diverse unità motoria influisce notevolmente sulla prognosi. Nella SLA, l'esordio spinale con sintomi che iniziano negli arti superiori e inferiori conduce alla morte in circa 3-5 anni4. Al contrario, l'insorgenza di bulbare, a cominciare dalla degenerazione di MNs craniali, ha una prognosi peggiore. Inoltre, la percentuale di insorgenza di bulbare è significativamente più alta nei pazienti con mutazioni nelle proteine leganti l'RNA FUS e TDP-43 rispetto agli individui con mutazioni SOD15. Quasi la totalità dei protocolli alternativi di differenziazione MN si basa sull'attività dell'acido retinoico (RA), che conferisce un carattere spinale per differenziare iPSCs6,7,8. Ciò limita la possibilità di studiare fattori intrinseci, che potrebbero essere protettivi in specifici sottotipi di MN9,10.
Coerentemente con un precedente lavoro in cellule staminali embrionali di topo11, abbiamo recentemente dimostrato che nell'espressione ectopica iPSCs umana di Ngn2, Isl1 e LHX3 (Nil) induce un'identità spinale MN, mentre Ngn2 e Isl1 più PHOX2A (NIP) specificano MNS craniali12. Abbiamo quindi sviluppato un protocollo efficiente, che porta alla produzione di MNs umani dotati di proprietà funzionali in un turnaround di 12 giorni. Lo scopo di questo metodo è quello di ottenere, in un breve lasso di tempo e senza la necessità di purificazione (ad esempio, da FACS), popolazioni di cellule altamente arricchite per gli MNs con identità spinale o craniale.
Protocollo
1. manutenzione degli IPSC umani
-
Preparazione di piastre rivestite in matrice
- Scongelare un flaconcino da 5 mL di matrice (vedere tabella dei materiali) a 4 ° c durante la notte. Le scorte di matrici originali sono disponibili in diverse concentrazioni di scorte e le aliquote sono effettuate in base al fattore di diluizione indicato sulla scheda tecnica, specifica per il lotto individuale. È importante tenere il flaconcino e i tubi ghiacciati per evitare la gelificante prematura della matrice. Distribuire la matrice in aliquote in criotubi pre-refrigerati sul ghiaccio. Congelare le aliquote inutilizzate a-20 ° c.
- Mettere una aliquota sul ghiaccio per circa 2 h per scongelare.
- Diluire l'aliquota della matrice con 20 mL di DMEM/F12 freddo in un tubo conico 50 mL.
- Miscelare bene e erogare 1 mL di matrice diluita in piatti da 35 mm (quantità equivalenti per superficie di altri piatti).
- Tenere i piatti contenenti la matrice diluita per 1 h a temperatura ambiente per consentire il rivestimento.
Nota: I piatti, sigillati con parafilm, possono essere conservati a 4 ° c per un massimo di 2 settimane.
-
Preparazione della soluzione di riserva (20 ml) e 1x aliquote di lavoro del reagente di dissociazione delicata delle cellule (vedere tabella dei materiali).
- Sciogliere la polvere a 10 mg/mL in PBS (CA2 +/mg2 + free).
- Filtrare sterilizzare attraverso una membrana filtrante di 0,22 μm.
- Preparare 20 aliquote (1 mL ciascuna) e conservare a-20 ° c.
- Prima dell'uso, diluire un'aliquota in PBS (CA2 +/mg2 + free) a 1 mg/ml (1x aliquote di lavoro).
Nota: 1x le aliquote di lavoro possono essere conservate a 4 ° c per un massimo di 2 settimane.
-
Passeggio iPSCs umani.
- Prima di iniziare: se conservato a 4 ° c, piastre pre-calde rivestite con matrice nell'incubatore a 37 ° c per 20-30 min. preriscaldamento a temperatura ambiente la quantità di supporto iPSC umano (vedere tabella dei materiali) necessaria. Preriscaldare il DMEM/F12.
- Mezzo di coltura aspirato.
- Sciacquare gli IPSC con PBS (CA2 +/mg2 + free).
- Aggiungere 1x soluzione di dissociazione delicata (0,5 mL per un piatto da 35 mm). Incubare a 37 ° c fino a quando i bordi delle colonie iniziano a staccarsi dalla piastra, di solito 3-5 min.
- Aspirare la soluzione di dissociazione delicata, facendo attenzione a non staccare le colonie di iPSC.
- Lavare le cellule con DMEM/F12 (2 mL per un piatto 35 mm) e aspirare facendo attenzione a non staccare le cellule. Ripeti questo passaggio ancora una volta.
- Aggiungere un supporto iPSC umano (1 mL per un piatto da 35 mm).
- Staccare delicatamente le colonie con un sollevatore cellulare e trasferirla in un tubo da 15 mL.
- Rompere delicatamente i grumi delle cellule pipettando su e giù con un P1000 pipettatore 3-4 volte.
- Aspirare il surnatante dalla piastra (e) rivestita con matrici.
- Semina le cellule nel volume di coltura appropriato del medium iPSC umano. Il rapporto di divisione può variare da linea a linea ed è di circa 1:4-1:8. Cambiare il mezzo ogni giorno.
2. generazione di linee iPSC di NIL e NIP inducible
-
Trasfezione cellulare.
- Sciacquare le cellule con PBS (CA2 +/mg2 + free).
- Aggiungere il reagente di dissociazione delle cellule (vedere tabella dei materiali) (0,35 ml per un piatto da 35 mm) e incubare a 37 ° c fino a separare le singole cellule (5-10 min).
- Completare delicatamente la separazione delle cellule pipettando su e giù con un P1000 pipettatore 3-4 volte.
- Raccogliere in un tubo da 15 mL e aggiungere PBS (CA2 +/mg2 + libero) a 10 ml. Conta le celle.
- Pellet 106 cellule e risospendere in 100 μl di tampone R (incluso nel kit elettroporazione cellulare, vedere tabella dei materiali).
- Aggiungere il DNA plasmide per la trasfezione: 4,5 μg di vettore traimabile (epB-BSD-TT-NIL o epB-BSD-TT-NIP12) e 0,5 μg del piggyBac trailamento plasmide13.
- Transfect con il sistema di elettroporazione delle cellule (Vedi tabella dei materiali) secondo le istruzioni del fabbricante e come descritto in precedenza3 con i seguenti parametri: 1.200 tensione di V, 30 ms di larghezza, 1 impulso. Seminare le cellule nel mezzo umano iPSC integrato con 10 μM Y-27632 (inibitore di roccia, vedere la tabella dei materiali) in un 6 mm piatto rivestito di matrice.
-
Selezione con antibiotici.
- Due giorni dopo la trasfezione, aggiungere 5 μg/ml di alla blasticina al mezzo di coltura.
- La maggior parte delle cellule non trasfite morirà entro 48 h di selezione blasticidina. Tenere le cellule in alla blasticina per almeno 7-10 giorni per contrastare-selezionare le cellule che non hanno integrato i transgeni nel genoma.
- Mantenere stabilmente le cellule trasfected come una popolazione mista, composto da cellule con diversi numero di transgeni e siti di integrazione diversi, o isolare singoli cloni.
- Preparare un piatto aggiuntivo per verificare l'effettiva espressione dei transgeni, su 1 μg/mL di induzione della doxiciclina, da RT-PCR con primer specifici per il Ngn2 (forward: TATGCACCTCACCTCCCCATAG; Inverso: GAAGGGAGGAGGGCTCGACT), come descritto in precedenza12.
- In questa fase, congelare le scorte delle nuove linee NIL-e NIP-iPSC nel mezzo di congelamento per gli iPSCs umani (vedere la tabella dei materiali).
3. differenziazione del motore Neuron
- Dissociare le cellule con il reagente di dissociazione cellulare come descritto (passi 2.1.1.-2.1.3.). Raccogli le cellule dissociate in un tubo da 15 mL e Diluisci con 5 volumi di DMEM/F12. Pellet le cellule e risospendere in umano iPSC medio integrato con 10 μM inibitore di roccia. Contare le cellule e il seme su piatti con rivestimento a matrice a una densità di 62.500 cellule/cm2.
- Il giorno dopo, sostituire il mezzo con DMEM/F12, integrato con 1x L-Glutammina stabile analogico, 1x aminoacido non essenziale (NEAA) integratore culturale cellulare e 0.5 x penicillina/streptomicina, e contenente 1 μg/mL doxiciclina. Questo è considerato come giorno 0 di differenziazione. Il giorno 1, aggiornare il mezzo e doxiciclina.
- Il giorno 2, cambiare il medium a Neurobasal/B27 Medium (mezzo Neurobasale integrato con 1x B27, 1x stabile di L-Glutammina analogico, 1x NEAA e 0.5 x penicillina/streptomicina), contenente 5 μM DAPT, 4 μM SU5402 e 1 μg/mL doxiciclina (vedere tabella dei materiali ). Rinfrescare il fluido e la doxiciclina ogni giorno fino al giorno 5.
-
Giorno 5: dissociazione delle cellule con reagente di dissociazione delle cellule (vedere tabella dei materiali).
- Sciacquare le cellule con PBS (CA2 +/mg2 + free).
- Aggiungere il reagente di dissociazione delle cellule (0,35 mL per un piatto da 35 mm) e incubare a 37 ° c fino a quando l'intero monostrato cellulare si separa dal piatto. Si noti che le singole celle non saranno separate durante l'incubazione.
- Aggiungere 1 mL di DMEM/F12 e raccogliere le cellule in un tubo da 15 mL.
- Completare delicatamente la separazione delle cellule pipettando su e giù con un P1000 pipettatore 10-15 volte.
- Aggiungere 4 mL di DMEM/F12 e contare le celle.
- In questa fase, congelare i progenitori dei neuroni del motore nel mezzo di congelamento delle cellule (vedere tabella dei materiali), secondo le istruzioni del produttore.
- Pellet le cellule e risospendere in mezzo neuronale (Neurobasal/B27 Medium integrato con 20 ng/mL BDNF, 10 ng/mL GDNF e 200 ng/mL acido L-ascorbico, vedere tabella dei materialis) completato con inibitore di roccia 10 μm.
- Semina le cellule su poli-ornitina/laminina-o in alternativa su supporti con rivestimento a matrice alla densità di 100.000 cellule/cm2. Utilizzare supporti in plastica μ-slide con coprioggetti in polimero (vedere tabella dei materiali) per l'analisi immunocolorazione.
- Il giorno 6, cambiare il mezzo con fresco neuronale medium privo di inibitore di roccia. Nei prossimi giorni, aggiornare metà del mezzo ogni 3 giorni. Il mezzo di coltura deve essere modificato con molta attenzione al fine di evitare il distacco dalla superficie.
4. analisi immunostaining
- Fissazione delle cellule. Sciacquare le cellule con PBS (con CA2 +/mg2 +) e incubare per 15 minuti in 4% paraformaldeide in PBS (con CA2 +/mg2 +) a temperatura ambiente.
Attenzione: La paraformaldeide è tossica e sospetta di causare il cancro. Evitare il contatto con la pelle e gli occhi e maneggiare sotto una cappa chimica. - Permeabilizzare con PBS (con CA2 +/mg2 +) contenente 0,1% Triton X-100 per 5 minuti a temperatura ambiente.
- Incubare per 30 minuti a temperatura ambiente in soluzione di blocco degli anticorpi (ABS: 3% BSA in PBS con CA2 +/mg2 +).
- Incubare per 1 h a temperatura ambiente con anticorpi primari in ABS: anti-TUJ1 (1:1000; coniglio) e anti-Oct4 (1:200; topo) o anti-CHAT (anti-colina acetiltransferasi; 1:150; capra). Vedere la tabella dei materiali.
- Incubare per 45 min a temperatura ambiente con appropriata coppia di anticorpi secondari in ABS: anti-topo Alexa Fluor 647 (1:250), anti-coniglio Alexa Fluor 594 (1:250) e anti-capra Alexa Fluor 488 (1:250). Vedere la tabella dei materiali.
- Incubare in 0,4 μg/mL DAPI per 5 minuti a temperatura ambiente per etichettare i nuclei.
- Montare le celle con il supporto di montaggio (vedere la tabella dei materiali) per l'imaging a un microscopio a fluorescenza.
5. caratterizzazione funzionale tramite patch-registrazioni morsetto
- Preparare la soluzione esterna HEPES-equilibrated (NES) come segue: 140 mM NaCl, 2,8 mM KCl, 2 mM CaCl2, 2 mm MgCl2, 10 mm HEPES e 10 mm di glucosio. Impostare l'osmolarità tra 290-300 mΩ. Regolare il pH a 7,3 utilizzando 1N NaOH e conservare la soluzione a 4 ° c.
- Preparare la soluzione interna: 140 mM K-gluconato, 2 mM NaCl, 5 mM BAPTA, 2 mM MgCl2, 10 mm HEPES, 2 mm mg-ATP, 0,3 mm na-GTP. Regolate il pH a 7,3 con 1M KOH e verificate che l'osmolarità sia fissata a circa 290 mΩ. Congelare la soluzione a-20 ° c in piccole aliquote.
- Prima di eseguire gli esperimenti, preriscaldare la soluzione NES in un bagno d'acqua a circa 28-30 ° c.
- Estrarre alcune Micropipette in borosilicato (ID 0,86 mm; OD 1,5 mm) con una resistenza alla punta: 5-6 MΩ e riempire con la soluzione intracellulare prima di montare nel supporto della pipetta.
Nota: Ricordarsi di legare i fili d'argento dell'elettrodo di registrazione e dell'elettrodo di riferimento in candeggina per almeno 30 minuti in modo da formare uno strato uniforme di AgCl sulla superficie del filo. - Trasferire la capsula di Petri nella camera di registrazione e lasciare che la camera con la soluzione NES a 1-2 mL/min. Lasciare che il flusso passa attraverso un riscaldatore in linea impostato a temperatura di 30 ° c al fine di mantenere la soluzione calda.
- Collocare la camera di registrazione elettrofisiologica sotto un microscopio verticale. Registra le correnti della membrana con l'amplificatore patch-clamp e Acquisisci i dati con un software appropriato.
- Aprire il software di controllo dell'amplificatore e impostare il guadagno del segnale al valore 1 e il filtro di Bessel a 10 kHz. Assicurarsi che il filtro di Bessel sia 2,5 volte inferiore alla frequenza di campionamento.
- Impostare i protocolli sperimentali per gli esperimenti di tensione-morsetto e corrente-Clamp nel software di registrazione.
- Nell'impostazione del protocollo, selezionare la modalità di stimolazione episodica e impostare la frequenza di campionamento a 25 kHz. Quindi, passare alla scheda forma d'onda e digitare la tensione o l'ampiezza e la lunghezza del passo corrente come segue.
- Per le correnti di sodio con Gate di tensione, utilizzare 15 gradini di tensione (50 ms di durata ciascuno) da-100 mV a + 40 mV (incrementi di 10 mV). Eseguire il protocollo dopo aver imposto alla cella patchato un potenziale di detenzione di-60 mV attraverso l'amplificatore. Analogamente, le correnti di potassio in tensione sono evocate da passi di tensione (250 ms di durata ciascuno) da-30 mV a + 50 mV (incrementi di 10 mV) che tengono la cella registrata a-40 mV.
- Per indagare le proprietà di cottura degli MNs craniali e spinali di iPSC, le celle di serraggio, in modalità di corrente-pinza, ad un potenziale di membrana di-70 mV e utilizzare 4 impulsi di corrente (1 s durata ciascuno) di aumentare l'ampiezza (da + 20 pA a + 80 PA; 20 pA incremento).
- Acquisisci per ogni corrente attivata dalla tensione delle celle, attività di cottura evocata e tre proprietà passive come capacità dell'intera cellula (cm), resistenza della membrana cellulare (RM) e potenziale di membrana di riposo (RMP).
Risultati
Una descrizione schematica del metodo di differenziazione è illustrata nella Figura 1. Gli iPSCs umani (WT I linea3) sono stati trasfusati con EPB-BSD-TT-Nil o EPB-BSD-TT-NIP, generando, dopo la selezione di blasticidina, linee cellulari stabili e inducibili12, di seguito denominate IPSC-nil e IPSC-NIP, rispettivamente. Differenziando le cellule sono state caratterizzate per l'espressione del marcatore pluripotenza Oct4 e il marcatore Pan-neuronale TUJ1. L'analisi immunostaining ha mostrato un'espressione uniforme di OCT4 in tutte le cellule al giorno 0, in assenza di positività TUJ1 (Figura 2a). Al giorno 3, abbiamo osservato una forte diminuzione del numero di cellule OCT4 positive, rispecchiate dall'espressione di TUJ1 in un sottoinsieme di iPSCs differenzianti (Figura 2B). Al giorno 5, nessuna espressione di OCT4 è stata osservata nella popolazione, che ha mostrato espressione coerente di TUJ1 e ha acquisito una morfologia neuronale (Figura 2C). I progenitori del motoneuron sono stati poi dissociati e riplaccati per un'ulteriore maturazione. Dopo 7 giorni (12 giorni dal giorno 0), le cellule espresse uniformemente TUJ1 e il marcatore del neurone motore maturo CHAT (Figura 3). Risultati simili sono stati ottenuti con due ulteriori linee commerciali iPSC (Vedi tabella dei materiali), utilizzando le stesse condizioni di coltura e differenziazione (Figura complementare S1 e Figura supplementare S2). Analogamente ai neuroni motori indotti da NIL da ESCs del topo11, gli IPSC umani indotti con nil e NIP hanno espresso bassi livelli di fattori di trascrizione Hox e possono essere modellati lungo l'asse rostro-caudale con acido retinoico (Figura S3 supplementare ).
Questi risultati sono stati ottenuti quando 62.500 cellule/cm2 sono state seminate all'inizio della differenziazione e indotte con 1 μm di doxiciclina al giorno 0. Ciò ha provocato la concentrazione ottimale di doxiciclina per ottenere la massima induzione dei transgeni senza tossicità evidente per le cellule (Figura supplementare S4A). Dopo la rimozione di doxiciclina al giorno 5, l'espressione dei transgeni è stata silenziata (Figura supplementare s4b). Abbiamo anche eseguito esperimenti pilota per stabilire la densità ottimale delle cellule a questo punto del protocollo. Abbiamo notato che variando questo parametro negli esperimenti di differenziazione parallela ha fornito risultati diversi. Con la densità iniziale abbassata a 31.250 cellule/cm2, la differenziazione era apparentemente normale, come valutato osservando la morfologia cellulare. Tuttavia, abbiamo notato resistenza alla dissociazione al giorno 5 (sezione 3,4) e ridotta redditività nella successiva fase di maturazione. Viceversa, quando la densità iniziale delle cellule è stata aumentata a 125.000 cellule/cm2, abbiamo osservato una differenziazione inefficiente, come valutato dalla mancanza di acquisizione della tipica morfologia Neuron-like. Ciò ha provocato una popolazione mista contenente solo una frazione minore di MNs, che avrebbe bisogno di ulteriore purificazione (ad esempio, da FACS). Abbiamo quindi stabilito che la densità ottimale per ottenere una popolazione pura di cellule neuronali, in grado di sopravvivere nella cultura per più di due mesi, è 62.500 cellule/cm2.
Abbiamo quindi valutato la maturazione funzionale dei neuroni del motore spinale derivato da iPSC NIL, caratterizzando le loro proprietà elettrofisiologiche (Figura 4), come precedentemente riportato per i neuroni del motore craniale di IPSC NIP-derivato12. Le registrazioni del morsetto di patch, in modalità di tensione e di corrente, sono state eseguite al giorno 7 della fase di maturazione degli MNs del protocollo (vedere Figura 1; tempo totale di differenziazione: 12 giorni) (Figura 4A). In questo momento, i neuroni del motore iPSC NIL hanno mostrato un potenziale di membrana di riposo leggermente inferiore (-30 ± 2 mV; n = 24) e un valore di capacitanza cellulare simile (+ 25 ± 2 pF; n = 25) rispetto agli MNs12di IPSC precedentemente segnalati. Poi, per caratterizzare più profondamente il grado di maturazione delle cellule differenziate, abbiamo indagato la loro capacità di evocare le correnti di sodio e potassio quando stimolato con una serie di impulsi di tensione. In questi esperimenti, i neuroni IPSC Nil hanno mostrato con successo le correnti di sodio dipendenti dalla tensione (Figura 4B) e le correnti di potassio dipendenti dalla tensione (Figura 4C), raggiungendo l'ampiezza di picco quando sono bloccate a un potenziale di membrana vicino a − 20 mV e + 50 mV, rispettivamente. I potenziali di equilibrio per na+ e K+, calcolati utilizzando l'equazione di nernst (www.physiologyweb.com/calculators/nernst_potential_calculator.html) con le soluzioni extracellulari e intracellulari precedentemente segnalate, erano + 110 mV e-102 mV rispettivamente. Inoltre, il 80% degli MNs iPSC NIL-derivati bloccati nella modalità di corrente-clamp sono stati in grado di innescare treni Spike quando iniettato con un impulso di corrente di + 60 pA o più (Figura 4D). La corrente minima necessaria per suscitare una cottura ripetitiva in più del 50% delle cellule registrate era + 40 pA (15 su 18 cellule; Figura 4 E). soglia Spike era-37,6 ± 0,8 MV e frequenza di cottura media a + 40 PA era di circa 7,9 ± 2,2 Hz (n = 18; Figura 4 F).
Nel complesso, questi dati suggeriscono che, analogamente a quanto riferito in precedenza agli MNs cranici di iPSC NIP12, gli MNS spinali derivati da IPSC-Nil hanno proprietà funzionali tipiche dei neuroni maturi.

Figura 1 : Protocollo di differenziazione del neurone del motore. La figura mostra una rappresentazione schematica del protocollo di differenziazione, dalla generazione di linee iPSC stabili con i vettori piggyBac al punto temporale dell'analisi funzionale riportata nel testo. Vengono mostrate le immagini di contrasto di fase rappresentative delle celle in diverse fasi del protocollo. Barre di scala = 50 μm. Si prega di cliccare qui per visualizzare una versione più grande di questa cifra.
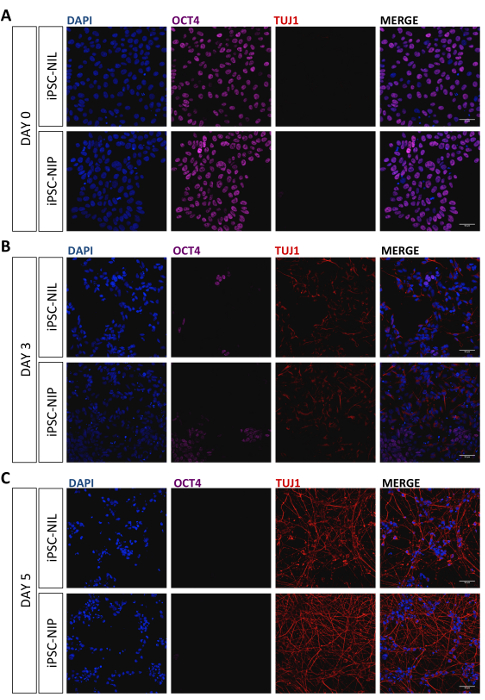
Figura 2 : Analisi immunocolorazione rappresentativa delle cellule differenzianti. le cellule IPSC-nil e IPSC-nip sono state analizzate mediante immunocolorazione per l'espressione del marcatore pluripotenza Oct4 (viola) e del marcatore Pan-neuronale TUJ1 (rosso) al giorno 0 (A), giorno 3 (B) e giorno 5 (C). I nuclei sono stati controcolorati con DAPI. Le immagini confocale sono state acquisite al microscopio confocale a scansione laser (vedere la tabella dei materiali) utilizzando un obiettivo 20x na 0,75 con zoom 2x, 1024 x 1024 pixel, dotato di 405 nm, 473 nm, 559 nm e 635 nm laser. Sono state utilizzate le impostazioni dei filtri per DAPI, Alexa Fluor 594 e Alexa Fluor 647. Barre di scala = 50 μm. Si prega di cliccare qui per visualizzare una versione più grande di questa cifra.

Figura 3 : Analisi immunocolorazione rappresentativa dei motoneuroni di origine IPSC. le cellule IPSC-nil e IPSC-nip sono state analizzate da immunocolorazione per l'espressione del marcatore Pan-neuronale TUJ1 (rosso) e il marcatore del neurone del motore chat (colina acetiltransferasi; verde) al giorno 12. I nuclei sono stati controcolorati con DAPI. Le immagini confocale sono state acquisite come descritto nella Figura 2 legenda. Barre di scala = 50 μm. Si prega di cliccare qui per visualizzare una versione più grande di questa cifra.
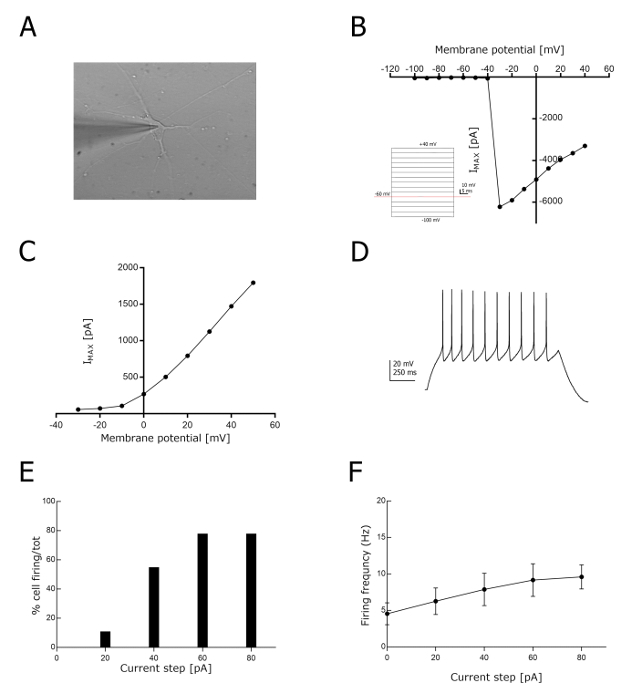
Figura 4 : Analisi funzionale dei neuroni del motore spinale e craniale. (A) campo luminoso immagine del morsetto di patch a cellula intera su neurone del motore spinale derivato da IPSC Nil. B) curva I/V rappresentativa per na+ corrente registrata in MNS derivati da IPSC Nil in risposta a una serie di incrementi di tensione crescenti (n = 26; potenziale di mantenimento pari a-60 MV). C) curva I/V rappresentativa per K+ registrata in MNS derivati da IPSC Nil in risposta a una serie di incrementi di tensione crescenti (n = 23; potenziale di mantenimento pari a-40 mV). (D) traccia rappresentativa di un treno di potenziali d'azione evocato in risposta ad un'iniezione di corrente di 1 s di + 60 PA. (E) istogramma che rappresenta la percentuale di MN derivato da IPSC che suscitano potenziali d'azione ad ogni impulso di corrente (n = 18). (F) istogramma che mostra la frequenza di cottura evocata ad ogni impulso di corrente (n = 18). La registrazione elettrofisiologica è stata eseguita sotto un microscopio verticale. Il sistema di registrazione delle correnti a membrana è indicato nella tabella dei materiali. Si prega di cliccare qui per visualizzare una versione più grande di questa cifra.
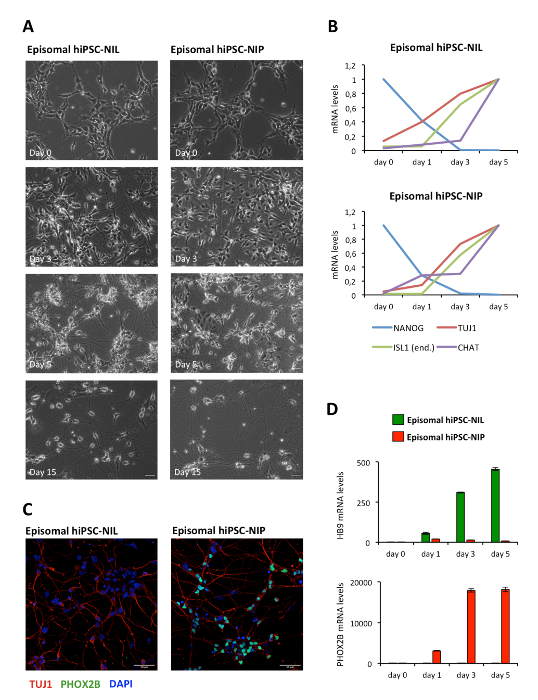
Figura complementare S1: differenziazione MN con hiPSCs episomiale. (A) campo chiaro immagini di differenziazione episomiale HIPSC-Nil (a sinistra) e episomiale HIPSC-NIP (a destra) nei punti temporali indicati. Barre di scala = 50 μm. B) analisi dell'espressione dei marcatori indicati nella differenziazione delle cellule Episomiali HIPSC-Nil (superiore) e episomica HIPSC-NIP (inferiore) in tempo reale QRT-PCR. Per ogni marcatore, il punto temporale con l'espressione più alta è stato utilizzato come campione di calibratore. I primer utilizzati per ISL1 sono specifici per il gene endogeno. C) immunostaining per il marcatore Pan-neuronale TUJ1 (rosso) e il marcatore MN cranico Phox2B (verde) in cellule differenziate (giorno 6) episomiale HIPSC-Nil (sinistra) e episomiale HIPSC-NIP (a destra). I nuclei sono controcolorati con DAPI. Barra di scala per tutti i pannelli: 50 μm. D) analisi dell'espressione di HB9 e Phox2B nella differenziazione delle cellule Episomiali HIPSC-Nil (superiore) e episomica HIPSC-NIP (inferiore) in tempo reale QRT-PCR. Il giorno 0 è stato utilizzato come campione del calibratore. I primer e i metodi della PCR sono riportati in de santis et al., 201812. Si prega di cliccare qui per visualizzare una versione più grande di questa cifra.
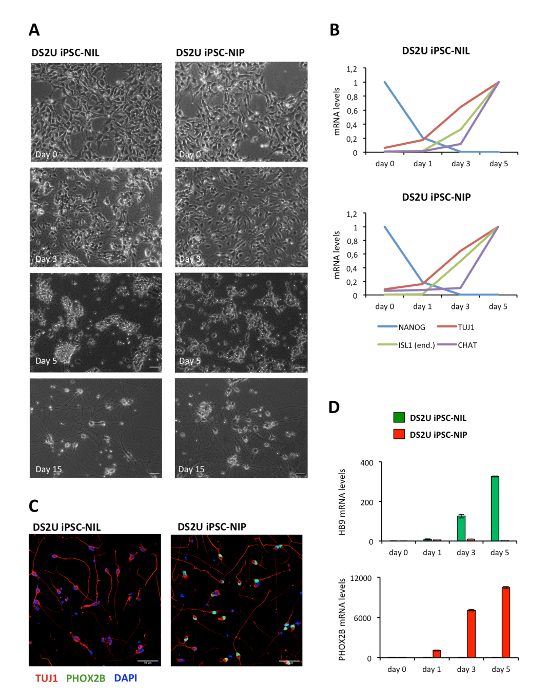
Figura supplementare S2: differenziazione MN utilizzando DS2U iPSCs. (A) campo chiaro immagini di differenziare DS2U-Nil (sinistra) e DS2U-NIP (destra) nei punti temporali indicati. Barre di scala = 50 μm. (B) analisi dell'espressione dei marcatori indicati nella differenziazione delle cellule DS2U-Nil (superiore) e DS2U-NIP (inferiore) in tempo reale QRT-PCR. Per ogni marcatore il punto temporale con l'espressione più alta è stato utilizzato come campione di calibratore. I primer utilizzati per ISL1 sono specifici per il gene endogeno. C) immunostaining per il marcatore Pan-neuronale TUJ1 (rosso) e il marcatore MN craniale Phox2B (verde) in cellule differenziate (giorno 6) DS2U-Nil (sinistra) e DS2U-NIP (destra). I nuclei sono controcolorati con DAPI. Barre di scala = 50 μm. (D) analisi dell'espressione di HB9 e Phox2B nella differenziazione delle celle DS2U-Nil (superiore) e DS2U-NIP (inferiore) in tempo reale QRT-PCR. Il giorno 0 è stato utilizzato come campione del calibratore. I primer e i metodi della PCR sono riportati in de santis et al., 201812. Si prega di cliccare qui per visualizzare una versione più grande di questa cifra.

Figura complementare S3: espressione genica HOX. (A) analisi dell'espressione di quattro diversi geni Hox (Hox a2, Hox B1, Hox a4, Hox B5) dopo 5 giorni di differenziazione delle cellule IPSC-nil e IPSC-Nil + ra in tempo reale QRT-PCR. IPSC-NIL al giorno 0 è stato utilizzato come campione del calibratore. (B) analisi dell'espressione di quattro diversi geni Hox (Hox a2, Hox B1, Hox a4, Hox B5) dopo 5 giorni di differenziazione delle cellule IPSC-NIP e IPSC-NIP + ra in tempo reale QRT-PCR. IPSC-NIP al giorno 0 è stato utilizzato come campione di calibratore. C) coppie di primer per PCR. Si prega di cliccare qui per visualizzare una versione più grande di questa cifra.
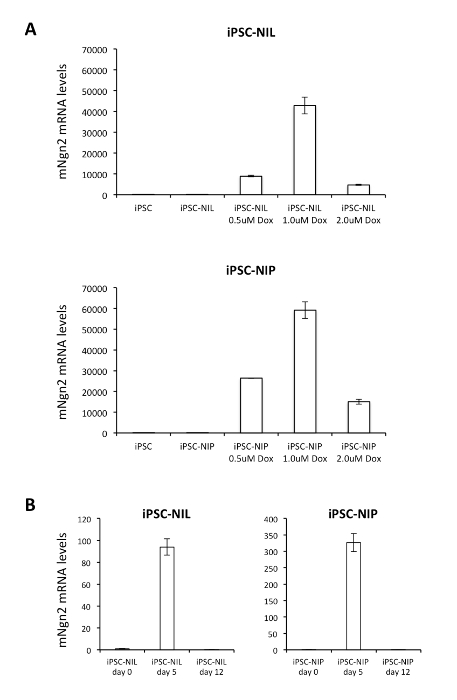
Figura complementare S4: analisi di induzione della doxiciclina. (A) analisi in tempo reale QRT-PCR dell'espressione di Ngn2 esogeno in IPSC-Nil (in alto) e IPSC-NIP (Bottom) cellule non trattate o coltivate per 24 h in presenza di doxiciclina a diversa concentrazione (0,5 μm, 1,0 μm, 2,0 μm). Ngn2 è stato analizzato con primer specifici per il gene del topo esogeno. La linea di iPSC parentale, priva di costrutti NIL e NIP, è stata inclusa nell'analisi come un controllo. L'espressione dei transgeni in iPSC-NIL e iPSC-NIP è stata trascurabile in assenza di doxiciclina. iPSC-NIL e iPSC-NIP al giorno 0 sono stati utilizzati come campioni di calibratore. (B) analisi in tempo reale QRT-PCR dell'espressione di Ngn2 esogena nella differenziazione delle cellule IPSC-Nil (sinistra) e IPSC-NIP (destra) nei punti temporali indicati del protocollo. Ngn2 è stato analizzato con primer specifici per il gene del topo esogeno. iPSC-NIL e iPSC-NIP al giorno 0 sono stati utilizzati come campioni di calibratore. I primer e i metodi della PCR sono riportati in de santis et al., 201812. Si prega di cliccare qui per visualizzare una versione più grande di questa cifra.
Discussione
Questo protocollo consente di convertire efficacemente gli IPSC umani in neuroni motori spinali e cranici grazie all'espressione ectopica di fattori di trascrizione specifici per lignaggio. Questi transgeni sono inducibili dalla doxiciclina e stabilmente integrati nel genoma grazie a un vettore basato su trasposti piggyBac. In una popolazione mista, una o più copie del vettore piggyBac saranno integrate in modo casuale nel genoma delle singole cellule, aumentando il rischio di alterazioni dell'integrità del genoma. Inoltre, una selezione progressiva dei subcloni di iPSC può avvenire nel tempo, con possibili conseguenze per la differenziazione e per l'analisi comparativa delle malattie e delle linee cellulari di controllo. Complessivamente, gli MNs derivati da iPSC ottenuti con questo protocollo non saranno adatti alla medicina rigenerativa. Tuttavia, il nostro metodo potrebbe essere particolarmente utile per la modellazione in vitro della malattia dei neuroni del motore. I principali punti di forza sono rappresentati dalle condizioni di coltura estremamente semplificate, dalla rapidità della conversione MN, dall'alto grado di maturazione degli MNs derivati dall'iPSC e dalla possibilità di ottenere sia gli MNs spinali che quelli cranici, come precedentemente dimostrato da analisi di specifici geni marker espressione12. La robustezza del protocollo è dimostrata dalla sua riproducibilità. Finora, abbiamo applicato con successo questo protocollo più di 30 volte per la generazione di MN spinale e/o craniale, valutando i risultati per marker e/o analisi funzionali. Abbiamo mantenuto con successo le colture di neuroni del motore per un massimo di due mesi senza evidente diminuzione della redditività. Inoltre, una volta ottenuta la linea iPSC traslabile stabilmente, ogni esperimento può essere avviato dall'induzione della doxiciclina senza la necessità di una nuova trasfezione. Anche i vettori virali non sono necessari. I risultati rappresentativi qui presentati sono stati ottenuti elettroporating iPSCs con il sistema di elettroporazione cellulare indicato nella tabella dei materiali. Tuttavia, altri metodi di elettroporazione potrebbero rappresentare opzioni alternative. Viceversa, nella nostra esperienza, i sistemi di trasfezione basati sulla lipofection non sono una buona opzione per le cellule staminali pluripotenti12. Le popolazioni di cellule ottenute alla fine del processo sono composte quasi esclusivamente di MNs, evitando la necessità di ulteriore purificazione (ad esempio, da FACS). Come abbiamo mostrato in precedenza12, il protocollo permette di ottenere 90% TUJ1-positive cellule, di cui 95% dove anche Phox2B positivo in colture di Nip-derivato.
Possiamo prevedere alcuni punti critici che devono essere accuratamente presi in considerazione. In primo luogo, la qualità della popolazione iniziale di IPSC è cruciale per garantire una conversione omogenea e coerente in MNs. colture contenenti una sostanziale frazione di differenziazione (oltre il 5-10%) deve essere evitato. Abbiamo impostato il protocollo utilizzando il supporto iPSC umano descritto nella tabella dei materiali come mezzo di manutenzione per iPSCs indifferenziati. Altri supporti definiti commercialmente disponibili potrebbero rappresentare opzioni alternative valide, nonostante non abbiamo affrontato sperimentalmente questo punto nel presente lavoro. Poiché la composizione dei media può influenzare il tasso di proliferazione della popolazione cellulare iniziale, l'adattamento del protocollo ad altri supporti di manutenzione potrebbe richiedere l'ottimizzazione della densità iniziale al giorno 0. Dopo la trasfezione, è importante mantenere le cellule sotto selezione antibiotica per almeno 2 settimane per ottenere linee cellulari stabili. Potrebbe essere opportuno che alcune applicazioni derivino linee clonali dopo l'integrazione di piggyBac, al fine di ottenere una popolazione più omogenea in termini di livelli di espressione transgenica e un migliore controllo dei siti di integrazione. La densità delle cellule al giorno 0, il tempo di doxiciclina aggiunta al mezzo, è un parametro cruciale. Come accennato nella sezione risultati rappresentativi, abbiamo stimato una densità ottimale delle cellule per garantire la riproducibilità. Non possiamo escludere che altre linee di cellule staminali pluripotenti potrebbero richiedere una diversa densità iniziale, che dovrebbe essere determinata empiricamente negli esperimenti pilota, poiché il tasso di duplicazione può variare significativamente tra le singole linee. Il passaggio medio a Neurobasal/B27 deve avvenire dopo 48 h dall'induzione della doxiciclina (sezione 3,3): la differenziazione può risultare più lenta e meno efficiente se le cellule non vengono tenute per 2 giorni interi nel supporto DMEM/F12. La dissociazione al giorno 5 deve essere eseguita senza sottolineare le cellule differenzianti. Il tempo di incubazione con il reagente di dissociazione delle cellule (fase 3,4) potrebbe differire da lotto a lotto e deve essere accuratamente stimato in esperimenti pilota. Dopo l'incubazione con il reagente di dissociazione cellulare, l'intero monostrato cellulare si distacca dalla piastra. Quindi, deve essere accuratamente dissociato dal pipettaggio, come descritto nella sezione 3,4, per preservare l'integrità delle cellule e per evitare lo stress meccanico, che potrebbe avere un impatto negativo sul mantenimento della coltura cellulare oltre D5. Infine, abbiamo notato che dopo la ri-placcatura MNs aderire meglio sulla coltura del tessuto plastica rispetto al vetro. I coprioggetti in polimero che assicurano un'aderenza ottimale delle cellule e proprietà ottiche adatte sono una buona opzione per applicazioni basate sulla microscopia.
Il nostro metodo rappresenta una "programmazione" diretta delle cellule pluripotenti in un destino MN, e non ricapitolerà le fasi intermedie attraverso le quali le cellule embrionali acquisiscono un'identità MN durante lo sviluppo in vivo, come la specifica iniziale per l'ectoderma neurale e patterning lungo gli assi dorso-ventrale e rostro-caudale. Pertanto, non sarebbe adatto a modellare la specifica MN umana in vitro per studiare, ad esempio, i meccanismi molecolari alla base della differenziazione. D'altra parte, il nostro protocollo permette di generare una considerevole quantità di MN spinale o craniale senza la necessità di ulteriore purificazione. Prendendo anche in considerazione il grado di maturazione raggiunto, questo rappresenta uno strumento utile per studiare la base molecolare delle malattie neurodegenerative del Neuron motorio. Negli ultimi anni, la comunità scientifica ha prodotto un numero consistente di linee iPSC con mutazioni patogene nei geni correlati alla malattia del motoneuron. Le collezioni di linee iPSC "programmabili" di MN potrebbero quindi essere facilmente generate dall'integrazione stabile dei moduli NIL e NIP in quelle linee iPSC mutanti. Possiamo immaginare, come possibile futura applicazione del metodo, la caratterizzazione dei determinanti cellulari-autonomi che conferiscono una diversa suscettibilità ai singoli sottotipi di MN, confrontando gli MNs cranici e spinali side-by-side ottenuti da iPSCs con lo stesso background genetico. Inoltre, una conversione efficace degli iPSCs in MNs in condizioni di coltura semplificate che necessitano di una manipolazione minima (cioè senza transizione attraverso corpi embrioidi) e che possono facilmente essere scalabili, potrebbe facilitare notevolmente la produzione automatizzata di farmaci ad alto rendimento approcci di screening per le malattie motoneuriche. La modellazione in vitro di malattie neurodegenerative ad esordio adulto può essere impegnativa a causa della natura fetale dei neuroni derivati dall'iPSC14. Le strategie precedenti concepite per accelerare l'invecchiamento degli MNs derivate dalla differenziazione convenzionale degli IPSC, come la sovraespressione di progerin15, potrebbero essere combinate con il nostro metodo per ottenere migliori modelli di malattia.
Il metodo descritto qui, basato sull'espressione inducibile dei fattori di trascrizione mediato da un vettore piggyBac, può essere applicato ad altri tipi di cellule indotte. In precedenza abbiamo dimostrato che le cellule muscolari scheletriche possono essere ottenute da iPSCs umani per espressione di BAF60c e MyoD16. Allo stesso modo, possiamo immaginare la possibilità di estendere il metodo ad altri tipi di cellule di interesse, tra cui altri sottotipi neuronali e astrociti, dall'espressione mediata da piggybac di insiemi appropriati di fattori di programmazione17,18, 19,20,21.
Divulgazioni
Gli autori non hanno nulla da rivelare
Riconoscimenti
Gli autori desiderano ringraziare l'imaging Facility presso Center for Life Nano Science, Istituto Italiano di tecnologia, per il supporto e la consulenza tecnica. Siamo grati ai membri del centro per la vita Nano Science per una discussione utile. Questo lavoro è stato parzialmente sostenuto da una sovvenzione di AriSLA (Pilot Grant 2016 "StressFUS") all'AR.
Materiali
| Name | Company | Catalog Number | Comments |
| 5-Bapta | Sigma-Aldrich | A4926-1G | chemicals for electrophysiological solutions |
| Accutase | Sigma-Aldrich | A6964-100ML | Cell dissociation reagent |
| anti-CHAT | EMD Millipore | AB144P | Anti-Choline Acetyltransferase. Primary antibody used in immunostaining assays. RRID: AB_2079751; Lot number: 2971003 |
| anti-goat Alexa Fluor 488 | Thermo Fisher Scientific | A11055 | Secondary antibody used for immonofluorescence assays. RRID: AB_2534102; Lot number: 1915848 |
| anti-mouse Alexa Fluor 647 | Thermo Fisher Scientific | A31571 | Secondary antibody used for immonofluorescence assays. RRID: AB_162542; Lot number: 1757130 |
| anti-Oct4 | BD Biosciences | 611202 | Primary antibody used in immunostaining assays. RRID: AB_398736; Lot number: 5233722 |
| anti-Phox2b | Santa Cruz Biotechnology, Inc. | sc-376997 | Primary antibody used in immunostaining assays. Lot number: E0117 |
| anti-rabbit Alexa Fluor 594 | Immunological Sciences | IS-20152-1 | Secondary antibody used for immonofluorescence assays |
| anti-TUJ1 | Sigma-Aldrich | T2200 | Primary antibody used in immunostaining assays. RRID: AB_262133 |
| B27 | Miltenyi Biotec | 130-093-566 | Serum free supplement for neuronal cell maintenance |
| Bambanker | Nippon Genetics | NGE-BB02 | Cell freezing medium, used here for motor neuron progenitors |
| BDNF | PreproTech | 450-02 | Brain-Derived Neurotrophic Factor |
| Blasticidin | Sigma-Aldrich | 203350 | Nucleoside antibiotic that inhibits protein synthesis in prokaryotes and eukaryotes |
| BSA | Sigma-Aldrich | A2153 | Bovine Serum Albumin. Blocking agent to prevent non-specific binding of antibodies in immunostaining assays |
| CaCl2 | Sigma-Aldrich | C3881 | chemicals for electrophysiological solutions |
| Clampex 10 software | Molecular Devices | Clampex 10 | Membrane currents recording system |
| Corning Matrigel hESC-qualified Matrix | Corning | 354277 | Reconstituted basement membrane preparation from the Engelbreth-Holm-Swarm (EHS) mouse sarcoma. Used for adhesion of iPSC to plastic and glass supports |
| CRYOSTEM ACF FREEZING MEDIA | Biological Industries | 05-710-1E | Freezing medium for human iPSCs |
| D-Glucose | Sigma-Aldrich | G5146 | chemicals for electrophysiological solutions |
| DAPI powder | Roche | 10236276001 | 4′,6-diamidino-2-phenylindole. Fluorescent stain that binds to adenine–thymine rich regions in DNA used for nuclei staining in immonofluorescence assays |
| DAPT | AdipoGen | AG-CR1-0016-M005 | Gamma secretase inhibitor |
| Dispase | Gibco | 17105-041 | Reagent for gentle dissociation of human iPSCs |
| DMEM/F12 | Sigma-Aldrich | D6421-500ML | Basal medium for cell culture |
| Doxycycline | Sigma-Aldrich | D9891-1G | Used to induce expression of transgenes from epB-Bsd-TT-NIL and epB-Bsd-TT-NIP vectors |
| DS2U | WiCell | UWWC1-DS2U | Commercial human iPSC line |
| E.Z.N.A Total RNA Kit | Omega bio-tek | R6834-02 | Kit for total extraction of RNA from cultured eukaryotic cells |
| GDNF | PreproTech | 450-10 | Glial-Derived Neurotrophic Factor |
| Gibco Episomal hiPSC Line | Thermo Fisher Scientific | A18945 | Commercial human iPSC line |
| Glutamax | Thermo Fisher Scientific | 35050038 | An alternative to L-glutamine with increased stability. Improves cell health. |
| Hepes | Sigma-Aldrich | H4034 | chemicals for electrophysiological solutions |
| iScript Reverse Transcription Supermix for RT-qPCR | Bio-Rad | 1708841 | Kit for gene expression analysis using real-time qPCR |
| iTaqTM Universal SYBR Green Supermix | Bio-Rad | 172-5121 | Ready-to-use reaction master mix optimized for dye-based quantitative PCR (qPCR) on any real-time PCR instrument |
| K-Gluconate | Sigma-Aldrich | G4500 | chemicals for electrophysiological solutions |
| KCl | Sigma-Aldrich | P9333 | chemicals for electrophysiological solutions |
| L-ascorbic acid | LKT Laboratories | A7210 | Used in cell culture as an antioxidant |
| Laminin | Sigma-Aldrich | 11243217001 | Promotes attachment and growth of neural cells in vitro |
| Laser scanning confocal microscope | Olympus | iX83 FluoView1200 | Confocal microscope for acquisition of immunostaining images |
| Mg-ATP | Sigma-Aldrich | A9187 | chemicals for electrophysiological solutions |
| MgCl2 | Sigma-Aldrich | M8266 | chemicals for electrophysiological solutions |
| Mounting Medium | Ibidi | 50001 | Mounting solution used for confocal microscopy and immunofluorescence assays |
| Multiclamp patch-clamp amplifier | Molecular Devices | 700B | Membrane currents recording system |
| Na-GTP | Sigma-Aldrich | G8877 | chemicals for electrophysiological solutions |
| NaCl | Sigma-Aldrich | 71376 | chemicals for electrophysiological solutions |
| NEAA | Thermo Fisher Scientific | 11140035 | Non-Essential Amino Acids. Used as a supplement for cell culture medium, to increase cell growth and viability. |
| Neon 100 μL Kit | Thermo Fisher Scientific | MPK10096 | Cell electroporation kit |
| Neon Transfection System | Thermo Fisher Scientific | MPK5000 | Cell electroporation system |
| Neurobasal Medium | Thermo Fisher Scientific | 21103049 | Basal medium designed for long-term maintenance and maturation of neuronal cell populations without the need for an astrocyte feeder layer |
| NutriStem-XF/FF | Biological Industries | 05-100-1A | Human iPSC culture medium |
| Paraformaldehyde | Electron Microscopy Sciences | 157-8 | Used for cell fixation in immunostaining assays |
| PBS | Sigma-Aldrich | D8662-500ML | Dulbecco's Phosphate Buffer Saline, with Calcium, with Magnesium |
| PBS Ca2+/Mg2+ free | Sigma-Aldrich | D8537-500ML | Dulbecco's Phosphate Buffer Saline, w/o Calcium, w/o Magnesium |
| Penicillin/Streptomycin | Sigma-Aldrich | P4333-100ML | Penicillin/Streptomicin solution used to prevent cell culture contamination from bacteria. |
| poly-ornithine | Sigma-Aldrich | P4957 | Promotes attachment and growth of neural cells in vitro |
| SU5402 | Sigma-Aldrich | SML0443-5MG | Selective inhibitor of vascular endothelial growth factor receptor 2 (VEGFR-2) |
| Triton X-100 | Sigma-Aldrich | T8787 | 4-(1,1,3,3-Tetramethylbutyl)phenyl-polyethylene glycol, t-Octylphenoxypolyethoxyethanol, Polyethylene glycol tert-octylphenyl ether. Used for cell permeabilization in immunostaining assays |
| Upright microscope | Olympus | BX51VI | Microscope for electrophysiological recording equipped with CoolSnap Myo camera |
| Y-27632 (ROCK inhibitor) | Enzo Life Sciences | ALX-270-333-M005 | Cell-permeable selective inhibitor of Rho-associated, coiled-coil containing protein kinase (ROCK). Increases iPSC survival |
| μ-Slide 8 Well | Ibidi | 80826 | Support for high–end microscopic analysis of fixed cells |
Riferimenti
- Dimos, J. T., et al. Induced Pluripotent Stem Cells Generated from Patients with ALS Can Be Differentiated into Motor Neurons. Science (New York, NY). 321 (5893), 1218 (2008).
- Ebert, A. D., et al. Induced pluripotent stem cells from a spinal muscular atrophy patient. Nature. 457 (7227), 277-280 (2009).
- Lenzi, J., et al. ALS mutant FUS proteins are recruited into stress granules in induced Pluripotent Stem Cells (iPSCs) derived motoneurons. Disease models & mechanisms. 8 (7), 755-766 (2015).
- Wijesekera, L. C., Leigh, P. N. Amyotrophic lateral sclerosis. Orphanet journal of rare diseases. 4 (3), 1-22 (2009).
- Yan, J., et al. Frameshift and novel mutations in FUS in familial amyotrophic lateral sclerosis and ALS/dementia. Neurology. 75 (9), 807-814 (2010).
- Boulting, G. L., et al. A functionally characterized test set of human induced pluripotent stem cells. Nature biotechnology. 29 (3), 279-286 (2011).
- Amoroso, M. W., et al. Accelerated high-yield generation of limb-innervating motor neurons from human stem cells. The Journal of neuroscience: the official journal of the Society for Neuroscience. 33 (2), 574-586 (2013).
- Maury, Y., et al. Combinatorial analysis of developmental cues efficiently converts human pluripotent stem cells into multiple neuronal subtypes. Nature biotechnology. 33 (1), 89-96 (2015).
- Allodi, I., et al. Differential neuronal vulnerability identifies IGF-2 as a protective factor in ALS. Scientific reports. 6, 25960 (2016).
- Kaplan, A., et al. Neuronal matrix metalloproteinase-9 is a determinant of selective neurodegeneration. Neuron. 81 (2), 333-348 (2014).
- Mazzoni, E. O., et al. Synergistic binding of transcription factors to cell-specific enhancers programs motor neuron identity. Nature neuroscience. 16 (9), 1219-1227 (2013).
- De Santis, R., Garone, M. G., Pagani, F., de Turris, V., Di Angelantonio, S., Rosa, A. Direct conversion of human pluripotent stem cells into cranial motor neurons using a piggyBac vector. Stem Cell Research. 29, 189-196 (2018).
- Yusa, K., Zhou, L., Li, M. A., Bradley, A., Craig, N. L. A hyperactive piggyBac transposase for mammalian applications. Proceedings of the National Academy of Sciences of the United States of America. 108 (4), 1531-1536 (2011).
- Sances, S., et al. Modeling ALS with motor neurons derived from human induced pluripotent stem cells. Nature Neuroscience. 16 (4), 542-553 (2016).
- Miller, J. D., et al. Human iPSC-based modeling of late-onset disease via progerin-induced aging. Cell stem cell. 13 (6), 691-705 (2013).
- Lenzi, J., et al. Differentiation of control and ALS mutant human iPSCs into functional skeletal muscle cells, a tool for the study of neuromuscolar diseases. Stem Cell Research. 17 (1), 140-147 (2016).
- Zhang, Y., et al. Rapid Single-Step Induction of Functional Neurons from Human Pluripotent Stem Cells. Neuron. 78 (5), 785-798 (2013).
- Theka, I., et al. Rapid generation of functional dopaminergic neurons from human induced pluripotent stem cells through a single-step procedure using cell lineage transcription factors. Stem cells translational medicine. 2 (6), 473-479 (2013).
- Pawlowski, M., et al. Inducible and Deterministic Forward Programming of Human Pluripotent Stem Cells into Neurons, Skeletal Myocytes, and Oligodendrocytes. Stem Cell Reports. 8 (4), 803-812 (2017).
- Li, X., et al. Fast Generation of Functional Subtype Astrocytes from Human Pluripotent Stem Cells. Stem Cell Reports. 11 (4), 998-1008 (2018).
- Nehme, R., et al. Combining NGN2 Programming with Developmental Patterning Generates Human Excitatory Neurons with NMDAR-Mediated Synaptic Transmission. Cell reports. 23 (8), 2509-2523 (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon