Method Article
3D ultrastruttura mitocondriale del muscolo di voli indiretti di Drosophila ha rivelato tramite tomografia dell'elettrone Serial-sezione
In questo articolo
Riepilogo
In questo protocollo, dimostriamo l'applicazione della tomografia elettronica di serie-sezione per delucidare la struttura mitocondriale nel muscolo di Drosophila voli indiretti.
Abstract
I mitocondri sono le centrali elettriche cellulari che producono ATP, i lipidi ed i metaboliti, come pure regolano la morte delle cellule e l'omeostasi del calcio. L'ultrastruttura di unico ricco di cristae doppia membrana di questo organello è elegantemente disposti a svolgere molteplici funzioni di partizionamento biomolecole. Ultrastruttura mitocondriale è intimamente collegato con varie funzioni; Tuttavia, i dettagli di queste relazioni struttura-funzione stanno solo cominciando a essere descritto. Qui, noi dimostrare l'applicazione della tomografia elettronica di serie-sezione per delucidare la struttura mitocondriale nel muscolo di Drosophila voli indiretti. Tomografia elettronica di serie-sezione può essere adattata a studiare qualsiasi struttura cellulare in tre dimensioni.
Introduzione
La microscopia elettronica è uno strumento prezioso per studiare il contesto strutturale delle assemblee subcellulare e organelli che eseguono processi cellulari. Sono stati sviluppati metodi per preservare l'ultrastruttura dei tessuti o delle cellule sia tramite la fissazione chimica con aldeidi o ad alta pressione congelamento (HPF) seguita da congelare sostituzione (FS)1,2. I blocchi campione incorporato possono essere sezionati, macchiati e osservati con un microscopio elettronico a trasmissione (TEM). L'esemplare di HPF poteva essere trattato anche sotto cryo-condizione, come da cryo-sezionamento o da fascio ionico focalizzato (FIB) fresatura e osservata da cryo-EM3,4.
Anche se sottile-sezione EM fornisce le comprensioni morfologiche informativi, le immagini 2D risultanti solo possono rivelare l'ultrastruttura di una particolare sezione. Come ultrastruttura è organizzato in un volume 3D rimane oscurato. Al fine di visualizzare l'ultrastruttura cellulare in tre dimensioni, un metodo di tomografia dell'elettrone è stato sviluppato dove serie di immagini di inclinazione sono stata acquisita e proiettata indietro per generare una ricostruzione tomografica5 (Figura 1). Possono essere raccolti una serie doppia inclinazione ruotando il campione di 90° e l'acquisizione di una seconda serie di inclinazione. Questo ridurrà mancante-Cuneo artefatti che derivano da angoli di campione limitato e migliorare la risoluzione del tomogramma.
Qui, descriviamo l'applicazione della tomografia elettronica di serie-sezione per studiare l'ultrastruttura mitocondriale di Drosophila voli indiretti del muscolo (IFM)6,7,8,9 . Al fine di ottenere ricostruzioni 3D che coprono i mitocondri intero (circa 2,5 µm di spessore), sezioni di serie sono state ottenute da blocchi di tessuto IFM di Drosophila . I tomogrammi di ogni sezione sono stati raccolti singolarmente utilizzando software di raccolta dati automatica. Ricostruzioni tomografiche sono stati generati e tomogrammi seriali si unirono con il pacchetto IMOD per ottenere un volume ricostruito di un mitocondrio intero. I tomogrammi riuniti sono stati analizzati dal software 3D. Le densità di cristae mitocondriali sono state segmentate per generare un modello di segmentazione che ha rivelato l'organizzazione in tre dimensioni.
Protocollo
1. sezione della drosofila tessuti utilizzando un microtomo lama vibrante
- Anestetizzare Drosophila sul ghiaccio e immergere ogni singolo fly in 1 mL di agarosio fusione basso al 4% in tampone fosfato. Consentire l'agarosio solidificare il ghiaccio. In genere 4-6 mosche sono stati elaborati.
- Utilizzare un microtomo lama vibrante per sezione del gel dell'agarosi incorporato della drosofila in fette con spessore di 100 µm e immergere in soluzione fissante contenenti glutaraldeide al 2,5% in tampone fosfato 0,1 M.
Nota: Vibratome sezionamento è preferito perché l'architettura del tessuto rimane più intatta rispetto ad altri metodi. In alternativa, pinzette di dissezione può essere utilizzato per sezionare IFM in soluzione fissante contenenti glutaraldeide al 2,5% in tampone fosfato 0,1 M.
2. preparare gli esemplari di EM del congelamento ad alta pressione e il metodo di sostituzione (HPF/FS) Freeze
- Lavare le sezioni di tessuto in 3 gocce (~ 150 µ l) di tampone fosfato, seguita da 2 gocce (~ 100 µ l) di tampone fosfato con BSA 20%. Quindi inserire sezioni in oro vettori per HPF riempito con buffer e 20% BSA.
- Caricare il campione contenente i vettori in un congelatore ad alta pressione secondo il manuale dell'utente.
- Dopo il congelamento, rilasciare i vettori da parte del titolare sotto azoto liquido e trasferire su un dispositivo di blocco-sostituzione raffreddato a-140 ° C.
- Eseguire il protocollo di congelamento-sostituzione come illustrato nella tabella 1, con le FS cocktail contenenti glutaraldeide 2%, 2% tetrossido di osmio e 0,1% acetato di uranile in acetone.
- Con attenzione rimuovere i campioni da parte dei vettori con un ago e incorporare gli esemplari in resina a temperatura ambiente. Polimerizzare la resina a 65 ° C per 16 h.
Nota: Protocolli di FS dovrebbero essere modificate per preparare altri tipi di campioni. HPF/FS è preferito preservare l'ultrastruttura e ridurre al minimo la perdita di contenuto cellulare.- In alternativa, applicare un protocollo di fissazione chimica. Difficoltà esemplari con glutaraldeide 2,5% durante la notte, lavare con buffer e poi fissare con tetrossido di osmio 1% per 2 h. lavaggio e disidratare con ascendente concentrazioni di etanolo e quindi infiltrarsi e incorporare gli esemplari in resina di Spurr prima polimerizzazione a 65 ° C per 16 h.
3. preparare serie-sezioni degli esemplari per tomografia elettronica
- Tagliare i blocchi campione per esporre la faccia di blocco desiderato che contiene il tessuto.
- Pretrattare le particelle d'oro (10 nm di diametro) con BSA 1% per 30 min. Wash e sospendere le particelle d'oro in tampone PBS. Sovrapposizione di particelle d'oro sulle griglie di rame slot rivestite con pellicola di carbonio per creare marker fiduciali.
- Controllare sotto TEM di avere sufficienti fiduciali indicatori (indicatori di almeno 5-10) nel campo di vista dell'acquisizione di tomografia.
- Tagliare le sezioni di serie, 200-250 nm di spessore, utilizzando un ultramicrotomo.
- Raccogliere le sezioni di serie sulle griglie di slot utilizzando un ciclo perfetto per sezioni sottili.
- Macchia le sezioni con citrato di piombo di Reynold per 10 min.
- Sovrapporre un secondo strato di particelle di oro fiduciale sulla parte superiore le sezioni.
4. raccogliere tomografia elettronica doppia inclinazione
- Caricare la griglia su un supporto di doppio-asse tomografia e inserire il microscopio elettronico a trasmissione operanti a 200 kV.
- Allineare il microscopio al fuoco eucentric.
- Installare il software di raccolta automatica dei dati. Regolare e allineare il fascio di elettroni con l'impostazione di imaging multi-scala. Fare riferimento al manuale utente per operazione dettaglio10 (Figura 2).
- Acquisire riferimenti di scuro e brillante di fotocamera in un'area vuota senza pellicola di carbonio sotto l'impostazione di collezione di tomografia.
- Raccogliere un Atlante di griglia a basso ingrandimento. Selezionare i mitocondri su sezioni seriali come obiettivi per la raccolta di tomografia.
- Acquisire una serie di inclinazione da-60 ° a + 60 ° con incrementi di 2 ° su asse-A per ogni destinazione.
Nota: Gli angoli di inclinazione saranno meccanicamente vincolati dal design del portapreparato. Il titolare si bloccherà il fascio ad alta inclinazione angoli. - Per raccogliere la seconda serie di inclinazione, ruotare il portacampioni 90°. Acquisire un nuovo Atlante. Selezionare posizioni corrispondenti e acquisire il tilt series su asse-B per ogni destinazione.
Nota: Altri pacchetti software, ad esempio SerialEM e Xplore3D, sono disponibili per la raccolta automatica dei dati.
5. ricostruire tomogrammi 3D e segmento sub-volumi utilizzando il Software
- Ricostruire i tomogrammi di doppio-asse inclinazione immagini usando IMOD software11.
- Fare riferimento al manuale utente per i dettagli di funzionamento.
- Allineare la serie di inclinazione individuale dalle posizioni di marker fiduciali oro. Ricostruire i tomogrammi dal metodo simultaneo iterativo ricostruzione tecnica (SIRT) oppure il metodo indietro proiezione per entrambi asse-A e asse-B, rispettivamente (Figura 3).
- Combinare tomogrammi di asse A e asse-B per generare un tomogramma double-tilt con ridotta mancante artefatto di Cuneo.
- Unire insieme i tomogrammi double-inclinazione delle sezioni di serie per ottenere ricostruzioni che copre il volume intero mitocondrio. Modello i divari tra le sezioni di serie dal programma IMOD.
- Tagliare le tomogrammi riuniti e bin una dimensione desiderata per la segmentazione di volume.
- Analizzare riuniti tomogrammi seriali usando un software 3D.
- Fare riferimento al manuale utente per i dettagli di funzionamento.
- Filtrare i tomogrammi con un filtro gaussiano (o altro metodo desiderato) per migliorare il contrasto delle caratteristiche e ridurre la densità di sfondo.
- Segmentare l'ultrastruttura dei mitocondri sia manualmente che automaticamente.
- Visualizzare modelli di segmentazione per consentire l'ispezione in 3D.
- Generare filmati utilizzando strumenti disponibili in 3D.
Risultati
Abbiamo applicato la tomografia elettronica di serie-sezione per analizzare le caratteristiche strutturali dei cristae mitocondriali che riflette il suo stato energetico e invecchiamento. Abbiamo mostrato che i mitocondri di Drosophila IFM formano un sistema integrato di rete i cristae e matrice in 3D (Figura 4)7. Inoltre, mosche mutanti con difetto di replica del DNA mitocondriale e un fenotipo di invecchiamento accelerato accumulato i mitocondri che conteneva sottosezioni della cipolla-come vorticoso nucleo (Figura 5)7.

Figura 1: illustrazione della ricostruzione di tomografia di elettroni da una serie di tilt. Nell'esperimento, un fascio di elettroni viene passato attraverso un oggetto 3D quando l'oggetto viene inclinato ai vari gradi. Per ogni condizione di inclinazione, una proiezione 2D è generata e catturata con una macchina fotografica. Le proiezioni 2D quindi vengono proiettate indietro per ricostruire l'oggetto 3D basato sul Teorema centrale fetta. Nella figura, lo stesso processo è indicato per un'immagine 2D originale che viene proiettata in una dimensione unica sotto vari angoli di inclinazione. Le proiezioni di 1D vengono quindi utilizzate per ricostruire l'immagine 2D. Clicca qui per visualizzare una versione più grande di questa figura.
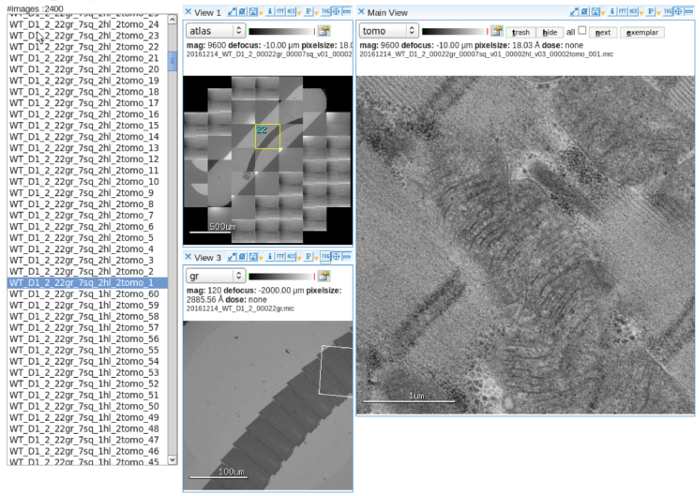
Figura 2: raccolta automatica dei dati. Un display Visualizzatore di immagine del software mostrando l'Atlante di una griglia di slot contenente sezioni di serie, multi-scala targeting dei mitocondri e acquisito inclina immagini. Barra della scala = 500 µm (pannello superiore sinistro), 100 µm (pannello in basso a sinistra), 1 µm (pannello di destra). Clicca qui per visualizzare una versione più grande di questa figura.

Figura 3: Tomografia elettronica di serie-sezione di un singolo mitocondrio nel muscolo di Drosophila voli indiretti. Micrografie 2D (A) e (B) tomogrammi 3D da sezioni di serie che coprono l'intero volume di un mitocondrio. (C), la serie-sezione riunita tomogrammi vengono proiettati per creare una sezione longitudinale, con l'asse z mostrato verticalmente. In particolare, tessuto di sezionamento ha portato alla perdita di materiale, lasciando spazi vuoti tra i tomogrammi Unite in join. Barra della scala = 500 nm. Clicca qui per visualizzare una versione più grande di questa figura.
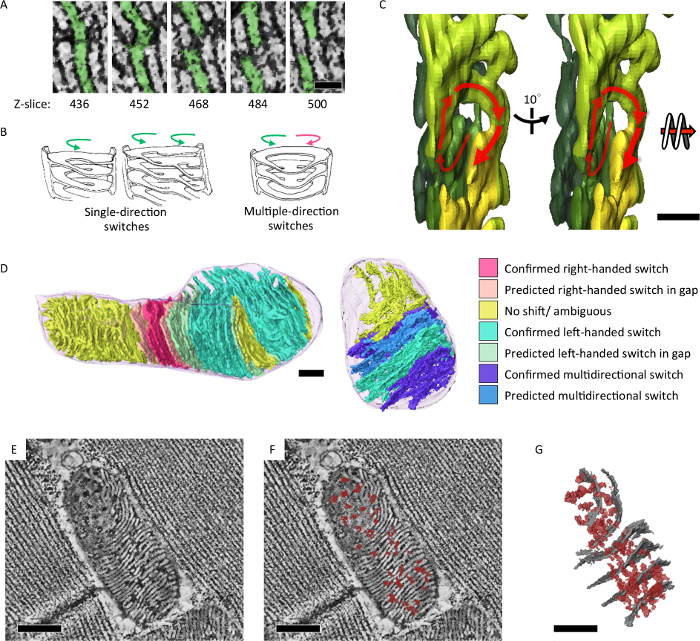
Figura 4: i cristae intra-mitocondri e rete di matrice ha rivelato in 3D integrati. (A) fette di ricostruzioni tomografiche mitocondriale dell'elettrone risultati commuta tra membrane lamellare attraverso l'asse z. (B) illustrazioni di cristae osservati modelli di spirali in mano e/o sinistri di commutazione. (C) segmentazione tomografica illustrando una spirale mancina in 3D Cristae (D) modelli di commutazione sono stati analizzati e colore-il rendering del modello di segmentazione (E, F). Tomografica fetta risultati confluency di matrice laterale (densità scuro, segnati in rosso) attraverso le membrane cristae (densità bianco). (G) segmentazione modello del tomogramma nella (E) risultati confluency di matrice laterale (in rosso) e i cristae rappresentativi (in grigio). Barra della scala = 50 nm (A), 200 nm (C) e 300 nm (D, E, F, G). La figura era la ristampa da Jiang et al. 7 Clicca qui per visualizzare una versione più grande di questa figura.

Figura 5: mitocondri con cipolla-come nuclei vorticosi accumulano nelle mosche con difetti di replica del DNA mitocondriale durante l'invecchiamento. Rappresentante fette tomografiche e la segmentazione corrispondente (pannelli di destra) Mostra una vista di sezione trasversale (in alto) e una vista di sezione longitudinale (in basso) di un nucleo vorticoso. Segmentazione del volume è indicato dal rendering colore arbitrario per evidenziare il nucleo vorticoso. Barra della scala: 200 nm. La figura era la ristampa da Jiang et al. 7 Clicca qui per visualizzare una versione più grande di questa figura.
| Passo | Temp | Tempo | Soluzione | |||
| 1 | -140 ° C a-9 0 ° C | 30 min | Azoto liquido | |||
| 2 | -90 ° C | a 96 ore | Cocktail di FS | |||
| 3 | -90 ° C a-60 ° C | 6 (5 ° C/h) | Cocktail di FS | |||
| 4 | -60 ° C | 12 hr | Cocktail di FS | |||
| 5 | -60 ° C a-25 ° C | 7 (5 ° C/h) | Cocktail di FS | |||
| 6 | -25 ° C | 12 hr | Cocktail di FS | |||
| 7 | -25 ° C a 0 ° C | 5 (5 ° C/h) | Cocktail di FS | |||
| 8 | 0 ° C | 1 hr x 3 volte | Acetone | |||
| 9 | Temperatura della camera | Infiltrazione di resina | ||||
Tabella 1: Protocollo di Freeze-sostituzione.
Discussione
In questo protocollo, descriviamo un workflow ottimizzato per l'applicazione di tomografia elettronica di serie-sezione per lo studio 3D ultrastruttura mitocondriale del muscolo di Drosophila voli indiretti. Conservazione della ultrastruttura del campione è la principale sfida tecnica per questo tipo di analisi. Al fine di preservare metodologico ultrastruttura due passaggi sono stati inclusi. In primo luogo, tessuto stato campionato dal sezionamento con vibrante microtomo lama al fine di mantenere il più possibile l'architettura del tessuto. In secondo luogo, un protocollo di HPF/FS è stato ottimizzato per preservare l'ultrastruttura organello durante la preparazione di blocchi campione incorporato. I campioni sono stati congelati sotto alta pressione, che abbassa il punto di congelamento dell'acqua e riduce la formazione di cristalli di ghiaccio che danneggiare ultrastruttura1. Campioni di spessore come 0,1 mm possono essere vetrificati istantaneamente e quindi sottoposti per congelare sostituzione per generare blocchi campione per l'analisi di EM. La migliore conservazione dell'ultrastruttura di HPF/FS è stata notata, rispetto ai metodi di fissazione chimica. Tramite questa serie di passaggi per la preparazione del campione, conservazione delle membrane mitocondriali doppie e i cristae membrane è stata migliorata drammaticamente.
Ottenere sezioni di serie dell'esemplare è il passo più impegnativo del metodo. Come il fascio elettronico ha un limitato potere di penetrazione, lo spessore della sezione è limitato a entrambi 250 nm o 500 nm utilizzando un TEM di funzionamento a 200 kV o 300 kV, rispettivamente. Poiché lo spessore di un mitocondrio può essere più di 2 µm, le sezioni di serie vengono richiesto di ottenere ricostruzioni volume completo. Tuttavia, riprendendo un numero sufficiente di sezioni di serie da occupare un'intero organello è una sfida tecnica. Tagliare il volto di blocco per essere il più piatto possibile tra le basi del trapezio può consentire una griglia di slot accogliere più sezioni e quindi coprire volumi più grandi. Inoltre, utilizzando un ciclo perfetto per sezioni sottili aumenta il tasso di successo di trasferire le sezioni seriale alla griglia.
Tomografia elettronica di serie-sezione può essere realizzata con standard attrezzature di anima di EM. Tuttavia, il metodo ha alcune inevitabili limiti che derivano da vincoli tecnici. Uno è che il materiale è inevitabilmente perso tra le sezioni di serie, lasciando spazi vuoti nella ricostruzione Unite in join. In secondo luogo è l'artefatto mancante di Cuneo, che si pone a causa degli angoli di inclinazione limitata che sono realizzabili. Questa restrizione si verifica perché il supporto del campione non può essere trasformata in una rotazione completa senza bloccare il fascio di elettroni. Nonostante queste limitazioni, tomografia elettronica di serie-sezione fornisce una risoluzione sufficiente per rivelare l'ultrastruttura cellulare e organello in 3D.
Per l'imaging di scala più piccola, cryo-elettrone la tomografia è una tecnologia emergente che può essere utilizzata per ottenere la struttura macromolecolare complessi e assiemi in situ risoluzione nm o Sub-angstrom in combinazione con sub-tomografica ricostruzione3. In questa applicazione, le cellule sono assottigliate di sezionamento o di fresatura sotto azoto liquido il fascio ionico focalizzato. I tomogrammi vengono raccolti in cryo-condizioni dove strutture molecolari sono conservate vicino lo stato nativo senza fissaggio chimico, disidratazione o incorporamento. A altra estremità della scala, per analizzare i volumi di grandi dimensioni tessuto a scapito della risoluzione, microscopia elettronica a scansione seriale blocco-viso è una modalità accattivante anche se richiede un strumento specifico4.
Divulgazioni
Gli autori dichiarano di non avere nessun concorrenti interessi finanziari.
Riconoscimenti
Gli studi sono stati effettuati in core EM presso l'Istituto di biologia cellulare e organismica e il nucleo di cryo-EM di Academia Sinica, Taipei, Taiwan. Il lavoro è stato supportato da Academia Sinica e la maggior parte.
Materiali
| Name | Company | Catalog Number | Comments |
| vibrating blade microtome | Leica | VT1200S | Tissue sectioning |
| high-pressure freezer | Leica | EM HPM100 | Specimen preparation |
| freeze-substitution device | Leica | EM AFS2 | Specimen preparation |
| ultramicrotome | Leica | EM UC7 | Ultra-thin sectioning |
| dual-axis tomography holder | Fischione | Model 2040 | tomography collection |
| transmission electron microscope | FEI | Tecnai F20 | tomography collection |
| CCD | Gatan | UltraScan 1000 | tomography collection |
| Leginon | NRAMM/AMI | tomography collection | |
| IMOD | Boulder Laboratory for 3-D Electron Microscopy of Cells | Tomography reconstruction | |
| Avizo 3D | FEI | Tomography analysis | |
Riferimenti
- Dahl, R., Staehelin, L. A. High-pressure freezing for the preservation of biological structure: theory and practice. J Electron Microsc Tech. 13 (3), 165-174 (1989).
- Sabatini, D. D., Bensch, K., Barrnett, R. J. Cytochemistry and electron microscopy. The preservation of cellular ultrastructure and enzymatic activity by aldehyde fixation. J Cell Biol. 17, 19-58 (1963).
- Rigort, A., et al. Micromachining tools and correlative approaches for cellular cryo-electron tomography. J Struct Biol. 172 (2), 169-179 (2010).
- Denk, W., Horstmann, H. Serial block-face scanning electron microscopy to reconstruct three-dimensional tissue nanostructure. PLoS Biol. 2 (11), e329 (2004).
- Lucic, V., Forster, F., Baumeister, W. Structural studies by electron tomography: from cells to molecules. Annu Rev Biochem. 74, 833-865 (2005).
- Soto, G. E., et al. Serial section electron tomography: a method for three-dimensional reconstruction of large structures. Neuroimage. 1 (3), 230-243 (1994).
- Jiang, Y. F., et al. Electron tomographic analysis reveals ultrastructural features of mitochondrial cristae architecture which reflect energetic state and aging. SciRep. 7, 45474 (2017).
- Cogliati, S., Enriquez, J. A., Scorrano, L. Mitochondrial Cristae: Where Beauty Meets Functionality. TrendsBiochemSci. 41 (3), 261-273 (2016).
- Friedman, J. R., Nunnari, J. Mitochondrial form and function. Nature. 505 (7483), 335-343 (2014).
- Suloway, C., et al. Fully automated, sequential tilt-series acquisition with Leginon. J Struct Biol. 167 (1), 11-18 (2009).
- Mastronarde, D. N., Held, S. R. Automated tilt series alignment and tomographic reconstruction in IMOD. J Struct Biol. 197 (2), 102-113 (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon