Method Article
Cavitazione di azoto e centrifugazione differenziale permette di monitorare la distribuzione periferica delle proteine di membrana in cellule coltivate
In questo articolo
Riepilogo
Qui presentiamo protocolli per omogeneizzazione detergente privo di cellule di mammiferi coltivate basato su azoto cavitazione e successiva separazione delle proteine citosoliche e membrana-limiti di ultracentrifugazione. Questo metodo è ideale per il monitoraggio il partizionamento periferici delle proteine di membrana tra solubili e frazioni della membrana.
Abstract
Le cellule coltivate sono utili per studiare la distribuzione subcellulare delle proteine, tra cui proteine di membrana periferici. Geneticamente codificato fluorescente proteine etichettate hanno rivoluzionato lo studio della distribuzione subcellulare della proteina. Tuttavia, è difficile quantificare la distribuzione con microscopia di fluorescenza, soprattutto quando le proteine sono parzialmente citosoliche. Inoltre, è spesso importante per lo studio di proteine endogene. Come immunoblots rimangono il gold standard per la quantificazione della distribuzione della proteina dopo frazionamento subcellulare dosaggi biochimici. Anche se ci sono kit commerciali che mirano a isolare citosolico o determinate frazioni di membrana, la maggior parte di questi kit si basano sull'estrazione con detersivi, che potrebbe non essere adatto per lo studio delle proteine di membrana periferici che facilmente vengono estratti dalle membrane. Qui presentiamo un protocollo privo di detergente per cellulare omogeneizzazione di azoto cavitazione e successiva separazione delle proteine citosoliche e di membrana-limitano dall'ultracentrifugazione. Confermiamo la separazione degli organelli subcellulari in solubile e frazioni di pellet attraverso diversi tipi di cellule e confrontare l'estrazione di proteine tra diversi metodi di omogeneizzazione meccanica non basati su detergente comune. Tra i numerosi vantaggi di azoto cavitazione è l'efficienza superiore di rottura cellulare con minimo danno fisico e chimico di organelli delicati. Combinato con ultracentrifugazione, azoto la cavitazione è un metodo eccellente per esaminare lo spostamento delle proteine di membrana periferici tra citosolico e frazioni della membrana.
Introduzione
Proteine cellulari possono essere suddivisi in due classi: quelli che sono associati con membrane e quelli che non sono. Proteine associate non-membrana si trovano nel citosol, nucleoplasma e lumina degli organelli come il reticolo endoplasmico (ER). Ci sono due classi di proteine di membrana-collegato, integrale e periferici. Proteine integrali di membrana sono noti anche come proteine transmembrana, poiché uno o più segmenti della catena del polipeptide si estende la membrana, in genere come un α-elica composta da amminoacidi idrofobici. Proteine transmembrana co-translationally vengono inseriti nelle membrane nel corso della loro biosintesi e rimangono così configurate fino a quando essi sono catabolizzate. Proteine di membrana periferico secondariamente sono spinti a membrane, solitamente come conseguenza di modificazione post-traduzionale con molecole idrofobe quali i lipidi. A differenza di proteine integrali di membrana, l'associazione di proteine di membrana periferico con le membrane cellulari è reversibile e può essere regolata. Molti funzione di proteine di membrana periferico in vie di segnalazione e regolamentato associazione con membrane è un meccanismo per attivando o inibendo una via. Un esempio di una molecola di segnalazione che è una proteina di membrana periferico è il piccolo GTPase, RAS. Dopo una serie di modificazioni post-traduzionali che includono la modifica con un lipido di farnesyl, modificate C-terminale di una proteina RAS matura inserisce l'opuscolo citoplasmatico della membrana cellulare. In particolare, la membrana plasmatica è dove il RAS si aggancia il suo effettore a valle RAF1. Per impedire l'attivazione costitutiva della chinasi di proteina mitogene-attivata (MAPK), livelli multipli di controllo RAS sono a posto. Oltre a rendering RAS inattivo idrolizzando il GTP in PIL, RAS attivo anche può essere rilasciato dalla membrana plasmatica da modifiche o interazioni con solubilizzanti fattori per inibire la segnalazione. Anche se fluorescente live imaging offre biologi cellulari l'opportunità di osservare la localizzazione subcellulare delle proteine di membrana periferico della proteina-etichetta fluorescente1, ci rimane un bisogno fondamentale per valutare l'associazione della membrana di proteine endogene semi-quantitativamente con semplici approcci biochimici.
L'appropriata valutazione biochimica delle proteine partizionamento tra membrana e frazioni solubili è criticamente dipendono da due fattori: cellulare omogeneizzazione ed efficiente separazione della membrana e frazioni solubili. Anche se alcuni protocolli, tra cui i kit commercializzati più ampiamente usati, dipendono dall'omogeneizzazione di detergente a base di cellule, questi metodi possono offuscare analisi tramite l'estrazione di proteine di membrana nei solubile fase2. Di conseguenza, non detergente basati, meccanici metodi di distruzione cellulare forniscono risultati più puliti. Ci sono diversi metodi di rottura meccanica delle cellule in coltura o raccolte dal sangue o organi. Questi includono lisata omogeneizzazione, rottura fine dell'ago, cuscinetti a sfera omogeneizzazione, sonicazione e cavitazione di azoto. Qui valutiamo cavitazione di azoto e confrontarlo con altri metodi. Cavitazione di azoto si basa sull'azoto che è dissolto nel citoplasma delle cellule ad alta pressione. Dopo l'equilibratura, la sospensione cellulare bruscamente è esposta alla pressione atmosferica tale che bolle di azoto si formano nel citoplasma che strappare aperto la cella in conseguenza del loro effervescenza. Se la pressione è sufficientemente elevata, effervescenza di azoto possa interferire con il nucleo3 e membrana associato organelli come i lisosomi4. Tuttavia, se la pressione è mantenuta abbastanza bassa, la decompressione altererà la membrana plasmatica ed ER ma non altri organelli, quindi versare sia cytosol e organelli citoplasmici intatti nell'omogenato viene designato il cavitate5. Per questo motivo, la cavitazione di azoto è il metodo di scelta per l'isolamento di organelli come i lisosomi e mitocondri.
Tuttavia, è anche un ottimo modo di preparare un omogeneato che può essere facilmente separato in membrana e frazioni solubili. Il recipiente a pressione (d'ora in poi chiamato "the bomb") utilizzato durante la cavitazione è costituito da un involucro di acciaio inox spessore che resiste ad alta pressione, con un ingresso per la consegna del gas dell'azoto da un serbatoio e un orificio di presa con valvola di scarico regolabile.
Cavitazione di azoto è stato utilizzato per l'omogeneizzazione delle cellule dal 1960s6. Nel 1961, Hunter e Commerfold7 stabilito cavitazione di azoto come una valida opzione per distruzione di tessuti di mammiferi. Da allora, i ricercatori hanno adattato la tecnica di varie cellule e tessuti con successo e cavitazione azoto è diventato un fiocco in più applicazioni, tra cui membrana preparazione8,9, nuclei e organello preparazione10,11ed estrazione biochimici labili. Attualmente, i biologi delle cellule più spesso impiegano altri metodi di omogeneizzazione delle cellule perché i benefici di omogeneizzazione di azoto non sono stati ampiamente pubblicizzati, bombe di azoto sono costose e c'è un equivoco che un numero relativamente elevato di celle è Obbligatorio. Protocolli per la cavitazione di azoto raggiungere omogeneati privo di cellule con nuclei intatti non sono stati pubblicati, e nelle valutazioni pubblicati più volumi di 20 mL di sospensione cellulare sono stati usati. Per adattare questa tecnica classica per soddisfare i requisiti attuali di lavorare con campioni di piccole dimensioni, vi presentiamo un protocollo modificato di cavitazione di azoto specificamente progettato per le cellule coltivate. Dopo cavitazione di azoto, l'omogeneizzato è separato in solubili (S) e frazioni di membrana (P) tramite centrifugazione differenziale, prima con un giro di basso-velocità per rimuovere i nuclei e cellule ininterrotte e poi con una rotazione ad alta velocità (> 100.000 x g) per separare membrane dalla frazione solubile. Analizziamo l'efficienza della separazione con immunoblots e confrontare cavitazione di azoto con altre tecniche di rottura meccanica. Studiamo anche l'effetto osmotico del buffer di omogeneizzazione durante la cavitazione di azoto.
Protocollo
1. buffer e attrezzature preparati
- Chill 45ml cella perturbazione bombe, provette da 15 mL e tubi ultracentrifugazione a 4 ° C.
- Preparare e chill 25 mL di tampone di omogeneizzazione per 2 x 10 7 cellule a 4 ° C. Aggiungi una proteasi inibitore compressa appena prima dell'uso.
Nota: Omogeneizzazione buffer contengono in genere KCl anziché NaCl per meglio riflettere composizione sale intracellulare. Buffer di omogeneizzazione utilizzato in questo protocollo è costituito da 10 mM HEPES pH 7.4, 10mm KCl e 1,5 mM MgCl 2 (in appresso denominato buffer di omogeneizzazione ipotonica). Maggior parte dei buffer può essere adattato per la cavitazione di azoto (Vedi discussione). - Preparare e chill 6 mL di 1x tampone Phosphate-Buffered salino (PBS) per campione a 4 ° C. Aggiungi della proteasi inibitore compresse freschi prima dell'uso.
Nota: Tampone PBS utilizzato nel presente protocollo è costituito da 10 mM Na 2 HPO 4 a pH 7.4, 1,8 mM KH 2 PO 4, 137 mM NaCl e 2.7 mM KCl. - Preparare e chill 4 mL di tampone di solubilizzazione per campione a 4 ° C. aggiungere una proteasi inibitore pastiglia appena prima dell'uso.
Nota: Solubilizzazione buffer utilizzato in questo protocollo è 1 tampone del saggio (RIPA) x Radioimmunoprecipitation, che consiste di 25 mM Tris a pH 7.4, 150 mM NaCl, 0.1% SDS, sodio desossicolato 0,5% e 1% NP-40. Vedere nota 4.6.
2. Raccolta di cella
- Grow 2 x 10 7 -10 x 10 9 celle di coltura del tessuto con la raccomandata di coltura per il tipo di cella. In genere, un piatto di 15 cm produce 2 x 10 7 cellule HEK-293 coltivate in DMEM al 90% confluency ( Figura 1).
- Rimuovere il mezzo di crescita dal vuoto.
- Per le cellule aderenti, mettere i piatti di cultura sul ghiaccio, lavare le cellule direttamente sui piatti cultura delicatamente con buffer di omogeneizzazione refrigerati due volte (10 mL di tampone per ogni piatto di 15cm a lavaggio) e raccogliere le cellule con un raschietto di grandi cellule in un volume adeguato di buffer di omogeneizzazione; Per cellule in sospensione, raccogliere e lavare il pellet cellulare in due volte di buffer per il refrigerate omogeneizzazione (10 mL di tampone per la cultura di 50 mL per lavaggio) con x 500 g di spin a 4 ° C per 5 min e risospendere il pellet cellulare lavato in un adeguato volume di tampone di omogeneizzazione su ghiaccio.
Nota: il volume dovrebbe essere basato sui requisiti di concentrazione nella proteina negli esperimenti previsti, nonché il volume minimo/massimo consentito della bomba di rottura delle cellule. Una linea guida generale è 2-10 x 10 7 cellule/mL, o a pellet circa 10 volumi della cella. Questo protocollo è ottimizzato per i tre piatti di 15 cm di cellule HEK-293 in 2 mL di buffer di omogeneizzazione.
3. Cavitazione di azoto
- trasferimento la sospensione cellulare a una bomba pulita e rilassante in un bagno di ghiaccio su scalpore piastra.
Attenzione: La bomba ha ad alta pressione, bassa temperatura, gas azoto – indossare protezione personale appropriato . - Inserire un micro ancoretta magnetica all'interno della bomba e accendere la piastra stir per mantenere omogeneità sospensione.
- Aggiungere una compressa di inibitore della proteasi alla sospensione e chiudere la bomba al produttore ' Istruzione s.
- Pressurizzare gradualmente la bomba con un serbatoio di gas di azoto per fabbricante ' s istruzione fino al manometro bomba legge 300-600 psi. Chiudere tutte le valvole e scollegare il serbatoio di azoto.
Nota: La pressione necessaria può variare con il tipo di cella. Qui abbiamo effettuato cavitazione a 350-400 psi per cellule HEK-293, NIH-3T3 e Jurkat. - Aspettare 20 minuti consentire l'azoto sciogliere e raggiungere l'equilibrio all'interno delle cellule.
- Rimuovere l'acqua in eccesso attorno alla valvola di scarico con un tovagliolo di stoffa. Aprire la valvola di scarico delicatamente per ottenere un rilascio goccia a goccia dell'omogeneato e raccogliere in un tubo da 15 mL pre-refrigerati.
Nota: Vicino alla fine della raccolta ci sarà un getto dell'omogeneato e gas emergerà con un sibilo. Assicurarsi che il gas non causa precedentemente raccolto cavitano per sparare fuori dal tubo (da qui l'uso di 15 mL tubi anziché provette da 1,5 mL). Una volta che il getto viene avviato, chiudere la valvola di scarico e aprire il rubinetto dell'azoto bruscamente per depressurizzare la bomba e raggiungere la cavitazione delle cellule restanti nella bomba. Aperta la bomba per cavitazione recupero e pulizia accurata.
Nota: Il cavitate finale dovrebbe avere un aspetto lattiginoso con schiuma sulla parte superiore. Mescolare delicatamente con un puntale per consentire la schiuma a scemare prima centrifugazione.
Nota: Esaminare il cavitate da microscopia di contrasto di fase per determinare l'efficienza di omogeneizzazione. Aggiungere una goccia di 15 µ l di cavitazione alla superficie di una diapositiva di microscopia e coprire con un vetrino coprioggetti. Ripetere il punto 3.4-3.6 solo se troppi ininterrotte cellule vengono rilevati con un obiettivo X 20.
Nota: Se il buffer di omogeneizzazione non contiene EDTA o EGTA, aggiungerlo ai raccolti cavitano ad una concentrazione finale di 1 mM entro 5 min dopo la dimissione.
4. Separazione delle frazioni della membrana e citosolica
- centrifuga cavitate a 500 x g per 10 min a 4 ° C per rimuovere cellule ininterrotte e nuclei.
Nota: Ripetere la fase di centrifugazione non visibile a pellet è prodotto e raccogliere il surnatante nucleare Post (PNS) evitando la schiuma che galleggia sulla parte superiore. Ri-Centrifugare la schiuma per raccogliere e combinare PNS, ulteriormente se necessario ( Figura 1). - Elaborare il PNS come desiderato per ottenere frazioni di interesse. Ai fini della separazione citosolico e frazioni della membrana, trasferire il PNS in una provetta ultracentrifuga ed eseguire ultracentrifugazione come desiderato. Questo protocollo è ottimizzato per un < 3,5 mL di campione in un tubo di policarbonato ultracentrifuga e per ultracentrifugazione a 350.000 x g per 1 h a 4 ° C.
- Raccogliere il surnatante (la frazione S) usando una pipetta da 1 mL.
- Risciacquare accuratamente il pellet con 3 mL di PBS freddo senza disturbarla. Rimuovere il PBS dal vuoto.
Nota: Se la contaminazione della frazione della membrana di proteine citosoliche è una preoccupazione maggiore rispetto alla perdita di campione, risospendere il pellet in 3 mL di PBS freddo e re-ultracentrifuga come descritto al punto 4.2. Rimuovere il PBS dal vuoto. - Risospendere il pellet completamente in un appropriato volume di tampone di solubilizzazione contenente detersivo di scelta. Per raggiungere l'equivalente di cella, utilizzare lo stesso volume di tampone di solubilizzazione come frazione citosolica.
Nota: Si consiglia di utilizzare 1 x RIPA buffer come buffer di solubilizzazione per estrazione di proteine di membrana efficiente. Se nessun test a valle è necessaria per la frazione della membrana, utilizzare 1 x laemmli tampone per estrazione di proteine di membrana maximal.
Nota: Consigliamo di sloggiare e trasferendo il pellet nel buffer di solubilizzazione in una provetta pulita in un rotatore tubo a 4 ° C per estrazione di proteine di membrana maximal.
Nota: Se la pallina è troppo appiccicosi (troppi lipidi) per essere rimosso in modo efficiente da thtubo di ultracentrifuga e nel buffer di solubilizzazione, suggeriamo lo snap il pellet di congelamento in azoto liquido e rapidamente sloggiare il pellet dal tubo ultracentrifuga con una spatola di metallo mini prima il pellet si scongela.
Nota: In alternativa, membrana pellet può essere solo risospese e non solubilizzati in un buffer non contenente detersivo per produrre una frazione P di sospensione delle vescicole di membrana, nel qual caso la centrifugazione passo in 4.6 non è richiesto. Tali frazioni di P sono utili quando indagare funzioni quali attività enzimatiche che dipendono dalla membrana associazione. - Centrifugare la sospensione a pellet completamente solubilizzata dal passaggio 4.5 a 20.000 x g in una centrifuga da tavolo per 10 min a 4 ° C. raccogliere il surnatante con una pipetta da 1 mL (la frazione di P) e scartare il pellet (lipidi insolubili).
- Effettuare saggi desiderate come macchiarsi occidentale con le frazioni citosoliche e/o membrana, o salvarli a-80 ° C per un uso futuro.
Risultati
La figura 2 Mostra il partizionamento delle proteine cellulari da PNS in frazione citosolica solubili (S) o frazione della membrana della pallina (P). Abbiamo esaminato tre linee cellulari rappresentative da diversi tipi di cellule: HEK-293 (epiteliali), NIH-3T3 (fibroblasti) e Jurkat (linfociti). Inibitore di Rho guanina dissociazione (RhoGDI) e recettore di cationi-indipendente mannosio-6-fosfato (CIMPR) sono stati utilizzati come controlli positivi per citosolico e frazioni della membrana, rispettivamente. Abbiamo confermato la separazione efficiente di citoplasma e membrana dopo cavitazione di azoto a ~ 350 psi nel buffer ipotonico seguita da ultracentrifugazione a 350.000 x g per 1 h. La proteina totale recuperato dalle frazioni di S e P sono stati quindi analizzate con marcatori per ER, endosomi e lisosomi, mitocondri, Golgi e altri organelli. Tutte le proteine transmembrane sono presenti nella frazione della membrana esclusivamente, tra cui Na+/k+ dell'atpasi e ricevitore di fattore di crescita epidermico (EGFR) dalla membrana del plasma, calnexin da ER, proteina di membrana lisosomiale-collegata 1 (LAMP1) da lisosomi, F0-atpasi da mitocondri e Golgin 97 dal trans Golgi network. Come previsto, molte proteine di membrana periferici che sono vagamente associato con la membrana, sono presenti sia nelle frazioni citosoliche e di membrana a vari gradi. Alcuni esempi includono l'antigene endosome in anticipo 1 (EEA1), Rab7/9 (late endosomes) ed esochinasi 1 (membrana esterna del mitocondrio). Questo dimostra l'utilità di questa tecnica nella valutazione della resistenza membrana associazione di proteine periferici. Il nostro laboratorio ha approfittato di questa tecnica di cavitazione per esaminare lo stato di associazione di membrana di diverse molecole segnalazione5,12,13,14. È interessante notare, abbiamo trovato calregulin quasi esclusivamente nella frazione solubile, suggerendo che i microsomi generati dalle membrane ER inoltre sono stati interrotti durante la cavitazione di azoto e le proteine nel lume ER vengono rilasciate alla frazione solubile. Al contrario, l'integrità dei mitocondri dopo cavitazione è dipenda dal tipo di cella. Abbiamo osservato F1-atpasi, una proteina periferica interna mitocondri, parzialmente nella frazione citosolica che vanno dal 2% per le cellule NIH 3T3-a ~ 35% per le cellule HEK-293 e Jurkat. Questo indica che azoto cavitazione a 350 psi con buffer ipotonico, non garantisce l'integrità mitocondriale. Nonostante tre cicli di 500 x g giri per generare il PNS da cavitate, non tutti i nuclei sono stati rimossi. Istone deacetilasi 1 (HDAC1) da nucleoplasma e fibrillarina dal nucleolo, sono state trovate sia in PNS e il pellet. La presenza di HDAC1 nel pellet e non nel surnatante suggerisce che il PNS è contaminato con i nuclei, piuttosto che la distruzione nucleare durante la cavitazione. Questo è coerenza con le relazioni che la bomba a 350 psi di pressurizzazione lascia i nuclei intatti10, anche se il nostro uso del buffer ipotonico può rendere i nuclei fragili, come discusso nella sezione successiva.
Abbiamo confrontato l'efficienza di omogeneizzazione di cavitazione di azoto con quella di altri metodi di interruzione fisica comune. Uguali quantità di cellule Jurkat sono stati sospesi nel buffer di omogeneizzazione ipotonico identici e sottoposti a metodi diversi disagi. Ci era perdita rilevabile di campioni in lisata cavitazione di omogeneizzazione e azoto (in entrambi i casi circa il 5-10% meno volume è raccolto). Tuttavia, la cavitazione di azoto ha dato la più alta efficienza di estrazione di proteine; passaggio dell'ago e lisata ha reso solo il 60% quanta proteina (Figura 3). Curiosamente, il recupero di alcune proteine come determinato da immunoblot non ha mostrato una differenza significativa fra i metodi differenti di rottura meccanica. Tuttavia, la resa di alcune proteine di membrana periferici quali esochinasi 1 e RAS era più alta nei campioni preparati con cavitazione di azoto. Questo supporta tale cavitazione di azoto potrebbe essere ottimale per omogeneizzazione durante l'analisi di proteine di membrana periferici. Recupero delle proteine HDAC1 di cavitazione di azoto è paragonabile ad altri metodi, suggerendo che i nuclei rimangono intatti in tutte le condizioni. Così, i dati nella Figura 3 dimostrano che cavitazione di azoto offre risultati superiori omogeneizzazione.
Successivamente, abbiamo confrontato l'efficienza di omogeneizzazione tra buffer ipotonico e lo stesso buffer ma fatto isotoniche con saccarosio o cloruro di sodio (Figura 4). Simili quantità di proteina sono stati recuperati dalle cellule Jurkat omogeneizzate nel buffer ipotonico contro NaCl isotonica tampone dopo cavitazione a 350 psi. Al contrario, meno proteine è stata recuperata nel PNS delle cellule interrotte nel buffer fatto isotonica con saccarosio. Più singole proteine esaminate da immunoblot misura questo modello. Unica eccezione era fibrillarina che relativamente è stato arricchito nel PNS generato nel buffer isotonico.
Per determinare se il nostro protocollo possa essere impiegato per indagare il partizionamento periferici delle proteine di membrana tra solubili e frazioni della membrana, abbiamo confrontato il partizionamento modelli tra le ANR Bandierina-etichettate wild type e suo mutante prenilazione-carenti, C186S ( Figura 5). Allo stato stazionario, il tipo selvaggio ANR era presente nelle membrane, simile alle proteine della membrana più periferici, anche se la parte recuperata nella frazione solubile è maggiore per altre proteine RAS14e cytosol. Quando le ANR sono privata della sua modifica prenil, si perde la capacità di associare con membrane e diventa interamente citosolica. I modelli attesi partizionamento delle ANR ha confermato che il nostro protocollo è una scelta affidabile per studiare la dinamica di partizionamento periferici delle proteine di membrana tra citosol e membrane.
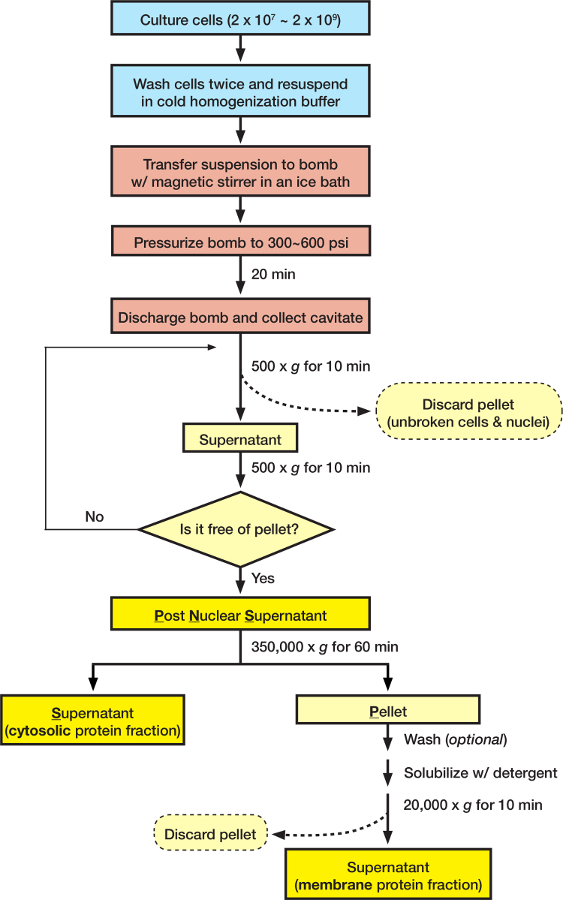
Figura 1: diagramma di flusso che riassume i passaggi 2 (blu), fase 3 (rosso) e 4 (giallo) del protocollo. Clicca qui per visualizzare una versione più grande di questa figura.
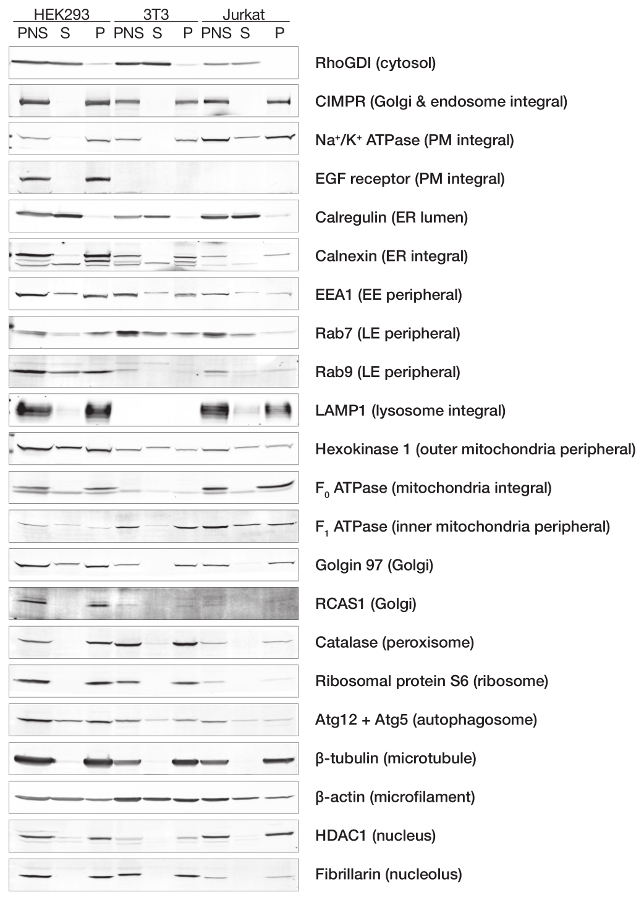
Figura 2: proteina partizionamento tra le frazioni citosoliche e di membrana. HEK-293, cellule NIH 3T3 e Jurkat sono stati sottoposti ad azoto cavitazione (~ 350 psi per 20 min, sospeso nel buffer ipotonico omogeneizzazione) e ultracentrifugazione (~ 350.000 x g per 1 h) come descritto nel protocollo. I livelli di proteina endogena in diverse frazioni sono stati analizzati da immunoblot. PNS: Antigene del cancro del ricevitore-legante espressa su cellule SiSo (RCA), nucleare surnatante, s: surnatante, p: pellet. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 3: confronto tra tecniche differenti di rottura meccanica. Quantità equivalente di cellule Jurkat sono stati sospesi nel buffer ipotonico e sottoposti a congelamento-scongelamento (3 cicli), needle omogeneizzazione di lisata di passaggio (5 passa, attraverso 28G½ ago), (15 passaggi, Kontes 2 mL lisata tubo con un pestello di macinare di tessuto, liquidazione dei 0.01-0.06 mm) o cavitazione azoto (~ 350 psi per 20 min). Livelli relativi di proteine endogene nel PNS sono stati analizzati da immunoblot per ciascun metodo. Le concentrazioni di proteina totale di PNS sono state quantificate da BCA assay e normalizzato al valore del PNS preparato dalla cavitazione di azoto. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 4: confronto tra buffer ipotonico e isotonico utilizzati durante la cavitazione azoto. Quantità equivalenti di Jurkat cellule sono stati sospesi nel buffer ipotonico, o buffer ipotonico completati con 8,5% di saccarosio o 150 mM NaCl e sottoposti a cavitazione azoto (~ 350 psi per 20 min). Livelli relativi di proteine endogene nel PNS sono stati analizzati da immunoblot per ogni buffer. Le concentrazioni di proteina totale di PNS erano quantificate dall'analisi BCA e normalizzate al valore del PNS preparato in tampone ipotonico. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 5: Farnesylated NRAS partizioni nelle frazioni sia P e S. Cellule HEK-293 transfected transitoriamente con plasmidi dirigere l'espressione della bandierina-etichettate le ANR wild type o mutanti di C186S. Proteina di partizionamento è stato effettuato come descritto nella Figura 2 e sono stati analizzati i livelli di proteina indicato da immunoblot. Riprodotto con permesso (Zhou, M et al., 2016. Originariamente pubblicato nel Journal of Cell Biology. https://Doi.org/10.1083/JCB.201604061). Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
I vantaggi della cavitazione di azoto rispetto ad altri metodi di rottura meccanica sono molteplici. Forse il vantaggio più significativo è la sua capacità a delicatamente ma efficacemente omogeneizzare campioni. I principi fisici di decompressione si raffredda campioni invece di generare riscaldamento locale danni quali ultrasuoni e attrito/tosatura basato su tecniche. La cavitazione è anche estremamente efficiente a perturbare la membrana plasmatica. Perché l'azoto bolle vengono generate all'interno di ogni singola cella al momento della decompressione, il processo di cavitazione è limitato meno le dimensioni della cella, modella indossa una taglia o concentrazione di esempio. Pertanto offre vantaggi aggiuntivi rispetto lisata basati su liquidazione o omogeneizzazione di passaggio dell'ago. Cavitazione di azoto offre anche risultati più coerenti come la stessa forza dirompente che è applicata in modo uniforme in tutto il campione può essere riprodotto con pressioni identiche. Inoltre, ogni cella avverte solo il processo di rottura per una sola volta e componenti sottocellulari sono, pertanto, non costantemente esposti alle forze dirompenti variabile. Questo limita la frammentazione artefactual degli organelli. Preoccupazione per l'ossidazione dei componenti cellulari labili causato da rottura anche è mitigato, come l'azoto è utilizzato per saturare la sospensione cellulare e sia privo di ossigeno supplementare. Le sollecitazioni fisiche e chimiche con restrizioni imposte dalla cavitazione di azoto rende una tecnica ideale per studiare labili enzimi e organelli fragili e la riproducibilità di questa tecnica di omogeneizzazione è adatto per la quantificazione.
Cavitazione di azoto offre anche notevole flessibilità per quanto riguarda la dimensione del campione, la composizione del buffer di omogeneizzazione e il grado di integrità dell'organello. Per scalare l'omogeneizzazione, si deve semplicemente distribuire recipienti a pressione di maggiori capacità, senza sacrificare la velocità, la convenienza o l'efficienza della decompressione. Può essere utilizzato qualsiasi buffer di omogeneizzazione per la cavitazione di azoto; scelta di buffer può essere adattato per la compatibilità con i requisiti di ogni esperimento. Si può progettare un buffer di omogeneizzazione che corrisponde il liquido intracellulare (ad es., di potassio e basso contenuto di sodio) per ridurre al minimo le alterazioni in organelli. Per esempio, abbiamo sviluppato un buffer di "relax" che riduce al minimo ex vivo polimerizzazione dell'actina e quindi aiuta nella raccolta delle vescicole citoplasmatiche intatte da granulociti5. Le caratteristiche osmotiche e ioniche del buffer possono anche essere modificate per controllare il grado di omogeneizzazione delle cellule. Inoltre, l'aggressività di rottura cellulare può essere controllata regolando la pressione di azoto. Una pressione moderata riduce la forza dirompente di preservare i nuclei, i mitocondri e altri organelli in uno stato integro, mentre ad alta pressione sconvolge questi organelli. Brock et al. segnalato il successo con rottura delle cellule di leucemia Basophilic ratto pur mantenendo la maggior parte dei nuclei intatto a 350 psi con tampone ipotonico10, che costituisce la base della pressione dell'azoto utilizzato nel nostro protocollo corrente.
Limitazioni di cavitazione di azoto includono il fatto che l'omogeneizzazione è uniforme in tutti i campioni e strutture differenti della membrana possono produrre vescicole di dimensioni simili; Questo può complicare ulteriore separazione della membrana plasmatica, lisci microsomi, endosomi e membrane del Golgi di centrifugazione in gradiente di densità tasso-zonale. Un'altra preoccupazione è che gli organelli diventano relativamente fragili dopo omogeneizzazione dovuto la rottura dall'interno. Questo metodo richiede pertanto un'attenta ottimizzazione per prevenire rotture organello in successive manipolazioni. Abbiamo mirato a risolvere alcuni di questi problemi in questo studio.
Separazione di citosolico e frazioni della membrana mediante centrifugazione è stato esplorato in rapporti limitati dettagliare il partizionamento delle frazioni proteiche con una manciata di proteina marcatori15,16. Il nostro metodo genera una separazione grezza delle proteine solubili e di membrana-limita e non comporta isolamento degli organelli particolari. Ancora, è ancora una preoccupazione legittima che le vescicole omogenee delle diverse membrane prodotte dalla cavitazione possono influenzare l'efficienza di separazione dall'ultracentrifugazione. Abbiamo presentato un'indagine completa di immunoblots dell'isolamento e la suddivisione dei comuni organelli sottocellulari dopo frazionamento con nostro protocollo. Come previsto, proteine citosoliche puramente come RhoGDI sono esclusivamente nella frazione solubile e proteine transmembrana sulla membrana plasmatica (PM) quali EGFR e Na+/k+ dell'atpasi, sono stati recuperati solo nel pellet. Proteine transmembrana su organelli come ER, mitocondri, Golgi e lisosomi inoltre sono stati recuperati nel pellet, confermando che proteine integrali di membrana da diverse membrane e organelli possono essere separati con successo da proteine solubili con il nostro metodo. Questa convalida fondamentale è cruciale per l'ulteriore analisi periferici delle proteine di membrana, che è lo scopo principale del presente protocollo.
La maggior parte delle proteine di membrana periferico esistono in entrambe le frazioni citosoliche e di membrana. Anche se spesso si tratta di una vera rappresentazione della loro localizzazione, può in alcuni casi rappresentare artefactual ridistribuzione di queste proteine dalla superficie delle membrane citoplasmatica alla frazione solubile durante il processo di omogeneizzazione, poiché la forza della loro associazione con membrane non è forte come quello di proteine integrali di membrana. Anche se la cavitazione di azoto riduce al minimo la ridistribuzione potenziale esponendo cellule una perturbazione di una tantum, delicata, uno dovrebbe essere conscio di tali manufatti di frazionamento biochimico e concentrarsi sui cambiamenti nella ripartizione delle proteine tra citosolico e le frazioni di membrana in condizioni sperimentali. Tuttavia, l'esame delle proteine di membrana periferici nelle varie fasi di maturazione endosomal è possibile con questo metodo. EEA1, un effettore Rab5 che media attracco delle vescicole rivestite di clatrina per l'early endosomes, si localizza la superficie citoplasmatica di early endosomes ed è stato recuperato principalmente nella frazione di pellet. Proteine Rab7 e Rab9 tardo-endosome-relative sono anche nella frazione di pellet. Tuttavia, rimane una quantità apprezzabile di Rab7/9 nel citosol. Perché RabGDI è in grado di interagire con prenilato Rabs ed eseguire il rendering di queste proteine altrimenti insolubili citosolico, proteine Rab tendono ad essere più citosolica di altre proteine di membrana periferici relativi endosoma.
A caratterizzare l'integrità nucleare e organello con omogeneizzazione di cavitazione di azoto, abbiamo immunoblotted le frazioni isolate per compartimenti sia luminal e citoplasmatici. In questa analisi, calregulin è completamente rilasciata la frazione solubile, dimostrando che ER è interrotto ai microsomi di forma e quindi perdite di contenuto luminale. Dato che una metà dell'area totale della membrana di una cellula eucariotica racchiude gli spazi labirintici dell'ER, è sorprendente che la sua vasta rete di sac-like o tubolare struttura non rimangono intatti durante l'espansione delle bolle di azoto. D'altra parte, la nostra analisi di integrità mitocondriale dopo cavitazione Mostra chiara dipendenza del tipo di cella, come abbiamo osservato F1-atpasi, una proteina che è marginalmente associata con la membrana interna dei mitocondri, nella frazione citosolica a vari livelli (~ 35% per HEK-293 e Jurkat, 2% per NIH-3T3). Questo indica che azoto cavitazione a 350 psi con buffer ipotonico non garantisce l'integrità mitocondriale. Infatti, altri hanno segnalato che, per preservare i mitocondri, cavitazione deve essere eseguita a pressioni inferiori a 150 psi17. Al contrario, abbiamo trovato che quel catalasi rimane nel lume dei perossisomi. Intrigante, proteine nucleari sono osservate nelle frazioni di PNS e pellet. In particolare, HDAC1, un deacetylase dell'istone presente in tutto il nucleoplasma, è assente nella frazione solubile, che indica che l'integrità nucleare non è compromessa. Ciò suggerisce che l'involucro nucleare non è rotto dalla cavitazione, come ci si aspetta il rilascio di HDAC1 nella soluble frazione in tale scenario. Resta probabile che i nuclei non vengono completamente rimossi durante il processo di preparazione del PNS, e la proteina rimane nel pellet dopo ultracentrifugazione. Questa probabilità è ulteriormente supportata dal fatto che nessuna perdita di DNA è individuata nella frazione solubile. Concludiamo, quindi, che utilizzando buffer ipotonico omogeneizzazione durante la cavitazione a ~ 350 psi con l'aggiunta di Mg2 + è in grado di mantenere l'integrità del nucleare in linee cellulari multiple.
Anche i nostri risultati dimostrano che i ribosomi raccolgono interamente in frazione di pellet della membrana, suggerendo che i ribosomi anche liberi nel citoplasma che sono non associati alle membrane ER possono essere completamente sedimentati in un buffer di non-saccarosio-cuscino dopo centrifugazione a 350.000 x g per 1 h. Parimenti, identificazione delle proteine autophagy-relative 12 (Atg12)-Atg5 coniugato come indicatore di autofagosomi, rivela rigorosa segregazione per le frazioni di pellet. Sua presenza nel citosol può essere causato dal fatto che il collegamento covalente di Atg12 e di Atg5 precede la formazione di autofagosomi. Proteine del citoscheletro sono difficili da valutare a causa della polimerizzazione ex vivo . Questo fenomeno può essere minimizzato con un buffer di relax ma nel protocollo qui descritto, la tubulina è probabile polimerizzata ex vivo su chelazione di calcio 18. Nelle nostre preparazioni tubulina è trovato nel pellet suggerendo polimerizzazione mentre l'actina è trovata nelle frazioni la P e la S.
Per caratterizzare potenziali vantaggi della cavitazione azoto nell'efficienza di omogeneizzazione, abbiamo esaminato alcune comuni tecniche di rottura meccanica. Metodi ottimizzati per campioni di tessuto come blender o mortaio/pestello non sono stati valutati. Sonicazione è stato escluso anche come è difficile interrompere la superficie della membrana mantenendo intatti gli organelli interni. La nostra analisi focalizzata sui seguenti metodi: gelo-disgelo, ago passaggio e omogeneizzazione lisata, come sono facili da eseguire e non richiedono attrezzature speciali, come un omogeneizzatore di cuscinetti a sfera. Cellule Jurkat sono state utilizzate in questa analisi a causa delle loro piccole dimensioni, che li rende difficili da omogeneizzare in modo efficiente con tradizionali tecniche di taglio liquide che dipendono dal gioco, come il passaggio dell'ago e lisata. Giudicato dalla concentrazione delle proteine totali recuperate, abbiamo trovato che metodi di perturbazioni indipendenti dimensione cella come azoto cavitazione e gelo-disgelo mostrano infatti superiore efficienza di estrazione di proteine. Simile analisi con celle di grandi dimensioni ha rivelato più piccole differenze nelle efficienze di omogeneizzazione, anche se la cavitazione di azoto è ancora superiore a metodi fisici alternativi testati (dati non mostrati).
Molti citosolico, proteine endosomal e citoscheletro sono state recuperate in modo efficiente con tutti i metodi testati. Al contrario, proteine sul ER e Golgi sono stati recuperati in modo ottimale utilizzando cavitazione di azoto. Il rendimento leggermente inferiore di F1-atpasi in cavitazione di azoto rispetto agli altri metodi possono riflettere il fatto che i mitocondri sono più probabili di rimanere intatta durante la cavitazione e pertanto più efficiente rimosso nella rotazione lenta velocità utilizzata per generare il PNS. L'osservazione che esochinasi 1, una proteina di membrana periferica trovato sia nel citosol e associato con la membrana mitocondriale esterna, si è comportata in maniera opposta rispetto la F1-ATPasi probabilmente riflette la labilità dell'associazione di esochinasi 1 relativo F1-atpasi, che sono sequestrato all'interno dei mitocondri. Cosa importante, anche se in grado di perturbare le celle con efficienze paragonabili, metodi alternativi di rottura meccanica ha dato relativamente povero recupero di alcune proteine di membrana periferici (esochinasi 1 e RAS). Così, la cavitazione è la nostra tecnica di omogeneizzazione di scelta allo scopo di indagare la partizione delle proteine di membrana periferici. Perché lisata omogeneizzazione è ampiamente usato per preparare i nuclei intatti, abbiamo usato lisata come un punto di riferimento per valutare l'integrità nucleare attraverso altri metodi di rottura meccanica. Nella nostra analisi, simili livelli di recupero delle proteine HDAC1 confermano che i nuclei rimangono intatti in Jurkat omogeneati preparati dalla cavitazione di azoto a ~ 350 psi con buffer ipotonico.
Era evidente che il buffer di omogeneizzazione utilizzato durante la cavitazione di azoto influenzano significativamente l'integrità di efficienza e organello di rottura. Mentre la composizione del buffer di omogeneizzazione deve essere ottimizzata per i requisiti dell'applicazione in questione, vale la pena notare che i ricercatori esecuzione di decompressione di azoto dei tessuti animali utilizzano buffer con una bassa pressione osmotica per estrarre nucleare contenuto. Hunter e Commerford ha mostrato i nuclei gonfiore e rottura quando le soluzioni sono state diluite e nuclei conservazione quando si utilizzano soluzioni isotoniche7 (che può essere raggiunto da qualsiasi aggiunta di sali inorganici come il cloruro di sodio o soluti organici come saccarosio o glicerolo). Il tampone isotonico standard utilizzato per isolare gli organelli sottocellulari diverso da nuclei da cellule di mammifero è 0,25 M saccarosio contenenti 1 mM EDTA e tamponata con Tris, HEPES o tRiCiNa a pH 7.0-7.6. Ma non tutte le cellule di cultura possono essere efficientemente omogeneizzate in uno dei buffer isotonico. Buffer ipotonico (solitamente 10 mM Tris, HEPES, ecc.) viene spesso utilizzato per fornire lo stress osmotico necessario per alcuni tipi di cellule per ottenere completa omogeneizzazione. Tuttavia, come accennato prima, buffer ipotonico tende a rendere i nuclei fragili e inclini a perdita di DNA quando EDTA è incluso. Per promuovere l'integrità nucleare, sostituiamo EDTA con KCl e basse concentrazioni di cationi bivalenti come MgCl2 o MgSO4. Mg2 + è solitamente preferibile Ca2 + perché quest'ultimo può attivare alcune fosfolipasi e proteasi e inibire la RNA polimerasi. Al termine della cavitazione EDTA o EGTA può aggiungersi torna a omogenati di chelare il catione, se necessaria per inibire la metalloproteasi.
Cellule Jurkat visualizzano un rapporto di nucleo al citoplasma relativamente grande, che li rende una linea cellulare ideale per studiare l'integrità nucleare. L'aumento dell'efficienza di omogeneizzazione con buffer ipotonico è minima quando si confrontano al buffer isotonico, completati con NaCl. Tuttavia, la cavitazione in un buffer isotonico completato con saccarosio recupera circa metà delle proteine estratte utilizzando una controparte di buffer ipotonico. Questo è improbabile da essere un risultato diretto di isotonicità, e la discrepanza tra utilizzando NaCl e saccarosio per raggiungere isotonicità richiede ulteriori indagini. Una possibile spiegazione è che sale sconvolge interazioni elettrostatiche tra proteine mentre non saccarosio. Così, anche la stessa tonicità, NaCl ha un'attività rilevante per estrazione di proteine che manca di saccarosio. With riguardo a preservare integrità nucleare, abbiamo trovato maggiore fibrillarina proteine recuperati con buffer isotonico, nonostante buffer isotonico teoricamente essere più protettivo dei nuclei. Curiosamente, la frazione mitocondriale è stata estratta in modo più efficiente con buffer isotonico, completati con NaCl, suggerendo che condizioni sale basse possono essere suboptimale per estrazione di mitocondri. Considerando la cavitazione con buffer ipotonico raggiunge il miglior equilibrio tra efficienza di omogeneizzazione e integrità nucleare, il buffer ipotonico diventi il buffer di scelta nel nostro protocollo di cavitazione. Tuttavia, cautela i lettori che nessun buffer è una garanzia di risultati ottimali omogeneizzazione. Il nostro protocollo dovrebbe servire come modello per l'ottimizzazione e pilota test con buffer desiderato, linee cellulari di interesse e altre variabili (pressione, composizione di buffer, ecc.) devono essere stabiliti per ottenere i migliori risultati.
Divulgazioni
Gli autori dichiarano di non avere nessun concorrenti interessi finanziari.
Riconoscimenti
Questo lavoro è stato finanziato dalla GM055279, CA116034 e CA163489.
Materiali
| Name | Company | Catalog Number | Comments |
| Cell Disruption Vessel (45 mL) | Parr Instrument | 4639 | Nitrogen cavitation Bomb |
| Dounce homogenizer (2 mL) | Kontes | 885300-0002 | Dounce pestle and tube |
| U-100 Insulin Syringe 28G½ | Becton Dickinson | 329461 | Needle |
| Atg12 antibody | Santa Cruz | 271688 | Mouse antibody, use at 1:1000 dilution |
| β-actin antibody | Santa Cruz | 47778 | Mouse antibody, use at 1:1000 dilution |
| β-tubulin antibody | DSHB | E7-s | Mouse antibody, use at 1:5000 dilution |
| Calnexin antibody | Santa Cruz | 23954 | Mouse antibody, use at 1:1000 dilution |
| Calregulin antibody | Santa Cruz | 373863 | Mouse antibody, use at 1:1000 dilution |
| Catalase antibody | Santa Cruz | 271803 | Mouse antibody, use at 1:1000 dilution |
| CIMPR antibody | Abcam | 124767 | Rabbit antibody, use at 1:1000 dilution |
| EEA1 antibody | Santa Cruz | 137130 | Mouse antibody, use at 1:1000 dilution |
| EGFR antibody | Santa Cruz | 373746 | Mouse antibody, use at 1:1000 dilution |
| F0-ATPase antibody | Santa Cruz | 514419 | Mouse antibody, use at 1:1000 dilution |
| F1-ATPase antibody | Santa Cruz | 55597 | Mouse antibody, use at 1:1000 dilution |
| Fibrillarin antibody | Santa Cruz | 374022 | Mouse antibody, use at 1:200 dilution |
| Golgin 97 antibody | Santa Cruz | 59820 | Mouse antibody, use at 1:1000 dilution |
| HDAC1 antibody | Santa Cruz | 81598 | Mouse antibody, use at 1:1000 dilution |
| Hexokinase 1 antibody | Cell Signaling Technology | 2024S | Rabbit antibody, use at 1:1000 dilution |
| Lamin A/C antibody | Santa Cruz | 376248 | Mouse antibody, use at 1:1000 dilution |
| LAMP1 antibody | DSHB | H4A3-c | Mouse antibody, use at 1:1000 dilution |
| Na+/K+ ATPase antibody | Santa Cruz | 48345 | Mouse antibody, use at 1:1000 dilution |
| Rab7 antibody | Abcam | 137029 | Rabbit antibody, use at 1:1000 dilution |
| Rab9 antibody | Thermo | MA3-067 | Mouse antibody, use at 1:1000 dilution |
| RCAS1 antibody | Santa Cruz | 398052 | Mouse antibody, use at 1:1000 dilution |
| RhoGDI antibody | Santa Cruz | 360 | Rabbit antibody, use at 1:3000 dilution |
| Ribosomal protein S6 antibody | Santa Cruz | 74459 | Mouse antibody, use at 1:1000 dilution |
| Sec61a antibody | Santa Cruz | 12322 | Goat antibody, use at 1:1000 dilution |
| Thickwall Polycarbonate ultracentrifuge tube | Beckman Coulter | 349622 | Sample tube for ultracentrifugation |
| TLK-100.3 rotor | Beckman Coulter | 349481 | rotor for ultracentrifugation |
| Optima MAX High-Capacity Personal Ultracentrifuge | Beckman Coulter | 364300 | ultracentrifuge |
| cOmplete protease inhibitor cocktail tablets | Roche | 11697498001 | protease inhibitors |
| Cell Scrapers with 25cm Handle and 3.0cm Blade | Corning | 353089 | large cell scraper |
| Magnetic Stir Bar | Fisher Scientific | 14-513-57SIX | micro stir bar |
| Ceramic-Top Magnetic Stirrer | Fisher Scientific | S504501AS | magnetic stirrer |
Riferimenti
- Miyawaki, A. Proteins on the move: insights gained from fluorescent protein technologies. Nat Rev Mol Cell Biol. 12 (10), 656-668 (2011).
- Bunger, S., Roblick, U. J., Habermann, J. K. Comparison of five commercial extraction kits for subsequent membrane protein profiling. Cytotechnology. 61 (3), 153-159 (2009).
- Simpson, R. J. Disruption of cultured cells by nitrogen cavitation. Cold Spring Harb Protoc. (11), (2010).
- Klempner, M. S., Mikkelsen, R. B., Corfman, D. H., Andre-Schwartz, J. Neutrophil plasma membranes. I. High-yield purification of human neutrophil plasma membrane vesicles by nitrogen cavitation and differential centrifugation. J Cell Biol. 86 (1), 21-28 (1980).
- Philips, M. R., et al. Low molecular weight GTP-binding proteins in human neutrophil granule membranes. Journal of Biological Chemistry. 266, 1289-1298 (1991).
- Wallach, D. F., Soderberg, J., Bricker, L. The phospholipides of Ehrlich ascites carcinoma cells: composition and intracellular distribution. Cancer Res. 20, 397-402 (1960).
- Hunter, M. J., Commerford, S. L. Pressure homogenization of mammalian tissues. Biochim Biophys Acta. 47, 580-586 (1961).
- Wallach, D. F., Kamat, V. B. Plasma and Cytoplasmic Membrane Fragments from Ehrlich Ascites Carcinoma. Proc Natl Acad Sci U S A. 52, 721-728 (1964).
- Birckbichler, P. J. Preperation of plasma membrane vesicles by nitrogen cavitation. TCA manual / Tissue Culture Association. 3 (3), 653-654 (1977).
- Brock, T. G., Paine, R., Peters-Golden, M. Localization of 5-lipoxygenase to the nucleus of unstimulated rat basophilic leukemia cells. J Biol Chem. 269 (35), 22059-22066 (1994).
- Dowben, R. M., Gaffey, A., Lynch, P. M. Isolation of liver and muscle polyribosomes in high yield after cell disruption by nitrogen cavitation. FEBS Letters. 2 (1), (1968).
- Dai, Q., et al. Mammalian prenylcysteine carboxyl methyltransferase is in the endoplasmic reticulum. J Biol Chem. 273 (24), 15030-15034 (1998).
- Wynne, J. P., et al. Rap1-interacting adapter molecule (RIAM) associates with the plasma membrane via a proximity detector. J Cell Biol. , (2012).
- Zhou, M., et al. VPS35 binds farnesylated N-Ras in the cytosol to regulate N-Ras trafficking. J Cell Biol. 214 (4), 445-458 (2016).
- Sim, D. S., Dilks, J. R., Flaumenhaft, R. Platelets possess and require an active protein palmitoylation pathway for agonist-mediated activation and in vivo thrombus formation. Arterioscler Thromb Vasc Biol. 27 (6), 1478-1485 (2007).
- Pace, P. E., Peskin, A. V., Han, M. H., Hampton, M. B., Winterbourn, C. C. Hyperoxidized peroxiredoxin 2 interacts with the protein disulfide- isomerase ERp46. Biochem J. 453 (3), 475-485 (2013).
- Annis, M. G., et al. Endoplasmic reticulum localized Bcl-2 prevents apoptosis when redistribution of cytochrome c is a late event. Oncogene. 20 (16), 1939-1952 (2001).
- Berkowitz, S. A., Wolff, J. Intrinsic calcium sensitivity of tubulin polymerization. The contributions of temperature, tubulin concentration, and associated proteins. J Biol Chem. 256 (21), 11216-11223 (1981).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon