Method Article
Imaging di cellule vive di cellule fungine per studiare modalità di entrata e localizzazione subcellulare delle defensine antifungino pianta
In questo articolo
Riepilogo
Defensine pianta gioca un ruolo importante nella difesa delle piante contro gli agenti patogeni. Per un uso efficace di questi peptidi antimicotici come agenti antifungini, comprendere le loro modalità di azione (MOA) è critico. Qui, è descritto un metodo di imaging di cellule vive per studiare aspetti critici di MOA di questi peptidi.
Abstract
Piccoli ricche di cisteina defensine sono uno dei più grandi gruppi di peptidi di difesa ospite presente in tutte le piante. Molti pianta defensine esibiscono potente in vitro attività antifungina contro un ampio spettro di patogeni fungini e pertanto hanno il potenziale per essere utilizzati come agenti antifungini in coltivazioni transgeniche. Al fine di sfruttare appieno il potenziale delle defensine impianto per controllo di malattie, è fondamentale per chiarire i loro meccanismi di azione (MOA). Con l'avvento delle tecniche di microscopia avanzata, formazione immagine della vivere-cella è diventato un potente strumento per comprendere le dinamiche di MOA antifungina della pianta defensine. Qui, un metodo di imaging microscopia confocale basato su cellule vive è descritti tramite due pianta fluorescente contrassegnati defensine (MtDef4 e MtDef5) in combinazione con le tinture fluorescenti vitale. Questa tecnica consente la visualizzazione in tempo reale e analisi degli eventi dinamici dell'internalizzazione di MtDef4 e MtDef5 nelle cellule fungine. D'importanza, questo test genera una ricchezza di informazioni, compresa interiorizzazione cinetica, modalità di entrata e localizzazione subcellulare di questi peptidi. Insieme ad altri strumenti biologici delle cellule, questi metodi hanno fornito approfondimenti critici la dinamica e la complessità di MOA di questi peptidi. Questi strumenti è utilizzabile anche per confrontare il MOA di questi peptidi contro funghi differenti.
Introduzione
Le piante hanno sviluppato un sofisticato sistema immunitario innato per difesa contro agenti patogeni microbici pianta1. Essi esprimono numerosi peptidi di difesa ospite codificato dal gene con attività antimicrobica presunta2. Infatti, molti di questi peptidi visualizzare attività antimicrobica in vitro3. Defensine costituiscono uno dei più grandi gruppi di peptidi di difesa ospite in pianta Unito4. Questi peptidi ricchi di cisteina, cationici presentano attività inibitoria della crescita potente contro fungine e Oomycota patogeni alle concentrazioni micromolar e rappresentano una delle prime linee di difesa contro questi agenti patogeni5,6. A causa della loro potente attività antifungina, defensine possono essere sfruttate in agribiotechnological applicazioni per generare colture resistenti alle malattie. La sovraespressione costitutiva delle diverse piante defensine è stata indicata per migliorare la resistenza alle malattie nei test serra e campo di colture transgeniche6. È importante definire i meccanismi di azione (MOA) di questi peptidi antimicotici al fine di sfruttare pienamente il loro potenziale come strumenti efficaci per la protezione delle colture. Tuttavia, il MOA delle defensine vegetali è capito male. La prova corrente suggerisce che essi presentano diversi MOA5,6,7,8. Alcuni defensine agiscono extracellularly sui funghi e destinazione particolare parete cellulare/plasma membrana residente sfingolipidi, compromettere l'integrità della membrana e attivare tossicità cellulare vie9,10,11. Recentemente, tuttavia, defensine antifungine che traslocano nelle cellule fungine sono stati scoperti12,13,14. Alcuni di questi defensine associare a membrana-residente bioattivi fosfolipidi, formano complessi oligomerici e permeabilize membrane plasmatiche15,16,17. Così, alcuni aspetti di MOA delle defensine pianta sono stati delucidati. Tuttavia, il MOA delle defensine pianta probabilmente coinvolge una serie complessa di eventi che non ancora sono stati identificati e integrati in un modello globale. In particolare, ci rimane una grave lacuna nella nostra comprensione dei bersagli cellulari di questi peptidi.
Con gli avanzamenti recenti nella microscopia tecnologie e lo sviluppo di nuove sonde fluorescenti, tecniche di imaging di cellule vive ora sono frequentemente utilizzati per studiare il MOA di peptidi antimicrobici (amp). Queste tecniche completano metodi ampiamente usati come immunolocalizzazione, microscopia elettronica, microscopia a forza atomica o di tomografia a raggi x18, che sono stati impiegati principalmente per analizzare gli effetti dei peptidi antimicotici sulla morfologia e crescita delle cellule fungine, compreso lo studio dell'integrità della parete cellulare, alterazioni nel pattern di crescita/ramificazione di celle, come pure la permeabilizzazione della membrana del plasma e uccidendo. Tuttavia, questi studi sono stati limitati a imaging cellule ad un certo punto di tempo dopo il trattamento con i peptidi anziché eseguire time-lapse imaging sulle stesse cellule per monitorare i cambiamenti dinamici in risposta alla sfida di defensine. Negli ultimi anni, uso di peptidi fluorescente contrassegnati in collaborazione con live cell imaging mediante microscopia confocale ha permesso la visualizzazione in tempo reale della dinamica delle interazioni AMP – microbo. Entrambi naturalmente purificata e peptidi antimicotici chimicamente sintetizzati possono essere contrassegnate con etichette fluorescenti (ad es., DyLight, rodamina, BODIPY o Alexa Fluor basato coloranti) e osservato direttamente durante la loro interazione con le cellule da Time-lapse formazione immagine di cellule vive. L'uso di questi peptidi con l'etichetta ha aumentato significativamente la nostra comprensione dei diversi aspetti del loro MOA inclusa la modalità di ingresso, localizzazione subcellulare, traffico intracellulare e siti d'azione antifungina nelle cellule fungine viventi 18.
Recentemente, parecchi studi hanno indicato che vari peptidi antimicotici compreso impianto defensine sono interiorizzati da vivere le cellule fungine12,14,19,20. Il MOA di questi defensine probabile che comportano interazioni con obiettivi intracellulari. Recentemente abbiamo segnalato l'azione antifungina di un impianto di defensine MtDef4 in due funghi ascomiceti, Neurospora crassa e Fusarium graminearum. MtDef4 è stato indicato per utilizzare diverse vie per l'entrata delle cellule fungine e localizzazione subcellulare in questi funghi14. Questo studio usato chimicamente sintetizzato tetrametil rodamina (TAMRA)-con l'etichetta MtDef4 in combinazione con le tinture fluorescenti vitale (la tintura di membrana selettiva, FM4-64; tintura membrana-permeante, verde SYTOX; il colorante reporter di morte delle cellule, ioduro di propidio) e inibitori metabolici. Queste analisi hanno dimostrato la cinetica di interiorizzazione dei MtDef4, i suoi meccanismi di trasporto intracellulare e suoi obiettivi sottocellulari14.
Qui, un protocollo per l'imaging di cellule vive usando la microscopia confocale è presentato. Il protocollo utilizza fluorescente identificati peptidi in combinazione con coloranti vitali fluorescenti per studiare le interazioni defensina-fungine di pianta, in particolare, i percorsi di spostamento e i bersagli intracellulari di defensine in cellule fungine.
Protocollo
1. etichettatura delle defensine con fluorofori
- Selezionare un fluoroforo che ha minimo effetto sulla proprietà antimicrobiche, nonché l'assorbimento e la localizzazione delle defensine all'interno della cellula vivente.
Nota: Selezionando il fluoroforo ottima dipende da specifici obiettivi sperimentali. Le proprietà spettrali e chimica, fotostabilità, dimensioni e carica del fluoroforo dovrebbe anche essere considerate. - Etichetta defensine con il fluoroforo selezionato utilizzando un peptide appropriato etichettatura kit disponibili dai fornitori commerciali. In alternativa, di sintetizzare chimicamente fluoroforo defensine. In questo studio, etichetta MtDef5 defensine con DyLight550 usando un'etichettatura commerciale kit secondo rodamina di protocollo e tetrametil del produttore (TAMRA)-con etichetta MtDef4 defensine è stata sintetizzata chimicamente commercialmente.
Nota: È consigliabile che i peptidi sintetizzati chimicamente fluoroforo taggato (soprattutto per brevi peptidi di 50 aminoacidi o meno) essere usato poiché il fluoroforo può essere collegato al residuo N - o C-terminale del peptide per garantire il minimo effetto sulla sua attività antimicrobica. Sintesi di peptidi lunghi con più di tre ponti disolfuro sono impegnativo. In questo caso, è consigliabile che il peptide essere etichettato con un fluoroforo ottima usando un adatto peptide disponibile in commercio kit di etichettatura. - Test in vitro l'attività antimicrobica della defensina etichettati e determinare la concentrazione inibitoria minima (MIC) contro i funghi per essere studiato.
Nota: In questo studio, il MIC di DyLight550-MtDef5 e TMR-MtDef4 era risoluto contro Neurospora crassa e Fusarium graminearum.
2. fungine culture e crescita medio
- Trasferimento conidi da una cultura di stock di N. crassa inclinano provetta contenente agar media21 di Vogel e incubare a temperatura ambiente sotto la luce per 5 giorni.
- F. graminearum PH-1 ceppo su piastre contenenti completa medio (CM)22 a 28 ° c per 5 giorni della coltura. Per la produzione di conidi, inoculare 4 spine (10 mm di diametro) di 5 giorno vecchia cultura di F. graminearum PH-1 in 50 mL di carbossimetil cellulosa (CMC) terreno (cellulosa carbossimetilica 15 g, 1 g di Estratto di lievito, 0,5 g MgSO4,7 H2O, 1 g di NH 4 NO3e 1 g KH2PO4) e la cultura per 4-7 giorni a 28 ° c in un agitatore rotativo a 180 giri/min.
3. preparazione della sospensione Conidial
- F. graminearum conidial sospensione
- Vortice la coltura liquida di F. graminearum e raccogliere conidi filtrando il coltura fungosa 1,5 mL attraverso due strati di materiale di filtrazione (come Miracloth) in una provetta da 2 mL microcentrifuga. Centrifugare la sospensione conidial presso 13.226 x velocità g in una microcentrifuga per 2 min.
- Scartare il surnatante e aggiungere 1 mL di acqua sterile per lavare la pallina.
- Centrifugare la sospensione conidial presso 13.226 x velocità g in una microcentrifuga per 2 min. scartare il supernatante e risospendere il pellet in 1 mL di 2 X SFM (sintetica Media fungine)23.
- Contare i conidi utilizzando un emocitometro sotto un microscopio chiaro.
- Regolare le sospensioni conidial a 105 conidi/mL con 2 X SFM.
- N. crassa conidial sospensione
- Trasferire una piccola quantità di cultura crescente (5 giorni) di N. crassa utilizzando un ciclo di inoculazione di un tubo del microcentrifuge contenente 2 mL di terreno liquido di Vogel.
- Vortice della sospensione conidial e filtrare attraverso materiale di filtrazione in un nuovo tubo del microcentrifuge.
- Centrifugare la sospensione conidial alla massima velocità in una microcentrifuga per 2 min. scartare il supernatante e risospendere il pellet conidial in 1 mL di terreno liquido di Vogel.
- Contare i conidi utilizzando un emocitometro sotto un microscopio chiaro.
- Regolare le sospensioni conidial a 105 conidi/mL con mezzo liquido di Vogel.
4. microscopia confocale e preparazione del campione
- Localizzazione subcellulare delle defensine MtDef4 nelle cellule fungine
- Pipettare 50 µ l di ogni sospensione conidial (105 conidi/mL) nel micropozzetto per 10 mm 35 mm fondo microtiter dei piatti di vetro. Per l'osservazione dei conidi, direttamente procedere al passaggio successivo, o per la preparazione di germlings, pipettare 50 µ l di N. crassa e F. graminearum conidi in piastre di coltura di 35mm e far germinare per 3-6 h a temperatura ambiente.
- Aggiungere 50 µ l di defensine fluorescente contrassegnati al MIC per ogni piatto di pozzetti e incubare per 2,5 h. Il MIC di defensine fluoroforo utilizzate qui (punto 1.3) è 3 µM per N. crassa e F. graminearum e il MIC di TMR-MtDef4 per N. crassa e F. graminearum era 1 µM e 12 µM, rispettivamente.
- Aggiungere 2 µ l di membrana selettiva tingere FM4-64 (concentrazione finale: 5 µM) nella cultura piatto e incubare per 30 min e montare immediatamente il microscopio confocale per l'imaging.
- Selezionare il laser a luce bianca (WLL). Utilizzare il due laser fonti 488 nm e 550 nm per eccitare tintura TMR-MtDef4 e FM4-64, rispettivamente. Rilevare la fluorescenza di TMR-MtDef4 a 580-700 nm e rilevare FM4-64 tintura a 690-800 nm.
Nota: Eseguire la microscopia confocale in camera oscura.
- Formazione immagine di lasso di tempo di interiorizzazione delle defensine MtDef5
- Pipettare 50 µ l di sospensione conidial N. crassa e F. graminearum (105 conidi/mL) in micropozzetti 10 mm 35 mm fondo microtiter dei piatti di vetro. Micropiastra 10mm è non rivestito e ha un vetro di copertura n. 1.5 nella parte inferiore. Per l'osservazione dei conidi, procedere direttamente al passaggio successivo. Per l'osservazione di germlings, incubare conidi per 3-6 h a temperatura ambiente prima di procedere con la microscopia.
- Accendere il WLL. Selezionare la linea di laser a 550 nm per eccitare il fluoroforo defensine usate qui (punto 1.3) e FM4-64 con 1,00% intensità e attivare i sensori corrispondenti. Le defensine fluoroforo utilizzata qui (punto 1.3) è stata rilevata a 560-600 nm e FM4-64dye è stata rilevata a 690-800 nm.
Nota: Utilizzare laser a bassa intensità per live cell imaging come intensità del laser alta può causare danni alle cellule fungine dal vivo. - Montare il piatto microtiter sul microscopio. Trovare le celle utilizzando 10 x obiettivi inizialmente, poi passare a x 100 / 1,44 obiettivo di olio per ingrandimenti maggiori.
Nota: Impostare la modalità di scansione xyzt (combinazione di modalità Time-lapse e Z-stack), posizione Z, Zoom, frequenza di acquisizione di immagini, ecc.
Nota: Aggiunta di defensine e fluoroforo dopo montaggio microtiter piatto nel microscopio può sfocare la regione di interesse. Pertanto, dopo l'aggiunta di defensine e fluoroforo alla striscia di micropozzetti, area di interesse deve essere riorientato che può causare ritardo nell'acquisizione delle immagini.
Risultati
Live imaging cellulare è stato effettuato per monitorare e confrontare l'internalizzazione e la localizzazione subcellulare delle due defensine, MtDef4 e MtDef5, da Medicago truncatula; in cellule fungine. TMR-MtDef4 è stato sintetizzato chimicamente mentre MtDef5 fu etichettata con Dylight550 (Dylight550-MtDef5). Conidi sono state incubate con entrambi defensine in combinazione con la tintura di membrana selettiva FM4-64. La figura 1 Mostra che TMR-MtDef4 propone diverse vie di traffico N. crassa rispetto al F. graminearum. Nel N. crassa, FM4-64 non co-localizza con la defensina ma piuttosto macchie le membrane dei vacuoli all'interno del quale le defensine è sequestrata. In F. graminearum, d'altra parte, TMR-MtDef4 non è localizzata all'interno di qualsiasi specifica membrana associato organelli ma è diffuso nel citoplasma (Figura 1).
Imaging di lasso di tempo di N. crassa cellule identificate con spettacoli di FM4-64 che le defensine è in grado di entrare nelle cellule fungine entro 30-40 min di trattamento (Figura 2) sia il fluoroforo defensine usate qui (punto 1.3). Al momento dell'entrata nelle cellule, le defensine fluoroforo utilizzata qui (punto 1.3) non localizzata all'interno di qualsiasi specifico organello ma prontamente diffonde nel citoplasma. Questo è in contrasto con TMR-MtDef4 che entra nelle cellule N. crassa e rimane intrappolato all'interno dei corpi vescicolari anche dopo 3 ore di trattamento (Figura 1).
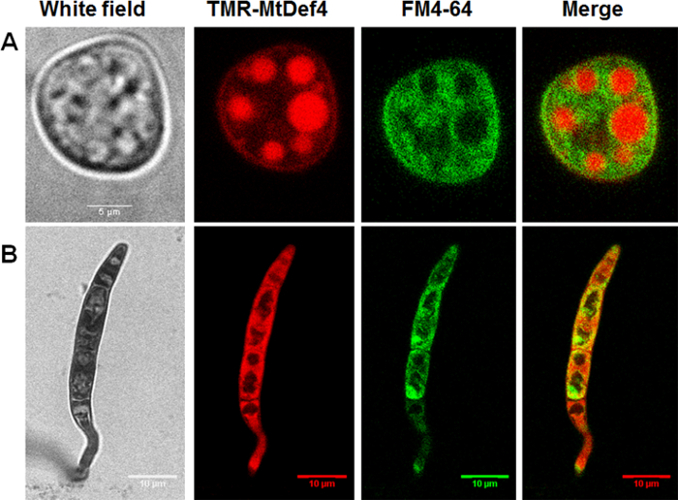
Figura 1: MtDef4 propone diverse vie di traffico N. crassa e F. graminearum. TMR-MtDef4 si localizza nei corpi vescicolari in N. crassa (A) ma è diffuso nel citoplasma di F. graminearum (B). Conidi di N. crassa e F. graminearum erano co-etichettati con 1 µM e 12 µM TMR-MtDef4 (rosso), rispettivamente e con FM4-64 (verde). Immagini sono state scattate dopo 3 ore di trattamento. Clicca qui per visualizzare una versione più grande di questa figura.
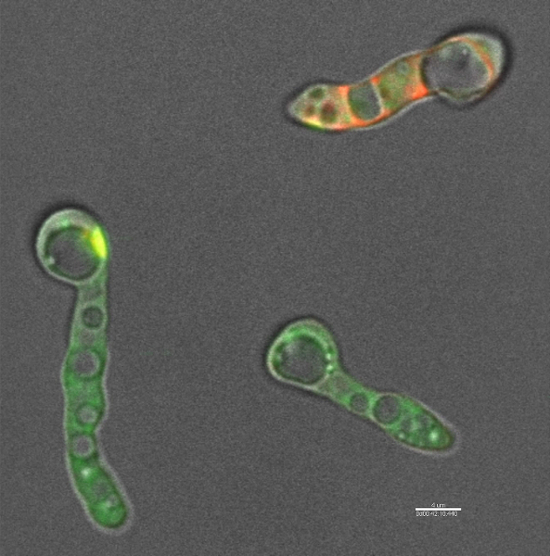
Figura 2: MtDef5 è interiorizzato dal N. crassa e si diffonde all'interno delle cellule. DyLight550-MtDef5 è interiorizzato nelle cellule N. crassa e diffonde nel citoplasma. N. crassa cellule co-sono state etichettate con 3 µM, DyLight550-MtDef5 (rosso) e FM4-64 (verde). Video è stato registrato per 2 h e 30 min. Il ritardo tra l'aggiunta di MtDef5 e a partire di acquisizione di immagini è stato 5 min scala bar = 4 µm. per favore clicca qui per visualizzare questo video. (Tasto destro per scaricare.)
Discussione
In questo studio, una metodologia di imaging affidabile cellule vive con l'uso delle defensine antifungine fluorescente identificate è stato descritta per studiare la cinetica di interiorizzazione di questi peptidi in cellule fungine e per determinare i loro obiettivi sottocellulari. Questo metodo è un potente strumento per visualizzare le dinamiche dell'interazione tra cellule fungine e defensine temporalmente e spazialmente.
Vari metodi sono stati usati per studiare l'internalizzazione e la localizzazione intracellulare della pianta defensine in cellule fungine. In questi metodi, cellule defensina-trattate sono solitamente fisso e poi trattate per immunolocalizzazione, microscopia elettronica o raggi x tomografia15,24,25. Inoltre, la maggior parte di queste tecniche sono stata limitata a cellule a un momento specifico, piuttosto che in tempo reale usando la formazione immagine time-lapse delle stesse cellule viventi per monitorare i cambiamenti dinamici che si svolgono in risposta alla sfida di defensine di imaging. La possibilità di visualizzare, analizzare e confrontare gli eventi dinamici che si svolgono nelle cellule fungine in tempo reale durante l'azione antifungina di defensine rende questa tecnica efficace ed emozionante. Inoltre, fornisce migliore comprensione di come la localizzazione intracellulare dinamica del peptide influisce la morfogenesi di singole cellule fungine con il tempo.
Uno degli aspetti importanti di questo metodo consiste nel determinare gli obiettivi sottocellulari usando i peptidi fluorescente contrassegnati con coloranti fluorescenti vitali (ad es. FM4-64; SG). Localizzazione sottocellulare determina l'ambiente in cui opera un peptide e rappresenta un passo importante per chiarire la sua partner di interazione, funzione e potenziali ruoli nel macchinario cellulare26,27.
Un limite minore di questa tecnica è che il peptide fluorescente esibisce spesso ridotta attività antifungina, confrontato con il peptide senza etichetta. Se il peptide è associata all'etichetta un kit etichettatura peptide commercialmente disponibili e Mostra la perdita completa di attività antifungina, è consigliato un peptide sintetizzato chimicamente etichettato con un fluoroforo piccolo al suo N - o C-terminale.
Nella cella Riepilogo, vivo imaging delle cellule fungine sfidato con un fluoroforo-etichetta defensine è uno strumento efficace che può fornire diretto, ad alta risoluzione spazio-temporale di MOA di un antimicotico defensine. Per studio MOA più preciso, questa tecnica può essere combinata con durata di fluorescenza imaging microscopia (FLIM)28 , che vi permetterà di misurare la cinetica di interazione di un peptide con altri peptidi, o con altre molecole con etichettate o cellulare costituenti in tempo reale. Ciò arricchirà la nostra comprensione dei modi peptidi antimicrobici lavorano quindi accelerare il loro sviluppo come agenti antifungini per uso nell'agricoltura e medicina.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Si ringraziano il Dr. R. Howard Berg, direttore del centro di microscopia integrato presso il Donald Danforth Plant Science Center, per la sua guida e aiutare con la microscopia confocale. Gli autori non hanno alcun conflitto di interessi per dichiarare.
Materiali
| Name | Company | Catalog Number | Comments |
| FM4-64 Dye | Life Technologies | T13320 | |
| DyLight 550 Antibody Labeling Kit | Thermo Scientific | 84530 | |
| Glass Bottom Microwell Dishes | Mat TeK | P35G-1.5-10-C | |
| Mira cloth | EMD Millipore Corp | 475855-1R | |
| SP8-X confocal microscope | Leica | ||
| ImageJ software | FiJi | For Image analysis | |
| Imaris software | Bitplane | For Image analysis |
Riferimenti
- Jones, J. D. G., Dangl, J. L. The plant immune system. Nature. 444 (7117), 323-329 (2006).
- Tavormina, P., De Coninck, B., Nikonorova, N., De Smet, I., Cammue, B. P. A. The Plant Peptidome: An expanding repertoire of structural features and biological functions. Plant cell. 27 (8), 2095-2118 (2015).
- Van Der Weerden, N. L., Bleackley, M. R., Anderson, M. A. Properties and mechanisms of action of naturally occurring antifungal peptides. Cell. and Mol. Life Sci. 70 (19), 3545-3570 (2013).
- Van der Weerden, N. L., Anderson, M. A. Plant defensins: Common fold, multiple functions. Fungal Biol Rev. 26 (4), 121-131 (2013).
- De Coninck, B., Cammue, B. P. A., Thevissen, K. Modes of antifungal action and in planta functions of plant defensins and defensin-like peptides. Fungal Biol Rev. 26 (4), 109-120 (2013).
- Kaur, J., Sagaram, U. S., Shah, D. Can plant defensins be used to engineer durable commercially useful fungal resistance in crop plants?. Fungal Biol. Rev. 25 (3), 128-135 (2011).
- Vriens, K., Cammue, B. P. A., Thevissen, K. Antifungal plant defensins: Mechanisms of action and production. Molecules. 19 (8), 12280-12303 (2014).
- Sagaram, U. S., Kaur, J., Shah, D. Antifungal plant defensins: Structure-activity relationships, modes of action, and biotech applications. ACS Symp. Ser. 1095, 317-336 (2012).
- Thevissen, K., Francois, I. E. J. A., Aerts, A. M., Cammue, B. P. A. Fungal sphingolipids as targets for the development of selective antifungal therapeutics. Curr. Drug Targets. 6 (8), 923-928 (2005).
- Thevissen, K., Kristensen, H. H., Thomma, B. P. H. J., Cammue, B. P. A., Francois, I. E. J. A. Therapeutic potential of antifungal plant and insect defensins. Drug Discov. Today. 12 (21-22), 966-971 (2007).
- Aerts, A. M., François, I. E. J. A., Cammue, B. P. A., Thevissen, K. The mode of antifungal action of plant, insect and human defensins. Cell. Mol. Life Sci. 65 (13), 2069-2079 (2008).
- Van Der Weerden, N. L., Lay, F. T., Anderson, M. A. The plant defensin, NaD1, enters the cytoplasm of Fusarium oxysporum hyphae. J. Biol. Chem. 283 (21), 14445-14452 (2008).
- Lobo, D. S., Pereira, I. B., et al. Antifungal Pisum sativum Defensin 1 Interacts with Neurospora crassa Cyclin F Related to the Cell Cycle. Biochemistry. 46 (4), 987-996 (2007).
- El-Mounadi, K., Islam, K. T., Hernandez-Ortiz, P., Read, N. D., Shah, D. M. Antifungal mechanisms of a plant defensin MtDef4 are not conserved between the ascomycete fungi Neurospora crassa and Fusarium graminearum. Mol. Microbiol. 100 (3), 542-559 (2016).
- Baxter, A. A., et al. The Tomato Defensin TPP3 Binds Phosphatidylinositol (4,5)-Bisphosphate via a Conserved Dimeric Cationic Grip Conformation To Mediate Cell Lysis. Mol. and Cell. Biol. 35 (11), 1964-1978 (2015).
- Kvansakul, M., et al. Binding of phosphatidic acid by NsD7 mediates the formation of helical defensin-lipid oligomeric assemblies and membrane permeabilization. Proc. Natl. Acad. Sci. 113, 11202-11207 (2016).
- Poon, I. K. H., et al. Phosphoinositide-mediated oligomerization of a defensin induces cell lysis. eLife. 3, e01808 (2014).
- Muñoz, A., Read, N. D. Live-cell imaging and analysis shed light on the complexity and dynamics of antimicrobial Peptide action. Front. Immunol. 3, 248 (2012).
- Hayes, B. M. E., et al. Identification and mechanism of action of the plant defensin NaD1 as a new member of the antifungal drug arsenal against candida albicans. Antimicrob. Agents Chemother. 57 (8), 3667-3675 (2013).
- Muñoz, A., Marcos, J. F., Read, N. D. Concentration-dependent mechanisms of cell penetration and killing by the de novo designed antifungal hexapeptide PAF26. Mol. Microbiol. 85 (1), 89-106 (2012).
- Eaton, C. J., et al. The guanine nucleotide exchange factor RIC8 regulates conidial germination through Gα proteins in Neurospora crassa. PLoS One. 7 (10), e48026 (2012).
- Leslie, J. F., Summerell, B. A. . The Fusarium. laboratory manual. , (2006).
- Broekaert, W. F., Terras, F. R. G., Cammue, B. P. A., Vanderleyden, J. An automated quantitative assay for fungal growth inhibition. FEMS Microbiol.Lett. 69 (1-2), 55-59 (1990).
- Sagaram, U. S., et al. Structural and functional studies of a phosphatidic acid-binding antifungal plant defensin MtDef4: Identification of an RGFRRR motif governing fungal cell entry. PLoS One. 8 (12), 1-22 (2013).
- Uchida, M., et al. Soft X-ray tomography of phenotypic switching and the cellular response to antifungal peptoids in Candida albicans. Proc. Natl. Acad. Sci. 106 (46), 19375-19380 (2009).
- Nair, R., et al. Better prediction of sub-cellular localization by combining evolutionary and structural information. Proteins Struct. Funct. Bioinform. 53 (4), 917-930 (2003).
- Scott, M. S., Calafell, S. J., Thomas, D. Y., Hallett, M. T. Refining protein subcellular localization. PLoS Comput. Biol. 1 (6), e66 (2005).
- Shagaghi, N., Bhave, M., Palombo, E., Clayton, A. Revealing the sequence of interactions of PuroA peptide with Candida albicans cells by live-cell imaging. Sci. Rep. 7, 43542 (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon