Method Article
Le proteine immunofluorescenza di endogeni ed esogeni Centromere-cinetocoro
In questo articolo
Riepilogo
Here we report protocols to detect endogenous and exogenous centromere-kinetochore proteins in human cells and quantify these protein levels at centromeres-kinetochores by indirect immunofluorescent staining through the use of fixation (paraformaldehyde, acetone, or methanol fixation).
Abstract
"Centromeres" and "kinetochores" refer to the site where chromosomes associate with the spindle during cell division. Direct visualization of centromere-kinetochore proteins during the cell cycle remains a fundamental tool in investigating the mechanism(s) of these proteins. Advanced imaging methods in fluorescence microscopy provide remarkable resolution of centromere-kinetochore components and allow direct observation of specific molecular components of the centromeres and kinetochores. In addition, methods of indirect immunofluorescent (IIF) staining using specific antibodies are crucial to these observations. However, despite numerous reports about IIF protocols, few discussed in detail problems of specific centromere-kinetochore proteins.1-4 Here we report optimized protocols to stain endogenous centromere-kinetochore proteins in human cells by using paraformaldehyde fixation and IIF staining. Furthermore, we report protocols to detect Flag-tagged exogenous CENP-A proteins in human cells subjected to acetone or methanol fixation. These methods are useful in detecting and quantifying endogenous centromere-kinetochore proteins and Flag-tagged CENP-A proteins, including those in human cells.
Introduzione
"I centromeri" sono stati classicamente definiti come regioni della ricombinazione meiotica soppressa nel campo della genetica e in seguito riconosciuto come la costrizione primaria dei cromosomi mitotici, che svolge un ruolo essenziale nel accurata segregazione dei cromosomi durante la mitosi. "cinetocori" sono stati descritti come strutture multistrato che si legano ai microtubuli alla superficie di centromeri, come rivelato dalla microscopia elettronica; "cinetocori" sono stati successivamente definiti come il complesso macromolecolare che localizza al centromero dei cromosomi mitotici. Nonostante una divergenza drammatica di sequenze di DNA centromeriche tra i vertebrati, la struttura e la composizione cinetocoro sono altamente conservati. Una interazione dinamica tra microtubuli del fuso e il cinetocore è necessario per la separazione fedele dei cromosomi durante la mitosi, e difetti di centromero-cinetocore piombo funzione per aneuploidie e quindi il cancro.
Il centromero nella maggior parte dei eucariotisenza sequenza di DNA definito, ma si compone di grandi array (0,3-5 MB) di DNA ripetitivo alphoid costituito da DNA α-satellite 171-bp. Salvo il lievito, identità centromero non è raggiunto dalla sequenza di DNA, ma per la presenza di un nucleosoma speciale che contiene la variante istone H3 CenH3 (centromero Proteina A [CENP-A] nell'uomo). 5 CENP-A nucleosomi localizzare la piastra interna di cinetocori mammiferi 7 e si legano al DNA α-satellite 171 bp. centromeri attivi richiedono CENP-A-contenente nucleosomi per dirigere l'assunzione di una rete costitutiva centromero-associato (Ccan) e le proteine del cinetocore, che insieme regolano l'attaccamento di cromosomi al fuso mitotico e la progressione del ciclo successivo attraverso il posto di blocco mandrino.
Alla luce degli elementi di cui sopra, CENP-A è stato proposto per essere il marchio epigenetica del centromero 8; Tuttavia, il processo con cui CENP-A è incorporato into DNA centromerico e dei fattori responsabili di questa incorporazione non sono ancora stati ben caratterizzati. Una breve dominio centromero-targeting (CATD) risiede nel istone piegare regione CENP-A, e la sostituzione della corrispondente regione H3 con il CATD è sufficiente H3 al centromero. 9 Diversi studi suggeriscono ruoli funzionali per post-traduzionale modifica (PTM) di CENP-A 12-16; Tuttavia, i meccanismi molecolari di queste PTM di CENP-A nel reclutamento di centromeri non sono ancora stati chiariti. Abbiamo precedentemente riportato che CUL4A-RBX1-COPS8 E3 ligasi attività è necessario per CENP-A K124 ubiquitinazione e la localizzazione di CENP-A per centromeri. 17
La scoperta e la caratterizzazione di proteine cinetocoro hanno portato a una nuova visione per quanto riguarda la segregazione dei cromosomi. 18 Più di 100 componenti cinetocoro sono stati identificati in cellule dei vertebrati dai vari approcci. 19,20 un sottoIn piedi di come cinetocori assemblare e la funzione viene anche dalla caratterizzazione delle funzioni cellulari di ciascuna rete centromero-cinetocore proteine e proteine-proteine all'interno delle cellule. 19 visualizzazione diretta e di imaging avanzate metodologie di microscopia a fluorescenza forniscono notevole risoluzione di componenti centromero-cinetocore e permettono osservazione diretta di componenti molecolari specifici dei centromeri e cinetocori. Inoltre, i metodi di immunofluorescenza indiretta (IIF) colorazione utilizzando anticorpi specifici sono fondamentali per queste osservazioni. Tuttavia, nonostante le numerose segnalazioni circa i protocolli di IIF, pochi problemi discussi in dettaglio di specifiche proteine centromero-cinetocore. 1-4 Così, lo sviluppo e la segnalazione metodi di IIF colorazione e un test quantitativo IIF per analizzare specificamente ogni proteina centromero-cinetocore è estremamente importante. In IIF colorazione, si deve procedere con il protocollo di colorazione per evitare la perdita della proteina di interesse oil resto della cellula. Tuttavia, la fissazione distrugge siti antigenici occasionalmente e diverse combinazioni anticorpo-antigene funziona male con un fissativo, ma molto bene con un altro, 21 e la scelta di fissativo dipende in gran parte della proteina (s) di interesse. Pertanto, diversi metodi di fissativi sono cruciali per IIF colorazione delle proteine centromero-cinetocore.
sono stati sviluppati metodi qui ottimizzate di immunofluorescenza indiretta (IIF) colorazione e un test per affrontare la localizzazione delle proteine centromero-cinetocore endogeni, tra cui CENP-A e esogeni proteine CENP-A-Flag con tag, e la quantificazione di queste proteine nelle cellule umane. Questi metodi possono essere applicati all'analisi delle proteine centromero-cinetocore in altre specie.
Protocollo
1. Colture cellulari e trasfezione
- Mettere un vetro di copertura (22 mm x 22 mm) in una lastra di polistirene 6-bene. cappotto Facoltativamente un vetro di copertura con Poli-L-lisina, 0,1% w / v, in acqua (vedi elenco dei materiali / attrezzature) per mantenere le cellule in mitosi sul vetro di copertura seguendo le istruzioni riportate di seguito:
Nota: Le condizioni ottimali devono essere determinati per ciascuna linea cellulare ed applicazione.- Asetticamente superficie cultura cappotto con poli-L-lisina, 0,1% w / v, in acqua (0,4 ml / pozzetto di una piastra da 6 pozzetti polistirene). Agitare delicatamente per garantire anche il rivestimento della superficie di cultura.
- Dopo 5 minuti, rimuovere la soluzione tramite aspirazione e lavare accuratamente la superficie con del tessuto acqua sterile cultura di grado.
- Secco, almeno 2 ore prima di introdurre cellule e mezzo.
- Cellule HeLa Seed 17 o cellule 17,22 su un vetro di copertura (22 mm x 22 mm) mettere in una lastra di polistirene 6-ben HeLa Tet-Off. Controllare che la densità cellulare è 5,4 x 10 5 per pozzetto. cellule di colturain DMEM ad alto glucosio con 10% FBS e 1% di penicillina-streptomicina.
Nota: Per ottenere risultati ottimali, empiricamente determinare la densità delle cellule da utilizzare in semina. - Incubare le cellule a 37 ° C in atmosfera al 5% di CO 2 per 18 ore.
Nota: Nel caso di cellule HeLa Tet-Off, la trascrizione del gene esogeno è attiva in assenza di induttore (cioè, tetraciclina / doxiciclina) quindi cellule di coltura senza tetraciclina / doxiciclina e trasfezione transiente con la sovraespressione vettoriale pTRM4 (Tabella 2 ), la cui trascrizione è regolata dal promotore TRE (vedi sotto). - Diciotto ore dopo la semina delle cellule, trasfezione come segue:
- Fai la soluzione A mescolando 1,5 microlitri siRNA oligo (20 micron azionari ricotto; Tabella 1) e / o 2.0 mcg plasmide (Tabella 2) in 50 microlitri ridotto medio sierico (vedere elenco dei materiali / attrezzature), e incubare a temperatura ambiente per 5 min .
Nota: In questa analisi,CA-UTR siRNA (una miscela di 5 'e 3' UTR siRNA; Tabella 1) sono state co-trasfettate per l'utilizzo in protocolli 3 e 4 (vedi la discussione). - Fai la soluzione B mescolando 0,75 ml trasfezione reagente I (vedere l'elenco dei materiali / Equipment) in 50 microlitri riduzione media di siero, e incubare a temperatura ambiente per 5 min.
Nota: un passaggio facoltativo è l'aggiunta di 1,0 ml di reagente di trasfezione II (vedi elenco dei materiali / attrezzature). - Mescolare le soluzioni A e B insieme, e incubare a temperatura ambiente per 15 min.
- Lavare le cellule coltivate una volta con PBS, e quindi aggiungere 500 microlitri ridotte medio siero in ciascun pozzetto della piastra di polistirene 6 pozzetti. Aggiungere la miscela delle soluzioni A e B (cioè, RNA e / o complesso DNA-lipide) direttamente a ciascuno dei singoli bene.
Nota: la concentrazione finale è di 3,3 mg / ml (plasmidi); 50 Nm (siRNA). - Incubare le cellule a 37 ° C in atmosfera al 5% di CO 2 per 4,5 ore. Cambiare il medio-alta-glucosio DMEM ingegnoh 10% FBS e 1% di penicillina-streptomicina.
- Incubare le cellule a 37 ° C in atmosfera al 5% di CO 2 per 48-72 ore dopo la trasfezione.
Nota: Per ottenere risultati ottimali, il periodo di incubazione per la crescita cellulare prima della fissazione deve essere determinato empiricamente, e l'esaurimento di proteine e / o di espressione deve essere confermata da analisi Western Blot (vedi Protocollo 6). - Se l'analisi delle cellule mitotico è di interesse, aggiungere paclitaxel (10 nM) a cellule in coltura 24 ore prima della fissazione, o aggiungere TN16 (0,5 micron) al cellule in coltura 2,5 ore prima della fissazione.
- Fai la soluzione A mescolando 1,5 microlitri siRNA oligo (20 micron azionari ricotto; Tabella 1) e / o 2.0 mcg plasmide (Tabella 2) in 50 microlitri ridotto medio sierico (vedere elenco dei materiali / attrezzature), e incubare a temperatura ambiente per 5 min .
2. cellulare Fissazione e colorazione di immunofluorescenza per rilevare proteine endogene centromero-cinetocore (Paraformaldeide fissazione)
- Preparazione di fissativo, tamponi e reagenti.
- Appena preparare 50 ml di soluzione di paraformaldeide al 4% in PBS, pH 7,4.
- Aggiungere 40 ml di 1 × PBS in un becher di vetro su un piatto mescolare in una cappa ventilata. Calore mentre Stirring a circa 60 ° C. Aggiungere 2 g di polvere di paraformaldeide al PBS riscaldata.
- Sollevare lentamente il pH aggiungendo 1 ml di 1 N NaOH in totale, perché la polvere non sciogliere immediatamente.
Nota: La soluzione cancella dopo l'aggiunta di NaOH. - Una volta paraformaldeide si è dissolta, fresco e filtrare la soluzione.
- Regolare il pH con 1 N HCl a pH 7.4 e il volume della soluzione con 1 × PBS a 50 ml.
Nota: Aliquote della soluzione possono essere congelati o conservati a 2-8 ° C per il tempo 1 settimana. La soluzione deve essere ghiacciata o conservato a 4 ° C fino a quando non deve essere utilizzato.
- Preparare 50 ml di tampone KB1, che consiste di 10 mM Tris-HCl, pH 7.5; 150 mM NaCl; 0,5% BSA; e 0,5% Triton X-100.
Nota: BSA dovrebbe essere rinnovato aggiunto.
Nota: Il buffer KB serie sono basate sul buffer KB descritto nelle precedenti relazioni 1,2,4. - Preparare 50 ml di tampone KB2, che consiste di 10 mM Tris-HCl,pH 7.5; 150 mM NaCl; e 0,5% BSA.
Nota: BSA dovrebbe essere rinnovato aggiunto. - Preparare 1 ml di tampone contenente KB3 DAPI (50 ng / ml).
- Preparare 100 ml di mezzo di montaggio, che consiste di 1 mg / ml p-fenilendiammina; 10% PBS, e il 90% glicerolo.
- Regolare il pH di 1 × PBS a 9,8 con NaOH 1 N, sciogliere p-fenilendiammina nella soluzione e quindi aggiungere glicerolo.
- Conservare 1 ml aliquote a -80 ° C. Proteggere dalla luce.
- Appena preparare 50 ml di soluzione di paraformaldeide al 4% in PBS, pH 7,4.
- Rimuovere il terreno di coltura per aspirazione a 48-72 ore dopo la trasfezione (vedi protocollo 1) per la fissazione delle cellule. Sciacquare le cellule una volta con PBS. Applicare PBS al lato dei pozzetti di coltura per non disturbare la superficie delle cellule.
Nota: Il punto di tempo ottimale per la fissazione delle cellule deve essere determinata empiricamente. - Fissare le cellule in 4% paraformaldeide in PBS per 30 minuti a 4 ° C. Lavare le cellule due volte con tampone KB2 rimuovere sufficientemente residuo 4% paraformaldeide.
- permeabilitàlize i campioni in tampone KB1 per 30 minuti a RT. Lavare le cellule una volta con tampone KB2, e aggiungere tampone KB2 per 5 minuti a RT per bloccare i siti supplementari di legami non specifici.
Nota: Buffer KB1 contribuisce in modo significativo al blocco. - Rimuovere un vetro di copertura (22 mm x 22 mm) da una lastra di polistirene 6-bene con pinze. Utilizzando una penna barriera idrofobica (vedi elenco dei materiali / attrezzature), disegnare un quadrato o un cerchio verde pallido per formare una barriera idrofobica attorno ad ogni campione di vetro di copertura. Non toccare o avvicinarsi troppo alle cellule con la penna di barriera idrofoba. Mettere il vetro di copertura in nuova lastra di polistirene 6-bene.
- Diluire un anticorpo primario per centromero o proteine kinetochore (rapporto di diluizione di 1: 100 a 1: 200; vedi anche elenco dei materiali / attrezzature) e sia un anticorpo anti-CENP-B (rapporto di diluizione di 1: 400) o un anti anticorpo centromero (ACA) (rapporto di diluizione di 1: 2000) come marcatore di posizione centromero in tampone KB2.
Nota: Per ottenere risultati ottimali, la concentrazione finalezione dell'anticorpo primario in questa soluzione deve essere determinato empiricamente. - Applicare un volume sufficiente (circa 30 ml) di anticorpo primario diluito immergere il campione cellulare. Incubare il campione di cellule per 1 ora a 37 ° C. Lavare le cellule 3 volte con tampone KB2.
- Utilizzando tampone KB2, diluire un anticorpo secondario fluoroforo coniugato (rapporto di diluizione di 1: 100 a 1: 200) diretto contro l'anticorpo primario.
Nota: Per ottenere risultati ottimali, la concentrazione finale di anticorpo secondario in questa soluzione deve essere determinato empiricamente. - Applicare un volume sufficiente (una goccia di circa 30 microlitri sul vetro di protezione) dell'anticorpo secondario diluito immergere il campione cellulare. Incubare il campione di cellule per 1 ora a 37 ° C. Lavare le cellule per 5 volte con tampone KB2 durante un periodo di 30 min (cinque lavaggi 6 min).
Nota: Per risultati ottimali (ad esempio, per una minima perdita di cellule), la condizione di lavaggio ottimale deve essere determinata empirically. - Applicare un volume sufficiente di KB3 tampone contenente DAPI (50 ng / ml) per immergere il campione cellulare. Incubare il campione di cellule per 5 minuti a RT. Lavare le cellule 1-2 volte con tampone KB2.
- Montare il vetro di copertura che contiene il campione di cellule sul micro diapositiva.
- Mettere una goccia di mezzo di montaggio al centro del micro diapositiva.
- Rimuovere il liquido dal campione cellulare e, con le mani o pinze, posizionare il campione nel centro del micro diapositiva. Evitare di bolle d'aria.
- Rimuovere l'eccesso di mezzo di montaggio con un tovagliolo di carta.
3. Fissazione delle cellule e la colorazione di immunofluorescenza di C-terminale Flag-tag CENP-A Proteine (Acetone fissazione)
- Preparazione
- Preparare ghiacciata 75% di acetone.
- Appena preparare 50 ml di PBS (pH 7,4) contenente 0,5% di latte scremato e 0,5% BSA.
- Appena preparare 50 ml di PBS (pH 7,4) contenente 0,1% di latte scremato e 0,1% BSA.
- Preparare 1 ml di PBS (pH 7.4) contenente DAPI (50-100 ng / ml).
- Preparare 100 ml di mezzo di montaggio come descritto in 2.1.5.
- Rimuovere il terreno di coltura per aspirazione a 48-72 ore dopo la trasfezione (vedi protocollo 1) per la fissazione delle cellule. Sciacquare le cellule una volta con PBS. Applicare PBS al lato del bene coltura per non disturbare la superficie delle cellule.
Nota: Il punto di tempo ottimale per la fissazione delle cellule deve essere determinata empiricamente. - Fissare le cellule in ghiaccio freddo 75% di acetone, e incubare le cellule per 10 minuti a -20 ° C. pile a secco sul vetro di copertura in una cappa aspirante per 30-60 minuti a temperatura ambiente.
Nota: Per ottenere risultati ottimali, il tempo necessario per la fissazione e la cella di essiccazione deve essere determinato empiricamente. - Utilizzando la penna barriera idrofoba (vedi elenco dei materiali / attrezzature), disegnare un quadrato o un cerchio verde pallido per formare una barriera idrofobica attorno ad ogni campione di vetro di copertura. Non toccare o avvicinarsi troppo alle cellule con la penna di barriera idrofoba.
- blocco nonspecific siti di legame sulle cellule aggiungendo PBS contenente 0,5% di latte scremato e 0,5% BSA per 5 minuti a RT.
- Utilizzando PBS contenente 0,1% di latte scremato e 0,1% BSA, diluire un anticorpo anti-Flag (1: 1.000 rapporto di diluizione) e sia un anticorpo anti-CENP-B (rapporto di diluizione di 1: 200) o ACA (rapporto 1: 2.000 diluizione ) come un indicatore di posizione centromero.
Nota: Per ottenere risultati ottimali, la concentrazione finale di anticorpo primario in questa soluzione deve essere determinato empiricamente. - Applicare un volume sufficiente (circa 30 ml) di anticorpo primario diluito immergere il campione cellulare. Incubare il campione di cellule per 1 ora a 37 ° C. Lavare le cellule per 5 volte con il tampone di bloccaggio per un periodo di 30 min.
Nota: Le celle possono essere risciacquati con PBS. Per ottenere risultati ottimali (cioè, per evitare la perdita di cellule), le condizioni di lavaggio ottimali devono essere determinati empiricamente. - Diluire un anticorpo secondario fluoroforo coniugato (rapporto di diluizione di 1: 100 a 1: 200) diretto controla ogni anticorpo primario in PBS contenente 0,1% di latte scremato e 0,1% BSA.
Nota: Per ottenere risultati ottimali, la concentrazione finale di anticorpo secondario in questa soluzione deve essere determinato empiricamente. - Applicare un volume sufficiente (circa 30 ml) di anticorpo secondario diluito immergere il campione cellulare. Incubare il campione di cellule per 1 ora a 37 ° C. Lavare le cellule 2 volte con PBS contenente 0,1% di latte scremato e 0,1% BSA.
Nota: PBS solo può anche essere utilizzato per lavare le cellule. - Applicare un sufficiente volume di PBS contenente DAPI (50-100 ng / ml) per immergere il campione cellulare. Incubare il campione di cellule per 5 minuti a RT. Lavare le cellule 1-2 volte con PBS.
- Montare il vetro di copertura contenente il campione cellulare sul micro diapositiva come descritto in 2.11.
4. cellulare Fissazione e colorazione di immunofluorescenza di N-terminale Flag-tag CENP-A Proteine (Metanolo fissazione)
- Preparazione
- Preparare metanolo ghiacciata.
- Preparare TBS (pH 7.4) contenente 4% siero di capra.
- Preparare 1 ml di TBS (pH 7,4) contenente DAPI (50-100 ng / ml).
- Preparare 100 ml di mezzo di montaggio come descritto in 2.1.5.
- Rimuovere il terreno di coltura per aspirazione a 48-72 ore dopo la trasfezione (vedi protocollo 1) per la fissazione delle cellule. Sciacquare le cellule una volta con TBS. Applicare TBS al lato dei pozzetti di coltura per non disturbare la superficie delle cellule.
Nota: Il punto di tempo ottimale per la fissazione delle cellule deve essere determinata empiricamente. - Fissare le cellule in metanolo ghiacciato, e incubare le cellule per 6 minuti a -20 ° C. Lavare le cellule due volte con TBS per rimuovere sufficientemente metanolo residuo.
- Utilizzando una penna barriera idrofobica (vedi elenco dei materiali / attrezzature), disegnare un quadrato o un cerchio verde pallido per creare una barriera idrofobica attorno ad ogni campione di vetro di copertura. Non toccare o avvicinarsi troppo alle cellule con la penna di barriera idrofoba.
- Bloccare non specifici siti di legame sul celLS di TBS aggiungendo contenente 4% di siero di capra. Incubare per 10 minuti a temperatura ambiente.
- Diluire un anticorpo anti-Flag (1: 1.000 diluizione) e sia un anticorpo anti-CENP-B (rapporto di diluizione di 1: 200) o ACA (1: 2.000 rapporto di diluizione) come marcatore di posizione centromero in TBS contenente 4% di siero di capra .
Nota: Per ottenere risultati ottimali, la concentrazione finale di anticorpo primario in questa soluzione deve essere determinato empiricamente. - Applicare un volume sufficiente (circa 30 ml) di anticorpo primario diluito immergere il campione cellulare. Incubare il campione di cellule per 1 ora a 37 ° C. Lavare le cellule per 5 volte con il tampone di bloccaggio per un periodo di 30 min. Per ottenere risultati ottimali (cioè, la perdita minima di cellule), la condizione di lavaggio ottimale deve essere determinata empiricamente.
- Diluire un anticorpo secondario fluoroforo coniugato (rapporto di diluizione di 1: 100 a 1: 200) diretto contro l'anticorpo primario in TBS contenente 4% siero di capra.
Nota: Per ottenere risultati ottimali, la Final concentrazione dell'anticorpo secondario in questa soluzione deve essere determinato empiricamente. - Applicare un volume sufficiente (circa 30 ml) di anticorpo secondario diluito immergere il campione cellulare. Incubare il campione di cellule per 1 ora a 37 ° C. Lavare le cellule 3 volte con il tampone bloccante.
- Applicare un volume sufficiente di TBS contenente DAPI (50-100 ng / ml) per immergere il campione cellulare. Incubare il campione di cellule per 5 minuti a RT. Lavare le cellule 1-2 volte con TBS.
- Montare il vetro di copertura che contiene il campione cellulare sul micro diapositiva come descritto in 2.11.
5. immunofluorescenza Immagine di osservazione, l'acquisizione, la quantificazione e analisi
- Osservare il campione di cellule attraverso un microscopio a fluorescenza motorizzato dotato di una lente 63X e 100X olio di immersione, una fonte di luce esterna compatta, e una fotocamera CCD digitale.
- Eseguire l'acquisizione delle immagini e l'elaborazione, tra cui deconvoluzione, con SoftwareA, o Software B1 e B2 (vedere l'elenco dei materiali / attrezzature). Si prega di consultare supplementare Codice Files (5.2.1) per tutti i comandi utilizzati in Software A. Per tutti i comandi utilizzati in Software B1 e B2, vedi (5.2.2), in Supplemental Codice File.
- Utilizzare un metodo precedentemente descritto 23-25 per quantificare i segnali di proteine centromero-cinetocore (ad esempio, rimanendo segnali di CENP-A al centromero) con le seguenti modifiche minori:
- Seleziona area delle proteine centromero-cinetocore e dello sfondo come segue:
- cell Mitotic: (regione centromero-cinetocore) Seleziona l'area di sovrapposizione con cromosomi colorati con DAPI; (regione di fondo) e area esterna cromosomi ma dentro l'identico singola cellula (regione cioè citosolico).
- cellula Interphase: (regione centromero-cinetocore) Seleziona l'area di sovrapposizione con cromatina macchiato con DAPI; (Regione di fondo) e la zona esterna della cromatina ma dentro l'identico singola cella ( cioè, regione citosolico).
Nota: vedere anche Supplemental file di codice (5.2.1.4.3) o (5.2.2.6.4) per la selezione zona con Software A o Software B2, rispettivamente.
- Quantificare la percentuale di segnali rimanenti ai centromeri con Software A o B. Per questa operazione, utilizzare la seguente formula:
segnali di proteine centromero-cinetocore Rimanendo al centromero-cinetocore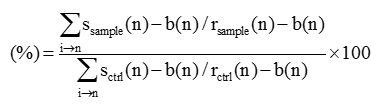
dove s è la luminosità del segnale dell'area selezionata, che è confermato da ACA o CENP-B colorazione, b è la luminosità segnale di fondo; r campione è i segnali ACA riferimento o CENP-B per siRNA (s) cellule -treated; e R ctrl è l'ACA di riferimento o segnali CENP-B per le cellule Luc siRNA-transfettate.
Nota: In questa analisi, i segnali CENP-B sono stati utilizzati come segnali di riferimento per CUL4A e RBX1 come descritto nelle precedenti relazioni. 17- Utilizzare un file di Excel per il calcolo dopo copiando e incollando i dati grezzi dal software del segnale quantitativa sopra descritta. Vedere anche supplementare file di codice: (5.2.1.4) e (5.2.2.6) per i dettagli. Un esempio di calcolo è mostrato nella Tabella 3.
- Analizzare almeno 20 cellule per eliminare variazione colorazione e acquisizione delle immagini per ogni livello di misurazione. Opzionalmente usare "valore medio" di rimanere segnali centromero-cinetocore per ogni analizzati cella per confrontare questi valori tra le diverse proteine centromero-cinetocore.
- Seleziona area delle proteine centromero-cinetocore e dello sfondo come segue:
6. Western Blot analisi delle proteine totali
- Risospendere le cellule in denaturazione tampone A (20 mM Tris-HCl, pH 7,4; 50 mM NaCl; 0,5% Nonidet P-40; 0,5% desossicolato; 0,5% SDS, 1 mM EDTA e completa proteasi inibitore cocktail gratuito-EDTA), 26 soggetti la sospensione di un sonicazione e il processo di congelamento-scongelamento, e la misuraconcentrazioni di proteine come segue:
- Per il processo di sonicazione, aggiungere 50 ml di tampone A per le cellule raccolte da due pozzetti di una piastra di polistirene 6 pozzetti a 48-72 ore dopo la trasfezione (vedi protocollo 1). Operare un sonicatore dotata di corno disgregatore e microtip (vedi elenco dei materiali / Equipment) per 15 sec durata intermittente impulsi totale (duty cycle del 50%) per un campione.
- Per il processo di congelamento-scongelamento, congelare cellule con azoto liquido e scongelare le cellule a temperatura ambiente.
- Misurare concentrazioni di proteine usando un saggio commerciale proteine reagenti I o II (vedi elenco dei materiali / attrezzature).
Nota: I lisati sono diluiti con rapporto di 1:10 nella misurazione delle concentrazioni di proteine sia con reagente I o II. A questa diluizione, la SDS presente nel tampone A mostra poca o nessuna interferenza in questa misura.
- Mescolare il lisato contenente 20-30 mg di proteine totali con tampone di caricamento 2 × 4 × o SDS-PAGE. 27 Lessare i campioni per5 min e poi caricarli su un 12,0% -15,0% denaturazione gel di SDS-poliacrilammide per l'elettroforesi.
- Trasferire le proteine separate mediante SDS-PAGE su una membrana PVDF utilizzando un metodo Western blotting descritto in precedenza. 17,24,27-31
- Bloccare i siti di legame non specifici sulla membrana con latte scremato 5% a 1 × PBS, e incubare la membrana con soluzioni di anticorpi primari diluiti per 1 ora a RT. Vedere elenco dei materiali / attrezzature per informazioni dettagliate (ad esempio, il rapporto di diluizione) di ogni anticorpo primario.
- Dopo aver lavato le membrane 3-4 volte (ogni 3 a 5 minuti di incubazione con agitazione) con PBS-T tampone (1 × PBS e 0,1% Tween-20), incubare la membrana in una miscela di vicino infrarosso (IR) fluorescenti anticorpi secondari coniugati colorante (rapporto di diluizione di 1: 20.000), anticorpi secondari coniugati DyLight (rapporto di diluizione di 1: 20.000), e / o perossidasi di rafano (HRP) coniugata anticorpi secondari (diluito in PBS-T; dilutiil rapporto di 1: 10.000) per 1 ora a RT. Vedere elenco dei materiali / attrezzature per informazioni dettagliate (ad esempio, il rapporto di diluizione) di ogni anticorpo secondario.
- Lavare la membrana 3 volte, e quindi acquisire la membrana per analizzare le proteine con il sistema di imaging a raggi infrarossi e / o l'imager chemiluminescenza per il rilevamento immunoblot (vedi elenco dei materiali / attrezzature).
- Per utilizzare il sistema di imaging a raggi infrarossi, vedi (6.4.1) in Supplemental Codice File.
- Per utilizzare il imager chemiluminescenza, vedi (6.4.2) in Supplemental Codice File. Utilizzare un substrato ultrasensibile migliorato chemiluminescenza (ECL) (vedi elenco dei materiali / Equipment) per questo sistema.
Risultati
Immunofluorescenza di endogena CENP-A supporta l'ipotesi che CUL4A-E3 ligasi è richiesto per la localizzazione di CENP-A per centromeri
I nostri studi recenti hanno dimostrato che CUL4A-RBX1-COPS8 E3 ligasi attività è necessario per ubiquitylation di lisina 124 (K124) sulla CENP-A e la localizzazione di CENP-A per centromeri. 17 Inizialmente, il complesso interfase-centromero (ICEN) è stato isolato da anti-CENP-a: nativo immunoprecipitazione della cromatina, 32-34 ed è stato ipotizzato che alcune delle proteine ICEN possono svolgere un ruolo nella localizzazione CENP-a per centromeri. Pertanto, gli esperimenti knockdown siRNA sono stati effettuati per lo screening per proteine la cui assenza di espressione indotta la delocalizzazione di CENP-A a centromeri 17 (dati non riportati): in modo asincrono crescenti cellule HeLa sono state trasfettate con Cullin 4A (CUL4A) siRNA o RBX1 siRNA per i tempi necessario per l'esaurimento della proteina ottimale (48 hR per CUL4A e 72 ore per RBX1). Cellule trasfettate con CUL4A siRNA mostrato una significativa delocalizzazione di CENP-A a centromeri (Figura 1A-C). Come si vede nella figura 2G, i livelli di proteina di CENP-A in lisati cellulari totali di cellule CUL4A siRNA-trasfettate erano simili a livelli CENP-A in lisati da luciferasi (Luc), le cellule siRNA-transfettate alle stesse condizioni di coltura. La possibilità di effetti off-target del CUL4A siRNA è stato escluso, perché l'espressione ectopica di CUL4A-Flag salvato la riduzione di CENP-A a centromeri quando CUL4A siRNA mirati 3 'UTR (Figura 2A-C). I livelli di proteina di CENP-A in lisati cellulari totali sono stati confermati per essere simile a livelli di CENP-A in lisati da cellule Luc siRNA transfettate alle stesse condizioni di coltura indipendentemente dalla fase del ciclo cellulare (figure 1B, 2G); pertanto, la possibilità che la deplezione CUL4A causato CENP-A degradazione delle proteine è stato eliminato.
Cullin-RING-E3 ubiquitina ligases (CRL) sono la classe più importante di ubiquitina ligasi 35 e contengono 3 elementi principali: un'impalcatura Cullin, una proteina dito anulare (RBX1 o RBX2), e un enzima E2 ubiquitina-carica che viene assunto da RBX1 o RBX2, 36 uN ANELLO finger proteina assume anche un adattatore substrato che pone substrati in prossimità l'enzima E2 per facilitare il trasferimento ubiquitina. 36 siRNAs RBX1 indotto una riduzione significativa di CENP-a a centromeri (Figura 1D-F) nelle condizioni in cui i livelli di proteina di CENP-a in lisati cellulari totali di cellule siRNA-trasfettate RBX1 erano simili a livelli CENP-a in lisati da cellule Luc siRNA-trasfettate (Figura 2G). Degradazione di CENP-A proteina mediante deplezione RBX1 o dalla combinazione di CUL4A e deplezione RBX1 nelle stesse condizioni di coltura non è stato osservato (Figura 2G). La possibilità di effetto off-bersaglios di RBX1 siRNA è stato escluso, perché l'espressione ectopica di Flag-RBX1 salvato la riduzione di CENP-A a centromeri quando RBX1 siRNA mirati 3 'UTR (Figura 2D-F). Questi risultati hanno suggerito che CUL4A-RBX1-E3 ligasi è specificamente necessario per la localizzazione di CENP-A per centromeri.
Immunofluorescenza di esogeno CENP-A - proteine flag indica che CENP-A K124 ubiquitinazione è essenziale per la localizzazione di CENP-A per centromeri
In precedenza, la nostra analisi spettrometria immunoprecipitazione-massa ha dimostrato che la lisina 124 (K124) di CENP-A-Flag è ubiquitylated in cellule HeLa. 17 Nella struttura cristallina del CENP-A nucleosoma, K124 risiede nel elica α3, anche se il sito è non all'interno della regione CATD. 17 Inoltre, K124 è conservata tra mammiferi, uccelli, lucertole, piante, e un gruppo di funghi (per esempio, gemmading lievito). 17 CENP-A mutanti lisina (figura 3A) sono stati costruiti e testati la loro capacità di localizzare al centromero. Abrogazione sostanziale della localizzazione centromerica del esogeno CENP-A mutante K124R con segnali diffuse in cellule HeLa sia mitotiche e interfase stato osservato (Figura 3B, C). Centromere localizzazione non è stata significativamente influenzata né da mutazioni K9A (K9 corrisponde a dell'istone H3 K9 metilazione) né la mutazione K77R (K77 è un sito unico in lisina CATD) (Figura 3B, C).
Sulla base di questi risultati, si propongono due ipotesi per quanto riguarda la funzione in vivo di CENP-A ubiquitinazione a K124. In primo luogo, il ruolo di K124 ubiquitinazione è per proteolisi ubiquitina-mediata per eliminare overexpressed e / o mislocalized CENP-A a eucromatina. In secondo luogo, il ruolo di CENP-A ubiquitinazione sul K124 è per il caricamento di CENP-A sul centromeri. Per testare la fpossibilità irst, sono state affrontate le stabilità di CENP-A-Flag wild type e il mutante K124R così come endogena CENP-A dopo cicloesimide (CHX) trattamento di CUL4A- o cellule RBX1-impoverito. Questi dati suggeriscono che ubiquitylation del K124 è probabilmente non è coinvolto nella proteolisi ubiquitina-mediata per eliminare overexpressed e / o mislocalized CENP-A (dati non riportati). 17. Inoltre, è stato confermato che la mutazione K124R abroga bande monoubiquitylation e diubiquitylation putativi, sia in in vivo e in saggi di ubiquitinazione in vitro (dati non riportati). 17 Collettivamente, questi dati suggeriscono che il complesso CUL4A-RBX1 contribuisce alla ubiquitylation "segnalazione", che è richiesto per CENP-a localizzazione a centromeri.
Immunofluorescenza di esogeno Flag - CENP-A proteine indica che la fusione monoubiquitin è sufficiente per caricareCENP-A K124R a centromeri
Sulla base dei risultati di cui sopra, è stato ipotizzato che monoubiquitin covalentemente legato serve come un segnale per CENP-A carico su centromeri. Per verificare questa ipotesi, un N-terminale di Flag-tag e C-terminale ubiquitina-fuso wild-type CENP-A e un mutante K124R CENP-A è stato costruito (Figura 4A). Il mutante monoubiquitin Ub (K48R), che manca di un importante sito per polyubiquitylation 37-39 è stato utilizzato anche per prevenire ubiquitina-fuso CENP-A proteina da potenziale polyubiquitylation. Catturando segnali immunofluorescenza Anti-Flag, la localizzazione centromerica delle proteine codificate da questi costrutti è stato testato. Mentre Flag-CENP-A (K124) sostanzialmente abrogato centromero la localizzazione di CENP-A (Figura 4B e 4C, colonna [6]), sia Flag-CENP-A (WT) e Flag-CENP-A (WT) -UB ( WT) mantenuto la loro localizzazione centromerica (Figura 4B e 4C, colonne [1] e [2]). IlBandiera CENP-A-(K124R) -UB (K48R) proteina presumibilmente imitato monoubiquitylated CENP-A, come questa proteina sostanzialmente ripristinato localizzazione per centromeri (Figura 4C, confrontare le colonne [5] e [6]) in modo più efficiente di quanto ha fatto CENP-A (K124R) -UB (WT) (Figura 4C, confrontare le colonne [4] - [6]). Questi dati hanno dimostrato che monoubiquitylation è sufficiente per l'assunzione di CENP-A per centromeri.

Figura 1. Analisi di immunofluorescenza di endogena CENP-A supporta l'ipotesi che CUL4A-E3 ligasi è richiesto per la localizzazione di CENP-A per centromeri (I dati sono stati adattati da Niikura et al. 17). (A) CUL4A siRNA indotta delocalizzazione di CENP-A a centromeri. cellule HeLa sono state trasfettate con CUL4A o luciferasi siRNA (Luc) e incubate per 48 ore (Tabella 1). Barra della scala rappresenta 10 micron. (B) Analisi Western blot di HeLa lisati cellulari tutto utilizzando la stessa condizione di cultura come in (A). Le cellule sono state raccolte 48 ore dopo la trasfezione con siRNA o CUL4A Luc siRNA (Tabella 1). GAPDH servito come controllo di carico. (C) quantificata endogeni CENP-A segnali a centromeri mostrato in (A). La normalizzazione dei segnali è stata effettuata utilizzando cellule Luc siRNA-trasfettate, e sono esposte le percentuali di media (± SD). **** P <0,0001 vs cellule Luc siRNA-trasfettate (test t). (D) RBX1 siRNA indotta delocalizzazione di CENP-A da centromeri. Cellule HeLa sono state trasfettate con RBX1 o Luc siRNA (s) e incubate per 72 ore (Tabella 1). Barra della scala rappresenta 10 micron. (E) Analisi Western Blot di HeLa lisati cellulari utilizzando tutta la stessa condizione della cultura come in (D). Le cellule sono state raccolte 72 ore dopo la transfezione con RBX1 oR Luc siRNA (s) (Tabella 1). GAPDH servito come controllo di carico. (F) quantificata segnali CENP-A endogeni a centromeri mostrati in (D). La normalizzazione dei segnali è stata effettuata utilizzando cellule Luc siRNA-trasfettate, e sono esposte le percentuali di media (± SD). **** P <0,0001 vs cellule Luc siRNA-trasfettate (test t). Clicca qui per vedere una versione più grande di questa figura.

Figura 2. Informazione di riferimento relative alla figura 1 centromeri (Figure sono stati adattati da Niikura et al. 17). (A) esogena CUL4A-Flag salvato la delocalizzazione di CENP-A quando CUL4A siRNA di mira il 'UTR 3. HeLa Tet-Off celLS sono stati fissati a 48 ore dopo la cotrasduzione con siRNA (CUL4A # 2: 3 'UTR bersaglio o Luc; Tabella 1) più il costrutto plasmide (pTRM4-CUL4A-Flag o vettore; tabella 2). 4 colori immunostaining: colorazione per DAPI (blu); Bandiera; endogena CENP-B (rosso); e endogena CENP-A (verde), è stata eseguita e le cellule Flag-positivi sono stati ordinati, ma le immagini Flag sono omessi per semplicità. Nota: CUL4A # 2 è l'obiettivo identico a quello mostrato nella Figura 1A-C. Barra della scala rappresenta 10 micron. (B) Analisi Western Blot di HeLa Tet-Off lisati cellulari intere seguendo la stessa condizione di cultura come mostrato in (A). GAPDH servito come controllo di caricamento. Segnali (C) CENP-A a centromeri mostrati in (A) sono stati quantificati mediante microscopia dopo la selezione di cellule di isolare quelli legati ad un anticorpo anti-Flag. La normalizzazione dei segnali è stata eseguita con cellule trasfettate con siRNA Luc più vettore, e sono esposte le percentuali medi (± DS).**** P <0,0001 vs cellule trasfettate con siRNA Luc più vettore (colonna sinistra, test t). (D) esogena Flag-RBX1 salvato la delocalizzazione di CENP-A al centromero quando RBX1 siRNA di mira il 3 'UTR . Cellule HeLa sono stati fissati a 72 ore dopo la cotrasduzione con siRNA (RBX1 # 2: 3 'UTR bersaglio o Luc; Tabella 1) più il costrutto plasmide (pcDNA3-Flag-RBX1 o vettore; tabella 2). 4 colori immunostaining: colorazione per DAPI (blu); Bandiera; endogena CENP-B (rosso); e endogena CENP-A (verde), è stata eseguita e le cellule Flag-positivi sono stati ordinati, ma le immagini Flag sono omessi per semplicità. Barra della scala rappresenta 10 micron. (E) Analisi Western Blot di HeLa lisati cellulari tutto seguendo la stessa condizione di cultura come in (D). GAPDH servito come controllo di carico. (F) segnali CENP-A a centromeri mostrati in (D) sono stati quantificati al microscopio dopo la selezione di cellule per isolare quelli legati da un n anticorpo anti-Flag. La normalizzazione dei segnali è stata eseguita con cellule trasfettate con siRNA Luc più vettore, e sono esposte le percentuali medi (± DS). **** P <0,0001 vs cellule trasfettate con siRNA Luc più vettore (colonna sinistra, test t). (G) L'esaurimento delle CUL4A e RBX1 non ha influenzato i livelli di endogena CENP-A proteina. I livelli di endogena CENP-A proteina sono stati misurati in HeLa lisati cellulari intero raccolte 72 ore dopo la trasfezione con CUL4A, RBX1, CUL4A più RBX1, o Luc siRNA. Quarantotto ore dopo la trasfezione, le cellule erano o non trattata (Asyn) o trattato con paclitaxel (+ tasse) per indurre l'arresto in mitosi, ed erano coltivate per 24 ore. GAPDH servito come controllo di carico. Si prega di cliccare qui per vedere una versione più grande di questa figura.
32 / 53732fig3highres.jpg "width =" 700 "/>
Figura 3. Analisi immunofluorescenza di esogeni proteine CENP-A-Flag ha indicato che CENP-A K124 ubiquitinazione è necessario per CENP-A localizzazione a centromeri (I dati sono stati adattati da Niikura et al. 17). (A) sovraespressione di CENP-A- costrutti Flag (WT e mutanti KR) è stata confermata dall'analisi Western blot di HeLa Tet-Off lisati cellulari interi. Le cellule sono state raccolte 48 ore dopo la trasfezione con pTRM4-CENP-A-Flag WT, i mutanti KR, o pTRM4 vettore (Tabella 2). Sovraespressione di CENP-A-Flag è stata rilevata da un anticorpo anti-Flag. GAPDH servito come controllo di caricamento. Putativo CENP-A-Flag dimero (##) e CENP-A-Flag monomero (#) sono mostrati. Nota: SDS-resistenti dimeri CENP-A sono stati segnalati in precedenza 40,41 (B) La CENP-A mutante K124R mostrato delocalizzazione diffusa da centromeri.. I segnali provenienti da DAPI (blu), Bandiera (green), e endogena CENP-B (rosso, un centromero di controllo posizione) sono mostrati. Nota: A differenza endogena CENP-A in cellule trasfettate con CUL4A o RBX1 siRNA, segnali diffusi apparsi in cellule che overexpressed esogena CENP-A K124R-Flag come un fenotipo della impossibilità di essere mirata a centromeri, presumibilmente perché il suo livello di espressione è di circa 10 - a 25 volte superiore a quella del endogena CENP-a (dati non mostrati) 17 bar Scala rappresenta 10 micron (C) istogrammi dei modelli di localizzazione mostrati in (B)... Più di 50 / cellule pro prometafase e metafase e più di 200 cellule in interfase per esperimento sono stati contati (n ≥ 3 esperimenti). sono mostrati Le percentuali medi (± DS). "Altri (Non-centromero)" indica cellule danneggiate per lo più, le cellule morte, o cellule con localizzazione nucleolare (solo in interfase), presumibilmente a causa di trasfezione o di altri trattamenti. *** P <0.001 vs CENP-A WT-Flag (test t). Clicca qui per vedere una versione più grande di questa figura.
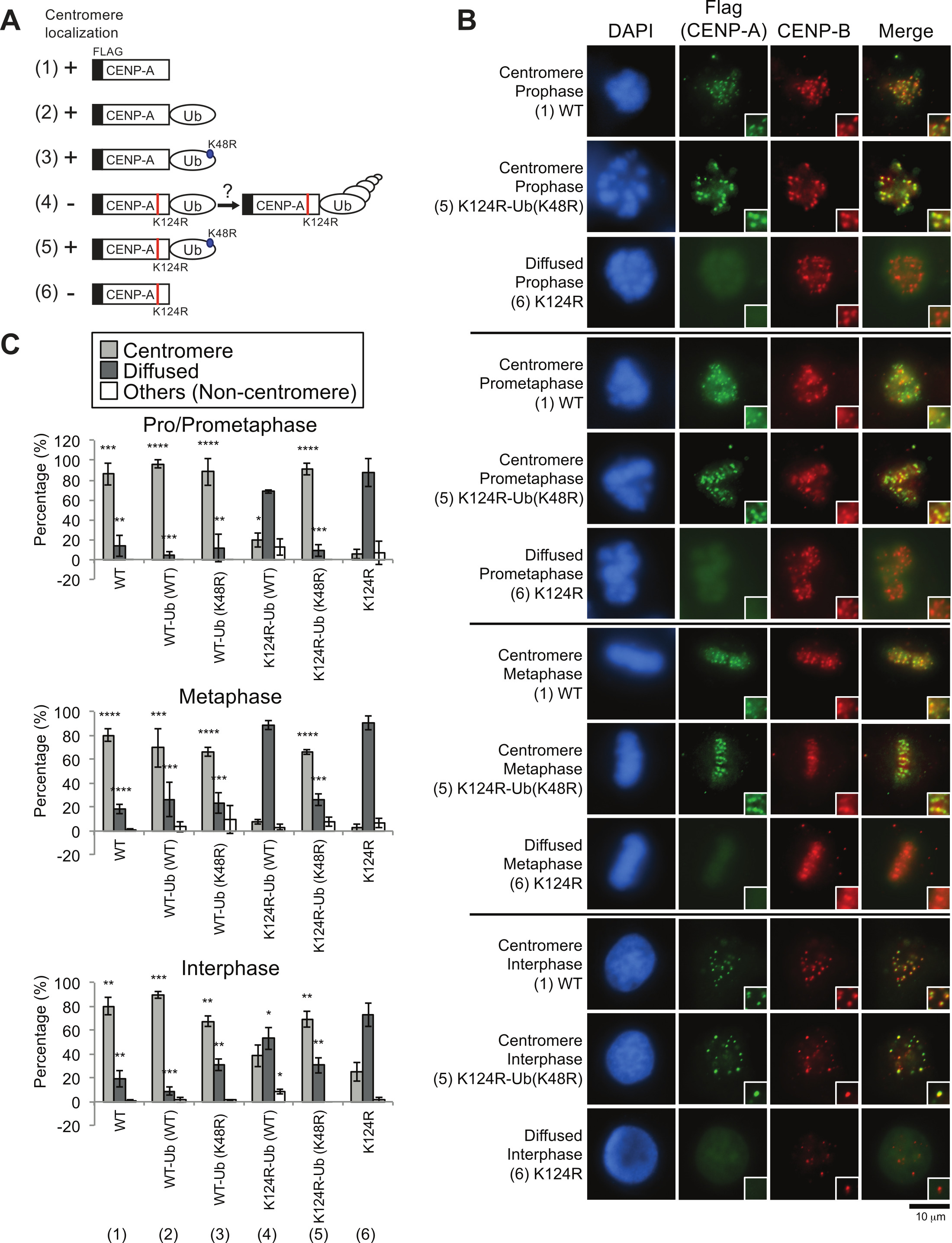
Figura 4. Analisi immunofluorescenza di esogeni proteine Flag-CENP-A ha indicato che CENP-A fusione monoubiquitin salvato delocalizzazione della CENP-A mutante K124R da centromeri (figure sono stati adattati da Niikura et al. 17). (A) cartoni schematica di ciascun costrutto utilizzato in questo studio (vedi anche tabella 2). Il sito di mutazione K124R su CENP-A (rosso) e il sito di mutazione K48R su monoubiquitin (blu) vengono visualizzati. (1) WT: Flag-CENP-A WT, (2) WT-Ub (WT): Flag-CENP-A WT-Ub (WT), (3) WT-Ub (K48R): Flag-CENP-A WT -UB (K48R), (4) K124R-Ub (WT): Flag-CENP-A K124R-Ub (WT), (5) K124R-Ub (K48R): Flag-CENP-A K124R-Ub (K48R), (6) K124R: Flag-CENP-A K124R (B) esogena CENP-Una fusione monoubiquitin salvato delocalizzazione della CENP-A mutante K124R da centromeri.. DAPI (blu), Bandiera (verde), e endogena CENP-B (rosso, un centromero di controllo posizione) sono mostrati. Bar scala rappresenta 10 micron. (C) istogrammi dei modelli di localizzazione mostrato in (C). Più di 50 / cellule pro prometafase e metafase e più di 200 cellule in interfase per esperimento sono stati contati (n ≥ 3 esperimenti). sono mostrati Le percentuali medi (± DS). **** P <0,0001 e *** P <0.001 vs non-fuso Flag-CENP-A K124R (colonna [6]) (test t). Si prega di cliccare qui per vedere una versione più grande di questa figura.
| bersaglio siRNA | Indicazione nel presente studio | tipo di destinazione | il numero di database siRNA | Sequenza Forward (s) | Fonte / riferimento | |
| Luciferasi (GL3) | - | 1 bersaglio | RKK9 | CUUACGCUGAGUACUUCGAdTdT | Elbashir et al., (2001) | |
| CENP-A | CA-UTR | Piscina siRNA (2 obiettivi miscela) | RKK375 / 5 'UTR T1 | CGAGCGGCGCGGACUUCUGCCdTdT | Niikura et al., (2015) | |
| RKK383 / 3 'UTR T1 | UCCUGCACCCAGUGUUUCUGUdGdT | Niikura et al., (2015) | ||||
| CUL4A | # 1 | 1 bersaglio | CRKK178 / RKK309 / T1 | AGCGAUCGUAAUCAAUCCUGA | Niikura et al., (2015) | |
| # 2 (3'UTR) | 1 bersaglio | CRKK181 / RKK331 / t4 | AUGCGGGUUUGAAAUAUGACA | Niikura et al., (2015) | ||
| RBX1 / ROC1 | # 1 | piscina siRNA (4 bersagli miscela) | CRKK198 / RKK411 / T1 | GAAGCGCUUUGAAGUGAAA | Niikura et al., (2015) | |
| CRKK198 / RKK413 / t2 | GCAUAGAAUGUCAAGCUAA | Niikura et al., (2015) | ||||
| CRKK198 / RKK415 / t3 | GCAAGAAGCGCUUUGAAGU | Niikura et al., (2015) | ||||
| CRKK198 / RKK417 / t4 | CAACAGAGAGUGGGAAUUC | Niikura et al., (2015) | ||||
| # 2 (3 'UTR) | 1 bersaglio | CRKK206 / RKK425 / T8 | UUCCCUGCUGUUACCUAAUUA | Niikura et al., (2015) | ||
Tabella 1. sequenze siRNA utilizzati in questo studio.
| numero B | Caratteristica rilevante (s) | Fonte / riferimento |
| B288 | pTRM4 | Niikura et al., (2006) |
| B2067 | pTRM4-umano CENP-A-Flag | Niikura et al., (2015) |
| B2281 | pTRM4-umano CENP-A K9A-Flag | Niikura et al., (2015) |
| B2387 | pTRM4-umano CENP-A K77R-Flag | Niikura et al., (2015) |
| B2388 | pTRM4-human CENP-A K124R-Flag | Niikura et al., (2015) |
| B2512 | pTRM4-Flag-umano CENP-A | Niikura et al., (2015) |
| B2579 | CENP-A pTRM4-Flag-umano K124R | Niikura et al., (2015) |
| B2513 | pTRM4-Flag-umano CENP-A-Ub (WT) | Niikura et al., (2015) |
| B2559 | pTRM4-Flag-umano CENP-A K124R-Ub (WT) | Niikura et al., (2015) |
| B2515 | pTRM4-Flag-umano CENP-A-Ub (K48R) | Niikura et al., (2015) |
| B2560 | pTRM4-Flag-umano CENP-A K124R-Ub (K48R) | Niikura et al., (2015) |
| B2759 | pTRM4-umano CUL4A-Flag | Niikura et al., (2015) |
| B2624 | RBX1 pcDNA3-Flag-umano | Niikuraet al., (2015) |
| B1491 | pcDNA3-Flag | Niikura et al., (2015) |
Tabella 2. vettori plasmidi utilizzati in questo studio.
| R (anti-CENP-B) del campione | B (anti-CENP-B) | Campione R (sottratto) | Rapporto (campione S: campione R) | Rapporto Corretto (campione S: campione R) |
| 520 | 514 | 6 | -4,167 | 0.000 |
| 535 | 445 | 90 | -1,033 | 0.000 |
| 515 | 431 | 84 | 0,274 | 0,274 |
| 562 | 483 | 79 | 0,506 | 0,506 |
| 902 | 562 | 340 | 0,509 | 0,509 |
| 1203 | 703 | 500 | -0,560 | 0.000 |
| 1014 | 589 | 425 | -0,819 | 0.000 |
| 768 | 510 | 258 | -1,345 | 0.000 |
| 555 | 458 | 97 | -1,845 | 0.000 |
| 781 | 576 | 205 | -1,146 | 0.000 |
| 556 | 436 | 120 | 0,758 | 0,758 |
| 534 | 493 | 41 | -1,634 | 0.000 |
| 702 | 543 | 159 | 0.667 | 0.667 |
| 482 | 429 | 53 | -1.000 | 0.000 |
| 654 | 531 | 123 | 0.740 | 0.740 |
| 1190 | 607 | 583 | 0,384 | 0,384 |
| 552 | 454 | 98 | 0.969 | 0.969 |
| 489 | 413 | 76 | 0,539 | 0,539 |
| 511 | 452 | 59 | -3,186 | 0.000 |
| 485 | 475 | 10 | -2,700 | 0.000 |
| 481 | 441 | 40 | -1,475 | 0.000 |
| Somma | 5.347 | |||
| Media | 0.255 | |||
| R (anti-CENP-B) ctrl | B (anti-CENP-B) | R Ctrl (sottratto) | Ratio (S ctrl: R ctrl) | Rapporto Corretto (S ctrl: R ctrl) |
| 543 | 483 | 60 | 3.017 | 3.017 |
| 526 | 466 | 60 | 2.667 | 2.667 |
| 532 | 460 | 72 | 1.792 | 1.792 |
| 507 | 457 | 50 | 3.520 | 3.520 |
| 491 | 458 | 33 | 4.273 | 4.273 |
| 545 | 464 | 81 | 2.160 | 2.160 |
| 518 | 484 | 34 | 3.559 | 3.559 |
| 461 | 404 | 57 | 2.404 | 2.404 |
| 487 | 447 | 40 | 3.550 | 3.550 |
| 534 | 477 | 57 | 3.965 | 3.965 |
| 528 | 456 | 72 | 3.097 | 3.097 |
| 707 | 601 | 106 | 1.868 | 1.868 |
| 615 | 528 | 87 | 2.828 | 2.828 |
| 660 | 481 | 179 | 1.620 | 1.620 |
| 565 | 476 | 89 | 1.607 | 1.607 |
| 527 | 455 | 72 | 3.583 | 3.583 |
| 560 | 474 | 86 | 2.930 | 2.930 |
| 510 | 451 | 59 | 4.017 | 4.017 |
| 729 | 586 | 143 | 2.678 | 2.678 |
| 634 | 576 | 58 | 3.655 | 3.655 |
| 507 | 476 | 31 | 3.935 | 3.935 |
| Somma | 62,725 | |||
| Media | 2.987 | |||
| Rimanendo segnali CENP-A al centromero (%) | 8.5 |
. Tabella 3. Un esempio di calcolo delle rimanenti segnali CENP-A al centromero (%) Un esempio di Pro / Prometafase in Figura 2F viene mostrato: campione è RBX1 # 2 (3'UTR) + Vec (colonna centrale in Figura 2F ) ed il controllo è Luc + Vec (colonna di sinistra in Figura 2F). Come risultato, rimanendo segnali CENP-A acentromero (%) = (0.255 / 2.987) x 100 = 8,5% è ottenuto (o [5,347 / 62,725] x 100 = 8,5% con lo stesso numero di cellule totale [vale a dire, 21 celle] analizzati tra due campioni). Si noti che se il valore B è più di valore S (ad esempio, se il valore è negativo sottratto), valore del rapporto corretto è impostato su 0,00.
Discussione
Negli ultimi anni molti studi hanno sviluppato diversi saggi di microscopia quantitativa per le cellule fisse. 42 I progressi nel campo della biologia centromero-cinetocore spesso richiede una comprensione della funzione centromero-specifica o specifiche cinetocoro delle proteine di cui subcellulare regolazione spazio-temporale riflette le funzioni mutevoli queste proteine durante il ciclo cellulare. Quindi, qui abbiamo sviluppato metodi di IIF colorazione e un test quantitativo IIF per analizzare specificamente i livelli relativi di endogeni ed esogeni proteine CENP-A, che può essere applicabile in campioni trattati in modo diverso. Attualmente abbiamo raggiunto con successo IIF colorazione del endogena CENP-I, CENP-H, KNL1, Hec1, e Ska1 utilizzando protocollo 2 in questo studio (vedi Niikura et al 24 e dati non riportati;. Elenco dei materiali / Attrezzature di informazioni di anticorpi) . In futuro, si può applicare lo stesso metodo per quantificare il livello di diverse proteine centromero-cinetocore (entrambi endogendogeni e Flag-tagged proteine esogene) scegliendo gli anticorpi specifici, tuttavia, i nostri metodi IIF non coprono la rilevazione di queste proteine nelle cellule viventi o in una determinata cella singola durante tutto il ciclo cellulare. Poiché queste procedure richiedono cellule da fissare ed elaborati su vetrini separati, abbiamo introdotto segnali di riferimento (ad esempio, ACA o CENP-B) al fine di normalizzazione al fine di confrontare abbastanza segnali tra i campioni diversi e aumentare la precisione del test. Poiché ACA comporta o segnali endogeni ed esogeni / CENP-A, CENP-B come segnale di riferimento al fine di normalizzazione sarebbe più appropriato per il confronto di quanto sarebbe ACA in termini di precisione del dosaggio quantitativo di segnali CENP-A. La regione amino-terminale di CENP-B si lega al motivo di 17 bp della sequenza scatola CENP-B in DNA alphoid, e la regione carbossi-terminale di CENP-B costituisce homodimers. 43,44 CENP-B non colocalize completamente con CENP-A e / o altri centrproteine Omere-cinetocoro, tuttavia, è strettamente associato con loro. 45 Mentre immunofluorescenza con anticorpi anti-CENP-C offre in genere due punti discreti, colorazione con anti-CENP-B appare spesso come una singola barra luminosa che collega entrambi i centromeri sorella. 46 Tuttavia , nella nostra macro-scala di osservazione microscopica, alcuni segnali CENP-B a quanto pare si sovrappongono con i segnali CENP-a, in particolare nelle fasi mitotiche precedenti (profase-prometafase di ritardo metafase), anche in parte, a seconda dell'angolo di osservazione e il tipo di fissativo usato (dati non mostrati). Al contrario, in un dosaggio quantitativo IIF di segnali CENP-A, proteina localizzazione dei segnali di riferimento non deve essere influenzata da esaurimento e / o disfunzione di CENP-A per un'analisi imparziale, anche se la localizzazione della maggior parte delle proteine centromero-cinetocore dipende CENP-a localizzazione a centromeri. assemblaggio cinetocoro 47,48 CENP-a-indipendente è stabilita sostituendo le regioni di DNA-binding di CENP-C E CENP-T con i domini cromosomici-targeting alternativi che reclutano queste proteine a loci ectopica. 49,50 Inoltre, un recente lavoro ha indicato che i componenti interni cinetocoro CENP-C e CENP-T agire in parallelo per reclutare la rete KMN a cinetocori. 51-54 Inoltre, Nishino et al. hanno suggerito che le forme complesse CENP-TWSX una struttura nucleosomi simile unica per generare contatti con il DNA, l'estensione del 'codice istonico' al di là di proteine nucleosoma canoniche. 55 Poiché il centromero localizzazione dei CENP- C dipende dalla localizzazione centromero di CENP-a, 47,48 CENP-TWSX o particolare CENP-T potrebbe essere un altro candidato proteina (s) per fornire segnali di riferimento in un dosaggio quantitativo IIF di CENP-a. Tuttavia, il candidato per la segnalazione di riferimento deve essere provato con attenzione prima del test per determinare se la localizzazione in centromeri è influenzata da esaurimento e / o disfunzione di CENP-A. analoga considerazionedovrebbe essere dato per analisi quantitative IIF di altre proteine centromero-cinetocore: localizzazione delle proteine di segnali di riferimento non deve essere influenzata da esaurimento e / o disfunzione della proteina bersaglio per un'analisi imparziale. Nello studio precedente, abbiamo usato ACA come segnale di riferimento per il dosaggio quantitativo IIF di altre proteine centrali outer cinetocoro. 24 ACA poteva essere una soluzione più appropriata di singolo CENP-B come un controllo di posizione, nonché un segnale di riferimento, se ACA segnali non sono interessati da esaurimento e / o disfunzione della proteina bersaglio, ma colocalizzazione della proteina bersaglio con CENP-B è scarsa come detto sopra.
Bodor et al. hanno riferito che centromeriche livelli CENP-A sono regolati da azione di massa, cioè, la quantità di centromerica CENP-A varia in modo direttamente proporzionale al contenuto cellulare. 56 Essi hanno inoltre dimostrato che l'induzione transiente di CENP-A espressione porta ad un rapido aumento della centromeric-A livello di CENP. Al contrario, abbiamo osservato una grande popolazione di cellule con centromero-localizzato, Flag-tag CENP-A WT dopo cotrasfezione del vettore di espressione pTRM4 con CA-UTR siRNA (una miscela di 5 'e 3' UTR siRNA; Tabella 1) (non dati mostrato); questa popolazione era più grande di quella osservata dopo la transfezione con il solo vettore di espressione pTRM4. Il livello di esogeno Flag-tag CENP-A proteina la cui espressione è stata guidata da pTRM4, era di circa 1,0 a 1,4 ordini di grandezza (dai 10 ai 25 volte) superiore a quella di endogena CENP-A (dati non mostrati). Si dice che l'espressione di esogena CENP-A WT di sopra di una determinata soglia potrebbe influenzare negativamente il corretto localizzazione centromeric.
Nelle attuali protocolli, fissazione è un passaggio critico per mantenere struttura cellulare e l'ambiente come situazione prossimale possibile alla stato nativo. Una volta che le cellule sono fissi, si deve procedere con il protocollo di colorazione per evitare la perdita della proteina di interiposo o il resto della cellula. Tuttavia, la fissazione distrugge siti antigenici occasionalmente e diverse combinazioni anticorpo-antigene funziona male con un fissativo, ma molto bene con un altro. 21 A causa di questa variazione, la scelta di fissativo dipende in gran parte della proteina (s) di interesse. La lunghezza di tempo di fissaggio può influenzare significativamente la rilevazione della proteina (s) mediante immunocolorazione, e fissazione breve è generalmente migliore di mantenere antigenicità. 57 Pertanto, nello stabilire procedure di colorazione per una nuova destinazione di altre proteine centromero-cinetocore, specialmente incerta localizzazione, o quando si utilizza un nuovo anticorpo, dovrebbe testare diversi metodi di fissazione e tamponi per trovare la condizione ottimale. Nelle attuali protocolli, sono stati selezionati due classi di fissativi popolari: fissativi aldeide (protocollo 2) e solventi organici (protocolli 3 e 4). La purezza dei fissativi è estremamente importante. Nel protocollo 4, 4% di siero di capra viene utilizzato especially di bloccare i recettori IgG sulla superficie cellulare per ridurre fondo aspecifica. 57 Generalmente, il siero per bloccare i recettori IgG dovrebbe essere da una specie non correlata con l'anticorpo primario e preferibilmente dalla stessa specie come anticorpo secondario. 57
In ciascuno degli attuali protocolli, la scelta di anticorpo primario è il passo più critico nel raggiungimento immunocolorazione successo. L'anticorpo con la più grande specificità, la purezza, l'affinità e l'avidità è desiderato. Una buona concentrazione di partenza per gli anticorpi monoclonali è generalmente 1-5 ug / ml. Diluizioni tipiche di preparazione madre di anticorpi policlonali vanno da 1:20 e 1:. 500 57 Bisogna determinare empiricamente la concentrazione appropriata della anticorpo primario e il diluente per ogni campione. I campioni devono essere cullati ma non secchi durante l'incubazione per la colorazione omogenea. lavaggio estensivo tra il incubazione dell'anticorpo primario e uno secondariontibody può essere richiesto per evitare la reazione incrociata con le immunoglobuline di altre specie. Tuttavia, la condizione di lavaggio ottimale deve essere determinata empiricamente per evitare la perdita di cellule, in particolare le cellule in mitosi, che completano e si staccano quando scossa (vale a dire, mitotico shake-off). 58 Per avere successo, un anticorpo secondario deve essere scelto per l'associazione a l'anticorpo primario con alta affinità. Ad esempio, per evitare il legame non specifico della porzione Fc dell'anticorpo secondario ai recettori IgG Fc, Fb (ab ') 2 frammenti possono essere utilizzati al posto di tutta anticorpi. 57 Per molti anticorpi secondari, tempo di incubazione può essere minimizzato 30 min a temperatura ambiente per ridurre lo sfondo non specifico.
Oltre al protocollo 5, scansione laser confocale microscopio potrebbe essere vantaggioso nel rilevare e analizzare la localizzazione e la funzione delle proteine centromero-cinetocore con le proprietà fluorescenti. Come mostrato nella lista dei materiali / Equipment, Alexa Fluor 488 e Alexa Fluor 594 sono probabilmente i coloranti fluorescenti utilizzate più frequentemente nel presente protocollo. Per rilevare due e o più proteine centromero-cinetocore / diverse nel campione, si deve garantire che gli anticorpi utilizzati per rilevare una proteina non impediscono la rilevazione della seconda proteina. 57 Nel presente protocolli, due anticorpi primari sono mescolati e applicati allo stesso tempo. Tuttavia, si dovrebbe ottimizzare le condizioni di immunoistochimica per ogni proteina separatamente prima di applicare gli anticorpi primari o secondari insieme: se le due proteine sono in stretta prossimità, come nel caso di due componenti centromero-cinetocore, un anticorpo primario può strutturalmente prevenire il legame del secondo anticorpo primario. Un'altra preoccupazione di rilevamento del segnale multipla è il problema di sovrapposizione spettrale: la difficoltà di impedire segnale da un colorante "perde" nel canale spettrale di un altro. Questo problema diventa più difficile per conventionFiltro al interferenza imposta per risolvere il numero di coloranti aumenta o se il campione contiene segnali overenhanced (ad esempio, ACA o anti-Flag segnali nel presente studio), compresa l'interferenza di autofluorescenza. Risoluzione dei problemi di autofluorescenza è stata condotta in altri studi. 57,59,60 Nel presente studio, ottimizzando set di filtri fluorescenti con i controlli etichetta singola in ogni canale è sufficiente nella maggior parte dei casi per evitare questi problemi (vedi sotto).
In ciascuna delle presenti protocolli, controlli appropriati sono essenziali. Ogni nuovo anticorpo primario dovrebbe essere caratterizzato prima dell'inizio immunocolorazione. La specificità dell'anticorpo deve essere confermata da analisi Western Blot, se applicabile. Inoltre, conferma che la colorazione non è causato da legame non specifico alla superficie cellulare deve essere ottenuto, e la concentrazione ottimale di un particolare anticorpo primario lavoro dovrebbe essere determinato attraverso l'uso di diluizioni seriali(Cioè, una curva vincolante dovrebbe essere sviluppato). Un controllo negativo (cioè, l'assenza dell'anticorpo primario dal processo di colorazione) dovrebbe essere inclusa. Un'altra opzione di controllo negativo è la sostituzione della normale IgG dalla stessa specie per l'anticorpo primario. Per il rilevamento di più etichette, bisogna preparare i controlli senza anticorpi secondari (il controllo di sfondo), così come i controlli etichetta singola per evitare artefatti sovrapposizione spettrale (cioè, sanguinare-through, crossover, diafonia, o tintura "perdite", come descritto in precedenza ). Si può quantificare la frazione "perdite" confrontando segnali attraverso i filtri "sbagliati" con il segnale corretto, utilizzando queste proporzioni per rimuovere computazionalmente tutti "fuoriuscita", e calcolando la distribuzione reale e / o co-localizzazione del marcatore fluorescente. 60 il controllo di fondo dovrebbe essere esaminata in modo indipendente, con ogni canale per impostare i limiti di guadagno del segnale e l'offset di essere adapted per la rappresentazione finale. Tutti i canali che verranno utilizzati per ottenere un'immagine di un campione multiple-label devono essere sottoposti a correzione del fondo indipendente, perché il livello di autofluorescenza in ciascun canale varia notevolmente. Le "perdite" controlli come descritto sopra sono necessari per determinare la quantità di guadagno del segnale possibile in ogni canale senza rilevare "perdite" in canali adiacenti. 60
Divulgazioni
Gli autori dichiarano di non avere interessi finanziari concorrenti.
Riconoscimenti
Questo lavoro è stato sostenuto da NIH concedere GM68418.
Materiali
| Name | Company | Catalog Number | Comments |
| Lipofectamin 2000 | Life Technologies/Invitrogen | 11668 | transfection reagent I |
| Lipofectamin RNAiMAX | Life Technologies/Invitrogen | 13778 | transfection reagent II |
| Opti-MEM I | Life Technologies/Invitrogen | 31985 | Reduced serum media, warm in 37 °C water bath before use |
| High-glucose DMEM (Dulbecco’s modified Eagle’s medium) | Life Technologies/BioWhittaker | 12-604 | high-glucose DMEM, warm in 37 °C water bath before use |
| Fetal Bovine Serum, certified, heat inactivated, US origin | Life Technologies/Gibco | 10082 | FBS (fetal bovine serum) |
| Poly-L-Lysine SOLUTION | SIGMA-SLDRICH | P 8920 | Poly-L-Lysine, 0.1% w/v, in water |
| UltraPure Distilled Water | Life Technologies/Invitrogen/Gibco | 10977 | Sterile tissue culture grade water |
| Micro Cover glass (22 mm x 22 mm) | Surgipath | 105 | Cover glass (22 mm x 22 mm) |
| 6 Well Cell Culture Cluster | Fisher/Corning Incorporated | 07-200-83 | 6-well polystyrene plate |
| Penicillin, Streptomycin; Liquid | Fisher/Gibco | 15-140 | Penicillin-streptomycin |
| PAP PEN | Binding Site | AD100.1 | Hydrophobic barrier pen (for a water repellant barrier in immunofluorescent staining) |
| Paclitaxel (Taxol) | SIGMA-SLDRICH | T7402 | Taxol for mitotic cell analysis |
| TN-16, microtubule inhibitor (TN16) | Enzo Life Sciences | BML-T120 | TN16 for mitotic cell analysis |
| BSA (bovine serum albumin) | SIGMA-SLDRICH | A7906 | Blocking reagent |
| Triton X-100 | SIGMA-SLDRICH | T8787 | Detergent for permeabilization |
| Paraformaldehyde | SIGMA-SLDRICH | P6148 | Fixation reagant |
| DAPI | SIGMA-SLDRICH | D9542 | For nuclear staining |
| p-phenylenediamine | SIGMA-SLDRICH | P6001 | For mounting medium |
| VWR Micro Slides, Frosted | VWR International | 48312-013 | Micro slides |
| Anti-CENP-A antibody | Stressgen/Enzo Life Sciences | KAM-CC006 | Mouse monoclonal antibody; dilution ratio of 1:100 (IIF), 1:5000 (WB) |
| Anti-CENP-B antibody | Novus Biologicals | H00001059-B01P | Mouse monoclonal antibody; dilution ratio of 1:200 (IIF, methanol/acetone fixation)-1:400 (IIF, paraformaldehyde fixation) |
| Anti-CENP-B antibody | abcam | ab25734 | Rabbit polyclonal antibody; dilution ratio of 1:200 (IIF, methanol/acetone fixation)-1:400 (IIF, paraformaldehyde fixation) |
| Anti-centromere antibody (ACA) | Fitzgerald Industries International, Inc. | 90C-CS1058 | Human centromere antiserum; dilution ratio of 1:2000 (IIF) |
| Anti-CENP-H antibody | Bethyl Laboratories | BL1112 (A400-007A) | Rabbit polyclonal antibody; dilution ratio of 1:200 (IIF) |
| Anti-CENP-H antibody | BD | 612142 | Mouse monoclonal antibody; dilution ratio of 1:200 (IIF) |
| Anti-CENP-I antibody | N/A, Dr. Katusmi Kitagawa | N/A, Dr. Katusmi Kitagawa | Rabbit polyclonal antibody; dilution ratio of 1:1000 (IIF); Niikura et al., Oncogene, 4133-4146 (2006) |
| Anti-KNL1 antibody | Novus Biologicals | NBP1-89223 | Rabbit polyclonal antibody; dilution ratio of 1:100 (IIF) |
| Anti-Hec1 antibody | Novus Biologicals / GeneTex | NB 100-338 / GTX70268 | Mouse monoclonal antibody; dilution ratio of 1:100 (IIF) |
| Anti-Hec1 antibody | GeneTex | GTX110735 | Rabbit polyclonal antibody; dilution ratio of 1:100 (IIF) |
| Anti-Ska1 antibody | abcam | ab46826 | Rabbit polyclonal antibody; dilution ratio of 1:100 (IIF) |
| Anti-Flag antibody | SIGMA-ALDRICH | F3165 | Mouse monoclonal antibody; dilution ratio of 1:1000 (IIF), 1:5000 (WB) |
| Anti-Flag antibody | SIGMA-ALDRICH | F7425 | Rabbit polyclonal antibody; dilution ratio of 1:1000 (IIF), 1:5000 (WB) |
| Anti-CUL4A antibody | N/A, Dr. Pradip Raychaudhuri | N/A, Dr. Pradip Raychaudhuri | Rabbit polyclonal antibody; dilution ratio of 1:3000 (WB); Shiyanov et al., The Journal of biological chemistry, 35309-35312 (1999) |
| Anti-RBX1 antibody | Cell Signaling | 4397 | Rabbit polyclonal antibody; dilution ratio of 1:2000 (WB) |
| Anti-GAPDH antibody | Chemicon | MAB374 | Mouse monoclonal antibody; dilution ratio of 1:5000 (WB) |
| Alexa Fluor 488 Goat Anti-Mouse IgG | Life Technologies/Invitrogen | A11001 | fluorophore-conjugated secondary antibody (Affinity-purified secondary antibody) |
| Alexa Fluor 594 Goat Anti-Mouse IgG | Life Technologies/Invitrogen | A11005 | fluorophore-conjugated secondary antibody (Affinity-purified secondary antibody) |
| Alexa Fluor 488 Goat Anti-Rabbit IgG | Life Technologies/Invitrogen | A11008 | fluorophore-conjugated secondary antibody (Affinity-purified secondary antibody) |
| Alexa Fluor 594 Goat Anti-Rabbit IgG | Life Technologies/Invitrogen | A11012 | fluorophore-conjugated secondary antibody (Affinity-purified secondary antibody) |
| Non fat powdered milk (approved substitution for carnation powdered milk) | Fisher Scientific | NC9255871 (Reorder No. 190915; Lot# 90629) | Skim milk |
| Leica DM IRE2 motorized fluorescence microscope | Leica | motorized fluorescence microscope | |
| HCX PL APO 63x oil immersion lens | Leica | LEICA HCX PL APO NA 1.40 OIL PH 3 CS | 63X oil immersion lens |
| HCX PL APO 100x oil immersion lens | Leica | LEICA HCX PL APO NA 1.40 OIL PHE | 100X oil immersion lens |
| Leica EL6000 compact light source | Leica | External compact light source for fluorescent excitation | |
| ORCA-R2 Digital CCD camera | Hamamatsu | C10600-10B | digital CCD camera |
| Openlab version 5.5.2 Scientific Imaging Software | Perkin Elmer/Improvision | For image observation, acquisition, quantification, and analysis | |
| Velocity version 6.1.1 3D Image Analysis Software | Perkin Elmer/Improvision | For image observation, acquisition, quantification, and analysis | |
| Complete EDTA-free protease inhibitor cocktail | Roche | 11873580001/11836170001 | Protease inhibitor cocktail tablets |
| PlusOne 2-D Quant Kit | Amersham Biosciences | 80-6483-56 | Commercial protein assay reagent I for measurement of protein concentration (compatible with 2% SDS) |
| Bio-Rad Protein Assay | Bio-Rad | 500-0006 | Commercial protein assay reagent II for measurement of protein concentration (compatible with 0.1% SDS) |
| Immobilon-FL | EMD Millipore | IPFL00010 | PVDF membrane for transferring |
| IRDye 800CW Goat Anti-Mouse IgG | LI-COR Biosciences | 926-32210 | IR fluorescent dye-conjugated secondary antibody (Affinity-purified secondary antibody); dilution ratio of 1:20000 (IIF) |
| IRDye 680 Goat Anti-Rabbit IgG | LI-COR Biosciences | 926-32221 | IR fluorescent dye-conjugated secondary antibody (Affinity-purified secondary antibody); dilution ratio of 1:20000 (IIF) |
| Goat anti-Mouse IgG DyLight 549 | Fisher Scientific | PI35507 | DyLight-conjugated secondary antibodyIR fluorescent dye-conjugated secondary antibody (Affinity-purified secondary antibody); dilution ratio of 1:20000 (IIF) |
| Goat anti-Rabbit DyLight 649 | Fisher Scientific | PI35565 | DyLight-conjugated secondary antibodyIR fluorescent dye-conjugated secondary antibody (Affinity-purified secondary antibody); dilution ratio of 1:20000 (IIF) |
| Goat anti-mouse IgG-HRP | Santa Cruz | SC-2005 | HRP-conjugated secondary antibodyDyLight-conjugated secondary antibodyIR fluorescent dye-conjugated secondary antibody (Affinity-purified secondary antibody); dilution ratio of 1:10000 (IIF) |
| Goat anti-rabbit IgG-HRP | Santa Cruz | SC-2004 | HRP-conjugated secondary antibodyDyLight-conjugated secondary antibodyIR fluorescent dye-conjugated secondary antibody (Affinity-purified secondary antibody); dilution ratio of 1:10000 (IIF) |
| Openlab version 5.5.2. Scientific Imaging Software | Improvision/PerkinElmer | Software A | |
| Volocity version 6.3 3D Image Analysis Software (Volocity Acquisition) | PerkinElmer | Software B1 | |
| Volocity version 6.3 3D Image Analysis Software (Volocity Quantification) | PerkinElmer | Software B2 | |
| Branson SONIFIER 450 | Sonicator | ||
| Branson Ultrasonics sonicator Microtip Step, Solid, Threaded 9.5 mm | VWR Scientific Products Inc. | 33995-325 | Disruptor horn for sonication |
| Branson Ultrasonics sonicator Microtip Tapered 6.5 mm | VWR Scientific Products Inc. | 33996-185 | Microtip for sonication |
| Odyssey CLx Infrared imaging System | LI-COR Biosciences | Infrared imaging system for immunoblot detection | |
| Image Studio Analysis Software Ver 4.0 | LI-COR Biosciences | Software C | |
| Molecular Imager Versadoc MP4000 System | Bio-Rad | Chemiluminescence imager for immunoblot detection | |
| Quantity One 1-D analysis software | Bio-Rad | Software D | |
| SuperSignal West Femto Maximum Sensitivity Substrate | Thermo | 34095 | Ultra-sensitive enhanced chemiluminescent (ECL) substrate |
Riferimenti
- DeLuca, J. G., et al. Hec1 and nuf2 are core components of the kinetochore outer plate essential for organizing microtubule attachment sites. Molecular biology of the cell. 16, 519-531 (2005).
- Earnshaw, W. C., Halligan, N., Cooke, C., Rothfield, N. The kinetochore is part of the metaphase chromosome scaffold. J Cell Biol. 98, 352-357 (1984).
- Hoffman, D. B., Pearson, C. G., Yen, T. J., Howell, B. J., Salmon, E. D. Microtubule-dependent changes in assembly of microtubule motor proteins and mitotic spindle checkpoint proteins at PtK1 kinetochores. Molecular biology of the cell. 12, 1995-2009 (2001).
- Regnier, V., et al. CENP-A is required for accurate chromosome segregation and sustained kinetochore association of BubR1. Mol Cell Biol. 25, 3967-3981 (2005).
- Bernad, R., Sanchez, P., Losada, A. Epigenetic specification of centromeres by CENP-A. Exp Cell Res. 315, 3233-3241 (2009).
- Black, B. E., Cleveland, D. W. Epigenetic centromere propagation and the nature of CENP-a nucleosomes. Cell. 144, 471-479 (2011).
- Warburton, P. E., et al. Immunolocalization of CENP-A suggests a distinct nucleosome structure at the inner kinetochore plate of active centromeres. Curr Biol. 7, 901-904 (1997).
- Karpen, G. H., Allshire, R. C. The case for epigenetic effects on centromere identity and function. Trends Genet. 13, 489-496 (1997).
- Black, B. E., et al. Structural determinants for generating centromeric chromatin. Nature. 430, 578-582 (2004).
- Black, B. E., et al. Centromere identity maintained by nucleosomes assembled with histone H3 containing the CENP-A targeting domain. Mol Cell. 25, 309-322 (2007).
- Fachinetti, D., et al. A two-step mechanism for epigenetic specification of centromere identity and function. Nat Cell Biol. 15, 1056-1066 (2013).
- Zeitlin, S. G., Shelby, R. D., Sullivan, K. F. CENP-A is phosphorylated by Aurora B kinase and plays an unexpected role in completion of cytokinesis. The Journal of cell biology. 155, 1147-1157 (2001).
- Zhang, X., Li, X., Marshall, J. B., Zhong, C. X., Dawe, R. K. Phosphoserines on maize CENTROMERIC HISTONE H3 and histone H3 demarcate the centromere and pericentromere during chromosome segregation. The Plant cell. 17, 572-583 (2005).
- Goutte-Gattat, D., et al. Phosphorylation of the CENP-A amino-terminus in mitotic centromeric chromatin is required for kinetochore function. Proc Natl Acad Sci U S A. 110, 8579-8584 (2013).
- Bailey, A. O., et al. Posttranslational modification of CENP-A influences the conformation of centromeric chromatin. Proc Natl Acad Sci U S A. 110, 11827-11832 (2013).
- Samel, A., Cuomo, A., Bonaldi, T., Ehrenhofer-Murray, A. E. Methylation of CenH3 arginine 37 regulates kinetochore integrity and chromosome segregation. Proc Natl Acad Sci U S A. 109, 9029-9034 (2012).
- Niikura, Y., et al. CENP-A K124 Ubiquitylation Is Required for CENP-A Deposition at the Centromere. Dev Cell. , (2015).
- Chan, G. K., Liu, S. T., Yen, T. J. Kinetochore structure and function. Trends in cell biology. 15, 589-598 (2005).
- Hori, T., Okada, M., Maenaka, K., Fukagawa, T. CENP-O class proteins form a stable complex and are required for proper kinetochore function. Molecular biology of the cell. 19, 843-854 (2008).
- Fukagawa, T., Earnshaw, W. C. The centromere: chromatin foundation for the kinetochore machinery. Developmental cell. 30, 496-508 (2014).
- Kedersha, N., Grainger, D. . The Proteintech Blog.Proteintech. , (2012).
- Clontech Laboratories, Inc. . HeLa Tet-Off Advanced Cell Line. , (2012).
- Meraldi, P., Sorger, P. K. A dual role for Bub1 in the spindle checkpoint and chromosome congression. EMBO J. 24, 1621-1633 (2005).
- Niikura, Y., et al. 17-AAG, an Hsp90 inhibitor, causes kinetochore defects: a novel mechanism by which 17-AAG inhibits cell proliferation. Oncogene. 25, 4133-4146 (2006).
- Yang, Z., et al. Silencing mitosin induces misaligned chromosomes, premature chromosome decondensation before anaphase onset, and mitotic cell death. Mol Cell Biol. 25, 4062-4074 (2005).
- Wang, H., et al. Histone H3 and H4 ubiquitylation by the CUL4-DDB-ROC1 ubiquitin ligase facilitates cellular response to DNA damage. Mol Cell. 22, 383-394 (2006).
- Lamb, J. R., Tugendreich, S., Hieter, P. Tetratrico peptide repeat interactions: to TPR or not to TPR?. Trends Biochem Sci. 20, 257-259 (1995).
- Kitagawa, K., Skowyra, D., Elledge, S. J., Harper, J. W., Hieter, P. SGT1 encodes an essential component of the yeast kinetochore assembly pathway and a novel subunit of the SCF ubiquitin ligase complex. Mol Cell. 4, 21-33 (1999).
- Niikura, Y., Dixit, A., Scott, R., Perkins, G., Kitagawa, K. BUB1 mediation of caspase-independent mitotic death determines cell fate. J Cell Biol. 178, 283-296 (2007).
- Niikura, Y., Kitagawa, K. Identification of a novel splice variant: human SGT1B (SUGT1B). DNA Seq. 14, 436-441 (2003).
- Niikura, Y., Ogi, H., Kikuchi, K., Kitagawa, K. BUB3 that dissociates from BUB1 activates caspase-independent mitotic death (CIMD). Cell Death Differ. 17, 1011-1024 (2010).
- Ando, S., Yang, H., Nozaki, N., Okazaki, T., Yoda, K. CENP-A, -B, and -C chromatin complex that contains the I-type alpha-satellite array constitutes the prekinetochore in HeLa cells. Mol Cell Biol. 22, 2229-2241 (2002).
- Izuta, H., et al. Comprehensive analysis of the ICEN (Interphase Centromere Complex) components enriched in the CENP-A chromatin of human cells. Genes to cells : devoted to molecular & cellular mechanisms. 11, 673-684 (2006).
- Obuse, C., et al. Proteomics analysis of the centromere complex from HeLa interphase cells: UV-damaged DNA binding protein 1 (DDB-1) is a component of the CEN-complex, while BMI-1 is transiently co-localized with the centromeric region in interphase. Genes Cells. 9, 105-120 (2004).
- Merlet, J., Burger, J., Gomes, J. E., Pintard, L. Regulation of cullin-RING E3 ubiquitin-ligases by neddylation and dimerization. Cell Mol Life Sci. 66, 1924-1938 (2009).
- Bennett, E. J., Rush, J., Gygi, S. P., Harper, J. W. Dynamics of cullin-RING ubiquitin ligase network revealed by systematic quantitative proteomics. Cell. 143, 951-965 (2010).
- Antonelli, A., et al. Efficient inhibition of macrophage TNF-alpha production upon targeted delivery of K48R ubiquitin. Br J Haematol. 104, 475-481 (1999).
- Codomo, C. A., Furuyama, T., Henikoff, S. CENP-A octamers do not confer a reduction in nucleosome height by AFM. Nat Struct Mol Biol. 21, 4-5 (2014).
- Thrower, J. S., Hoffman, L., Rechsteiner, M., Pickart, C. M. Recognition of the polyubiquitin proteolytic signal. The EMBO journal. 19, 94-102 (2000).
- Yoda, K., et al. Human centromere protein A (CENP-A) can replace histone H3 in nucleosome reconstitution in vitro. Proc Natl Acad Sci U S A. 97, 7266-7271 (2000).
- Shelby, R. D., Vafa, O., Sullivan, K. F. Assembly of CENP-A into centromeric chromatin requires a cooperative array of nucleosomal DNA contact sites. J Cell Biol. 136, 501-513 (1997).
- Majumder, S., Fisk, H. A. Quantitative immunofluorescence assay to measure the variation in protein levels at centrosomes. J Vis Exp. , (2014).
- Masumoto, H., Masukata, H., Muro, Y., Nozaki, N., Okazaki, T. A human centromere antigen (CENP-B) interacts with a short specific sequence in alphoid DNA, a human centromeric satellite. J Cell Biol. 109, 1963-1973 (1989).
- Yoda, K., Kitagawa, K., Masumoto, H., Muro, Y., Okazaki, T. A human centromere protein, CENP-B, has a DNA binding domain containing four potential alpha helices at the NH2 terminus, which is separable from dimerizing activity. J Cell Biol. 119, 1413-1427 (1992).
- Sugata, N., et al. Human CENP-H multimers colocalize with CENP-A and CENP-C at active centromere--kinetochore complexes. Hum Mol Genet. 9, 2919-2926 (2000).
- Earnshaw, W. C., Ratrie, H., Stetten, G. Visualization of centromere proteins CENP-B and CENP-C on a stable dicentric chromosome in cytological spreads. Chromosoma. 98, 1-12 (1989).
- Goshima, G., Kiyomitsu, T., Yoda, K., Yanagida, M. Human centromere chromatin protein hMis12, essential for equal segregation, is independent of CENP-A loading pathway. J Cell Biol. 160, 25-39 (2003).
- Liu, S. T., Rattner, J. B., Jablonski, S. A., Yen, T. J. Mapping the assembly pathways that specify formation of the trilaminar kinetochore plates in human cells. J Cell Biol. 175, 41-53 (2006).
- Gascoigne, K. E., Cheeseman, I. M. T time for point centromeres. Nat Cell Biol. 14, 559-561 (2012).
- Gascoigne, K. E., et al. Induced ectopic kinetochore assembly bypasses the requirement for CENP-A nucleosomes. Cell. 145, 410-422 (2011).
- Nishino, T., et al. CENP-T provides a structural platform for outer kinetochore assembly. EMBO J. 32, 424-436 (2013).
- Malvezzi, F., et al. A structural basis for kinetochore recruitment of the Ndc80 complex via two distinct centromere receptors. EMBO J. 32, 409-423 (2013).
- Schleiffer, A., et al. CENP-T proteins are conserved centromere receptors of the Ndc80 complex. Nat Cell Biol. 14, 604-613 (2012).
- Rago, F., Gascoigne, K. E., Cheeseman, I. M. Distinct organization and regulation of the outer kinetochore KMN network downstream of CENP-C and CENP-T. Curr Biol. 25, 671-677 (2015).
- Nishino, T., et al. CENP-T-W-S-X forms a unique centromeric chromatin structure with a histone-like fold. Cell. 148, 487-501 (2012).
- Bodor, D. L., et al. The quantitative architecture of centromeric chromatin. Elife. 3, e02137 (2014).
- Oliver, C., Rapely, R., Walker, J. M. Ch 58. Molecular Biomethods Handbook. , 1063-1079 (2008).
- Terasima, T., Tolmach, L. J. Changes in x-ray sensitivity of HeLa cells during the division cycle. Nature. 190, 1210-1211 (1961).
- Levenson, G. B. R., Vo-Dinh, T. u. a. n. Ch 8. Biomedical Photonics Handbook. , 8-19 (2003).
- Sanderson, J. . Fluorescence bleed-though. , (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon