Method Article
Misura High Throughput di extracellulare DNA di uscita e quantitativa NET Formazione in neutrofili umani
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
High throughput assays are presented that in combination provide excellent tools to quantitate NET release from human neutrophils.
Abstract
Neutrophil granulocytes are the most abundant leukocytes in the human blood. Neutrophils are the first to arrive at the site of infection. Neutrophils developed several antimicrobial mechanisms including phagocytosis, degranulation and formation of neutrophil extracellular traps (NETs). NETs consist of a DNA scaffold decorated with histones and several granule markers including myeloperoxidase (MPO) and human neutrophil elastase (HNE). NET release is an active process involving characteristic morphological changes of neutrophils leading to expulsion of their DNA into the extracellular space. NETs are essential to fight microbes, but uncontrolled release of NETs has been associated with several disorders. To learn more about the clinical relevance and the mechanism of NET formation, there is a need to have reliable tools capable of NET quantitation.
Here three methods are presented that can assess NET release from human neutrophils in vitro. The first one is a high throughput assay to measure extracellular DNA release from human neutrophils using a membrane impermeable DNA-binding dye. In addition, two other methods are described capable of quantitating NET formation by measuring levels of NET-specific MPO-DNA and HNE-DNA complexes. These microplate-based methods in combination provide great tools to efficiently study the mechanism and regulation of NET formation of human neutrophils.
Introduzione
NET formation is a novel mechanism by which neutrophils fight pathogens.1 The core of NETs is nuclear DNA.1 This DNA network is associated with neutrophil granule proteins and histones.1 The main form of NET formation requires the death of neutrophils characterized by chromatin decondensation, disappearance of granular and nuclear membranes, translocation of neutrophils elastase to the nucleus, citrullination of histones and finally the spill of DNA-based NETs.2 NETs entrap and kill a wide variety of microbes and are an essential part of the innate immune weapon repertoire. Uncontrolled NET formation has, however, been linked to numerous autoinflammatory diseases.3,4 Despite their increasingly established relevance, little is known about the mechanism and regulation of NET release.
Neutrophils dying by releasing NETs are different from apoptotic or necrotic neutrophils.3,5 NET-releasing neutrophils show several features that are characteristic for NET formation. Granule components are associated with DNA in NETs.1 Myeloperoxidase (MPO) and human neutrophil elastase (HNE) are both found in primary granules in resting cells but are translocated to the nucleus to bind to DNA in NETs.1 MPO-DNA and HNE-DNA complexes are specific for NETs, do not occur in apoptotic or necrotic neutrophils.1,3,5 Chromatin decondensation is another feature typical for NETosis.2 NET release also requires citrullination of histones by peptidyl aminidase 4 (PAD4).6 Citrullinated histones are hallmarks of neutrophils that underwent NET release.6
Here three methods are introduced that in combination provide excellent tools to quantitate NETs on a high-throughput scale. The first assay has been used on the field with different changes and quantitates extracellular DNA release in a microplate format. The second and third assays provide confirmation of NETs by measuring NET-specific MPO-DNA and HNE-DNA complexes.
Protocollo
L'Institutional Review Board della Università della Georgia ha approvato lo studio soggetto umano per raccogliere sangue periferico di volontari sani (UGA # 2012-10769-06). 5,7,8 I volontari hanno firmato il modulo di consenso informato richiesto prima di prelievo di sangue. La ricerca svolta in questo articolo è in conformità con le linee guida etiche per la ricerca medica che coinvolge soggetti umani della Dichiarazione di Helsinki.
1. Isolamento di neutrofili dal sangue periferico umano (Figura 1)
Nota: Ci sono diversi modi per isolare neutrofili dal sangue periferico. Il protocollo che segue fornisce una possibilità. Questo protocollo produce un gran numero di neutrofili non attivati umani capaci di liberare NET dopo stimolazione esterna. Utilizzare 40 ml di sangue intero ottenuto da volontari sani mediante prelievo venoso. 7,9
- Aliquota del sangue 20 ml in due provette da 50 ml coniche. Aggiungere 10 ml 6% destrano. Mescolare delicatamente.
- Dopo 20 min di trasferimento del leucociti-rich fase superiore senza prendere i globuli rossi pellet in clean 50 ml provette coniche, e riempire con PBS sterile.
- Centrifugare a 400 xg 10 min. Risospendere il pellet in 4 ml di PBS sterile.
- Preparare un 5-passo gradiente di Percoll 85% -80% -75% -70% -65% in due provette da 15 ml coniche e strato 2 ml di sospensione di leucociti in cima ad ogni sfumatura.
- Centrifuga 800 xg per 30 minuti con freni sbloccati.
- Raccogliere tutte le cellule (neutrofili) negli strati al 75% / 80% e l'interfaccia 70% / 75%.
- Lavare le cellule due volte in PBS (350 xg, 10 min, RT) e contare le cellule utilizzando un emocitometro.
2. Misura di Cinetica di extracellulare DNA di uscita Utilizzando un micropiastre fluorimetro
Nota: Questo metodo consente la misurazione delle variazioni nella fluorescenza di una membrana impermeabile DNA-binding dye indicativa di rilascio DNA su un formato micropiastra a 96 pozzetti (Figura 2).
- Preparare una sospensione di neutrofili nel terreno di coltura (HBSS +1% (v / v) di siero autologo + 5 mM di glucosio) ad una concentrazione di 2 x 10 6 cellule / ml.
- Aggiungere 5 mmol / L del impermeabile tintura DNA-binding membrana. Mescolare delicatamente.
- Aliquota neutrofili umani (50 pl / pozzetto) sui 96 pozzetti nero, fondo trasparente micropiastre. Assicurarsi che il terreno di coltura copre l'intero fondo del pozzo. In caso contrario, toccare delicatamente la piastra sul lato.
- Riscaldare i neutrofili piatto contenente per 10 minuti a 37 ° C.
- Nel frattempo preparare soluzioni di stimoli al doppio della loro concentrazioni finali da utilizzare (in 37 ° C terreno di coltura caldo). Come utilizzare un controllo positivo netto neutrofili umani con 100 nM forbolo-miristato-acetato (PMA) per 4 ore a innescare il rilascio massima NET. 5,8 stimolazione PMA per 4 ore assicura rilascio massima NET mentre la formazione NET spontanea rimane bassa 5,8.
- Preparare il fluorimetro micropiastra per la misurazione (4 ore, 37 ° C, 530 nm per l'eccitazione e 590 nm per l'emissione).
- Stimolare neutrofili con l'aggiunta di una soluzione di stimolo 50 ml a 50 sospensioni neutrofili microlitri. Utilizzare 0,5 mg / ml saponina in un pozzetto per misurare segnale di rilascio del DNA massima.
- Mettere piatto in lettore e misurare le variazioni di fluorescenza per 4 ore ad ogni 2 min senza agitazione.
- Per l'analisi dei dati, calcolare gli aumenti normalizzati a fluorescenza nel tempo.
- Sottrarre basale fluorescenza dai valori endpoint per tutti i pozzetti compresi saponina (Figura 2A, B). Aumento della saponina fluorescenza dovrebbe essere il segnale più alto. Si è indicato come "rilascio DNA massima" (Figura 2A).
- Calcolare le percentuali di rilascio di DNA nei campioni sconosciuti dividendo aumenti di fluorescenza dei campioni sconosciuti dal rilascio del DNA massima.
- Risultati medi di repliche e li presentano come "% del rilascio di DNA massima" (Figura 2C).
3. La quantificazione della Forma NETzione da MPO-DNA e HNE-DNA ELISA saggi
Nota: Questi test modificato (MPO-DNA) o stabilito (HNE-DNA) nel nostro laboratorio di quantificare la formazione NET misurando i livelli di MPO-DNA e complessi HNE-DNA 5.
- Coat 96 pozzetti di alto vincolante capacità piastre per una notte con anticorpi di cattura (50 ml / pozzetto): anti-MPO (1: 2.000 diluizione) o anti-HNE (1: 2.000).
- Lavare le piastre ELISA tre volte con PBS (200 pl / pozzetto) e bloccarli con 5% di BSA e 0,1% di albumina di siero umano per 2 ore a temperatura ambiente (200 pl / pozzetto). Lavare tre volte con PBS.
- neutrofili seme su 96 pozzetti ad una densità di 100.000 cellule / pozzetto in 100 microlitri terreno di coltura, e stimolare la formazione NET. Incubare le cellule: 4 ore per i 37 ° C.
- Eseguire un digestione DNasi limitata con l'aggiunta di 2 U / ml DNasi per supernatanti neutrofili, mescolare bene. Tenerli a temperatura ambiente.
- Arrestare il DNasi dopo 15 min con l'aggiunta di 11 ml di 25 mM EGTA (concentrazione finale 2,5 mm). Mescolare bene. raccogliere supernatants in tubi microcentrifuga pulite. campioni Spin per sbarazzarsi di detriti cellulari a 300 xg per 5 minuti a temperatura ambiente. Trasferire le loro surnatanti in tubi microcentrifuga pulito. Tenerli in ghiaccio fino al momento.
- Utilizzare una aliquota di un preparato in precedenza "standard NET".
Nota: Il NET-standard è un mix di surnatanti DNasi-digerito di neutrofili PMA-stimolati ottenuti da almeno 5 diversi donatori umani indipendenti.- Per utilizzare lo stesso standard per lungo tempo, raccogliere e un'aliquota un grande volume di surnatanti neutrofili dal singolo donatore. Raccolta per esempio 2 ml di surnatanti DNasi-digerito di neutrofili PMA-stimolati si tradurrà in 200 aliquote (10 ml ciascuno) sufficienti per 200 piastre ELISA. Dati che caratterizzano lo standard NET si trovano in figura 5.
- Stimolare neutrofili umani con 100 nM PMA per 4 ore e applicare DNasi digerire ai loro surnatanti secondo passi 3.4-3.5.
- Raccogliere, piscina, aliquota (10 ml) e la connessionesurnatanti ze (-20 ° C) dei neutrofili.
- Per preparare il "NET-standard", disgelo un'aliquota / donatore ottenuto da almeno cinque donatori separati, mescolarli e tenerli in ghiaccio.
- Eliminare i campioni scongelati e usare quelli freschi per il prossimo esperimento.
- Preparare una diluizione 1: 2 di serie del NET standard in PBS + EGTA.
- Diluire i campioni DNase1-digerite (standard e supernatanti sconosciuti) 20 volte in PBS + EGTA e li aliquota su piastre ELISA rivestiti con anticorpi di cattura.
- Incubare le piastre ELISA per tutta la notte a 4 ° C e lavarli tre volte con PBS.
- Applicare la soluzione anticorpo di rilevazione (100 l / pozzetto): perossidasi di rafano coniugato anticorpo anti-DNA (1: 500, mouse). Incubare per 1 ora al buio.
- Dopo quattro lavaggi con PBS aggiungere TMB substrato della perossidasi: 100 ml / pozzetto, 30 min. Colorazione blu è un'indicazione per l'attività perossidasi (NET presenti).
- Arrestare reazione aggiungendo 100 microlitri / pozzetto 1 M HCl. IlLe soluzioni blu si accende di colore giallo (Figura 5A).
- Leggere l'assorbanza a 450 nm con un fotometro per micropiastre.
- Per l'analisi dei dati, calcolare la quantità di MPO-DNA o complessi HNE-DNA rispetto allo standard NET.
- Sottrarre il valore di densità ottica sfondo del terreno di coltura da tutti i campioni.
- Tracciare i valori di assorbanza (asse X) dei campioni di riferimento diluiti contro il loro contenuto netto relativa (asse Y) (Figura 5A).
- Utilizzando la gamma insaturi di questa curva standard stabilire una linea di tendenza (usare soluzione migliore esponenziale dal software utilizzato) (Figura 5A). L'equazione di questa linea di tendenza determina la conversione dei valori di DO per quantitativi quantità di reti.
- Inserire i valori OD misurati dei incognite nell'equazione trend-line da 3.13.3 che darà la quantità di reti nel campione sconosciuto come percentuale del prezzo netto standard (Figura 5A).
- Calcolaremedie di repliche (triplicato) e presentare i dati finali come "quantità di MPO-DNA o complessi HNE-DNA (% del campione)" (figura 5c).
Risultati
Le figure in questo manoscritto descrivono il metodo di isolamento dei neutrofili, procedure sperimentali, e presentano risultati rappresentativi con spiegazione di analisi dei dati. La Figura 1 mostra le fasi sequenziali di preparazione neutrofili umani. Questo protocollo rappresenta solo un modo possibile di isolamento dei neutrofili. Si produce grandi quantità di neutrofili in grado di rilasciare NET sulla stimolazione di riposo. La figura 2 mostra come funziona il test di rilascio del DNA basato sulla fluorescenza. Utilizzando una fluorescenza Lettore di micropiastre cinetica di rilascio di DNA nei neutrofili umani sono seguite a 96 pozzetti. Figura 3 illustra ogni fase dell'analisi dei dati. In primo luogo, il calcolo del segnale del DNA massima nei neutrofili trattati con saponina (Figura 3A). Aumento della fluorescenza è sempre calcolato come differenza tra il valore di fluorescenza più alto e il livello di fondo di fluorescenza ( Figura 3A). Questo viene calcolato per ciascun pozzetto. L'esempio mostra i dati rappresentativi di neutrofili umani lasciati non stimolate o attivati con PMA o P. aeruginosa (PAO1) (Figura 3B). P. aeruginosa innesca la formazione di NET in PMN umani. 5,8,10 E 'stato dimostrato con diversi P. aeruginosa ceppi compresi PAO1 che solo i batteri (senza neutrofili) non vincolano il colorante DNA di rilevamento utilizzato. 8 PAO1 P. aeruginosa è stato ottenuto da ATCC, coltivate in Luria-Bertani medio overnight, raccolte in fase tardiva-esponenziale, lavate in terreno di coltura e usato per stimolare neutrofili ad una molteplicità di infezione (MOI) di 10: 1 (Figura 3B, C, 4B, C). Medie di aumento della fluorescenza di pozzi repliche sono calcolati, normalizzato fluorescenza massima (saponina) e sono espressi come "rilascio DNA (% max)" (Figura 3C). La figura 3D mostra FLUORES rappresentativiimmagini Cence di neutrofili non trattati o PMA-stimolate.
Figura 4A illustra lo schema che spiega i principi della MPO-DNA e HNE-DNA test ELISA. La figura 4B mostra immagini rappresentative di immunofluorescenza di non stimolate, PMA- e neutrofili umani PAO1-attivato. Extracellulare DNA mostra morfologia tipica NET e co-localizza con MPO e citrullinato H4 istone, un marker di formazione NET. 6 Figura 4C mostra MPO-DNA e HNE-DNA rilascio nei neutrofili umani nelle stesse condizioni precedenti (come la figura 3B, 3C e 4B). IThe "NET" metodi ELISA sono stati precedentemente caratterizzati. 5 Brevemente, 2 U / ml trattamento DNasi (equivalente di 1 mg / concentrazione ml di DNasi utilizzato) per 15 min a RT portato massimi MPO-DNA e HNE-DNA segnali . 5 campioni da corsa (100 ng / corsia) su un gel di agarosio ha rivelato che la lunghezza del DNANella gamma dei rendimenti inferiori kb ottimale ELISA segnale. 5 del DNA overdigestion (DNasi posologia superiore a 20 U / ml) o l'omissione o la cattura o l'anticorpo di rilevazione aboliti i segnali ELISA. 5
Inoltre, qui i saggi e lo "standard NETWORK" sono ulteriormente caratterizzato (Figura 5A). Il NET-standard contiene in media 19,5 ug / ml di DNA extracellulare (misurata usando uno standard con concentrazioni di DNA note), 399 ng / ml MPO e 103,4 ng / ml HNE (ELISAkits commerciali) (n = 8). Sulla base dei pesi molecolari totali di MPO (84 kDa) e HNE (29,5 kDa) e il fatto che 1 mg DNA contiene 9,91 x 10 14 nucleotidi, il NET-standard contiene 109 HNE molecole e 147 MPO molecole per 1000 nucleotidi (kb DNA ). Due-fold esperimenti di diluizione seriali della rete standard hanno rivelato che le gamme dinamiche dei dosaggi sia del MPO-DNA e HNE-DNA ELISA sono tra 19,0-609,0 ng /Le concentrazioni ml di DNA (Figura 5B). Diluizione inferiore a 32 volte dello standard NET portato in saturazione, mentre diluizioni superiori a 1.024 volte, non erano diversi dallo sfondo (Figura 5B). Trattamento della norma NET con inibitori della proteasi non ha cambiato i risultati ottenuti dai test MPO-DNA e HNE-DNA che indicano che le proteasi non interferire con questi test (Figura 5C). Così, i risultati ottenuti nello stesso campione NET standard da parte MPO-DNA o HNE-DNA ELISA saggi fortemente correlato con l'altro (Figura 5D). Per dimostrare che surnatanti PMN congelati possono essere utilizzati anche in questi test senza perdere efficienza, alcune aliquote della rete standard sono stati lasciati non trattati o altri sono stati esposti a una o due di congelamento / scongelamento cicli eseguiti a -20 ° C o -80 ° C (1 intervallo di ora). Nessuno dei trattamenti di congelamento / scongelamento colpito i MPO-DNA o HNE-DNA risultati (Figura 5e). In sintesi, laNET-standard fornisce un punto di riferimento affidabile in questi test NET-quantificazione consentono il confronto quantitativo dei risultati netti tra esperimenti indipendenti.

Figura 1:. Isolamento di granulociti neutrofili umani neutrofili sono stati isolati da sangue periferico di volontari secondo il protocollo descritto. Il sangue contenente anticoagulante è stato usato per isolare neutrofili di sedimentazione destrano e centrifugazione in gradiente. Il sangue senza anticoagulante è stato usato per preparare il siero. Viabilità delle preparazioni dei neutrofili è valutata mediante test di esclusione Trypan Blue. Neutrofili purezza è valutata cytospin o citometria a flusso (CD16, CD66abcd cellule doppio positive). Clicca qui per vedere una versione più grande di questa figura.
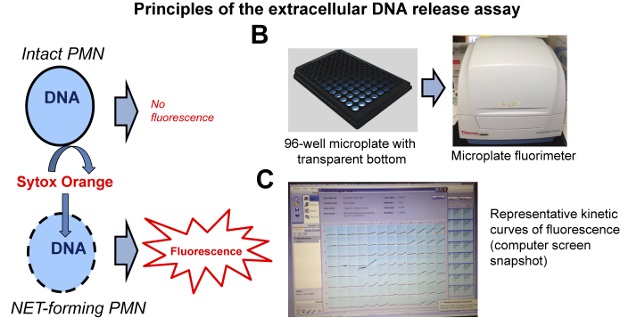
Figura 2:. Principi del extracellulare DNA saggio di rilascio (A) fluorescenza si ottiene solo in neutrofili cui plasma e membrane nucleari sono stati compromessi e il colorante DNA-binding membrana impermeabile in grado di legarsi al loro DNA. (B) Il test viene eseguito su nero 96 pozzetti utilizzando un fluorimetro micropiastra. (C) Il fluorimetro registra le curve di fluorescenza in tempo reale in ogni pozzetto. Uno dei risultati di rappresentanza in mostrato. Cliccate qui per vedere una versione più grande di questa figura.
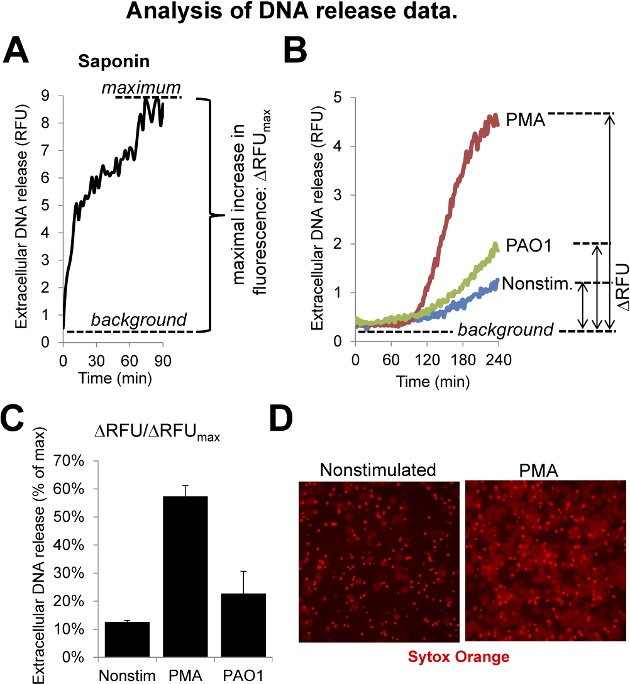
Figura 3: Analisi del DNA di rilascio dei dati (. A) saponina viene utilizzato per creare il valore massimo in quanto lisa interamente neutrofili ed espone loro DNA. La cinetica di fluorescenza indotta saponina è mostrato. (B) Cinetica di rilascio di neutrofili DNA indotta da PMA (100 nM) e batteri (Pseudomonas aeruginosa PAO1, 10 MOI). (C) normalizzato riassume i dati di rilascio di DNA ottenuti in una esperimenti usando quadriplicates. (D) immagini di fluorescenza rappresentativi di non trattate e PMA-stimolati neutrofili umani (Sytox Arancione, 4 ore di incubazione). Clicca qui per vedere una versione più grande di questa figura.

Figura 4: MPO-DNA e HNE-DNA ELISA saggi:. Principio e Rappresentante Risultati (A) Schema di spiegareing come il MPO-DNA e il lavoro saggi HNE-DNA ELISA. (B) I risultati rappresentativi delle immagini di immunofluorescenza preparati sui neutrofili umani esposti a 100 nm PMA o P. aeruginosa ceppo PAO1 (10 MOI) per 4 ore. Extracellulare DNA (DAPI, blu) co-localizza in reti con MPO (verde) e H4 istone citrullinato (rosso). Un risultato rappresentativo, n = 3. (C) rilascio NET stata misurata nei neutrofili umani esposti agli stessi stimoli come sopra dalla MPO-DNA e saggi HNE-DNA ELISA. Media ± SEM, n = 3. Fate clic qui per vedere una versione più grande di questa figura.

Figura 5: Caratterizzazione dei "NET-standard" (A) diluizioni seriali. (1: 2) dello standard NET sono stati preparatie sottoposti a MPO-DNA ELISA. valori di OD misurati sono tracciate contro% del contenuto netto standard. "X" rossa indica un valore OD misurata di un campione sconosciuto. freccia grigia indica come "concentrazione NET" (blu "X") del campione sconosciuto è determinato utilizzando la gamma centrale della curva standard montata. Concentrazioni (B) DNA in campioni NET standard serialmente diluiti sono stati determinati e tracciati contro i valori di OD misurati del test ELISA MPO-DNA o HNE-DNA. aree grigie indicano le gamme dinamiche dei saggi. (C) il trattamento con inibitori della proteasi (cocktail di inibitori delle proteasi, 1%) non pregiudica l'esito della MPO-DNA o test HNE-DNA (NET-standard). Media ± SEM, n = 2 (D) Correlazione di MPO-DNA e risultati HNE-DNA (OD 450 nm) di diluiti campioni "NET-standard" nelle loro gamme dinamiche (vedi 5B). (E) di gelo e disgelo cicli (-20 ° C o -80 ° C; oNE o due) non incidono i risultati di MPO-DNA e saggi HNE-DNA (NET standard, media ± SEM, n = 2). Si prega di cliccare qui per vedere una versione più grande di questa figura.
Discussione
NET rappresentano un affascinante romanzo meccanismo attraverso il quale i neutrofili uccidere gli agenti patogeni. 1 Sebbene la letteratura di NET ha continuato ad ampliarsi nel corso degli ultimi dieci anni dalla loro scoperta, diverse questioni importanti relative al loro ruolo nella biologia, meccanismo e regolazione rimangono poco chiari. metodologia appropriata deve essere sviluppato per misurare NET, questo meccanismo antimicrobico molto singolare. Questo articolo vengono descritti metodi che possono essere utilizzati per quantificare NET in modo elevato throughput. Il primo test segue cinetiche di fluorescenza del colorante DNA-binding membrana impermeabile. Questo test fornisce numerose informazioni sulla pendenza e la velocità di rilascio di DNA dai neutrofili. Questa informazione è fondamentale nelle indagini che studiano eventi all'inizio di segnalazione che portano alla formazione di NET. Tutte queste informazioni si perde in saggi endpoint misurando il rilascio di DNA al termine dell'incubazione. Il saggio extracellulare a base di colorante DNA non è specifico per le reti. essomisura solo rilascio del DNA. Per confermare NET, è ampiamente accettato per eseguire immunofluorescenza sui neutrofili NET-formatura e mostrare co-localizzazione del DNA sia con marcatori granuli (MPO, HNE) o istoni. La quantificazione dei dati immunofluorescenza è però molto noioso e soggettiva.
Qui, una modifica di un saggio ELISA precedentemente sviluppato rilevare complessi MPO-DNA è presentato. A simili ELISA misura test complessi HNE-DNA che rappresentano misure specifiche NET è anche descritto. 3,5 Entrambi i test sono stati caratterizzati in dettaglio. 5 La MPO-DNA e HNE-DNA ELISA test sono quantitative e NET-specifica allo stesso tempo . 5 Queste caratteristiche non sono soddisfatte con qualsiasi altro metodo corrente. I saggi sono facili da eseguire e fornire i dati riproducibili. 5 Un passo DNasi digestione limitata è incluso che garantisce la gestione rapida e standardizzata di NET direttamente sul neutrofili. 5 Questi test sono adatti a COMpare più campioni contemporaneamente. Nelle nuove, misure di controllo è dimostrata la cicli di gelo / disgelo e proteasi non influenzano il risultato dei test MPO-DNA e HNE-DNA. Non è noto, tuttavia, se le proteine note per legano al DNA in NET (compresi istoni e LL-37) 1,11 potrebbero interferire con questi test.
Passaggi critici dei protocolli presentati sono i seguenti. E 'importante raccogliere i neutrofili che sono in una di riposo, stato di riposo e non rilasciano grandi quantità di NET spontaneamente. La mancanza di un passo lisi ipotonica per rimuovere i globuli rossi nel protocollo di isolamento dei neutrofili presentato è fondamentale per ottenere i neutrofili non attivati. Per inibire l'attivazione dei neutrofili dopo che le cellule erano state purificate, è anche importante per risospendere neutrofili in un mezzo contenente siero propria del loro donatore. Per ottenere cellule quiescenti ma è anche fondamentale per utilizzare reagenti pyrogen- e privo di contaminazione durante la preparazione dei neutrofili. SPONTAnea rilascio NET di neutrofili durante il test di rilascio del DNA extracellulare potrebbe anche accadere. Aggiunta di 1% di siero autologo al terreno di coltura è importante per evitare che. dosi superiori o inferiori del siero autologo potrebbero essere testati per ottenere risultati ottimali. È critico durante la MPO-DNA e saggi HNE-DNA ELISA che chiarificati DNA non dovrebbe essere troppo digerito dal DNasi perché comporta la perdita completa del segnale. Pertanto, la dose ottimale dovrebbe essere determinata per ogni nuovo lotto DNasi. I parametri della digestione DNA potrebbero essere leggermente modificati per ottenere segnali possibile il massimo ELISA. E 'anche importante mescolare il DNasi e NET accuratamente per garantire che la DNasi avrà accesso chiaro al DNA. Quando la raccolta Le reti digerito, lavare il pozzo ampiamente per raccogliere tutte le reti.
Il limite di rilevazione del DNA del MPO-DNA e saggi HNE-DNA è di circa 10-20 DNA ng / ml (Figura 5B). Siero e DNA libero livelli plasmatici di severAl cento a mille ng / ml sono stati riportati in malattie (cancro, lupus eritematoso sistemico, infezione del miocardio) che sono stati associati con la formazione NET anormale. 12-14 Ciò suggerisce che il test MPO-DNA e HNE-DNA ELISA hanno anche il potenziale essere in grado di quantificare NET in campioni clinici umani. Questa futura applicazione del MPO-DNA e saggi HNE-DNA ELISA consentirà di collegare quantitativamente reti per patogenesi della malattia.
Divulgazioni
The authors have nothing to disclose.
Riconoscimenti
Special thanks to the personnel of the University of Georgia Health Center laboratory for their continuous support of our work on isolating human neutrophils. This work was supported by the start-up fund of Dr. Rada provided by UGA Office of Vice President for Research.
Materiali
| Name | Company | Catalog Number | Comments |
| Anti-Human Neutrophil Elastase Rabbit Ab | Calbiochem | 481001 | 1:2,000x coated |
| Anti-Myeloperoxidase Ab (Rabbit) | Millipore | 07-496 | 1:2,000x coated |
| DNase-1 | Roche | 10-104-159-001 | 1 µg/ml used for digestion |
| 20 mM EGTA/ PBS | Sigma-Aldrich | E3889-25G | |
| 2.5 mM EGTA/PBS | Sigma-Aldrich | E3889-25G | |
| Cell death detection ELISA Anti-DNA POD | Roche | 11544675001 | 1:500x |
| Eon Microplate Spectrophotometer | Biotek | ||
| Gen5 All-in-One microplate software | Biotek | analytical tool (ELISA) | |
| Sytox orange | Life Technology | S11368 | 0.2% final concentration/volume |
| 1 M Hepes | Cellgro | 25-060-Cl | Use 10 mM final concentration. |
| 1 M glucose | Sigma | Use 5 mM final concentration. | |
| HBSS | Corning | 21-023-CM | |
| Varioskan Flash Ver.2.4.3 | Thermoscientific | ||
| PMA | Sigma | P 8139 | 100 nM final used |
| ELISA Plate | Greiner bio-one | 655061 | |
| Conical tubes 15 ml | Thermoscientific | 339650 | |
| Conical tubes 50 ml | Thermoscientific | 339652 | |
| Percoll (pH 8.5-9.5) | Sigma | P 1644 | Sodium Chloride, Sigma, S7653-250G |
| Dextran | Spectrum | D1004 | |
| RPMI 1640 media | Corning Cellgro | 17-105-CV | |
| 96 well assay plate black plate clear bottom | Costar | 3603 |
Riferimenti
- Brinkmann, V., et al. Neutrophil extracellular traps kill bacteria. Science. 303, 1532-1535 (2004).
- Fuchs, T. A., et al. Novel cell death program leads to neutrophil extracellular traps. J Cell Biol. 176, 231-241 (2007).
- Kessenbrock, K., et al. Netting neutrophils in autoimmune small-vessel vasculitis. Nat Med. 15, 623-625 (2009).
- Leffler, J., et al. Degradation of neutrophil extracellular traps co-varies with disease activity in patients with systemic lupus erythematosus. Arthritis Res Ther. 15, R34(2013).
- Yoo, D. G., Floyd, M., Winn, M., Moskowitz, S. M., Rada, B. NET formation induced by Pseudomonas aeruginosa cystic fibrosis isolates measured as release of myeloperoxidase-DNA and neutrophil elastase-DNA complexes. Immunol Lett. 160, 186-194 (2014).
- Wang, Y., et al. Histone hypercitrullination mediates chromatin decondensation and neutrophil extracellular trap formation. J Cell Biol. 184, 205-213 (2009).
- Pang, L., Hayes, C. P., Buac, K., Yoo, D. G., Rada, B. Pseudogout-associated inflammatory calcium pyrophosphate dihydrate microcrystals induce formation of neutrophil extracellular traps. J Immunol. 190, 6488-6500 (2013).
- Yoo, D. G., et al. Release of cystic fibrosis airway inflammatory markers from Pseudomonas aeruginosa-stimulated human neutrophils involves NADPH oxidase-dependent extracellular DNA trap formation. J. Immunol. 192, 4728-4738 (2014).
- Brinkmann, V., Laube, B., Abu Abed,, Goosmann, U., Zychlinsky, C., A, Neutrophil extracellular traps, how to generate and visualize them. J. Vis. Exp. , (2010).
- Rada, B., et al. Pyocyanin-enhanced neutrophil extracellular trap formation requires the NADPH oxidase. PLoS One. 8, e54205(2013).
- Neumann, A., et al. The antimicrobial peptide LL-37 facilitates the formation of neutrophil extracellular traps. Biochem J. 464, 3-11 (2014).
- Leon, S. A., Shapiro, B., Sklaroff, D. M., Yaros, M. J. Free DNA in the serum of cancer patients and the effect of therapy. Cancer Res. 37, 646-650 (1977).
- Raptis, L., Menard, H. A. Quantitation and characterization of plasma DNA in normals and patients with systemic lupus erythematosus. J Clin Invest. 66, 1391-1399 (1980).
- Chang, C. P., et al. Elevated cell-free serum DNA detected in patients with myocardial infarction. Clin Chim Acta. 327, 95-101 (2003).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Tutti i diritti riservati
Utilizziamo i cookies per migliorare la tua esperienza sul nostro sito web.
Continuando a utilizzare il nostro sito web o cliccando “Continua”, accetti l'utilizzo dei cookies.
