Method Article
Site-specific Ingegneria batterica Cromosoma: ΦC31 integrasi Mediated Cassette Exchange (IMCE)
In questo articolo
Riepilogo
Un metodo rapido ed efficace per integrare DNA estraneo di interesse in pre-fatti ceppi accettori, chiamati ceppi pista di atterraggio, è descritta. Il metodo permette integrazione sito-specifica di una cassetta DNA nel locus ingegnerizzato atterraggio di un dato ceppo, tramite coniugazione e l'espressione del ΦC31 integrasi.
Abstract
Il cromosoma batterico può essere utilizzato per mantenere stabilmente il DNA estraneo nel mega-base gamma 1. Integrazione nel cromosoma elude questioni quali la replicazione del plasmide, la stabilità plasmide, incompatibilità plasmide, e il plasmide varianza numero di copie. Questo metodo utilizza il site-specific integrasi dal fago Streptomyces (Φ) C31 2,3. Il ΦC31 integrasi catalizza una ricombinazione diretto tra due specifici siti del DNA: ATTB e attP (34 e 39 bp, rispettivamente) 4. Tale ricombinazione è stabile e non ripristinare 5. Una "pista" (LP) costituito da una sequenza spectinomicina gene di resistenza, aadA (SPR), e la E. coli ß-glucuronidasi gene (uidA) affiancato da siti attP è stato integrato nei cromosomi di Sinorhizobium meliloti, Ochrobactrum anthropi e tumefaciens in una regione intergenica, l'ampC locus, e il locus teta, rispettivamente. S. meliloti viene utilizzato in questo protocollo. Vettori contenenti siti donatori mobilizzabile attB fiancheggiano una stuffer rosso proteina fluorescente (RFP) gene e un gene di resistenza agli antibiotici sono stati costruiti. In questo esempio la gentamicina pJH110 resistente plasmide viene utilizzato. Il gene RFP 6 può essere sostituito con un costrutto desiderata con Sph I e Pst I. In alternativa, un costrutto sintetico fiancheggiata da siti attB possono essere subclonato in un vettore mobilizzabile come pK19mob 7. L'espressione del gene ΦC31 integrasi (clonato da pHS62 8) è guidata dal promotore lac, su una vasta gamma mobilizzabile ospite plasmide pRK7813 9.
Un protocollo accoppiamento tetraparental viene utilizzato per trasferire la cassetta nel ceppo donatore LP sostituendo i marcatori nella sequenza LP con la cassetta donatore. Queste cellule sono trans-integrants. Trans-integrants sono formate con una efficienza tipica di 0,5%. Trans-integrants trovano tipicamente entro i primi 500-1.000 colonie schermati dalla sensibilità agli antibiotici o blu-nero screening utilizzando 5-bromo-4-cloro-3-indolil-beta-D-glucuronico (X-Gluc). Questo protocollo contiene le procedure di accoppiamento e di selezione per la creazione e isolare trans integranti.
Protocollo
1. Produzione di Cultura
- Preparare liquidi sterili: TY 10 (5 g / l triptone, 3 g / l di estratto di lievito, 0,44 g / l di cloruro di calcio diidrato), e LB 11 (10 g / l triptone, 5 g / l di estratto di lievito, 5 g / cloruro di sodio, pH 7).
- Inoculare da una singola colonia: SmUW227 (S. meliloti LP-ceppo: verranno descritte altrove, ceppo dettagli costruttivi disponibili su richiesta) () in 5 ml di TY media con 50 ug / ml spectinomicina. Inoculare le seguenti ceppi in 5 ml di liquidi LB, che integra con l'antibiotico data: E. coli DH5a 12 contenente pJC2, il plasmide di espressione integrasi, con 10 pg / ml di tetraciclina; E. coli MT616 13, il mobilizzatore, con 10 pg / ml cloramfenicolo, e E. coli DH5a contenente pJH110, la cassetta plasmide donatore, con 5 pg / ml di gentamicina.
- Incubare E. ceppi di E. notte a 37 ° C con costante agitazione. Incubare S. meliloti deformazione a 30 ° C per due giorni con agitazione. È possibile ottenere una S. meliloti cultura per tutta la notte con un inoculo più grande (1:500 sottocultura di una cultura satura).
2. Cultura preparazione e miscelazione
- Lavare 1,5 ml di coltura di ciascun ceppo raccogliendo il pellet cellulare mediante centrifugazione a 17.000 xg per 30 secondi e risospeso in 1 ml di NaCl sterile 0,85%; ripetere una volta.
- Risospendere il pellet in 100 microlitri di sterile allo 0,85% NaCl.
- Individualmente individuare 10 pl della cultura lavato per ogni ceppo su una pianura piastra di agar TY. Questi punti sono punti di controllo.
- Aggiungere 40 pl di ciascun ceppo esclusa la E. coli DH5a 12 contenente pJC2 in una provetta sterile, mescolare, e spot 120 microlitri di questa miscela in una pianura piastra di agar TY. Questo punto è il no-integrasi controllo negativo.
- Aggiungere 40 pl di ciascun ceppo in un t sterileUbe, mix, e spot 160 microlitri di questa miscela in una pianura piastra di agar TY. Questo luogo è il punto di accoppiamento IMCE.
- Lasciare punti asciugare in una cappa a flusso laminare.
- Piastre di tenuta e incubare a 30 ° C per una notte.
3. Isolamento di Trans-integrants
- Punti di controllo Streak e spot IMCE su piastre di agar TY integrato con 200 mg / ml di streptomicina (SmUW227 si basa su Rm1021 che trasporta una mutazione conferisce resistenza di alto livello alla streptomicina 14) e 30 mg / ml di gentamicina.
- Per il calcolo del rendimento IMCE, risospendere circa ¼ del punto di accoppiamento IMCE in 500 microlitri sterile NaCl allo 0,85%. Effettuare diluizioni seriali di 10 volte a 10 -7. Piatto diluizioni 10 -2 a 10 -5 su piastre di agar TY integrati con 200 mg / ml di streptomicina e 30 mg / ml di gentamicina, di selezionare per trans integranti. Piatto diluizioni 10 -3 through 10 -7 su piastre di agar TY integrati con 200 mg / ml di streptomicina, per selezionare sia per i trans integranti e dei destinatari potenziali, cioè dei destinatari totali.
- Per l'isolamento di markerless trans integrants risospendere circa un quarto del punto di accoppiamento IMCE in 500 pl di sterile 0,85% NaCl. Effettuare diluizioni seriali di 10 volte a 10 -6. Diluizioni tavola 10 -4 a 10 -6 su quattro piastre di agar TY integrati con 200 mg / ml di streptomicina e 200 mg / ml X-Gluc.
- Incubare le piastre a 30 ° C per 3 giorni.
- Visualizza RFP trans integrants sotto la luce verde (525 nm) e attraverso un filtro rosso (> 610 nm) 15.
- Calcolare l'efficienza IMCE per cento come CFU di trans-integrants CFU rispetto al totale dei destinatari. Circa la metà di trans-integrants sono sottoposti vero scambio cassette che li rende bianchi e spectinomicina sensibile.
- Per trovare i markerless trans integrants (wqui il vettore donatore non contiene Gm r come in pJH110) schermo per una colonia bianco (un periodo di incubazione prolungato, 1-2 giorni supplementari a temperatura ambiente, aumenterà la differenziazione delle colonie tramite X-Gluc, ciò è necessario in S. meliloti SmUW227), confermare la sensibilità del spectinomicina della colonia mediante screening su una piastra di agar TY senza antibiotici e una piastra di agar TY contenente 100 ug / ml spectinomicina.
4. Risultati rappresentativi
Dopo tre giorni di incubazione su TY integrato con streptomicina e gentamicina le strisce di controllo non dovrebbe avere alcuna crescita. La striscia IMCE dovrebbe avere crescita confluente sulla striscia testa e molte colonie sulla seconda striscia, come mostrato nella Figura 2. L'efficienza di trans-integrazione, espressa come percentuale di trans-integrants ai destinatari totale, dovrebbe essere nell'intervallo di 0,5%. Circa la metà di trans-integrants sarà spectinomicina sensibili e nerodimostrando di avere subito vero e proprio scambio cassetta. Trans-integrants contenenti il donatore tester rfp cassetta dalla pJH110 dovrebbe visualizzare distinguibile fluorescenza RFP se visto sotto la luce verde (525 nm) e attraverso un filtro rosso (> 610 nm) 15.
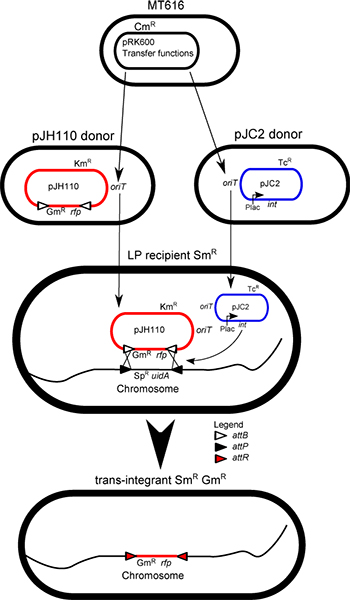
. Figura 1 illustrazione miscela coniugazione: Con l'aiuto della espressione dei geni di trasferimento di pRK600, tutti i plasmidi sono trasferiti in modo casuale da una cella all'altra. Questo trasferimento comporta la creazione di trans-integrants nella miscela, attraverso l'acquisizione LP-ceppo dei due plasmidi necessari per IMCE, l'integrasi (int) pJC2 plasmide helper e la pJH110 plasmide donatore. La cassetta del donatore non replicante plasmide donatore (pJH110) viene scambiata attraverso ΦC31 integrasi con i marcatori del LP-cassetta sul cromosoma, con conseguente perdita dei marcatori LP (SpR e uidA) e il mantenimento della cassetta donatore (RFP e Gm r) nel risultante trans-integrante.
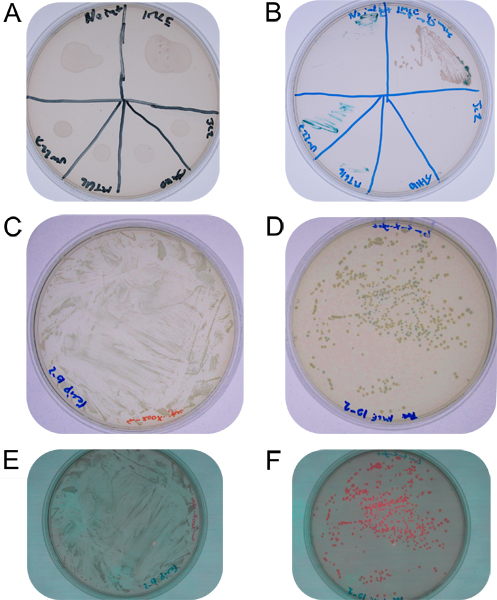
Figura 2. A:. Macchia piastra di accoppiamento in modo non selettivo piastra TY mostrando miscele di cellule secche sulla superficie agar B: punti di accoppiamento seminata su streptomicina, gentamicina-X-Gluc piatto, dall'alto a sinistra in senso orario alcun controllo integrasi, IMCE in S. meliloti, E. coli DH5a contenente il controllo pJC2, E. coli DH5a contenente pJH110 controllo, E. coli MT616 controllo e S. . meliloti UW227 di controllo C: 10 -2 diluizione di risospensione accoppiamento punto sul TY streptomicina-X-agar Gluc D:. diluizione 10 -2 di risospensione accoppiamento punto sul TY streptomicina-gentamicina-X-Gluc agar (colonie blu sono ricombinanti singoli, colonie bianche sono stati sottoposti a vere. scambio di cassette) E: 10 -2 diluizione di risospensione punto di accoppiamento sulla streptomicina TY-X-agar Gluc che mostra la mancanza di fluorescenza F:. diluizione 10 -2 di risospensione accoppiamento punto sul TY streptomicina-gentamicina-X-agar Gluc che mostra due livelli di fluorescenza (più brillanti colonie corrispondono a colonie blu e hanno una maggiore espressione rfp presumibilmente a causa di promotore leggere, anche se dalla sequenza vettoriale, dove le colonie che hanno subito true-cassette contengono RFP scambio solo con il suo promotore immediata senza read-through da parte del promotore lac in il vettore, che è assente.).
Discussione
La tecnica IMCE consente l'integrazione efficace di una singola cassetta DNA attB affiancato nella LP-locus di un ceppo precedentemente progettato. Una volta che il costrutto desiderato viene clonato al posto di RFP per creare la cassetta donatore, la tecnica non richiede successiva purificazione del DNA e trasformazione, che rende molto robusto. È fondamentale che controlli crescita appropriati sono inclusi, per essere certi della resistenza agli antibiotici è dovuta alla creazione di trans-integrants e fattori non altri.
IMCE produce trans integrants a circa il 0,5% di efficienza. Al contrario, crossover doppia via ricombinazione omologa avviene ad una frequenza di circa 10 -6. Recombineering via λ-rosso 16, un altro sistema basato su fago, richiede omologia specifico, e ha variato efficacia fuori di E. coli 17. Ancora un'altra opzione, Tn7 ricombinazione sito-specifica richiede attTn7 18 siti , che sono raramente presenti al di fuori della γ-proteobatteri. Il ΦC31 integrasi ha dimostrato efficace attività negli ospiti disparati 4,19. Sebbene l'uso di ΦC31 dell'integrasi richiede ingegneria preventiva di un ospite, essa non è limitata a certi phyla. IMCE presenta i vantaggi di efficienza e modularità dato che ogni cassetta donatore possono potenzialmente essere integrato in qualsiasi LP-strain. E 'ideale per gli studi in cui una libreria singolo clone deve essere funzionalmente proiettati in più host a causa dei requisiti di espressione genica o complementazione genetica in singola copia si voglia.
La dimensione del costrutto da integrare è limitata dalla dimensione del DNA che può essere clonato con successo nel vettore donatore tramite ligazione. Vettori di donatori contenenti destinazioni Gateway 20 può essere utilizzato, e sono particolarmente utili per le grandi inserto fosmid / cosmide librerie. I marcatori di resistenza agli antibiotici nei vettori donatori possono anche essere utilizzati per selezionare per tegli assemblaggio di frammenti sovrapposti DNA messaggio IMCE dall'incrocio di due differenti trans integrants via di trasduzione, o elettroporazione genomica 21. Questa considerazione di design dovrebbe consentire il montaggio di costrutti molto ampie LP-locus nel LP-strain. Questo potrebbe essere particolarmente utile per l'incorporazione dei costrutti genici sintetici per applicazioni in ingegneria metabolica o biologia sintetica.
Divulgazioni
Non ci sono conflitti di interesse dichiarati.
Riconoscimenti
Per CM Margaret Smith per aver gentilmente fornire il clone integrasi
Un sostegno finanziario a partire da:
Genome Canada / Genome Prairie
NSERC Discovery e borse di studio progetti strategici
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reattivo | Azienda | Numero di catalogo | Comments |
| Streptomicina | Bioshop Canada Inc. | STP101 | |
| Spectinomicina | Bioshop Canada Inc. | SPE201 | |
| Gentamicina | Bioshop Canada Inc. | GTA202 | |
| Choramphenicol | Bioshop Canada Inc. | CLR201 | |
| Tetracycline | Bioshop Canada Inc. | TET701 | |
| Kanamicina | Bioshop Canada Inc. | KAN201 | |
| Batteriologica agar grade | Bioshop Canada Inc. | AGR001 | |
| Triptone | Bioshop Canada Inc. | TRP402 | |
| Estratto di lievito | Bioshop Canada Inc. | YEX401 | |
| Cloruro di sodio | Bioshop Canada Inc. | SOD001 | |
| Cloruro di calcio | Bioshop Canada Inc. | CCL444 | |
| X-Gluc | Oro Biotechnology Inc. | G1281C1 | |
| MT616 ceppo E. coli | Disponibile su richiesta | Utilizzato anche al di fuori del nostro laboratorio | |
| PJC2 ceppo E. coli | In casa, disponibile su richiesta | ||
| PJH110 ceppo E. coli | In casa, a disposizione dal richt | ||
| SmUW227 ceppo | In casa, disponibile su richiesta |
Riferimenti
- Itaya, M., Tsuge, K., Koizumi, M., Fujita, K. Combining two genomes in one cell: Stable cloning of the Synechocystis PCC6803 genome in the Bacillus subtilis 168 genome. Proc. Natl. Acad. Sci. U.S.A. 102, 15971-15976 (2005).
- Kushtoss, S., Rao, R. N. Analysis of the Integration Function of the Streptomycete Bacteriophage FC31. J. Mol. Biol. 222, 897-908 (1991).
- Brown, W. R., Lee, N. C., Xu, Z., Smith, M. C. Serine recombinases as tools for genome engineering. Methods. 53, 372-379 (2011).
- Groth, A. C., Olivares, E. C., Thyagarajan, B., Calos, M. P. A phage integrase directs efficient site-specific integration in human cells. Proc. Natl. Acad. Sci. U.S.A. 97, 5995-6000 (2000).
- Rowley, P. A., Smith, M. C., Younger, E. A motif in the C-terminal domain of FC31 integrase controls the directionality of recombination. Nuc. Acid. Res. 36, 3879-3891 (2008).
- Campbell, R. E. A monomeric red fluorescent protein. Proc. Natl. Acad. Sci. U.S.A. 99, 7877-7882 (2002).
- Schafer, A. Small mobilizable multi-purpose cloning vectors derived from the Escherichia coli plasmids pK18 and pK19: selection of defined deletions in the chromosome of Corynebacterium glutumicum. Gene. 145, 69-73 (1994).
- Thorpe, H. M., Smith, M. C. In vitro site-specific integration of bacteriophage DNA catalyzed by a recombinase of the resolvase/invertase family. Proc. Natl. Acad. Sci. 95, 5505-5510 (1998).
- Jones, J. D., Gutterson, N. An efficient mobilizable cosmid vector, pRK7813, and its use in a rapid method for marker exchange in Pseudomonas fluorescens strain HV37a. Gene. 61, 299-306 (1987).
- Beringer, J. E. R Factor transfer in Rhizobium leguminosarum. J. Gen. Microbiol. 84, 188-198 (1974).
- Lennox, E. S. Transduction of linked genetic characters of the host by bacteriophage P1. Virology. 1, 190-206 (1955).
- Hanahan, D. Studies on transformation of Escherichia coli with plasmids. J. Mol. Biol. 166, 557-580 (1983).
- Charles, T. C., Finan, T. M. Genetic map of Rhizobium meliloti megaplasmid pRmeSU47b. J. Bacteriol. 172, 2469-2476 (1990).
- Leigh, J. A., Signer, E. R., Walker, G. C. Exopolysaccharide-deficient mutants of Rhizobium meliloti that form ineffective nodules. Proc. Natl. Acad. Sci. U.S.A. 82, 6231-6235 (1985).
- Heil, J. R., Nordeste, R. F., Charles, T. C. The fluorescence theatre: a cost-effective device using theatre gels for fluorescent protein and dye screening. Can. J. Microbiol. 57, 339-342 (2011).
- Datsenko, K. A., Wanner, B. L. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products. Proc. Natl. Acad. Sci. U.S.A. 97, 6640-6645 (2000).
- Lesic, B., Rahme, L. G. Use of the lambda Red recombinase system to rapidly generate mutants in Pseudomonas aeruginosa. BMC Mol. Biol.. 9, 20-20 (2008).
- Choi, K. -H., Schweizer, H. P. mini-Tn7 insertion in bacteria with single attTn7 sites: example Pseudomonas aeruginosa. Nat. Protocols. 1, 153-161 (2006).
- Thomason, L. C., Calendar, R., Ow, D. W. Gene insertion and replacement in Schizosaccharomyces pombe mediated by the Streptomyces bacteriophage FC31 site-specific recombination system. Molecular Genetics and Genomics. 265, 1031-1038 (2001).
- Katzen, F. Gateway recombinational cloning: a biological operating system. Expert Opin. Drug Discovery. , 571-586 (2007).
- Charles, T. C., Doty, S. L., Nester, E. W. Construction of Agrobacterium strains by electroporation of genomic DNA and its utility in analysis of chromosomal virulence mutations. Appl. Environ. Microbiol. 60, 4192-4194 (1994).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon