Method Article
Extraction optimisée d’ADN de haute qualité à partir de lésions athéroscléreuses humaines fixées au formol et incluses dans de la paraffine
Dans cet article
Résumé
Ici, nous présentons un protocole d’extraction semi-automatisée de l’ADN à partir de lésions fixées au formol et incluses dans la paraffine des artères carotides humaines. La lyse tissulaire est effectuée sans xylène toxique, qui est suivie d’un protocole d’extraction automatisé de l’ADN, comprenant une deuxième étape de lyse, la liaison de l’ADN à des particules paramagnétiques pour la liaison à base de cellulose, des étapes de lavage et l’élution de l’ADN.
Résumé
Les tissus fixés au formol et inclus dans de la paraffine (FFPE) représentent une source précieuse pour les analyses moléculaires et les études génomiques cliniques. Ces tissus sont souvent pauvres en cellules ou difficiles à traiter. Par conséquent, les acides nucléiques doivent être soigneusement isolés. Ces dernières années, diverses méthodes d’isolement de l’ADN ont été mises au point pour les tissus de nombreuses maladies, principalement le cancer. Malheureusement, l’ADN génomique extrait des tissus FFPE est fortement dégradé en raison de la réticulation entre les brins d’acide nucléique et les protéines, ainsi que des ruptures aléatoires en séquence. Par conséquent, la qualité de l’ADN de ces échantillons est nettement réduite, ce qui rend difficile la réalisation d’analyses moléculaires ultérieures. D’autres problèmes avec les tissus difficiles sont, par exemple, le manque de cellules dans les lésions athéroscléreuses humaines calcifiées et les tissus adipeux, les petites biopsies cutanées et, par conséquent, la faible disponibilité des acides nucléiques souhaités, comme c’est également le cas dans les tissus anciens ou fixés.
Dans nos laboratoires, nous avons mis au point une méthode d’extraction de l’ADN à partir de lésions athéroscléreuses fixées au formol, à l’aide d’un système d’isolement semi-automatisé. Nous avons comparé cette méthode à d’autres protocoles d’extraction disponibles dans le commerce et nous nous sommes concentrés sur d’autres analyses en aval. La pureté et la concentration de l’ADN ont été mesurées par spectrométrie et fluorométrie. Le degré de fragmentation et la qualité globale ont été évalués.
La quantité et la qualité d’ADN les plus élevées ont été obtenues avec le protocole d’ADN sanguin modifié pour le système d’extraction automatisé, au lieu du protocole commercial FFPE. Grâce à ce protocole étape par étape, les rendements en ADN des échantillons FFPE étaient en moyenne quatre fois plus élevés et moins d’échantillons ont échoué au processus d’extraction, ce qui est essentiel lorsqu’il s’agit de biopsies de petits vaisseaux. Des tailles d’amplicon de 200 à 800 pb ont pu être détectées par PCR. Cette étude montre que bien que l’ADN obtenu à partir de notre tissu FFPE soit très fragmenté, il peut toujours être utilisé pour une amplification et un séquençage réussis de produits plus courts. En conclusion, entre nos mains, la technologie automatisée semble être le meilleur système pour l’extraction d’ADN, en particulier pour les petits échantillons de tissus FFPE.
Introduction
La fixation au formol suivie d’un enrobage de paraffine (FFPE) est une procédure standard pour la conservation à long terme d’échantillons pathologiques dans les biobanques1. Ces échantillons constituent une source précieuse pour les études histologiques ainsi que pour les analyses moléculaires, en particulier les études génétiques2. D’autres avantages des mouchoirs FFPE sont un meilleur stockage à long terme, des coûts réduits et des conditions de stockage plus faciles. Notre intention ici est de fournir un protocole fiable et facile à utiliser pour l’isolement reproductible des acides nucléiques à partir de petites quantités de coupes FFPE, car l’extraction d’ADN de haute qualité est la première étape cruciale d’un large éventail de techniques moléculaires et les tissus FFPE sont la source d’échantillons la plus disponible.
Les nouvelles approches scientifiques, telles que le séquençage de nouvelle génération (NGS) et les approches de recherche « omiques », nécessitent une haute qualité d’acides nucléiques 3,4,5. L’extraction d’ADN à partir d’échantillons de tissus FFPE reste une entreprise difficile. La qualité et la quantité de l’ADN provenant d’échantillons FFPE peuvent varier considérablement en fonction de son âge et de ses conditions de fixation. Le formol, le composé le plus fréquemment utilisé, conduit à la réticulation ADN-protéine 6,7,8 et provoque une rupture aléatoire non spécifique dans la séquence nucléotidique9. Cela peut avoir un impact significatif sur les analyses génomiques en aval, car la réticulation peut désactiver l’amplification de la réaction en chaîne par polymérase (PCR) 6,10. En raison des contaminants pendant le processus de fixation, la pureté de l’ADN isolé à partir d’échantillons FFPE est souvent limitée. Au cours des dernières années, diverses méthodes d’isolement de l’ADN ont été mises en place, principalement à partir d’échantillons de tissus cancéreux 2,11,12,13.
En général, les protocoles d’extraction des acides nucléiques à partir de tissus FFPE peuvent être différenciés en trois groupes principaux. Le premier groupe de méthodes, le plus couramment utilisé, comprend les systèmes de colonnes à base de silice disponibles dans le commerce14. Le deuxième groupe implique des méthodes d’extraction manuelles en phase organique avec du phénol et du chloroforme, décrites pour la première fois par Joseph Sambrook et David W. Russell15. En tant que troisième groupe, des systèmes automatisés ont été mis en place au cours des dernières années, tels que les systèmes de manipulation de liquides ainsi que les systèmes paramagnétiques à base de particules16. Chacun des trois systèmes nommés présente des avantages et des inconvénients différents, tels que des produits chimiques dangereux (c’est-à-dire le xylène, le phénol, le chloroforme), des coûts élevés17, de la main-d’œuvre18 et la consommation de temps19. En particulier, pour les échantillons de tissus difficiles ainsi que pour les analyses à haut débit, la normalisation, la reproductibilité, la consommation de temps, la main-d’œuvre et les coûts relativement faibles sont les caractéristiques les plus pertinentes pour trouver une méthode appropriée pour l’isolement des acides nucléiques20. Les méthodes d’extraction automatisées sont connues pour donner des résultats plus reproductibles et sont plus sensibles pour les petites biopsies. De plus, moins de tissu ou de sang est nécessaire et le risque d’obstruction du système dû à des quantités élevées de paraffine est réduit. Bien que les machines pour l’extraction automatisée des acides nucléiques et les kits nécessaires soient plus chères que les méthodes manuelles, elles convainquent toujours en raison des processus d’extraction moins problématiques. La recherche documentaire fournit de nombreuses publications qui illustrent une comparaison directe entre les méthodes manuelles, basées sur des colonnes et automatisées d’extraction d’ADN et d’ARN à partir de différents tissus et organismes, tels que les plantes, les animaux et les humains, ainsi que des cellules en culture 20,21,22. Il existe également des preuves présentes dans la littérature montrant que l’ADN et l’ARN isolés de tissus congelés de 10 ans peuvent être utilisés pour des analyses en aval telles que la PCR, la PCR quantitative, le NGS, les analyses de méthylation et le clonage 9,23,24,25,26.
Le problème majeur, par exemple, des tissus vasculaires humains âgés, ainsi que des biopsies de petits tissus, en particulier des échantillons FFPE, est le manque de cellules dans les lésions athéroscléreuses hautement calcifiées, ce qui conduit par conséquent à de faibles concentrations d’acides nucléiques1. Bien que plusieurs méthodes d’extraction d’ADN à partir de tissus FFPE aient déjà été établies et soient largement utilisées, les méthodes manuelles de préparation des échantillons nécessitent un long temps de manipulation27 et des réactifs toxiques tels que le xylène ou le phénol sont nécessaires pour la déparaffinisation2. Comme nous l’avons décrit, le processus de déparaffinisation est une étape cruciale qui prend du temps (p. ex., environ 30 min) et qui affecte considérablement la qualité et la quantité de l’ADN extrait (p. ex., effets toxiques sur l’ADN, tels que la fragmentation et la dégradation de la solution de déparaffinisation et les températures élevées)28. De nouveaux protocoles d’extraction d’ADN récemment développés se concentrent sur l’utilisation d’autres solutions de déparaffinisation non toxiques, de stratégies de réparation et de technologies de billes automatisées. En particulier, les méthodes automatisées et semi-automatisées se sont avérées efficaces dans l’extraction de l’ADN avec une récupération efficace, l’absence de contamination croisée et des performances faciles29. Nous avons établi un protocole qui surmonte ces limitations. Par conséquent, notre technique permet une réduction du temps de traitement et de manipulation selon les normes quantitatives et qualitatives les plus élevées.
En particulier pour les analyses reproductibles à haut débit telles que le génotypage, les études épigénomiques et le séquençage de l’ARN, la manipulation d’échantillons FFPE avec des systèmes de purification sur colonne est souvent difficile et chronophage (par exemple, de longues étapes de déparaffinisation, le colmatage de la colonne et de longs temps de manipulation). Le colmatage des membranes de silice en raison d’une grande quantité de paraffine est le principal problème. D’autres circonstances peuvent aggraver l’isolement des acides nucléiques de haute qualité : de petites quantités de tissus, telles que des micro-biopsies de la peau, de petits tissus de souris, des tissus très gras ou calcifiés sous forme de plaques, des tissus ossifiés et des échantillons âgés. En particulier dans le domaine du diagnostic et de la médecine légale, les systèmes automatisés et semi-automatisés tels que la manipulation de liquides ou les méthodes d’extraction paramagnétiques basées sur les particules sont devenus de plus en plus essentiels au cours des dernières années30,31, principalement en raison des temps de manipulation relativement courts et de la possibilité de normalisation. La plupart des protocoles déjà publiés fonctionnent parfaitement pour les tissus lisses avec des quantités élevées ou moyennes de cellules, tels que les biopsies tumorales ou les tissus végétaux 13,22,32. La littérature sur les méthodes semi-automatisées à base de particules utilisées pour isoler l’ADN de tissus relativement difficiles à manipuler tels que les cellules uniques fixes, les vaisseaux calcifiés, les tissus riches en collagène et les tissus adipeux à faible nombre de cellules n’est que mal décrite33.
Dans cette étude, une méthode semi-automatisée optimisée pour l’isolement de l’ADN à partir de coupes intégrées à la paraffine vasculaire est décrite, en la comparant à deux protocoles manuels basés sur des colonnes. La quantité, la pureté et l’étendue de la fragmentation de l’ADN ont été utilisées pour la validation. Le protocole d’ADN sanguin disponible dans le commerce a été utilisé comme point de départ et les étapes manuelles du système semi-automatisé ont ensuite été optimisées pour l’utilisation du FFPE ainsi que des échantillons de tissus frais congelés provenant de tissus humains et animaux, en combinant les étapes du FFPE et du protocole tissulaire. L’étape automatisée de ce protocole est préinstallée sur l’instrument et dépend du kit utilisé (ici, le kit ADN sanguin). Avec le système semi-automatisé à cartouche décrit, il est possible d’isoler l’ADN du sang, des tissus fraîchement congelés, des tissus fixés au formol et même des cellules uniques avec le même protocole, la même machine, le même kit et les mêmes consommables, au lieu d’utiliser des protocoles et des kits différents pour l’instrument, comme cela est recommandé par l’entreprise. Il n’y a que des différences mineures dans les protocoles, comme un tampon et des temps d’incubation pour les différentes applications, ce qui rend ce protocole très utile pour extraire l’ADN de toutes sortes de tissus. Notre protocole est principalement optimisé pour les tissus vasculaires humains calcifiés, pauvres en cellules et fibreux, mais peut bien sûr être utilisé et optimisé pour toutes sortes de tissus difficiles mentionnés ci-dessus.
En résumé, pour les chercheurs dans le domaine cardiovasculaire travaillant sur l’athérosclérose (par exemple, l’aorte, les artères carotides, les artères coronaires), nous fournissons un protocole point par point facile à utiliser pour l’extraction semi-automatisée d’ADN à partir d’échantillons vasculaires FFPE.
Protocole
L’autorisation de collecter des échantillons d’athérosclérose carotidienne humaine dans notre biobanque a été approuvée par le comité d’éthique de l’hôpital local (2799/10, Ethikkommission der Fakultät für Medizin der Technischen Universität München, Munich, Allemagne). Un consentement éclairé écrit a été obtenu de tous les patients. Les expériences ont été réalisées conformément aux principes de la Déclaration d’Helsinki.
1. Préparation des tissus
- Préparez 5 à 8 coupes de tissu de 10 μm à partir de l’échantillon FFPE avec le microtome et transférez-le dans un tube de 1,5 mL. Il n’est pas nécessaire de réduire l’excès de paraffine du bloc.
REMARQUE : Des sections simples plus fines au lieu d’une grande section accélèrent la réaction du tampon. Jetez les premières sections en raison de l’exposition à O2 . Pour des échantillons plus grands, il est également possible d’utiliser moins de sections. - Centrifugez ces tubes dans une centrifugeuse de paillasse, réglée à 5 000 x g pendant 1 minute à température ambiante pour prélever chaque échantillon au fond du tube.
ATTENTION : Une centrifugation trop longue entraîne une coagulation de l’échantillon et complique la lyse.
2. Dissolution et déparaffinisation des lipides
REMARQUE :Cette étape est nécessaire pour la déparaffinisation et la digestion des lipides. Le tampon utilisé est moins toxique que les solutions de déparaffinisation commerciales.
- Ajouter 300 μL du tampon d’incubation disponible dans le commerce et 6 μL de 1-thioglycérol dans chaque tube.
REMARQUE : N’utilisez pas plus de 300 μL car il s’agit du volume maximum de la cartouche du système d’automatisation. - Vortex pendant 10 s et incuber l’échantillon pendant 10 min à 80 °C et 500 tr/min dans un bloc chauffant pour solubiliser la paraffine.
ATTENTION : Le tissu doit être complètement dissous à la fin. Si nécessaire, faites plusieurs fois tourbillonner pendant l’incubation.
3. Digestion des échantillons et des protéines
REMARQUE : La digestion native avec la protéase K est cruciale pour avoir des extraits d’ADN propres sans protéines. Il réduit également les protéines contaminantes présentes. De plus, les nucléases sont également détruites pour sauver l’ADN34. Cette étape de nuit est également nécessaire pour une digestion complète de l’échantillon.
- Laissez refroidir l’échantillon à 60 °C, puis ajoutez 30 μL de la solution de protéinase K fournie.
- Agitez à nouveau et incubez le mélange à 65 °C et 500 tr/min pendant la nuit (4 à 20 h) dans un bloc chauffant. Vortex les échantillons pendant l’incubation de temps en temps pour une digestion complète des échantillons.
REMARQUE : L’incubation d’une nuit donne de meilleurs résultats. Il est recommandé d’effectuer des étapes de mélange toutes les 30 à 60 minutes. En fin de compte, il ne devrait pas y avoir de morceau de tissu visuel à l’intérieur du tube.
4. Lyse cellulaire
- Ajouter 400 μL du tampon de lyse, fourni dans la trousse de sang et vortex sous peu.
- Incuber à nouveau l’échantillon à 65 °C pendant 30 min à 500 tr/min.
- Laissez l’échantillon refroidir à température ambiante. La paraffine durcira sur le dessus.
ATTENTION : Ne vortex pas à nouveau, pour garder la paraffine séparée de l’échantillon. Sinon, la paraffine est mélangée au tissu, ce qui détruit l’échantillon. L’échantillon sera prélevé à l’étape 6.1.
5. Préparation des cartouches pré-distribuées
- Mettez l’appareil sous tension, ainsi que la tablette associée.
- Démarrez l’application logicielle et cliquez sur le bouton Porte pour ouvrir l’instrument.
- Retirez le rack de l’instrument et insérez la cartouche préremplie dans le rack de la sonde. Assurez-vous que la cartouche s’enclenche deux fois lorsqu’elle est en place et retirez la feuille d’étanchéité.
- Ajoutez le piston dans le dernier (8) puits de la cartouche. Il sert de pointe de pipette dans l’instrument.
- Remplissez les tubes d’élution de 0,5 ml fournis avec 65 μL de tampon d’élution fourni avec le kit. Laissez les tubes ouverts et insérez-les dans la position dédiée dans la partie avant du rack, après la cartouche.
REMARQUE : Le volume minimum pour l’élution est de 60 μL. Le système perdra 5 à 10 μL du volume d’élution ajouté.
6. Extraction automatisée de l’ADN
- Percez soigneusement la paraffine sur le dessus du tube de 1,5 mL à partir de l’étape 4.3 pour atteindre l’échantillon propre au fond du tube sans le mélanger à nouveau avec de la paraffine.
- Transvasez l’ensemble du mélange (730 μL) de l’échantillon préparé dans le premier puits de la cartouche.
- Insérez le rack dans la machine d’extraction d’ADN automatisée. Assurez-vous que le rack se verrouille d’abord à l’arrière de la machine, puis à l’avant.
- Démarrez la course en cliquant sur le bouton orange Démarrer en haut à gauche dans le logiciel. Une fenêtre avec différents protocoles préinstallés s’ouvrira. Sélectionnez Protocole d’ADN sanguin sur l’instrument. Vérifiez que le piston, le tube d’élution et l’échantillon ont été ajoutés en cliquant sur oui dans le logiciel. La porte de l’instrument se fermera automatiquement et le cycle commencera (le voyant deviendra vert). La course durera environ 38 min. Aucun étalonnage supplémentaire n’est nécessaire.
REMARQUE : Surveillez jusqu’à ce que le système ait ramassé les pistons pour tous les échantillons dans le rack. Si ce n’est pas le cas, le système s’arrête automatiquement et le protocole de la machine doit être redémarré. - Assurez-vous que le système effectue l’étape de lyse automatisée dans le premier puits de la cartouche, suivie des étapes de lavage dans les puits 3 à 7. Il n’y a pas d’autre étape de programmation nécessaire. Le programme complet est préinstallé par l’entreprise.
- Une fois cela fait, assurez-vous que le système élue l’ADN dans les tubes d’élution préparés via le piston ajouté. Les particules magnétiques restent dans le piston. Le piston à la fin retourne au dernier puits de la cartouche.
7. Terminez la course
- Une fois le fonctionnement terminé (la machine affiche un voyant vert clignotant), ouvrez l’instrument en cliquant sur le bouton d’ouverture (panneau de porte) et retirez le rack du système.
- Jetez les cartouches.
- Réinsérez le rack vide dans l’instrument et fermez la porte à l’aide du bouton de porte dans le coin supérieur droit. Fermez l’application logicielle et éteignez la machine, ainsi que la tablette.
- Stockez les éluats à -20 °C pour un stockage à long terme ou à 4 °C pour un stockage à court terme, ou utilisez-les directement pour l’analyse en aval ou les mesures de concentration.
Résultats
Pour l’établissement du protocole, 5 blocs de tissus FFPE de patients atteints d’athérosclérose de l’artère carotide ont été utilisés. L’ADN a été isolé à l’aide d’un protocole semi-automatisé optimisé (kit C) ainsi que de deux protocoles manuels basés sur des colonnes disponibles dans le commerce (kit A et kit B, voir le tableau des matériaux). L’extraction de l’ADN avec les kits A et B a été réalisée selon le protocole du fabricant. Le seul changement qui a été apporté au protocole des deux kits disponibles dans le commerce (kit A et kit B) : la déparaffinisation au début a été effectuée deux fois en raison de grandes quantités de paraffine entourant les sections. Après déparaffinisation avec du xylène, l’ADN a été lié à la membrane-colonne et lavé trois fois. Après séchage de la membrane par centrifugation, l’ADN a été élué avec 60 μL du tampon d’élution fourni.
Pour déterminer la quantité optimale d’échantillons nécessaires pour obtenir des résultats reproductibles chez chaque patient, différentes quantités de matériel ont été prélevées. Des coupes de tissus consécutives de 2 x 10 μm (20 μm au total), 5 x 10 μm (50 μm au total) et 8 x 10 μm (80 μm au total) ont été utilisées (figure 1). La qualité et la quantité de l’ADN génomique total isolé à l’aide du système automatisé et des systèmes à deux claviers basés sur des colonnes ont été évaluées à la fois par spectrométrie, par un système spectrométrique de lecteur de microplaques et par fluorométrie. L’analyse de fragmentation a été effectuée à l’aide de la technologie automatisée de bande d’écran et de la réaction en chaîne par polymérase (PCR). L’analyse statistique a été effectuée à l’aide d’un logiciel disponible dans le commerce (voir la Table des matières).
Le protocole semi-automatisé conduit à une plus grande quantité d’ADN extrait
À l’aide de la spectrométrie de microvolume et de la spectrométrie de lecteur de plaques ainsi que de la fluorométrie, comme le montre la figure 2, la quantité totale d’ADN génomique a été comparée pour différentes quantités de coupes de tissus (20 μm, 50 μm, 80 μm au total). Une augmentation linéaire de la concentration d’ADN a été obtenue avec le kit semi-automatisé C à cartouche, avec les trois méthodes de quantification. Pour les kits manuels A et B, aucune différence significative n’a été observée entre les sections de 20 μm, 50 μm et 80 μm. La concentration moyenne des cinq échantillons de tissus mesurée par spectrométrie de microvolume et par technologie de lecture de microplaques pour l’ADN extrait avec le protocole semi-automatisé (figure 2A, B, tableau 1) a montré une concentration significative, quatre fois plus élevée (45 ng/μL) par rapport aux protocoles A et B basés sur des colonnes (p = 0,016). Le kit A a atteint des concentrations moyennes de 12,5 ng/μL, tandis que le kit B n’a atteint que 8,5 ng/μL. La figure 3 montre la qualité de l’ADN extrait avec les trois méthodes mentionnées. En résumé, il n’y a pas de grande différence de qualité. Le kit semi-automatisé présente une qualité plus stable, les quantités mesurées sont comparables. Des résultats similaires ont été obtenus en utilisant des mesures fluorimétriques de la concentration d’ADN (figure 2C). Les concentrations moyennes d’ADN à l’aide du kit A ont été réduites à 3 ng/μL par rapport à l’analyse spectrométrique. En résumant la quantité d’ADN, le protocole automatisé s’est avéré être la méthode la plus efficace pour les échantillons de cellules et de tissus faibles.
Le protocole d’extraction automatisé permet d’obtenir une meilleure qualité de l’ADN extrait
De plus, la qualité de l’ADN isolé à l’aide des trois protocoles différents a été évaluée par spectrophotométrie, à l’aide d’un spectrophotomètre et d’un lecteur de microplaques, en calculant le rapport A260/280 (Figure 3A, B et Tableau 2). Les acides nucléiques ont leur absorbance maximale à 260 nm. La pureté de l’ADN de haute qualité se situe généralement dans la gamme des valeurs A260/280 de 1,6 à 1,8, comme en témoignent les deux lignes pointillées de la figure3 A, B35. Le rapport moyen du kit manuel sur colonne A était de 1,92 ± 0,6 en spectrométrie et de 2,58 ± 1,2 mesuré avec la technologie des lecteurs de microplaques. Le kit B présentait des ratios plus petits (1,45 ± 0,3 et 1,85 ± 0,7). L’ADN isolé avec le kit semi-automatisé C a montré des rapports de pureté de 1,4 ± 0,07 en spectrométrie et de 1,68 ± 0,05 calculés avec le lecteur de microplaques. Dans les deux mesures, la méthode d’isolement automatisée a montré moins de variation dans le rapport de pureté que les kits manuels, mais seul le kit A a montré des différences significatives dans l’analyse du test t apparié en raison de l’énorme diffusion (p = 0,0025 et p < 0,001). En ce qui concerne la mesure des microplaques, tous les échantillons isolés avec le système automatisé ont satisfait aux critères de pureté (rapports entre 1,6 et 1,8), tandis que le kit A et le kit B ont échoué dans de nombreux cas à ces critères (p < 0,001). La spectrométrie (figure 3A) a mis en évidence que tous les échantillons isolés avec le système de cartouche optimisé (rapport minimum = 1,36) n’atteignaient pas le seuil de pureté défini (lignes pointillées, indiquant le rapport A260/280 de 1,6 et 1,8), indiquant une contamination par le tampon. De plus, une énorme variation du rapport calculé pour les kits A et B a été observée. Par conséquent, l’isolement semi-automatisé de l’ADN fournit des échantillons de qualité comparable et reproductible, alors que la qualité de l’ADN fluctue considérablement avec les méthodes d’isolement basées sur des colonnes. Un autre contrôle de la qualité a été effectué à l’aide du calcul DV200 (figure 3A, tableau 2). Ici, le système d’extraction manuel A basé sur une colonne a montré une valeur de 90 % ± 3,8 pour les fragments supérieurs à 200 pb, alors que le kit B n’a montré que 85 % ± 7,1 (p = 0,01). De plus, dans cette analyse, l’ADN extrait avec le protocole semi-automatisé a montré le pourcentage le plus élevé de fragments supérieurs à 200 pb (92 % ± 3,6).
En résumé, la qualité de l’ADN extrait avec le système semi-automatisé était plus stable et reproductible, tandis que l’ADN isolé avec les systèmes basés sur des colonnes présentait plus de variations dans les rapports de qualité. Le DV200 indiquait une qualité similaire pour les kits A et C, tandis que le kit B avait moins de fragments de plus de 200 pb.
La fragmentation de l’ADN est indépendante du protocole utilisé
La fragmentation de l’ADN a été évaluée à l’aide d’une technologie de bande d’écran semi-automatisée. La figure 4A montre que des gels représentatifs montrent l’étendue de la fragmentation de tous les échantillons utilisés dans cette étude. Les couleurs sombres indiquent une densité plus élevée, tandis que les images grisées indiquent des densités plus faibles. Ici, des tendances similaires ont été observées pour tous les échantillons et dans les trois kits. Les trois premiers échantillons montrent la région la plus sombre (ce qui équivaut à la plus grande accumulation d’ADN fragmenté), dans la partie inférieure du gel représentant les plus petits fragments d’ADN, les échantillons des patients 4 et 5 montrent plus de fragments (plus denses) dans la partie supérieure du gel, indiquant moins d’ADN fragmenté. La figure 4B montre la différence de fragmentation de l’ADN à l’aide de différentes quantités de matériau (20 μm, 50 μm et 80 μm) pour un échantillon représentatif en utilisant les trois stratégies d’isolement. Les électrophérogrammes montrent la distribution des fragments correspondante. Les données sont résumées dans le tableau 3. Ici, la seule différence visuelle entre les trois kits a été trouvée dans les graphiques des sections de 20 μm, le kit automatique montrant un électrophérogramme plus clair avec plus d’ADN détecté. Pour les coupes de 50 μm et 80 μm, aucune différence visible dans l’électrophérogramme n’a été observée entre les différents kits utilisés dans l’étude.
La figure 5A montre la distribution moyenne de la taille des fragments pour le kit de système de cartouche semi-automatisé C ainsi que pour les deux kits manuels A et B. La distribution moyenne de la taille des fragments pour le système automatisé montre les résultats suivants : 5 % des fragments d’ADN étaient inférieurs à 200 pb, 62 % étaient de taille comprise entre 200 pb et 1500 pb et 29 % des fragments d’ADN étaient plus longs que 1 500 pb. Un pourcentage plus élevé de fragmentation accrue de l’ADN a été mesuré avec le kit B (12 %) et le kit A (8 %). Les fragments de taille moyenne étaient similaires pour le kit A (62 %) et le kit B (57 %). En ce qui concerne les fragments d’ADN plus longs, aucune différence significative (kit A 26 %, kit B 27 %) n’a été observée. Cela conduit à la conclusion que l’ADN isolé avec le kit semi-automatisé optimisé (kit C) semble être moins fragmenté que l’ADN isolé avec des kits basés sur des colonnes.
Le pic de fragmentation (quantité d’ADN la plus élevée pour une certaine taille de fragment) dans les trois kits a été analysé pour chacun des cinq échantillons de tissus (figure 5B). Aucune différence entre les trois méthodes d’isolement n’a pu être détectée concernant la fragmentation globale de l’ADN. Il convient de noter que, quel que soit le kit utilisé, les échantillons 1 à 3 présentent un pic maximal moyen compris entre 280 pb et 620 pb, tandis que les échantillons 4 et 5 sont moins fragmentés avec un pic de fragmentation moyen compris entre 1 200 pb et 1 800 pb. Ces résultats indiquent que la fragmentation de l’ADN est affectée par la qualité des échantillons eux-mêmes.
L’ADN extrait avec le kit semi-automatisé est adapté à l’analyse PCR
En fin de compte, huit échantillons d’ADN isolés avec le protocole optimisé ont été utilisés pour l’analyse en aval effectuant une PCR de différentes longueurs de fragments. Des amorces appropriées pour le gène de l’entretien de la bêta-actine ont été choisies. Une lignée de cellules musculaires lisses primaires a servi de contrôle (Figure 6). En affichant les fragments détectables dans l’électrophorèse sur gel d’agarose, des échantillons avec des fragments maximaux de grande taille (500 pb à 800 pb pour les échantillons 1, 2 et 8) ainsi que des échantillons avec seulement 200 pb de fragments maximaux ont été détectés (échantillons 6 et 7), indépendamment de la méthode d’extraction utilisée.
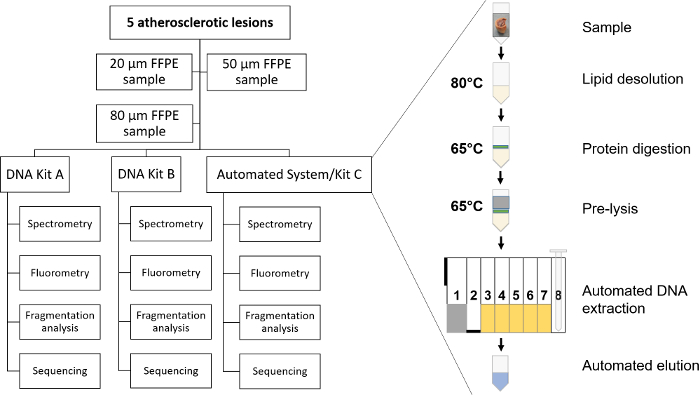
Figure 1 : Flux de travail expérimental. Les principales étapes de la préparation des échantillons et de l’extraction semi-automatisée de l’ADN sont présentées dans le contexte des échantillons utilisés et en comparaison avec les kits d’extraction manuelle de l’ADN A et B. Veuillez cliquer ici pour voir une version agrandie de cette figure.
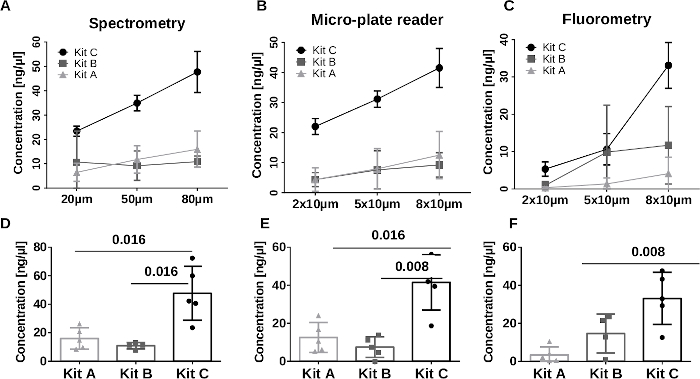
Figure 2 : Mesure de la concentration. La partie supérieure (A-C) montre les concentrations d’ADN mesurées par spectrométrie, technologie de lecture de microplaques et fluorométrie à l’aide de sections de 20 μm, 50 μm et 80 μm (chacune, n = 5) pour les trois kits différents utilisés. La partie inférieure montre la distribution des concentrations d’ADN de cinq échantillons (80 μm) mesurés par spectrométrie (D), technologie de lecteur de microplaques (E) et fluorométrie (F). Les valeurs p sont calculées à l’aide du test t apparié, p < 0,05 a été considéré comme significatif. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 3 : Pureté de l’ADN isolé. La pureté de l’ADN a été évaluée en calculant le rapport A260/280 dans deux spectromètres différents (A,B) pour les trois kits différents (n = 15). Les lignes pointillées indiquent le rapport optimal (1,6-1,8). (C) montre le DV200 (fragments >200 pb) en % calculé par analyse de fragmentation. Les valeurs p sont calculées à l’aide du test de Mann-Whitney et le graphique montre que p < 0,05 a été considéré comme significatif. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 4 : Images représentatives de l’analyse de fragmentation. La fragmentation de chaque échantillon individuel 1 à 15 est illustrée dans les traces de bande d’écran. Pour chaque échantillon (n = 5), différentes quantités d’ADN provenant de coupes de 20 μm, 50 μm et 80 μm ont été analysées séparément (n = 3 pour chaque échantillon) (A). (B) montre la fragmentation de l’ADN d’un échantillon représentatif pour des coupes de 20 μm, 50 μm et 80 μm pour les trois kits. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 5 : Distribution de fragments par analyse de bande d’écran. (A) montre la distribution des fragments courts, moyens et longs dans les échantillons isolés avec différents kits. (B) montre le pic de fragmentation maximal de l’ADN isolé à partir des échantillons 1 à 5 tracés pour les kits individuels. Pour chaque kit, les sections de 20 μm, 50 μm et 80 μm de chaque échantillon sont regroupées. Les valeurs p sont calculées à l’aide d’un test t multiple apparié et le graphique montre que P < 0,05 a été considéré comme significatif. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 6 : Analyse de fragmentation par PCR. Cette électrophorèse sur gel montre les produits PCR avec différentes longueurs d’amplicon du gène de maintien de la bêta-actine pour huit échantillons différents et une lignée cellulaire de contrôle. Les voies sont nommées comme suit : 1 = 100 pb, 2 = 200 pb, 3 = 300 pb, 4 = 400 pb, 5 = 500 pb, 6 = 600 pb, 7 = 700 pb, 8 = 800 pb, 9 = 1 000 pb et 10 = 1 300 pb. Veuillez cliquer ici pour voir une version agrandie de ce chiffre.
| Kit A | Kit B | Kit C | ||
| Spectromètre | Concentration maximale [ng/μl] | 26.2 | 13.25 | 72.35 |
| Concentration minimale [ng/μl] | 8.45 | 7.75 | 23.55 | |
| Concentration moyenne [ng/μl] | 15.96 | 10.89 | 47.72 | |
| Lecteur de microplaques | Concentration maximale [ng/μl] | 24 | 13.9 | 56.2 |
| Concentration minimale [ng/μl] | 5.2 | 0 | 18.6 | |
| Concentration moyenne [ng/μl] | 12.5 | 7.4 | 41.5 | |
| Fluorimètre | Concentration maximale [ng/μl] | 10.3 | 23.4 | 47.6 |
| Concentration minimale [ng/μl] | 0.211 | 0.842 | 12.5 | |
| Concentration moyenne [ng/μl] | 3.369 | 14.64 | 33.1 | |
Tableau 1 : Résultats de l’analyse quantitative. Ce tableau montre la concentration minimale, maximale et moyenne de l’ADN isolé chez les cinq patients avec les trois kits différents. Les concentrations ont été mesurées par spectrométrie à l’aide d’un microspectromètre et d’un lecteur de microplaques, ainsi que par fluormétrie.
| Échantillon | μm | Trousse | Microplaque 260/280 | Spectromètre 260/280 | DV200 [ %] |
| Patient 1 | 20 | Kit A | 6 | 1.65 | 90.72 |
| Patient 1 | 50 | Kit A | 1.8 | 1.71 | 90 |
| Patient 1 | 80 | Kit A | 2.09 | 1.345 | 88.98 |
| Patient 2 | 20 | Kit A | 1.88 | 1.53 | 87.14 |
| Patient 2 | 50 | Kit A | 2.29 | 1.465 | 92.46 |
| Patient 2 | 80 | Kit A | 4.36 | 2.68 | 84.78 |
| Patient 3 | 20 | Kit A | 1.91 | 1.85 | 89.24 |
| Patient 3 | 50 | Kit A | 2.21 | 1.92 | 90.86 |
| Patient 3 | 80 | Kit A | 2.06 | 1.675 | 94.59 |
| Patient 4 | 20 | Kit A | 2.27 | 1.86 | 95.72 |
| Patient 4 | 50 | Kit A | non détectable | 3.385 | 87.13 |
| Patient 4 | 80 | Kit A | 2.33 | 1.905 | 95.64 |
| Patient 5 | 20 | Kit A | 1.6 | 1.51 | 82.87 |
| Patient 5 | 50 | Kit A | 1.79 | 1.525 | 91.76 |
| Patient 5 | 80 | Kit A | 3.47 | 2.765 | non détectable |
| Patient 1 | 20 | Kit B | 1.88 | non détectable | 80.23 |
| Patient 1 | 50 | Kit B | 0.73 | 1.43 | 75.72 |
| Patient 1 | 80 | Kit B | 1.44 | 1.585 | 75.64 |
| Patient 2 | 20 | Kit B | 2.63 | 1.45 | 79.75 |
| Patient 2 | 50 | Kit B | 1.92 | 1.765 | 87.14 |
| Patient 2 | 80 | Kit B | 1.56 | 1.52 | 91.01 |
| Patient 3 | 20 | Kit B | non détectable | 1.335 | 78.00 |
| Patient 3 | 50 | Kit B | 2.28 | 1.63 | 83.21 |
| Patient 3 | 80 | Kit B | 2 | 1.67 | 78.61 |
| Patient 4 | 20 | Kit B | 0.59 | 0.625 | 94.37 |
| Patient 4 | 50 | Kit B | 2.29 | 1.78 | 92.97 |
| Patient 4 | 80 | Kit B | non détectable | non détectable | non détectable |
| Patient 5 | 20 | Kit B | 2.63 | 1.41 | 83.15 |
| Patient 5 | 50 | Kit B | non détectable | 1.005 | 92.06 |
| Patient 5 | 80 | Kit B | 2.21 | 1.69 | 94.51 |
| Patient 1 | 20 | Kit C | 1.69 | 1.495 | 90.73 |
| Patient 1 | 50 | Kit C | 1.72 | 1.555 | 94.43 |
| Patient 1 | 80 | Kit C | 1.72 | 1.565 | 88.71 |
| Patient 2 | 20 | Kit C | 1.64 | 1.4 | 92.75 |
| Patient 2 | 50 | Kit C | 1.63 | 1.455 | 93.26 |
| Patient 2 | 80 | Kit C | 1.72 | 1.51 | 94.34 |
| Patient 3 | 20 | Kit C | 1.63 | 1.4 | 91.78 |
| Patient 3 | 50 | Kit C | 1.7 | 1.505 | 93.61 |
| Patient 3 | 80 | Kit C | 1.74 | 1.595 | 94.61 |
| Patient 4 | 20 | Kit C | 1.62 | 1.36 | 92.89 |
| Patient 4 | 50 | Kit C | 1.71 | 1.495 | 94.2 |
| Patient 4 | 80 | Kit C | 1.76 | 1.575 | 95.17 |
| Patient 5 | 20 | Kit C | 1.57 | 1.36 | 80.55 |
| Patient 5 | 50 | Kit C | 1.66 | 1.455 | 92.65 |
| Patient 5 | 80 | Ki t C | 1.69 | 1.45 | 90.29 |
Tableau 2 : Résultats de l’analyse qualitative. Les rapports A260/280 calculés avec un micro-spectromètre ainsi qu’un lecteur de microplaques sont indiqués pour chaque échantillon (n = 15) et les trois méthodes d’extraction différentes. Le DV200 (fragments de plus de 200 pb) calculé par analyse de fragmentation est également indiqué pour les kits A, B et C.
| Échantillon | μm | Trousse | Fragments % <200bp | Fragments % 200-1500 bp | Fragments % >1500bp | Fragments max peak (bp) |
| Patient 1 | 20 | Kit A | 9.04 | 77.08 | 13.64 | 484 |
| Patient 1 | 50 | Kit A | 10.14 | 77.03 | 12.97 | 378 |
| Patient 1 | 80 | Kit A | 10.91 | 76.56 | 12.42 | 393 |
| Patient 2 | 20 | Kit A | 11.01 | 67.04 | 20.1 | 589 |
| Patient 2 | 50 | Kit A | 6.09 | 68.67 | 23.79 | 628 |
| Patient 2 | 80 | Kit A | 11.54 | 66.07 | 18.71 | 560 |
| Patient 3 | 20 | Kit A | 10.22 | 77.29 | 11.95 | 412 |
| Patient 3 | 50 | Kit A | 8.52 | 76.93 | 13.93 | 465 |
| Patient 3 | 80 | Kit A | 4.79 | 77.5 | 17.09 | 580 |
| Patient 4 | 20 | Kit A | 2.7 | 55.77 | 39.95 | 1331 |
| Patient 4 | 50 | Kit A | 7.6 | 54.3 | 32.83 | 1183 |
| Patient 4 | 80 | Kit A | 2.5 | 61.06 | 34.58 | 1185 |
| Patient 5 | 20 | Kit A | 12.32 | 36.52 | 46.35 | 1895 |
| Patient 5 | 50 | Kit A | 5.34 | 36.37 | 55.39 | 1856 |
| Patient 5 | 80 | Kit A | non détectable | non détectable | non détectable | non détectable |
| Patient 1 | 20 | Kit B | 18.51 | 69.28 | 10.95 | 283 |
| Patient 1 | 50 | Kit B | 23.58 | 65.24 | 10.48 | 254 |
| Patient 1 | 80 | Kit B | 22.42 | 67.14 | 8.5 | 257 |
| Patient 2 | 20 | Kit B | 16.21 | 59.88 | 19.87 | 523 |
| Patient 2 | 50 | Kit B | 9.39 | 63.7 | 23.44 | 678 |
| Patient 2 | 80 | Kit B | 7.45 | 67.62 | 23.39 | 666 |
| Patient 3 | 20 | Kit B | 18.53 | 65.15 | 12.85 | 370 |
| Patient 3 | 50 | Kit B | 15.96 | 72.12 | 11.09 | 316 |
| Patient 3 | 80 | Kit B | 20.7 | 71.49 | 7.12 | 266 |
| Patient 4 | 20 | Kit B | 3.09 | 57.44 | 36.93 | 1296 |
| Patient 4 | 50 | Kit B | 3.69 | 52.2 | 40.77 | 1373 |
| Patient 4 | 80 | Kit B | non détectable | non détectable | non détectable | non détectable |
| Patient 5 | 20 | Kit B | 9.86 | 34.96 | 48.19 | 1831 |
| Patient 5 | 50 | Kit B | 3.76 | 31.42 | 60.64 | 2002 |
| Patient 5 | 80 | Kit B | 1.93 | 33.24 | 61.27 | 2015 |
| Patient 1 | 20 | Kit C | 8.88 | 81.47 | 9.26 | 358 |
| Patient 1 | 50 | Kit C | 5.92 | 83.95 | 10.48 | 374 |
| Patient 1 | 80 | Kit C | 11.02 | 80.18 | 8.53 | 334 |
| Patient 2 | 20 | Kit C | 5.18 | 74.75 | 18 | 492 |
| Patient 2 | 50 | Kit C | 4.96 | 75.67 | 17.59 | 458 |
| Patient 2 | 80 | Kit C | 3.14 | 72.46 | 21.88 | 441 |
| Patient 3 | 20 | Kit C | 7.15 | 79.05 | 12.73 | 393 |
| Patient 3 | 50 | Kit C | 5.85 | 78.02 | 15.59 | 365 |
| Patient 3 | 80 | Kit C | 2.59 | 68.54 | 26.07 | 458 |
| Patient 4 | 20 | Kit C | 3.94 | 54.49 | 38.4 | 1433 |
| Patient 4 | 50 | Kit C | 1.42 | 45.24 | 48.96 | 1634 |
| Patient 4 | 80 | Kit C | 0.76 | 37.03 | 58.14 | 1849 |
| Patient 5 | 20 | Kit C | 13.02 | 40.78 | 39.77 | non détectable |
| Patient 5 | 50 | Kit C | 2.72 | 39.08 | 53.57 | 1813 |
| Patient 5 | 80 | Ki t C | 5.25 | 31.87 | 58.42 | 2024 |
Tableau 3 : Résultats de l’analyse de fragmentation. Ici, le pourcentage de fragments inférieurs à 200 pb, supérieurs à 1 500 pb et compris entre 200 pb et 1 500 pb sont indiqués pour les cinq patients, les différentes quantités de matériau de départ (20 μm, 50 μm et 80 μm) et les trois méthodes d’isolement différentes. Les pourcentages inférieurs à 2 % ont été désignés comme non détectables.
Discussion
Les méthodes d’extraction de l’ADN pour les tissus FFPE varient en qualité et en quantité d’ADN isolé, ce qui affecte inévitablement la performance des analyses ultérieures en aval. Ainsi, l’automatisation devient impérative pour améliorer le flux de travail et la standardisation, ainsi que la gestion de la qualité. Par conséquent, dans la présente étude, une méthode semi-automatisée d’extraction d’ADN à partir d’échantillons FFPE a été évaluée, démontrant de meilleurs résultats que les autres protocoles manuels testés basés sur des colonnes.
Afin d’optimiser la méthode semi-automatisée décrite, notre objectif était d’avoir un protocole général pour chaque type de liquide tissulaire et corporel, ainsi que pour les échantillons fixes. Par conséquent, nous avons utilisé le kit ADN Blood disponible dans le commerce de la société (Table of Materials) pour l’optimisation. Tout d’abord, nous avons inclus une étape d’incubation pour la déparaffinisation, en utilisant le tampon d’incubation disponible dans le commerce et le 1-thioglycérol du même fabricant. Dans différents tests concernant le temps d’incubation et la température de fusion optimale de la paraffine, nous avons choisi une étape d’incubation de 10 minutes à 80 °C comme prévu dans d’autres kits FFPE disponibles dans le commerce. En ce qui concerne la quantité de tranches de FFPE, la plupart des entreprises recommandent des tranches de 2 μm x 10 μm pour l’extraction de l’ADN. Comme on le sait dans cette ampleur, il est extrêmement difficile d’isoler l’ADN d’un échantillon contenant une petite quantité de cellules. Par conséquent, nous avons essayé différentes quantités de coupes de tissu fixe et avons constaté que l’utilisation de 50 μm de tissu au total (de préférence dans des sections uniques de 5 μm à 10 μm) donnait les meilleurs résultats. Cela convient en tout cas aux tissus avec de faibles quantités de cellules, mais les tissus avec des quantités plus élevées de cellules peuvent également être traités de cette manière. Les systèmes basés sur des colonnes, en particulier, présentent ici une forte limitation en cas de colmatage de la membrane de silice. Il a été suggéré que la digestion de la protéinase K dure entre 4 et 20 heures. Ici, nous avons découvert qu’un temps d’incubation de 4 à 6 h est en effet acceptable, mais qu’une incubation d’une nuit conduit à une qualité et une quantité d’ADN plus suffisantes. Pour une meilleure lyse cellulaire, il est également apparu utile d’ajouter 10 μL supplémentaires de protéinase K le lendemain et d’incuber à nouveau pendant 1 h. Comme vitesse d’agitation/mélange, nous avons choisi plus que les 300 tr/min suggérés, car nous avons vu qu’un mélange plus fort (500 tr/min à 600 tr/min) conduit à une meilleure digestion de la protéinase K, et donc à des quantités plus élevées d’ADN. De plus, des étapes de mélange supplémentaires (telles que le vortex ou l’agitation à pleine vitesse) pendant l’incubation permettent une meilleure extraction de l’ADN. Contrairement au protocole FFPE original, nous avons choisi un temps d’incubation supplémentaire après l’ajout du tampon de lyse à l’échantillon, la lyse étant plus suffisante à 65°C. La dernière étape d’optimisation du protocole a été l’ajout d’un autre tampon d’incubation. Le système nécessite au moins 60 μL de tampon d’élution. Le système automatisé étant connu pour perdre 5 μL à 10 μL, nous choisissons d’avoir un volume d’élution de 65 μL.
Ce protocole comporte quelques étapes cruciales à noter. La première consiste à régler la bonne température pour solubiliser la paraffine. 80 °C semble être la meilleure température pour une fonte optimale. Il est également crucial de refroidir l’échantillon à 60 °C avant d’ajouter la protéinase K pour éviter la dégradation de cette enzyme. Ensuite, il est important de ne pas vortex à nouveau l’échantillon après le temps d’incubation de 30 minutes avec le tampon de lyse, car la paraffine doit durcir sur le dessus. Sinon, l’échantillon sera contaminé par de la paraffine. Comme dernière étape, il est également important d’ajouter un volume supplémentaire de tampon d’élution, car le système perd 5 μL à 10 μL. Nous pensons que 65 μL de tampon d’élution sont une bonne quantité. Si des quantités plus élevées d’ADN sont attendues, le volume peut bien sûr être augmenté.
La méthode d’isolement semi-automatisée optimisée a fourni les concentrations les plus élevées d’ADN génomique total dans tous les échantillons FFPE. En utilisant la même quantité de volume d’élution, le rendement total d’ADN était également quatre fois plus élevé. De plus, les kits basés sur des colonnes n’ont montré aucune augmentation du rendement en ADN en utilisant des sections de tissus plus épaisses, tandis que le système automatisé a démontré une augmentation linéaire de la concentration d’ADN associée à l’augmentation de la quantité de tissu utilisée. Ces différences peuvent s’expliquer principalement par l’encrassement de la paraffine dans les colonnes et la diminution conséquente de la capacité de liaison à l’ADN. Nos données suggèrent également qu’une plus grande quantité de tissu FFPE, dans notre cas des coupes de 80 μm, n’est pas nécessaire lors de l’isolement de l’ADN avec le système semi-automatisé. Nous avons pu montrer qu’une quantité déjà plus petite (sections de 20 μm) conduit à un rendement suffisant d’ADN. Ainsi, de précieux matériaux tissulaires peuvent être économisés. De plus, l’ADN extrait avec le protocole semi-automatisé remplissait, dans la plupart des cas, le critère de pureté requis pour un ADN de haute qualité (rapport A260/280 entre 1,6 et 1,8). En revanche, l’ADN obtenu à partir de systèmes d’isolement basés sur des colonnes a montré une grande variation dans la pureté de l’ADN et moins d’échantillons correspondent au rapport optimal mentionné.
Sarnecka et al. ont récemment décrit qu’un système basé sur des particules magnétiques est plus suffisant que des systèmes basés sur des colonnes12. Khokhar et ses collègues ont pu démontrer que le système automatisé basé sur d’autres protocoles est plus suffisant pour les amplicons plus petits et largement robuste en termes de quantité et de pureté des extraits par rapport à d’autres méthodes36. Nos échantillons ont confirmé ces résultats montrant une distribution élevée dans le rapport A260/280 de l’ADN isolé par les autres kits.
En ce qui concerne l’analyse de fragmentation, huit extraits d’ADN obtenus à partir de ces trois méthodes commerciales ont été utilisés pour l’amplification par PCR. Différentes longueurs de fragments d’un gène d’entretien ménager (bêta-actine) ont été amplifiées et visualisées à l’aide de l’électrophorèse sur gel. Il n’y avait qu’une légère variation entre les échantillons individuels, ce qui peut être attribué au fait que tous les tissus ont un stade similaire d’athérosclérose. Cette hypothèse a été confirmée par l’histologie (données non présentées). De plus, ces données montrent que tous les échantillons ont été traités avec du formol de la même manière standard. Aucune différence significative n’a été observée en ce qui concerne la longueur des fragments obtenue à partir des différents échantillons, indépendamment de la méthode utilisée pour l’extraction de l’ADN. Par conséquent, la fragmentation du tissu FFPE ne dépend pas de la méthode utilisée pour l’isolement de l’ADN, mais est plutôt affectée par la variabilité des échantillons humains et le traitement au formol.
Étant donné que le système semi-automatisé utilisé est décrit comme un déménageur de particules magnétiques, et non comme un manipulateur de liquides, il offre également certains avantages par rapport aux autres systèmes semi-automatisés. Le principal avantage est le risque minimal de contamination croisée, car il n’y a pas de manipulation de liquide ou d’éclaboussures pendant le traitement des échantillons. Un autre point que nous voulons mentionner est la grande quantité d’échantillons, qui peuvent être traités en une seule fois dans le système, sans avoir beaucoup plus de temps de manipulation. L’utilisation d’un système semi-automatisé entraîne également une probabilité d’erreur d’utilisation plus faible qu’avec les méthodes basées sur des colonnes ou à séparation de phase. En particulier pour les analyses de biobanques et les travaux de diagnostic ainsi que pour la manipulation de tissus difficiles, la méthode semi-automatisée décrite ci-dessus est fortement recommandée. Dans ces domaines, il est très important que différents travailleurs et parfois même des personnes inexpérimentées obtiennent tous des résultats rapides, faciles et reproductibles. D’autres avantages du système sont le protocole généralement applicable, qui peut être utilisé pour tous les types de tissus ainsi que pour le sang, d’autres liquides corporels et aussi des cellules avec une seule modification mineure dans la première étape du protocole, où aucun tampon d’incubation n’est nécessaire.
Même si nos résultats ont clairement démontré la supériorité du système automatisé d’extraction d’ADN à partir de FFPE par rapport aux protocoles manuels, il est à noter que nous n’avons comparé que trois kits disponibles dans le commerce. Par conséquent, notre conclusion ne concerne que les méthodes testées dans notre étude. De plus, le nombre d’échantillons était faible et nous n’avons utilisé que des plaques d’athérosclérose à des fins de comparaison. Afin de généraliser les résultats de notre étude, d’autres échantillons de tissus en plus grand nombre seraient nécessaires.
Néanmoins, le système automatisé semble avoir un avantage considérable sur les kits manuels, en particulier pour les échantillons contenant une faible quantité d’acides nucléiques attendus. De plus, le système lui-même présente certaines limites. Par exemple, comme mentionné ci-dessus, il est nécessaire d’utiliser au minimum 60 μl pour l’élution, car le piston a besoin de cette quantité pour éluer l’ADN. De plus, la perte de volume d’élution est plus élevée pour le système semi-automatisé que pour les systèmes sur colonne. La méthode automatisée a une perte d’environ 10 μl de volume d’élution, tandis que les méthodes manuelles n’ont une perte que de 5 μl. De plus, les coûts plus élevés doivent être mentionnés. Mais pour les analyses à haut débit, le système semi-automatisé offre toujours les meilleurs avantages par rapport aux coûts. Comme dernière limitation du système, il est important de noter que l’extraction automatisée et semi-automatisée est un système fermé autonome. Il est bien sûr possible d’isoler, par exemple, l’ADN et les protéines, ou aussi l’ARN et l’ADN d’un seul échantillon, mais ce n’est pas si facile et souvent utilisé avec des méthodes manuelles de séparation de phase. Tous les tampons sont adaptés au kit et ne sont pas utilisables pour d’autres méthodes, contrairement aux réactifs Trizol. Enfin, il est également important de mentionner qu’en général, l’ADN des échantillons FFPE n’est pas adapté au séquençage du génome entier car la fragmentation des acides nucléiques est trop forte.
En conclusion, dans cette étude, nous démontrons qu’une méthode d’isolement de l’ADN entièrement automatisée pourrait être un excellent outil pour réduire le temps de manipulation et la quantité de matériel de départ. Aussi, il semble être le meilleur choix pour réduire les risques de contamination entre les échantillons. De plus, la flexibilité du système automatisé permet de traiter non pas un mais jusqu’à 48 échantillons par cycle, ce qui fait de cette méthode un outil utile pour les futures analyses à haut débit d’échantillons avec un faible nombre de cellules et donc une petite quantité d’acides nucléiques. La qualité et la quantité de l’ADN isolé à l’aide du système automatisé semblent supérieures à celles des autres kits manuels et l’utilisation dans d’autres analyses telles que le transfert de Southern, le génotypage de nucléotides uniques, les analyses de clonage, les tests de méthylation ainsi que l’amplification par PCR sont possibles.
Déclarations de divulgation
Les auteurs déclarent qu’il n’y a pas de conflit d’intérêts.
Remerciements
La mise en place du protocole d’extraction automatisée de l’ADN a été soutenue par le Dr Paul Muschler de la société Promega. Nous remercions Paul Muschler pour son soutien et sa contribution scientifique. Nous remercions également notre collègue le Dr Moritz von Scheidt (Centre cardiaque allemand de Munich) de nous avoir fourni l’instrument Maxwell et d’avoir soutenu la partie expérimentale. Toutes les expériences ont été réalisées dans les laboratoires du Centre allemand de cardiologie (Munich, Allemagne) et du Klinikum rechts der Isar (Munich, Allemagne). La recherche a été financée par la DFG (PE 900/6-1).
matériels
| Name | Company | Catalog Number | Comments |
| 1.5 ml tubes for sample incubation | Eppendorf, Hamburg, Germany | 30120086 | |
| 1-Thioglycerol | Promega, Walldorf, Germany | A208 | |
| Agilent tape station software 3.2 | Agilent, Waldbronn, Germany | ||
| dsDNA HS Kit | ThermoFisher Scientific, Schwerte, Germany | Q32851 | |
| FFPE DNA Purification Kits (Kit A) | Norgene Biotek, Heidelberg, Germany | 47400 | |
| FFPE tissue samples n=5 | Munich Vascular Biobank,Munich, Germany | ||
| GeneRead DNA FFPE Kit (Kit B) | Qiagen, Hilden, Germany | 180134 | |
| Heating blocks, set to 80°C and 65°C | VWR,Darmstadt,Germany | 460-0250 | |
| High Sensitivity D5000 reagents | Agilent, Waldbronn, Germany | 5067-5593 | |
| High Sensitivity D5000 ScreenTape | Agilent, Waldbronn, Germany | 5067-5592 | |
| Incubation Buffer | Promega, Walldorf, Germany | D920 | |
| Maxwell Blood Kit RSC including: Lysis Buffer, Elution Buffer, Proteinase K | Promega, Walldorf, Germany | AS1400 | |
| Maxwell RSC 48 Instrument | Promega, Walldorf, Germany | AS8500 | |
| Microcentrifuge | Eppendorf, Hamburg, Germany | ||
| NanoDrop 2000c Spectrometer | ThermoFisher Scientific, Schwerte, Germany | ND-2000C | |
| Optical caps | Agilent, Waldbronn, Germany | 401425 | |
| Optical tube strips | Agilent, Waldbronn, Germany | 401428 | |
| Pipettors and pipette tips | Eppendorf, Hamburg, Germany | ||
| Prism 6 for statistics, version 6.01 | GraphPad Inc., San Diego, California | ||
| Qubit 3.0 Fluorometer | ThermoFisher Scientific, Schwerte, Germany | Q33216 | |
| TapeStation 4200 | Agilent, Waldbronn, Germany | ||
| Tecan Infinite M200 Pro | Tecan, Männedorf, Swizerland | IN-MNANO |
Références
- Pelisek, J., et al. Biobanking: objectives, requirements, and future challenges-experiences from the munich vascular biobank. Journal of Clinical Medicine. 8 (2), 251(2019).
- Goelz, S. E., Hamilton, S. R., Vogelstein, B. Purification of DNA from formaldehyde fixed and paraffin embedded human tissue. Biochemical and Biophysical Research Communications. 130 (1), 118-126 (1985).
- Busch, A., Eken, S. M., Maegdefessel, L. Prospective and therapeutic screening value of non-coding RNA as biomarkers in cardiovascular disease. Annals of Translational Medicine. 4 (12), (2016).
- Kandpal, R. P., Saviola, B., Felton, J. The era of 'omics unlimited. BioTechniques. 46 (5), 351-355 (2009).
- McDonough, S. J., et al. Use of FFPE-derived DNA in next generation sequencing: DNA extraction methods. PloS One. 14 (4), (2019).
- Gilbert, M. T. P., et al. The isolation of nucleic acids from fixed, paraffin-embedded tissues-which methods are useful when. PloS One. 2 (6), (2007).
- Williams, C., et al. A high frequency of sequence alterations is due to formalin fixation of archival specimens. The American Journal of Pathology. 155 (5), 1467-1471 (1999).
- Solomon, M. J., Varshavsky, A. Formaldehyde-mediated DNA-protein crosslinking: a probe for in vivo chromatin structures. Proceedings of the National Academy of Sciences. 82 (19), 6470-6474 (1985).
- Gillio-Tos, A., et al. Efficient DNA extraction from 25-year-old paraffin-embedded tissues: study of 365 samples. Pathology. 39 (3), 345-348 (2007).
- Srinivasan, M., Sedmak, D., Jewell, S. Effect of fixatives and tissue processing on the content and integrity of nucleic acids. The American Journal of Pathology. 161 (6), 1961-1971 (2002).
- van Eijk, R., Stevens, L., Morreau, H., van Wezel, T. Assessment of a fully automated high-throughput DNA extraction method from formalin-fixed, paraffin-embedded tissue for KRAS, and BRAF somatic mutation analysis. Experimental and Molecular Pathology. 94 (1), 121-125 (2013).
- Sarnecka, A. K., et al. DNA extraction from FFPE tissue samples–a comparison of three procedures. Contemporary Oncology. 23 (1), 52(2019).
- Kalmár, A., et al. Comparison of automated and manual DNA isolation methods for DNA methylation analysis of biopsy, fresh frozen, and formalin-fixed, paraffin-embedded colorectal cancer samples. Journal of Laboratory Automation. 20 (6), 642-651 (2015).
- McCormick, S. F. Multilevel adaptive methods for partial differential equations. (SIAM). , (1989).
- Sambrook, J., Russell, D. W. Purification of nucleic acids by extraction with phenol: chloroform. Cold Spring Harbor Protocols. 2006 (1), 4455(2006).
- Berensmeier, S. Magnetic particles for the separation and purification of nucleic acids. Applied Microbiology and Biotechnology. 73 (3), 495-504 (2006).
- Petrigh, R. S., Fugassa, M. H. DNA extraction and a cost-effective detection method for Echinococcus granulosus protoscoleces. Veterinary Parasitology. 198 (3-4), 410-413 (2013).
- Lee, J. H., Park, Y., Choi, J. R., Lee, E. K., Kim, H. S. Comparisons of three automated systems for genomic DNA extraction in a clinical diagnostic laboratory. Yonsei Medical Journal. 51 (1), 104-110 (2010).
- Rohland, N., Siedel, H., Hofreiter, M. A rapid column-based ancient DNA extraction method for increased sample throughput. Molecular Ecology Resources. 10 (4), 677-683 (2010).
- Gutiérrez-López, R., Martínez-de la Puente, J., Gangoso, L., Soriguer, R. C., Figuerola, J. Comparison of manual and semi-automatic DNA extraction protocols for the barcoding characterization of hematophagous louse flies (Diptera: Hippoboscidae). Journal of Vector Ecology: Journal of the Society for Vector Ecology. 40 (1), 11-15 (2015).
- Seiler, C., et al. Nucleic acid extraction from formalin-fixed paraffin-embedded cancer cell line samples: a trade off between quantity and quality. BMC Clinical Pathology. 16 (1), 17(2016).
- Harada, S. Sample Preparation Techniques for Soil, Plant, and Animal Samples. , Springer. 125-138 (2016).
- Haile, S., et al. Automated high throughput nucleic acid purification from formalin-fixed paraffin-embedded tissue samples for next generation sequence analysis. PloS One. 12 (6), 0178706(2017).
- Fujii, T., et al. Evaluation of DNA and RNA quality from archival formalin-fixed paraffin-embedded tissue for next-generation sequencing-Retrospective study in Japanese single institution. Pathology International. , (2020).
- Ludyga, N., et al. Nucleic acids from long-term preserved FFPE tissues are suitable for downstream analyses. Virchows Archiv: An International Journal of Pathology. 460 (2), 131-140 (2012).
- Niland, E. E., McGuire, A., Cox, M. H., Sandusky, G. E. High quality DNA obtained with an automated DNA extraction method with 70+ year old formalin-fixed celloidin-embedded (FFCE) blocks from the indiana medical history museum. American Journal of Translational Research. 4 (2), 198(2012).
- Riemann, K., et al. Comparison of manual and automated nucleic acid extraction from whole-blood samples. Journal of Clinical Laboratory Analysis. 21 (4), 244-248 (2007).
- Steinau, M., Patel, S. S., Unger, E. R. Efficient DNA extraction for HPV genotyping in formalin-fixed, paraffin-embedded tissues. The Journal of Molecular Diagnostics. 13 (4), 377-381 (2011).
- Dundas, N., Leos, N. K., Mitui, M., Revell, P., Rogers, B. B. Comparison of automated nucleic acid extraction methods with manual extraction. The Journal of Molecular Diagnostics. 10 (4), 311-316 (2008).
- Okello, J. B., et al. Comparison of methods in the recovery of nucleic acids from archival formalin-fixed paraffin-embedded autopsy tissues. Analytical Biochemistry. 400 (1), 110-117 (2010).
- Witt, S., Neumann, J., Zierdt, H., Gébel, G., Röscheisen, C. Establishing a novel automated magnetic bead-based method for the extraction of DNA from a variety of forensic samples. Forensic Science International: Genetics. 6 (5), 539-547 (2012).
- Ivanova, N. V., Fazekas, A. J., Hebert, P. D. Semi-automated, membrane-based protocol for DNA isolation from plants. Plant Molecular Biology Reporter. 26 (3), 186(2008).
- Sengüven, B., Baris, E., Oygur, T., Berktas, M. Comparison of methods for the extraction of DNA from formalin-fixed, paraffin-embedded archival tissues. International Journal of Medical Sciences. 11 (5), 494(2014).
- Miller, S. A., Dykes, D. D., Polesky, H. F. A simple salting out procedure for extracting DNA from human nucleated cells. Nucleic Acids Research. 16 (3), 1215(1988).
- Wilfinger, W. W., Mackey, K., Chomczynski, P. Effect of pH and ionic strength on the spectrophotometric assessment of nucleic acid purity. BioTechniques. 22 (3), 474-481 (1997).
- Khokhar, S. K., Mitui, M., Leos, N. K., Rogers, B. B., Park, J. Y. Evaluation of Maxwell 16 for automated DNA extraction from whole blood and formalin-fixed paraffin embedded (FFPE) tissue. Clinical Chemistry and Laboratory Medicine (CCLM). 50 (2), 267-272 (2012).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon