Method Article
Optimierte hochwertige DNA-Extraktion aus formalinfixierten, in Paraffin eingebetteten humanen atherosklerotischen Läsionen
In diesem Artikel
Zusammenfassung
Hier stellen wir ein Protokoll für die halbautomatische DNA-Extraktion aus formalinfixierten, in Paraffin eingebetteten Läsionen menschlicher Halsschlagadern vor. Die Gewebelyse wird ohne toxisches Xylol durchgeführt, gefolgt von einem automatisierten DNA-Extraktionsprotokoll, das einen zweiten Lyseschritt, die Bindung der DNA an paramagnetische Partikel für die Bindung auf Zellulosebasis, Waschschritte und die DNA-Elution umfasst.
Zusammenfassung
Formalin-fixierte, in Paraffin eingebettete (FFPE) Gewebe stellen eine wertvolle Quelle für molekulare Analysen und klinische genomische Studien dar. Diese Gewebe sind oft arm an Zellen oder schwer zu verarbeiten. Daher müssen Nukleinsäuren sorgfältig isoliert werden. In den letzten Jahren wurden verschiedene Methoden zur DNA-Isolierung für Gewebe aus vielen Krankheiten, vor allem Krebs, etabliert. Leider wird genomische DNA, die aus FFPE-Geweben extrahiert wird, aufgrund der Vernetzung zwischen Nukleinsäuresträngen und Proteinen sowie durch zufällige Brüche in der Sequenz stark abgebaut. Daher ist die DNA-Qualität dieser Proben deutlich reduziert, was sie zu einer Herausforderung für weitere molekulare Downstream-Analysen macht. Weitere Probleme bei schwierigen Geweben sind z.B. der Mangel an Zellen in verkalkten atherosklerotischen Läsionen und Fettgewebe des Menschen, kleine Hautbiopsien und folglich eine geringe Verfügbarkeit der gewünschten Nukleinsäuren, wie es auch in altem oder fixiertem Gewebe der Fall ist.
In unseren Laboren haben wir eine Methode zur DNA-Extraktion aus formalinfixierten atherosklerotischen Läsionen unter Verwendung eines halbautomatischen Isolationssystems etabliert. Wir verglichen diese Methode mit anderen kommerziell erhältlichen Extraktionsprotokollen und konzentrierten uns auf weitere nachgelagerte Analysen. Reinheit und Konzentration der DNA wurden mittels Spektrometrie und Fluorometrie gemessen. Bewertet wurden der Grad der Fragmentierung und die Gesamtqualität.
Die höchste DNA-Quantität und -Qualität wurde mit dem modifizierten Blut-DNA-Protokoll für das automatisierte Extraktionssystem anstelle des kommerziellen FFPE-Protokolls erreicht. Mit diesem Schritt-für-Schritt-Protokoll waren die DNA-Ausbeuten von FFPE-Proben im Durchschnitt viermal höher und weniger Proben bestanden den Extraktionsprozess, was bei Biopsien kleiner Gefäße von entscheidender Bedeutung ist. Amplikongrößen von 200–800 bp konnten mittels PCR nachgewiesen werden. Diese Studie zeigt, dass die aus unserem FFPE-Gewebe gewonnene DNA zwar stark fragmentiert ist, aber dennoch für eine erfolgreiche Amplifikation und Sequenzierung kürzerer Produkte verwendet werden kann. Zusammenfassend lässt sich sagen, dass die automatisierte Technologie in unseren Händen das beste System für die DNA-Extraktion zu sein scheint, insbesondere für kleine FFPE-Gewebeproben.
Einleitung
Die Formalinfixierung mit anschließender Paraffineinbettung (FFPE) ist ein Standardverfahren zur Langzeitkonservierung pathologischer Proben im Biobanking1. Diese Proben stellen eine wertvolle Quelle für histologische Studien sowie molekulare Analysen, insbesondere genetische Studien,dar 2. Weitere Vorteile von FFPE-Geweben sind eine bessere Langzeitlagerung, geringere Kosten und einfachere Lagerbedingungen. Unsere Absicht ist es, ein zuverlässiges und einfach zu handhabendes Protokoll für die reproduzierbare Nukleinsäureisolierung aus kleinen Mengen von FFPE-Schnitten bereitzustellen, da eine qualitativ hochwertige DNA-Extraktion der erste entscheidende Schritt in einer Vielzahl von molekularen Techniken ist und FFPE-Gewebe die am besten verfügbare Probenquelle sind.
Neue wissenschaftliche Ansätze, wie z. B. Next-Generation-Sequencing (NGS) und "Omics"-Forschungsansätze, erfordern eine hohe Qualität der Nukleinsäuren 3,4,5. Die Extraktion von DNA aus FFPE-Gewebeproben ist nach wie vor eine Herausforderung. DNA aus FFPE-Proben kann je nach Alter und Fixierungsbedingungen in Qualität und Quantität stark variieren. Formalin, die am häufigsten verwendete Verbindung, führt zur DNA-Protein-Vernetzung 6,7,8 und bewirkt einen unspezifischen zufälligen Bruch in der Nukleotidsequenz9. Dies kann sich erheblich auf nachgelagerte genomische Analysen auswirken, da die Vernetzung die Amplifikation der Polymerase-Kettenreaktion (PCR) deaktivieren kann 6,10. Aufgrund von Verunreinigungen während des Fixierungsprozesses ist die Reinheit der aus FFPE-Proben isolierten DNA oft eingeschränkt. In den letzten Jahren wurden verschiedene Methoden zur DNA-Isolierung etabliert, meist aus Krebsgewebeproben 2,11,12,13.
Generell können Protokolle für die Extraktion von Nukleinsäuren aus FFPE-Gewebe in drei Hauptgruppen unterschieden werden. Die erste, am häufigsten verwendete Gruppe von Verfahren umfasst kommerziell erhältliche Säulensysteme auf Kieselsäurebasis14. Die zweite Gruppe umfasst manuelle Extraktionsmethoden in der organischen Phase mit Phenol und Chloroform, die erstmals von Joseph Sambrook und David W. Russellbeschrieben wurden 15. Als dritte Gruppe haben sich in den letzten Jahren automatisierte Systeme etabliert, wie z.B. Liquid-Handling-Systeme sowie paramagnetische partikelbasierte Systeme16. Jedes der drei genannten Systeme hat unterschiedliche Vor- und Nachteile, wie z. B. gefährliche Chemikalien (d. h. Xylol, Phenol, Chloroform), hohe Kosten17, Arbeitskraft18 und Zeitaufwand19. Insbesondere für die schwierigen Gewebeproben sowie Hochdurchsatzanalysen sind Standardisierung, Reproduzierbarkeit, relativ geringer Zeit-, Personal- und Kostenaufwand die wichtigsten Merkmale bei der Suche nach einem geeigneten Verfahren zur Nukleinsäureisolierung20. Es ist bekannt, dass automatisierte Extraktionsmethoden bessere reproduzierbare Ergebnisse liefern und für kleine Biopsien empfindlicher sind. Darüber hinaus wird weniger Gewebe oder Blut benötigt und das Risiko einer Verstopfung des Systems durch hohe Mengen an Paraffin wird reduziert. Obwohl Maschinen zur automatisierten Nukleinsäureextraktion und die benötigten Kits im Vergleich zu manuellen Methoden teurer sind, überzeugen sie dennoch durch weniger problematische Extraktionsprozesse. Die Literaturrecherche bietet eine Vielzahl von Publikationen, die einen direkten Vergleich zwischen manuellen, säulenbasierten und automatisierten DNA- und RNA-Extraktionsmethoden aus verschiedenen Geweben und Organismen wie Pflanzen, Tieren und Menschen sowie Zellen in Kultur veranschaulichen 20,21,22. Es gibt auch Hinweise in der Literatur, die zeigen, dass DNA und RNA, die aus 10 Jahre altem schockgefrorenem Gewebe isoliert wurden, für nachgelagerte Analysen wie PCR, quantitative PCR, NGS, Methylierungsanalysen und Klonierungverwendet werden können 9,23,24,25,26.
Das Hauptproblem bei z.B. gealtertem humanem Gefäßgewebe sowie kleinen Gewebebiopsien, insbesondere bei FFPE-Proben, ist der Mangel an Zellen in den stark verkalkten atherosklerotischen Läsionen, was folglich zu niedrigen Konzentrationen von Nukleinsäurenführt 1. Obwohl mehrere Methoden zur DNA-Extraktion aus FFPE-Gewebe bereits etabliert und weit verbreitet sind, erfordern die manuellen Probenvorbereitungsmethoden eine lange praktische Zeit27 und toxische Reagenzien wie Xylol oder Phenol sind für die Entparaffinisierung erforderlich2. Wie beschrieben, ist der Entparaffinierungsprozess ein entscheidender, zeitaufwändiger Schritt (z. B. etwa 30 Minuten), der die Qualität und Quantität der extrahierten DNA deutlich beeinflusst (z. B. toxische Wirkungen auf die DNA, wie z. B. Fragmentierung und Abbau der Entparaffinierungslösung und hohe Temperaturen)28. Kürzlich entwickelte neue DNA-Extraktionsprotokolle konzentrieren sich auf die Verwendung anderer ungiftiger Entparaffinierungslösungen, Reparaturstrategien und automatisierter Bead-Technologien. Insbesondere hat sich gezeigt, dass automatisierte und halbautomatische Methoden bei der DNA-Extraktion erfolgreich sind, da sie eine effiziente Rückgewinnung, keine Kreuzkontamination und eine einfache Leistung aufweisen29. Wir haben ein Protokoll etabliert, das diese Einschränkungen überwindet. Dadurch ermöglicht unsere Technik eine Reduzierung der Bearbeitungs- und Hands-on-Zeit bei höchsten quantitativen und qualitativen Standards.
Insbesondere bei reproduzierbaren Hochdurchsatzanalysen wie Genotypisierung, epigenomischen Studien und RNA-Sequenzierung ist die Handhabung von FFPE-Proben mit säulenbasierten Aufreinigungssystemen oft schwierig und zeitaufwändig (z. B. lange Entparaffinisierungsschritte, Säulenverstopfung und lange Hands-on-Zeiten). Das Hauptproblem ist die Verstopfung der Kieselsäuremembranen durch den hohen Paraffingehalt. Weitere Umstände, die die Isolierung von hochwertigen Nukleinsäuren verschlechtern können, sind kleine Gewebemengen wie Mikrobiopsien der Haut, kleines Mausgewebe, sehr fettiges oder verkalktes Gewebe wie Plaques, verknöchertes Gewebe und gealterte Proben. Insbesondere in der Diagnose und Forensik sind automatisierte und halbautomatische Systeme wie Liquid Handling oder paramagnetische partikelbasierte Extraktionsmethoden in den letzten Jahren immer wichtiger geworden30,31, vor allem aufgrund der relativ geringen Hands-on-Zeiten und der Möglichkeit der Standardisierung. Die meisten der bereits veröffentlichten Protokolle eignen sich perfekt für glatte Gewebe mit hohen oder mittleren Zellmengen, wie z. B. Tumorbiopsien oder Pflanzengewebe 13,22,32. Die Literatur über Methoden für halbautomatische partikelbasierte Methoden, die zur Isolierung von DNA aus relativ schwer zu handhabendem Gewebe wie fixierten Einzelzellen, verkalkten Gefäßen, kollagenreichem Gewebe und Fettgewebe mit geringer Zellzahl verwendet werden, ist nur unzureichend beschrieben33.
In dieser Arbeit wird eine optimierte halbautomatische Methode zur DNA-Isolierung aus vaskulären Paraffin-eingebetteten Schnitten beschrieben und mit zwei manuellen säulenbasierten Protokollen verglichen. Für die Validierung wurden die DNA-Menge, die Reinheit und das Ausmaß der Fragmentierung verwendet. Als Ausgangspunkt diente das kommerziell erhältliche Blut-DNA-Protokoll, und die manuellen Schritte des halbautomatischen Systems wurden anschließend für die Verwendung von FFPE sowie frischen gefrorenen Gewebeproben aus menschlichem und tierischem Gewebe optimiert, wobei Schritte aus dem FFPE- und dem Gewebeprotokoll kombiniert wurden. Der automatisierte Schritt dieses Protokolls ist auf dem Gerät vorinstalliert und hängt vom verwendeten Kit (hier dem Blut-DNA-Kit) ab. Mit dem beschriebenen halbautomatischen kartuschenbasierten System ist es möglich, DNA aus Blut, frisch gefrorenem Gewebe, formalinfixiertem Gewebe und sogar einzelnen Zellen mit dem gleichen Protokoll, der gleichen Maschine, dem gleichen Kit und den gleichen Verbrauchsmaterialien zu isolieren, anstatt verschiedene Protokolle und Kits für das Instrument zu verwenden, wie es vom Unternehmen empfohlen wird. Es gibt nur geringfügige Unterschiede in den Protokollen, wie z.B. einen Puffer und einige Inkubationszeiten für die verschiedenen Anwendungen, was dieses Protokoll sehr nützlich für die Extraktion von DNA aus allen Arten von Geweben macht. Unser Protokoll ist in erster Linie für verkalktes, zellarmes und fibröses menschliches Gefäßgewebe optimiert, kann aber natürlich für alle Arten von schwierigen Geweben, die oben erwähnt wurden, verwendet und weiter optimiert werden.
Zusammengefasst bieten wir Forschern im kardiovaskulären Bereich, die sich mit Atherosklerose (z. B. Aorta, Halsschlagadern, Koronararterien) befassen, ein einfach zu bedienendes Punkt-für-Punkt-Protokoll für die halbautomatische DNA-Extraktion aus vaskulären FFPE-Proben.
Protokoll
Die Erlaubnis zur Entnahme von atherosklerotischen Proben der menschlichen Halsschlagader in unserer Biobank wurde von der örtlichen Ethikkommission des Krankenhauses (2799/10, Ethikkommission der Fakultät für Medizin der Technischen Universität München, München, Deutschland) genehmigt. Von allen Patienten wurde eine schriftliche Einverständniserklärung eingeholt. Die Versuche wurden in Übereinstimmung mit den Grundsätzen der Deklaration von Helsinki durchgeführt.
1. Vorbereitung des Gewebes
- Bereiten Sie 5–8 Gewebeschnitte von 10 μm aus der FFPE-Probe mit dem Mikrotom vor und übertragen Sie es in ein 1,5-ml-Röhrchen. Es ist nicht notwendig, den Überschuss an Paraffin aus dem Block zu reduzieren.
HINWEIS: Dünnere Einzelabschnitte anstelle eines großen Abschnitts beschleunigen die Pufferreaktion. Die ersten Abschnitte wegen O2-Exposition verwerfen. Bei größeren Proben ist es auch möglich, weniger Schnitte zu verwenden. - Zentrifugieren Sie diese Röhrchen in einer Tischzentrifuge mit einer Temperatur von 5.000 x g für 1 Minute bei Raumtemperatur, um jede Probe am Boden des Röhrchens zu sammeln.
ACHTUNG: Eine zu lange Zentrifugation führt zur Gerinnung der Probe und erschwert die Lyse.
2. Lipidauflösung und Entparaffinisierung
HINWEIS:Dieser Schritt ist für die Entparaffinisierung und den Lipidaufschluss erforderlich. Der verwendete Puffer ist weniger toxisch als kommerzielle Entparaffinierungslösungen.
- Geben Sie 300 μl des handelsüblichen Inkubationspuffers und 6 μl 1-Thioglycerin in jedes Röhrchen.
HINWEIS: Verwenden Sie nicht mehr als 300 μl, da dies das maximale Volumen der Patrone des Automatisierungssystems ist. - 10 s vortexen und die Probe 10 min bei 80 °C und 500 U/min in einem Heizblock inkubieren, um Paraffin zu lösen.
ACHTUNG: Das Gewebe sollte sich am Ende vollständig aufgelöst haben. Falls erforderlich, während der Inkubation mehrmals vortexen.
3. Proben- und Proteinverdauung
HINWEIS: Der native Verdau mit Protease K ist entscheidend, um saubere DNA-Extrakte ohne Proteine zu erhalten. Es reduziert auch alle vorhandenen kontaminierenden Proteine. Darüber hinaus werden auch Nukleasen zerstört, um die DNAzu retten 34. Dieser Schritt über Nacht ist auch für den vollständigen Probenaufschluss erforderlich.
- Lassen Sie die Probe auf 60 °C abkühlen und fügen Sie dann 30 μl der mitgelieferten Proteinase K-Lösung hinzu.
- Erneut vortexen und die Mischung bei 65 °C und 500 U/min über Nacht (4–20 h) in einem Heizblock inkubieren. Während der Inkubation werden die Proben von Zeit zu Zeit vortext, um einen vollständigen Probenaufschluss zu erzielen.
HINWEIS: Die Inkubation über Nacht führt zu besseren Ergebnissen. Mischschritte werden alle 30–60 Minuten empfohlen. Am Ende sollte sich kein sichtbares Gewebestück im Inneren des Röhrchens befinden.
4. Zelllyse
- Geben Sie 400 μl des Lysepuffers, der im Blutkit enthalten ist, hinzu und wirbeln Sie ihn in Kürze ein.
- Die Probe erneut bei 65 °C für 30 min mit 500 U/min inkubieren.
- Lassen Sie die Probe auf Raumtemperatur abkühlen. Das Paraffin härtet oben aus.
ACHTUNG: Nicht erneut vortexen, um das Paraffin von der Probe getrennt zu halten. Andernfalls wird das Paraffin mit dem Gewebe vermischt, wodurch die Probe zerstört wird. Die Probe wird in Schritt 6.1 entnommen.
5. Vorbereitung der vordosierten Kartuschen
- Schalten Sie das Gerät und den zugehörigen Tablet-Computer ein.
- Starten Sie die Software-App und klicken Sie auf die Tür-Taste, um das Instrument zu öffnen.
- Entfernen Sie das Gestell vom Gerät und setzen Sie die vorgefüllte Kartusche in das Sondengestell ein. Stellen Sie sicher, dass die Kartusche beim Einrasten zweimal einrastet und entfernen Sie die Siegelfolie.
- Setzen Sie den Kolben in die letzte (8) Vertiefung der Kartusche ein. Er dient als Pipettenspitze im Instrument.
- Füllen Sie die mitgelieferten 0,5 mL Elutionsröhrchen mit 65 μl Elutionspuffer, die mit dem Kit geliefert werden. Lassen Sie die Röhrchen offen und setzen Sie sie in die dafür vorgesehene Position im vorderen Teil des Racks nach der Kartusche ein.
HINWEIS: Das Mindestvolumen für die Elution beträgt 60 μL. Das System verliert 5–10 μl des hinzugefügten Elutionsvolumens.
6. Automatisierte DNA-Extraktion
- Durchstechen Sie vorsichtig das Paraffin auf dem 1,5-ml-Röhrchen aus Schritt 4.3, um an die saubere Probe am Boden des Röhrchens zu gelangen, ohne es erneut mit Paraffin zu vermischen.
- Die gesamte Mischung (730 μl) der vorbereiteten Probe wird in die erste Vertiefung der Patrone übertragen.
- Setzen Sie das Gestell in die automatisierte DNA-Extraktionsmaschine ein. Stellen Sie sicher, dass das Gestell zuerst hinten und danach vorne einrastet.
- Starten Sie den Lauf, indem Sie in der Software auf die orangefarbene Schaltfläche Start oben links klicken. Es öffnet sich ein Fenster mit verschiedenen vorinstallierten Protokollen. Wählen Sie Blut-DNA-Protokoll auf dem Instrument. Bestätigen Sie, dass der Kolben, das Elutionsrohr und die Probe hinzugefügt wurden, indem Sie in der Software auf Ja klicken. Die Klappe des Instruments schließt sich automatisch und der Lauf beginnt (das Licht leuchtet grün). Die Fahrt dauert ca. 38 Minuten. Es ist keine weitere Kalibrierung erforderlich.
HINWEIS: Beobachten Sie, bis das System die Kolben für alle Proben im Rack aufgenommen hat. Ist dies nicht der Fall, stoppt das System automatisch und das Maschinenprotokoll muss neu gestartet werden. - Stellen Sie sicher, dass das System den automatisierten Lyseschritt in der ersten Vertiefung der Kartusche durchführt, gefolgt von den Waschschritten in den Vertiefungen 3 bis 7. Es ist kein weiterer Programmierschritt erforderlich. Das komplette Programm wird von der Firma vorinstalliert.
- Stellen Sie anschließend sicher, dass das System die DNA in den vorbereiteten Elutionsröhrchen über den hinzugefügten Kolben eluiert. Die magnetischen Partikel bleiben im Kolben. Der Kolben geht am Ende zurück in die letzte Vertiefung der Patrone.
7. Beende den Lauf
- Wenn der Lauf abgeschlossen ist (das Gerät blinkt grün), öffnen Sie das Instrument, indem Sie auf die Schaltfläche zum Öffnen (Türschild) klicken, und entfernen Sie das Gestell aus dem System.
- Entsorgen Sie die Patronen.
- Setzen Sie das leere Gestell wieder in das Instrument ein und schließen Sie die Tür über den Türknopf in der oberen rechten Ecke. Schließen Sie die Software-App und schalten Sie das Gerät sowie den Tablet-Computer aus.
- Lagern Sie die Eluate bei -20 °C für die Langzeitlagerung oder bei 4 °C für die Kurzzeitlagerung oder verwenden Sie sie direkt für nachgelagerte Analysen oder Konzentrationsmessungen.
Ergebnisse
Für die Erstellung des Protokolls wurden 5 FFPE-Gewebeblöcke von Patienten mit Arteriosklerose der Halsschlagader verwendet. Die DNA wurde sowohl mit einem optimierten halbautomatischen Protokoll (Kit C) als auch mit zwei kommerziell erhältlichen manuellen säulenbasierten Protokollen (Kit A und Kit B, siehe Materialtabelle) isoliert. Die DNA-Extraktion mit Kit A und B wurde gemäß dem Protokoll des Herstellers durchgeführt. Die einzige Änderung, die im Protokoll der beiden kommerziell erhältlichen Kits (Kit A und Kit B) vorgenommen wurde: Die Deparaffinisierung zu Beginn wurde aufgrund der hohen Paraffinmengen in der Umgebung der Schnitte zweimal durchgeführt. Nach der Entparaffinierung mit Xylol wurde die DNA an die Säulenmembran gebunden und dreimal gewaschen. Nach dem Trocknen der Membran durch Zentrifugation wurde die DNA mit 60 μl des bereitgestellten Elutionspuffers eluiert.
Um die optimale Menge an Proben zu bestimmen, die für reproduzierbare Ergebnisse von jedem Patienten benötigt wird, wurden unterschiedliche Materialmengen entnommen. Es wurden konsekutive Gewebeschnitte von 2 x 10 μm (insgesamt 20 μm), 5 x 10 μm (insgesamt 50 μm) und 8 x 10 μm (insgesamt 80 μm) verwendet (Abbildung 1). Die Qualität und Quantität der gesamten genomischen DNA, die mit dem automatisierten System und den zweimanuellen säulenbasierten Systemen isoliert wurde, wurden sowohl mit Spektrometrie, einem spektrometrischen System mit Mikroplatten-Readern als auch mit Fluorometrie bewertet. Die Fragmentierungsanalyse wurde mit Hilfe der automatisierten Screen-Tape-Technologie und der Polymerase-Kettenreaktion (PCR) durchgeführt. Die statistische Auswertung erfolgte mit kommerziell erhältlicher Software (siehe Materialtabelle).
Das halbautomatische Protokoll führt zu einer höheren Menge der extrahierten DNA
Mit Hilfe der Mikrovolumenspektrometrie und der Plattenleserspektrometrie sowie der Fluorometrie, wie in Abbildung 2 gezeigt, wurde die Gesamtmenge an genomischer DNA für die verschiedenen Mengen an Gewebeschnitten (insgesamt 20 μm, 50 μm, 80 μm) verglichen. Ein linearer Anstieg der DNA-Konzentration wurde mit dem kartuschenbasierten halbautomatischen Kit C mit allen drei Quantifizierungsmethoden erzielt. Für die manuellen Kits A und B wurden keine signifikanten Unterschiede zwischen 20 μm-, 50 μm- und 80 μm-Abschnitten beobachtet. Die mittlere Konzentration aller fünf Gewebeproben, die mit Mikrovolumenspektrometrie und Mikroplatten-Reader-Technologie für DNA gemessen wurden, die mit dem halbautomatischen Protokoll extrahiert wurde (Abbildung 2A, B, Tabelle 1), zeigte eine signifikante, viermal höhere Konzentration (45 ng/μl) im Vergleich zu den säulenbasierten Protokollen A und B (p = 0,016). Kit A erreichte mittlere Konzentrationen von 12,5 ng/μL, während Kit B nur 8,5 ng/μL erreichte. Abbildung 3 zeigt die Qualität der extrahierten DNA mit den drei genannten Methoden. Zusammenfassend lässt sich sagen, dass es keinen großen Qualitätsunterschied gibt. Der halbautomatische Bausatz zeigt eine stabilere Qualität, die gemessenen Mengen sind vergleichbar. Ähnliche Ergebnisse wurden durch fluorimetrische Messungen der DNA-Konzentration erzielt (Abbildung 2C). Die mittleren DNA-Konzentrationen mit Kit A wurden im Vergleich zur spektrometrischen Analyse weiter auf 3 ng/μL reduziert. Fasst man die Menge an DNA zusammen, so erwies sich das automatisierte Protokoll als die effizienteste Methode für Proben aus niedrigem Zellgewebe.
Automatisiertes Extraktionsprotokoll führt zu einer besseren Qualität der extrahierten DNA
Darüber hinaus wurde die Qualität der mit den drei verschiedenen Protokollen isolierten DNA spektrophotometrisch bewertet, sowohl mit einem Spektralphotometer als auch mit einem Mikroplatten-Reader, wobei das Verhältnis A260/280 berechnet wurde (Abbildung 3A, B und Tabelle 2). Nukleinsäuren haben ihr Absorptionsmaximum bei 260 nm. Die Reinheit hochwertiger DNA liegt im Allgemeinen im Bereich von A260/280-Werten von 1,6 bis 1,8, wie die beiden gestrichelten Linien in Abbildung 3A,B 35 zeigen. Das mittlere Verhältnis des manuellen säulenbasierten Kits A betrug 1,92 ± 0,6 in der Spektrometrie und 2,58 ± 1,2, gemessen mit der Mikroplatten-Reader-Technologie. Kit B zeigte kleinere Verhältnisse (1,45 ± 0,3 und 1,85 ± 0,7). DNA, die mit dem halbautomatischen Kit C isoliert wurde, zeigte Reinheitsverhältnisse von 1,4 ± 0,07 in der Spektrometrie und 1,68 ± 0,05, berechnet mit dem Mikroplatten-Reader. In beiden Messungen zeigte die automatisierte Isolationsmethode eine geringere Variation des Reinheitsverhältnisses als die manuellen Kits, aber nur Kit A zeigte aufgrund der enormen Streuung signifikante Unterschiede in der gepaarten t-Test-Analyse (p = 0,0025 und p < 0,001). Bei der Mikrotiterplatten-Messung erfüllten alle Proben, die mit dem automatisierten System isoliert wurden, die Reinheitskriterien (Verhältnisse zwischen 1,6 und 1,8), während Kit A und Kit B diese Kriterien in vielen Fällen nicht erfüllten (p < 0,001). Die Spektrometrie (Abbildung 3A) zeigte, dass nicht alle Proben, die mit dem optimierten Kartuschensystem isoliert wurden (Mindestverhältnis = 1,36), den definierten Reinheitsschwellenwert erreichten (gestrichelte Linien, die das A260/280-Verhältnis von 1,6 und 1,8 anzeigen), was auf eine Pufferkontamination hinweist. Darüber hinaus wurde eine große Variation des berechneten Verhältnisses für die Kits A und B beobachtet. Folglich liefert die halbautomatische DNA-Isolierung Proben mit vergleichbarer und reproduzierbarer Qualität, während die DNA-Qualität bei säulenbasierten Isolationsmethoden stark schwankt. Eine weitere Qualitätsprüfung wurde mit Hilfe der DV200-Berechnung durchgeführt (Abbildung 3A, Tabelle 2). Hier zeigte das manuelle säulenbasierte Extraktionssystem A einen Wert von 90% ± 3,8 für Fragmente größer als 200 bp, während Kit B nur 85% ± 7,1 (p = 0,01) zeigte. Auch in dieser Analyse zeigte DNA, die mit dem halbautomatischen Protokoll extrahiert wurde, den höchsten Prozentsatz an Fragmenten, die größer als 200 bp waren (92% ± 3,6).
Zusammenfassend lässt sich sagen, dass die Qualität der mit dem halbautomatischen System extrahierten DNA stabiler und reproduzierbarer war, während die mit den säulenbasierten Systemen isolierte DNA eine größere Variation der Qualitätsverhältnisse aufwies. DV200 zeigte eine ähnliche Qualität für die Kits A und C, während Kit B weniger Fragmente aufwies, die größer als 200 bp waren.
Die DNA-Fragmentierung ist unabhängig vom verwendeten Protokoll
Die Fragmentierung der DNA wurde mit halbautomatischer Screen-Tape-Technologie bewertet. In Abbildung 4A sind repräsentative Gele dargestellt, die das Ausmaß der Fragmentierung aller in dieser Studie verwendeten Proben anzeigen. Dunkle Farben weisen auf eine höhere Dichte hin, während grau schattierte Bilder auf eine niedrigere Dichte hinweisen. Hier wurden ähnliche Muster für alle Proben und in allen drei Kits beobachtet. Die ersten drei Proben zeigen den dunkelsten Bereich (was der höchsten Ansammlung der fragmentierten DNA entspricht), im unteren Teil des Gels, der die kleineren DNA-Fragmente darstellt, zeigen Proben von Patient 4 und 5 mehr (dichtere) Fragmente im oberen Teil des Gels, was auf eine weniger fragmentierte DNA hinweist. In Abbildung 4B ist der Unterschied in der DNA-Fragmentierung mit verschiedenen Materialmengen (20 μm, 50 μm und 80 μm) für eine repräsentative Probe unter Verwendung aller drei Isolationsstrategien dargestellt. Elektropherogramme zeigen die entsprechende Fragmentverteilung. Die Daten sind in Tabelle 3 zusammengefasst. Hier wurde der einzige visuelle Unterschied zwischen den drei Kits in den Diagrammen der 20-μm-Abschnitte gefunden, wobei das automatisierte Kit ein klareres Elektropherogramm mit mehr detektierter DNA zeigte. Für 50 μm und 80 μm Schnitte wurde kein sichtbarer Unterschied im Elektropherogramm zwischen den verschiedenen in der Studie verwendeten Kits festgestellt.
Abbildung 5A zeigt die mittlere Verteilung der Fragmentgrößen für das halbautomatische Kartuschensystem Kit C sowie für die beiden manuellen Kits A und B. Die mittlere Verteilung der Fragmentgrößen für das automatisierte System zeigt folgende Ergebnisse: 5 % der DNA-Fragmente waren kleiner als 200 bp, 62 % der DNA-Fragmente zwischen 200 bp und 1500 bp und 29 % der DNA-Fragmente waren länger als 1.500 bp. Ein höherer Prozentsatz der augmentierten DNA-Fragmentierung wurde mit Kit B (12 %) und Kit A (8 %) gemessen. Mittelgroße Fragmente waren bei Kit A (62 %) und Kit B (57 %) ähnlich. Hinsichtlich der längeren DNA-Fragmente wurde kein signifikanter Unterschied (Kit A 26%, Kit B 27%) beobachtet. Dies führt zu der Schlussfolgerung, dass DNA, die mit dem optimierten halbautomatischen Kit (Kit C) isoliert wurde, weniger fragmentiert zu sein scheint als DNA, die mit säulenbasierten Kits isoliert wurde.
Der Fragmentierungspeak (höchste DNA-Menge für eine bestimmte Fragmentgröße) in allen drei Kits wurde für jede der fünf Gewebeproben analysiert (Abbildung 5B). Es konnten keine Unterschiede zwischen den drei Isolierungsmethoden hinsichtlich der gesamten DNA-Fragmentierung festgestellt werden. Unabhängig vom verwendeten Kit zeigen die Proben 1 bis 3 einen durchschnittlichen maximalen Peak zwischen 280 bp und 620 bp, während die Proben 4 und 5 mit einem durchschnittlichen Fragmentierungspeak zwischen 1.200 bp und 1.800 bp weniger fragmentiert sind. Diese Ergebnisse deuten darauf hin, dass die DNA-Fragmentierung von der Qualität der Proben selbst beeinflusst wird.
Die mit dem halbautomatischen Kit extrahierte DNA ist für die PCR-Analyse geeignet
Letztendlich wurden acht DNA-Proben, die mit dem optimierten Protokoll isoliert wurden, für die nachgelagerte Analyse verwendet, die eine PCR mit unterschiedlichen Fragmentlängen durchführte. Es wurden geeignete Primer für das Beta-Aktin-Haushaltsgen ausgewählt. Als Kontrolle diente eine primäre Zelllinie der glatten Muskulatur (Abbildung 6). Durch die Darstellung der nachweisbaren Fragmente in der Agarose-Gelelektrophorese wurden sowohl Proben mit großen maximalen Fragmenten (500 bp – 800 bp für die Proben 1, 2 und 8) als auch Proben mit nur 200 bp maximalen Fragmenten (Proben 6 und 7) detektiert, unabhängig von der verwendeten Extraktionsmethode.
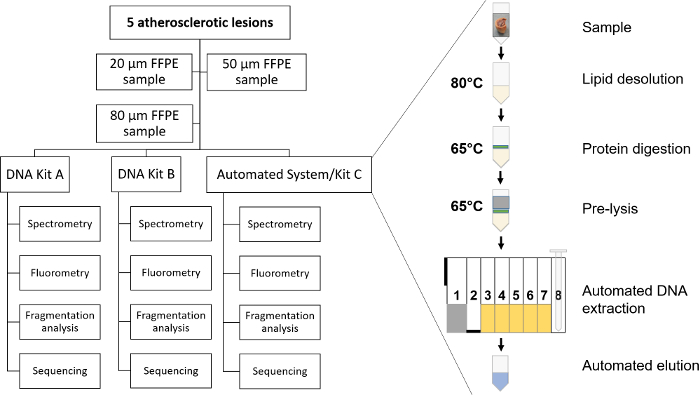
Abbildung 1: Experimenteller Arbeitsablauf. Die Hauptschritte der Probenvorbereitung und der halbautomatischen DNA-Extraktion werden im Kontext der verwendeten Proben und im Vergleich zu den manuellen DNA-Extraktionskits A und B dargestellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
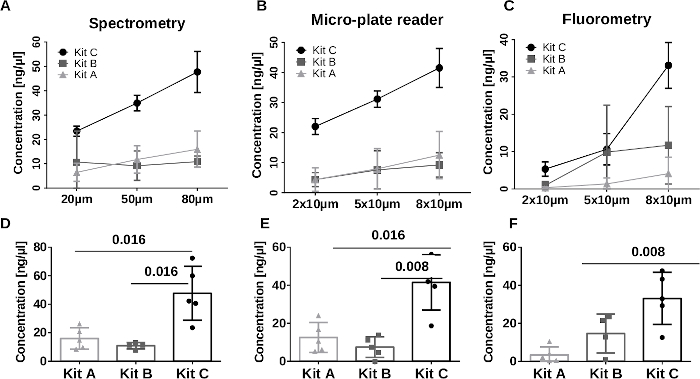
Abbildung 2: Konzentrationsmessung. Der obere Teil (A-C) zeigt die DNA-Konzentrationen, die mit Spektrometrie, Mikroplatten-Reader-Technologie und Fluorometrie in 20-μm-, 50-μm- und 80-μm-Schnitten (jeweils n = 5) für alle drei verschiedenen verwendeten Kits gemessen wurden. Der untere Teil zeigt die Verteilung der DNA-Konzentrationen von fünf Proben (80 μm), die mit Spektrometrie (D), Mikroplatten-Reader-Technologie (E) und Fluorometrie (F) gemessen wurden. p-Werte werden mit dem gepaarten t-Test berechnet, p < 0,05 wurde als signifikant angesehen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: Reinheit der isolierten DNA. Die Reinheit der DNA wurde durch Berechnung des Verhältnisses A260/280 in zwei verschiedenen Spektrometern (A,B) für alle drei verschiedenen Kits (n = 15) bewertet. Gestrichelte Linien zeigen das optimale Verhältnis (1,6–1,8). (C) zeigt den DV200 (Fragmente >200 bp) in %, berechnet mittels Fragmentierungsanalyse. Die p-Werte werden mit dem Mann-Whitney-Test berechnet und in der Grafik entsprechend dargestellt, p < 0,05 wurde als signifikant angesehen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Repräsentative Bilder aus der Fragmentierungsanalyse. Die Fragmentierung jeder einzelnen Probe 1–15 ist in den Siebbandspuren dargestellt. Für jede Probe (n = 5) wurden unterschiedliche DNA-Mengen aus 20 μm, 50 μm und 80 μm Schnitten separat analysiert (n = 3 für jede Probe) (A). (B) zeigt die DNA-Fragmentierung einer repräsentativen Probe für 20 μm, 50 μm und 80 μm Abschnitte für alle drei Kits. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 5: Fragmentverteilung durch Screen-Tape-Analyse. (A) zeigt die Verteilung von kurzen, mittleren und langen Fragmenten innerhalb der Proben, die mit verschiedenen Kits isoliert wurden. (B) zeigt den maximalen Fragmentierungspeak der DNA, die aus den Proben 1 bis 5 isoliert wurde, die für die einzelnen Kits aufgetragen wurden. Für jedes Kit werden die 20 μm, 50 μm und 80 μm großen Schnitte jeder Probe gepoolt. p-Werte werden mit einem gepaarten Mehrfach-t-Test berechnet und in der Grafik dargestellt, p < 0,05 wurde als signifikant angesehen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 6: Fragmentierungsanalyse mit PCR. Diese Gelelektrophorese zeigt die PCR-Produkte mit unterschiedlichen Amplikonlängen des Beta-Aktin-Housekeeping-Gens für acht verschiedene Proben und eine Kontrollzelllinie. Die Fahrspuren werden wie folgt benannt: 1 = 100 bp, 2 = 200 bp, 3 = 300 bp, 4 = 400 bp, 5 = 500 bp, 6 = 600 bp, 7 = 700 bp, 8 = 800 bp, 9 = 1.000 bp und 10 = 1.300 bp. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Bausatz A | Bausatz B | Bausatz C | ||
| Spektrometer | Maximale Konzentration [ng/μl] | 26.2 | 13.25 | 72.35 |
| Min. Konzentration [ng/μl] | 8.45 | 7.75 | 23.55 | |
| mittlere Konzentration [ng/μl] | 15.96 | 10.89 | 47.72 | |
| Mikroplatten-Reader | Maximale Konzentration [ng/μl] | 24 | 13.9 | 56.2 |
| Min. Konzentration [ng/μl] | 5.2 | 0 | 18.6 | |
| mittlere Konzentration [ng/μl] | 12.5 | 7.4 | 41.5 | |
| Fluorometer | Maximale Konzentration [ng/μl] | 10.3 | 23.4 | 47.6 |
| Min. Konzentration [ng/μl] | 0.211 | 0.842 | 12.5 | |
| mittlere Konzentration [ng/μl] | 3.369 | 14.64 | 33.1 | |
Tabelle 1: Ergebnisse der quantitativen Analyse. Diese Tabelle zeigt die minimale, maximale und mittlere Konzentration der DNA, die von den fünf Patienten mit den drei verschiedenen Kits isoliert wurde. Die Konzentrationen wurden spektrometrisch mit Mikrospektrometer und Mikroplatten-Reader sowie fluorometrisch gemessen.
| Probe | μm | Bausatz | Mikroplatte 260/280 | Spektrometer 260/280 | DV200 [%] |
| Patient 1 | 20 | Bausatz A | 6 | 1.65 | 90.72 |
| Patient 1 | 50 | Bausatz A | 1.8 | 1.71 | 90 |
| Patient 1 | 80 | Bausatz A | 2.09 | 1.345 | 88.98 |
| Patient 2 | 20 | Bausatz A | 1.88 | 1.53 | 87.14 |
| Patient 2 | 50 | Bausatz A | 2.29 | 1.465 | 92.46 |
| Patient 2 | 80 | Bausatz A | 4.36 | 2.68 | 84.78 |
| Patient 3 | 20 | Bausatz A | 1.91 | 1.85 | 89.24 |
| Patient 3 | 50 | Bausatz A | 2.21 | 1.92 | 90.86 |
| Patient 3 | 80 | Bausatz A | 2.06 | 1.675 | 94.59 |
| Patient 4 | 20 | Bausatz A | 2.27 | 1.86 | 95.72 |
| Patient 4 | 50 | Bausatz A | nicht nachweisbar | 3.385 | 87.13 |
| Patient 4 | 80 | Bausatz A | 2.33 | 1.905 | 95.64 |
| Patient 5 | 20 | Bausatz A | 1.6 | 1.51 | 82.87 |
| Patient 5 | 50 | Bausatz A | 1.79 | 1.525 | 91.76 |
| Patient 5 | 80 | Bausatz A | 3.47 | 2.765 | nicht nachweisbar |
| Patient 1 | 20 | Bausatz B | 1.88 | nicht nachweisbar | 80.23 |
| Patient 1 | 50 | Bausatz B | 0.73 | 1.43 | 75.72 |
| Patient 1 | 80 | Bausatz B | 1.44 | 1.585 | 75.64 |
| Patient 2 | 20 | Bausatz B | 2.63 | 1.45 | 79.75 |
| Patient 2 | 50 | Bausatz B | 1.92 | 1.765 | 87.14 |
| Patient 2 | 80 | Bausatz B | 1.56 | 1.52 | 91.01 |
| Patient 3 | 20 | Bausatz B | nicht nachweisbar | 1.335 | 78.00 |
| Patient 3 | 50 | Bausatz B | 2.28 | 1.63 | 83.21 |
| Patient 3 | 80 | Bausatz B | 2 | 1.67 | 78.61 |
| Patient 4 | 20 | Bausatz B | 0.59 | 0.625 | 94.37 |
| Patient 4 | 50 | Bausatz B | 2.29 | 1.78 | 92.97 |
| Patient 4 | 80 | Bausatz B | nicht nachweisbar | nicht nachweisbar | nicht nachweisbar |
| Patient 5 | 20 | Bausatz B | 2.63 | 1.41 | 83.15 |
| Patient 5 | 50 | Bausatz B | nicht nachweisbar | 1.005 | 92.06 |
| Patient 5 | 80 | Bausatz B | 2.21 | 1.69 | 94.51 |
| Patient 1 | 20 | Bausatz C | 1.69 | 1.495 | 90.73 |
| Patient 1 | 50 | Bausatz C | 1.72 | 1.555 | 94.43 |
| Patient 1 | 80 | Bausatz C | 1.72 | 1.565 | 88.71 |
| Patient 2 | 20 | Bausatz C | 1.64 | 1.4 | 92.75 |
| Patient 2 | 50 | Bausatz C | 1.63 | 1.455 | 93.26 |
| Patient 2 | 80 | Bausatz C | 1.72 | 1.51 | 94.34 |
| Patient 3 | 20 | Bausatz C | 1.63 | 1.4 | 91.78 |
| Patient 3 | 50 | Bausatz C | 1.7 | 1.505 | 93.61 |
| Patient 3 | 80 | Bausatz C | 1.74 | 1.595 | 94.61 |
| Patient 4 | 20 | Bausatz C | 1.62 | 1.36 | 92.89 |
| Patient 4 | 50 | Bausatz C | 1.71 | 1.495 | 94.2 |
| Patient 4 | 80 | Bausatz C | 1.76 | 1.575 | 95.17 |
| Patient 5 | 20 | Bausatz C | 1.57 | 1.36 | 80.55 |
| Patient 5 | 50 | Bausatz C | 1.66 | 1.455 | 92.65 |
| Patient 5 | 80 | Ki t C | 1.69 | 1.45 | 90.29 |
Tabelle 2: Ergebnisse der qualitativen Analyse. Die mit Mikrospektrometer und Mikroplatten-Reader berechneten A260/280-Verhältnisse werden für jede Probe (n = 15) und die drei verschiedenen Extraktionsmethoden angezeigt. DV200 (Fragmente größer als 200 bp), die über die Fragmentierungsanalyse berechnet wurden, wird auch für die Kits A, B und C angezeigt.
| Probe | μm | Bausatz | Fragmente % <200bp | Fragmente % 200-1500 bp | Fragmente % >1500bp | Maximale Spitze der Fragmente (bp) |
| Patient 1 | 20 | Bausatz A | 9.04 | 77.08 | 13.64 | 484 |
| Patient 1 | 50 | Bausatz A | 10.14 | 77.03 | 12.97 | 378 |
| Patient 1 | 80 | Bausatz A | 10.91 | 76.56 | 12.42 | 393 |
| Patient 2 | 20 | Bausatz A | 11.01 | 67.04 | 20.1 | 589 |
| Patient 2 | 50 | Bausatz A | 6.09 | 68.67 | 23.79 | 628 |
| Patient 2 | 80 | Bausatz A | 11.54 | 66.07 | 18.71 | 560 |
| Patient 3 | 20 | Bausatz A | 10.22 | 77.29 | 11.95 | 412 |
| Patient 3 | 50 | Bausatz A | 8.52 | 76.93 | 13.93 | 465 |
| Patient 3 | 80 | Bausatz A | 4.79 | 77.5 | 17.09 | 580 |
| Patient 4 | 20 | Bausatz A | 2.7 | 55.77 | 39.95 | 1331 |
| Patient 4 | 50 | Bausatz A | 7.6 | 54.3 | 32.83 | 1183 |
| Patient 4 | 80 | Bausatz A | 2.5 | 61.06 | 34.58 | 1185 |
| Patient 5 | 20 | Bausatz A | 12.32 | 36.52 | 46.35 | 1895 |
| Patient 5 | 50 | Bausatz A | 5.34 | 36.37 | 55.39 | 1856 |
| Patient 5 | 80 | Bausatz A | nicht nachweisbar | nicht nachweisbar | nicht nachweisbar | nicht nachweisbar |
| Patient 1 | 20 | Bausatz B | 18.51 | 69.28 | 10.95 | 283 |
| Patient 1 | 50 | Bausatz B | 23.58 | 65.24 | 10.48 | 254 |
| Patient 1 | 80 | Bausatz B | 22.42 | 67.14 | 8.5 | 257 |
| Patient 2 | 20 | Bausatz B | 16.21 | 59.88 | 19.87 | 523 |
| Patient 2 | 50 | Bausatz B | 9.39 | 63.7 | 23.44 | 678 |
| Patient 2 | 80 | Bausatz B | 7.45 | 67.62 | 23.39 | 666 |
| Patient 3 | 20 | Bausatz B | 18.53 | 65.15 | 12.85 | 370 |
| Patient 3 | 50 | Bausatz B | 15.96 | 72.12 | 11.09 | 316 |
| Patient 3 | 80 | Bausatz B | 20.7 | 71.49 | 7.12 | 266 |
| Patient 4 | 20 | Bausatz B | 3.09 | 57.44 | 36.93 | 1296 |
| Patient 4 | 50 | Bausatz B | 3.69 | 52.2 | 40.77 | 1373 |
| Patient 4 | 80 | Bausatz B | nicht nachweisbar | nicht nachweisbar | nicht nachweisbar | nicht nachweisbar |
| Patient 5 | 20 | Bausatz B | 9.86 | 34.96 | 48.19 | 1831 |
| Patient 5 | 50 | Bausatz B | 3.76 | 31.42 | 60.64 | 2002 |
| Patient 5 | 80 | Bausatz B | 1.93 | 33.24 | 61.27 | 2015 |
| Patient 1 | 20 | Bausatz C | 8.88 | 81.47 | 9.26 | 358 |
| Patient 1 | 50 | Bausatz C | 5.92 | 83.95 | 10.48 | 374 |
| Patient 1 | 80 | Bausatz C | 11.02 | 80.18 | 8.53 | 334 |
| Patient 2 | 20 | Bausatz C | 5.18 | 74.75 | 18 | 492 |
| Patient 2 | 50 | Bausatz C | 4.96 | 75.67 | 17.59 | 458 |
| Patient 2 | 80 | Bausatz C | 3.14 | 72.46 | 21.88 | 441 |
| Patient 3 | 20 | Bausatz C | 7.15 | 79.05 | 12.73 | 393 |
| Patient 3 | 50 | Bausatz C | 5.85 | 78.02 | 15.59 | 365 |
| Patient 3 | 80 | Bausatz C | 2.59 | 68.54 | 26.07 | 458 |
| Patient 4 | 20 | Bausatz C | 3.94 | 54.49 | 38.4 | 1433 |
| Patient 4 | 50 | Bausatz C | 1.42 | 45.24 | 48.96 | 1634 |
| Patient 4 | 80 | Bausatz C | 0.76 | 37.03 | 58.14 | 1849 |
| Patient 5 | 20 | Bausatz C | 13.02 | 40.78 | 39.77 | nicht nachweisbar |
| Patient 5 | 50 | Bausatz C | 2.72 | 39.08 | 53.57 | 1813 |
| Patient 5 | 80 | Ki t C | 5.25 | 31.87 | 58.42 | 2024 |
Tabelle 3: Ergebnisse der Fragmentierungsanalyse. Hier wird für alle fünf Patienten der prozentuale Anteil der Fragmente kleiner als 200 bp, größer als 1.500 bp und zwischen 200 bp und 1.500 bp dargestellt, die unterschiedlichen Mengen an Ausgangsmaterial (20 μm, 50 μm und 80 μm) und die drei verschiedenen Isolationsmethoden. Prozentsätze unter 2 % wurden als nicht nachweisbar bezeichnet.
Diskussion
DNA-Extraktionsmethoden für FFPE-Gewebe variieren in Qualität und Quantität der isolierten DNA, was sich zwangsläufig auf die Leistung weiterer nachgelagerter Analysen auswirkt. Daher wird die Automatisierung immer wichtiger, um den Workflow und die Standardisierung sowie das Qualitätsmanagement zu verbessern. Daher wurde in der vorliegenden Studie eine halbautomatische Methode zur DNA-Extraktion aus FFPE-Proben evaluiert, die bessere Ergebnisse als die anderen getesteten manuellen säulenbasierten Protokolle zeigte.
Um die beschriebene halbautomatische Methode zu optimieren, war es unser Ziel, ein allgemeines Protokoll für jede Art von Gewebe und Körperflüssigkeit sowie fixierte Proben zu haben. Daher haben wir zur Optimierung das kommerziell erhältliche DNA Blood Kit der Firma (Table of Materials) verwendet. Zunächst haben wir einen Inkubationsschritt zur Entparaffinierung eingebaut, wobei der handelsübliche Inkubationspuffer und 1-Thioglycerin des gleichen Herstellers verwendet wurden. In verschiedenen Tests bezüglich der Inkubationszeit und der optimalen Schmelztemperatur des Paraffins haben wir uns für einen 10-minütigen Inkubationsschritt bei 80 °C entschieden, wie er auch in anderen kommerziell erhältlichen FFPE-Kits vorgesehen ist. Hinsichtlich der Anzahl der FFPE-Scheiben empfehlen die meisten Unternehmen 2 μm x 10 μm Scheiben für die DNA-Extraktion. Wie in dieser Größenordnung bekannt, ist es äußerst schwierig, DNA aus Proben zu isolieren, die nur eine geringe Zellmenge enthalten. Daher probierten wir verschiedene Mengen an fixierten Gewebeschnitten aus und stellten fest, dass die Verwendung von insgesamt 50 μm Gewebe (am besten in Einzelschnitten von 5 μm bis 10 μm) die besten Ergebnisse zeigte. Dies ist auf jeden Fall für Gewebe mit geringen Zellmengen geeignet, aber auch Gewebe mit höheren Zellanteilen kann so behandelt werden. Insbesondere säulenbasierte Systeme zeigen hier eine hohe Limitierung mit Verstopfung der Silica-Membran. Es wurde vermutet, dass der Proteinase-K-Verdau zwischen 4 und 20 Stunden dauert. Hier fanden wir heraus, dass eine Inkubationszeit von 4–6 Stunden zwar in Ordnung ist, aber die Inkubation über Nacht zu einer ausreichenderen Qualität und Quantität der DNA führt. Für eine bessere Zelllyse erschien es auch hilfreich, am nächsten Tag weitere 10 μL Proteinase K hinzuzufügen und erneut 1 h zu inkubieren. Als Schüttel-/Mischgeschwindigkeit wählten wir mehr als die empfohlenen 300 U/min, da wir gesehen haben, dass ein stärkeres Mischen (500 U/min – 600 U/min) zu einer besseren Proteinase-K-Verdauung und damit zu höheren DNA-Mengen führt. Auch zusätzliche Mischschritte (wie Vortexen oder Schütteln bei voller Geschwindigkeit) während der Inkubation führen zu einer besseren DNA-Extraktion. Im Gegensatz zum ursprünglichen FFPE-Protokoll wählten wir eine weitere Inkubationszeit nach Zugabe von Lysepuffer zur Probe, wobei die Lyse bei 65 °C ausreichender war. Der letzte Optimierungsschritt des Protokolls war die Zugabe eines weiteren Inkubationspuffers. Das System benötigt mindestens 60 μl Elutionspuffer. Da das automatisierte System dafür bekannt ist, 5 μl bis 10 μl zu verlieren, haben wir uns für ein Elutionsvolumen von 65 μl entschieden.
Dieses Protokoll enthält einige wichtige Schritte, die zu beachten sind. Die erste besteht darin, die richtige Temperatur einzustellen, um das Paraffin zu lösen. 80 °C scheinen die beste Temperatur für ein optimales Schmelzen zu sein. Es ist auch wichtig, die Probe vor der Zugabe von Proteinase K auf 60 °C abzukühlen, um einen Abbau dieses Enzyms zu verhindern. Als nächstes ist es wichtig, die Probe nach der 30-minütigen Inkubationszeit mit Lysepuffer nicht erneut zu wirbeln, da das Paraffin oben aushärten muss. Andernfalls wird die Probe mit Paraffin kontaminiert. Als letzten Schritt ist es auch wichtig, etwas zusätzliches Elutionspuffervolumen hinzuzufügen, da das System 5 μl bis 10 μl verliert. Wir denken, dass 65 μl Elutionspuffer eine gute Menge sind. Werden höhere Mengen an DNA erwartet, kann das Volumen natürlich erhöht werden.
Die optimierte halbautomatische Isolierungsmethode lieferte die höchsten Konzentrationen an genomischer Gesamt-DNA in allen FFPE-Proben. Bei gleicher Menge an Elutionsvolumen war auch die Gesamtausbeute an DNA viermal höher. Darüber hinaus zeigten die säulenbasierten Kits keine Steigerung der DNA-Ausbeute bei dickeren Gewebeschnitten, während das automatisierte System einen linearen Anstieg der DNA-Konzentration in Verbindung mit der erhöhten Menge an verwendetem Gewebe zeigte. Diese Unterschiede lassen sich vor allem durch die Paraffinverstopfung in den Säulen und die daraus resultierende Abnahme der DNA-Bindungskapazität erklären. Unsere Daten deuten auch darauf hin, dass eine größere Menge an FFPE-Gewebe, in unserem Fall 80 μm-Schnitte, bei der Isolierung von DNA mit dem halbautomatischen System nicht erforderlich ist. Wir konnten zeigen, dass bereits eine kleinere Menge (20 μm Schnitte) zu einer ausreichenden Ausbeute an DNA führt. So kann wertvolles Gewebematerial eingespart werden. Darüber hinaus erfüllte DNA, die mit dem halbautomatischen Protokoll extrahiert wurde, in den meisten Fällen das erforderliche Reinheitskriterium für qualitativ hochwertige DNA (A260/280-Verhältnis zwischen 1,6 und 1,8). Im Gegensatz dazu zeigte DNA, die aus säulenbasierten Isolationssystemen gewonnen wurde, große Schwankungen in der DNA-Reinheit und weniger Proben entsprachen dem erwähnten optimalen Verhältnis.
Sarnecka et al. haben kürzlich beschrieben, dass ein auf magnetischen Teilchen basierendes System ausreichender ist als säulenbasierte Systeme12. Khokhar und Kollegen konnten zeigen, dass das automatisierte System, das auf einem anderen Protokoll basiert, für kleinere Amplikons ausreichender und in Bezug auf Menge und Reinheit der Extrakte im Vergleich zu anderen Methoden weitgehend robust ist36. Unsere Proben bestätigten diese Ergebnisse und zeigten eine hohe Verteilung im A260/280-Verhältnis der DNA, die von den anderen Kits isoliert wurde.
In Bezug auf die Fragmentierungsanalyse wurden acht DNA-Extrakte, die mit diesen drei kommerziellen Methoden gewonnen wurden, für die PCR-Amplifikation verwendet. Unterschiedliche Fragmentlängen eines Housekeeping-Gens (Beta-Aktin) wurden amplifiziert und mittels Gelelektrophorese sichtbar gemacht. Es gab nur geringe Unterschiede zwischen den einzelnen Proben, die darauf zurückzuführen sind, dass alle Gewebe ein ähnliches Stadium der Atherosklerose aufweisen. Diese Vermutung wurde durch die Histologie bestätigt (Daten nicht gezeigt). Darüber hinaus zeigen diese Daten, dass alle Proben auf die gleiche Standardweise mit Formalin behandelt wurden. Es wurden keine signifikanten Unterschiede in Bezug auf die Fragmentlänge beobachtet, die aus den verschiedenen Proben unabhängig von der für die DNA-Extraktion verwendeten Methode gewonnen wurde. Die Fragmentierung von FFPE-Gewebe hängt also nicht von der Methode der DNA-Isolierung ab, sondern wird durch die Variabilität der menschlichen Proben und die Formalinbehandlung beeinflusst.
Da das verwendete halbautomatische System als Magnetpulver-Mover, nicht als Liquid Handler bezeichnet wird, bietet es auch einige Vorteile gegenüber anderen halbautomatischen Systemen. Der Hauptvorteil ist das minimale Risiko einer Kreuzkontamination, da während der Probenverarbeitung kein Liquid Handling oder Spritzen auftritt. Ein weiterer Punkt, den wir erwähnen möchten, ist die hohe Anzahl an Proben, die in einem Durchgang des Systems verarbeitet werden können, ohne viel mehr Hands-on-Zeit zu haben. Der Einsatz eines halbautomatischen Systems führt zudem zu einer geringeren Wahrscheinlichkeit von Anwenderfehlern als bei den säulenbasierten oder Phasentrennverfahren. Insbesondere für Biobanking-Analysen und diagnostische Arbeiten sowie für den Umgang mit schwierigen Geweben ist die oben beschriebene halbautomatische Methode sehr zu empfehlen. In diesen Bereichen ist es sehr wichtig, dass verschiedene Mitarbeiter und manchmal auch unerfahrene Personen zu schnellen, einfachen und reproduzierbaren Ergebnissen kommen. Weitere Vorteile des Systems sind das allgemein anwendbare Protokoll, das sowohl für jede Art von Gewebe als auch für Blut, andere Körperflüssigkeiten und auch Zellen mit nur einer geringfügigen Änderung im ersten Schritt des Protokolls verwendet werden kann, bei dem kein Inkubationspuffer benötigt wird.
Auch wenn unsere Ergebnisse die Überlegenheit des automatisierten Systems zur DNA-Extraktion aus FFPE gegenüber den manuellen Protokollen deutlich gezeigt haben, ist es erwähnenswert, dass wir nur drei kommerziell erhältliche Kits verglichen haben. Unsere Schlussfolgerung bezieht sich daher nur auf die in unserer Studie getesteten Methoden. Darüber hinaus war die Anzahl der Proben gering und wir verwendeten nur atherosklerotische Plaques zum Vergleich. Um die Ergebnisse unserer Studie zu verallgemeinern, wären weitere Gewebeproben in größerer Zahl notwendig.
Nichtsdestotrotz scheint das automatisierte System einen erheblichen Vorteil gegenüber den manuellen Kits zu haben, insbesondere für Proben mit einer geringen Menge an zu erwartenden Nukleinsäuren. Auch das System selbst hat gewisse Einschränkungen. Zum Beispiel ist es, wie oben erwähnt, notwendig, mindestens 60 μl für die Elution zu verwenden, da der Kolben diese Menge benötigt, um die DNA zu eluieren. Außerdem ist der Verlust des Elutionsvolumens bei den halbautomatischen Systemen höher als bei den säulenbasierten Systemen. Die automatisierte Methode hat einen Verlust von etwa 10 μl Elutionsvolumen, während die manuellen Methoden nur einen Verlust von 5 μl aufweisen. Auch die höheren Kosten müssen erwähnt werden. Für Analysen mit hohem Durchsatz bietet das halbautomatische System jedoch immer noch den besten Nutzen im Vergleich zu den Kosten. Als letzte Einschränkung des Systems ist es wichtig zu beachten, dass es sich bei der automatisierten und halbautomatischen Extraktion um ein in sich geschlossenes geschlossenes System handelt. Es ist natürlich möglich, z.B. DNA und Protein, oder auch RNA und DNA aus einer Probe zu isolieren, aber es ist nicht so einfach und wird oft mit manuellen Phasentrennungsmethoden verwendet. Alle Puffer sind an das Kit angepasst und können im Gegensatz zu Trizol-Reagenzien nicht für andere Methoden verwendet werden. Zum Schluss ist es auch wichtig zu erwähnen, dass DNA aus FFPE-Proben im Allgemeinen nicht für die Sequenzierung des gesamten Genoms geeignet ist, da die Fragmentierung der Nukleinsäuren zu stark ist.
Zusammenfassend zeigen wir in dieser Studie, dass eine vollautomatische DNA-Isolierungsmethode ein hervorragendes Werkzeug sein könnte, um die Hands-on-Zeit und die Menge an Ausgangsmaterial zu reduzieren. Außerdem scheint es die beste Wahl zu sein, um das Kontaminationsrisiko zwischen den Proben zu reduzieren. Darüber hinaus ermöglicht die Flexibilität des automatisierten Systems die Verarbeitung nicht nur einer, sondern bis zu 48 Proben pro Lauf, was diese Methode zu einem nützlichen Werkzeug für zukünftige Hochdurchsatzanalysen von Proben mit geringer Zellzahl und damit geringer Menge an Nukleinsäuren macht. Die Qualität und Quantität der isolierten DNA mit dem automatisierten System scheint anderen manuellen Kits überlegen zu sein, und auch der Einsatz in weiteren Analysen wie Southern Blot, Einzelnukleotid-Genotypisierung, Klonierungsanalysen, Methylierungsassays sowie PCR-Amplifikation ist möglich.
Offenlegungen
Die Autoren erklären, dass kein Interessenkonflikt besteht.
Danksagungen
Die Etablierung des Protokolls für die automatisierte DNA-Extraktion wurde von Dr. Paul Muschler von der Firma Promega unterstützt. Wir danken Paul Muschler für seine Unterstützung und seinen wissenschaftlichen Beitrag. Wir danken auch unserem Kollegen Dr. Moritz von Scheidt (Deutsches Herzzentrum München) für die Bereitstellung des Maxwell-Instruments und die Unterstützung des experimentellen Teils. Alle Experimente wurden in den Laboren des Deutschen Herzzentrums (München, Deutschland) und des Klinikums rechts der Isar (München, Deutschland) durchgeführt. Die Forschung wurde von der DFG gefördert (PE 900/6-1).
Materialien
| Name | Company | Catalog Number | Comments |
| 1.5 ml tubes for sample incubation | Eppendorf, Hamburg, Germany | 30120086 | |
| 1-Thioglycerol | Promega, Walldorf, Germany | A208 | |
| Agilent tape station software 3.2 | Agilent, Waldbronn, Germany | ||
| dsDNA HS Kit | ThermoFisher Scientific, Schwerte, Germany | Q32851 | |
| FFPE DNA Purification Kits (Kit A) | Norgene Biotek, Heidelberg, Germany | 47400 | |
| FFPE tissue samples n=5 | Munich Vascular Biobank,Munich, Germany | ||
| GeneRead DNA FFPE Kit (Kit B) | Qiagen, Hilden, Germany | 180134 | |
| Heating blocks, set to 80°C and 65°C | VWR,Darmstadt,Germany | 460-0250 | |
| High Sensitivity D5000 reagents | Agilent, Waldbronn, Germany | 5067-5593 | |
| High Sensitivity D5000 ScreenTape | Agilent, Waldbronn, Germany | 5067-5592 | |
| Incubation Buffer | Promega, Walldorf, Germany | D920 | |
| Maxwell Blood Kit RSC including: Lysis Buffer, Elution Buffer, Proteinase K | Promega, Walldorf, Germany | AS1400 | |
| Maxwell RSC 48 Instrument | Promega, Walldorf, Germany | AS8500 | |
| Microcentrifuge | Eppendorf, Hamburg, Germany | ||
| NanoDrop 2000c Spectrometer | ThermoFisher Scientific, Schwerte, Germany | ND-2000C | |
| Optical caps | Agilent, Waldbronn, Germany | 401425 | |
| Optical tube strips | Agilent, Waldbronn, Germany | 401428 | |
| Pipettors and pipette tips | Eppendorf, Hamburg, Germany | ||
| Prism 6 for statistics, version 6.01 | GraphPad Inc., San Diego, California | ||
| Qubit 3.0 Fluorometer | ThermoFisher Scientific, Schwerte, Germany | Q33216 | |
| TapeStation 4200 | Agilent, Waldbronn, Germany | ||
| Tecan Infinite M200 Pro | Tecan, Männedorf, Swizerland | IN-MNANO |
Referenzen
- Pelisek, J., et al. Biobanking: objectives, requirements, and future challenges-experiences from the munich vascular biobank. Journal of Clinical Medicine. 8 (2), 251(2019).
- Goelz, S. E., Hamilton, S. R., Vogelstein, B. Purification of DNA from formaldehyde fixed and paraffin embedded human tissue. Biochemical and Biophysical Research Communications. 130 (1), 118-126 (1985).
- Busch, A., Eken, S. M., Maegdefessel, L. Prospective and therapeutic screening value of non-coding RNA as biomarkers in cardiovascular disease. Annals of Translational Medicine. 4 (12), (2016).
- Kandpal, R. P., Saviola, B., Felton, J. The era of 'omics unlimited. BioTechniques. 46 (5), 351-355 (2009).
- McDonough, S. J., et al. Use of FFPE-derived DNA in next generation sequencing: DNA extraction methods. PloS One. 14 (4), (2019).
- Gilbert, M. T. P., et al. The isolation of nucleic acids from fixed, paraffin-embedded tissues-which methods are useful when. PloS One. 2 (6), (2007).
- Williams, C., et al. A high frequency of sequence alterations is due to formalin fixation of archival specimens. The American Journal of Pathology. 155 (5), 1467-1471 (1999).
- Solomon, M. J., Varshavsky, A. Formaldehyde-mediated DNA-protein crosslinking: a probe for in vivo chromatin structures. Proceedings of the National Academy of Sciences. 82 (19), 6470-6474 (1985).
- Gillio-Tos, A., et al. Efficient DNA extraction from 25-year-old paraffin-embedded tissues: study of 365 samples. Pathology. 39 (3), 345-348 (2007).
- Srinivasan, M., Sedmak, D., Jewell, S. Effect of fixatives and tissue processing on the content and integrity of nucleic acids. The American Journal of Pathology. 161 (6), 1961-1971 (2002).
- van Eijk, R., Stevens, L., Morreau, H., van Wezel, T. Assessment of a fully automated high-throughput DNA extraction method from formalin-fixed, paraffin-embedded tissue for KRAS, and BRAF somatic mutation analysis. Experimental and Molecular Pathology. 94 (1), 121-125 (2013).
- Sarnecka, A. K., et al. DNA extraction from FFPE tissue samples–a comparison of three procedures. Contemporary Oncology. 23 (1), 52(2019).
- Kalmár, A., et al. Comparison of automated and manual DNA isolation methods for DNA methylation analysis of biopsy, fresh frozen, and formalin-fixed, paraffin-embedded colorectal cancer samples. Journal of Laboratory Automation. 20 (6), 642-651 (2015).
- McCormick, S. F. Multilevel adaptive methods for partial differential equations. (SIAM). , (1989).
- Sambrook, J., Russell, D. W. Purification of nucleic acids by extraction with phenol: chloroform. Cold Spring Harbor Protocols. 2006 (1), 4455(2006).
- Berensmeier, S. Magnetic particles for the separation and purification of nucleic acids. Applied Microbiology and Biotechnology. 73 (3), 495-504 (2006).
- Petrigh, R. S., Fugassa, M. H. DNA extraction and a cost-effective detection method for Echinococcus granulosus protoscoleces. Veterinary Parasitology. 198 (3-4), 410-413 (2013).
- Lee, J. H., Park, Y., Choi, J. R., Lee, E. K., Kim, H. S. Comparisons of three automated systems for genomic DNA extraction in a clinical diagnostic laboratory. Yonsei Medical Journal. 51 (1), 104-110 (2010).
- Rohland, N., Siedel, H., Hofreiter, M. A rapid column-based ancient DNA extraction method for increased sample throughput. Molecular Ecology Resources. 10 (4), 677-683 (2010).
- Gutiérrez-López, R., Martínez-de la Puente, J., Gangoso, L., Soriguer, R. C., Figuerola, J. Comparison of manual and semi-automatic DNA extraction protocols for the barcoding characterization of hematophagous louse flies (Diptera: Hippoboscidae). Journal of Vector Ecology: Journal of the Society for Vector Ecology. 40 (1), 11-15 (2015).
- Seiler, C., et al. Nucleic acid extraction from formalin-fixed paraffin-embedded cancer cell line samples: a trade off between quantity and quality. BMC Clinical Pathology. 16 (1), 17(2016).
- Harada, S. Sample Preparation Techniques for Soil, Plant, and Animal Samples. , Springer. 125-138 (2016).
- Haile, S., et al. Automated high throughput nucleic acid purification from formalin-fixed paraffin-embedded tissue samples for next generation sequence analysis. PloS One. 12 (6), 0178706(2017).
- Fujii, T., et al. Evaluation of DNA and RNA quality from archival formalin-fixed paraffin-embedded tissue for next-generation sequencing-Retrospective study in Japanese single institution. Pathology International. , (2020).
- Ludyga, N., et al. Nucleic acids from long-term preserved FFPE tissues are suitable for downstream analyses. Virchows Archiv: An International Journal of Pathology. 460 (2), 131-140 (2012).
- Niland, E. E., McGuire, A., Cox, M. H., Sandusky, G. E. High quality DNA obtained with an automated DNA extraction method with 70+ year old formalin-fixed celloidin-embedded (FFCE) blocks from the indiana medical history museum. American Journal of Translational Research. 4 (2), 198(2012).
- Riemann, K., et al. Comparison of manual and automated nucleic acid extraction from whole-blood samples. Journal of Clinical Laboratory Analysis. 21 (4), 244-248 (2007).
- Steinau, M., Patel, S. S., Unger, E. R. Efficient DNA extraction for HPV genotyping in formalin-fixed, paraffin-embedded tissues. The Journal of Molecular Diagnostics. 13 (4), 377-381 (2011).
- Dundas, N., Leos, N. K., Mitui, M., Revell, P., Rogers, B. B. Comparison of automated nucleic acid extraction methods with manual extraction. The Journal of Molecular Diagnostics. 10 (4), 311-316 (2008).
- Okello, J. B., et al. Comparison of methods in the recovery of nucleic acids from archival formalin-fixed paraffin-embedded autopsy tissues. Analytical Biochemistry. 400 (1), 110-117 (2010).
- Witt, S., Neumann, J., Zierdt, H., Gébel, G., Röscheisen, C. Establishing a novel automated magnetic bead-based method for the extraction of DNA from a variety of forensic samples. Forensic Science International: Genetics. 6 (5), 539-547 (2012).
- Ivanova, N. V., Fazekas, A. J., Hebert, P. D. Semi-automated, membrane-based protocol for DNA isolation from plants. Plant Molecular Biology Reporter. 26 (3), 186(2008).
- Sengüven, B., Baris, E., Oygur, T., Berktas, M. Comparison of methods for the extraction of DNA from formalin-fixed, paraffin-embedded archival tissues. International Journal of Medical Sciences. 11 (5), 494(2014).
- Miller, S. A., Dykes, D. D., Polesky, H. F. A simple salting out procedure for extracting DNA from human nucleated cells. Nucleic Acids Research. 16 (3), 1215(1988).
- Wilfinger, W. W., Mackey, K., Chomczynski, P. Effect of pH and ionic strength on the spectrophotometric assessment of nucleic acid purity. BioTechniques. 22 (3), 474-481 (1997).
- Khokhar, S. K., Mitui, M., Leos, N. K., Rogers, B. B., Park, J. Y. Evaluation of Maxwell 16 for automated DNA extraction from whole blood and formalin-fixed paraffin embedded (FFPE) tissue. Clinical Chemistry and Laboratory Medicine (CCLM). 50 (2), 267-272 (2012).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten