Method Article
Adsorption de spores comme un système d’affichage recombinants pour les Enzymes et les antigènes
Dans cet article
Résumé
Ce protocole porte sur l’utilisation des spores bactériennes comme un outil de nanobiotechnological « en direct » pour adsorber les molécules hétérologues avec diverses activités biologiques. Les méthodes pour mesurer l’efficacité d’adsorption sont également indiqués.
Résumé
Les spores bactériennes est une cellule métaboliquement quiescente, formée par une série de couches protectrices entourant un cytoplasme déshydraté. Cette structure particulière rend la spore extrêmement stable et résistant et a suggéré l’utilisation de la spore comme une plate-forme pour afficher des molécules hétérologues. Jusqu’ici, une variété d’antigènes et d’enzymes ont été affichés sur les spores de Bacillus subtilis et de quelques autres espèces, tout d’abord par une approche recombinante et, ensuite, par une méthode simple et efficace de recombinants. Le système d’affichage recombinants repose sur l’adsorption directe de hétérologue molécules sur la surface des spores, en évitant la construction de souches recombinantes et la libération des bactéries génétiquement modifiés dans l’environnement. Molécules adsorbées sont stabilisées et protégés par l’interaction avec les spores, qui limite la dégradation rapide des antigènes et la perte de l’activité enzymatique à des conditions défavorables. Une fois utilisé, spore-adsorbé enzymes peuvent être collectées facilement avec une réduction minimale de l’activité et réutilisés pour tours de réaction supplémentaire. Dans le présent document est montré comment s’adsorber les molécules modèles à spores purifiées de b. subtilis, comment évaluer l’efficacité d’adsorption et la collecte des spores usagés pour les recycler pour nouvelles réactions.
Introduction
Systèmes d’affichage visent à présenter des molécules biologiquement actives sur la surface des micro-organismes et de trouver des applications dans une variété de domaines, des industriels aux biotechnologies médicales et écologiques. En plus des phages1,2 et cellules de divers Gram - positifs et espèces3,4,5,6,7, spores bactériennes ont également été proposé comme systèmes d’affichage par deux approches8,9.
En raison de sa structure particulière, à savoir un cytoplasme déshydraté, entouré par une série de couches protectrices10, la spore offre plusieurs avantages par rapport aux phages et cellule dotés d’écran systèmes8,9. Un premier avantage provient de l’extrême robustesse et stabilité des spores dans des conditions qui seraient préjudiciables à tous les autres cellules10,11. Enzymes et spore-affiche les antigènes sont stables après une conservation à température ambiante12 et protégée de la dégradation à pH faible et des températures élevées,13. Un deuxième avantage des spores est la sécurité de nombreuses espèces sporulées. B. subtilis, b. clausii, b. coagulanset plusieurs autres espèces sont utilisées dans le monde entier comme probiotiques et sont trouvent sur le marché à usage humain ou animal pour des décennies14,15. Cet enregistrement de sécurité exceptionnel est une évidente exigence générale pour un système d’affichage de surface et est particulièrement pertinent lorsque le système est destiné à l’usage humain ou animal,16. Un tiers, un avantage important d’un système d’affichage basé sur spore est qu’il n’a pas de limites pour la taille de la molécule qui doit être exposé. Dans les systèmes phage, une grosse protéine hétérologue peut affecter la structure de la capside, tandis que dans les systèmes basés sur les cellules, il peut affecter la structure de la membrane ou limite/qui risqueraient de la membrane translocation étape17. Les couches protectrices entourant la spore sont composent de plus de 70 différents protéines10 et sont suffisamment souples pour accepter les grosses protéines étrangères sans défaut structurel évident ou une impotence fonctionnelle8. En outre, avec les deux systèmes d’affichage axée sur les spores, la translocation de la membrane de la protéine hétérologue n’est pas requis8,9. En effet, les protéines hétérologues sont soit produites dans le cytoplasme de la cellule-mère et assemblés sur le spore qui se forme dans le même cytoplasme ou adsorbés sur les spores mûres8,9.
Affichage de spore a été initialement obtenu en développant un système génétique pour l’ingénieur de la spore surface18. Ce système génétique reposait sur i) la construction d’une fusion de gènes entre le gène codant pour une protéine de capside de spore (utilisée comme support) et le gène codant pour la protéine d’être affiché - la présence des signaux transcription et traduction de l’endogène gène contrôlera l’expression de la fusion et ii) l’intégration du gène sur le chromosome de b. subtilis d’accorder la stabilité génétique chimérique. Une variété d’antigènes et d’enzymes ont été affichées par cette approche recombinante, à l’aide de diverses protéines de surface des spores comme porteurs et visant à diverses applications possibles, allant de vaccin muqueux à biocatalyseur, biocapteurs, bioremédiation, ou bioanalytiques outil8,13.
Plus récemment, une approche différente de l’affichage de la spore a été développé19. Ce deuxième système est recombinants et repose sur l’adsorption spontanée et extrêmement serrée des molécules sur la surface de spore9. 19,d’antigènes20 et enzymes de13,21 ont été exposés avec efficacité et ont révélé que cette méthode est significativement plus efficace que celui recombinant. Cette approche non permet l’affichage des protéines dans la forme native20 et peut également être utilisée avec autoclave, mort des spores19. Le mécanisme moléculaire de l’adsorption n’a pas été entièrement précisé encore. La charge négative et l’hydrophobicité de la spore ont été proposés en tant que propriétés pertinentes pour l’adsorption13,19,22. Récemment, il a été démontré qu’une protéine de modèle, la protéine auto-fluorescente rouge (RDRF) des coraux Acropora, lorsque adsorbés à la spore, a pu s’infiltrer à travers les couches de surface localisation dans la couche intérieure de23. S’est avéré vrai pour les autres protéines, la localisation interne des protéines hétérologues pourrait expliquer leur stabilité accrue lorsque adsorbé à spores23.
Dans une étude récente, deux enzymes catalysant deux étapes successives de la voie de dégradation de xylan s’affichaient indépendamment sur les spores de b. subtilis et incubés ensemble, ont été en mesure d’effectuer les deux étapes de dégradation21. Spores recueillis après la réaction étaient encore actifs et en mesure de continuer la dégradation xylane lors de l’addition du substrat frais21. Même si une perte d’environ 15 % du produit final a été observée dans la deuxième réaction21, la réutilisabilité des enzymes adsorbés pour unique, ainsi que plusieurs étapes, les réactions constitue un atout important du système d’affichage de la spore.
Pan et al.24 a signalé une approche supplémentaire pour afficher des protéines hétérologues sur la surface des spores : protéines hétérologues (une protéine endoglucanase et une bêta-galactosidase un) produits dans la cellule-mère au cours de la sporulation étaient spontanément enfermé dans le manteau formant des spores, sans avoir besoin d’un transporteur. Ce système d’affichage supplémentaires spore est une combinaison des deux approches décrites jusqu'à présent. En effet, il est recombinant puisque les protéines hétérologues ont été conçus pour être exprimé dans la cellule-mère au cours de la sporulation, tandis que leur Assemblée au sein de la couche a été spontanée et, par conséquent, non24. Cependant, l’efficacité de l’affichage de cette approche supplémentaire reste à être testés et comparés avec les deux autres approches en utilisant les mêmes protéines hétérologues.
Le présent protocole exclut les processus de production de spores et de purification, qui ont été décrits abondamment ailleurs24. Il comprend la réaction d’adsorption, l’évaluation de l’efficacité d’adsorption par microscopie-dot-blot et de fluorescence, et le recyclage des enzymes adsorbés de réaction supplémentaire de tours.
Protocole
1. réaction d’adsorption
- Incuber 2, 5 et 10 µg de RDRF avec 2 x 109 de spores purifiées de b. subtilis sauvage dans 200 µL de liaison tampon, citrate de sodium de 50 mM, pH 4,0 (16,7 mM du citrate de sodium dihydraté ; acide citrique 33,3 mM) pendant 1 h à 25 ° C sur un agitateur basculant (Figure 1) .
- Centrifuger les mélanges de liaison (13 000 x g pendant 10 min) pour fractionner les granulés (P2, P5 et P10) et des surnageants (S2, S5 et S10). Stocker les surnageants d’évaluation indirecte de l’efficacité d’adsorption décrite à l’étape 3.2.
- Laver les granules 2 x avec 200 µL de tampon et remettre dans 100 µL de liaison de la mémoire tampon et utilisent pour l’analyse suivante contraignants.
Remarque : La réaction de liaison se produit préférentiellement à un pH plus bas que le point isoélectrique de la protéine ; en général, 1,5 M tampon phosphate salin (PBS), pH 4.0, ou 50 mM du citrate de sodium, pH 4,0, sont utilisés.
2. directe évaluation de l’efficacité d’adsorption
-
Extraction des protéines de surface et analyse par western blot
- Prendre 50 µL de la remise en suspension des spores RDRF-adsorbé (P2, P5 et P10 de l’étape 1.3) et ajouter 50 µL de 2 x dodécylsulfate de sodium (SDS)-le dithiothréitol (DTT) (0,1 M Tris-HCl, pH 6,8 ; 2 % SDS ; 0,1 M TNT) pour solubiliser des protéines de surface de spore.
- Utilisez les RDRF gratuit, purifiée et extraits des mêmes quantités de spores qui ne sont pas adsorbés à la protéine comme témoins positifs et négatifs, respectivement.
- Après 45 min d’incubation à 65 ° C, centrifuger (13 000 x g pendant 10 min), les mélanges et analyser 10 µL de l’extraits (surnageants) de protéines par western blot avec l’anticorps monoclonal anti-son anticorps reconnaissant son étiquette actuelle du N-terminal de RDRF ( Figure 2A).
-
Observations de microscopie fluorescente
Remarque : En utilisant des protéines hétérologues fluorescentes ou effectuer une analyse de l’immunofluorescence, il est possible de localiser et de quantifier les molécules adsorbées par microscopie à fluorescence.- Prenez 5 µL de resuspension des spores adsorbé à l’étape 1.3 et ajouter 95 µL de PBS 1 x, pH 4.0, pour obtenir environ 1 x 106 spores/µL.
- Déposer 5 µL de suspension sur une lame de microscope, recouvrir d’un lamelle couvre-objet précédemment traité par poly-l-lysine pour 30 s et l’observe sous un microscope à fluorescence.
- Pour chaque champ, enregistrer l’image de microscopie à contraste de phase et l’image de microscopie de fluorescence (Figure 2B).
Remarque : Vous pouvez également fluorescents spores peuvent être analysés par cytofluorimetry. Remettre un total de 106 spores adsorbé ou non-adsorbé avec la protéine hétérologue dans 1 mL de PBS 1 x, pH 4.0 et analyse la suspension à l’aide d’un cytomètre de flux (Figure 2C).
-
Analyse de données à l’aide de ImageJ
- Ouvrir les images de microscopie de fluorescence avec ImageJ logiciel (http://rsbweb.nih.gov/ij/) et vérifiez que toutes les images sont au format 8 bits (Image | Type | 8 bits).
- Ajustez le contraste si nécessaire (Image | Ajuster | Luminosité contraste) et vérifiez que l’échelle de l’image est PIXEL (Image | Définissez l’échelle).
- Dans le menu analyser, sélectionnez définir les mesures. Assurez vous d’avoir la zone, la Densité intégréeet Signifie gris valeur sélectionnée.
- Tracer une ligne autour de la spore d’intérêt en utilisant les outils de dessin et de sélection (c.-à-d., le cercle, le polygone, ou forme libre) (Figure 3).
- Sélectionnez la mesure dans le menu analyser (ou appuyez sur cmd + M). Une boîte de popup avec un stack de valeurs pour cette première spores s’affiche (Figure 3).
- Répétez ces deux étapes pour au moins 50 autres spores dans le champ de vision choisis doivent être mesurés.
- Sélectionner plusieurs régions sans spores (qui n’ont aucune fluorescence) et recommencez la mesure ; ce sera le fond.
Remarque : La taille n’est pas importante. Ces contre-valeurs de fluorescence de fond servira à soustraire manuellement l’arrière-plan. - Sélectionnez toutes les données dans la fenêtre de résultats et de copier les résultats dans une feuille de calcul.
- Calculer la moyenne de la zone des spores sélectionnés et des valeurs de fluorescence de fond et de Densité intégrée et utilisez-les pour obtenir la fluorescence de total par cellule (FCCT) corrigée à l’aide de la formule : CTCF = moyenne intégrés Densité - (surface moyenne x moyenne fluorescence de fond).
NOTE : Sinon, si toutes les spores apparaissent bien séparés, il est possible d’analyser toutes les spores du champ image en utilisant la fonction Analyser les particules, suivant les instructions de ImageJ. Pour éviter que le logiciel lit une fluorescence spécifique ou agrégats de spore, la dimension des particules doit être réglé à pixelˆ2 = 50-200 (Figure 4).
3. indirecte évaluation de l’efficacité d’adsorption
- Préparer des dilutions successives pour la protéine purifiée.
- Préparer un premier tube de 1,5 mL contenant 250 µL de RDRF purifiée à une concentration finale de 0,5 ng/µL, en utilisant le tampon de liaison. Ce volume est suffisant pour charger deux voies.
- Effectuer six double série de dilutions de 250 µL (volume final) chacune, en utilisant le tampon de liaison.
- Préparer des dilutions en série pour les échantillons de surnageants contenant la fraction RDRF indépendant de la réaction d’adsorption (S2, S5 et S10 de l’étape 1.2) double.
- Mettre 100 µL de chaque surnageant dans un tube de 1,5 mL et ajouter 100 µL de tampon de liaison. Effectuer six double série de dilutions de 200 µL (volume final) de chaque, en utilisant le tampon de liaison.
- Couper une nitrocellulose membrane (coupure de 0,45 µm), 9 x 10 cm dans la taille, pour couvrir la zone 5 (nombre d’échantillons) x 6 points (nombre de dilutions). La membrane ne doit pas dépasser le bord du joint de l’appareil de dot blot.
- Placer la membrane tremper dans l’appareil de la tache de dot. Éliminez les bulles d’air emprisonnés entre la membrane et le joint. Couvrir la fraction inutilisée de l’engin avec le film de ruban ou de la paraffine pour empêcher l’air de se déplacer par le biais de ces puits.
- Assembler l’appareil de la tache de dot comme indiqué par le fabricant.
- Si le vide est utilisé lors de l’assemblage, réhydrater la membrane avec 100 µL de solution 1 PBS x / puits pour garantir la liaison uniforme de l’antigène et éviter les auréoles ou un signal de détection faible.
- Retirez délicatement le tampon des puits par le vide. Dès que la solution tampon s’écoule de tous les puits, arrêter la pompe à vide et débranchez-le.
- Charger la norme dans les deux voies plus extérieures et les échantillons au milieu (Figure 5). Remplir les puits appropriés avec 100 µL de chaque dilution. Le même volume utilisé pour chaque puits devrait assurer une filtration homogène de tous les puits de l’échantillon.
- Mettre en marche la pompe à vide pendant 2 min, arrêter et ensuite, laisser l’échantillon filtrer à travers la membrane par écoulement gravitaire.
- Laver tous les puits avec 100 µL de 1 x PBS et laissez la pompe fonctionner pendant encore 5 min après le tampon de lavage a été entièrement vidangée de l’appareil.
- Avec le vide, desserrer les vis et ouvrir avec précaution l’appareil de dot blot.
- Éteindre l’aspirateur, prendre la membrane et le traiter selon un protocole de transfert de western.
- Effectuer une analyse densitométrique du filtre, à l’aide de logiciels appropriés, tels que ImageJ.
- Mesurer la densité intégrée de chaque point de leur expliquant avec un cercle du même espace et en utilisant la commande Analyze/mesure .
- Faire une correction de fond de l’image, dessiner un cercle dans une zone vide et sa densité intégrée de mesure ou à l’aide de la commande de Processus/soustraction arrière-plan .
- La densité intégrée des points standards en corrélation avec la quantité de protéines chargées et d’obtenir une courbe d’étalonnage (R2 valeurs d’étalonnage courbes doivent être à 0,95).
- La courbe d’étalonnage permet d’extrapoler la concentration de RDRF de chaque point de l’échantillon.
- Calculer la concentration de la RDRF restant dans les fractions non liées.
Remarque : Pour assurer la fermeture correcte de l’appareil de dot blot et, par conséquent, au sujet la membrane à une pression uniforme, serrer les vis en suivant un schéma croisé en diagonale et, ensuite, ouvrir la pompe à vide pour serrer les vis plus fortement.
4. la collection des spores et la réutilisation
- Pour le recyclage de la spore adsorbée, effectuer deux réactions d’adsorption de 2,0 x 10 spores9 purifié avec 10 µg de purifiée GH10-XA xylanase ou 10 µg de purifiée GH3-XT β-xylosidase, tel que décrit dans les étapes 1.1-1.3 (Figure 6A).
- Recueillir des granulés contenant les spores adsorbé à l’enzyme, les remettre en suspension dans 50 µL de la mémoire tampon d’optimale pour l’analyse de la réaction enzymatique (tampon de phosphate de sodium de 50 mM à pH 6,5 ; 2,48689 g/L Na2HPO4, 4,88991 g/L NaH2PO4), et mélanger l’ensemble pour obtenir un mélange de 100 µL de spores adsorber GH10-XA ou GH3-XT.
- Ajoutez le substrat (5 mg/mL 4-O-methyl-d-glucuronod-xylan [MGX]) et de laisser les réactions enzymatiques ont lieu pendant 16 h à 65 ° C.
- Centrifuger le mélange réactionnel (à 15 min à 13 000 x g) et stocker le liquide surnageant contenant le produit de la réaction enzymatique.
- Resuspendre le culot dans 100 µL de tampon de phosphate de sodium frais 50 mM à pH 6,5 en présence d’un nouveau support (MGX) (Figure 6B).
Remarque : Pour adsorber plus d’une enzyme, il est possible d’adsorber les deux ensemble ou les uns indépendamment. La dernière possibilité facilite l’analyse quantitative de l’efficacité d’adsorption et de l’activité de chaque enzyme, ce qui permet un équilibre stoechiométrique de chaque enzyme nécessaire pour les réactions.
Résultats
Adsorption réussie peut être évaluée par western Blot. Lors de la réaction, le mélange est fractionné par centrifugation et lavé, et la fraction de pellet (Figure 1) est utilisée pour extraire les protéines de surface. L’extrait est fractionné par électrophorèse sur gel de polyacrylamide SDS (PAGE), électrotransféré sur une membrane de polyfluorure de vinylidène (PVDF) et a réagi contre les anticorps primaires et secondaires. La présence de protéines de la taille attendue, seulement dans la voie chargée avec un extrait des spores adsorbés, témoigne d’une réaction d’adsorption réussie (Figure 2A).
L’efficacité de la réaction d’adsorption peut être évaluée par des méthodes directes et indirectes. L’évaluation directe de l’efficacité d’adsorption dépend de la protéine hétérologue qui a été utilisée et peut être réalisée par microscopie de fluorescence (Figure 2B) et cytofluorimetry (Figure 2C) sur la fraction de pellet après le fractionnement de la réaction d’adsorption. Une quantification des signaux fluorescents présents sur les spores peut être effectuée en utilisant le logiciel ImageJ (Figure 3 et Figure 4). Une analyse indirecte de l’efficacité d’adsorption peut être effectuée par une dot blot analyse (Figure 5A) de la fraction surnageante contenant la protéine non liée (Figure 1). Une analyse densitométrique de la protéine non liée (Figure 5B) permettra ensuite aux scientifiques de calculer indirectement la quantité de protéines adsorbé sur les spores.
Deux réactions successives peuvent être catalysées par un mélange de spores affichant l’une des deux enzymes spécifiques (Figure 6A). L’enzyme adsorbée peut être enlevées avec une étape de centrifugation simple, lavés et incubés avec frais pour un nouveau cycle de réaction (Figure 6B).

Figure 1:régime général de l’expérience de l’adsorption. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
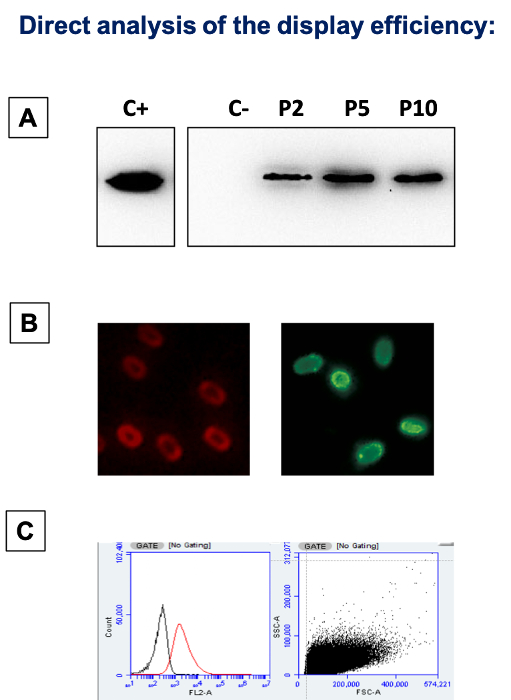
Figure 2 : Direct analyse de l’efficacité de l’affichage RDRF. (A) Western blot avec anticorps RDRF spécifique. C + = gratuit RDRF purifié ; C - = un extrait de protéines de spores non adsorbés à la protéine ; P2/P5/P10 = protéines extraites des spores de b. subtilis adsorbés avec 2, 5 et 10 mg de RDRF, respectivement. (B) la microscopie des spores adsorbées par Immunofluorescence. Immunoreactions ont été réalisées avec un anticorps primaire reconnaissant la protéine adsorbée et un anticorps secondaire fluorescent conjugué avec l’isothiocyanate de fluorescéine (FITC). Le panneau de gauche montre la fluorescence rouge RDRF intrinsèque, que le panneau de droite montre la fluorescence verte d’anticorps secondaire conjugué FITC. (C) analyse d’écoulement par cytométrie en flux de spores adsorbés. Les spores ont réagi avec les anticorps spécifiques RDRF et anticorps secondaires conjugué FITC et, ensuite, ont été analysés par cytofluorimetry. L’analyse a été réalisée sur la population entière de spore (10 000 événements, non vannés). Dans le panneau gauche, spores non-adsorbés sont indiquées dans des spores noires RDRF-adsorbés en rouge. Le panneau de droite montre l’intrigue de point de diffusion vers l’avant et latéraux (FSC-SSC). S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 3: manuelle quantification du signal fluorescent par ImageJ. Une image de microscope de fluorescence des spores, adsorbé avec la protéine fluorescente rouge RDRF, analysée avec le logiciel ImageJ. Un cercle jaune a été établi autour d’une spore pour obtenir des données densitométriques (image agrandie). La boîte de popup montre les résultats de l’analyse densitométrique de la spore sélectionnée. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 4: la quantification simultanée du signal fluorescent par ImageJ. (A) boîte contextuelle obtenue lorsque vous sélectionnez Analyser les particules. Une valeur d’intervalle de 50 à 200 pixels ^ 2 doit être réglé pour spores23. (B) Segmentation de l’image de la Figure 3A après avoir utilisé l' Analyse de particules (à gauche) et les résultats de l’analyse densitométrique relative (à droite). S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 5: Dot blot analyse densitométrique. (A) Dot blot avec des dilutions de le RDRF purifiée en deux exemplaires (Std1 et Std2) et le surnageant (S10, S5 et S2) de la réaction d’adsorption avec 10, 5 et 2 µg de RDRF, respectivement23. (B), le dot blot de panneau A est utilisé pour l’analyse densitométrique. Les cercles indiquent la zone utilisée pour quantifier la densité des signaux. Le panneau sur la droite indique un exemple des résultats obtenus avec l’analyse densitométrique. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 6: Conversion de xylane par spores réutilisables. (A) le régime général de dégradation de xylane. (B) Spores affichant la xylanase ou les enzymes de β-xylosidase, lorsqu’il est mélangé ensemble, catalysent la dégradation en deux étapes de xylane. Après la réaction, l’échantillon est fractionné par centrifugation. Le surnageant contient le produit de la réaction, tandis que le culot contient des enzymes liées aux spores qui peuvent être réutilisés en ajoutant le substrat frais. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Discussion
Ce protocole d’adsorption de spore est très simple et intuitive. La réaction est strictement dépendante du pH de la mémoire tampon de réaction et l’efficacité d’adsorption est optimale lorsque le pH acide (pH 5,0 ou plus bas). À des conditions de pH neutre, l’efficacité d’adsorption est faible, et à un pH alcalin, adsorption ne peut survenir. Adsorption optimale est obtenue en utilisant un volume de 200 µL dans des tubes de 1,5 mL (ou garder un ratio similaire) sur un agitateur basculant.
L’adsorption est très serrée, et lavages avec un tampon à pH même de la mémoire tampon de réaction ne causent pas de toute libération des protéines adsorbés. Lavages avec tampons alcalins peuvent entraîner un minimum (généralement moins de 15 %26) version de la protéine adsorbée.
L’évaluation indirecte de l’efficacité d’adsorption par dot-blot est fiable si plusieurs dilutions de protéine purifiée et indépendante sont analysées et l’analyse densitométrique est correctement réalisée. Aucun signe de dégradation des protéines hétérologues n’a été rapporté25. L’évaluation directe de l’efficacité d’adsorption dépend grandement de la protéine qui est adsorbée. Si la protéine est auto-fluorescente ou fluorescent étiqueté, une analyse assistée par ImageJ fournit une détermination quantitative de la fluorescence et des quantités de molécules fluorescentes présente sur les spores. Si la protéine possède une activité enzymatique, un dosage enzymatique spécifique pourrait fournir une indication des quantités de protéine présente sur les spores. Cependant, on sait que l’activité enzymatique liée à spores peut être augmentée par un effet de stabilisation en raison de l’interaction avec la spore13. Si la protéine adsorbée n’est pas fluorescente et n’est pas une activité enzymatique, l’efficacité d’adsorption peut être évaluée par dot-blot sur les spores extorquées dans des conditions drastiques.
Une collection de spores utilisés peut être faite par une procédure très simple. Une étape de lavage avec le tampon de réaction peut être importante d’éliminer les sous-produits de la réaction, tandis que l’addition du substrat frais est essentielle d’initier une nouvelle réaction21.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Ce travail a été soutenu par « Finanziamento di Ricerca di Ateneo » à L. Baccigalupi, titre du projet « SP-LAY : des spores bactériennes platefrome direct pour l’affichage des protéines ».
matériels
| Name | Company | Catalog Number | Comments |
| 0.1% Poly-L-lysine solution | Sigma | P8920 | |
| 0.45 µm Nitrocellulose Blotting Membrane | Sartorius | M_Blotting_Membranes | |
| 100× objective UPlanF1 | Olympus | microscope equipment | |
| Bacillus subtilis strain NCIB3610 | Bacillus Genetic Stock Center | 3A1 | |
| BD ACCURI C6 PLUS | BD | flow cytometer | |
| BX51 | Olympus | Fluorescent microscope | |
| Clarity | Biorad | 1705060 | |
| DP70 digital camera a | Olympus | microscope equipment | |
| Goat anti-Rabbit IgG (H+L) Cross-Adsorbed Secondary Antibody, FITC | Thermo fisher | F-2754 | |
| Goat Anti-Rabbit IgG H&L (HRP) | Abcam | ab6721 | |
| Monoclonal Anti-polyHistidine−Peroxidase antibody | Sigma | A7058-1VL | used to detect adsorbed proteins presenting a 6xhistidine-tag at C- or N- terminal |
| SecureSlip glass coverslip | Sigma | S1815-1PAK | |
| Superwhite Uncharged Microscope Slides | VWR | 75836-190 | |
| U-CA Magnification Changer | Olympus | microscope equipment |
Références
- Felici, F., et al. Peptide and protein display on the surface of filamentous bacteriophage. Biotechnology Annual Review. 1, 149-183 (1995).
- Cortese, R., et al. Identification of biologically active peptides using random libraries displayed on phage. Current Opinion in Biotechnology. 6, 73-80 (1995).
- Sousa, C., Cebolla, A., de Lorenzo, V. Enhanced metalloadsorption of bacterial cells displaying poly-His peptides. Nature Biotechnology. 14, 1017-1020 (1996).
- Richins, R., Kaneva, I., Mulchandani, A., Chen, W. Biodegradation of organophosphorus pesticides by surface-expressed organophosphorus hydrolase. Nature Biotechnology. 15, 984-987 (1997).
- Wu, J. Y., et al. Expression of immunogenic epitopes of hepatitis B surface antigen with hybrid flagellin proteins by a vaccine strain of Salmonella. Proceedings of the National Academy of Sciences of the United States of America. 86, 4726-4730 (1989).
- Newton, S. M., Jacob, C. O., Stocker, B. A. Immune response to cholera toxin epitope inserted in Salmonella flagellin. Science. 244, 70-72 (1989).
- Fischetti, V. A., Medaglini, D., Pozzi, G. Gram-positive commensal bacteria for mucosal vaccine delivery. Current Opinion in Biotechnology. 7, 659-666 (1996).
- Isticato, R., Ricca, E. Spore surface display. Microbiology Spectrum. 2 (5), (2014).
- Ricca, E., et al. Mucosal vaccine delivery by non-recombinant spores of Bacillus subtilis. Microbial Cell Factories. 13, 115(2014).
- McKenney, P. T., Driks, A., Eichenberger, P. The Bacillus subtilis assembly and functions of the multilayered coat. Nature Reviews Microbiology. 11, 33-44 (2013).
- Knecht, L. D., Pasini, P., Daunert, S. Bacterial spores as platforms for bioanalytical and biomedical applications. Analytical and Bioanalytical Chemistry. 400, 977-989 (2011).
- Isticato, R., Cangiano, G., De Felice, M., Ricca, E. Display of molecules on the spore surface. Bacterial Spore Formers: Probiotics and Emerging Applications. Ricca, E., Henriques, A. O., Cutting, S. M. , Horizon Bionsciences. Norfolk, UK. 193-200 (2004).
- Sirec, T., et al. Adsorption of β-galactosidase of Alicyclobacillus acidocaldaricus wild type and mutant spores of Bacillus subtilis. Microbial Cell Factories. 11, 100(2012).
- Cutting, S. M. Bacillus probiotics. Food Microbiology. 28, 214-220 (2011).
- Baccigalupi, L., Ricca, E., Ghelardi, E. Non-LAB Probiotics: Spore Formers. Probiotics and Prebiotics: Current Research and Future Trends. Venema, K., Do Carmo, A. P. , Caister Academic Press. 93-103 (2014).
- Cutting, S. M., Hong, H. A., Baccigalupi, L., Ricca, E. Oral Vaccine Delivery by Recombinant Spore Probiotics. International Reviews of Immunology. 28, 487-505 (2009).
- Lee, S. Y., Choi, J. H., Xu, Z. Microbial cell-surface display. Trends in Biotechnology. 21, 45-52 (2003).
- Isticato, R., et al. Surface display of recombinant proteins on Bacillus subtilis spores. Journal of Bacteriology. 183, 6294-6301 (2001).
- Huang, J. M., et al. Mucosal delivery of antigens using adsorption to bacterial spores. Vaccine. 28, 1021-1030 (2010).
- Isticato, R., et al. Non-recombinant display of the B subunit of the heat labile toxin of Escherichia coli wild type and mutant spores of Bacillus subtilis. Microbial Cell Factories. 12, 98(2013).
- Mattossovich, R., et al. Conversion of xylan by recyclable spores of Bacillus subtilis thermophilic enzymes. Microbial Cell Factories. 16, 218(2017).
- Pesce, G., et al. Surface charge and hydrodynamic coefficient measurements of Bacillus subtilis by optical tweezers. Colloids and Surfaces B: Biointerfaces. 116, 568-575 (2014).
- Donadio, G., et al. Localization of a red fluorescence protein adsorbed on wild type and mutant spores of Bacillus subtilis. Microbial Cell Factories. 15, 153(2016).
- Pan, J. G., Choi, S. K., Jung, H. C., Kim, E. J. Display of native proteins on Bacillus subtilis spores. FEMS Microbiology Letters. 358, 209-217 (2014).
- Cutting, S., Vander Horn, P. B. Genetic analysis. Molecular Biological Methods for Bacillus. Harwood, C., Cutting, S. , John Wiley & Sons. Chichester, UK. (1990).
- Lanzilli, M., et al. Display of the peroxiredoxin Bcp1 of Sulfolobus solfataricus probiotic spores of Bacillus megaterium. New Biotechnology. 46, 38(2018).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon