Method Article
Multiplex Cytokine profilage des splénocytes de souris stimulés à l’aide d’une plateforme axée sur la cytométrie de flux perle Immunoassay
Dans cet article
Résumé
Ce protocole décrit la quantification des cibles multiples de cytokines simultanément dans les surnageants de culture de tissus prélevés de splénocytes de souris stimulés à l’aide de perle multiplex immunodosage base plate-forme et un cytomètre en flux.
Résumé
Perle immunologiques utilisent le même principe de base que les immuno-essais "sandwich". Capture de billes, qui peuvent être différenciés par la taille et l’intensité de fluorescence allophycocyanin interne (APC), sont conjugués d’anticorps spécifiques d’un analyte spécifique. Ensuite, un groupe sélectionné de capture définies perle ensembles est incubé avec un échantillon biologique contenant des analytes cibles spécifiques à l’anticorps de capture. Un anticorps de détection biotinylé cocktail est ajouté, ce qui conduit à la formation des sandwichs de perle-analyte-détection anticorps de capture.
Enfin, streptavidine-phycoérythrine (SA-PE) est ajoutée, qui se lie à l’anticorps de détection biotinylés, fournissant les intensités du signal fluorescent proportionnellement à la quantité d’analyte lié. Le PE fluorescent signal des régions de l’analyte spécifique perles quantifiée à l’aide de cytométrie en flux et les concentrations d’analytes particulière sont déterminées à l’aide du logiciel d’analyse de données et la courbe d’étalonnage générée lors de l’essai.
Dans cette expérience, nous utilisons un panneau de cytokines de souris T helper pour quantifier simultanément la concentration de 13 cibles de cytokines distinctes dans les surnageants de culture de tissus prélevés de splénocytes de souris cultivées dans différentes conditions de stimulation.
Introduction
En tant que molécules de signalisation solubles, cytokines médier les processus hautement coordonnées et multifactorielles qui régissent les réactions immunologiques de l’hôte. Ils sont exprimés durant toutes les étapes du processus inflammatoire, de l’initiation à la résolution et régulent un réseau d’interactions complexes qui comporte leur propre synthèse et celle de leurs récepteurs cellulaires1. Ce réseau de communication est posé avec une complexité qui dépasse les relations synergiques ou antagonistes qui peuvent exister entre les différents composants. En effet, nombreuses cytokines sont connus pour partager les fonctions redondantes ou au moins partiellement superposés2,3.
Approches de la biologie des systèmes intégrés qui quantifient simultanément plusieurs analytes de cytokine fournissent actuellement une compréhension plus complète de le cytokinomes unique qui orchestrent les réponses immunitaires sous-jacente aux maladies multiples dispose de4,5. Ces États de maladie s’étendent d’une inflammation généralisée au cancer, neuro-dégénératives et les maladies cardiovasculaires6,7,8,9.
Ces réseaux de cytokines peut être interrogées efficacement à l’aide de LEGENDplex perle immunologiques. Ces tests sont basés sur le même principe que le sandwich enzyme-linked Immunosorbent Assay (ELISA) et utilisent des microsphères de fluorescence encodé avec des anticorps de capture de façon covalente à leur surface. Ces anticorps sont immobilisées sur des surfaces beaucoup plus petites que celles requises par les formats traditionnels de ELISA. Cela permet à ces tests d’être interprétée avec beaucoup moins volume de l’échantillon, alors qu’en même temps réduisant non-spécifiques contraignant et fournissant multiplexés analyse plusieurs analytes.
Le test décrit dans ce protocole utilise cette technologie pour doser jusqu'à 13 cibles simultanément. Données de cet essai peuvent être obtenues en utilisant une grande variété de cytomètres couramment disponibles et contrairement à d’autres tests disponibles ne nécessitent pas l’utilisation de l’instrumentation de test spécifique dédiée. Avec un catalogue en pleine expansion des panneaux de l’analyte validés, ces tests ont été utilisés dans plusieurs travaux de recherche biomédicale projets10,11,12,13.
Pour démontrer la facilité et l’utilité du format dosage, dans cette expérience que nous permet de quantifier un panel de cytokine souris T helper 13 distincts des concentrations de cytokine de surnageants de culture de tissus obtenus à partir des splénocytes de souris cultivées sous multiple conditions de stimulation. Outre les surnageants de culture de tissus, ce test peut également être effectué en utilisant des échantillons de sérum ou de plasma.
Protocole
toutes les expérimentations animales ont été effectuées conformément aux recommandations de NIH contenues dans le Guide pour le soin et l’utilisation des animaux de laboratoire et ont été approuvées par l’IACUC à BioLegend.

figure 1 : filtre plaque test procédure Résumé. Le protocole pour la réalisation de l’essai à l’aide de plaques de filtration est représenté par un diagramme illustrant les composants du coffret de clés et les étapes d’incubation. s’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
1. préparation de l’échantillon biologique
Remarque : le choix de l’emplacement anatomique et le volume de prélèvement de sang est laissée à la discrétion de chaque chercheur individuel et n’affectera pas le résultat du test. Cependant, une fois qu’elles sont obtenues, les échantillons doivent être manipulés selon les étapes ci-dessous.
- Préparation des échantillons de sérum
- recueillir le volume souhaité de sang dans le tube de collection par défaut et lui permettre de coaguler pendant au moins 30 min.
- Centrifuger l’échantillon pendant 10 min à 1 000 x g à la température ambiante.
- Supprimer sérum et test immédiatement ou partie aliquote dans des tubes de microcentrifuge de polypropylène et de conserver les échantillons à ≤ -20 ° C.
- Préparation des échantillons de Plasma
- recueillir le volume souhaité de sang à l’aide de l’acide éthylènediaminetétraacétique (EDTA) enduit tubes de prélèvement sanguin.
- Centrifugation pendant 10 min à 1 000 x g à la température ambiante dans les 30 minutes de la collection.
- Supprimer le plasma et test immédiatement ou partie aliquote dans des tubes de microcentrifuge de polypropylène et de conserver les échantillons à ≤ -20 ° C.
- Préparation d’échantillons de surnageant de Culture tissulaire
- Centrifuger l’échantillon pendant 10 min à 1 500 g à 4 ° C pour éliminer les cellules et les débris.
- Recueillir le surnageant et test immédiatement ou aliquote dans des tubes de microcentrifuge de polypropylène et de conserver à ≤ -20 ° C.
- Dégel des congelés prélèvements biologiques
- complètement décongeler les échantillons biologiques décongelées sur la glace et agiter brièvement avant utilisation. Évitez plus de 2 cycles de gel/dégel.
Remarque : Les échantillons utilisés dans le présent protocole ont été obtenus en cultivant les splénocytes de souris BALB/c (1 x 10 6 cellules à 37 ° C + 5 % de CO 2 dans diverses conditions : non stimulés, LPS (100 ng/mL), αCD3 (1 µg/mL de plaque-enduit) + αCD28 (1 µg/mL soluble), PMA (20 ng/mL) + ionomycine (500 ng/mL). Les surnageants de culture ont été prélevés après 48 h et traitée de la manière décrite à la section 1.3.
- complètement décongeler les échantillons biologiques décongelées sur la glace et agiter brièvement avant utilisation. Évitez plus de 2 cycles de gel/dégel.
2. Préparation du réactif
- Perles de Preparation of Pre-mixed Antibody-Immobilized
- un prémélangés perles bouteille pendant 1 min dans un bain sonicateur à température ambiante et puis vortexer pendant 30 avant de s de l’utiliser. Si aucun bain sonicateur n’est disponible, augmenter le temps de vortex à 1 min.
- Préparation du tampon de lavage
- tampon de lavage apportez le 20 x (20 x PBS avec 1 % Tween-20) fourni avec le kit à température ambiante et vortex d’apporter tous les sels en solution.
- Diluer 25 mL 20 x tampon de lavage avec 475 mL d’eau désionisée. Portion non utilisée de magasin entre 2 ° C et 8 ° C pendant environ un mois.
- Préparation de la matrice B (pour utilisation avec du sérum et Plasma échantillons seulement)
- Ajouter 5,0 mL de tampon Assay (PBS avec 1 % de BSA) inclus dans le kit pour le flacon lyophilisé matrice B. autoriser au moins 15 min pour reconst complet itution, puis vortexer pour bien mélanger. Restes reconstitué matrice B peut être conservée à ≤-70 ° C pendant environ un mois.
3. Préparation standard
Remarque : chaque analyte dans ce panneau a une concentration supérieure standard de 10 000 pg/mL.
- Ajouter 250 µL de tampon de reconstituer le lyophilisé souris Th Cytokine Standard Cocktail. Mélanger au vortex brièvement et laisser la fiole pour reposer à température ambiante pendant 10 min.
- Transférer le cocktail standard dans un tube de microcentrifuge polypropylène étiqueté " C7 ". Il sera utilisé comme le plus haut standard.
- 6 étiquette polypropylène microtubes à centrifuger comme C6, C5, C4, C3, C2 et C1.
- Ajouter 75 µL de tampon à chacun de ces tubes.
- Transfert de 25 µL de la C7 standard supérieur au tube de C6 et mélanger bien au vortex. Ce sera la norme C6.
- Continuer à effectuer la série des dilutions de 1:4 à l’aide d’une pipette de nouvelle astuce pour chaque tube d’ajouter 25 µL de la norme précédente à la 75 µL de tampon dans le tube standard suivant le plus bas, suivi au vortex pour obtenir les normes C5, C4, C3 , C1 et C2. Utilisation du tampon d’essai comme la norme 0 pg/mL (C0).
4. Dilution de l’échantillon
Remarque : expériences préliminaires de pilotes à l’aide de ce test avec des dilutions multiples peuvent être nécessaires afin de déterminer le facteur de dilution plus approprié pour un ensemble particulier d’échantillons biologiques. Un facteur de dilution adéquate produira les calculs de concentration de l’échantillon qui se trouvent dans les limites de la courbe standard. Les étapes suivantes visent comme lignes directrices et peuvent devoir être déterminées empiriquement selon le type d’échantillon.
- Diluer sérum ou plasma échantillons 2 fois avec du tampon (e.g. diluer 50 µL de l’échantillon avec 50 µL de tampon) dans des tubes de microcentrifuge polypropylène.
NOTE : Si plus la dilution de l’échantillon est nécessaire, dilutions devraient être faites avec matrice B au lieu du tampon afin d’assurer une mesure précise. Ajout des échantillons de sérum ou de plasma sans dilution seront traduira par la précision de dosage faible et peuvent obstruer la plaque de filtre. Matrice B est composé de sérum de souris groupés appauvri de cibles d’essai endogène. Il est utilisé comme un échantillon de sérum ou de plasma diluant afin d’éviter les effets de matrice, qui sont connus pour la sensibilité analytique de l’affect d’immunoessais. - Test de cellules échantillons de surnageant de culture sans dilution.
NOTE : Les niveaux de l’analyte peuvent varier considérablement d’un échantillon à l’autre. Si nécessaire, diluer les échantillons surnageants à l’aide d’une préparation fraîche de leur milieu de culture cellulaire correspondant ou tampon.
5. Mode opératoire
Remarque : le test peut être effectué dans les plaques de filtration en polypropylène, tubes micro FACS ou fond V microplaques. La procédure de dosage de plaque de filtre est recommandée en raison de bon échantillon à l’autre, cohérence, test de robustesse et facilité de manipulation. Cette procédure nécessite une unité de filtration sous vide pour le lavage (fourni par l’utilisateur final).
- Tous les réactifs laisser se réchauffer à température ambiante (20-25 ° C) avant utilisation.
- Ensemble la plaque de filtre sur une couverture plaque retournée à tout moment au cours des étapes d’installation et d’incubation de dosage, afin que le bas de la plaque ne touche pas toutes les surfaces, car il pourrait causer des fuites.
- Garder la plaque en position verticale pendant le mode OPERATOIRE entière, y compris les étapes de lavage, pour éviter de perdre les perles.
- Garder la plaque dans le sombre ou enveloppé avec du papier aluminium pour toutes les étapes de l’incubation.
- Exécuter tous les échantillons et étalons comme des doublons, disposées sur la plaque dans un ordre séquentiel.
- Pré mouillage la plaque de filtre en ajoutant 100 µL de 1 x tampon de lavage à chaque puits et laisser reposer pendant 1 min à température ambiante (si vous utilisez le filtre fond microplaques).
- Volume de tampon de supprimer à l’aide de la tubulure de vide (5-10 s). Ne pas dépasser 10 " Hg du vide. Épongez l’excès lavage Tampon du fond de la plaque en appuyant sur la plaque sur une pile de serviettes en papier propre. Placer la plaque au-dessus du couvercle de la plaque retournée.
Remarque : Les étapes 5.1-5.2 peuvent être omises si vous effectuez le test en utilisant un fond V microplaque ou micro FACS tubes. - Échantillons surnageant de culture cellulaire, ajouter 25 µL de tampon dans chaque puits. Ajouter 25 µL de chaque étalon aux puits standards. Ajouter 25 µL de chaque échantillon dans les puits échantillon.
- Pour mesurer les échantillons de sérum ou de plasma, ajouter 25 µL de matrice B aux puits standards. Ajouter 25 µL de tampon aux puits d’échantillons. Ajouter 25 µL de chaque étalon aux puits standards. Ajouter 25 µL de chaque sérum dilué ou un échantillon de plasma dans les puits échantillon.
- Vortex mixte perles pour 30 s. Ajouter 25 µL de perles mélangées dans chaque puits, en secouant la bouteille perle par intermittence afin d’éviter la perle s’installer. Le volume final devrait être de 75 µL à chaque puits après l’ajout de perles.
- Sceller la plaque avec un film adhésif. Enrouler la plaque entière, y compris la couverture de la plaque retournée, une feuille d’aluminium. Placer la plaque sur un agitateur de plaque, fixez-la et agiter à environ 500 tr/min pendant 2 h à température ambiante. Pour éviter des fuites, ne pas appliquer une pression positive sur le film adhésif.
- Sans inversion, placer la plaque sur la tubulure de vide et appliquer le vide comme avant et ajouter 200 µL de 1 x tampon de lavage à chaque puits.
- Supprimer le contenu des puits assay plaque de filtration sous vide. Tamponnez le tampon de lavage excessif du bas de la plaque avec un tampon absorbant ou essuie-tout. Répétez cette étape une fois de plus.
NOTE : Si l’analyse est réalisée à l’aide d’un FACS de microplaque ou micro V-bas tubes skip étapes 5.12 et 5.13. Au lieu de cela Centrifuger la plaque à 1 000 x g pendant 5 min à température ambiante, puis retirez le surnageant à l’aide d’une pipette multicanaux. - Ajouter 25 µL d’anticorps de détection (voir la Table des matières) dans chaque puits.
- Sceller la plaque avec un film adhésif fraîches. Enrouler la plaque entière, y compris la couverture de la plaque retournée, une feuille d’aluminium.
- Placer la plaque sur un agitateur de plaque et l’agiter à environ 500 tr/min pendant 1 h à température ambiante.
- Sans aspiration, ajouter 25 µL de réactif de SA-PE directement dans chaque puits. Aucune dilution du réactif n’est nécessaire.
- Sceller la plaque avec un film adhésif fraîches. Enrouler la plaque entière, y compris la couverture de la plaque retournée, une feuille d’aluminium.
- Placer la plaque sur un agitateur de plaque et l’agiter à environ 500 tr/min pendant 30 min à température ambiante.
- Répéter l’étape ci-dessus 5.13.
- Ajouter 200 µL de 1 x tampon de lavage à chaque puits. Remettre en suspension les perles sur un agitateur de plaque pendant 1 min.
- à l’aide d’une pipette multicanaux, transférer les échantillons de la plaque de filtre aux tubes de FACS pour lire les échantillons sur un cytomètre.
NOTE : Volume de l’échantillon peut être augmenté de 200 µL à 300 µL en ajoutant un supplément de 100 µL de 1 x tampon de lavage à chaque tube pour éviter l’échantillon sec. Si les échantillons nécessaires peuvent être conservés à 4 ° C abri de la lumière et analysé le lendemain. Cependant, conservation de l’échantillon prolongée peut conduire à signal réduit.
6. Flow cytomètre Set-up
Remarque : afin de générer des données fiables, le cytomètre de flux doit être correctement configuré avant l’acquisition de données. Ce processus peut varier pour les utilisateurs individuels à certains égards selon la configuration de l’instrument particulier, sur que l’analyse est réalisée. Un processus de réglage généralisé pour un cytomètre capable de mesurer des PE et des APC et équipé à la fois un laser 488 633 nm est abordée ci-dessous.
- Lancer le logiciel de flux cytomètre et acquisition selon le fabricant ' instructions s fournies avec l’appareil.
- Créer un terrain en dot à FSC (forward scatter) sur l’axe des abscisses et SSC (diffusion latérale) de l’axe y. Mode linéaire la valeur FSC et SSC.
- Créer une seconde intrigue dot avec du PE sur l’axe x et l’APC pour l’axe y. Cette parcelle doit être réglée sur un mode d’affichage basé sur un journal.
- Vortex le flacon de perles brutes inclus dans le kit pour 30 s pour remettre en suspension les perles. Ces billes contiennent un colorant APC interne et se composent de deux populations de taille.
- Transférer 400 µL des billes brutes dans un nouveau tube de FACS.
- Régler le débit du cytomètre de flux à faible.
- Exécuter les perles brutes, ajuster soigneusement le gain et la tension pour FSC et SSC afin que les deux populations de taille de ces perles sont visiblement séparés et faciles à porte.
- Régler le seuil FSC pour exclure les éléments indésirables (c'est-à-dire les bulles de débris ou de l’air).
- Intrigue dans the FSC vs SSC, dessinez une porte qui inclut toutes les populations de perle.
Remarque : Les perles brutes contiennent deux populations de taille des perles, le plus petit " une perles " et la plus grande " perle B " régions. - Afficher les populations perle dépendants de l’intrigue FSC vs SSC sur le second tracé dot avec du PE sur l’axe x et l’APC pour l’axe y.
- Régler la tension de PMT pour le canal de fluorescence APC afin que le signal de l’APC pour toutes les populations de perle a une intensité de fluorescence médian (MFI) qui se situe entre 1 x 10 1 et 5 × 10-3.
- Vortex le flacon de l’installation de PE de perles pour 30 s pour remettre en suspension les perles.
- Perles de transférer 400 µL du PE dans un nouveau tube de FACS.
- Remplacer les premières perles tube depuis le cytomètre de flux avec le PE perles tube.
- Ajuster la tension photomultiplicateur (PMT) pour le PE fluorescence canal réglage afin que l’IMF des perles PE se situe entre la gamme spécifique du lot trouvée figurant sur le flacon de perles PE.
Remarque : Les perles de configuration PE contiennent des perles de la taille d’une population (" un perles " seulement).
7. Acquisition de données
Remarque : les procédures exactes associées à l’acquisition de données sur un instrument donné peuvent varier et dépendent de la cytomètre ' s spécifications de configuration et le logiciel d’interface utilisé. Les instructions ci-dessous sont donc destinées à mettre en évidence les étapes nécessaires à prendre lors de l’essai quel que soit le cytomètre employée dans l’essai de.
- Vérifier le débit cytomètre est toujours fixé à faible.
- Définir le nombre d’événements de perle à acquérir pour environ 300 par analyte. Pour un 13-plex, panneau, cela équivaut à l’acquisition de 3 900 événements combinés des deux populations de taille de perles (perles de A + B).
- Vortex chaque échantillon pendant 5 s avant l’analyse par.
- Lecture des échantillons. Lors de la lecture des échantillons, affectez le cytomètre mode d’installation tout d’abord et attendre jusqu'à ce que la population de billes est stabilisée avant de passer au mode d’acquisition.
- Utiliser des noms simples avec une numérotation consécutive pour les fichiers de données pour faciliter l’analyse des données.
- Exporter uniquement les événements dépendants (régions de billes A + B) plutôt que du nombre total d’événements.
- Stocker tous les fichiers de la FCS dans le même dossier pour chaque dosage. Si plusieurs essais en cours d’exécution, créer un dossier séparé pour chaque dosage.
Remarque : les fichiers de la FCS générées sur le cytomètre en flux doivent être analysées en utilisant le logiciel d’analyse de données, qui peut être téléchargé gratuitement 14.
- Installer le logiciel d’analyse de données sur un PC.
- Transférer tous les fichiers de test FCS à l’ordinateur qui contient le logiciel d’analyse.
- Branchez le dongle clé de licence (inclus dans le kit) sur un port USB de l’ordinateur.
- Lancer le logiciel d’analyse de données.
- Cliquez sur le bleu " ajouter des fichiers " bouton situé en haut de l’écran.
- Accédez au dossier qui contient les fichiers de FCS de l’analyse dans la fenêtre pop-up qui s’affiche.
- Cliquez et faites glisser tous les fichiers de test FCS pop-up fenêtre vers l’affichage du logiciel. Tous les fichiers doivent maintenant apparaître dans une liste.
- Cliquez sur le vert " prochaine " bouton en bas à droite de l’écran. Faites un clic gauche et maintenez pour faire glisser la petite courbe standard bleu boutons (C7 à C0) à leurs fichiers FCS correspondantes dans la liste pour définir la courbe standard.
- Cliquez sur le vert " prochaine " bouton en bas à droite de l’écran pour ouvrir le mécanisme de sélection fenêtre pop-up.
- Sur le côté gauche de la fenêtre de blocage, entrez les noms des deux A et régions perle B dosage d’analytes cibles et leur ID associés perle (trouvé dans le manuel fourni avec le test) dans l’ordre croissant.
- Sélectionner l’outil de blocage du haut de la fenêtre pop-up et l’utiliser pour dessiner 2 portes dans le complot FSC vs SSC, un autour des perles A et l’autre autour des perles de B.
- Revue de l’APC vs PE des intrigues qui apparaissent en dessous de l’intrigue FSC vs SSC qui apparaissent. Il y aura 6 bandes d’analyte dans les perles A (terrain à gauche) et 7 bandes d’analyte dans les perles de B (bonne intrigue).
Remarque : Portes peut être redessiné manuellement en sélectionnant l’outil Gomme en haut et cliquer sur pour supprimer un portail donné. L’outil de blocage peut alors être sélectionné et utilisé pour appliquer manuellement une porte dans la région de bande désirée. - Cliquez sur le vert " OK " bouton pour fermer le blocage fenêtre pop-up et de revenir à la liste de fichiers FCS.
- Définir les dilutions apportées aux échantillons biologiques par la droite en cliquant sur un fichier donné, sélectionner " Dilution plier " dans le menu et entrée la valeur correcte.
- Cliquez sur le vert " Run " bouton en bas à droite de l’écran pour générer les courbes standards pour l’essai et calculer les concentrations d’échantillons biologiques inconnus.
Résultats
Ce protocole montre comment une plateforme de test immunologique axée sur la perle peut être utilisée pour quantifier la cytokine profils provenant d’échantillons biologiques à l’aide d’un cytomètre en flux simultanément. Les échantillons biologiques utilisés dans cet exemple sont les surnageants de culture de cellules de splénocytes de souris qui avaient été préalablement incubées dans diverses conditions d’activation, bien que le format d’essai a également été validé pour utilisation avec des échantillons de sérum ou de plasma.
Les données générées par le test sont utilisées pour construire les courbes d’étalonnage pour tous les analytes dans le panneau multiplex. Le logiciel d’analyse de données classifie chaque analyte particulier basé sur une taille de perle unique et l’intensité de l’APC et quantifie les à l’aide d’un journaliste de PE. Les plages dynamiques, ainsi que la sensibilité de l’essai est démontrés lorsque tracées sur une échelle logarithmique en Figure 2 a. Le logiciel utilise les données de test et un algorithme d’ajustement de courbe de 5 paramètres pour calculer avec précision non seulement les concentrations d’analytes dans utilisateur fourni des échantillons biologiques, mais aussi les limites de détection sur deux extrémités haute et basse de toutes les courbes d’étalonnage (tableau 1 ). Le format décrit dans le présent protocole prévoit également la précision de dosage très robuste. Dans la Figure 2 b et 2C données sont présentées illustrant la variabilité intra-essai de chaque analyte. Deux concentrations distinctes de protéines standard (haute et basse) ont été analysées lors d’un essai avec 16 répétitions pour chaque échantillon. Figure 2 et 2D représentent la variabilité inter-essai des analytes cibles de trois tests indépendants. Comme avec les expériences de précision intra-essai, hautes et basses concentrations standards ont été analysées avec 3 réplique dans chacune des expériences indépendantes. Variabilité minime dans la concentration calculée de protéines cibles est clairement visible.
Afin de démontrer l’utilité des tests de cytométrie de flux axées sur le talon en interrogeant les profils d’expression des cytokines, des splénocytes de souris ont été incubées dans diverses conditions d’activation. Outre un contrôle non stimulée, les cellules ont été incubées également avec les vinyles, les anticorps anti-CD3/anti-CD28, ou phorbol 12-myristate 13-acétate et ionomycine. Les surnageants de culture de cellules ont été récoltées 48 h plus tard et analysée à l’aide du panneau de cytokine souris T helper. Les résultats de la quantification de cette expérience sont représentées dans la Figure 3 et l’effet des conditions de stimulation sur les concentrations de cytokines est clairement délimitée.
Les résultats décrits ci-dessus sont typiques, aussi longtemps que l’expérimentateur a bon laboratoire technique et suit le protocole d’essai prévu. Sources d’erreur dans ce format d’essai peuvent survenir de déviations dans les temps d’incubation, les volumes de réactifs, de lavage, ou de certains paramètres de PMT sur le cytomètre en flux utilisé pour analyser les échantillons. Dans la Figure 4 a un scatter plot d’un essai réussi montre 2 populations distinctes de perle qui sont clairement définies. Cela peut être comparée à la Figure 4 b, où adhérence médiocre de lavage et de protocole ont conduit à des agrégats de perle qui brouillent les deux populations. En outre, comme en témoignent les débris dans le quadrant inférieur gauche, cytomètre total événements ont été exportées pour analyse, et non le format préféré des événements dépendants uniquement. Cytomètre adéquat mis en place est essentiel pour produire des données fiables dans ce test. Dans PMT bonne Figure 4 paramètres permettent la résolution de chacun des 6 analytes dans le chenal de classification d’APC. Certains paramètres de PMT seront traduira par des données semblable à celle présentée dans la Figure 4 où le canal de l’APC a des populations de perle avec des intensités de classification qui se chevauchent. Cela annulerait le test puisqu’il sera impossible de calculer les concentrations d’analyte sans les populations de perles de dosage clairement déterminées.
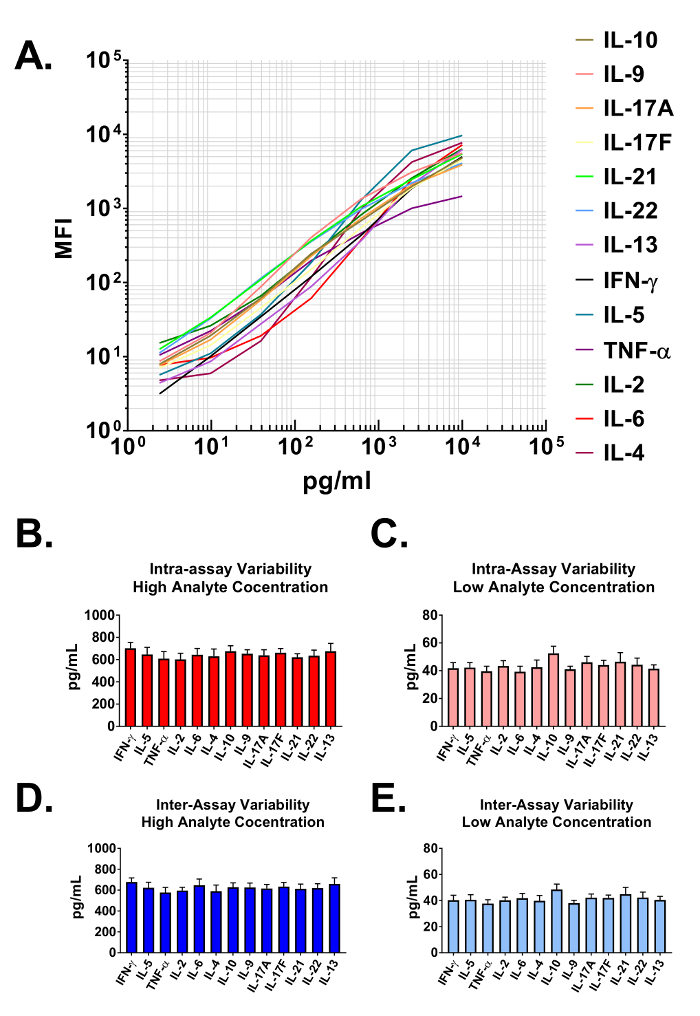
Figure 2 : gammes courbe Standard et précision essai. (A) une standard courbe générée à l’aide du panneau de cytokine souris T helper. Une courbe d’étalonnage doit être exécutée avec chaque test. (B-C) Précision intra-essai. Deux échantillons avec des concentrations différentes de protéines cibles (haute et basse) ont été analysées lors d’un essai avec 16 répétitions pour chaque échantillon. Données sont représentées sous la moyenne + l’écart-type. (D-E) Précision inter-essai. Deux échantillons avec des concentrations différentes de protéines cibles (haute et basse) ont été analysés dans les trois essais indépendants avec 3 répétitions pour chaque échantillon. Données sont représentées sous la moyenne + l’écart-type. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
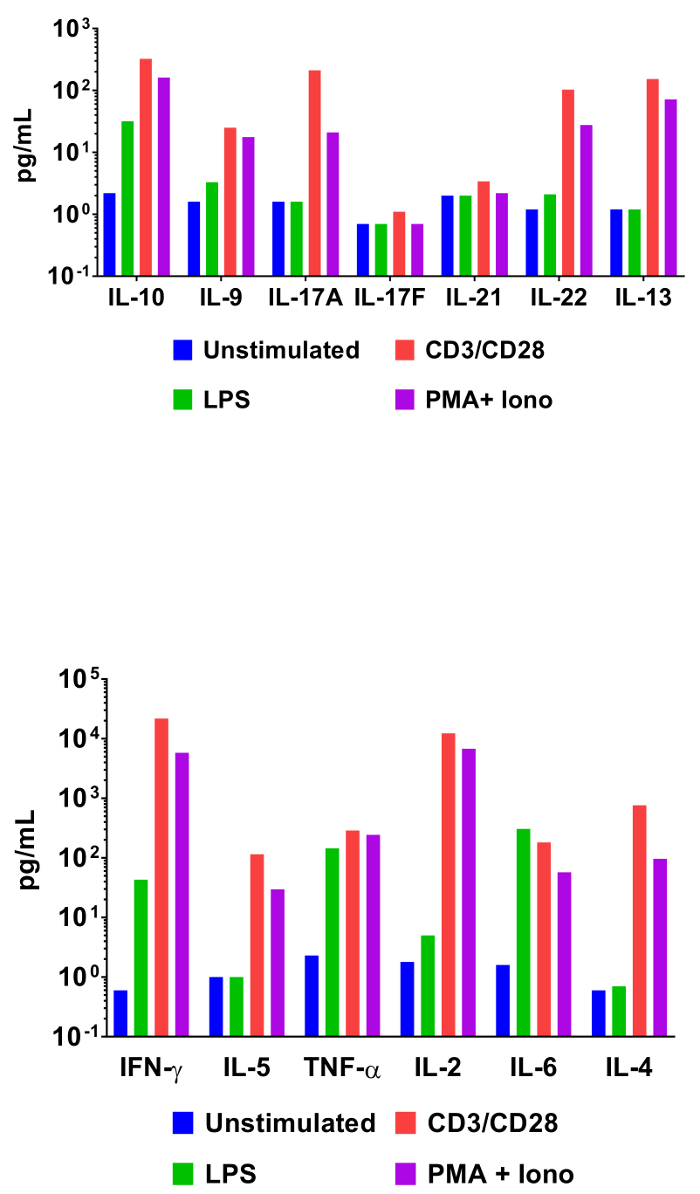
Figure 3 : Quantification de résultats représentant. Splénocytes de souris (1 x 106 cellules) sont cultivées à 37 ° C + 5 % de CO2 dans diverses conditions : non stimulés, LPS (100 ng/mL), αCD3 (1 µg/mL de plaque-enduit) + αCD28 (1 µg/mL soluble), PMA (20 ng/mL) + ionomycine (500 ng/mL). Les surnageants de culture ont été prélevés après 48 h puis quantifiée à l’aide du panneau de cytokine souris T helper. Profils d’expression des cytokines différentielle en réponse aux conditions de stimulation sont clairement visibles. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
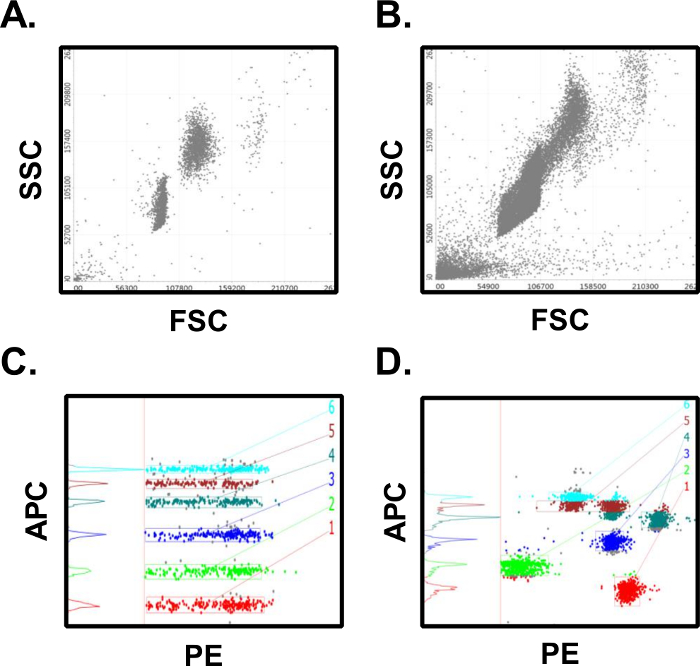
Figure 4 : résulte de succès contre les essais infructueux. (A) des essais réussis auront des populations distinctes de perle pour les populations de microsphères. (B) les essais infructueux aura séparation des populations pauvres, les agrégats de perle et trop d’événements collectés. (C) les essais réussis ont clairement défini intensités dans le canal de fluorescence de classification qui sont faciles à la porte et à quantifier. (D) les essais infructueux seront ont mal séparé et classification des intensités qui se chevauchent plusieurs populations de perle, ce qui rend difficile le dosage. Bon nombre de ces erreurs peuvent être évitées en respectant soigneusement le protocole d’essai. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
| Courbe d’étalonnage | Sensibilité (pg/mL) | ||||
| Analyte | Formule forme | CV | R2 | Min. | Max. |
| IFN-Γ | 5-P.log(4.43, 14.40, 0.54, 9.11, 0.01) | 1,77 % | 0.99 | 2.1 | 18105.0 |
| IL-5 | 5-P.log(4.36, 12.68, 0.64, 5.73, 0.36) | 1,32 % | 1 | 1,9 | 14514.0 |
| TNF-Α | 5-P.log(4.36, 11.63, 0.79, 5.45, 0.37) | 1.48 % | 0.99 | 1,9 | 20109.0 |
| IL-2 | 5-P.log(4.46, 12.62, 0.78, 5.07, 0.34) | 1,34 % | 1 | 1,9 | 25109.0 |
| IL-6 | 5-P.log(4.58, 11.66, 0.64, 5.77, 0.37) | ||||
Tableau 1 : ajustement de la courbe d’étalonnage et de sensibilité. Le logiciel d’analyse de données a été utilisé pour générer des courbes standard pour tous les analytes contenus dans l’essai multiplex. Cette analyse entièrement automatique s’applique à une courbe de 5 paramètres robuste montage algorithme selon les normes de série de dilution de l’essai et a également été utilisée pour définir la sensibilité en utilisant les limites théoriques de détection.
Discussion
Le format d’essai décrit dans le présent protocole peut fournir robuste quantification des profils de médiateur soluble dans des échantillons biologiques fournis l’expérimentateur a bon laboratoire technique et adhère au protocole recommandé.
Il y a plusieurs étapes clés qui doivent être suivies pour garantir des résultats fiables. Tout d’abord, les normes recombinants doivent pouvoir reconstituer dans du tampon le recommandé à plein temps et après seulement dilué dans des tubes en polypropylène. Tubes polystyrène peuvent favoriser l’adhérence des protéines de la paroi des tubes, qui peuvent conduire à des signaux faibles lors de la génération de la courbe d’étalonnage. Deuxièmement, secouer pendant les étapes d’incubation adéquate est essentielle. Sans agitation appropriée, les caractéristiques de liaison, des perles d’essai peuvent être gravement entravées. En outre, lavage correcte entre les étapes d’incubation est également requis. L’expérimentateur doit s’assurer que les excès réactifs soient écartés les puits avant de commencer l’étape suivante dans le protocole. Paramètres d’instruments pour le cytomètre en flux utilisés pour interroger les perles de dosage doivent être optimisés aussi bien. En particulier, les paramètres de PMT doivent être réglées de manière séparation appropriée de perle et des plages dynamiques larges pour les courbes standards. Enfin, juste avant d’être analysés sur le cytomètre, les billes doivent être mixés pour assurer une suspension adéquate et d’éviter la formation d’agrégats qui peuvent fausser les résultats.
Ce protocole peut être potentiellement modifié pour analyser des homogénats de tissus ainsi que les types d’échantillons examinés dans ce manuscrit, pourvu que le chercheur est prêt à optimiser leur protocole de lyse avant d’effectuer des essais de plaque grand, plein. La technique réelle dépendra bien sûr de type tissulaire ; Cependant, en général le protocole doit utiliser un tampon de pH neutre contenant des concentrations physiologiques des sels ioniques, sans dénaturation des produits chimiques et préférence pas de détergent. Si un détergent doit être utilisé, les concentrations de détergent non ionique doivent être maintenues au minimum (par exemple pas plus de 1 %). La mémoire tampon doit également contenir des antiprotéases suffisante pour empêcher la dégradation protéolytique de protéines cibles. Quel que soit le protocole de lyse utilisé, les derniers préparatifs doivent être centrifugés pour enlever les particules avant l’analyse.
Au sujet des limites de dosage, les concentrations d’analytes cibles dans types d’échantillons biologiques peuvent varier grandement. Afin de correctement quantifier les concentrations de la substance à analyser, ils doivent être comprise entre les valeurs inférieure et supérieure pour la courbe d’étalonnage. Extrapolation des valeurs de la courbe n’est pas nécessairement fiable et n’est pas recommandé. Les enquêteurs doivent donc optimiser dilutions de leurs échantillons particuliers dans des expériences pilotes avant d’exécuter une analyse complète-plaque. En outre, la préparation minutieuse des échantillons biologiques à doser est primordiale pour le succès de ce format de test. Échantillons lipémiques, hémolysés, ou contenant des débris de la protéine seront affectent les interactions anticorps antigène qui définissent le principe essentiel de ce test immunologique et restituer des résultats ininterprétables.
Ce test est significatif en ce qui concerne les méthodes de quantification existantes pour plusieurs raisons. Lorsque exécuté correctement, le format du test peut doser jusqu'à 13 analytes auprès d’un échantillon en utilisant des volumes beaucoup plus faible qu’il faudrait analyser l’échantillon à l’aide des tests ELISA traditionnelles. Ce format d’essai ne nécessite pas instrumentation dédiée afin d’être exécutée. Il s’agit d’un avantage par rapport aux autres immunologiques perle disponibles dans le commerce comme le dosage peut être exécuté à l’aide de n’importe quel nombre de cytomètres couramment utilisés.
Une enquête scientifique sur le rôle joué par les cytokines et facteurs sécrétés réseaux en santé et la maladie se développe. Le format de test décrit dans ce manuscrit peut être un outil précieux pour les chercheurs qui cherchent à comprendre ces phénomènes complexes et multiformes. Programmes de recherche biomédicale active commencent à explorer le rôle des réseaux de cytokines de l’inflammation tels que généralisés des zones non seulement6, mais aussi en contexte des États de maladie spécifiques telles que l’athérosclérose, le cancer et neuro-inflammation 15,16,17. Sans aucun doute, enquête va continuer à croître dans ces domaines comme la recherche de nouvelles thérapies traiter ces affections chroniques se développe. La nécessité d’une quantification précise et fiable des médiateurs solubles associés à ces maladies demeurera une priorité de recherche critique.
Déclarations de divulgation
Les auteurs sont employés par BioLegend, qui fabrique les réactifs décrits dans ce manuscrit. Vigene Tech, Inc. a développé le logiciel d’analyse de données LEGENDplex qui est utilisé pour analyser les données générées par le test décrit dans ce manuscrit.
Remerciements
Les auteurs tiennent à souligner les contributions des biomarqueurs et Immuno-essais équipe de développement de produits pour le développement du dosage. En outre, nous tenons à remercier Vigene Tech, Inc. pour leurs efforts de collaboration dans la conception de progiciels des analyse les données.
matériels
| Name | Company | Catalog Number | Comments |
| Allegra 6R Centrifuge with MICROPLUS Carrier Adaptor | Beckman Coulter | 366816 | For LEGENDplex assays is run in microtubes, V-or U-bottom 96-well plate (optional) |
| LEGENDplex Mouse T helper Cytokine Panel | BioLegend | 740005 | Multiplex Immunoassay (13-plex) |
| Anti-mouse CD3ε antibody | BioLegend | 100314 | Clone 145-2C11, low endotoxin, azide free format |
| Anti-mouse CD28 antibody | BioLegend | 102112 | Clone 37.51, low endotoxin, azide free format |
| Vacuum Pump | EMD Millipore | WP6111560 | For LEGENDplex assays using filter plates (recommended) |
| Vacuum Manifold | EMD Millipore | MSVMHTS00 | For LEGENDplex assays using filter plates (recommended) |
| Sterile Disposable Reagent Reservoirs | Fisher Scientific | 07-200-130 | Or equivalent |
| Polypropylene MicroFACS Tubes | Fisher Scientific | 11-842-90 | For LEGENDplex assays is run in microtubes, V-or U-bottom 96-well plate (optional) |
| Pipette Kit | Fisher Scientific | 14-388-100 | Four pipette sizes (0.2-2µL, 2-20µL, 20-200µL, 100-1000µL) |
| Multi-Channel Pipette | Fisher Scientific | FA10011G | capable of dispensing 5 μL to 200 μL |
| Microplate Shaker | Fisher Scientific | 88880023 | Or equivalent |
| Microcentrifuge | Fisher Scientific | 75002410 | Or equivalent |
| FACS tubes | Fisher Scientific | NC9885747 | 12 x 75 mm round bottom |
| PMA (Phorbol 12-myristate 13-acetate) | Sigma-Aldrich | P8139 | |
| Lipopolysaccharide (LPS) | Sigma-Aldrich | L-8274 | Escherichia coli O26:B6 |
| Ionomycin | Sigma-Aldrich | I0634 | |
| Branson B200 Ultrasonic Cleaner | Sigma-Aldrich | Z305359 | Or equivalent |
| Microcentrifuge tubes | Sigma-Aldrich | Z666505 | 1.5 mL polypropylene tubes |
| Flow Cytometer | Various | Various | Cytometer equipped with single laser (488nm blue) or two lasers (488nm blue or 532nm green + 633nm Red) capable of reading emission wavelengths at 575nm and 660nm |
| 96-Well Polypropylene Plate | VWR | 82050-662 | For LEGENDplex assays is run in microtubes, V-or U-bottom 96-well plate (optional) |
Références
- Stanley, A. C., Lacy, P. Pathways for cytokine secretion. Physiology (Bethesda). 25, 218-229 (2010).
- Nicola, N. A. Cytokine pleiotropy and redundancy: a view from the receptor. Stem Cells. 12, (1994).
- Miyajima, A., Hara, T., Kitamura, T. Common subunits of cytokine receptors and the functional redundancy of cytokines. Trends Biochem Sci. 17, 378-382 (1992).
- Costantini, S., Castello, G., Colonna, G. Human Cytokinome: a new challenge for systems biology. Bioinformation. 5, 166-167 (2010).
- Capone, F., et al. Serum Cytokinome Profile Evaluation: A Tool to Define New Diagnostic and Prognostic Markers of Cancer Using Multiplexed Bead-Based Immunoassays. Mediators Inflamm. , 3064643(2016).
- Schett, G., Elewaut, D., McInnes, I. B., Dayer, J. M., Neurath, M. F. How cytokine networks fuel inflammation: Toward a cytokine-based disease taxonomy. Nat Med. 19, 822-824 (2013).
- Tse, E., Kwong, Y. L. T-cell lymphoma: Microenvironment-related biomarkers. Semin Cancer Biol. 34, 46-51 (2015).
- Reale, M., Greig, N. H., Kamal, M. A. Peripheral chemo-cytokine profiles in Alzheimer's and Parkinson's diseases. Mini Rev Med Chem. 9, 1229-1241 (2009).
- Vistnes, M., Christensen, G., Omland, T. Multiple cytokine biomarkers in heart failure. Expert Rev Mol Diagn. 10, 147-157 (2010).
- Gobel, K., et al. Blood coagulation factor XII drives adaptive immunity during neuroinflammation via CD87-mediated modulation of dendritic cells. Nat Commun. 7, 11626(2016).
- Palm, A. K., Friedrich, H. C., Kleinau, S. Nodal marginal zone B cells in mice: a novel subset with dormant self-reactivity. Sci Rep. 6, 27687(2016).
- Yu, Y., et al. The transcription factor Bcl11b is specifically expressed in group 2 innate lymphoid cells and is essential for their development. J Exp Med. 212, 865-874 (2015).
- Pelly, V. S., et al. IL-4-producing ILC2s are required for the differentiation of TH2 cells following Heligmosomoides polygyrus infection. Mucosal Immunol. 9, 1407-1417 (2016).
- LEGENDplex Multiplex Assays. , Available from: https://www.biolegend.com/legendplex (2017).
- Autieri, M. Pro- and anti-inflammatory networks in atherosclerosis. ISRN Vascular Medicine. 2012, (2012).
- West, N. R., et al. Emerging cytokine networks in colorectal cancer. Nat Rev Immunol. 15, 615-629 (2015).
- Becher, B., Spath, S., Goverman, J. Cytokine networks in neuroinflammation. Nat Rev Immunol. 17, 49-59 (2017).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon