Method Article
Créer et appliquer une référence afin de faciliter la Discussion et la Classification des protéines dans un groupe diversifié
Dans cet article
Résumé
Ce protocole vise à développer une référence pour les protéines divergentes dans un groupe qui n’a pas de critères cohérents pour la classification et la nomenclature. Cette référence facilitera l’analyse et la discussion du groupe dans son ensemble et peut être utilisée en plus de noms établis.
Résumé
Protéines apparentées qui ont été étudiés dans des laboratoires différents utilisant des différents organismes n’ont pas un système uniforme de la nomenclature et classification, rendant difficile de discuter de l’ensemble du groupe et de placer de nouvelles séquences dans le contexte approprié. Développer une référence qui donne la priorité aux fonctionnalités importantes séquences liées à structure et/ou de l’activité peut être utilisée en plus des noms établis pour ajouter quelque cohérence à un groupe diversifié de protéines. Cet article utilise la super-famille des (CS-αβ) hélice alpha cystéine stabilisé à titre d’exemple pour montrer comment une référence générée en tableur peut clarifier les relations entre les protéines existantes dans la superfamille, ainsi que faciliter l’ajout de nouveaux séquences. Il montre également comment la référence peut aider à affiner les alignements de séquences générées dans les logiciels couramment utilisés, dont l’incidence sur la validité des analyses phylogénétiques. L’utilisation d’une référence sera probablement plus utile pour les groupes de protéines qui incluent des séquences très divergentes d’un vaste éventail de taxons, avec des fonctionnalités qui ne sont pas suffisamment couverts par les analyses moléculaires.
Introduction
Nom de la protéine devrait refléter est caractéristiques et relation à d’autres protéines. Malheureusement, les noms sont généralement affectés au moment de la découverte et que les recherches se poursuivent, la compréhension de l’ensemble des contextes peut-être changer. Cela peut conduire à plusieurs noms si une protéine a été identifiée séparément par plus d’un laboratoire, à des changements dans la nomenclature ou dans les caractéristiques considérés comme définitif lorsque vous affectez le nom et le nom n’est plus suffisamment différenciant la protéine d’autres personnes.
Les défensines invertébrés offrent un bon exemple de la dégénérescence dans la nomenclature et la classification. Les premiers défensines d’invertébrés ont été signalés par les insectes, et le nom « defensin insecte » a été proposée basée sur l’homologie perçue à mammifères défensines1,2. Le terme defensin est toujours utilisé, même s’il est maintenant clair que défensines invertébrés et mammifères ne partagent pas un ancêtre commun3,4. Selon les espèces, un invertébré « defensin » peut avoir six ou huit des cystéines (qui forment trois ou quatre ponts disulfures) et une variété d’activités antimicrobiennes. Pour compliquer la situation, protéines ayant les mêmes caractéristiques que les défensines ne sont pas toujours appelés « défensines, » tels que le cremycins récemment identifié de Caenorhabditis remanei5. En outre, défensines gros invertébrés sont plus susceptibles de concerner évolutionnaire vertébrée β-défensines que d’autres invertébrés défensines6. Malgré cela, les chercheurs s’appuient parfois sur le nom « defensin » pour déterminer quelles séquences devraient être inclus dans les analyses.
Des études structurales ont révélé la similitude entre insectes défensines et scorpion toxines7, et le pli de CS-αβ a été établi par la suite comme la caractéristique structurelle de l’insecte défensines8. Cette bergerie définit la superfamille (CS-αβ) scorpion toxine dans la Classification structurale des protéines (SCOP) base de données9, qui comprend actuellement cinq familles : insectes défensines, toxines scorpion à chaîne courte, toxines de longue chaîne scorpion, MGD-1 (à partir de mollusque) et plante défensines. Cette superfamille est synonyme de la cis-défensines décrit récemment4 et super-famille 3.30.30.10 dans le CATH/Gene database 3D10,11. Études de divers invertébrés, plantes et champignons que les noms des protéines qui contiennent cette bergerie ne sont pas clairement liées à afficher numéro de cystéine ou dessin de collage, activité antimicrobienne ou histoire évolutive12.
Le manque de cohérence et de critères clairs rendent difficile de nommer et classer les séquences vient d’être identifié dans cette superfamille. Un obstacle majeur à comparer les protéines dans cette superfamille est que cystéines sont comptés à l’égard de chaque séquence individuel (la première cystéine dans chaque séquence est C1), sans aucun moyen pour expliquer le rôle structurel. Cela signifie que seules les séquences avec le même nombre de cystéines peuvent être comparés. Il y a peu conservation de séquence autre que les cystéines formant le pli de CS-αβ, ce qui complique les alignements et les analyses phylogénétiques. En développant un système de numérotation qui priorise les caractéristiques structurales, superfamille séquences peuvent être plus facilement par rapport et alignés. Caractéristiques conservées, ainsi que ceux définissant les sous-groupes, peuvent être visualisées rapidement, et de nouvelles séquences peuvent être placés plus facilement dans le contexte approprié.
Cet article utilise un tableur (par exemple, Excel) pour générer une référence à la numérotation de la super-famille des CS-αβ. Il montre comment cela clarifie les comparaisons entre les séquences et l’applique aux nouvelles séquences de CS-αβ identifiés des tardigrades. À l’aide de la super-famille des CS-αβ à titre d’exemple, le protocole a été écrit pour fournir des orientations pour l’utilisation de séquences d’intérêt ; Cependant, il n’est pas prévu pour être précis de cette superfamille ou de séquences riches en cystéine. Cette méthode sera probablement plus utile pour les groupes de protéines qui ont été étudiés séparément dans des taxons divergentes et/ou ont peu homologie de séquence globale, ayant des caractéristiques distinctes qui peut ne pas être facilement reconnu par le logiciel d’analyse moléculaire. Cette méthode requiert certaines décisions a priori au sujet des caractéristiques importantes, il sera d’une utilité limitée si aucune caractéristiques importantes n’ont été identifiées. L’objectif principal est de montrer comment une visualisation simple des relations séquence peut être atteints. Cela peut ensuite servir à informer l’alignement de séquences et d’analyse, mais si l’alignement et l’analyse sont les principaux objectifs, une méthode de code à barres serait une alternative appropriée qui a plus de capacité pour l’automatisation,13. La méthode actuelle affiche les caractéristiques de chaque peptide sous une forme linéaire, donc il ne sera pas utile pour la visualisation directe de la structure 3D.
Protocole
1. déterminer les caractéristiques de définition du groupe de protéines d’intérêt
- consulter des publications antérieures pour déterminer s’il existe un consensus sur les fonctions qui sont nécessaires pour être considérés comme faisant partie du groupe. Prendre note des incohérences ou divergences d’opinion entre les groupes de recherche et comprennent des caractéristiques qui peuvent servir à différencier un sous-groupe de l’autre.
- Si la littérature antérieure ne traite pas les caractéristiques qui définissent, utiliser des séquences qui sont considérés comme représentatifs du groupe comme point de départ pour identifier les caractéristiques conservées.
2. Collecter les séquences
- si les commentaires ont été écrits, qui comprennent des analyses de séquences qui représentent le groupe, inclure ces séquences dans le jeu de données brute. Récupérer les séquences à l’aide de numéros référencés dans la littérature et enregistrer dans une séquence standard, programme d’édition (par exemple, EditSeq dans la suite Lasergene ou un des nombreux disponibles pour gratuit en ligne).
- Si le groupe en question a été défini dans l’une des bases de données structurelles, comprennent les séquences de que la base de données répertorie comme faisant partie du groupe. récupérer les séquences à l’aide de numéros fournis dans la base de données et l’enregistrer dans un ordre standard d’édition programme, comme indiqué ci-dessus.
NOTE : par exemple, les séquences regroupées autour de la super-famille des CS-αβ (toxine ressemblant scorpion) dans la base de données de la SCOP se trouvent ici : http://scop.mrc-lmb.cam.ac.uk/scop/data/scop.b.h.c.h.html. - effectuer base locale Alignement Search Tool (BLAST) 14 recherches dans des bases de données publiques, en ligne, structurelles ou disponibles via le Centre National pour Biotechnology Information (NCBI) de trouver des séquences qui peuvent n’avoir pas été inclus dans la littérature bases de données. Pour la plupart résultats complets, utilisez les deux la protéine BLAST (blastp) et traduit souffle avec programmes de protéine de requête (tblastn) ; Ce sont tous deux disponibles à : https://blast.ncbi.nlm.nih.gov/Blast.cgi.
- Des séquences d’utilisation connus pour faire partie du groupe d’intérêt sous forme de séquences de la requête. Copiez-collez la séquence dans la zone de recherche en haut ou fournir un identificateur de numéro ou gi d’adhésion GenBank, si elles sont disponibles.
- Choisir la base de données dans le menu déroulant. Choisissez les séquences de protéines non redondant (nr) pour blastp et étiquettes de séquence pour tblastn exprimées.
- Recherche de résultats dans des taxons particuliers dans l’organisme en entrant l’organisme ou le nom du taxon et en choisissant dans la liste qui s’affiche lors de la frappe. Pour ajouter des organismes complémentaires ou taxons d’exclure, cliquez sur le " + " touche un autre champ s’affiche. Exclure des taxons non désirées dans la zone de l’organisme en tapant le nom de l’organisme ou taxon, choisissant dans la liste qui s’affiche tout en tapant et en cochant le " Exclude " encadré sur la droite.
- Accéder aux paramètres supplémentaires en cliquant sur " les paramètres d’algorithme " près du bas de la page. Laisser par défaut sauf s’il existe une justification permettant de modifier un paramètre.
- Cliquez le " BLAST " bouton pour exécuter l’analyse, il peut prendre un certain temps pour que les résultats apparaissent. En général, récupérer des hits avec une valeur s’attendre (ou e-value) de " -05 " ou mieux et sauvegarder dans une séquence standard, programme d’édition.
- Si toutes les réponses sont au-dessus de ce seuil, exécutez de nouveau la recherche avec un nombre accru de séquences cibles (dans la section de paramètres d’algorithme) pour obtenir toutes les séquences pertinentes.
- Si nécessaire, coupez les séquences pour exclure les informations non pertinentes (p. ex., le pli de CS-αβ s’applique uniquement au peptide mature). Identifier les peptides signaux et pro-peptides pour enlever en utilisant ProP 15 (disponible en ligne), ou SignalP pour sophistiquer signal peptide prédiction 16 (disponible en ligne).
3. Générer une référence à une feuille de calcul basé sur les importantes caractéristiques qu’ont été identifiées
- identifier les caractéristiques du groupe d’intérêt. Par exemple, utilisez le pli de CS-αβ définitivement établi par la structure de solution d’insecte defensin A du Phormia terraenovae ( Figure 1) 8.
- Cette bergerie comprend un motif plus petit appelé l' hélice stabilisée cystéine (CSH) 17 ; identifier ce motif par un CXXXC (où X est n’importe quel acide aminé) en amont d’un CXC qui forment deux ponts disulfures ( Figure 1 , solid pink lines).
NOTE : Pour compléter le motif CS-αβ, un troisième pont disulfure est formé à partir des cystéines supplémentaires placés devant chaque moitié du motif CSH ( Figure 1, pointillé des lignes roses).
- Cette bergerie comprend un motif plus petit appelé l' hélice stabilisée cystéine (CSH) 17 ; identifier ce motif par un CXXXC (où X est n’importe quel acide aminé) en amont d’un CXC qui forment deux ponts disulfures ( Figure 1 , solid pink lines).
- Entrer dans ces définissant les caractéristiques sur une feuille de calcul. Voir la Figure 2.
- Colonnes d’utilisation pour les éléments conservés et pour représenter les espaces entre ces caractéristiques. Garder les colonnes assez larges pour adapter les numéros et s’assurer qu’ils ont une largeur uniforme. Réglez la largeur avec la " Format | Largeur de colonne " fonction ( Figure 2, Flèche Rose).
- Utiliser les lignes pour le nom de la séquence.
- Lorsqu’une séquence a la particularité, renseignez la zone à l’aide de la fonction de remplissage ( Figure 2, carré rose). Pour l’espacement entre les fonctions, entrez le nombre d’acides aminés dans la zone entre et le laisser vide. Par exemple, à l’aide de l’insecte defensin séquence donne une référence qui comprend six cystéines, avec espacement défini entre C2 et C3 et entre C5 et C6.
- Ajouter des séquences représentant qui ont été précédemment établis en tant que membres du groupe basé sur les bases de données structurelles et de la littérature.
NOTE : par exemple, la littérature antérieure et la base de données de la SCOP identifient plusieurs groupes pour l’inclusion : insectes défensines, toxines scorpion à chaîne courte, toxines de longue chaîne scorpion, MGD-1 plante défensines, nématode ABFs, drosomycins de drosophile, et macins. La littérature recense aussi une séquence bactérienne avec seulement quatre cystéines qui pourraient représenter l’ancêtre de cette superfamille 18. L’ajout de ces séquences augmente le nombre des cystéines dans le renvoi de six à dix, mais maintient l’alignement des éléments structurels importants ( Figure 3).- Pour ajouter une fonctionnalité qui est susceptible de définir un sous-groupe de séquences (par exemple, une cystéine supplémentaire), utilisez le " Insert " fonction ( Figure 3, la flèche rose).
- S’il existe des fonctionnalités manquantes à partir d’une séquence donnée, laissez la case vide et combinez-le avec boîtes représentant les acides aminés intervenants. Si nécessaire, fusionner les cellules à l’aide de la fonction fusionner et centrer ( Figure 3, boîte rose).
- Continuer ajoutant des séquences aux groupes pour obtenir une meilleure image de la variation dans chaque groupe de la superfamille des plus grande. Résumer les caractéristiques de groupe pour faciliter les comparaisons ( Figure 4).
- Lorsque le nombre d’acides aminés entre les principales caractéristiques varie, utilisez un trait d’Union pour indiquer une plage, par exemple les 6-12 (acides aminés de 6 à 12) et une barre oblique pour indiquer / ou, par exemple 7/10 (7 ou 10 acides aminés).
- Choisir une voie pour annoter les caractéristiques des séquences qui peuvent être utiles mais ne sont pas assez souvent à inclure dans la référence. Par exemple, puisque cystéines sont importants dans cette superfamille, étiquette supplémentaires cystéines ( Figure 4, boîtes de roses).
- Ajouter newlséquences de y-identifiés dans la feuille de calcul utilisant les séquences établis comme guide. Par exemple, ajouter des séquences de tardigrades (jaune) montre que les séquences tardigrade appartiennent à différents groupes de la superfamille ( la Figure 5 présente des résumés au lieu d’une ligne pour chaque séquence aux fins de l’espace).
- Présentent une variabilité au sein d’un groupe taxonomique en réorganisant les lignes ( Figure 6).
4. Utiliser la référence à affiner les alignements d’acides aminés
Remarque : il existe de nombreux programmes qui peuvent être utilisés pour les alignements multiples de séquences, mais cette démonstration utilise l’analyse moléculaire de la génétique évolutive (MEGA6) 19 car il est disponible pour téléchargement gratuit.
- Télécharger et installer le logiciel.
- Commencer un nouveau tracé en MEGA en sélectionnant " Edit/Build alignement " sous l’onglet alignement Select " créer un nouveau tracé de la " dans la boîte qui apparaît, puis cliquez sur " OK. " puis sélectionnez " Protein. "
- Select " Insérez la séquence du fichier " dans le " Edit " menu permettant d’importer les séquences de.
NOTE : Séquences devront être au format FASTA pour importation dans MEGA. Les couleurs d’arrière-plan qui représentent les types de différents acides aminés sont utilisés par défaut, mais cette option peut être désactivée en vertu de la " écran " menu. - Une fois que toutes les séquences sont entrés, cliquez sur l’icône de bras fléchis, puis " protéine aligner " pour aligner les séquences à l’aide de l' algorithme MUSCLE 20.
Remarque : ClustalW est également disponible.- Si un message disant que rien n’a été sélectionné pop et souhaite sélectionner tout, cliquez sur " OK. "
- Remarque : cela ouvre une fenêtre qui permet de modifier certains paramètres, mais ils devraient seulement être changés il y a raison de le faire. Cette analyse utilise un sous-ensemble des séquences analysées dans un précédent fascicule 12.
- Contrôle l’alignement fondé sur les caractéristiques importantes ; Notez que la barre supérieure au-dessus des séquences affiche toutes les colonnes où l’acide aminé est complètement conservée (*). Voir Figure 7. Voir que l’alignement initial ne montre que trois des quatre cystéines conservées ( Figure 7, boîtes de roses) ; regardant vers le bas de la colonne, la séquence AlCRP est clairement mal alignées ( Figure 7, Flèche Rose).
- De se débarrasser de l’écart important entre le j’et le C conservé, sélectionnez les tirets et appuyez sur la " supprimer " clé. Pas en évidence des acides aminés, ou ils seront ainsi supprimés.
- Pour déplacer des acides aminés vers la droite, mettez en surbrillance et appuyez sur la barre d’espace.
- Remarque que le AlCRP a maintenant les cystéines structurels alignés et que le dernier C du motif CXXXC est conservée tout au long de l’alignement ( Figure 8). Ajuster l’alignement nécessaire de hiérarchiser les éléments les plus importants des séquences.
5. Comparer les groupes identifiés à l’aide de la référence aux résultats d’Analyses phylogénétiques
- d’alignements préliminaires, déterminer quelles séquences doivent être inclus dans une analyse phylogénétique ; pour un petit nombre de séquences, cette étape peut n’est pas nécessaire.
- Garder un fichier alignement comprenant toutes les séquences, mais pour une analyse phylogénétique, supprimer les séquences redondantes ( Figure 9, rose boîtes Voir la paires de séquences redondantes).
- Si le jeu de données comprend un grand nombre de séquences, exécuter une analyse préliminaire et certains représentants de groupes qui a toujours forment un clade.
- Déterminer le meilleur modèle de substitution des acides aminés.
- Exporter l’alignement au format MEGA (sous l’onglet données).
- Allez dans le menu modèles, puis sélectionnez " trouver meilleur ADN/protéine modèle. " choisir le fichier juste sauvé et ouvrez-le ; Ceci ouvrira une fenêtre qui a certains paramètres qui peuvent être changées.
- Utiliser les paramètres par défaut sauf si il y a une raison de les changer. Cliquez sur " calculer " pour commencer l’analyse.
- Exécuter une analyse de probabilité maximale (ML) en MEGA.
- Choisir " arbre de probabilité maximale construction/Test " dans le menu de phylogénie.
- Choisir le modèle déterminé à être le meilleur ajustement pour les données de l’étape 5.2 (la sortie donnera le modèle de substitution ainsi que le meilleur " taux des sites " paramètre).
- Choisir 1 000 bootstrap réplique pour obtenir les mesures de soutien pour l’arbre.
- Click " calculer " pour exécuter l’analyse ; MEGA a un " Tree Explorer " de visualiser l’arborescence.
- Exécuter une analyse bayésienne dans MrBayes logiciels libres 21.
Remarque : Un manuel de MrBayes est également disponible sur ce site. Ceci est destiné à fournir des étapes de base et n’est pas un guide complet pour l’analyse phylogénétique bayésienne.- Exporter l’alignement méga format PAUP (Nexus) dans le même dossier que le programme MrBayes.
- Open MrBayes et type " exe nom de fichier " (par exemple, " exe Alignment.nex ").
- Spécifier les paramètres de modèle et de l’analyse. Choisir soit le modèle spécifié à l’étape 5.2 ou choisir les " mixtes " qui sera essayer différents modèles et de faire rapport à la fréquence du modèle dans les arbres avec les meilleures probabilités postérieures (prset aamodelpr = mixte). Type " showmodel " de déclarer les paramètres actuels du modèle et " aider mcmc " pour afficher les paramètres actuels, avec une brève explication de chaque.
- Définir le nombre de générations à l’aide de la " mcmcp ngen = " commande (1 million est typique).
- Type " mcmc " pour commencer l’analyse.
- Lorsque le nombre de générations est terminée, le programme vous demandera d’ajouter plusieurs générations. Si l’écart moyen des fréquences de split est inférieure à 0,1, tapez no. Si c’est au-dessus de 0,1, l’analyse devrait être autorisée à continuer ou certains paramètres doivent être changés (voir le manuel).
- Utiliser la " sumt " commande pour générer l’arborescence des fichiers.
- Lorsque l’analyse est terminée et un arbre de consensus est généré, l’arbre peut être visualisé dans FigTree (disponible en ligne).
- Comparer les arbres pour voir si les méthodes génèrent des résultats cohérents.
Remarque : Certaines séquences ne fournissent pas beaucoup d’informations : les arbres ne soit pas bien résolues et les branches peuvent avoir un minimum de soutien ( Figure 10). - Comparer les arbres pour les groupes identifiés à l’aide de la référence pour voir si les analyses phylogénétiques des ces groupes de soutien.
Résultats
Groupes de séquences de la super-famille des CS-αβ rapportées dans la littérature sont affichés dans la Figure 4. Les appariements de cystéine basées sur la numérotation pour chaque séquence suggèrent cinq groupes principaux (tableau 1, colonne du milieu). Le groupe 1 a 6 cystéines qui de disulfure de trois obligations et comprend des séquences d’insectes, les arachnides, les mollusques, les nématodes et les champignons. Les groupes 2, 3 et 4 ont 8 cystéines qui forment quatre ponts disulfures. Groupe 2 comprend les insectes, arachnides et séquences d’usine ; Groupe 3 comprend les arachnides, mollusque et séquences de nématodes ; et regroupe 4 séquences de cnidaires, annélides, mollusques et les champignons. Groupe 5 comprend les 10 macins de cystéine. Certaines séquences ne correspondait pas tout à fait ces patrons, mais ont été généralement plus près d’un groupe que les autres.
Groupes 1 et 2 semblent partager deux liaisons : C2-C5 et C3-C6 ; Cependant, commencer la numérotation de chaque séquence avec sa première cystéine ne reconnaît pas le contexte structural des obligations. C2-C5 dans le groupe 1 des séquences formulaires, l’un des deux liens dans le motif CSH, tandis que le C2-C5 en groupe 2 séquences forme la liaison finale nécessaire pour stabiliser le pli de CS-αβ. La liaison homologue pour le groupe 1 C2-C5 est Group2 C3-C6, qui n’est pas évident, d’après la numérotation. Il n’est pas aussi évidente que dans le groupe 3, la liaison C2-C6 joue le même rôle structurel.
En utilisant des séquences de la littérature produite une référence avec un total de dix cystéines. Le motif CSH est formé à partir des obligations C3-C8 et C9-C4, avec C2-C6 complétant le pli de CS-αβ. Renumérotation des paires de cystéine basés sur des chiffres de référence précise les liens présents dans chaque séquence (tableau 1, colonne de droite). Il est maintenant évident que toutes les séquences de C2-C6, C8-C3 et C4-C9, reflétant le pli structural qui définit la superfamille. L’utilisation d’une référence permettant une comparaison facile entre les séquences qui ont nomenclature incompatible et critères de classification ambigu. Il peut également aider à identifier les caractéristiques qui définissent un sous-groupe de séquences. Par exemple, la liaison C1-C7 peut différencier macins d’autres membres de la superfamille, rendant approprié de classer les séquences avec ce lien comme « macins » plutôt que « défensines » (tableau 1 et Figure 4).
La fouille de bases de données publiques en ligne révélé seize séquences de tardigrades qui ont clairement le CS-αβ plier, huit de Hypsibius dujardini et Milnesium tardigradum. Quatre des nouvelles séquences ont six cystéines, neuf ont huit, on a neuf et deux ont dix. Ce qui donne très peu d’information, mais en alignant les séquences à la référence, il devient clair que tardigrade séquences avec le même nombre de cystéines n’ont pas toujours les cystéines structurellement important au même endroit dans la séquence ( Figure 5 et Figure 6). L’alignement avec la référence permet également l’inférence de la liaison des modèles (tableau 2, déduit des modes de liaison entre parenthèses). Certaines des séquences tardigrade fit clairement modèles 1-4. D’autres sont plus semblables à l’ancêtre bactérien proposée, scorpion Cl-toxine ou une famille de peptides de type defensin fongiques. Modèle 2 peut avoir deux sous-groupes, l’un représenté par scorpion Na + toxines, drosomycin et plante défensines et l’autre par scorpion Cl-toxines. Poursuite des travaux sur la fonction des protéines tardigrade sont nécessaire pour déterminer si certains devraient être considérés toxines plutôt que des défensines.
Les analyses phylogénétiques sont souvent utilisés pour étudier comment un groupe de protéines peut-être avoir évolué. Les séquences de la super-famille des CS-αβ sont généralement courtes et très divergentes ; les arbres qui en résultent sont souvent mal résolus et offrent peu d’indications. Arbres les ML et bayésienne pour le sous-ensemble des séquences analysées ici étaient mal résolus, avec le faible soutien de nombreux clades (Figure 10, supplémentaire fichiers 1 - 4). C’est une pratique courante pour afficher uniquement les niveaux « bootstrap » plus de 70 (ou probabilités postérieures sur 0,7), mais Figure 10 conserve tous les numéros pour démontrer les globalement faibles niveaux de soutien. Cinq groupes ont été pris en charge au-dessus de 70/0,7 dans au moins un des deux arbres : (a) un 6c et une toxine de scorpion 8C ; (b) macins ; tiques (c) et le scorpion défensines ; (d) plante défensines ; et (e) 6C défensines d’insectes, d’arachnides et de mollusques. Dans l’arborescence de ML, clade e comprend également une toxine 8c et un 8C tardigrade defensin, mais le soutien a été très faible (Figure 10 a). En général, ceux-ci reflètent les catégories définies à l’aide de la numérotation de cystéine de référence mais montrent également que les séquences avec des nombres différents de cystéine dans un grand groupe taxonomique peuvent être plus étroitement liés que les séquences avec le même modèle de différents groupes. Alors que seul un petit nombre de séquences ont été utilisé dans cette étude, une analyse de plus de 250 séquences n’élimine pas le manque de résolution (supplémentaire fichiers 5 - 8)12. L’alignement de référence de feuille de calcul peut offrir une visualisation plus facile des similitudes avec pertinence structurelle ou fonctionnelle par rapport à des arbres phylogénétiques.
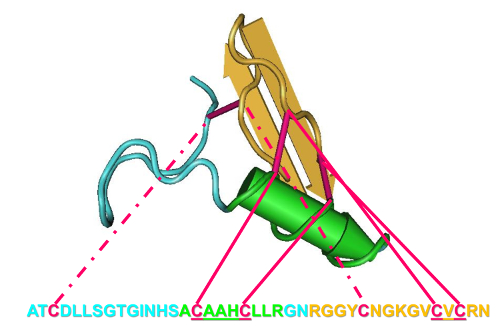
Figure 1 : Définir la séquence et les caractéristiques structurales de la superfamille des CS-αβ. Acides aminés et la structure 3D sont codées par couleur : boucle (bleu), alpha-helix (vert), bêta-feuilles (or) et les liaisons disulfide (rose). S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 2 : Six-cystéine question PREJUDICIELLE basé sur la séquence des insectes Defensin. Les colonnes indiquent les cystéines conservées (C1-C6) et, pour le motif CSH, le nombre de conservé amino acides entre les cystéines. Les boîtes pleines indiquent que la séquence a la cystéine donnée et les nombres indiquent acides aminés entre les cystéines. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
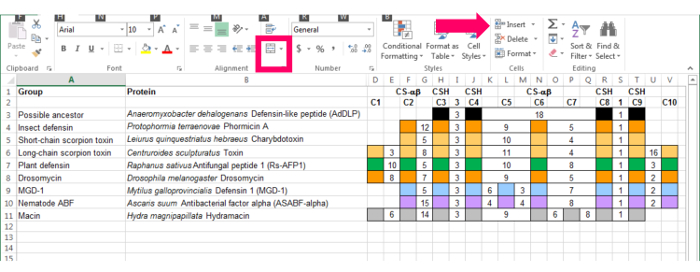
Figure 3 : Raffiné Ten-cystéine référence basée sur les séquences représentant des groupes de la superfamille des CS-αβ. Les colonnes indiquent les cystéines conservées et les acides aminés entre eux. Cystéines qui contribuent au motif CSH (C3, C4, C8 et C9) et au bercail CS-αβ (C2 et C6) sont étiquetés. Séquences sont couleur par groupe taxonomique : arachnides (orange clair), bactéries (noir), cnidaires (gris), Hexapoda (orange), mollusques (bleu), Nematoda (violet) et Plantae (vert). S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
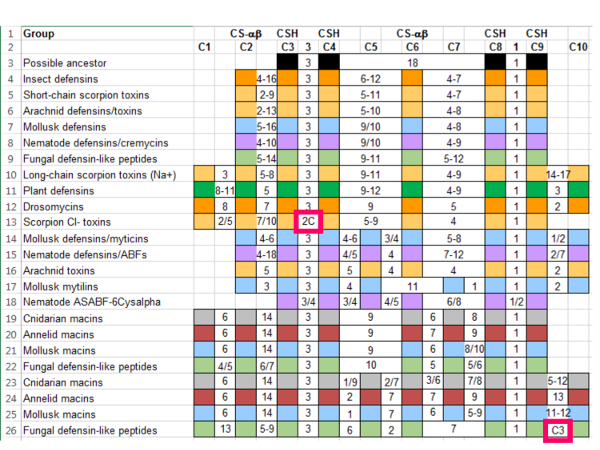
Figure 4 : Résumé de CS-αβ superfamille des séquences alignées avec référence de caractéristiques d’un groupe. Les colonnes indiquent les cystéines conservées et les acides aminés entre eux. Cystéines qui contribuent au motif CSH (C3, C4, C8 et C9) et au bercail CS-αβ (C2 et C6) sont étiquetés. Séquences sont couleur par groupe taxonomique : Annelida (rouge foncé), Arachnida (orange clair), bactéries (noir), cnidaires (gris), champignons (vert clair), Hexapoda (orange), mollusques (bleu), Nematoda (violet) et les plantes (vert). Chiffres séparés par un tiret indiquent une gamme d’intermédiaires acides aminés ; chiffres séparés par un slash représentent soit / ou. Un « C » indique une cystéine supplémentaire qui n’intervient pas avec une fréquence suffisante pour justifier l’ajout de la référence. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
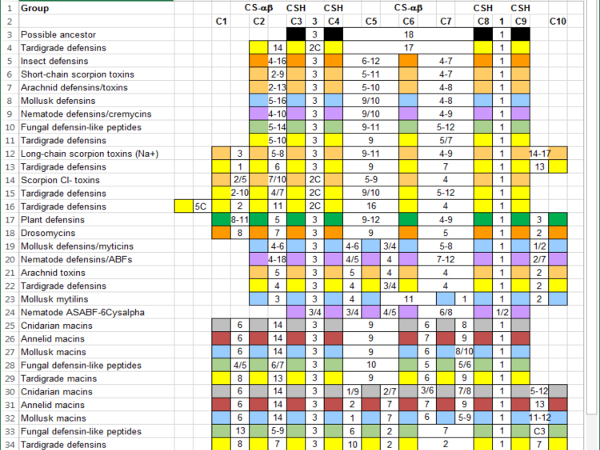
Figure 5 : Ajout de séquences de Tardigrade CS-αβ superfamille alignement avec référence de caractéristiques d’un groupe. Les colonnes indiquent les cystéines conservées et les acides aminés entre eux. Cystéines qui contribuent au motif CSH (C3, C4, C8 et C9) et au bercail CS-αβ (C2 et C6) sont étiquetés. Séquences sont couleur par groupe taxonomique : Annelida (rouge foncé), Arachnida (orange clair), bactéries (noir), cnidaires (gris), champignons (vert clair), Hexapoda (orange), mollusques (bleu), Nematoda (violet), Plantae (vert) et Tardigrada (jaune). Chiffres séparés par un tiret indiquent une gamme d’intermédiaires acides aminés ; chiffres séparés par un slash représentent soit / ou. Un « C » indique une cystéine supplémentaire qui n’intervient pas avec une fréquence suffisante pour justifier l’ajout de la référence. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
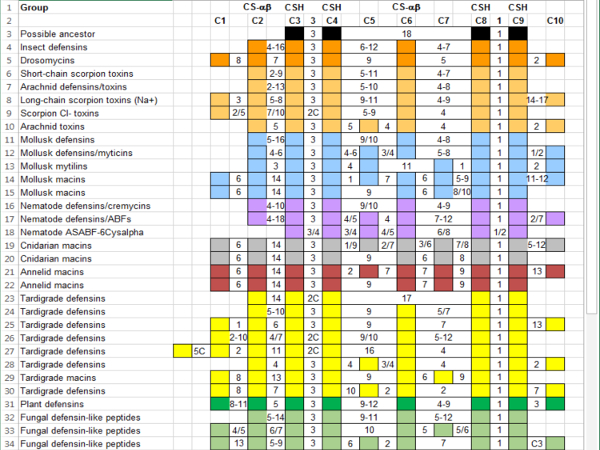
Figure 6 : Ajout de séquences de Tardigrade CS-αβ superfamille alignement avec référence par groupe taxinomique. Les colonnes indiquent les cystéines conservées et les acides aminés entre eux. Cystéines qui contribuent au motif CSH (C3, C4, C8 et C9) et au bercail CS-αβ (C2 et C6) sont étiquetés. Séquences sont couleur par groupe taxonomique : Annelida (rouge foncé), Arachnida (orange clair), bactéries (noir), cnidaires (gris), champignons (vert clair), Hexapoda (orange), mollusques (bleu), Nematoda (violet), Plantae (vert) et Tardigrada (jaune). Chiffres séparés par un tiret indiquent une gamme d’intermédiaires acides aminés ; chiffres séparés par un slash représentent soit / ou. Un « C » indique une cystéine supplémentaire qui n’intervient pas avec une fréquence suffisante pour justifier l’ajout de la référence. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 7 : à l’aide de la séquence mal alignée automatisé alignement. Acides aminés conservés dans toutes les séquences sont indiqués par * dans la ligne au-dessus de la première séquence (décrite dans les boîtes de roses). AlCRP n’est pas conforme. L’écart doit être retiré pour aligner correctement le C (flèche rose). S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 8 : Raffinement manuelle du tracé conserve les caractéristiques importantes structurellement des séquences. AlCRP est maintenant correctement alignée (flèche rose), et le motif CXXXC est entièrement conservé pour les séquences (boîtes de roses). S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
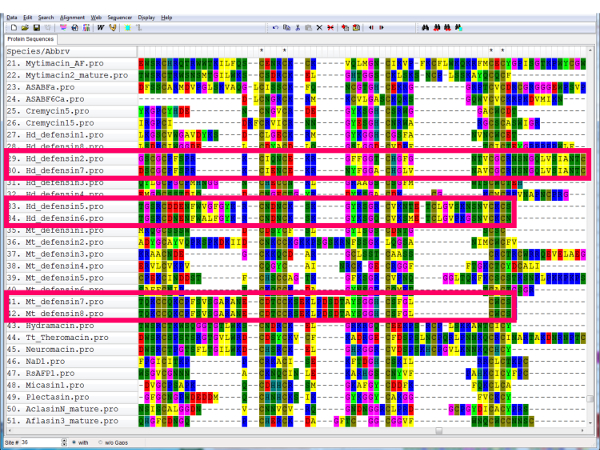
Figure 9 : Séquences redondantes dans un alignement. S’il y a des paires de séquences presque identiques (boîtes de roses), on peut être enlevé, puisque ceux-ci seront probablement toujours regrouper ensemble dans et contribuent peu à la topologie globale de l’arbre. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 10 : Comparaison des arbres produit des Analyses phylogénétiques. (A) Maximum analyse de probabilité en méga, avec 1.000 bootstrap réplique utilisant la WAG + G + j’ai le modèle. (B) une analyse bayésienne avec 1 000 000 générations en utilisant le paramètre de modèle mixte. Clades pris en charge à 70/0,7 sont indiquées en traits pleins de roses ; pointillés roses montrent des clades pris en charge à 70/0,7 dans l’autre arbre. b un 6c et une toxine de scorpion 8C ; (b) macins ; tiques (c) et le scorpion défensines ; (d) plante défensines ; et (e) 6C défensines d’insectes, d’arachnides et de mollusques. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Tableau 1 : groupes au sein de la super-famille des CS-αβ basée sur la structure d’appariement cystéine. Cinq modèles de base de formation de la liaison sont affichés à l’aide des numéros internes (colonne du milieu) ou les numéros de référence (colonne de droite). Toxines Scorpion Cl - ASABF 6Cys-alpha et un groupe de peptides fongiques sont placés sur le modèle que mproche possible de l’ost. Une cystéine non inclus dans la référence est indiquée par un exposant des cystéines avant/après (par exemple, C3/4 se situe entre C3 et C4).

Tableau 2 : ajout de Tardigrade CS-αβ séquences aux groupes de motif cystéine-appariement. Tardigrade défensines et macins (en gras) sont placés dans les groupes déjà établis lorsque c’est possible. Certaines séquences tardigrade peuvent montrer un modèle spécifique de groupe. Une cystéine non inclus dans la référence est indiquée par un exposant des cystéines avant/après (par exemple, C3/4 se situe entre C3 et C4). La notation « 2C1» indique il y a deux cystéines en amont de référence C1.
Supplémentaire fichier 1 (S1) : alignement de ce Dataset dans MEGA. S’il vous plaît cliquez ici pour télécharger ce fichier.
Fichier supplémentaire 2 (S2) : fichier méga arbre de Maximum de vraisemblance pour ce Dataset. S’il vous plaît cliquez ici pour télécharger ce fichier.
Fichier supplémentaire 3 (S3) : alignement de cet ensemble de données au Format de Nexus pour MrBayes. S’il vous plaît cliquez ici pour télécharger ce fichier.
Fichier supplémentaire 4 (S4) : fichier de Consensus de l’analyse MrBayes de ce Dataset. S’il vous plaît cliquez ici pour télécharger ce fichier.
Fichier supplémentaire 5 (S5) : alignement des séquences de CS-αβ 250 en MEGA. S’il vous plaît cliquez ici pour télécharger ce fichier.
Supplémentaires fichier 6 (S6) : arbre de probabilité maximale de 250 séquences CS-αβ. S’il vous plaît cliquez ici pour télécharger ce fichier.
Fichier complémentaire 7 (S7) : alignement de 250 CS-αβ séquences dans Nexus formater pour MrBayes. S’il vous plaît cliquez ici pour télécharger ce fichier.
Fichier complémentaire 8 (S8) : fichier de Consensus de l’analyse MrBayes de 250 séquences CS-αβ. S’il vous plaît cliquez ici pour télécharger ce fichier.
Discussion
Les critères pour nommer une protéine au sein d’un groupe doivent être clairs, mais ce n’est pas toujours le cas. Les séquences qui ont le CS-αβ plier ont été étudiés dans plusieurs laboratoires en utilisant une variété d’organismes, ayant pour résultat différents systèmes de nomenclature, ainsi que différents niveaux de qualification. Tentative d’imposer une toute nouvelle nomenclature n’est pas raisonnable et se traduirait par une grande confusion lors de la consultation de la littérature antérieure. Une référence système de numérotation peut être utilisée en plus du nom d’une protéine de préciser ses caractéristiques par rapport à la superfamille.
Groupes de protéines avec des critères clairs pour la dénomination et la classification seront probablement pas bénéficient de générer une référence à une feuille de calcul, même si il peut être utile de résumer un grand nombre de séquences et la visualisation des caractéristiques importantes. Logos et alignements de séquences sont utiles pour enquêter sur le niveau de conservation à chaque site, mais n’accordent-elles pas activement les caractéristiques de séquence importantes pour la structure ou la fonction. L’exemple de CS-αβ axée sur la structure, mais des acides aminés qui forment un site de liaison pourrait également être intégrées comme une caractéristique déterminante. Caractéristiques de séquence qui confèrent des activités antimicrobien/toxiques spécifiques des peptides CS-αβ sont identifiées, ces peuvent être ajoutés à la référence à préciser des groupes basés sur l’activité. Bien que seulement les peptides matures prédits ont été utilisés dans cet exemple, si la présence d’un peptide signal ou Pro-peptide est importante, cette information peut être ajoutée pour chaque séquence. Spécifiques d’insertion ou délétions, ainsi que des endroits intron, peuvent également être incluses si elles sont susceptibles d’être informatif. Un avantage d’utiliser MrBayes pour l’analyse phylogénétique est qu’il n’est pas limité aux données moléculaire-it peut analyser les données codant pour d’autres caractéristiques qui peuvent avoir une importance évolutive. Ceux-ci peuvent être codées comme présentes ou absentes, fournissant davantage d’informations que la séquence seule.
Collecter les séquences pertinentes est une étape cruciale du protocole. Selon la portée de l’étude et la répartition des membres du groupe, ceci pouvant couvrir les grands groupes taxonomiques. Si l’objectif est de comprendre tout un groupe de protéines, de considérer que certaines séquences peuvent survenir en dehors de l’espèce qui ils sont habituellement rapportées de. Si un taxon est déjà bien représenté et séquences supplémentaires sont redondantes ou improbables, leur exclusion de la recherche peut être approprié. Un base règle du pouce pour récupérer des hits dans une recherche BLAST est d’utiliser un seuil de -05 pour la valeur de e. La valeur de e est le nombre de résultats à attendre du hasard. Alors qu’elle est adaptée à certaines situations, s’il y a un groupe de séquences qui est très divergent mais actions caractéristiques spécifiques, il peut être moins fiable-it peut extraire des séquences qui ressemblent mais ne faire pas avoir voulu les caractéristiques, et il ne peut pas retourner les séquences qui ont les caractéristiques essentielles, mais qui sont très divergentes. Il y a des façons possibles d’aborder cette question. La première consiste à examiner les séquences identifiées dans la recherche qui sont sous le seuil de-05 pour déterminer s’il satisfait aux critères d’inclusion. Deuxièmement, s’il y a suffisamment d’informations, utilisez Position spécifique itérée BLAST (PSI-BLAST)22 ou Pattern-Hit a lancé BLAST (PHI-BLAST)23. PSI-BLAST utilise les résultats d’une recherche initiale pour générer un nouveau modèle pour le prochain tour et peut parfois trouver des séquences divergentes que la recherche initiale n’a pas identifié. PHI-BLAST nécessite un modèle être soumis avec la séquence requête. Cela limite les séquences récupérées à ceux contenant le motif d’intérêt. Cet outil est particulièrement utile si un motif unique pour le groupe peut être clairement identifié.
Un alignement précis est essentiel pour l’analyse phylogénétique ; interprétations des arbres n’est valables que lorsqu’ils sont générés à l’aide d’alignement bon. À l’aide de la référence d’informer l’alignement peut permettre d’éviter les erreurs qui ne sont évidentes que lorsque la structure ou l’activité sont considérés. Séquence de redondance devront être définis pour le projet. Deux séquences qui semblent redondants peut-être pas phylogénétique fins s’ils sont des taxons très divergentes ou sont presque identiques en séquence, mais ont des propriétés structurales ou fonctionnelles différentes. S’il y a ambiguïté au sujet desquelles les séquences doivent être inclus, alignements multiples peuvent être générés et analysés séparément pour voir comment l’alignement change inférences phylogénétiques d’impact. La méthode présentée ici n’élimine pas la nécessité pour le réglage manuel des alignements, mais il peut aider à préciser comment les séquences doivent être alignées et pourraient éventuellement être utilisés conjointement avec une technique plus sophistiquée de codage à barres a été décrite déjà13.
Pour la référence utile, il est important d’identifier les caractéristiques qui ne sont pas actuellement évidents à partir de la seule séquence. Par exemple, considérez l’impossibilité de comparer la cystéine collage de motifs entre les séquences avec des nombres différents de cystéines lors de chaque séquence est numéroté à l’égard de lui-même. L’objectif est de faciliter la comparaison et discussion, ne pas d’ajouter une autre couche de confusion. Ceci peut impliquer plusieurs itérations de la référence et arrêt appelle pour décider quelles fonctionnalités à inclure. Il est à espérer que l’adoption d’une méthode commune de discuter des séquences divergentes dans un groupe augmentera la compréhension du groupe dans son ensemble.
Déclarations de divulgation
L’auteur n’a rien à divulguer.
Remerciements
Peptide antimicrobien tardigrade en cours recherche est appuyée par un financement intra-muros du Bureau de recherche de l’Université du Midwest et parrainé des programmes (ORSP). L’ORSP n’avait aucun rôle dans la conception de l’étude, la collecte de données, analyse, interprétation ou préparation du manuscrit.
matériels
| Name | Company | Catalog Number | Comments |
| BLAST webpage | https://blast.ncbi.nlm.nih.gov/Blast.cgi | ||
| EditSeq (Lasergene suite) | DNASTAR | https://www.dnastar.com/t-allproducts.aspx | |
| Excel 2013 | Microsoft | ||
| FigTree | http://tree.bio.ed.ac.uk/software/figtree/ | ||
| MEGA | www.megasoftware.net | ||
| MrBayes | http://mrbayes.sourceforge.net/ | ||
| SCOP database | http://scop.mrc-lmb.cam.ac.uk/scop/ |
Références
- Matsuyama, K., Natori, S. Purification of Three Antibacterial Proteins from the Culture Medium of NIH-Sape-4, an Embryonic Cell Line of Sarcophaga peregrina. J Biol Chem. 263 (32), 17112-17116 (1988).
- Lambert, J., et al. Insect immunity: Isolation from immune blood of the dipteran Phormia terranovae. of two insect antibacterial peptides with sequence homology to rabbit lung macrophage bactericidal peptides. PNAS. 86 (262-266), (1989).
- Dimarcq, J. -. L., Bulet, P., Hetru, C., Hoffmann, J. Cysteine-rich antimicrobial peptides in invertebrates. Biopolymers. 47, 465-477 (1998).
- Shafee, T. M. A., Lay, F. T., Hulett, M. D., Anderson, M. A. The Defensins Consist of Two Independent, Convergent Protein Superfamilies. Mol Biol Evol. 33 (9), 2345-2356 (2016).
- Zhu, S., Gao, B. Nematode-derived drosomycin-type antifungal peptdies provide evidence for plant-to-ecdysozoan horizontal transfer of a disease resistance gene. Nat Commun. 5, (2014).
- Zhu, S., Gao, B. Evolutionary origin of b-defensins. Dev. Comp. Immunol. 39, 79-84 (2013).
- Bonmatin, J. -. M., et al. Two-dimensional 1H NMR study of recombinant insect defensin A in water: Resonance assignments, secondary structure and global folding. J Biomol NMR. 2 (3), 235-256 (1992).
- Cornet, B., et al. Refined three-dimensional solution structure of insect defensin A. Structure. 3 (5), 435-448 (1995).
- Murzin, A. G., Brenner, S. E., Hubbard, T., Chothia, C. SCOP: a structural classification of proteins database for the investigations of sequences and structures. J Mol Biol. 247, 536-540 (1995).
- Sillitoe, I., et al. CATH: comprehensive structural and functional annotations for genome sequences. Nucleic Acids Res. 43, 376-381 (2015).
- Lam, S. D., et al. Gene3D: expanding the utility of domain assignments. Nucleic Acids Res. 44, 404-409 (2016).
- Tarr, D. E. K. Establishing a reference array for the CS-ab superfamily of defensive peptides. BMC Res Notes. 9, 490 (2016).
- Shafee, T. M. A., Robinson, A. J., van der Weerden, N., Anderson, M. A. Structural homology guided alignment of cysteine rich proteins. SpringerPlus. 5 (27), (2016).
- Altschul, S. F., Gish, W., Miller, W., Myers, E. W., Lipman, D. J. Basic Local Alignment Search Tool. J Mol Biol. 215 (3), 403-410 (1990).
- Duckert, P., Brunak, S., Blom, N. Prediction of proprotein convertase cleavage sites. Protein Eng Des Sel. 17 (1), 107-112 (2004).
- Petersen, T. N., Brunak, S., von Heijne, G., Nielsen, H. SignalP 4.0:discriminating signal peptides from transmembrane regions. Nat Methods. 8, 785-786 (2011).
- Kobayashi, Y., et al. The cysteine-stabilized a-helix: A common structural motif of ion-channel blocking neurotoxic peptides. Biopolymers. 31, 1213-1220 (1991).
- Gao, B., del Carmen Rodriguez, M., Lanz-Mendoza, H., Zhu, S. AdDLP, a bacterial defensin-like peptide, exhibits anti-Plasmodium. activity. Biochem Biophys Res Commun. 387, 393-398 (2009).
- Tamura, K., Stecher, G., Peterson, D., Filipski, A., Kumar, S. MEGA6: Molecular Evolutionary Genetics Analysis. Mol Biol Evol. 30 (12), 2725-2729 (2013).
- Edgar, R. C. MUSCLE: multiple sequence alignment with high accuracy and high throughput. Nucleic Acids Res. 32 (5), 1792-1797 (2004).
- Ronquist, F., Huelsenbeck, J. P. MrBayes 3: Bayesian phylogenetic inference under mixed models. Bioinformatics. 19 (12), 1572-1574 (2003).
- Altschul, S. F., et al. Gapped BLAST and PSI-BLAST: a new generation of protein database search programs. Nucleic Acids Res. 25 (17), 3389-3402 (1997).
- Zhang, Z., et al. Protein sequence similarity searches using patterns as seeds. Nucleic Acids Res. 26 (17), 3986-3990 (1998).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon