Method Article
L'identification d'un épitope linéaire des cellules B monoclonales d'anticorps reconnus par le peptide à balayage assistée
Dans cet article
Résumé
Here, the authors present a simple and efficient protocol to define a linear antigenic epitope using a purified monoclonal antibody and peptide scanning through dot-blot hybridization. The identified epitope can then be used in therapeutic and diagnostic applications.
Résumé
L'identification d'un epitope antigénique par le système immunitaire permet la compréhension du mécanisme de protection d'anticorps neutralisants qui peuvent faciliter le développement de vaccins et de médicaments peptidiques. balayage peptidique est une méthode simple et efficace qui mappe carrément l'épitope linéaire reconnu par un anticorps monoclonal (mAb). Ici, les auteurs présentent une méthodologie de détermination de l'épitope impliquant des protéines recombinantes en série tronquées, la conception de peptides synthétiques et hybridation dot-blot pour la reconnaissance antigénique du virus de la nécrose nerveuse protéine d'enveloppe en utilisant un mAb neutralisant. Cette technique repose sur l'hybridation dot-blot de peptides synthétiques et des mAb sur un fluorure de polyvinylidène (PVDF). La région antigénique minimum d'une protéine d'enveloppe virale reconnue par le RG-M56 mAb peut être réduite par l'étape par étape taillée cartographie peptidique sur un épitope de peptide 6-mer. En outre, le balayage d'alanine mutagenèse et les résidus sousconstitution peut être effectuée pour caractériser l'importance de liaison de chaque résidu d'acides aminés constituant l'épitope. Les résidus flanquant le site d'épitope ont été trouvés à jouer un rôle critique dans la régulation peptide de conformation. Le peptide d'épitope identifié peut être utilisé pour former des cristaux de complexes peptide-anticorps épitope pour une étude de diffraction des rayons X et de la compétition fonctionnelle ou pour la thérapeutique.
Introduction
Dans le système immunitaire, la recombinaison V, D et J pour les anticorps segments permet de créer des variations considérables des régions déterminant la complémentarité (CDR) pour la liaison à des antigènes différents pour protéger l'hôte contre une infection pathogène. La défense de neutralisation des anticorps dirigés contre des antigènes dépend de la complémentarité spatiale entre les CDRs de l'anticorps et les épitopes des antigènes. Par conséquent, une compréhension de cette interaction moléculaire facilitera la conception d'un vaccin prophylactique et le développement peptide médicament thérapeutique. Cependant, cette interaction de neutralisation peut être influencée à la fois par de multiples domaines antigéniques à partir d'un seul antigène et par plusieurs CDR d'anticorps, ce qui rend par conséquent le procédé de détermination de l'épitope plus complexe. Heureusement, le développement de la technologie des hybridomes, qui fusionne les cellules productrices d'anticorps individuelles avec des cellules de myélome, permet de diviser un lot de cellules en permanence à secrete un anticorps spécifique, connu comme un anticorps monoclonal (mAb) 1. Les cellules d'hybridome produisent ces mAb, à haute affinité pures pour se lier à un domaine antigénique unique d'un antigène spécifique. La relation de l'antigène-anticorps mis en place, plusieurs approches, y compris le balayage des peptides, peut être utilisé pour déterminer l'épitope d'un antigène en utilisant son anticorps monoclonal correspondant. Les développements récents dans la technologie de peptide synthétique ont fait la technique de balayage de peptide plus accessible et plus commode d'exécuter. En bref, un ensemble de peptides synthétiques se chevauchant sont produites selon une séquence de l'antigène cible et sont associés à une membrane supporté sur un solide pour mAb hybridation. balayage Peptide non seulement offre un moyen simple pour cartographier la région de liaison de l'anticorps, mais facilite également l'acide aminé (aa) la mutagenèse par balayage de résidus ou de substitution pour évaluer l'interaction de liaison entre chaque résidu aa du peptide d'épitope et les CDR de l'anticorps.
Ici, la présente étude décrit un protocole pour l'identification efficace de l'épitope linéaire du mérou jaune virus de la nécrose nerveuse (YGNNV) protéine d'enveloppe en utilisant une neutralisation mAb 2, 3, 4. Le protocole comprend mAb préparation, la construction et l'expression de protéines recombinantes tronquées en série, la conception de peptides se chevauchant de synthèse, hybridation dot-blot, le balayage d'alanine et la mutagenèse par substitution. Compte tenu du coût élevé de la synthèse peptidique, l'étape consistant à tronquer en série les protéines recombinantes d'une protéine cible souhaitée a été modifiée, et la région antigénique a été réduit à environ 100 à 200 résidus aa avant la synthèse réseau peptidique analyse dot-blot a été réalisée.
Protocole
1. Préparation de l'anticorps monoclonal
- Culture les RG-M56 cellules hybridomes monoclonaux de souris 2 dans un milieu sans sérum dans des flacons de 175T à 37 ° C avec 5% de CO 2 supplément. Recueillir le surnageant lorsque la couleur du milieu devient jaune après cinq jours d'incubation.
REMARQUE: Les cellules d'hybridome sont cultivées dans un milieu exempt de sérum pour éviter la contamination anticorps à partir du sérum de foetus bovin. - Centrifuger le surnageant à 4500 xg pendant 30 min à 4 ° C et jeter le culot de débris cellulaires.
- Ajouter 2 ml d'agarose de protéine G (fourni sous forme d'une suspension à 50%) sur une colonne de 5 ml et on équilibre avec 10 volumes de résine (10 ml) de PBS glacé.
- Charge 200 ml du surnageant d'anticorps (étape 1.2) sur la colonne et jeter le pass-through.
- Ajouter 10 ml de PBS glacé à la colonne pour la laver. Répéter deux fois.
- Ajouter 10 ml de glycine 50 mM, pH 2,7 à la colonne pour éluer l'anticorps de la protéine G associée. Coltionner des fractions de 900 ul dans un tube de microcentrifugation contenant 100 pi de 10 x tampon de neutralisation (Tris 1 M, 1,5 M de NaCl et 1 mM d'EDTA, pH 8,0).
- Stocker l'anticorps purifié dans 50% de glycérol avec 0,03% de NaN3 à -20 ºC.
2. Construction et expression de protéines recombinantes tronquées en série
- Préparer un mélange de réaction PCR: 5 pi de tampon 10x Pfu, 0,2 mM de chaque dNTP, 0,2 uM d' amorce avant 3, 0,2 uM d' amorce inverse 3, 2 mM de MgSO 4, 1 ng de pET20b-1A59 ADN 3 de plasmide et 2,5 U ( unité) de l' ADN polymerase Pfu; ajouter ddH 2 O jusqu'à un volume final de 50 ul.
- échantillons exécuter dans un cycleur thermique automatique en utilisant les paramètres suivants: Cycle 1 (94 ° C pendant 5 min); cycles 2-36 (94 ° C pendant 30 s, 63 ° C pendant 30 s et 72 ° C pendant 60 s); et le cycle 37 (72 ° C pendant 7 min).
- Extraire la réaction en chaîne par polymérase (PCR) produits en utilisant un kit de purification PCR 5 pour faciliter la digestion par enzyme de restriction suivante.
- Digérer les fragments d'ADN amplifiés par PCR avec les enzymes de restriction Nde I et Xho I et ligaturé chacun de ces fragments d'ADN dans Nde I et Xho I enzyme clive pET-20b (+) vecteur. Transformer les constructions dans Escherichia coli cellules DH-5a-compétente 6.
- Préparer le mélange de digestion dans un tampon de digestion (20 mM de Tris-acétate, 10 mM de Mg (CH 3 COO) 2 mM KCH 50 3 COO, et 1 mM de DTT, pH 7,9) avec 1 pg d'ADN amplifié par PCR ou pET-20b (+) ADN vecteur et 2 U de restriction Nde I et Xho I enzymes dans un volume final de 20 ul.
- Mélanger le mélange de digestion en douceur et tourner rapidement vers le bas. Incuber à 37 ° C pendant 2 h dans un bain sec pour assurer la découpe complète de la restriction assises.
- Extraire les fragments d'ADN digéré par une enzyme de restriction en utilisant un kit de purification de PCR 5 afin de faciliter la construction du plasmide suivant.
- Préparer le mélange de ligature dans un tampon de ligature (Tris 66 mM, MgCl2 5 mM, ATP 1 mM et 5 mM de DTT, pH 7,5) avec 100 ng d'prédigéré ADN, amplifié par PCR, 10 ng de prédigéré pET-20b (+) ADN vecteur et 5 U d'ADN ligase T4 dans un volume final de 10 ul.
- Mélanger le mélange de ligature doucement et tourner rapidement vers le bas. Incuber à 16 ° C pendant 18 h dans un bain d'eau.
- Mettez 10 pi des échantillons de ligature dans 100 pi des cellules DH-5α-compétentes et mélanger doucement avant de placer le tube de microcentrifugation sur la glace pendant 30 min. Placer le tube à centrifuger dans un bain sec à 42 ° C pendant 90 s pour provoquer un choc thermique 7. Immédiatement transférer le tube sur de la glace pendant 2 min.
- Ajouter 900 ul de Luria-Bertani (LB) (1% de Bacto tryptone, 0,5% de Bacto levure Extract et 0,5% de NaCl, pH 7,0) dans le tube. Incuber à 37 ° C avec 150 rpm agitation pendant 45 min. Sédimenter les cellules par centrifugation à 4000 x g pendant 10 minutes et éliminer le surnageant.
- Remettre en suspension le culot avec 50 ul de bouillon LB et étaler chaque transformation sur des plaques pré-chauffé LB contenant 100 ug / ml d'ampicilline. Incuber les plaques à 37 ° C pendant 16 h.
- Ramassez une seule colonie en utilisant une pointe de 200 pi et le placer dans 3 ml de bouillon LB contenant 100 ug / ml d'ampicilline dans un lâche couvert 15 ml tube. Incuber à 37 ° C avec 150 rpm agitation pendant 12 h.
- Extraire l'ADN plasmidique 8 de chaque culture et de la séquence en utilisant le promoteur T7 et les amorces T7 terminator pour confirmer la séquence 9.
- Après confirmation de la séquence, transformer 10 ng d'ADN à partir de ces pET-20b (+) des plasmides avec des longueurs de YGNNV gène de protéine d'enveloppe variable dans la souche BL-21 (DE3) de E. coli en nousment la méthode de choc thermique 7. Suivez les étapes 2.3.6-2.3.8 pour effectuer la transformation.
- Transférer une seule colonie de E. coli transformée par BL-21 cellulaire dans 3 ml de bouillon LB contenant 100 ug / ml d' ampicilline dans un bouchée non hermétiquement 15 ml tube. Incuber la culture à 37 ° C avec 150 rpm agitation.
- On refroidit la culture à 25 ° C lorsque la DO600 de la culture est d' environ 0,6 et ajouter de l' IPTG à une concentration finale de 0,4 mM pour induire l'expression de la protéine recombinante. Incuber la culture pour un supplément de 4 h à 25 ° C avec 200 rpm agitation.
- Transférer 1 ml de la culture dans un tube de microcentrifugeuse. Sédimenter les cellules par centrifugation à 12 000 x g pendant 1 min et jeter le surnageant.
- Remettre en suspension le culot cellulaire dans 100 pi de tampon de dénaturation (8 M d'urée, du phosphate de sodium 20 mM et du NaCl 0,5 M, pH 7,4) par pipetage de haut en bas avec une micropipette. Mélanger par tourbillonnement vigoureux.
NOTE: L'échantillon de solutions devraient maintenant être semi-transparent, un peu collante, et prêt pour l'essai d'hybridation dot-blot.
3. Conception et synthèse de peptides chevauchants
- Conception et synthèse 3 série peptides 20-mères que chaque chevauchement avec son successeur par 10 résidus aa de la région 195-338 aa de la protéine d'enveloppe de YGNNV pour affiner la région d'épitope de RG-M56 mAb par point blot.
- Conception et synthétiser des peptides 3 trois 8-mer (195 VNVSVLCR 202, 197 VSVLCRWS 204 et 199 VLCRWSVR 206) avec un chevauchement de 6 résidus aa sur le prochain peptide synthétique pour affiner la région d'épitope de 195-206 aa par point blot.
- Conception et synthèse 3 7-mer (196 NVSVLCR 202 et 195 VNVSVLC 201), 6-mer (195 VNVSVL 200, 196 NVSVLC 201 et 197 VSVLCR 202) et 5-mères peptides (195 VNVSV 199, 196 NVSVL 200, 197 VSVLC 201 et 198 SVLCR 202) avec un chevauchement de 6, 5 et 4 résidus aa, respectivement, sur des leurs peptides voisins afin de minimiser la région de l'épitope par point blot.
4. Dot-blot Hybridation
- Dissoudre chaque peptide synthétisé dans du diméthylsulfoxyde (DMSO) à une concentration finale de 10 mg / ml.
Remarque: Afin de surmonter la solubilité varie de peptides synthétiques, les peptides de synthèse doit être dissous dans du DMSO. DMSO est un bon solvant pour dissoudre complètement les peptides hydrophobes ou hydrophiles. - Faire tremper la membrane de fluorure de polyvinylidène (PVDF) avec du methanol pendant 2 min.
NOTE: membrane de PVDF est jusqu'à résistante au DMSO 100%; d'autres peuvent ne pas être aussi. - Équilibrer la membrane de PVDF avec du tampon modifié Towbin (Tris 25 mM, glycine 192 et 0,1% de SDS, pH 8,3) pour2 min.
NOTE: 10-20% (v / v) de méthanol peut être ajouté à un tampon Towbin modifié afin d'améliorer les résultats de transfert. - Rincez un morceau de papier de chromatographie avec le tampon Towbin modifié. Placer la membrane PVDF sur le papier de Chromatographie. Attendre jusqu'à ce que le tampon Towbin modifié a disparu de la surface de la membrane de PVDF avant de passer à l'étape suivante.
- Ajouter 2 ul de chaque échantillon de peptide sur la membrane avec une pointe de 10 ul. Sécher à l'air de la membrane PVDF sur le papier de chromatographie pendant 10 min. Ajouter chaque échantillon de peptide lentement et progressivement sur la membrane pour éviter une trop grande diffusion.
- Bloquer la membrane dans du tampon TBST (0,05% (v / v) de Tween-20, Tris 20 mM et NaCl 150 mM, pH 7,4) avec 5% de lait écrémé pendant 30 minutes à température ambiante avec agitation douce.
- Ajouter le RG-M56 mAb à une dilution finale de 1: 1000 dans le tampon TBST à 5% de lait écrémé à la membrane. Incuber la membrane à 37 ° C pendant 1 h avec agitation douce.
- Retirer l'anticorps ainsirésolu-. Laver la membrane dans le tampon TBST pendant 5 minutes en agitant doucement. Répéter deux fois.
- Ajouter l'anticorps secondaire (chèvre anti-souris IgG Fc, la phosphatase alcaline conjuguée) à une dilution finale de 1: 5000 dans le tampon TBST à 5% de lait écrémé à la membrane. Incuber la membrane à 37 ° C pendant 1 h avec agitation douce.
- Jeter la solution d'anticorps. Laver la membrane dans le tampon TBST pendant 5 minutes en agitant doucement. Répéter deux fois l'étape de lavage.
- Développer la membrane avec une solution de substrat BCIP / NBT à la température ambiante pendant 15 min dans l'obscurité. Arrêtez le développement par lavage de la membrane avec ddH 2 O lorsque le signal apparaît.
- Sécher à l'air la membrane et capturer l'image de transfert en point en utilisant un système d'image.
- Mesurer l'intensité de chaque dot blot en utilisant un logiciel d'analyse d' image 3.
5. Numérisation Alanine et Substitution
- Concevoir et synthétiser 3 alanine et methides peptides de substitution onine. Remplacer chaque résidu aa avec alanine pour le peptide 8-mer 195 VNVSVLCR 202 et synthétiser des peptides 195 ANVSVLCR 202, 195 VAVSVLCR 202, 195 VNASVLCR 202, 195 VNVAVLCR 202, 195 VNVSALCR 202, 195 VNVSVACR 202, 195 VNVSVLAR 202 et 195 VNVSVLCA 202 . Remplacer résidu aa leucine 200 en méthionine pour créer le SJNNV génotype épitope peptide 195 VNVSVMCR 202.
- Suivez la section 4 pour effectuer un balayage de l'alanine et la substitution mutagenèse dot blot.
Résultats
Le but de cette expérience était d'identifier un épitope par dot blot en utilisant mAb. Pour affiner rapidement et efficacement en bas de la région antigénique reconnu par mAb, la pleine longueur et YGNNV protéines d'enveloppe recombinante en série tronquée avec une fusion tag 6xHis à l'extrémité C-terminale ont été exprimées à partir d' un système d'expression E. coli PET 10 (figure 1A). Les protéines recombinantes résultantes ont été repérés sur la membrane de PVDF en utilisant RG-M56 mAb et anti-6xHis anticorps pour hybridation dot-blot. Le blot de point a révélé des signaux positifs contre 1-338 aa (pleine longueur), 51-338 aa et 195-338 aa, mais pas 1-100 aa ou 1-200 aa protéines recombinantes (Figure 1B). La matrice de points hybridée contre l'anticorps anti-6xHis et a confirmé l'expression de toutes les protéines recombinantes. Ces données indiquent que l'épitope de reconnaissance RG-mAb M56 se trouve dans un 144-aa protéine recombinante near la protéine C-terminale de la couche YGNNV (195-338 aa).
Par la suite, série de peptides 20-mères avec 10 résidus aa-longue chevauche leur voisin a été conçu et synthétisé à partir de la séquence de 144 aa protéine recombinante pour l'analyse de peptides pour affiner la zone d'épitope. Ces peptides synthétiques ont été repérés sur une membrane de PVDF et soumis à hybridation dot-blot en utilisant RG-M56 mAb. Le résultat a montré que des signaux positifs sur le peptide 195-214 aa et le témoin positif, 195-338 aa protéine recombinante (figure 2A). Que l'épitope est situé dans le peptide 195-214 aa région, mais pas le peptide de la région 205-224 aa, trois séries de peptides 8-mères avec des résidus de 6 aa aa chevauchement à partir des résidus 195-206 (peptides 195-202 aa 197- 204 aa et 199-206 aa) ont été conçus et synthétisés. les résultats d'hybridation Dot-blot ont montré des signaux positifs sur le peptide 195-202 aa et le positif de contrôle peptide 195-214 aa utilisantRG-M56 mAb (figure 2B).
Balayage Alanine et la substitution mutagenèse ont été réalisées pour évaluer la spécificité de chaque résidu aa de l'épitope 8-mer, 195 VNVSVLCR 202. Chaque résidu aa du peptide 8-mer a été remplacé individuellement par l'alanine. L'alanine réseau mutation peptide a ensuite été placé sur une membrane de PVDF en utilisant le peptide 195-202 aa comme témoin positif. Analyse Dot-blot a indiqué que les trois remplaçant mutations, V197A, V199A et C201A, abolies l'affinité de liaison du RG-M56 mAb (figure 3A). Bien que aa résidu 200 dans l'épitope SJNNV génotype est la méthionine, par opposition à une leucine dans les quatre autres épitopes de génotype Betanodavirus, la séquence SJNNV génotype, 195 VNVSVMCR 202, a montré une affinité de liaison positive contre RG-M56 mAb, comme celle du contrôle positif (figure 3A). Ce résultat indique que les épitopes of tous les génotypes Betanodavirus peuvent être reconnus par RG-M56 mAb. L'affinité de liaison de chaque résidu aa participant au site épitopique a en outre été quantifié en mesurant l'intensité de signal du point de transfert de chaque substitution d' alanine en utilisant un logiciel d'analyse d'images (figure 3B). Les intensités des V197A, V199A, et des substitutions de C201A ont été réduites à 10,2%, 18,6% et 8,5%, respectivement, par rapport à ceux du témoin positif (100%), alors que le V195A, S198A, L200A, R202A, et substitutions L200M ont montré des intensités plus élevées ou similaires comme celui du contrôle positif. Il convient de noter que la force influant sur la substitution N196A est ambiguë, avec une réduction de 37,4% dans le témoin positif. Ces résultats indiquent que V197, v199, et C201 sont des résidus essentiels pour la liaison du RG-M56 mAb.
Les résultats balayage d'alanine de mutagenèse révèle que les résidus aa V195, N196 et R202 peutêtre remplacé par une alanine, ce qui implique que la région d'épitope pourrait encore être rétréci vers le bas aux deux extrémités. Par conséquent, l'étape par étape taillée cartographie peptidique à 7-mer, 6-mer, et 5-mer étape de peptides synthétiques a été conçu pour réduire au minimum la région antigénique. Le signal positif était présent sur le peptide synthétique 6-mer 196 NVSVLC 201, mais pas sur les 7-mer et 5-mères peptides synthétiques (Figure 4). Ces données indiquent que l'épitope minimal de NNV protéine d'enveloppe reconnue par RG-M56 mAb est le peptide 6-mer, 196 NVSVLC 201.
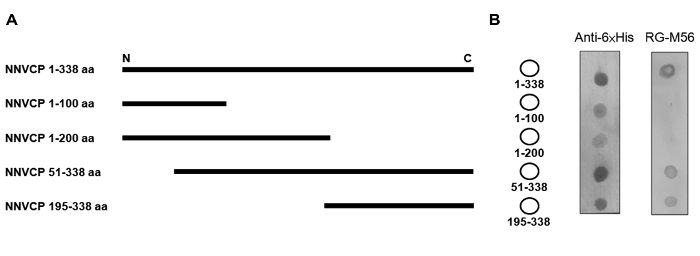
Figure 1: Réduction de la région d'épitope en utilisant des protéines recombinantes en série tronqués et un anticorps monoclonal. (A) Carte de NNVCPs recombinantes en série tronquées. NNVCP 1-338 aa est la protéine d'enveloppe pleine longueur. (B) Dot-bloAnalyse t NNVCPs de recombinaison. Gauche: Carte des YGNNV manteau protéines recombinantes en série tronqués sur la membrane de PVDF. Moyen: L'analyse dot-blot a été réalisée en utilisant un anticorps anti-6xHis. Droit: L'analyse dot-blot a été réalisée en utilisant RG-M56 mAb. NNVCP: virus nerveux de nécrose protéine d'enveloppe; aa: acide aminé; PVDF: polyfluorure de vinylidène; N: extrémité N-terminale; C: C-terminale; mAb: anticorps monoclonal. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.

Figure 2: Cartographie fine de la région d'épitope en utilisant des peptides synthétiques. (A) A gauche: les séquences d' acides aminés des peptides synthétiques 20-mères ont été alignés sur NNVCP 195-338 aa; chaque peptide précédent avait un 10 aa-résidus de long chevauchement avec le peptide suivant. Droite: Carte de la synthetic peptides sur la membrane de PVDF. Recombinant NNVCP 195-338 aa été utilisé comme témoin positif. L'analyse dot-blot a été réalisée en utilisant RG-M56 mAb. (B) A gauche: des séquences d'acides aminés des peptides synthétiques 8-mères, 195-202 aa, 197-204 aa et 199-206 aa; chaque peptide précédent avait un 6 aa résidus de long chevauchement avec le peptide suivant. Peptide synthétique 195-214 aa été utilisé comme témoin positif. Droit: L'analyse dot-blot a été réalisée en utilisant RG-M56 mAb. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.

Figure 3: numérisation Alanine et la substitution mutagenèse du 195 VNVSVLCR 202 épitope. Séquences (A) d' acides aminés de mutagenèse de substitution et de l' analyse dot-blot. Chaque aa residue de la région d'épitope de 195 à 202 aa, a été remplacé par l'alanine individuellement. la substitution L200M est la séquence de génotype SJNNV dans la région d'épitope de la protéine d'enveloppe Betanodavirus. Peptide synthétique 195-202 aa été utilisé comme témoin positif. Les résidus aa remplacés sont soulignés. (B) Quantification de l'affinité de liaison de signal dot blot. L'intensité du signal du témoin positif (aa 195-202) a été déterminée comme étant de 100%. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.

Figure 4: Détermination de l' épitope minimum. Le 7-mer (A), 6-mer (B), et 5-mer (C) peptides synthétiques 195-202 aa ont été utilisés pour identifier l'épitope minimum reconnu par RG-M56 mAb. Pepti synthétiquede 195-202 aa été utilisé comme témoin positif. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
Discussion
Ce protocole offre une technique simple et rapide pour identifier un épitope linéaire mAb reconnu. Prenant en considération le coût de la synthèse peptidique et l'efficacité des peptides de synthèse de la production, de la région antigénique de la protéine d'enveloppe du virus a été réduite par l'expression de protéines recombinantes tronquées en série avant l'analyse de balayage de peptides. En tant que tel, le système d'expression pET de E. coli fiable et efficace a été utilisé pour produire ces protéines recombinantes tronquées en série, comme des protéines recombinantes ayant des poids moléculaires compris entre 10 et 50 kDa peuvent être facilement exprimés par ce système. De cette manière, l'épitope peut être facilement réduit à une région plus facile à gérer aa 100-200. Le vecteur pET-20b (+) a été choisi spécifiquement, car il contient une séquence qui code pour l'expression de 6xHis-étiquettes, permettant ainsi aux protéines produites 6xHis-tag fusion à immunodétectées à l'aide d'un anticorps anti-6xHis pour confirmer l'expression de la p recombinantroteins. Les protéines d'enveloppe recombinantes produites ont ensuite été analysées en utilisant le RG-mAb M56 par l'intermédiaire d'un test d'hybridation dot-blot. Une autre méthode de détermination de l' épitope de la protéine recombinante est de purifier les protéines recombinantes exprimées par Chromatographie d'affinité sur ions métalliques immobilisés 10, séparer les protéines recombinantes par électrophorèse sur gel de polyacrylamide-SDS, et effectuer une analyse par transfert de Western 3.
De plus en plus finement cartographier l'emplacement de l'épitope déterminé par les résultats de l'analyse par hybridation dot-blot en utilisant des protéines recombinantes en série tronqués, des peptides synthétiques se chevauchant avec des tailles différentes ont été conçues. Parmi les différentes longueurs possibles de peptide synthétique pour synthétiser, 20-mer avec 10 aa-résidus de long peptides chevauchants ont été choisis d'abord dans le balayage de peptide, à la fois pour leur haute pureté de synthèse (à environ 90%) et leur longueur de peptide, assez pour la recherche de l'e continuepitope reconnu par l' anticorps des cellules B 11. Notez que la précision et la pureté des peptides synthétisés se détériorent que le peptide synthétisé est plus longue. De cette façon, la région d'épitope a été rapidement réduite à environ 10 résidus d'aa en longueur. Après série de peptides 8-mères chevauchants ont été étudiés, la région d'épitope linéaire a été défini au peptide 195-202 aa. Par la suite, le balayage d'alanine de la 8-mère épitope peptidique révèle la force de liaison d'affinité critique de chaque résidu aa, ce qui permet la recherche de l'épitope minimal. Les rôles essentiels de la V197, v199, et les résidus aa C201 implique que la région d'épitope linéaire comprend au moins 5 résidus aa, de 197 à 201. De plus, résidus aa V195, N196, et R202 peuvent être remplacés par l'alanine, sans perdre complètement la liaison affinité, ce qui indique que la région d'épitope peut être ramené à 7, 6, 5 ou même des résidus aa en longueur. séquences petits peptides peuvent être facilement et économiquement synthétisés pour la recherche linéaire (continuous) epitopes. Cependant, cette technique de balayage de peptide synthétique ne convient pas pour la détermination d'un épitope discontinu d'un anticorps, sauf si elle est combinée avec l' épitope excision et l' analyse spectrométrique de masse 12.
Dans ce protocole, une technique d'hybridation dot-blot a été utilisé pour rechercher l'épitope linéaire d'un mAb. Dot-blot hybridation est une méthode simple mais efficace. Au début de la recherche, lorsque la région d'épitope est rétréci vers le bas à partir d'un paysage à grande échelle, la principale préoccupation est d'observer soit un signal positif ou négatif après l'hybridation de l'anticorps à une protéine liée à la membrane cible, comme la plupart des mAb seulement se lier à un épitope spécifique d'une protéine antigénique (figures 1 et 2). Cependant, lorsqu'on utilise hybridation dot-blot pour explorer la disponibilité de liaison de chaque résidu aa dans la région d'épitope contre l'anticorps, par exemple par substitution d'alanine mutagénèse,l'intensité du signal de chaque résidu aa substitué déterminé par dot blot doit être pris en compte dans la signification globale de liaison. Que l' intensité du signal peut être facilement quantifié en utilisant le logiciel d'analyse d'images (figure 3B) ou d' un densitomètre. En variante, un dosage immuno-sorbant lié à une enzyme (ELISA) peut être réalisée afin de quantifier le degré d'affinité de liaison et la puissance du signal résultant 3.
Dans l'étude précédente, la seule protéine d'enveloppe du virus de la nécrose nerveuse non-enveloppe était immuno-reconnue par 10 mAb avec une valeur élevée de l' indice de neutralisation entre 6,5 à 4,5 (log 10 NI) 2. La capacité de reconnaissance hautement spécifique des anticorps monoclonaux ont encore été utilisés pour le développement de l'une étape, le kit de diagnostic rapide immunochromatographique pour la détection des poissons infectés NNV-13. L'épitope antigénique de nerveux protéine d'enveloppe du virus de la nécrose a été reconnu par le RG-M18mAb comme un peptide 8-mer, 195 VNVSVLCR 202 3, par lequel un nouveau récepteur NNV a été identifié (données non publiées). Dans la présente étude, l'épitope de la nécrose nerveuse protéine d'enveloppe du virus a également été réduit à un peptide 6-mer, 196 NVSVLC 201, par l'autre mAb, RG-M56.
Il est inattendu que deux peptides 7-mer (196 NVSVLCR 202 et 195 VNVSVLC 201) contenant 6-mer peptide 196 NVSVLC 201 ne sont pas reconnus par le RG-M56 mAb (figure 4A). Une interprétation raisonnable est que, bien que les résidus entourant V195 et R202 ne peuvent pas contribuer directement à l'interaction de liaison entre l'anticorps et l'épitope, les résidus flanquants influent sur la formation de la conformation peptidique approprié pour la reconnaissance de l'anticorps. L'apparition de V195 ou R202 à leur terminus flanquant seul peut contorsionner le peptide synthétiqueconformation pour la reconnaissance d'anticorps et la liaison. La conformation de l' épitope est entraîné par la force des deux extrémités, V195 et R202, qui sont équilibrés les uns contre les autres dans le peptide synthétique 8-mer, 195 VNVSVLCR 202, et contrecarré dans le peptide synthétique 6-mer, 196 NVSVLC 201. Les résidus aa, V195, N196 et R202, peuvent être remplacés individuellement par l'alanine sans complètement perdre la capacité de liaison, et donc, les résultats balayage d'alanine de mutagenèse indiquent que ces trois résidus aa ne peuvent pas jouer un rôle important dans la reconnaissance et la liaison des RG -M56 mAb. Cependant, après la coupe d' un plus résidu du peptide synthétique 6-mer, 196 NVSVLC 201, le peptide 5-mer, 197 VSVLC 201, sans le N196 dans la région N-terminale flanquant, perd la capacité d'être reconnu et lié par RG -M56 mAb (figure 4C). Ce résultat suggère que le résidu N196 peut aussi pjeter un rôle important dans la région adjacente de l'épitope pour stabiliser la conformation de l'épitope correct afin de faciliter la reconnaissance et la liaison de RG-M56 mAb. L'importance des résidus adjacents qui entourent la région d'épitope ont également été explorées par d'autres études de liaison antigène-anticorps. L'importance des résidus adjacents aa autour de la région d'épitope α-bungarotoxine pour la liaison des anticorps a été étudiée en utilisant différentes substitutions d'AA au sein du même sous-site cholinergique. Ils ont ensuite été évalués comme étant soit essentielle, influent, ou non influents 14. On a également constaté que la spécificité de l'épitope reconnu par les anticorps de mucine associée à un carcinome épithéliales peut être encore influencée par les résidus aa flanquantes. Ces effets peuvent présenter des obstacles conformationnels qui peuvent entraver la liaison d'un anticorps à un épitope 15.
L'épitope 6-mer, 196 NVSVLC 201 </ Sub>, a des caractéristiques très hydrophobes, avec quatre résidus hydrophobes, y compris deux Valines (197 et 199), une leucine (200), et une cystéine (201) (forme réduite). Résidus V197, v199, et C201 sont essentiels pour la reconnaissance RG-M56 mAb et contraignant, tel que déterminé par l'alanine mutagenèse par balayage. La région d'épitope est situé à l' un des huit brins ß antiparallèles du domaine de la coquille (S-domaine) de la protéine d'enveloppe 16 NNV. Fait intéressant, l'épitope ne semble pas sur le domaine de la saillie à l'extérieur, mais se cache dans la structure gelée-roll du S-domaine sous les autres brins ß anti-parallèles. L'épitope, avec sa haute hydrophobie, peut obtenir un microenvironnement plus stable dans cette dépression. Par ailleurs, le VNVSVLCR 195 202 3 peptidique de cet épitope a été trouvée pour empêcher la propagation du virus du mérou géant de la nécrose nerveuse du mérou dans les cellules du cerveau. Par conséquent, ce peptide epitope a été suggéré comme un competitor impliqués dans le domaine de liaison de récepteur nécessaire pour l' entrée virale 3. Il a été émis l' hypothèse que les inhibiteurs d'entrée des peptides comprenant des résidus hydrophobes et / ou amphiphiles peuvent altérer la conformation physique et la chimie des interfaces de la membrane cellulaire et peuvent empêcher la fusion des membranes cellulaires et virales 17. En outre, de nombreux inhibiteurs d'entrée de peptides synthétiques ont démontré de fortes propriétés inhibitrices contre diverses infections virales 17, 18. Ainsi, le peptide d'épitope identifié par des résidus hydrophobes et une forte inhibition de l'entrée contre l'infection NNV peut faciliter le développement de médicaments peptidiques thérapeutiques.
Déclarations de divulgation
The authors have no conflicts of interest related to this report.
Remerciements
The authors thank Miss Ching-Chun Lin and Miss Diana Lin of the Core Facility of the Institute of Cellular and Organismic Biology (ICOB) of Academia Sinica for offering their expertise on peptide synthesis and DNA sequencing, respectively. This study was supported by Academia Sinica.
matériels
| Name | Company | Catalog Number | Comments |
| Hybrid-SFM medium | Gibco | 12045-076 | |
| Dulbeccos's Phophate-Buffered Saline (PBS) | Gibco | 21600-069 | |
| Pfu DNA Polymerase | Thermo Scientific | EP0502 | Including buffers |
| T4 DNA Ligase | Roche | 10799009001 | Including buffers |
| NdeI | New England Biolabs | R0111S | Including buffers |
| XhoI | New England Biolabs | R0146S | Including buffers |
| pET-20b(+) vector | Novagen, Merck Millipore | 69739 | |
| E.coli DH-5α competent cell | RBC Bioscience | RH617 | |
| E.coli BL-21(DE3) competent cell | RBC Bioscience | RH217 | |
| Ampicillin | Amresco | 0339-25G | |
| LB broth | Invitrongen | 12780-052 | |
| Isopropylthio-β-D-thiogalactoside (IPTG) | MDBio, Inc. | 101-367-93-1 | |
| Methanol | Merck Millipore | 106009 | |
| Polyoxyethylene 20 Sorbitan Monolaurate (Tween-20) | J.T.Baker | X251-07 | |
| Dimethyl sulfoxide (DMSO) | Sigma | D2650 | |
| Glycine | Amresco | 0167-5KG | |
| Tris | Affymetrix, USB | 75825 | |
| NaCl | Amresco | 0241-1KG | |
| EDTA | Amresco | 0105-1KG | |
| Glycerol | Amresco | 0854-1L | |
| NaN3 | Sigma | S2002-500G | |
| BCIP/NBT | PerkinElmer | NEL937001PK | |
| Goat Anti-Mouse IgG, Fc fragment antibody | Jackson ImmunoResearch | 115-055-008 | |
| Immobilon-P (Polyvinylidene fluoride, PVDF) | Merck Millipore | IPVH00010 | |
| Protein G Agarose Fast Flow | Merck Millipore | 16-266 | |
| QIAquick PCR Purification kit | Qiagen | 28106 | |
| UVP BioSpectrum 600 Image System | UVP | n/a | |
| VisionWorks LS Analysis Software Ver 6.8 | UVP | n/a | |
| MyCycler thermal cycler | BioRad | 1709713 |
Références
- Milstein, C., Kohler, G. Clonal variations of myelomatous cells (proceedings). Minerva Med. 68 (50), 3453 (1977).
- Lai, Y. S., et al. In vitro neutralization by monoclonal antibodies against yellow grouper nervous necrosis virus (YGNNV) and immunolocalization of virus infection in yellow grouper Epinephelus awoara (Temminck & Schlegel). J Fish Dis. 24 (4), 237-244 (2001).
- Chen, C. W., Wu, M. S., Huang, Y. J., Cheng, C. A., Chang, C. Y. Recognition of Linear B-Cell Epitope of Betanodavirus Coat Protein by RG-M18 Neutralizing mAB Inhibits Giant Grouper Nervous Necrosis Virus (GGNNV) Infection. PLoS One. 10 (5), 0126121 (2015).
- Lai, Y. -. S., et al. Propagation of yellow grouper nervous necrosis virus (YGNNV) in a new nodavirus-susceptible cell line from yellow grouper, Epinephelus awoara (Temminck & Schlegel), brain tissue. J Fish Dis. 24 (5), 299-309 (2001).
- Lougee, E., Morjaria, S., Shaw, O., Collins, R., Vaughan, R. A new approach to HLA typing designed for solid organ transplantation: epityping and its application to the HLA-A locus. Int J Immunogenet. 40 (6), 445-452 (2013).
- Radulovich, N., Leung, L., Tsao, M. S. Modified gateway system for double shRNA expression and Cre/lox based gene expression. BMC Biotechnol. 11, 24 (2011).
- Froger, A., Hall, J. E. Transformation of plasmid DNA into E. coli using the heat shock method. J Vis Exp. (6), e253 (2007).
- Pronobis, M. I., Deuitch, N., Peifer, M. The Miraprep: A Protocol that Uses a Miniprep Kit and Provides Maxiprep Yields. PLoS One. 11 (8), e0160509 (2016).
- Metzker, M. L. Emerging technologies in DNA sequencing. Genome Res. 15 (12), 1767-1776 (2005).
- Chiu, C. C., John, J. A., Hseu, T. H., Chang, C. Y. Expression of ayu (Plecoglossus altivelis) Pit-1 in Escherichia coli: its purification and immunohistochemical detection using monoclonal antibody. Protein Expr Purif. 24 (2), 292-301 (2002).
- Atassi, M. Z. Antigenic structures of proteins. Their determination has revealed important aspects of immune recognition and generated strategies for synthetic mimicking of protein binding sites. Eur J Biochem. 145 (1), 1-20 (1984).
- Opuni, K. F., et al. Mass spectrometric epitope mapping. Mass Spectrom Rev. , (2016).
- Chang, C. Y., Chiu, C. C., Christopher John, J. A., Liao, I. C., Leaño, E. M. . The Aquaculture of Groupers. , 207-224 (2008).
- Conti-Tronconi, B. M., et al. Alpha-bungarotoxin and the competing antibody WF6 interact with different amino acids within the same cholinergic subsite. Biochemistry. 30 (10), 2575-2584 (1991).
- Briggs, S., Price, M. R., Tendler, S. J. Fine specificity of antibody recognition of carcinoma-associated epithelial mucins: antibody binding to synthetic peptide epitopes. Eur J Cancer. 29 (2), 230-237 (1993).
- Chen, N. C., et al. Crystal Structures of a Piscine Betanodavirus: Mechanisms of Capsid Assembly and Viral Infection. PLoS Pathog. 11 (10), e1005203 (2015).
- Badani, H., Garry, R. F., Wimley, W. C. Peptide entry inhibitors of enveloped viruses: The importance of interfacial hydrophobicity. Biochim Biophys Acta. , (2014).
- Qureshi, N. M., Coy, D. H., Garry, R. F., Henderson, L. A. Characterization of a putative cellular receptor for HIV-1 transmembrane glycoprotein using synthetic peptides. AIDS. 4 (6), 553-558 (1990).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon