Method Article
Automatisé de robotique Liquid Handling assemblage de dispositifs modulaires ADN
Dans cet article
Résumé
Est présenté ici, un workflow automatisé pour effectuer le montage de « dispositif » ADN modulaire en utilisant une méthode d’assemblage des ADN clonage modulaire sur les robots de manutention liquide. Le protocole utilise un outil logiciel intuitif pour générer des listes déroulantes gestionnaire liquide pour combinatoire génération de bibliothèque dispositif ADN, qui nous montrent l’utilisation de deux plates-formes de manipulation de liquides.
Résumé
Les progrès récents dans les techniques d’assemblage modulaires ADN ont permis à des biologistes synthétiques tester beaucoup plus de la vente « espace de conception » représenté par « devices », créés en tant que combinaisons des éléments génétiques distincts. Cependant, l’assemblage manuel de si grand nombre de périphériques est beaucoup de temps, erreurs et coûteux. La sophistication croissante et l’échelle de recherche en biologie synthétique nécessite une manière efficace et reproductible pour accueillir la construction du dispositif à grande échelle, complexe et haut débit.
Ici, un protocole d’Assemblée d’ADN en utilisant la technique modulaire clonage (MoClo) de Type-IIS endonucléase de restriction fondée est automatisé sur deux plates-formes robotiques liquide-manutention. Des robots automatisés de gestion liquide nécessitent attention, souvent fastidieuse optimisation des paramètres de pipetage pour liquides de différentes viscosités (p. ex. enzymes, ADN, l’eau, tampons), ainsi que la programmation explicite pour assurer une aspiration correcte et distribution des parties de l’ADN et réactifs. Cela rend manuel script écrit pour des assemblages complexes tout aussi problématiques que l’assemblage manuel ADN et nécessite un outil logiciel qui peut automatiser la génération du script. À cette fin, nous avons développé un outil logiciel web-basé, http://mocloassembly.com, pour générer des bibliothèques combinatoires de dispositif ADN de base ADN parties téléchargées sous forme de fichiers Genbank. Nous fournissons un accès à l’outil, et un fichier d’exportation de notre logiciel gestionnaire liquide qui comprend optimisé classes liquides, des paramètres de matériel de laboratoire et plan de pont. Toutes les parties de l’ADN utilisées sont disponibles par le biais de Addgene, et leurs cartes numériques sont accessibles via le registre de glace de Boston University BDC. Ensemble, ces éléments constituent une base pour d’autres organisations automatiser les expériences de clonage modulaires et protocoles similaires.
Le workflow automatisé de l’Assemblée des ADN présenté ici permet la production haut-débit, automatisée et reproductible des dispositifs de l’ADN et réduit le risque d’erreur humaine découlant de pipetage manuel répétitif. Données de séquençage montrent l’assemblage automatisé des ADN réactions générées à partir de ce flux de travail sont environ 95 % de réponses correctes et nécessitent que peu de 4 % comme du temps de beaucoup de pratique, par rapport à la préparation manuelle de réaction.
Introduction
Les dispositifs génétiques biologiques synthétiques plus tôt telles que le Collins interrupteur à bascule1 et la Elowitz repressilator2 a démontré que les systèmes biologiques pourraient être avant machinées pour avoir des fonctions spécifiques, déterministes. Depuis lors, les biologistes synthétiques ont s’est efforcé d’ingénieur systèmes vivants pour réaliser les fonctionnalités progressivement plus complexes au service de biomatériaux3, biothérapeutiques4,5,6, biocarburants 7,8et biodétection demandes9,10,11. Réaliser ces applications via combinant modulaire ADN « pièces » dans « devices » avec des fonctionnalités spécifiques a été l’un des principaux objectifs de la biologie synthétique. Pour ce processus à l’échelle, il doit y avoir une technique qui permet la création de dispositifs complexes de grandes bibliothèques de pièces dans un temps-efficace, rentable et surtout, façon reproductible.
Un tel processus d’Assemblée expansive est justifié parce que le domaine ne dispose pas actuellement une compréhension complète des règles guidant la conception des systèmes biologiques avec succès et la composition. Cette situation est aggravée par insuffisamment caractérisés ADN pièces12, manque de compatibilité et la composabilité des parties13et inattendues, indésirables les interactions entre les composantes génétiques au sein de dispositifs synthétiques14, 15. en l’absence de modélisation prédictive fiable, éléments génétiques synthétiques fonctionnels sont arrivés par essais et erreurs, qui exige des dizaines ou même des centaines de puissance du signal d’entrée-sortie variantes d’un dispositif prévu sont projetés et la « meilleure » est sélectionné pour la composition avec éléments en aval16. Même si moderne normalisées des modes d’assemblage ADN telles que Golden Gate17et clonage modulaire18,19,20 simplifier ce processus, un expert expérimental est toujours nécessaire pour effectuer chaque protocole. Comme périphériques synthétiques grandissent en taille et complexité, l’espace total disponible conception sera devenu trop grand pour construire et tester manuellement21et le processus sera trop artisanale pour des progrès significatifs qui sont réplicable à apporter dans le domaine.
Jusqu'à l’avènement de la biologie synthétique et référentiels partie biologiques telles que l’iGEM pièces Registre (http://partsregistry.org), les parties génétiques de l’inventaire JBEI d’éléments composables,22et23de SynBioHub, n’étaient pas stockés dans aucun format normalisé de l’Assemblée. Seulement une petite poignée de pièces nécessaires pour être cloné par projet et ainsi, le volume du clonage fait était faible et la réalisation d’un dispositif assemblé était réalisable et insignifiant par rapport aux objectifs réels de la recherche. Clonage moléculaire a été souvent ad hoc et exécutée à l’aide de recueils de restriction basées sur la disponibilité site et endonucléase de restriction, plutôt que suite à un processus normalisé. L’absence de normalisation rend impraticable pour automatiser n’importe quel protocole de clonage tel qu’il était peu probable que la prochaine réaction de clonage suivrait un protocole identique. Par ailleurs, automatiser l’assemblage d’ADN nécessaire investissement monétaire important dans l’équipement (liquide leur infrastructure connexe, logiciel et matériel de laboratoire et les robots de manutention) ainsi que l’investissement de temps pour la génération d’instructions exact paramètres pour gérer les différentes classes de liquides en cours de traitement et de la série précise d’instructions destinées à exécuter ces protocoles. À petite échelle des efforts de clonage ne justifiaient pas ces frais. La combinaison de modèles d’appareil génétique plus volumineux et plus complexes couplé avec montage normalisé protocoles24,25 crée un environnement où l’automatisation de ces processus est très pratique. Faible coût robotique comme la Opentrons OT-One26 apparaissent également qui permet même modeste financé par labs pour accéder à cette technologie. En outre, « nuage » laboratoires de27 dont Transcriptic et Emerald Cloud Lab mais aussi académique « biofoundries » tels que la fonderie de génome Edinburgh, l’iBioFab de l’UIUC, et la fonderie de MIT-Broad, harnais robotique pour assembler diverses séries de dessins pour une variété de clients rapidement et à plusieurs reprises tout en conservant un référentiel commun de base ADN primitives et des technologies d’assemblage pour les futures commandes.
Un des plus grands défis en automatisant le processus d’assemblage de l’ADN est la génération des commandes pipetage pour le gestionnaire de liquide. Tandis que les interfaces de logiciels pour ces appareils sont généralement faciles à utiliser, complexe pipetage instructions comme celles qui sont nécessaires pour l’assemblage de l’ADN combinatoire exigent le scientifique explicitement spécifier chaque aspiration et passer commande manuellement. Cela crée un goulot d’étranglement majeur dans le flux de travail et laisse le processus de génération de script vulnérable aux erreurs de pipetage mêmes comme si l’Assemblée ont été effectuée manuellement. Cela nécessite un outil logiciel qui permet d’automatiser toutes les parties de ce processus, de la conception de la bibliothèque de l’appareil, pour générer les instructions de pipetage et fournissant le chercheur avec les configurations de plaque/réactif requises pour assembler. Dans ce travail, nous misons sur notre outil de logiciel pour automatiser la conception d’une petite bibliothèque d’appareil combinatoire de l’ADN, ainsi que le mélange des réactifs (tampon, l’eau, enzymes) et des parties de l’ADN (Figure 1 b) en 96 un pot modulaire ADN Assemblée des réactions. Utilisation de l’outil ne nécessite aucune expérience en programmation préalable, est évolutif et haut débit et combinatoire par défaut. Nous montrent que clonage réactions préparés sur deux plates-formes différentes manipulation de liquides automatique rendement correct vérifier-séquence clones avec des fréquences comparables aux réactions préparé manuellement (95 %) et avec beaucoup moins de temps pratique.
Protocole
1. préciser les parties à utiliser dans l’ADN Device Library et générer des liquides/utilisateur gestionnaire d’Instructions [15 min]
- À l’aide de n’importe quel navigateur web, accédez à mocloassembly.com et télécharger des fichiers de Genbank pour toutes les parties de l’ADN qui seront inclus dans la conception de dispositif ADN combinatoire.
- Une fois que tous les fichiers ont été téléchargés, sélectionner des parties d’ADN désirées et glissez-les sur la toile vierge, plaçant les types de partie dans l’ordonnance définitive prévue des parties de l’ADN.
NOTE : Collections de pièces, ainsi que les parties séparées, peuvent être sélectionnées et placées sur la toile. Aussi, commander des pièces de l’ADN telles que 5' et 3' surplombe pour chaque match de la partie. - Cliquez sur « Monter » en bas à droite de la page.
Remarque : L’outil générera uniquement valables, constructibles assemblys basés sur les surplombs de quatre paires de bases qui entourent chaque partie à la digestion par l’enzyme BsaI. Si aucun périphérique ADN constructible n’existe basée sur les parties téléchargées par le scientifique, l’outil indique qu’aucun assembly n’ont été trouvés. -
Accédez à l’onglet « Plans » et télécharger des fichiers générés par l’outil. Ces fichiers comprendront :
- Plaque lisible des cartes pour le scientifique préparer des échantillons d’ADN, ainsi que les réactifs nécessaires pour les réactions
- Une « liste de choix » pour le gestionnaire de liquid
- Fichiers de Genbank entièrement annotés pour tous les périphériques de l’ADN à assembler
NOTE : Liquid handling bras positionnement lorsque vous accédez à tout nouveau matériel de laboratoire doit être testée. Consulter le manuel du fabricant pour obtenir des instructions détaillées sur la façon d’ajuster le bras pipetage dans le X, Y et Z de positionnement axes selon les besoins.
2. préparer l’ADN de plasmide et réactifs pour l’assemblage [3 jours]
- [Jour 1] À l’aide d’une anse à inoculation stérile et travaille près d’une flamme nue ou dans une hotte à flux laminaire, strie stocks bactérienne de glycérol sur LB-gélose additionnée de l’antibiotique approprié. Procéder ainsi pour toutes les pièces nécessaires d’ADN, en veillant à stériliser la boucle entre chaque échantillon. Incuber les plaques à 37 ° C pendant la nuit.
NOTE : Frozen bactérienne glycérol stocks devraient être conservés sur glace autant que possibles. Cycles de gel-dégel répétés abaisser la viabilité du stock et devraient être évitées. - [Jour 2] À l’aide d’une pointe de pipette stérile, un cure-dent ou Anse à inoculation et travailler près d’une flamme nue ou dans une hotte à flux laminaire, ensemencer 3 mL de bouillon LB (complété avec l’antibiotique approprié) avec une seule colonie des plaques LB-agar préparé en 2.1. Incuber les cultures pendant une nuit à 37 ° C en agitant à 300 tr/min.
- [Jour 3] Purifier l’ADN plasmidique des cultures bactériennes à l’aide de n’importe quel kit de purification de plasmide prép mini disponibles dans le commerce.
- En utilisant le fichier MoClo_Setup.xlsx fourni, diluer chaque échantillon d’ADN plasmidique à une concentration de 20 fmol/µL dans l’eau ou tampon TE.
- Suivant la carte de la plaque du fichier PDF généré par l’outil de montage, placer le volume de chaque partie d’ADN dilué dans le puits correspondant sur une plaque à 96 puits PCR-longe le plein. Maintenez cette SetupPlate sur la glace jusqu'à ce que nécessaire, ou sceller avec un opercule adhésif et conserver à-20 ° C.
-
Sur la glace, préparer la réaction mastermix comportant les éléments suivants : pour chaque 20 µL de réaction ajouter 2 µL de 10 x T4 DNA ligase tampon, 0,5 µL de T4 DNA Ligase (SC) et 1 µL d’enzyme BsaI. Une feuille de calculatrice est incluse dans le fichier MoClo_Setup.xlsx afin d’aider avec ceci.
- Distribuer l’enzyme mastermix dans les puits appropriés d’une nouveau plein-jupe PCR plaque à 96 puits, suivant le plan de la plaque pour la ReagentPlate dans le fichier PDF généré. Garder cette ReagentPlate sur la glace ou sur un froid-bloc de 96 puits.
Remarque : Le ReagentPlate doit être prêts lorsque vous êtes prêt à exécuter l’assembly sur le gestionnaire de liquid.
- Distribuer l’enzyme mastermix dans les puits appropriés d’une nouveau plein-jupe PCR plaque à 96 puits, suivant le plan de la plaque pour la ReagentPlate dans le fichier PDF généré. Garder cette ReagentPlate sur la glace ou sur un froid-bloc de 96 puits.
3. Exécutez le Script de l’Assemblée sur le gestionnaire Liquid [Variable]
- Placez le SetupPlate(s), le ReagentPlate(s) (sur un froid-bloc de 96 puits) et le nombre nécessaire de longe le plein vides plaques PCR 96 puits sur le pont du gestionnaire liquid. La plaque d’immatriculation vide sera la OutputPlate(s) où les réactions sont assemblées.
- Préparer le gestionnaire liquid maitrise des logiciels de création d’instances de chaque plaque d’échantillon et réactif préparé, en vous assurant de les nommer exactement tels qu’ils apparaissent sur les cartes de plaque générés par mocloassembly.com, y compris une cuvette propre, eau désionisée étiqueté " Réservoir '.
- À l’aide de la commande « Worklist » dans le logiciel de contrôle, chargez le fichier de .gwl généré par notre outil de logiciel, suivie d’une autre commande « Liste des tâches » qui va exécuter le fichier .gwl chargé dans la première commande.
- Exécutez le script à l’aide de la commande « Exécuter » du logiciel contrôleur.
NOTE : Laissez toujours le robot gestionnaire liquid terminer l’exécution d’un script avant d’essayer d’accéder à l’espace de pont. - Enlever toutes les plaques du pont gestionnaire liquide. Restant d’ADN peut-être être sauvegardé en scellant les SetupPlate(s) avec film étanchéité aluminium et stockage à-20 ° C. Sceller le OutputPlate(s) avec film adhésif, placer dans un thermocycleur ou le bloc chauffant et exécuter avec les paramètres suivants du cycle :
37 ° C pendant 2 h, 50 ° C pendant 5 min, 80 ° C pendant 10 min, tenir à 4 ° C
Remarque : Une fois terminé, le cyclage thermique réaction OutputPlate(s) peuvent être stockés à-20 ° C jusqu'à ce qu’ils sont prêts à se transformer.
4. transformer les réactions [1 jour]
-
Décongeler le nombre requis de compétent e. coli cellule aliquotes nécessaires (10 µL/réaction) sur la glace.
Remarque : Afin de maintenir la stérilité, les étapes suivantes devraient être effectuées près d’une flamme nue ou dans une hotte à flux laminaire.- Alors que les cellules sont en fonte, préparer LB-gélose (contenant antibiotique approprié) par pipetage 50µl de 0,1 M IPTG et 50 µL de 20 mg/mL X-GAL sur la surface. Faire un mélange maître si un grand nombre de réactions de placage. Enduire les plaques uniformément à l’aide d’une baguette de verre stériles ou des perles de verre et laisser les plaques se reposer à 37 ° C pendant au moins 15 min avant l’ensemencement de bactéries.
NOTE : Également ajouter IPTG et X-Gal à agar liquide avant les plaques sont versés, à 2 mM IPTG et concentration 40 µg/mL de la finale de X-Gal. Entreposer ces plaques de lumière directe comme le X-Gal est sensible à la lumière.
- Alors que les cellules sont en fonte, préparer LB-gélose (contenant antibiotique approprié) par pipetage 50µl de 0,1 M IPTG et 50 µL de 20 mg/mL X-GAL sur la surface. Faire un mélange maître si un grand nombre de réactions de placage. Enduire les plaques uniformément à l’aide d’une baguette de verre stériles ou des perles de verre et laisser les plaques se reposer à 37 ° C pendant au moins 15 min avant l’ensemencement de bactéries.
- Sur la glace, aliquote de 10 µL de cellules compétentes pour chaque réaction à une nouvelle plaque PCR 96 puits. Il s’agit de la plaque de Transformation.
- Ajouter 1 à 3 µL de chaque réaction de la OutputPlate(s) à la correspondant bien à la plaque de Transformation et incuber sur glace pendant 5 min.
- Sceller la plaque de Transformation avec adhésif choc de film et de la chaleur dans un thermocycleur à 42 ° C pendant 30 s. Ensuite, placez immédiatement la plaque sur la glace pendant 2 min.
- Pour les mêmes puits de la plaque de Transformation, ajouter 150 µL de médias SOC, sceller avec un joint adhésif en aluminium et incuber à 37 ° C en agitant à 900 tr/min pendant 1 h.
- Plaque de la totalité du contenu de chaque puits de la plaque de Transformation sur les plaques de LB-agar préparés en 4.1.1 à l’aide d’une baguette de verre stériles ou des perles de verre pour enrober uniformément la surface de la plaque. Incuber les plaques à 37 ° C pendant la nuit.
5. cloner vérification [2 jours]
-
Préparer un ou plusieurs blocs de 96 puits puits profond culture avec 1,5 mL de bouillon LB (contenant antibiotique approprié).
- Semblable à l’étape 2.2, ensemencer les culture de puits profonds ou des blocs avec des colonies blanches unique de chaque plaque LB-agar de réactions transformées.
- Sceller les culture ou des blocs avec une étanchéité perméable au gaz et incuber une nuit à 37 ° C en agitant à 900 tr/min.
Remarque : Le modulaire clonage technique utilise bleue-blanche de dépistage, donc positif UFC apparaîtra blanche sur la LB-gélose, tandis que les vecteurs de destination vide apparaît en bleus.
- Isoler l’ADN de plasmide de cultures bactériennes (comme dans 2.3) et soumettre pour Sanger séquençage de clones de vérifier.
Résultats
Nous démontrons l’assemblage automatisé modulaire de 96 appareils d’ADN provenant de diverses régions d’ADN base (Figure 1 b) à l’aide de deux plates-formes de traitement liquide robotique automatisé. Chaque unité de transcription est une disposition linéaire du promoteur, site de liaison ribosomique, gène et terminateur transcriptionnel, cloné dans un vecteur de destination spécifique. TUs sont un élément clé de nombreux circuits génétiques hiérarchique dessins28,29,30 et sont donc une preuve naturelle de concept pour cette approche. Vérifier à la séquence des clones peuvent être réalisés en environ 5 jours de bout en bout, et on présente un aperçu du flux de travail présenté dans la Figure 1 a.
À l’aide de l’outil décrit et au protocole, nous avons capturé plusieurs métriques utiles dans la détermination de la plateforme de montage optimale étant donnée un nombre défini de réactions clonage d’être générés par le scientifique. Figure 2 a montre une comparaison entre les temps de montage de réaction à travers tous les trois modalités. Temps d’exécution est le temps total pour compléter toutes les étapes de pipetage nécessaires pour assembler les 96 réactions, qui comprend la distribution de toutes les parties de l’ADN et les réactifs. Temps de pratique se réfère à la durée totale, qu'un homme a été manuellement impliqué dans la préparation des réactions clonage ou d’installation du logiciel exécutant le gestionnaire liquid. Figure 2 b compare les coûts entre les différentes méthodes. Les coûts présentés sont pour une seule réaction établie par chaque méthode et inclure le prix des enzymes et des pointes de pipette jetable, compte tenus du volume plus faible réaction typique (20 µL pour gestionnaire de liquide, 10 µL de 250 manuel, nL pour distributeur acoustique). Les clones unique de toutes les 96 réactions pour manuelles et liquides préparés gestionnaire d’ensembles ont été séquencés, avec un sous-ensemble de 12 séquencé pour les réactions de distributeur acoustique (Figure 2c). Séquences correctes ont été obtenues pour 95 % des 96 ensembles gestionnaire manuelle et liquide. 83 % du sous-ensemble échantillon distributeur acoustique étaient correctes, cependant les deux réactions finales n’a pas à céder des colonies blanches, probables due à une homogénéisation insuffisante des gouttelettes après la distribution de l’ADN et réactifs était complet. Figure 2d illustre clonage efficacité de réaction, tel que mesuré par le ratio des colonies blanches au nombre total de colonies, qui sont comparables entre manuellement (94 %) et liquides assemblés en gestionnaire des réactions (84 %). Fait intéressant, lorsque la révision à la baisse le volume final de réaction avec le distributeur acoustique, nous avons remarqué une baisse marquée en efficacité de réaction lors de réactions ont été préparées à des volumes plus petits que 1 µL (Supplemental Figure 1 & 2 de Table supplémentaire). Ceci est probablement dû à l’évaporation au cours de la réaction cyclage thermique, qui peut entraîner une augmentation des concentrations de sels dans la réaction.
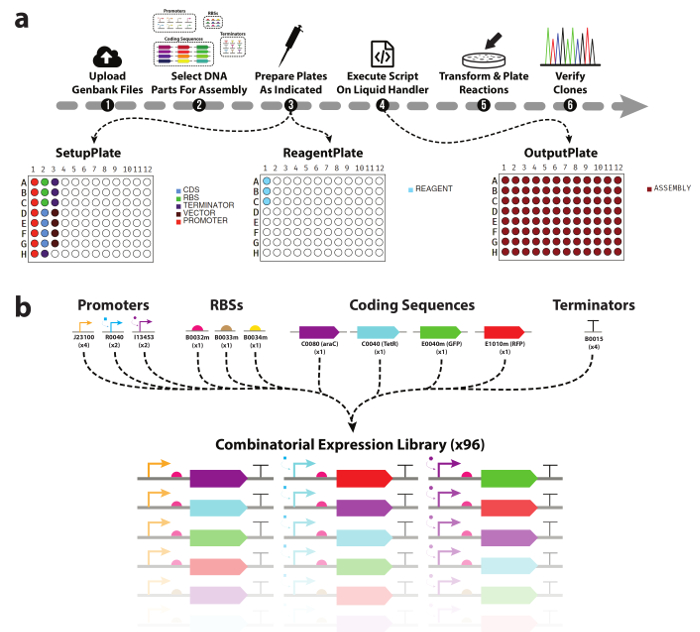
La figure 1. Workflow Assemblée des Bibliothèque de l’appareil d’automatisé ADN.
(a) un aperçu du flux de travail expérimental présentée dans ce travail. (1) chercheurs d’abord charger les fichiers Genbank pour toutes les parties de l’ADN et vecteurs de destination qu’il souhaite utiliser. (2) ensuite, les pièces de l’ADN à inclure dans l’assembly sont sélectionnées. (3) l’outil génère alors une liste de choix pour un gestionnaire automatisé de liquid, ainsi que des cartes de plaque aider avec une alimentation manuelle avec des parties de l’ADN et enzyme mastermix. (4) en utilisant l’ADN & plaques de réactifs, ainsi que la liste déroulante générée, chercheurs exécutent l’assembly des réactions clonage sur le gestionnaire de liquid. (5) une fois terminé, les réactions sont transformées et plaquées pour analyse en aval. (b) des pièces de liste de l’ADN utilisées dans ce travail. Un total de trois promoteurs, trois ribosomique accepteurs, quatre codage de séquences et un terminateur transcriptionnel servaient génèrent la bibliothèque combinatoire de 96 TUs. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
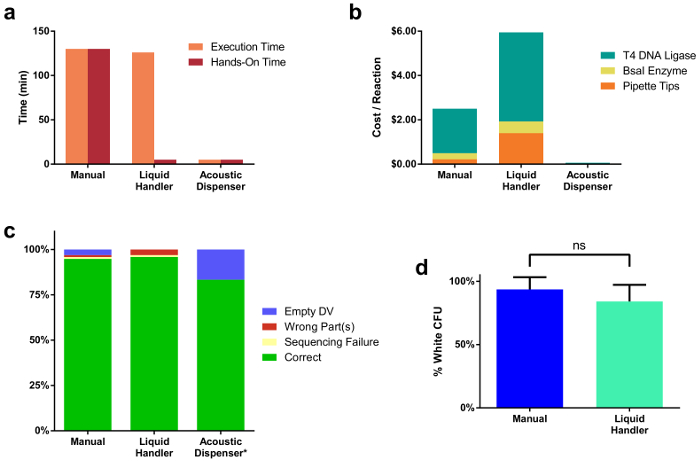
La figure 2. Comparaisons entre les trois modalités de l’Assemblée de réaction différents.
(a) temps de préparation de réaction pour 96 réactions assemblé manuellement, via un gestionnaire liquid ou par un distributeur acoustique. Assemblage manuel a pris 2 h 10 min, ce qui était pratique fois. Le gestionnaire liquid a pris une quantité semblable de temps (2 h 6 min) pour exécuter les commandes de pipetage, cependant qu’une fraction de ce temps (5 min) était pratique. Le distributeur acoustique a eu beaucoup moins de temps pour exécuter le transfert de liquides (5 min) et a pris le temps de pratique minimal (5 min). (b) tarif à la seule réaction de clonage pour chaque méthode de l’assembly. Ce prix comprend le coût des enzymes utilisés, ainsi que des pointes de pipette. (c) séquençage résulte de colonies individuelles de 96 tous assemblés TUs. Le pourcentage de clones correctes était comparable à travers tous les modes d’assemblage. Notez que seul un sous-ensemble (12) des assemblées complet 96 ont été transformées du distributeur acoustique préparé des échantillons. Deux réactions n’a pas à céder des colonies blanches, probablement due à une homogénéisation insuffisante des gouttelettes de mastermix ADN & enzyme dans le OutputPlate. (d) comparaison du clonage des efficacités de réaction entre manuelles et liquides préparés gestionnaire réactions. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Supplémentaire Figure 1. Efficacité de réaction diminue avec abaissement volume réactionnel.
Efficacité de réaction baisse nettement lorsque la révision à la baisse des volumes de réaction ci-dessous 1 µL. Cette tendance est observée avec deux concentrations différentes d’ADN finales ; Cependant, les scientifiques peuvent choisir d’utiliser plus petits volumes pour économiser sur les coûts de réactif si efficacité inférieure peut être tolérée. S’il vous plaît cliquez ici pour télécharger ce fichier.
La Table 1.
Tableau des paramètres de classe liquide ADN et ENZYME pour divers volumes de pipetage. S’il vous plaît cliquez ici pour télécharger ce fichier.
La Table 2.
Calculs de réaction de l’efficacité. Nombre d’UFC brut est donnés pour 12 des 96 réactions transformées préparées manuellement et via le gestionnaire de liquid. Numéros de l’UFC sont également fournis pour les essais de petits volumes de réaction finale sur la distributeur acoustique. S’il vous plaît cliquez ici pour télécharger ce fichier.
Discussion
En conclusion, l’automatisation du processus complet pour la création sur le dispositif ADN, de la conception in silico de manipulation de liquides, est un objectif viable à l’heure actuelle la technologie. Logiciel et robotique moderne permettent la création de workflows qui sont rentables, temps-efficace et évolutive, tout en produisant également davantage de résultats constamment reproductible que les méthodes manuelles. Tandis que les automatismes ne sont pas forcément le choix le plus rentable pour l’exécution d’un protocole, il améliore la reproductibilité expérimentale et libère temps précieux chercheur. Toutefois, selon le matériel utilisé, l’utilisation de l’automatisation peut parfois conduire coût et temps d’exécution bien en deçà de ce qui peut être obtenu par des méthodes manuelles conventionnelles. En outre, automatisation capture les protocoles explicitement dans une manière formalisée prévention ad hoc, artisanal, et anecdotique meilleure pratique basée expérimentation. Ici l’assemblage automatisé des dispositifs modulaires d’ADN est démontrée, et le protocole, fichiers électroniques et les ressources physiques d’ADN nécessaires pour le lecteur effectue ces et semblables, des expériences sur leurs propres sont fournis. Nous espérons que la disponibilité de notre outil, et la publication du présent protocole servira en tant que ressource et déplacer le champ vers un avenir plus transparent et communautaire dans le domaine des procédés d’assemblage de l’ADN et de la robotique de manipulation de liquides.
L’utilité du matériel d’automatisation est largement tributaire de la configuration et les capacités de ce matériel. Par exemple, notre gestionnaire liquide utilise déplacement fluide système pour actionner les aspirer et distribuer les commandes. Les pistons que le liquide du circuit en voiture sont relativement importante 1 mL seringues qui, bien qu’utiles pour une gamme des volumes, impose une limite inférieure de 2 µL pour un dosage précis des réactifs. En conséquence, nous avons intensifié le volume total du clonage réactions mises en place sur le gestionnaire liquid à 20 µL, depuis chaque commande devait être ≥ 2 µL de renoncer. Ce doublé efficacement le coût par réaction liquide réactions gestionnaire-préparée, mais la quantité de temps de pratique nécessaires à l’exécution de ces réactions est significativement réduite. Pour tenter de résoudre ce problème, nous avons répété l’installation de réaction pour toutes les 96 réactions sur un distributeur de liquide acoustique. Cet appareil utilise l’énergie sonore pour distribuer le liquide directement d’une plaque à l’autre et peut atteindre dispense bien au-dessous des volumes (2.5 nL) ce qui est possible avec le déplacement d’air standard base pipettes manuelles. À l’aide de ce dispositif, nous avons pu réduire le volume total de nos réactions à 250 nL, une réduction de 40 fois par rapport aux réactions manuellement préparées de 10 µL. En raison du faible volume distribué et l’absence de changements de pointe entre pipetage, le distributeur acoustique a été en mesure de générer les mêmes 96 réactions en une fraction du temps (< 5 min). Les plus petits volumes de réaction également enregistrer sur réactifs gaspillées, étant donné que nous transformons en général seulement 1-3 µL de la réaction. Cela étant dit, l’utilisation de matériel de bureautique, en conjonction avec un logiciel intuitif, peut faire la génération d’un grand nombre de réactions Assemblée ADN accessible à un plus large public universitaire.
Ici, nous démontrent l’utilité de notre outil de logiciel, cependant, il existe un certain nombre de caractéristiques qui contribuerait à élargir son utilité. Tout d’abord, chaque fois que l’outil est utilisé pour générer un assembly combinatoire, nouvelles plaques de partie d’ADN doivent être générés, exigeant que le scientifique à remplir manuellement ces plaques pour chaque assembly exécuté. Au lieu de cela, il serait utile, si le scientifique pourrait spécifier l’emplacement des pièces dans une plaque d’ADN pour être utilisés dans l’assemblage. Cela permettrait l’utilisation du haut débit plasmide kits de purification d’ADN étant donné que les chercheurs pouvaient ensemencer les cultures d’un kit comme la bibliothèque de MoClo CIADÎR et purifier tous les échantillons ensemble tout en conservant l’emplacement des puits de chaque partie spécifiée dans le kit. Deuxièmement, l’outil actuellement prend uniquement en charge l’utilisation de plaques à 96 puits. Pour les grands projets, où plusieurs centaines Dispositifs d’ADN doivent être construites, le nombre d’ADN, réactif et plaques de destination peut dépasser la capacité de pont du gestionnaire liquide. Ce problème pourrait être, au moins partiellement, allégé par le support pour les formats de plaque densité plus élevées (384 ou 1536 puits). Enfin, l’outil supporte actuellement seulement un seul type de gestionnaire liquide et stratégie de l’Assemblée de l’ADN. S’il est relativement facile de convertir les instructions de l’outil-produit liquide gestionnaire dans un format de distributeur acoustique à l’aide du tableur, nous espérons étendre la prise en charge native de nombreux gestionnaires de liquides différents qui élargiraient considérablement l’applicabilité, comme compatibilité avec les autres techniques d’assemblage ADN communs tiens Gibson Assemblée31.
Une partie cruciale de cet écosystème d’assemblage automatisé est un ensemble d’outils logiciels qui se traduit par des plans de haut niveau de l’Assemblée des protocoles amical automation qui sont explicitement planifiées pour s’exécuter sur les robots de manipulation de liquides. Bien qu’il existe un certain nombre d’outils logiciels qui permettent aux chercheurs de concevoir des assemblages in silico , y compris Benchling, MoClo Planner et Raven32, peu ont la capacité de traduire ces dessins dans des instructions exécutables à exécuter sur un liquide gestionnaire. À celle fin, tels que PR-PR Automation et marionnettiste33,34,35 ont commencé à autoriser ces outils de travail. En outre, les entités commerciales travaillant dans ce domaine cherchent à introduire des « laboratoires de nuage », qui offrent des services expérimentaux à de grands groupes d’utilisateurs finaux via automation. Le protocole décrit dans cet article pourrait servir un morceau de le quelconque de ces efforts fournis qu’ils sont présentés en tant que service.
Assemblage automatisé des dispositifs de l’ADN est d’une valeur immédiate et évidente à la biologie synthétique, notre protocole est utile pour l’ensemble de la communauté des biologistes moléculaires ainsi. Automatiser l’assemblage de l’ADN permet un grand nombre de connu, mais périphériques similaires, génétiques sont créés en parallèle et peut permettent la synthèse rapide de bibliothèques d’expression pour le criblage et des fins de test en études du développement pharmaceutique. Nous espérons que notre outil de logiciel sera plus axée sur la combinatoire ADN Assemblée ses efforts plus accessible et constituer une ressource utile pour la biologie synthétique, tant qu’ensemble de la communauté universitaire.
Déclarations de divulgation
Densmore est président et cofondateur du réseau Automation, Inc. Timmons et McCarthy Lattice employés. Treillis fait des logiciels et services pour l’automatisation d’un grand nombre du processus décrit.
Remerciements
Nous remercions Swapnil Bhatia, Alejandro Pelaez et Johnson Lam de travail sur le projet de marionnettiste, ainsi que Swati Carr, Rachael Smith et Thomas Costa pour obtenir de l’aide ce manuscrit. Ce travail a été financé par la NSF carrière prix #1253856. Il est aussi financé par les expéditions de NSF en calcul prix #1522074.
matériels
| Name | Company | Catalog Number | Comments |
| Hardware / Software | |||
| Freedom EVO 150 Liquid Handling Robot | Tecan | Custom liquid handler fitted with an 8-channel pipetting arm http://lifesciences.tecan.com/products/liquid_handling_and_robotics/freedom_evo | |
| Freedom EVOware Standard (Version 2.4 Service Pack 2) | Tecan | Software used to control the Freedom EVO 150 liquid handler http://lifesciences.tecan.com/products/software/freedom_evoware | |
| Echo 550 | Labcyte | Acoustic Liquid Dispenser http://www.labcyte.com/products/liquidhandling/echo-550-liquid-handler | |
| Sorvall Legend RT | Sorvall | Large benchtop swing-bucket sentrifuge | |
| MasterCycler Pro | Eppendorf | 950040015 | Thermocycler with 96-well heat block https://online-shop.eppendorf.us/US-en/PCR-44553/Cyclers-44554/Mastercycler-pro-PF-5193.html |
| ECHOTHERM Chilling/Heating Dry Bath | Torrey Pines Scientific | Heating/Chilling block for EVO 150 deck https://www.torreypinesscientific.com/products/chilling-and-heating-dry-baths/echotherm-ric20-series-remote-controlled-chillingheating-dr | |
| Tabletop Microcentrifuge 5418 | Eppendorf | 5418000017 | Stardard 18-well microcentrifuge https://online-shop.eppendorf.com/OC-en/Centrifugation-44533/Centrifuges-44534/Centrifuges-5418--5418R-PF-9257.html |
| Name | Company | Catalog Number | Comments |
| Resources | |||
| mocloassembly.com | Lattice Automation | Web-tool for combinatorial DNA assembly mocloassembly.com | |
| CIDAR MoClo Parts Kit | AddGene | 1000000059 | Kit of bacterial glycerol stocks for all DNA parts used in this study https://www.addgene.org/cloning/moclo/densmore/ |
| CIDAR ICE Registry | CIDAR Lab | Registry of plasmid DNA maps https://ice.cidarlab.org/folders/8 https://synbiohub.programmingbiology.org/public/bubdc_ice/bubdc_ice_folder_8/current | |
| Name | Company | Catalog Number | Comments |
| Labware | |||
| 50 μL Conductive Tips | Tecan | 30 057 818 | Sterile 50 μL conductive tips for Tecan liquid handler http://lifesciences.tecan.com/products/consumables/disposable_tips/liquid_handling_disposable_tips |
| 2 mL Deep 96-well Culture Plates | USA Scinetific | 5678-0285 | Bacterial culture plates used for culturing of large numbers of samples http://www.thermoscientific.com/en/product/nunc-1-3-2-0ml-deepwell-plates-shared-wall-technology.html |
| 1.5 mL Microcentrifuge Tubes | USA Scinetific | 1615-5599 | Disposable microcentrifuge tubes http://www.usascientific.com/Seal-Rite-1.5-ml-tube-colors.aspx |
| Breathe Easier sealing membrane | Sigma-Aldrich | Z763624-100EA | Breathable sealing membrane for bacterial culture plates http://www.sigmaaldrich.com/catalog/product/sigma/z763624?lang=en®ion=US |
| Full-Skirted, Low-Profile, 96-Well PCR Plates | GeneMate | T-3183-2 | PCR plates used for all steps https://www.bioexpress.com/store/catalog/product.jsp?catalog_number=T-3183-R |
| Alluminum Sealing Foil for PCR Plates | GeneMate | T-2451-1 | Alluminum seals for PCR plate storage https://www.bioexpress.com/store/catalog/product.jsp?catalog_number=T-2451-1 |
| Polyolefin Sealing Film for PCR Plates | GeneMate | T-2450-1 | Plastic seals for PCR plates during cycling https://www.bioexpress.com/store/catalog/product.jsp?catalog_number=T-2450-1 |
| PCR Cooler | Eppendorf | 22510525 | 96-well cold block https://online-shop.eppendorf.us/US-en/Temperature-Control-and-Mixing-44518/Accessories-44520/PCR-Cooler-PF-55940.html |
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| GenCatch Plasmid DNA Mini-Prep Kit | Epoch Life Sciences | 2160250 | Plasmid DNA purification kit http://www.epochlifescience.com/Product/PurificationKit/dna_mini.aspx |
| T4 DNA Ligase (HC) | Promega | M1794 | High concentration T4 DNA Ligase https://www.promega.com/products/cloning-and-dna-markers/molecular-biology-enzymes-and-reagents/t4-dna-ligase/?catNum=M1794 |
| BbsI Restriction Enzyme | New England Biolabs | R0539L | BbsI enzyme at 10,000 units/ml https://www.neb.com/products/r0539-bbsi |
| BsaI Restriction Enzyme | New England Biolabs | R0535L | BsaI enzyme at 10,000 units/ml https://www.neb.com/products/r3535-bsai-hf |
| T4 DNA Ligase Buffer Pack | Promega | C1263 | 10x T4 DNA ligase buffer https://www.promega.com/products/cloning-and-dna-markers/cloning-tools-and-competent-cells/t4-dna-ligase/ |
| Isopropyl-ß-D-thiogalactopyranoside (IPTG) | Zymo Research | I1001-25 | 0.5M IPTG Solution http://www.zymoresearch.com/buffers-solutions/chemicals/isopropyl-ss-d-thiogalactopyranoside-iptg |
| 5-bromo-4-chloro-3-indolyl ß-D-galactopyranoside (X-GAL) | Zymo Research | X1001-25 | 20 mg/ml X-GAL solution http://www.zymoresearch.com/buffers-solutions/chemicals/5-bromo-4-chloro-3-indolyl-ss-d-galactopyranoside-x-gal |
| Kanamycin Sulfate | Zymo Research | A1003-25 | 35 mg/ml Kanamycin solution http://www.zymoresearch.com/buffers-solutions/antibiotics/kanamycin-sulfate |
| Carbenicillin (Disodium Salt) | Fisher | BP26481 | 1 g Carbenicillin (Ampicillin analog) https://www.fishersci.com/shop/products/carbenicillin-disodium-salt-fisher-bioreagents-3/p-25005#?keyword=carbenicillin |
| SOC Broth Media | Teknova | S0225 | Powder media used to make SOC broth http://www.teknova.com/SOC-BROTH-MEDIA-p/s0225.htm |
| LB Broth (Lennox) Media | Sigma-Aldrich | L3022-1KG | Powder media used to make LB broth http://www.sigmaaldrich.com/catalog/product/sigma/l3022?lang=en®ion=US |
| LB Broth with agar (Lennox) Media | Sigma-Aldrich | L2897-1KG | LB with agar mix used for making solid media plates http://www.sigmaaldrich.com/catalog/product/sigma/l2897?lang=en®ion=US |
| Alpha-Select Gold Efficiency Competent Cells | Bioline | BIO-85027 | High efficiency chemically competent E. coli cells http://www.bioline.com/us/alpha-select-gold-efficiency.html |
| Name | Company | Catalog Number | Comments |
| Primers | |||
| Primer VF | 5'-TGCCACCTGACGTCTAAGAA-3' Primers used for Sanger sequencing and colony PCRs | ||
| Primer VR | 5'-ATTACCGCCTTTGAGTGAGC-3' Primers used for Sanger sequencing and colony PCRs |
Références
- Gardner, T. S., Cantor, C. R., Collins, J. J. Construction of a genetic toggle switch in Escherichia coli. Nature. 403 (6767), 339-342 (2000).
- Elowitz, M. B., Leibler, S. A synthetic oscillatory network of transcriptional regulators. Nature. 403 (6767), 335-338 (2000).
- Lutolf, M. P., Hubbell, J. A. Synthetic biomaterials as instructive extracellular microenvironments for morphogenesis in tissue engineering. Nat Biotechnol. 23 (1), 47-55 (2005).
- Anderson, J. C., Clarke, E. J., Arkin, A. P., Voigt, C. A. Environmentally controlled invasion of cancer cells by engineered bacteria. J Mol Biol. 355 (4), 619-627 (2006).
- Xie, Z., Wroblewska, L., Prochazka, L., Weiss, R., Benenson, Y. Multi-input RNAi-based logic circuit for identification of specific cancer cells. Science. 333 (6047), 1307-1311 (2011).
- Ro, D. K., et al. Production of the antimalarial drug precursor artemisinic acid in engineered yeast. Nature. 440 (7086), 940-943 (2006).
- Georgianna, D. R., Mayfield, S. P. Exploiting diversity and synthetic biology for the production of algal biofuels. Nature. 488 (7411), 329-335 (2012).
- Savage, D. F., Way, J., Silver, P. A. Defossiling fuel: how synthetic biology can transform biofuel production. ACS Chem Biol. 3 (1), 13-16 (2008).
- Fussenegger, M., et al. Streptogramin-based gene regulation systems for mammalian cells. Nat Biotechnol. 18 (11), 1203-1208 (2000).
- Boorsma, M., et al. A temperature-regulated replicon-based DNA expression system. Nat Biotechnol. 18 (4), 429-432 (2000).
- Malphettes, L., et al. A novel mammalian expression system derived from components coordinating nicotine degradation in arthrobacter nicotinovorans pAO1. Nucleic Acids Res. 33 (12), e107(2005).
- Brophy, J. A., Voigt, C. A. Principles of genetic circuit design. Nat Methods. 11 (5), 508-520 (2014).
- Lou, C., Stanton, B., Chen, Y. J., Munsky, B., Voigt, C. A. Ribozyme-based insulator parts buffer synthetic circuits from genetic context. Nat Biotechnol. , (2012).
- Cardinale, S., Arkin, A. P. Contextualizing context for synthetic biology--identifying causes of failure of synthetic biological systems. Biotechnol J. 7 (7), 856-866 (2012).
- Carr, S. B., Beal, J., Densmore, D. M. Reducing DNA context dependence in bacterial promoters. PLoS One. 12 (4), e0176013(2017).
- Yeung, E., Ng, A., Kim, J., Sun, Z. Z., Murray, R. M. Decision and Control (CDC), 2014 IEEE 53rd Annual Conference on. , 5405-5412 (2014).
- Engler, C., Gruetzner, R., Kandzia, R., Marillonnet, S. Golden gate shuffling: a one-pot DNA shuffling method based on type IIs restriction enzymes. PLoS One. 4 (5), e5553(2009).
- Weber, E., Engler, C., Gruetzner, R., Werner, S., Marillonnet, S. A modular cloning system for standardized assembly of multigene constructs. PLoS One. 6 (2), e16765(2011).
- Iverson, S. V., Haddock, T. L., Beal, J., Densmore, D. M. CIDAR MoClo: Improved MoClo Assembly Standard and New E. coli Part Library Enable Rapid Combinatorial Design for Synthetic and Traditional Biology. ACS Synth Biol. 5 (1), 99-103 (2016).
- Casini, A., Storch, M., Baldwin, G. S., Ellis, T. Bricks and blueprints: methods and standards for DNA assembly. Nat Rev Mol Cell Biol. 16 (9), 568-576 (2015).
- Bhatia, S. P., Smanski, M., Voigt, C. A., Densmore, D. M. Genetic design via combinatorial constraint specification. ACS Synth Biol. , (2017).
- Ham, T. S., et al. Design, implementation and practice of JBEI-ICE: an open source biological part registry platform and tools. Nucleic Acids Res. 40 (18), e141(2012).
- Madsen, C., et al. The SBOL Stack: A Platform for Storing, Publishing, and Sharing Synthetic Biology Designs. ACS Synth Biol. 5 (6), 487-497 (2016).
- Knight, T. Draft standard for BioBrick biological parts. , (2007).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nat Methods. 6 (5), 343-345 (2009).
- Ma, A. C., et al. FusX: A Rapid One-Step Transcription Activator-Like Effector Assembly System for Genome Science. Hum Gene Ther. 27 (6), 451-463 (2016).
- Check Hayden, E. The automated lab. Nature. 516 (7529), 131-132 (2014).
- Nielsen, A. A., et al. Genetic circuit design automation. Science. 352 (6281), aac7341(2016).
- Woodruff, L. B. A., et al. Registry in a tube: multiplexed pools of retrievable parts for genetic design space exploration. Nucleic Acids Res. 45 (3), 1553-1565 (2017).
- Hasty, J., McMillen, D., Collins, J. J. Engineered gene circuits. Nature. 420 (6912), 224-230 (2002).
- Gibson, D. G. Enzymatic assembly of overlapping DNA fragments. Methods Enzymol. 498, 349-361 (2011).
- Appleton, E., Tao, J., Haddock, T., Densmore, D. Interactive assembly algorithms for molecular cloning. Nat Methods. 11 (6), 657-662 (2014).
- Vasilev, V., Liu, C., Haddock, T., Bhatia, S., Adler, A., Yaman, F., Beal, J., Babb, J., Weiss, R., Densmore, D. A Software Stack for Specification and Robotic Execution of Protocols for Synthetic Biological Engineering. SynBERC Fall Retreat, Harvard University. , (2011).
- Beal, J., et al. An end-to-end workflow for engineering of biological networks from high-level specifications. ACS Synth Biol. 1 (8), 317-331 (2012).
- Bhatia, S., Densmore, D. Pigeon: a design visualizer for synthetic biology. ACS Synth Biol. 2 (6), 348-350 (2013).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon
Nous utilisons des cookies afin d'améliorer votre expérience sur notre site web.
En continuant à utiliser notre site ou en cliquant sur le bouton ''continuer'', vous acceptez l'utilisation de cookies.