Method Article
Incorporation spécifiques résidus de noncanonical acides aminés en protéines de modèle à l'aide d'un
Dans cet article
Résumé
An easy-to-use, cell-free expression protocol for the residue-specific incorporation of noncanonical amino acid analogs into proteins, including downstream analysis, is presented for medical, pharmaceutic, structural and functional studies.
Résumé
L'ensemble canonique des acides aminés conduit à un niveau exceptionnellement large éventail de fonctionnalités de protéines. Néanmoins, l'ensemble des résidus impose encore des limitations sur les applications de protéines potentielles. L'incorporation d'acides aminés non canoniques peut élargir ce périmètre. Il existe deux approches complémentaires pour l'incorporation d'acides aminés non canoniques. Pour l'incorporation spécifique du site, en plus des mécanismes endogènes de translation canoniques, une paire aminoacyl-ARNt synthétase-ARNt orthogonal doit être prévu qui ne réagit pas avec ceux canoniques. Par conséquent, un codon qui ne soit pas affecté à un acide aminé canonique, habituellement un codon d'arrêt est également nécessaire. Cette expansion du code génétique permet l'incorporation d'un aminoacide non canonique à un seul site donné au sein de la protéine. Le travail présenté ici décrit l'incorporation spécifique de résidus où le code génétique est réaffecté au sein du système de traduction endogène. Le mécanisme de traduction d'unccepts l'acide aminé noncanonical comme un substitut pour l' incorporer à des endroits canoniquement prescrits, à savoir, toutes les occurrences d'un acide aminé dans la protéine canonique sont remplacées par une noncanonical. L'incorporation d'acides aminés non canoniques peuvent modifier la structure des protéines, ce qui provoque des propriétés physiques et chimiques considérablement modifiés. Non canoniques des analogues d'acides aminés agissent souvent comme des inhibiteurs de la croissance cellulaire pour les hôtes d'expression , car ils modifient les protéines endogènes, ce qui limite la production de protéines in vivo. In vivo incorporation d'acides aminés non canoniques toxiques dans des protéines demeure particulièrement difficile. Ici, une approche sans cellule pour un remplacement complet de L-arginine par l'acide aminé L-canavanine noncanonical est présenté. Elle contourne les difficultés inhérentes à l'expression in vivo. En outre, un protocole pour préparer des protéines cibles pour l'analyse spectrale de masse est inclus. Il est montré que la L-lysine peut être remplacée par la L-hydroxy-lysine,mais avec une efficacité moindre. En principe, tout analogue d'aminoacide non canonique peut être incorporé en utilisant la méthode présentée tant que la endogène in vitro système de traduction reconnaît.
Introduction
Le code génétique est universel à la biosphère. Il code pour un ensemble de 20 acides aminés canoniques, qui est parfois prolongé par sélénocystéine 1 ou 2 pyrrolysine. Il est ribosome qui se traduit par le code génétique à l'aide d'ARNt dans des chaînes d'acides aminés qui se replient en protéines. Les groupes fonctionnels des acides aminés canoniques, en combinaison avec des modifications post - traductionnelles, contribuent à une gamme exceptionnellement large de la fonction de la protéine 3,4. En principe, les limitations fonctionnelles dues à l'ensemble limité d'acides aminés canoniques peuvent être surmontés en incorporant en outre, des acides aminés non canoniques (NCAAs) qui permettent de nouvelles chimies et de nouvelles fonctionnalités 3,4.
Il existe deux approches complémentaires pour l'incorporation de NCAAs: le site- ou l'incorporation spécifique de résidus. La première méthode implique des difficultés techniques considérables, puisque l'ensemble canonique de aminoacyl-ARNt-synthetases (RAA) et ARNt doivent être élargis par une paire aaRS-ARNt orthogonal qui ne doit pas interagir avec la machinerie de traduction endogène. Sur la base de l'ingénierie minutieuse, cette approche intègre les NCAAs que des mutations ponctuelles au niveau des sites de protéines désirées. Incorporation de NCAAs spécifique au site est génétiquement codé par un codon qui ne sont pas affectés à un acide aminé canonique (CAA), généralement un codon stop 5-9. Cette méthode implique des changements dans la fonction à un site donné , plutôt que sur l'ensemble de la protéine 10-13.
En revanche, l'incorporation spécifique au résidu repose sur la reconnaissance erronée de l'aminoacide non canonique par la machinerie de traduction canonique. L'incorporation se produit en raison de l'absence de spécificité de substrat des aaRS. L'incorporation spécifique de résidus de NCAAs, construit sur les travaux de Cohen et ses collègues 14, a conduit à d'importantes applications 3,10, entre les bio-orthogonale étiquetage 15-17 de protéinesou élucidation de la structure des protéines en cristallographie aux rayons X 18.
Comme aaRS naturels préfèrent généralement leur acide aminé apparenté sur une ncaa isostructurales, efficace in vivo incorporation spécifique de résidus nécessite généralement un hôte d'expression auxotrophe pas capable de synthétiser l'analogue canonique de la NCAA. Les cellules hôtes sont cultivées dans un milieu de croissance qui fournit seulement une faible concentration du PQA analogue. Son épuisement en combinaison avec la supplémentation consécutive avec la NCAA oblige l'hôte d'expression pour incorporer la NCAA dans la protéine de modèle à de multiples sites canoniquement prescrits. Contrairement à l'approche spécifique du site, ce qui a généralement un impact profond sur la structure de la protéine entière, conduisant à considérablement modifié les propriétés physiques et chimiques des protéines 19,20. Cependant, la plupart des NCAAs sont des inhibiteurs de croissance de l'hôte d'expression 3, car elles sont incorporées dans beaucoup d' autres proteins en plus de ceux d'intérêt au cours de l'expression du gène recombinant. Cela limite clairement l'approche in vivo. L'incorporation in vivo d'acides aminés qui sont toxiques ou qui ont une forte influence sur la structure des protéines demeure particulièrement difficile. Cependant, ces molécules sont parmi les plus prometteuses pour concevoir des protéines avec des fonctions extraordinaires.
Un exemple est la, noncanonical, naturel L-canavanine toxiques (Can), un analogue de L-arginine (Arg). Elle affecte et bloque Arg voies réactionnelles réglementaires et catalytiques associés, et sa présence dans la cellule vivante peut entraîner la mort immédiate 3,21-23. Son incorporation dans les protéines à des positions d'arginine peut réduire la stabilité des protéines 21-23. En raison de la toxicité résultant, l' expression de canavanine contenant des protéines dans Escherichia coli (E. coli) et d' autres hôtes d'expression commune reste un défi. Pour ces raisons, complète in vivo incorporation de Can à toutes les positions Arg a été confirmée de manière appropriée une seule fois 24, en utilisant un système de production unique protéine élaborée. Cependant, Can a été proposée comme un agent anti-cancer 25-27, et comme un stimulateur pour les maladies auto - immunes chez les humains 28. En outre, il fait l' objet de diverses études sur ses propriétés anti-métabolique, antibactériens, antifongiques et antivirales 25. Ces propriétés soulèvent une demande pour efficace et facile à exécuter des méthodes pour exprimer Peut contenant des protéines pour pharmacie, médicaux et des études fonctionnelles.
Bien que de nombreux problèmes qui sont liés à la production in vivo peuvent être contournés en utilisant des systèmes d'expression sans cellules, dans les approches in vitro spécifiques de résidus ont seulement été mal exploré. L'incorporation sans cellules spécifiques reste d'un L-tryptophane analogique 29 et multiple NCAAs 30 ont été rapportés. Ces méthodes sont basées sur la très efficpolymérase ient T7 RNA. La T7 ARN polymérase du bactériophage comporte en forme de transcription, ce qui réduit la fonctionnalité génétique par rapport à la transcription endogène.
L'incorporation spécifique du résidu complet de Can dans une protéine de modèle à toutes les positions Arg a été récemment rapporté 31, en utilisant un système d'expression sans cellule 32. Une légère modification du même système a permis l' incorporation spécifique du site de différents analogues de pyrrolysine dans une protéine modèle via codon stop suppression 33. Le système exempt de cellules utilisé 31 - 33 est basé sur un ensemble E. coli système de transcription-traduction. Néanmoins, il permet l' expression des protéines aussi efficacement que dans les systèmes de bactériophage courant (0,5 à 1 mg / ml de protéine recombinante) 32 tout en conservant une grande partie de la modularité originale de transcription-traduction.
Dans ce travail, un protocole détaillé est fourni sur la manière dont le résidincorporation spécifique de NCAAs-ue peut être réalisée en utilisant tout cela E. système sans cellule coli 32. En outre, de nouvelles mesures pour préparer les protéines exprimées pour une évaluation appropriée par spectroscopie de masse HPLC-ESI sont proposées. Pour développer les propriétés de ce système sans cellule, ce travail ne se réfère pas seulement à l'incorporation publiée de Can 31 , mais présente également de nouvelles données relatives à la L-lysine analogique L-hydroxy-lysine noncanonical.
Le protocole suivant pour l'incorporation spécifique du résidu d'NCAAs est une adaptation d'un protocole publié récemment dans JoVE 34. Le dernier protocole décrit comment effectuer l'expression sans cellule très efficace avec des acides aminés standard. En outre, il présente la préparation de l'extrait exempt de cellules brut, la solution d'acide aminé, la solution d'énergie de stock et le tampon de l'énergie utilisée dans cette approche. Le protocole qui suit se concentre sur les étapes modifiées par rapport à la précédente protocol afin de permettre l'incorporation spécifique de résidus de NCAAs. pipettes étalonnées, conseils faible liaison pipettes et tubes micro-centrifugeuse sont recommandées pour la préparation. Dans ce qui suit, les abréviations IUPAC pour les acides aminés sont utilisés.
Protocole
Attention! S'il vous plaît consulter toutes les fiches de données de sécurité des matériaux pertinents (MSDS) avant utilisation. Plusieurs des produits chimiques utilisés sont extrêmement toxiques. Équipement de protection individuelle est nécessaire (eyeshield, masque anti-poussière, des gants, une blouse de laboratoire, pantalon de longueur, fermé-orteil chaussures) ainsi que de travailler dans une hotte.
1. Préparation de la solution d'acides aminés
- Préparation de la solution mère de la NCAA (168 mM)
NOTE: La préparation de la solution mère de la NCAA est décrit pour l'analogue Arg Can comme un exemple. adapter en conséquence les valeurs d'autres NCAAs.- Placer un tube de 1,5 ml de réaction sur une microbalance. Peser 46,1 mg de Can à l'intérieur du tube de réaction pour la préparation de 1 ml d'une solution 168 mM. Utiliser une microspatule stérile. Pour un mélange racémique de la NCAA, le double de la concentration de la solution mère.
- Ajouter 977 pi de ddH stérile 2 O. Bien vortex jusqu'à Can est in dissolution complète.
NOTE: Pour un volume total de solution de 1 ml, le volume physique de l'acide aminé dissous doit être compensée. Pour tous les acides aminés, l' estimation de la moitié de la masse solide en mg que l'augmentation du volume correspondant de pi (100 mg solide aura un volume de 50 ul de la solution) 35. La plupart des acides aminés peuvent être dissous à cette concentration. Dans le cas contraire, réduire la concentration jusqu'à dissolution complète. - Utiliser directement la solution mère de NCAA pour la préparation des solutions d'acides aminés dans la section 1.2 ou le flash congeler dans l' azote liquide et de le stocker à -20 ° C. ATTENTION! Pour la sécurité, porter un eyeshield et Cryo-gants à l' abri de liquide éclaboussures d'azote.
- Préparation des solutions d'acides aminés
NOTE: Pour la préparation des solutions d'acides aminés, utiliser l'échantillonneur d'acides aminés fournissant les isomères L des 20 CAAS en stock séparésolution (1,5 ml, tamponnées avec HEPES / KOH, <0,1% de NaN3, pH 7,5), chacune à une concentration de 168 mM, à l' exception de la L-Leucine (140 mM). Pour une préparation artisanale de ces solutions mères (tamponné avec KOH), suivez ce protocole 35.- Décongeler les solutions mères des 20 CaaS (échantillonneur d'acides aminés ou préparé selon 35) et de la NCAA (préparé dans la section 1.1) à la température ambiante.
- Après décongélation, vortex fréquemment les solutions mères pour redissoudre des acides aminés précipités. Etant donné que certains acides aminés sont plus difficiles à dissoudre, les incuber dans un bloc chauffant à 37 ° C jusqu'à dissolution complète. Cys peut ne pas dissoudre complètement. Placez tous les acides aminés sur la glace, sauf pour Asn, Phe et Cys - garder ces à la température ambiante pour éviter la précipitation.
- Utilisez les valeurs suivantes pour utiliser un septième de la trousse complète.
NOTE: réduire de manière appropriée pour travailler avec de plus petits volumes et de sauvegarder des parties du kit pour d'autres expériences. Redoubler d'efforts pour incorporer l'ion de NCAAs en protéines modèles à grande échelle. Pour éviter la décongélation fréquents, ce qui est de nature à réduire la stabilité des acides aminés, aliquoter des solutions mères d'acides aminés individuels dans des volumes de 200 ul. Ce volume aliquote de 200 pi et les volumes aliquotes utilisés dans l'étape 1.2.4.1 compte des pertes dues à pipetage. - Tout d'abord, préparer une solution maître de mélange d'acides aminés qui sera divisé à l'étape 1.2.4.3 pour finaliser la préparation des 3 composés différemment des solutions d'acides aminés (sections 1.2.5 - 1.2.7). Dans ces solutions, on concentre l'ensemble des acides aminés à 6 mM, à l'exception Leu (5 mM).
- 1,4 ml de transfert stérile ddH 2 O dans un tube à centrifuger de 15 ml. Mettez-le sur la glace. Ajouter 175 pi de chaque solution d'acide aminé de stock. Ajouter un après l' autre, à l' exception de la solution mère du PQA (par exemple, Arg) pour être remplacé par le ncaa (par exemple, peut -il ). vortex soigneusement après chaque addition et de mettre la solution de retour sur la glace.
REMARQUE:Leu est à 5 mM dans les solutions d'acides aminés composition différente 3, par rapport à 6 mm pour les autres acides aminés. La concentration réduite ne diminue pas l'efficacité d'expression. Calamine jusqu'à 6 mm convient aussi. - Transférer les solutions acides aminés des actions dans l'ordre suivant pour éviter la précipitation: Ala, Arg, Asn, Asp, Gln, Glu, Gly, His, Ile, Lys, Met, Phe, Pro, Ser, Thr, Val, Trp, Tyr, Leu, et Cys. Rappelez - vous de ne pas ajouter la solution mère du PQA (par exemple, Arg) qui est analogue à la NCAA (par exemple, Can). Enfin, le fond vortex. Incuber à 37 ° C pour rendre la solution aussi claire que possible.
- Divisez cette solution maître de mélange d'acides aminés en trois volumes égaux de 1,35 ml. Transfert chacun des volumes fractionnés dans 1,5 ml tubes de réaction. Gardez-les sur de la glace.
- 1,4 ml de transfert stérile ddH 2 O dans un tube à centrifuger de 15 ml. Mettez-le sur la glace. Ajouter 175 pi de chaque solution d'acide aminé de stock. Ajouter un après l' autre, à l' exception de la solution mère du PQA (par exemple, Arg) pour être remplacé par le ncaa (par exemple, peut -il ). vortex soigneusement après chaque addition et de mettre la solution de retour sur la glace.
- Préparer une solution d'acides aminés qui se compose de l'ensemble des 20 caas à une concentration de 6 mM chacun, à l'exception Leu qui est de 5 mM. Pour le premier volume of 1,35 ml, à la suite de la scission à l' étape 1.2.4.3, ajouter 50 ul de la solution mM 168 du cAA (par exemple, Arg) qui est analogue à la NCAA (par exemple, peuvent -ils ). Bien vortex.
- Remettre sur la glace. Aliquoter ce 1,4 ml de solution dans des volumes de 16 ul dans des tubes de réaction. A noter que ce volume de solution conduit à environ 85 aliquotes. Marquez ces aliquotes "+ Caa" (par exemple, + Arg).
- Flash geler les aliquotes dans l' azote liquide et conserver à -80 ° C. ATTENTION! Pour la sécurité, porter un eyeshield et Cryo-gants pour être protégés contre les projections d'azote liquide.
- Préparer une solution d'acide aminé qui est composé de 19 CaaS sauf pour le cAA (par exemple, Arg) qui est analogue à la NCAA (par exemple, Can). Ajouter chaque acide aminé à une concentration de 6 mM, à l'exception Leu (5 mM).
- Ajouter 50 ul de ddH 2 O stérile au second volume de 1,35 ml, à la suite de la scissionà l'étape 1.2.4.3. Bien vortex et remis sur la glace. Aliquoter ce 1,4 ml de solution dans des volumes de 16 ul dans des tubes de réaction. A noter que ce volume de solution conduit à environ 85 aliquotes. Marquez ces aliquotes "- cAA" (par exemple, - Arg).
- Flash geler les aliquotes dans l' azote liquide et conserver à -80 ° C. ATTENTION! Pour la sécurité, porter un eyeshield et Cryo-gants pour être protégés contre les projections d'azote.
- Préparer un mélange d'acides aminés qui contient 19 CAAS et la NCAA (par exemple, Can) qui remplace celui canonique (par exemple, Arg). Ajouter chaque acide aminé à une concentration de 6 mM, à l'exception Leu (5 mM). Pour 1,35 ml dernier volume, à la suite de la scission à l' étape 1.2.4.3, ajouter 50 ul de la mM solution du ncaa 168 (par exemple, Can). Étiqueter "+ ncaa" (par exemple, + Can). Bien vortex et remis sur la glace.
- Aliquoter ce 1,4 ml de solution dans des volumes de 16 & #181; l dans des tubes de réaction. A noter que ce volume de solution conduit à environ 85 aliquotes. Marquez ces aliquotes "+ ncaa" (par exemple, + Can).
- Flash geler les aliquotes dans l' azote liquide et conserver à -80 ° C. ATTENTION! Pour la sécurité, porter un eyeshield et Cryo-gants pour être protégés contre les projections d'azote.
NOTE: Les 16 volumes aliquotes ul utilisés dans les étapes 1.2.5.1, 1.2.6.1 et 1.2.7.1 sont légèrement plus élevés que nécessaire pour tenir compte des pertes dues à pipetage.
2. Tampon énergie Préparation
NOTE: Chaque lot d'extrait brut est unique et nécessite des concentrations de Mg et K-glutamate 34 optimisé. Le volume de l' aliquote de l' extrait brut dépend de la concentration en protéines 34. Utilisez le modèle de calcul prévu (Supplemental Material 1) pour des valeurs différentes. Trouvez d'autres instructions supplémentaires Matériau 1 légende de la figure, 'explicationIning comment utiliser ce modèle.
- Préparer et conserver à -80 ° C la solution 14x de l' énergie et des aliquotes d'extrait brut selon le protocole non modifié 34. Calibrer l'extrait brut en fonction des concentrations de Mg et K-glutamate pour optimiser l' efficacité de l' expression 34.
REMARQUE: La composition finale de 14x solution d'énergie est la suivante: HEPES 700 (pH 8) mM, ATP 21 mM, GTP 21 mM, CTP 12,6 mM, UTP 12,6, 2,8 mg / ml d'ARNt, 3,64 mM de CoA réductase, mM NAD 4,62, 10,5 mM AMPc, 0,95 mM d'acide folinique, 14 mM de spermidine, 420 mM 3-PGA. - Thaw sur la glace, la solution 100 mM Mg-glutamate disponible, solution mère 3 M K-glutamate, solution 14x d'énergie et 40% de PEG-8000 pour préparer le mélange maître. Gardez-les sur de la glace.
- Mélanger 9,18 pi de solution mère 100 mM Mg-glutamate, 3,06 ul de solution mère 3 M K-glutamate, 21.86 pi de 14x solution énergétique, 15,3 pi de 40% de PEG-8000 et de 1,6 ul stériles ddH 2 O dans un tube de réaction. Bien vortexer ce mâter mélanger après chaque addition, et le garder sur la glace.
- Aliquoter le mélange maître (51 pi) dans des volumes de 16 pi (3 aliquotes) dans des tubes de réaction. Fréquemment vortex le mélange maître pendant aliquotage. Flash geler les aliquotes dans l'azote liquide.
NOTE: Le volume aliquote de 16 pi, ainsi que le volume de mélange maître sont légèrement plus élevés que nécessaire pour tenir compte des pertes dues à pipetage. - Utilisez une passoire pour recueillir les tubes tampons d'énergie. Stocker les tubes à -80 ° C. ATTENTION! Porter un eyeshield et Cryo-gants pour être protégés contre les projections d'azote.
3. Préparation et exécution de réactions sans cellule pour incorporation de résidus spécifiques de NCAAs
- Tout d' abord, préparer la solution d'ADN vecteur dans le trou DDH 2 O.
- Pour l' expression de la protéine hautement efficace, utiliser le vecteur d'expression pBEST-OR2-OR1-Pr-UTR1-deGFP-T500 32. Cloner le gène codant pour la protéine modèle dans ce vecteur 36,37 </ Sup>.
REMARQUE: Vous pouvez également utiliser d' autres promoteurs qui sont reconnus par σ 70 aussi bien, mais il faut noter que l' efficacité de l' expression peut être réduite. - Transformer 37,38 vecteur dans E. coli KL 740 32 (Yale SCGC #: 4382), purifient amplifié vecteur ADN 37,39 et de quantifier la concentration de la solution d'ADN 40-42. Conserver la solution d'ADN à -20 ° C ou directement l'utiliser pour la préparation de la réaction cellulaire des frais (étapes 3.4.1, 3.4.2 et 3.4.3).
- Pour l' expression de la protéine hautement efficace, utiliser le vecteur d'expression pBEST-OR2-OR1-Pr-UTR1-deGFP-T500 32. Cloner le gène codant pour la protéine modèle dans ce vecteur 36,37 </ Sup>.
- Calibrer l'efficacité d'expression sans cellule en fonction de la concentration utilisée vecteur de construction selon le protocole non modifié 34. Utilisez la concentration optimale qui conduit à plus hauts rendements en protéines pour la préparation de réaction exempt de cellules (étapes 3.4.1, 3.4.2 et 3.4.3).
NOTE: La préparation des réactions sans cellules est illustré en utilisant un nM ADN vecteur solution mère 90 qui conduit à une concentration de vecteur finale de 10 nM dans la libre celluleréaction et suit les valeurs optimales précitées de Mg et K-glutamate, ainsi que le volume de l'aliquote d'extrait. Utilisez le modèle de calcul des valeurs différentes. - Décongeler sur la glace 3 aliquots d'extrait brut chacun des 30 volumes ul (préparé selon le protocole non modifié 34), une amino solution d'acide aliquote marquée "+ CAA" (par exemple + Arg), 1 amino solution d'acide aliquote marquée "- CAA" (par exemple , , - Arg) et 1 amino solution d'acide aliquote marquée "+ ncaa" (par exemple, + Can) (préparé dans les sections 1.2.5 - 1.2.7), 3 tampons d'énergie aliquotes (préparé à l' article 2) et la solution d'ADN de vecteur ( préparé dans la section 3.1).
NOTE: L'extrait brut est légèrement visqueux et il peut contenir des bulles d'air. Retirer les bulles d'air par centrifugation à 10.000 xg pendant 30 secondes à 4 ° C. Mettez aliquotes d'extrait brut de retour sur la glace. - Préparer 3 réactions sans cellules différemment composées (chaque volume final de 90 pi) en mélangeant l'extrait brut (33,33%), l'énergieun tampon (16,67%), 1 des 3 composés différemment des aliquotes d'acide aminé de la solution (16,67%) et une solution d'ADN vecteur. Eventuellement, ajouter d' autres biomolécules (ADN, protéines, ARNt, etc.), mais aussi de réduire de manière appropriée le volume de ddH 2 O.
- Préparer la réaction acellulaire référence (90 ul) exprimant la protéine modèle non modifié.
- Ajouter 15 ul de tampon d'énergie, 15 ul de la solution aliquote d'acides aminés marquée "+ CAA" (par exemple + Arg), 10 pi de 90 nM d'une solution d'ADN de vecteur et 20 pi de ddH stérile 2 O 30 pl de brut extrait. Mélanger par pipetage vers le haut et vers le bas et légèrement vortex après addition de chaque ingrédient.
- Aliquoter les 90 ul de réaction exempt de cellules en 15 volumes égaux de 6 pi. Transférer chacun des 15 volumes dans un tube de réaction séparé. Fermer les tubes et les remettre sur la glace. Étiquetez tous les tubes de réaction comme «CFR (+ cAA)" (par exemple, CFR (+ Arg)).
- Préparer la réaction négative de contrôle exempt de cellules (90 pi), où ni le cAA (par exemple, Arg) , ni l'analogue noncanonical (par exemple, peuvent -ils ) sont ajoutés.
- Ajouter 15 pi de tampon d'énergie, 15 pl de la solution aliquote d'acide aminé marqué "- cAA" (par exemple, - Arg), 10 pl de 90 nM solution d'ADN de vecteur et 20 pi de ddH stérile 2 O à 30 pi de brut extrait. Mélanger par pipetage vers le haut et vers le bas et légèrement vortex après addition de chaque ingrédient.
- Aliquoter les 90 ul de réaction exempt de cellules en 15 volumes égaux de 6 pi. Transférer chacun des 15 volumes dans un tube de réaction séparé. Fermer les tubes et les remettre sur la glace. Étiquetez tous les tubes de réaction comme «CFR (-cAA)" (par exemple, CFR (Arg)).
- Préparer la réaction sans cellules (90 pi) qui est censé résidu spécifiquement incorporer la NCAA (par exemple, Can) dans la protéine modèle exprimé.
- Ajouter 15pi de tampon d'énergie, 15 ul de la solution aliquote d'acides aminés marquée "+ ncaa" (par exemple, + Can), 10 pi de 90 solution d'ADN nm et 20 pi de ddH stérile 2 O 30 pl d'extrait brut. Mélanger par pipetage vers le haut et vers le bas et légèrement vortex après addition de chaque ingrédient.
NOTE: L'efficacité de l'expression peut être réduite par comparaison à l'expression des acides aminés standards. Si nécessaire, il suffit de l'échelle le volume de réaction exempt de cellules. - Aliquoter les 90 ul de réaction exempt de cellules en 15 volumes égaux de 6 pi. Transférer chacun des 15 volumes dans un tube de réaction séparé. Fermer les tubes et les remettre sur la glace. Pour les volumes de réaction a augmenté, aliquoter en conséquence dans d'autres volumes de 6 pi. Étiquetez tous les tubes de réaction comme «CFR (+ ncaa)" (par exemple, CFR (+ Can)).
- Ajouter 15pi de tampon d'énergie, 15 ul de la solution aliquote d'acides aminés marquée "+ ncaa" (par exemple, + Can), 10 pi de 90 solution d'ADN nm et 20 pi de ddH stérile 2 O 30 pl d'extrait brut. Mélanger par pipetage vers le haut et vers le bas et légèrement vortex après addition de chaque ingrédient.
- Préparer la réaction acellulaire référence (90 ul) exprimant la protéine modèle non modifié.
- Incuber tous les tubes à 29 ° CO / N.
NOTE: Seuls les petits volumes de réaction permettent diff adéquates d'oxygèneusion dans la réaction qui est crucial pour l'expression des protéines très efficace. Les volumes de réaction de plus de 10 pi nécessitent l' oxygénation active à travers l' agitation 34. Pour les gros volumes, diviser la réaction en volumes inférieurs à 15 pi. - Après l'expression sans cellule, piscine toute identique composé des réactions fendues sans cellules de 6 pi. Tout d' abord, la piscine tous les 15 réactions fendues sans cellules de 6 pi étiquetés «CFR (+ cAA)" (par exemple, CFR (+ Arg)). Ensuite, la piscine tous les 15 réactions fendues sans cellules de 6 pi étiquetés «CFR (-cAA)" (par exemple, CFR (Arg)). Enfin, la piscine tous les 15 réactions fendues sans cellules de 6 pi étiquetés «CFR (+ ncaa)" (par exemple, CFR (+ Can)). Pour les volumes de réaction a augmenté, en conséquence en commun d'autres volumes de 6 pi.
- Vérifier le niveau de la protéine modèle d'expression dans tous les trois mis en commun, différemment composée des réactions sans cellules en effectuant dénaturant SDS-PAGE 43,44 (section 4.1).
REMARQUE: Utilisez cette métho d pour une évaluation préliminaire de l'expérience d'incorporation. - Préparer les protéines modèles exprimés sans cellules pour une analyse appropriée par spectroscopie de masse HPLC-ESI (section 4.4). Tout d' abord, purifier 45 les (section 4.2). Enfin, échanger le tampon (section 4.3) pour éviter le bruit de fond élevé lors de la spectroscopie de masse.
NOTE: Les sections 3.4.1, 3.4.2 et 3.4.3 conduisent à des conditions typiques sans cellules réaction 34: 08/09 à 09/09 mg / ml de protéine (extrait brut), 04.05 à 10.05 mM Mg-glutamate, 40-160 mM K-glutamate, 1 mM de chaque acide aminé à l'exception de la leucine, de la leucine 0,83 mM, HEPES 50 mM, 1,5 mM d'ATP et GTP mM de CTP 0,9 et UTP, 0,2 mg / ml d'ARNt, 0,26 mM de CoA réductase, mM NAD 0,33 AMPc 0,75 mM , 0,068 mM d'acide folinique, mM spermidine 1, 30 mM 3-PGA, 2% de PEG-8000 et 10 nM pBEST-OR2-OR1-Pr-UTR1-gene_of_model_protein-T500. Si on le désire, une autre procédure de préparation de réaction exempt de cellules peut être conduite qui conduit à des conditions de réaction ci-dessus.
- SDS-PAGE des réactions acellulaires
NOTE: Effectuer dénaturant SDS-PAGE pour une analyse rapide et préliminaire des protéines exprimées sans aucune autre purification ou d'extraction, et d'exécuter les étapes suivantes.- Décongeler la norme de protéines sur la glace. Veiller à ce qu'elle est constituée de protéines ayant des poids moléculaires similaires à la protéine exprimée modèle pour sa localisation sur le gel (étape 04/01/13).
Remarque: ici, la norme utilisée fournit des protéines sur une large gamme de poids moléculaires (myosine: 212 kDa, de liaison au maltose, une protéine β-galactosidase: 158 kDa, β-galactosidase: 116 kDa, Phosphorylase b: 97 kDa, l'albumine sérique : 66 kDa, déshydrogénase glutamique: 56 kDa, la protéine de liaison-maltose: 43 kDa, la thiorédoxine réductase: 35 kDa, triosephosphate: 27 kDa, inhibiteur de la trypsine: 20 kDa, lysozyme: 14 kDa, aprotinine: 7 kDa, l'insuline A: 3 kDa, la chaîne B: 2 kDa). - Puisque les réactions sans cellules sont légèrement visqueux en raison de la forte concentration de protéines, les diluer avec ddH stérile 2 O 5 à 10 fois avant de SDS-PAGE pour assurer la migration de gel approprié et pour éviter la saturation après coloration. Afin de ne pas perdre trop de l' échantillon, diluer 1 pl de réaction libre cellule dans 4 pi de ddH stérile 2 O. Préparer la dilution dans des tubes de réaction, bien vortex et peu tourner le liquide vers le bas avec une mini-centrifugeuse pour 2 - 3 sec à 2000 x g.
- Ajouter 5 pi de tampon 2x de chargement à 5 pi de réaction sans cellule diluée. Bien vortex et centrifuger avec une mini-centrifugeuse pour 2 - 3 sec à 2000 x g.
REMARQUE: Ici, après addition, les biomolécules sont dissous dans 62,5 mM de Tris / Cl -, 10% de glycerol, 2% de SDS et 0,0025% de bleu de bromophénol à pH 6,8, une composition typique. L'utilisation d'autres colorants de chargement qui conduisent à une dilution légèrement différente conditions peuvent convenir aussi bien. - Bien agiter au vortex le niveau de la protéine et transférer 15 pl de celui-ci dans un tube de réaction.
NOTE: Selon la taille du gel et pour d'autres normes de protéines, le volume recommandé peut différer de ce qui précède. - Placer les tubes de réaction dans un bloc chauffant. Vérifiez si les couvercles des tubes sont bien fermés. Chauffer à 95-100 ° C pendant 3 - 5 min. Faites ceci pour dénaturer les protéines et pour permettre SDS-enroulant autour de l'épine dorsale de protéine.
NOTE: Certaines normes de protéines ne doit pas être chauffée, voir les instructions du fournisseur. - Pendant ce temps, transférer le courant de tampon dans la chambre d'électrophorèse sur gel. Le contenu de la mémoire tampon est en cours d'exécution 1x Tris 25 mM, glycine 192 mM, SDS à 0,1% à pH 8,3 (avec du HCl).
- Fixer la préfabrication 4-20% de gradient de Tris-Glycine SDS-gel (10 cm x 10 cm x 1 mm) à la chambre d'électrophorèse. Retirer le peigne et rincer les puits avec une seringue chargée avec un tampon en cours d'exécution.
NOTE: Le stro de concentration de gelngly dépend de la taille et de la nature de la protéine exprimée modèle. La concentration au- dessus de la sépare E. coli de protéines de l' extrait brut ainsi que des protéines de faible poids moléculaire modèle. gels auto-cast conviennent aussi bien. - Retirer les tubes du bloc de chauffage. Spin le liquide vers le bas avec une mini-centrifugeuse pour 2 - 3 sec à 2000 x g. Peu de temps vortex et répéter filer vers le bas pour les 2 - 3 sec à 2000 x g.
- Transférer 15 pi de protéine étalon (24 - protéine 48 ug) et 10 pi (11-22 ug de protéine) de chaque échantillon dans les puits et commencer l'électrophorèse. Ici, SDS-PAGE est effectuée à 125 V et 20 - 40 mA pendant environ 90 min, un protocole typique.
- extraire délicatement le gel de la cassette. Transfert dans la solution de fixation (50% de méthanol, d' acide acétique à 10%, 40% ddH 2 O) pendant 30 min. ATTENTION! Le méthanol est toxique par inhalation et par contact avec la peau. Porter des gants de protection et à travailler sous une hotte.
- Transférer le gel dans une solution de coloration (0,025% bleu de Coomassie G-250, l' acide acétique à 10%, 90% ddH 2 O). Colorer pendant 60 min.
- Transférer le gel dans la solution de décoloration (acide acétique à 10%, 90% ddH 2 O) pendant 60-120 min.
NOTE: Fixation, coloration et de décoloration dépendent fortement de la taille et de la nature de la protéine exprimée. Ce protocole applique à une large gamme de protéines de poids moléculaires plutôt petites 46. - Déplacer le gel sur un transparent mat sur un fond blanc qui produit un contraste approprié pour les bandes de protéines colorées.
- Décongeler la norme de protéines sur la glace. Veiller à ce qu'elle est constituée de protéines ayant des poids moléculaires similaires à la protéine exprimée modèle pour sa localisation sur le gel (étape 04/01/13).
- La purification des protéines modèles étiquette His 45 des réactions acellulaires
Remarque: Pour la purification des protéines, il existe plusieurs méthodes qui donnent des résultats similaires. Ce protocole purifie les protéines modèles exprimés sans cellules qui ont une polyhistidine-tag C-terminal (His-tag). Il utilise un kit de purification appropriée pour des protéines qui sont Expressed dans les petits volumes de réaction de l'expression des protéines sans cellules. Il est identique pour la purification des protéines modèles natifs ou modifiés. Par conséquent, il est généralement décrit sur la base d'une réaction exempte de cellules unique.- Pour une HPLC-ESI analyse par spectroscopie de masse approprié, d'extraire et de purifier les protéines modèles de la réaction exempt de cellules et d'exécuter les étapes suivantes.
- Mélanger des volumes égaux de 90 - 150 pi de tampon de liaison His-90 et - 150 pl de réaction acellulaire. Pipeter haut et en bas, suivi par tourbillonnement doux.
NOTE: L'utilisation de His-tampon de liaison est recommandé. Cependant, le milieu réactionnel acellulaire peut être un matériau de départ aussi longtemps que la protéine modèle est soluble, le pH est compris entre 7,5 à 8, de l'imidazole / Sa concentration est <10 mM, la concentration des agents réducteurs forts est <15 mM et non métal- des agents chélateurs sont présents. Si votre volume de mélange dépasse 300 pi, diviser en volumes égaux et aliquote thèses volumes en différents rdes tubes de eaction pour les étapes de purification suivantes. - Préparer le système de colonne. Bien vortex la solution mère du gel Son affinité jusqu'à ce que la résine de gel soit complètement dissous. Transférer 250 pi de résine de gel dans la colonne. Utiliser un embout de pipette de 1 ml pour la résine de gel visqueux. Placer la colonne dans un tube de collecte.
- Centrifugeuse tube de colonne / collection pendant 5 - 10 sec à 13.000 - 15.000 x g. Assurez-vous que la résine de gel est complètement déchargée. Sinon, prolonger la durée de centrifugation par supplémentaire 5-10 sec, mais attention que le gel d'affinité est pas trop sèche. En conséquence de prolonger la durée de centrifugation dans les étapes 4.2.1.5, 4.2.1.7 et 4.2.1.9.
NOTE: La résine de gel est complètement déchargée si elle devient raide et pas surnageant reste sur le dessus. - Transférer 150 - 300 ul du tampon réactionnel acellulaire / His de liaison à la colonne. Si le volume de mélange dépasse le volume recommandé, réparti sur plusieurs colonnes de spin. Resuspendre la résine de gel par frequent taraudage et tourbillonnement doux pendant un temps d'incubation d'au moins 2 min. Pour des volumes supérieurs à 200 pi, incuber pendant encore 1 - 2 min.
NOTE: Le temps d'incubation suffisant est cruciale pour la liaison des protéines modèles à la résine de gel. - Centrifugeuse tube de colonne / collection pendant 5 - 10 sec à 13.000 - 15.000 x g. Jeter l'écoulement et placer la colonne dans le tube de collecte.
- Ajouter 250 pi de His-tampon de lavage. Resuspendre la résine de gel en tapotant et en tourbillonnement doux.
- Centrifugeuse tube de colonne / collection pendant 5 - 10 sec à 13.000 - 15.000 x g. Jeter l'écoulement et placer la colonne dans le tube de collecte. Répétez l'étape 4.2.1.6 et centrifuger à nouveau pendant 5 - 10 sec à 13.000 - 15.000 x g.
- Placer la colonne dans un tube réactionnel de 1,5 ml standard. Ajouter 150 ul de tampon d'élution et de remettre en suspension la résine de gel en appuyant dessus et tourbillonnement doux. Réduisez le volume à 100 pl pour une plus grande protéine purifiée modèle concentrations après élution dans l'étape suivante 4.2.1.9.
REMARQUE: La teneur totale de la protéine purifiée modèle peut être réduite en raison d'une éventuelle élution incomplète de toutes les protéines liées à la résine gel, si le volume de tampon d'élution est réduite. - Centrifuger le tube de colonne / de réaction pendant 5 à 10 secondes à 13 000 - 15 000 x g pour diluer la protéine purifiée dans le tampon d'élution. Si diviser avant, regrouper toutes les solutions en une seule solution de stock.
- Avant d' exécuter l'échange de tampon, déterminer la concentration en protéine en utilisant des procédés standard, par exemple, le dosage de Bradford 47,48.
NOTE: Pour une spectroscopie de masse HPLC-ESI appropriée (section 4.4), utiliser des concentrations de protéines optimales qui sont habituellement d'environ 0,5 mg / ml avec des volumes requis de 20 - 50 pi. Si la concentration est inférieure à 0,2 mg / ml, on concentre la solution de protéines après l'échange de tampon en utilisant un concentrateur à vide en rotation. Dans ce cas, lisez d'abord la section 4.3.8 pour adapter de manière appropriée le buff échangeer.
- Mélanger des volumes égaux de 90 - 150 pi de tampon de liaison His-90 et - 150 pl de réaction acellulaire. Pipeter haut et en bas, suivi par tourbillonnement doux.
- Pour une HPLC-ESI analyse par spectroscopie de masse approprié, d'extraire et de purifier les protéines modèles de la réaction exempt de cellules et d'exécuter les étapes suivantes.
- Échange de tampon pour la spectroscopie de masse HPLC-ESI
NOTE: Échange le tampon d'élution His-tag pour éviter le bruit de fond élevé dans l' analyse par spectroscopie de masse en raison d' une forte concentration d'imidazole (> 150 mM) et d' autres sels tels que NaH 2 PO 4 (> 300 mM) ou NaCl (> 50 mM) 49. Le protocole d'échange de tampon suivant utilise un système de colonne de centrifugation de filtration sur gel préhydraté. Elle est identique exécutée pour protéines modèles natifs ou modifiés. Par conséquent, il est généralement décrit sur la base d'une réaction exempte de cellules unique. Notez qu'il existe d' autres méthodes d'échange de tampons faciles à effectuer, par exemple, les cartouches mini-dialyse.- Préparer 100 ml de tampon de stockage de protéines (50 mM de Tris-Cl, pH 8, NaCl 100 mM, et 10% de glycerol). Peser dans un flacon de 100 ml autoclavable 605,7 mg de Tris-Base, 584,4 mg de NaCl et ajouter 10 ml de glycérol. Remplir jusqu'à environ 80 ml et ajuster la valeur de pH à 8 par unNaOH dding. vérifier régulièrement la valeur du pH avec un pH-mètre. Enfin, remplir jusqu'à la marque de 100 ml.
REMARQUE: utiliser cette mémoire tampon pour stabiliser une large gamme de protéines de modèle et de ne pas perturber l'analyse spectroscopique de masse, aussi longtemps que la CLHP est effectuée avant la mesure spectroscopique de masse. Toutefois, en fonction de la nature de la protéine modèle exprimé, utiliser d'autres tampons qui pourraient être mieux adaptés. Si votre protéine cible est pas stable au pH 8, ajuster le pH en conséquence. Ne pas utiliser des concentrations plus élevées de glycérol. - Réchauffez les colonnes de spin de filtration sur gel pendant au moins 15 min à température ambiante.
- Remettre en suspension le gel préhydratée en tapotant doucement ou vortex et éliminer les bulles d'air.
- Tout d'abord enlever le bouchon de fond, puis prendre le capuchon loin. Placer la colonne dans un tube de lavage (au moins 2 ml). Transférer le tube colonne / lavage dans la centrifugeuse. Chaque colonne possède un repère d'orientation. Centrifuger à 1000 g pendant 2 min pour éliminer le tampon de stockage de gel. Disqueard l'écoulement.
REMARQUE: Faites attention à procéder dans cet ordre. Faire en sorte que la colonne présente la même orientation dans toutes les autres étapes de centrifugation. - Ajouter jusqu'à 400 ul du tampon de stockage de la protéine. Centrifugeuse pendant 2 min à 1000 x g. Répéter cela pour charger complètement le tampon de stockage de la protéine dans le gel. ajouter avec précaution jusqu'à 100 ul de la solution de la protéine purifiée modèle. Introduire à la pipette directement sur le centre du lit de gel.
REMARQUE: les volumes de solution de protéines purifiées, en particulier des protéines modifiées, pourraient être plus élevés. Diviser ces solutions sur plusieurs colonnes d'échange de mémoire tampon. Les protéines doivent avoir des poids moléculaires supérieurs à 5 kDa pour ce type d'échange de tampon. - Placer la colonne dans un tube de collecte et centrifuger pendant 2 minutes à 1000 x g.
Remarque: Cette étape conduit à la dilution de la protéine purifiée modèle dans le tampon de stockage de la protéine. Si diviser avant, regrouper toutes les solutions en une seule solution de stock. - ème gel Flashsolution de protéine de e dans de l' azote liquide et conserver à -80 ° C ou directement l' analyser par HPLC-ESI spectroscopie 50 de masse (section 4.4). ATTENTION! Porter un eyeshield et Cryo-gants pour être protégés contre les projections d'azote.
- Effectuez les étapes suivantes du protocole pour concentrer la solution de protéine à l' aide d' un vide concentrateur de filage qui s'évapore soigneusement le solvant 51.
REMARQUE: Ces étapes sont nécessaires si la concentration en protéines est trop faible pour la spectrométrie de masse HPLC-ESI (<0,2 mg / ml). La concentration en protéines du volume de la solution et le modèle est à peu près le même avant et après l'échange de tampon. Le procédé de concentration est décrite au moyen de l'exemple suivant: Une fois l'étiquette His purification, la protéine modèle est dissous dans 100 pi de son tampon d'élution (étape 4.2.1.9) et a une concentration de 0,07 mg / ml (étape 4.2.1.10) .- Faire en sorte que la concentration en protéine et le volume de la solution sont, après évaporation àmoins de 0,2 mg / ml et 20 ul, respectivement, diluées avant l'échange de tampon, le tampon de stockage protéique préparée à l'étape 4.3.1 au moins trois fois, mais au maximum de cinq fois. s'évaporer De même dans ce qui suit au moins deux tiers (66,67 pi) ou au plus quatre cinquièmes (80 pi).
NOTE: Dilution du tampon de stockage de protéines avant l'échange de tampon est crucial de prendre en compte que les valeurs de concentration de ce tampon (étape 4.3.1) sont encore réalisées en dépit de l'évaporation. Après dilution dans du tampon de stockage protéique, effectuer d'abord l'échange de tampon (étapes 4.3.2 - 4.3.6) avant d'exécuter les étapes ci-après 4.3.8.2 - 4.3.8.4. - Après l'échange de tampon, mettre les complètes 100 ul de solution de protéine de modèle dans un tube ouvert dans le concentrateur sous vide de filage.
REMARQUE: La protéine modèle est dissous dans le tampon de stockage de la protéine diluée. Incline vers le centre de rotation pour éviter la perte de liquide pendant la rotation. Veiller à ce que une ébauche ouverte du même volume deH 2 O est placé de manière symétrique pour équilibrer le système de filature. - Fermez le couvercle du concentrateur et commencer la rotation. Pour assurer la stabilité des protéines, se concentrer à la température ambiante ou à basse température jusqu'à 30 ° C.
NOTE: Le concentrateur utilisé accélère automatiquement à 170 xg et établit le vide. Dans ce qui suit, il accélère jusqu'à sa vitesse maximale de 240 x g. - Fréquemment interrompre le processus de concentration pour surveiller le volume de liquide. Arrêter la concentration, quand le volume de la solution restante est comprise entre 20 pi et 33 pi.
NOTE: adapter en conséquence les étapes 4.3.8.1 - 4.3.8.4 pour les autres volumes de solution (étape 4.2.1.9) et d'autres concentrations de protéines (étape 4.2.1.10). Flash geler la solution concentrée de protéines dans de l' azote liquide et conserver à -80 ° C, ou directement l' analyser par HPLC-ESI spectroscopie de masse 50. PRUDENCE! Porter un eyeshield et Cryo-gants pour être protégés contre les projections d'azote.
- Faire en sorte que la concentration en protéine et le volume de la solution sont, après évaporation àmoins de 0,2 mg / ml et 20 ul, respectivement, diluées avant l'échange de tampon, le tampon de stockage protéique préparée à l'étape 4.3.1 au moins trois fois, mais au maximum de cinq fois. s'évaporer De même dans ce qui suit au moins deux tiers (66,67 pi) ou au plus quatre cinquièmes (80 pi).
- Préparer 100 ml de tampon de stockage de protéines (50 mM de Tris-Cl, pH 8, NaCl 100 mM, et 10% de glycerol). Peser dans un flacon de 100 ml autoclavable 605,7 mg de Tris-Base, 584,4 mg de NaCl et ajouter 10 ml de glycérol. Remplir jusqu'à environ 80 ml et ajuster la valeur de pH à 8 par unNaOH dding. vérifier régulièrement la valeur du pH avec un pH-mètre. Enfin, remplir jusqu'à la marque de 100 ml.
- HPLC-ESI analyse par spectrométrie de masse 50 de protéines modèles
- Effectuer une séparation par HPLC de 5 - solution à 15 ul de protéine (préparé dans la section 4.3) sur une colonne C5 phase inverse (3 pm, 100 x 2,1 mm) avec un gradient de 20% - 90% d'acétonitrile et 0,1% d'acide formique pendant 25 min et après la spectrométrie de masse ESI avec détection sur un analysator temps de vol de masse dans la gamme de 300 - 3000 m / z.
NOTE: En fonction de la nature de la protéine modèle exprimé, utiliser d'autres solvants ou des colonnes qui pourraient être mieux adaptés. - Déconvolution mesuré les spectres de masse en utilisant un logiciel approprié 52 pour calculer les masses de charge nulle.
- Effectuer une séparation par HPLC de 5 - solution à 15 ul de protéine (préparé dans la section 4.3) sur une colonne C5 phase inverse (3 pm, 100 x 2,1 mm) avec un gradient de 20% - 90% d'acétonitrile et 0,1% d'acide formique pendant 25 min et après la spectrométrie de masse ESI avec détection sur un analysator temps de vol de masse dans la gamme de 300 - 3000 m / z.
Résultats
Ce protocole guide à travers l'incorporation spécifique du résidu exempt de cellules de NCAAs en protéines modèles. Il propose SDS-PAGE pour une évaluation préliminaire de l'expérience d'incorporation et d'autres mesures pour préparer les protéines modèles pour une HPLC-ESI analyse spectroscopique de masse appropriée.
Ici, des résultats représentatifs de l'incorporation sans cellules spécifiques résidu de l'analogue Arg Can, ainsi que la Lys analogique L-hydroxy-lysine (Hyl) sont présentés. Les différentes solutions d'acides aminés, le tampon d'énergie, le codage de l'ADN vecteur pour la protéine de modèle et des réactions sans cellules sont préparées comme décrit ci-dessus. La réaction sans cellule référence est fournie avec la solution d'acides aminés constituée des 20 CAAS. Pour chaque expérience, une réaction négative sans cellule est fournie avec la solution d'acide aminé qui n'a pas l'analogique canonique de la NCAA en question. Pour chaque approche, une réaction sans cellule exprime la protéine modèle en présence de la solution d'acides aminés, où le cAA est substitué par l'analogue non canonique. His-tag purification, l'échange de tampon et HPLC-ESI analyse spectrométrique de masse sont effectuées selon le protocole décrit ci-dessus.
La protéine de modèle est le C-terminal marqué par His deGFP 32, une version tronquée de 53 EGFP. Sa séquence d'acides aminés à une lettre peut être trouvée dans (Matériau supplémentaire 2). Cette protéine modèle contient 6 Arg et 18 positions Lys, respectivement. Le vecteur d'expression est pBEST-OR2-OR1-Pr-UTR1-deGFP-T500.
Dans le cas de l'incorporation complète de la NCAA, on peut supposer que la réaction de contrôle négatif ne se prononce pas deGFP, puisque l'un des 20 CaaS est manquant. Contrairement, deGFP doit être détectable dans les deux autres réactions: celle native dans le référence réaction exempt de cellules et la protéine modifiée dans la réaction sans cellule qui est fourni avec la NCAA.
La figure 1A montre l'évaluation préliminaire SDS-PAGE de l'expérience d'incorporation Can. La réaction cellulaire sans référence a le niveau d'expression de deGFP plus haut. Dans la réaction acellulaire qui est pourvue de canettes, deGFP est exprimé à des concentrations légèrement plus faible. Aucune expression de deGFP peut être détectée dans le témoin négatif. Ce résultat SDS-PAGE est une bonne indication pour une intégration réussie de Can dans le deGFP de protéine cible.
Pour prouver l'incorporation complète de l' hypothèse peut dans deGFP, les deux protéines modèles purifiés, visualisées sur la figure 1B, sont analysés par spectrométrie de masse HPLC-ESI. La figure 1C montre les spectres de masse déconvoluée des molécules de deGFP purifiés. La masse déconvoluée de degF P qui est exprimé dans la réaction sans cellule référence est 26,192.8 Da. Pour deGFP exprimé dans le Can contenant la réaction sans cellule une masse de 26202,5 Da apparaît. Les masses attendues pour le deGFP6Arg natif et le deGFP6Can modifié avec Arg étant entièrement remplacés par Can sont 26.193 Da et 26.204 Da, respectivement. La différence de masse de 1,5 Da pour deGFP6Can est dans l'erreur de déconvolution du spectre. Ainsi, l'intégration complète de Can en deGFP à tous les 6 positions Arg est confirmée.
Les deux pics d'intensité réduite correspondent à la deGFP6Arg native et deGFP6Can modifiés qui n'a pas atteint leur fluorophore mature. Le fluorophore est généré de façon autocatalytique par élimination d'une molécule H 2 O, suivie par une oxydation. Cela conduit à une augmentation de la masse de 20 Da si ce processus ne se déroule pas.
pg "/>
Figure 1. SDS-PAGE évaluation de l'expérience peut l' incorporation et la spectroscopie de masse HPLC-ESI des exprimés et purifiés de molécules deGFP sans cellules. (A) L' évaluation préliminaire de l'expérience peut l' incorporation en utilisant SDS-PAGE. De gauche à droite: norme de protéines, la réaction sans cellule référence, contrôle négatif et de réaction sans cellule fournissant Can au lieu de Arg. (B) SDS-PAGE après His-tag purification et échange de tampon des molécules deGFP exprimées. De gauche à droite: standard Protein, deGFP purifiée à partir de la réaction de référence, purifié deGFP de la réaction de Can contenant. (C) Confirmation de la pleine intégration de Can par spectroscopie de masse HPLC-ESI. Les masses attendus du deGFP natif et le deGFP modifié avec Arg entièrement remplacée par Can sont 26.193 Da et 26.204 Da, respectivement. Chaque spectre est normalisé à son intensité maximale (chiffres). Les positions des pics sont indiquésDa. Pour des fins de visualisation, les pistes de gel sont extraites des images de gel, réunis, sont convertis en format échelle de gris, la taille est optimisée et le contraste ainsi que la luminosité sont améliorées. Pistes de gel originales sont présentées dans Matériel supplémentaire 3. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
La figure 2A montre l'évaluation préliminaire SDS-PAGE de l'expérience d'incorporation Hyl. Dans la réaction acellulaire qui est munie Hyl, deGFP est exprimé. Contrairement à la première expérience, une faible bande de deGFP peut être observée dans la réaction témoin négatif. Cela peut être dû à des résidus Lys dans les réactions sans cellules. Cela permet une expression de deGFP faible dans la réaction de contrôle négatif, où ni Lys ni Hyl sont ajoutés.
Pour H La spectroscopie de masse API-ESI, les molécules deGFP de la réaction exempt de cellules munies Hyl sont purifiés et le tampon est échangé (figure 2B).

Figure 2. Évaluation SDS-PAGE de l'expérience d'incorporation Hyl (A) De gauche à droite:. Réaction sans cellule témoin négatif, la réaction sans cellule contenant Hyl et le niveau de la protéine. (B) SDS-PAGE après His-tag purification et échange de tampon des molécules deGFP exprimées. De gauche à droite: Purifié deGFP du Hyl contenant la réaction et le niveau de la protéine. Pour des fins de visualisation, les pistes de gel sont extraites des images de gel, réunis, sont convertis en format échelle de gris, la taille est optimisée et le contraste ainsi que la luminosité sont améliorées. pistes de gel originales sont présentées dans Matériel supplémentaire 3.f = "https://www-jove-com.remotexs.ntu.edu.sg/files/ftp_upload/54273/54273fig2large.jpg" target = "_ blank"> S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
La figure 3 représente le spectre de masse des molécules d'déconvoluée deGFP purifié. La figure 3A confirme l'hypothèse de résidus Lys déjà présents dans les réactions sans cellules. Le pic prédominant du spectre correspond à la native (masse attendue: 26193 Da) deGFP. Encore une fois, les molécules deGFP d'un 20 Da supérieur de masse qui ne développent leur fluorophore peuvent être détectés. Les résidus Lys sont préférentiellement chargés sur l'ARNt Lys en conduisant à un niveau d'expression élevé des deGFP18Lys indigènes lysyl-ARNt-synthétase.
La différence de masse entre Hyl et Lys est de 16 Da. Grâce à la présence de Hyl qui est en concurrence avec les résidus Lys toutes les espèces possibles sont générés deGFP (deGFP18Hyl,deGFP17Hly + 1Lys, ..., + deGFP16Hyl 2Lys) (figure 3B). Il est vrai que le pic de deGFP1Hyl + 17Lys chevauche le sommet de la deGFP native qui ne produit pas de son fluorophore (figure 3A) et la masse de certains pics diffère de plus de 2 Da de la masse attendue. Ces différences de masse peuvent être attribués à un bruit élevé en raison des faibles quantités de l'espèce deGFP. Cependant, Hyl est généralement constituée par le système acellulaire. D'autres améliorations ont à faire pour supprimer les résidus Lys dans les réactions sans cellules.

. Figure 3. HPLC-ESI spectroscopie de masse des molécules d'deGFP purifiées de la réaction sans cellules contenant Hyl (A) Les deGFP18Lys indigènes est principalement détectée (masses attendues: avec fluorophore 26.193 Da, sans fluorophore matures 26.213 Da). (B)Grossissement révèle l'existence de toutes les espèces possibles de deGFP (deGFP18Hyl, deGFP17Hly + 1Lys, deGFP16Hyl + 2Lys, ..., deGFP1Hyl + 17Lys). Leurs masses sont attendues 26.193 Da + N x 16 Da (N = 1, ..., 18). Le spectre est normalisé à son intensité maximale (chiffres). Les positions des pics sont indiquées dans Da. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.

Supplemental modèle de calcul 1. Matériel. Dans la section 2, la préparation du mélange maître tampon d'énergie est illustrée en utilisant aliquotes brut extrait de 30 ul et optimale MG- et concentrations K-glutamate de respectivement 3 mM et 30 mM. Cet exemple conduit à un volume de mélange maître qui donne 3 fractions aliquotes de tampon d'énergie. Dans section 3, la préparation de la réaction sans cellule est illustré à l' aide d' une solution à 90 nM d' ADN stock conduisant à une concentration d'ADN de vecteur optimal de 10 nM dans la réaction sans cellule. S'il vous plaît cliquer ici pour télécharger ce fichier.
Pour une traçabilité appropriée, l'utilisation du modèle de calcul est illustré par l'insertion de ces valeurs typiques qui diffèrent de l'exemple ci-dessus: le volume brut extrait: 28 pi, optimal Mg-glutamate: 2 mM, optimal K-glutamate: 40 mM, nombre des aliquotes tampons souhaitées: 100, concentration de vecteur optimal en réaction exempt de cellules: 8 nM, vecteur solution stock d'ADN: 150 nM.
Entrez d'abord 28 pi que le volume d'extrait brut dans le champ d'orange de la première section de modèle. Ensuite, entrez dans la seconde section de modèle 2 mM und 40 mM de Mg comme optimale et les concentrations de K-glutamate dans les champs oranges. En tenant compte de la valeur optimale de K et Mg-concentration, la composition d'un tampon d'énergie de 15 ul, ainsi qu'un correspondant, ajusté aliquote de 16 ul est calculée. Ci-dessous, en conséquence entrer 100 comme nombre souhaité d'aliquotes de tampon d'énergie (16 pi). Le modèle adapte les volumes des différents composants du tampon pour le mélange maître de 1700 pi comme suit: 204 pi de solution mère 100 mM Mg-glutamate, 136 pi de solution de stock 3 M K-glutamate, 728.73 ul de 14x solution d'énergie, 510 pi de 40% de PEG-8000 et 121.27 ul stérile ddH 2 O. Enfin, dans la troisième section de modèle, entrez 8 nM et 150 nM comme concentration de vecteur optimal dans la réaction sans cellule et respectivement, ADN vecteur concentration de la solution de stock. Le modèle adapte les volumes des différents composants qui doivent être ajoutés aux 28 ul d'extrait brut pour finaliser la préparation d'un 90 piréaction acellulaire comme suit: 15 ul de tampon d'énergie, 15 pl d'une des solutions d'acides aminés composition différente 3, 4,80 ul de solution d'ADN de vecteur (150 nm) et 27,20 pi de ddH stérile 2 O.
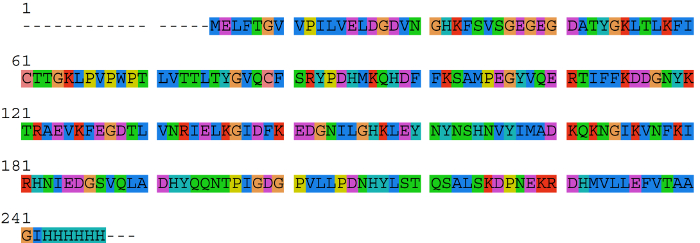
Matériel supplémentaire 2. Une lettre séquence d'acides aminés de la protéine modèle deGFP. Cette protéine modèle contient 6 Arg et 18 positions Lys. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.

Matériel supplémentaire 3. longueur et pistes de gel non modifiées qui correspondent aux images de gel présenté pleine figure 1 et 2. Les voies de gel présentées dans chaque individu s ubfigure sont extraits du même gel de polyacrylamide-SDS. Dans la figure 1 et 2 ces voies ont été réunies à des fins de présentation. Les poids moléculaires des bandes de protéines standards sont indiquées à côté des chiffres. (A.1) des pistes de gel de la figure 1A entières papillotées. De gauche à droite: norme de protéines, la réaction sans cellule référence, contrôle négatif et de réaction sans cellule fournissant Can au lieu de Arg. (A.2) des pistes de gel de la figure 1B entières papillotées. De gauche à droite: standard Protein, deGFP purifiée à partir de la réaction de référence, purifié deGFP de la réaction de Can contenant. (B.1) des pistes de gel de la figure 2A entières papillotées. De gauche à droite: réaction sans cellule témoin négatif, la réaction sans cellule contenant Hyl et le niveau de la protéine. (B.2) des pistes de gel de entières papillotées figure 2B. De gauche à droite: Purifié deGFP du Hyl contenant la réaction et le niveau de la protéine.d / 54273 / 54273supfig3large.jpg "target =" _ blank "> S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
Discussion
Un facile à utiliser le système d'expression sans cellule comme une stratégie viable au résidu spécifiquement incorporer NCAAs en protéines, est présenté. A cet effet, l'extrait brut est complété par de l'ADN codant vecteur pour la protéine d'intérêt, le tampon d'énergie et les acides aminés correspondants. A noter que le volume de l' aliquote de l' extrait brut dépend de l'extrait brut de la concentration en protéines 34. L'efficacité d'expression acellulaire est optimisée en fonction de la concentration de la construction d'ADN de vecteur. Les volumes des composants tampons d'énergie varie en fonction de la concentration optimale de Mg et K-glutamate afin de permettre à des rendements élevés de la protéine exprimée modèle acellulaire.
Une évaluation préliminaire de l'expérience d'incorporation peut être obtenue en effectuant une SDS-PAGE du milieu réactionnel acellulaire non purifié. Pour une analyse plus détaillée, la spectroscopie de masse HPLC-ESI est proposé comme un moyen pour vérifier complète, incor spécifique de résidusporation de la NCAA. Comme préparation pour ces derniers, les systèmes de colonnes de spin sont utilisés pour permettre His-tag purification et échange de tampon avec les petits volumes que nous utilisons dans ce protocole.
Y compris la spectroscopie de masse HPLC-ESI le protocole entier peut être effectuée dans les 2 jours. Il ne comprend pas les étapes particulièrement critiques. Cependant, les optimisations de concentration de Mg et K-glutamate, ainsi que du vecteur ADN sont cruciales afin d'exprimer des rendements élevés de la protéine modèle. L'utilisation du vecteur d'expression très efficace pBEST-OR2-Or1-Pr-UTR1-gene_of_model_protein-T500 est fortement recommandé. Elution de marquage His protéines est généralement due à une forte concentration d'imidazole (> 150 mM) et d' autres sels tels que NaH 2 PO 4 (> 300 mM) ou NaCl (> 50 mM) qui génèrent un bruit de fond élevé dans la masse analyse spectroscopique 49 . L'échange de ces tampons d'élution avec un tampon de stockage de protéine appropriée stabilise le modèle proteet considérablement réduit le bruit de fond lors de l'analyse par spectroscopie de masse.
Par conséquent, Can remplace Arg à six positions dans la protéine modèle. Dans le système d'expression, pas de résidus Arg peuvent être détectés. Cela simplifie l'incorporation spécifique de résidus d'analogues Arg par rapport à d' autres systèmes d'expression qui nécessitent de nouvelles stratégies d'épuisement 29,30. L'approche sans cellule présentée contourne les limites inhérentes aux approches in vivo qui sont dues à la toxicité Can ou une forte dépendance à l' égard séquence d'ARNm dans les stratégies 24,31 de production de protéines unique. Contrairement à l'emploi dans le système in vitro, le clivage in vivo de Can à homosérine et hydroxyguanidine ne se produit 31.
Cependant, le système acellulaire conserve une quantité suffisante de Lys en concurrence avec des analogues tels que Hyl. L'analyse par HPLC-ESI spectrométrie de masse montre que la protéine modèle contient à la fois, le canonical ainsi que l'analogue non canonique dans des proportions différentes. L'incorporation spécifique du résidu de Lys est possible en général, mais pour une substitution complète, de nouvelles stratégies d'épuisement, ou aaRS spécialement conçus et ARNt optimisé pour la reconnaissance de NCAAs doivent être développées.
Nous avons obtenu d'excellents rendements exprimés protéines modèles exempts de cellules, modifiées en ajoutant la NCAA à la même concentration que ceux canoniques. L'efficacité d'incorporation dépend de la nature de la ncaa à incorporer. Même les rendements plus élevés pourraient encore être réalisables en optimisant la concentration du ncaa.
Les résultats présentés démontrent l'applicabilité du système utilisé pour l'incorporation spécifique de résidus de NCAAs aussi longtemps qu'ils sont acceptés par le système de translation endogène canonique. Pour l'incorporation spécifique de résidus de NCAAs spécifiques, un des besoins supplémentaires pour vérifier si les résidus de la cAA perturba analogueb le système d'expression.
Systèmes de transcription-traduction exempts de cellules peuvent être conçus à partir de différents organismes pour répondre aux différentes demandes 54. Le tout E. coli transcription-traduction machineries du système sans cellule présentée ici permettent l'utilisation de bactériophages et E. promoteurs coli, et ils peuvent agir en parallèle ou consécutivement en cascades 55. L'applicabilité générale et la facilité d'utilisation font de la méthode, un outil puissant pour la recherche dans la toxicité des acides aminés et de l'application thérapeutique.
Déclarations de divulgation
The authors have nothing to disclose.
Remerciements
E.G. Worst and A. Ott acknowledge financial support by the Deutsche Forschungsgemeinschaft (DFG) within the collaborative research center SFB 1027 as well as Saarland University. E.G. Worst, A. Ott and V. Noireaux further acknowledge financial aid by the Human Frontiers Science Program Organization (HFSPO). The authors thank Tobias Baumann and Stefan Oehm (Institute of Chemistry, Technische Universität Berlin) for critical reading.
matériels
| Name | Company | Catalog Number | Comments |
| Protective eyewear | Sigma-Aldrich, St. Louis, USA | Z758841 | |
| Nitrile gloves (size S) | Sigma-Aldrich, St. Louis, USA | Z768960 | Catalog numbers other sizes: Z768979 for M, Z768987 for L and Z768995 for XL |
| Eppendorf Safe-Lock Tube 1.5 ml (PCR clean) | Eppendorf, Hamburg, Germany | 30123.328 | |
| Microbalance Discovery DV114CM | Ohaus, Greifensee, Switzerland | 80104140 | |
| Microspatula (L 6 5/8 in., stainless steel, rod diam. 0.09 in.) | Sigma-Aldrich, St. Louis, USA | Z243213 | |
| L-Canavanine | Sigma-Aldrich, St. Louis, USA | C9758 | Acute toxicity: wear eyeshields, dust mask, protective gloves |
| Hydroxylysine (racemic mixture) | Sigma-Aldrich, St. Louis, USA | H0377 | |
| Cryo-gloves (size S, water resistent) | Sigma-Aldrich, St. Louis, USA | Z183490 | Catalog numbers other sizes: Z183512 for M, Z183520 for L and Z183539 for XL |
| RTS Amino Acid Sampler | Biotechrabbit, Hennigsdorf, Germany | BR1401801 | For homemade preparation of amino acid stock solutions, follow this protocol35 and use the solid amino acid kit LAA21-1KT, L-proline (81709-25G), L-cysteine (30089-25G), L-histidine (53319-25G) and L-lysine (L5501-5G) (all from Sigma-Aldrich, St. Louis, USA) |
| HEPES | Sigma-Aldrich, St. Louis, USA | H6147 | |
| ATP | Sigma-Aldrich, St. Louis, USA | A8937 | |
| CTP | Affymetrix, Santa Clara, USA | 14121 | |
| GTP | Affymetrix, Santa Clara, USA | 16800 | |
| UTP | Affymetrix, Santa Clara, USA | 23160 | |
| tRNA (from E. coli, pack size 100 mg) | Sigma-Aldrich, St. Louis, USA | 10109541001 | Catalog number for pack size of 500 mg is 10109550001 |
| CoA | Sigma-Aldrich, St. Louis, USA | C4282 | |
| NAD (from yeast ) | Sigma-Aldrich, St. Louis, USA | N6522 | |
| cAMP | Sigma-Aldrich, St. Louis, USA | A9501 | |
| Folinic acid | Sigma-Aldrich, St. Louis, USA | F7878 | |
| 3-PGA | Sigma-Aldrich, St. Louis, USA | P8877 | |
| Mg-glutamate | Sigma-Aldrich, St. Louis, USA | 49605 | |
| K-glutamate | Sigma-Aldrich, St. Louis, USA | G1149 | |
| pBEST-OR2-OR1-Pr-UTR1-deGFP-T500 | Addgene, Cambridge, USA | Plasmid #40019 | |
| 4-20 % precast Tris-Glycine Gels (10 cm x 10 cm x 1 mm, 10 courses) | Anamed Elektrophorese, Groß-Bieberau / Rodau, Germany | TG 81610 | |
| SDS running buffer (10 x concentrate, 5000 ml) | Anamed Elektrophorese, Groß-Bieberau / Rodau, Germany | TG 50001 | |
| SDS loading buffer (2 x concentrate, 50 ml) | Anamed Elektrophorese, Groß-Bieberau / Rodau, Germany | TG 05002 | |
| Unstained protein marker, broad range (2-212 kDa) | New England Biolabs, Ipswich, USA | P7702S | |
| Methanol | Merck, Darmstadt, Germany | 1060091011 | Toxic by inhalation, in contact with skin and if swallowed: wear protective gloves and work under fume hood |
| Acetic acid (99.8 %) | VWR International, Darmstadt, Germany | 20104.447 | |
| Coomassie Blue G-250 (10 g) | Biozym Scientific, Hessisch Oldendorf, Germany | 902120 | |
| His-Spin Protein Miniprep kit | Zymo Research Europe, Freiburg, Germany | P2002 | Product also distributed by Zymo Research Corporation, Irvine, USA |
| Trizma Base | Sigma-Aldrich, St. Louis, USA | T1503 | |
| Hydrochloric acid | Sigma-Aldrich, St. Louis, USA | H1758 | |
| Glycerol, 99 % | VWR International, Darmstadt, Germany | 24397.296DB | |
| CentriPure Z25 mini spin columns | Genaxxon bioscience, Ulm, Germany | CP-0205-Z100 | |
| Sodium chloride | Sigma-Aldrich, St. Louis, USA | S9888 | |
| Concentrator 5301 | Eppendorf, Hamburg, Germany | 5301 000.210 | |
| 2xYT | MP biomedicals, Santa Ana, USA | 113012032 | |
| Bacto-Agar | BD Diagnostics, Franklin Lakes, USA | 214010 | |
| Bead-beating tubes (polypropylene microvials) | Biospec, Bartlesville, USA | 522S | |
| Beads, 0.1mm dia. | Biospec, Bartlesville, USA | 11079101 | |
| BL21 Rosetta 2 E. coli strain | Merck, Darmstadt, Germany | 71402 | |
| Bradford BSA protein assay Kit | Bio-Rad, München, Germany | 500-0201 | |
| Chloramphenicol | Sigma-Aldrich, St. Louis, USA | C1919 | |
| Cuvettes, 1.5ml | Thermo Fisher Scientific, Waltham, USA | 14-955-127 | |
| DTT | Sigma-Aldrich, St. Louis, USA | D0632 | |
| Micro Bio-Spin Chromatography Columns | Bio-Rad, München, Germany | 732-6204 | |
| Nunc 384-well optical bottom plates | Thermo Fisher Scientific, Waltham, USA | 142761 | |
| Nunc sealing tape | Thermo Fisher Scientific, Waltham, USA | 232701 | |
| PEG-8000 | Promega, Madison, USA | V3011 | |
| Potassium phosphate dibasic solution | Sigma-Aldrich, St. Louis, USA | P8584 | |
| Potassium phosphate monobasic solution | Sigma-Aldrich, St. Louis, USA | P8709 | |
| Slide-A-Lyzer dialysis cassettes, 10k MWCO (Kit) | Thermo Fisher Scientific, Waltham, USA | 66382 | |
| Spermidine | Sigma-Aldrich, St. Louis, USA | 85558 | |
| 1L centrifuge bottle | Beckman-Coulter, Brea, USA | A98813 | |
| 4L Erlenmeyer flask | Kimble Chase, Vineland (NJ), USA | 26500-4000 | |
| Avanti J-26XP centrifuge | Beckman-Coulter, Brea, USA | 393127 | Or centrifuge equivalent being able to centrifuge 1 l bottles. |
| Forma 480 orbital shaker | Thermo Fisher Scientific, Waltham, USA | 480 | Or shaker equivalent being able to shake chest-size 6 x 4 L . |
| JLA-8.1000 rotor | Beckman-Coulter, Brea, USA | 363688 | Or 5000 x g rotor equivalent for above centrifuge equivalent being able to centrifuge 1L-bottles. |
| Mini-Beadbeater-1 | Biospec, Bartlesville, USA | 3110BX | |
| Microfuge 22R refrigerated microcentrifuge | Beckman-Coulter, Brea, USA | 368831 | Or centrifuge equivalent being able to centrifuge 2 ml reaction tubes. |
| Heating block HLC HBT 130 | Labexchange, Burladingen, Germany | 24465 | Or heating block equivalent being able to heat samples in reaction tubes up to 100 °C |
| Eppendorf MiniSpin centrifuge | Eppendorf, Hamburg, Germany | 5452000018 | Or centrifuge equivalent being able to centrifuge 2 ml reaction tubes. |
| IKA Vortex 3 (4 mm orbital shaker diameter, 0 - 2500 rpm) | Sigma-Aldrich, St. Louis, USA | Z654760 | Or vortex equivalent |
| Scotsman AF103 ice flaker machine | Kälte-Berlin, Berlin, Germany | AF103 | Or ice flaker machine equivalent |
| MyTemp mini digital incubator | Sigma-Aldrich, St. Louis, USA | Z763314 | Or incubator equivalent being able to heat samples at 29 °C |
| EcoCell electrophoresis cell / chamber | Anamed Elektrophorese, Groß-Bieberau / Rodau, Germany | AN12005 | Or electrophoresis chamber equivalent being able to perform vertical gel electrophoresis with above precast gels or other used gels |
| Power-phor power supply for electrophoresis cell / chamber | Anamed Elektrophorese, Groß-Bieberau / Rodau, Germany | AN12001 | Or power supply equivalent being able to supply above used electrophoresis cell / chamber with power |
| VWR Signature Ergonomic High Performance Single-Channel Variable Volume Pipettors Starter Kit 1 | VWR International, Darmstadt, Germany | 613-5278 | Or equivalent micropipettes enabling to pipette similar volumes with the same precision |
| VWR Signature Ergonomic High Performance Single-Channel Variable Volume Pipettors Starter Kit 2 | VWR International, Darmstadt, Germany | 613-5279 | Or equivalent micropipettes enabling to pipette similar volumes with the same precision |
| Pipetman Tips Diamond D10ST (0.1 - 10 µl) | Gilson, Middleton, USA | F171101 | Or equivalent low-binding pipette tips enabling to pipette similar volumes with the same precision |
| Pipetman Tips Diamond D200ST (2 - 200 µl) | Gilson, Middleton, USA | F171301 | Or equivalent low-binding pipette tips enabling to pipette similar volumes with the same precision |
| Pipetman Tips Diamond D1000ST (100 - 1000 µl) | Gilson, Middleton, USA | F171501 | Or equivalent low-binding pipette tips enabling to pipette similar volumes with the same precision |
| 50 ml centrifuge tubes with screw caps (sterile) | VWR International, Darmstadt, Germany | 50-0156 | |
| 15 ml centrifuge tubes with screw caps (sterile) | VWR International, Darmstadt, Germany | 525-0150 | |
| 14 ml polypropylene tubes (round bottom, two-position vent stopper, sterile) | Greiner Bio-One, Frickenhausen, Germany | 187262 | |
| Discovery BIO Wide Pore C5 HPLC Column (3 µm particle size, L x I.D. 10 cm x 2.1 mm) | Sigma-Aldrich, St. Louis, USA | 567227-U | |
| Agilent 1260 HPLC machine | Agilent Technologies, Santa Clara, USA | G1312B | |
| 6500 Series Accurate-Mass Quadrupole Time-of-Flight (Q-TOF) LC/MS | Agilent Technologies, Santa Clara, USA | G6530BA | |
| Acetonitrile | Sigma-Aldrich, St. Louis, USA | 270717 | |
| FLUOstar Omega microplate reader | BMG Labtech, Ortenberg, Germany | 415-101 | Or microplate reader equivalent being able to measure the fluorescence of the expressed model protein |
| Hanna Checker pH meter | Sigma-Aldrich, St. Louis, USA | Z351091 | |
| Formic acid eluent additive for LC-MS | Sigma-Aldrich, St. Louis, USA | 56302 |
Références
- Böck, A., et al. Selenocysteine: the 21st amino acid. Mol. Microbiol. 5 (3), 515-520 (1991).
- Srinivasan, G., James, C. M., Krzycki, J. A. Pyrrolysine Encoded by UAG in Archaea: Charging of a UAG-Decoding Specialized tRNA. Science. 296 (5572), 1459-1462 (2002).
- Budisa, N. Prolegomena to Future Experimental Efforts on Genetic Code Engineering by Expanding Its Amino Acid Repertoire. Angew. Chem. Int. Ed. Engl. 43, 6426-6463 (2004).
- Wang, L., Xie, J., Schultz, P. G. Expanding the Genetic Code. Annu. Rev. Biophys. Biomol. Struct. 35, 225-249 (2006).
- Chin, J. W. Expanding and Reprogramming the Genetic Code of Cells and Animals. Annu. Rev. Biochem. 83, 379-408 (2014).
- Neumann, H. Rewiring translation - Genetic code expansion and its applications. FEBS Lett. 586 (15), 2057-2064 (2012).
- Liu, C. C., Schultz, P. G. Adding New Chemistries to the Genetic Code. Annu. Rev. Biochem. 79, 413-444 (2010).
- Goerke, A. R., Swartz, J. R. High-Level Cell-Free Synthesis Yields of Proteins Containing Site-Specific Non-Natural Amino Acids. Biotechnol. Bioeng. 102 (2), 400-416 (2009).
- Albayrak, C., Swartz, J. R. Cell-free co-production of an orthogonal transfer RNA activates efficient site-specific non-natural amino acid incorporation. Nucleic Acids Res. 41 (11), 5949-5963 (2013).
- Johnson, J. A., Lu, Y. Y., Van Deventer, J. A., Tirrell, D. A. Residue-specific incorporation of non-canonical amino acids into proteins: recent developments and applications. Curr. Opin. Chem. Biol. 14 (6), 774-780 (2010).
- Xiu, X., Puskar, N. L., Shanata, J. A. P., Lester, H. A., Dougherty, D. A. Nicotine binding to brain receptors requires a strong cation-pi interaction. Nature. 458 (7237), 534-537 (2009).
- Grünewald, J., et al. Mechanistic studies of the immunochemical termination of self-tolerance with unnatural amino acids. Proc. Natl. Acad. Sci. USA. 106 (11), 4337-4342 (2009).
- Nikić, I., Lemke, E. A. Genetic code expansion enabled site-specific dual-color protein labeling: superresolution microscopy and beyond. Curr. Opin. Chem. Bio. 28, 164-173 (2015).
- Munier, R., Cohen, G. N. Incorporation d'analogues structuraux d'aminoacides dans les protéines bactériennes. Biochim. Biophys. Acta. 21 (3), 592-593 (1956).
- Lepthien, S., Merkel, L., Budisa, N. In Vivo Double and Triple Labeling of Proteins Using Synthetic Amino Acids. Angew. Chem. Int. Ed. 49 (32), 5446-5450 (2010).
- Dieterich, D. C., Link, A. J., Graumann, J., Tirrell, D. A., Schuman, E. M. Selective identification of newly synthesized proteins in mammalian cells using bioorthogonal noncanonical amino acid tagging (BONCAT). Proc. Natl. Acad. Sci. USA. 103 (25), 9482-9487 (2006).
- Dieterich, D. C., et al. In situ visualization and dynamics of newly synthesized proteins in rat hippocampal neurons. Nat. Neurosci. 13 (7), 897-905 (2011).
- Hendrickson, W. A., Horton, J. R., LeMaster, D. M. Selenomethionyl proteins produced for analysis by multiwavelength anomalous diffraction (MAD): a vehicle for direct determination of three-dimensional structure. EMBO J. 9 (5), 1665-1672 (1990).
- Hoesl, M. G., et al. Lipase Congeners Designed by Genetic Code Engineering. ChemCatChem. 3 (1), 213-221 (2011).
- Bae, J. H., et al. Expansion of the Genetic Code Enables Design of a Novel "Gold" Class of Green Fluorescent Proteins. J. Mol. Biol. 328 (5), 1071-1081 (2003).
- Schachtele, C. F., Rogers, P. Canavanine death in Escherichia coli. J. Mol. Biol. 14 (2), 474-489 (1965).
- Rosenthal, G. A. The biological effects and mode of action of L-canavanine, a structural analogue of L-arginine. Q. Rev. Biol. 52 (2), 155-178 (1977).
- Rosenthal, G. A., Dahlman, D. L. Incorporation of L-Canavanine into Proteins and the Expression of Its Antimetabolic Effects. J. Agric. Food Chem. 39 (5), 987-990 (1991).
- Ishida, Y., Park, J. H., Mao, L., Yamaguchi, Y., Inouye, M. Replacement of All Arginine Residues with Canavanine in MazF-bs mRNA Interferase Changes Its Specificity. J. Biol. Chem. 288 (11), 7564-7571 (2013).
- Thomas, D. A., Rosenthal, G. A., Gold, D. V., Dickey, K. Growth Inhibition of a Rat Colon Tumor by L-Canavanine. Cancer Res. 46 (6), 2898-2903 (1986).
- Bence, A. K., Worthen, D. R., Adams, V. R., Crooks, P. A. The antiproliferative and immunotoxic effects of L-canavanine and L-canaline. Anticancer Drugs. 13 (3), 313-320 (2002).
- Bence, A. K., Crooks, P. A. The Mechanism of L-Canavanine Cytotoxicity: Arginyl tRNA Synthetase as a Novel Target for Anticancer Drug Discovery. J. Enzyme Inhib. Med. Chem. 18 (5), 383-394 (2003).
- Akaogi, J., et al. Role of non-protein amino acid L-canavanine in autoimmunity. Autoimmun. Rev. 5 (6), 429-435 (2006).
- Singh-Blom, A., Hughes, R. A., Ellington, A. D. An amino acid depleted cell-free protein synthesis system for the incorporation of non-canonical amino acid analogs into proteins. J. Biotechnol. 178, 12-22 (2014).
- Oh, S. -. J., Lee, K. -. H., Kim, H. -. C., Catherine, C., Yun, H., Kim, D. -. M. Translational Incorporation of Multiple Unnatural Amino Acids in a Cell-free Protein Synthesis System. Bioprocess. Eng. 19 (3), 426-432 (2014).
- Worst, E. G., Exner, M. P., De Simone, A., Schenkelberger, M., Noireaux, V., Budisa, N., Ott, A. Cell-free expression with the toxic amino acid canavanine. Bioorg. Med. Chem. Lett. 25 (17), 3658-3660 (2015).
- Shin, J., Noireaux, V. Efficient cell-free expression with the endogenous E. Coli RNA polymerase and sigma factor 70. J. Biol. Eng. 4 (8), (2010).
- Chemla, Y., Ozer, E., Schlesinger, O., Noireaux, V., Alfonta, L. Genetically expanded cell-free protein synthesis using endogenous pyrrolysyl orthogonal translation system. Biotechnol. Bioeng. 112 (8), 1663-1672 (2015).
- Sun, Z. Z., Hayes, C. A., Shin, J., Caschera, F., Murray, R. M., Noireaux, V. Protocols for Implementing an Escherichia coli Based TX-TL Cell-Free Expression System for Synthetic Biology. J. Vis. Exp. (79), e50762 (2013).
- Caschera, F., Noireaux, V. Preparation of amino acid mixtures for cell-free expression systems. Biotechniques. 58 (1), 40-43 (2015).
- Green, M. R., Sambrook, J. . Molecular Cloning: A Laboratory Manual. , (2012).
- Froger, A., Hall, J. E. Transformation of Plasmid DNA into E. coli Using the Heat Shock Method. J. Vis. Exp. (6), e253 (2007).
- Zhang, S., Cahalan, M. D. Purifying Plasmid DNA from Bacterial Colonies Using the Qiagen Miniprep Kit. J. Vis. Exp. (6), e247 (2007).
- Moreno, L. A., Cox, K. L. Quantification of dsDNA using the Hitachi F-7000 Fluorescence Spectrophotometer and PicoGreen Dye. J. Vis. Exp. (45), e2465 (2010).
- Desjardins, P., Conklin, D. NanoDrop Microvolume Quantitation of Nucleic Acids. J. Vis. Exp. (45), e2699 (2010).
- Sukumaran, S. Concentration Determination of Nucleic Acids and Proteins Using the Micro-volume Bio-spec Nano Spectrophotometer. J. Vis. Exp. (48), e2699 (2011).
- Laemmli, U. K. Cleavage of Structural Proteins during the Assembly of the Head of Bacteriophage T4. Nature. 227 (5259), 680-685 (1970).
- Hochuli, E., Bannwarth, W., Döbeli, H., Gentz, R., Stüber, D. Genetic Approach to Facilitate Purification of Recombinant Proteins with a Novel Metal Chelate Adsorbent. Nature Biotechnology. 6 (11), 1321-1325 (1988).
- Schägger, H., Aquila, H., Von Jagow, G. Coomassie blue-sodium dodecyl sulfate-polyacrylamide gel electrophoresis for direct visualization of polypeptides during electrophoresis. Anal. Biochem. 173 (1), 201-205 (1988).
- Bradford, M. M. A Rapid Method for the Quantitation of Microgram Quantities of Protein Utilizing the Principle of Protein-Dye Binding. Anal Biochem. 72, 248-254 (1976).
- Ernst, O., Zor, T. Linearization of the Bradford Protein Assay. J. Vis. Exp. (38), e1918 (2010).
- Banerjee, S., Mazumdar, S. Electrospray Ionization Mass Spectrometry: A Technique to Access the Information beyond the Molecular Weight of the Analyte. Int. J. Anal. Chem. 2012, 1-40 (2012).
- Fujiwara, K., Nomura, S. M. Condensation of an additive-free cell extract to mimic the conditions of live cells. PLoS One. 8 (1), e54155 (2013).
- Zhang, Z., Marshall, A. G. A universal algorithm for fast and automated charge state deconvolution of electrospray mass-to-charge ratio spectra. J. Am. Soc. Mass. Spectrom. 9 (3), 225-233 (1998).
- Li, X., Zhang, G., Ngo, N., Zhao, X., Kain, S. R., Huang, C. C. Deletions of the Aequorea victoria Green Fluorescent Protein Define the Minimal Domain Required for Fluorescence. J. Biol. Chem. 272 (45), 28545-28549 (1997).
- Gagoski, D., Polinkovsky, M. E., Mureev, S., Kunert, A., Johnston, W., Gambin, Y., Alexandrov, K. Performance benchmarking of four cell-free protein expression systems. Biotechnol. Bioeng. 113 (2), 292-300 (2016).
- Shin, J., Noireaux, V. An E. coli Cell-Free Expression Toolbox: Application to Synthetic Gene Circuits and Artificial Cells. ACS Synth. Biol. 1 (1), 29-41 (2012).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon