Method Article
Imagerie à haute résolution en direct du comportement des cellules dans le développement neuro-épithélium
Dans cet article
Résumé
L'imagerie des tissus embryonnaires en temps réel est difficile sur de longues périodes de temps. Nous présentons ici une analyse pour surveiller les changements cellulaires et sub-cellulaire de la moelle épinière de poulet pendant de longues périodes avec une résolution spatiale et temporelle. Cette technique peut être adaptée pour d'autres régions du système nerveux et de l'embryon en développement.
Résumé
Le cordon épinière embryonnaire se compose de vélo cellules progénitrices neurales qui donnent lieu à un grand pourcentage des cellules neuronales et gliales du système nerveux central (SNC). Bien que beaucoup est connu au sujet des mécanismes moléculaires qui motif de la moelle épinière et de susciter la différenciation neuronale 1, 2, nous manquons d'une compréhension profonde de ces événements précoces au niveau du comportement des cellules. Il est donc essentiel d'étudier le comportement des progéniteurs neuronaux en temps réel car ils subissent la neurogenèse.
Dans le passé, imagerie en temps réel des tissus embryonnaire précoce a été limitée par les cellules / tissus viabilité de la culture ainsi que les effets phototoxiques de l'imagerie de fluorescence. Ici, nous présentons un nouveau test pour l'imagerie des tissus tels pendant de longues périodes de temps, en utilisant un ex vivo roman protocole de culture tranche et à grand champ microscopie à fluorescence (Fig. 1). Cette approche permet d'atteindre à long terme time-lapse de surveillance de poussin embryonnaire scellules progénitrices de la moelle Pinal avec une résolution spatiale et temporelle.
Ce test peut être modifiée à l'image de toute une gamme de tissus embryonnaires 3, 4 En plus de l'observation des comportements cellulaires et sub-cellulaire, le développement du roman et des journalistes très sensibles pour l'activité des gènes (par exemple, la signalisation Notch 5) rend ce test un outil puissant permettant de comprendre comment de signalisation régule le comportement des cellules pendant le développement embryonnaire.
Protocole
1. Préparation de la vaisselle
- Plats utilisés pour la culture tranche sont à fond de verre (avec une lamelle de la base) (plats WillCo). Ces derniers sont placés sur du tissu optique dans une boîte de culture tissulaire de 6 cm pour garder le fond de verre propre.
- Le jour avant l'expérience, ajouter 2 ml de 0,1% de poly-L-lysine solution au plat à fond de verre et incuber pendant 5 minutes à température ambiante pour permettre à la poly-L-lysine recouvrir le fond de verre.
- Retirer la poly-L-lysine solution et rincer trois fois dans de l'eau désionisée, puis une fois dans l'éthanol à 70%.
- Laissez sécher une nuit. Les plats peuvent également être séché en chauffant légèrement dans un micro-ondes à faible puissance pendant 30-45 secondes.
2. L'électroporation d'embryons
- Incuber les œufs à 37 ° C à Hamburger-Hamilton (HH) étape 10 (~ 36 heures) (ou un autre stade de croissance désiré).
- Avant de commencer de préparer des aiguilles de verre (nous utilisons un Flaming / modèle de Brown p87 extracteur microcapillaire) et de rompre la pointe de l'needle à l'aide d'une pince fines sous une loupe binoculaire. La fin de l'aiguille doit être suffisamment pointu pour percer l'embryon tout en n'étant pas si étroite qu'elle entrave l'injection de la solution d'ADN.
- Oeufs de fenêtres et positionner les électrodes (5mm d'intervalle) de chaque côté de l'embryon
- Injecter de l'ADN (~ 0,025 à 0,5 ug / ul dans de l'eau déminéralisée coloré avec une petite quantité de vert rapide) dans le tube neural.
- Appliquer le courant - 12-17 V trois fois, la longueur d'impulsion 50ms avec 950 ms entre les impulsions.
- Nous utilisons de faibles concentrations d'ADN et des tensions d'électroporation bas pour obtenir l'expression de la mosaïque afin que nous puissions suivre les cellules individuelles.
- Couvrez la fenêtre dans la coquille d'oeuf avec scotch et assurez-vous qu'il est scellé.
- Laisser embryons de récupérer pendant 3-4 heures ou toute la nuit à 37 ° C.
3. Le collagène et de préparation moyen Slice Culture
- Préparer le mélange de collagène et d'un milieu de culture tranche d'environ une heure avant de trancher.
- Pour 300 t plype 1 collagène ajouter 100 ul solution à 0,1% d'acide acétique et 100 ml 5 x milieu L15. Vortex bien après chaque addition. La solution doit virer au jaune.
- Maintenant, ajoutez du bicarbonate de 15-20 pi de sodium à 7,5% et de manière approfondie vortex. La solution doit devenir légèrement rosée, et le volume de bicarbonate de sodium requise pour cela peut varier. Gardez sur la glace et maquillage frais à chaque fois.
- Pour 10 ml de milieu neurobasal ajouter B-27 supplément et Glutamax, à une concentration de 1 x final et une solution à 10 gentamicine ul.
- Lieu moyenne dans un incubateur à 37 ° C tamponnée avec 5% de CO 2. Laisser la partie supérieure du récipient lâche pour lui permettre de s'équilibrer avec le CO 2.
4. Culture Slice
- Retirer embryons à partir d'œufs et se laver dans du milieu L15.
- Lieu dans une boîte de culture tissulaire avec une couche de Sylgard au fond et les fixer à travers les entourant extra-embryonnaire de telle sorte que les membranes sont tendus.
- L'utilisation d'un microroknife, embryon tranche aussi droite que possible à travers la région d'intérêt. Pour la moelle épinière, des tranches doit être comprise entre 1-2 somites épais. Laissez tranches attachés à l'embryon pendant que vous coupez d'autres embryons afin de ne pas les perdre.
- Préparer une spatule 200 micropipette ul en coupant ~ 1 mm de la pointe et en joignant cette page à un p2 ou p10. Cette astuce va être utilisé pour transférer les tranches de la moelle épinière dans le plat à fond de verre. Un pourboire de 10 ul n'est pas assez large pour ramasser tranches.
- Manteau à l'intérieur de la pointe avec du collagène par pipetage 1 mix collagène ul préparé plus tôt. Laissez pendant 1-2 minutes, puis rincer à l'milieu L15. Cela permettra d'éviter le tissu de coller à l'intérieur de la pointe.
- Détachez tranche de l'embryon en utilisant un microknife et retirer de l'antenne en utilisant un p2 ou p10 munie d'une douille 200 pi et mis à 1 pl. Essayez de prendre place en tant que milieu le moins possible.
- Vortex du mélange de collagène et de goutte 5-8 pi de ce message à l'antenne de poly-L-lysine revêtu.
- ImmediaTely mis tranche embryon dans le collagène et la position en place avec une paire de pinces fines. Tranches doit être placé de sorte que le côté à imager est alignée avec la lamelle. Le tissu doit adhérer au revêtement poly-L-lysine sur la lamelle.
- Répétez jusqu'à ce que vous avez plusieurs tranches sur la lamelle. Nous pouvons généralement placer 6-9 tranches sur un plat.
- Une fois toutes les tranches sont en place, couvrir le plat et laissez le collagène de mettre pendant 20 minutes. Certaines des premières collagène placé peut-être déjà commencé à se dessécher. Pour éviter cela, ajoutez une petite quantité de L15 à ces avant de recouvrir.
- Une fois réglé, ajouter prudemment 2ml milieu de culture tranche qui a été équilibrée dans 5% de CO 2 à 37 ° C pendant au moins une heure. Des précautions doivent être prises pour ne pas déloger le collagène de la lamelle.
- Placer dans un 37 ° / 5% de CO 2 incubateur et permettent de récupérer les tranches pendant au moins 3 heures avant l'imagerie. S'il n'ya pas de placer le plat d'humidité, dans un esprit boîte de plexiglasha peu de papier absorbant humide dans un coin.
5. Tranches d'embryons d'imagerie
- Nous utilisons un Core DeltaVision grand champ microscope équipé d'une chambre de l'environnement StationMeteo à des tranches d'image. La chambre est maintenue constante à 37 ° C, avec un groupe CO 2 Dispositif de perfusion afin de maintenir la platine du microscope à 5% de CO 2/95% d'air.
- L'imagerie est normalement effectuée en utilisant un 40 x / 1,30 lentille à immersion d'huile et de NA images sont capturées avec un HQ2 Coolsnap refroidi caméra CCD.
- Z-sections sont capturés tous les 1,5 um à 45 um tissus. Le temps d'exposition doit être maintenue aussi basse que possible. Nous avons l'habitude d'exposer pour la 5-50 ms pour chaque section en Z. Les images sont capturées toutes les 7 minutes et jusqu'à 9 tranches peuvent être visités en utilisant le système de point précis DeltaVision de visiter fonction.
6. Les résultats représentatifs
Un exemple en accéléré séquence d'une cellule progénitrice moelle épinière estFig. 2a et Movie 1 correspondant. L'imagerie a été commencé sur une tranche de la moelle épinière de deux jours-vieux (stade HH 12) embryon. Cette cellule a été transfectées avec une construction exprimant GFP-αTubulin. Au cours de ce stade précoce, les cellules progénitrices neurales subissent principalement les divisions de mode géniteur-souches au cours de laquelle les cellules se divisent pour générer deux autres cellules progénitrices du cyclisme. Fig. Film 2b et correspondant 2 montre une cellule transfectée avec GFP-GPI (GPI ancrées GFP), le marquage de la membrane cellulaire. Cette cellule subit une division au cours de laquelle le processus de base se divise en deux et est également hérité par les cellules filles.
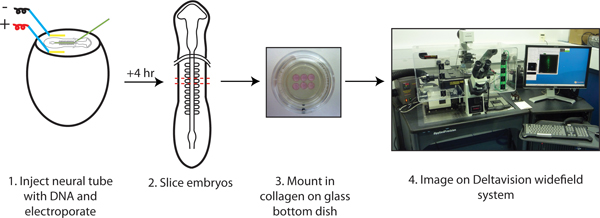
Figure 1. Protocole de culture Tranche d'imagerie time-lapse.

Figure 2. B portableehavior au cours du développement normal de la moelle épinière. (a) les cadres sélectionnés à partir de Film 1. Cellule exprimant la GFP-tubuline divise avec un plan de clivage qui est perpendiculaire à la surface apicale (2 h 20 min), générer deux cellules filles qui divisent une fois de plus (24 h 23 min et 25 h 54 min). (B) les cadres sélectionnés à partir de Movie 2. Cellules transfectées avec GFP-GPI subit la division cellulaire au cours de laquelle le processus de base est divisé (flèches blanches) et également hérité par les cellules filles.
Film 1. Cliquez ici pour voir le film .
Movie 2. Cliquez ici pour voir le film .
Discussion
Nous présentons ici une analyse de l'imagerie nouvelle time-lapse pour surveiller le comportement des cellules dans la culture tranche de poulet embryonnaire. Ce test permet l'imagerie haute résolution des tissus vivants pour un maximum de 70 heures, bien que les délais entre 24-48 heures sont plus faciles à capturer. L'utilisation d'un objectif NA élevés du pétrole et des intervalles relativement courts entre-temps des points permet l'acquisition d'images à haute résolution, ainsi que ce qui nous permet de surveiller le comportement des cellules qui se produit souvent rapidement, et peuvent facilement passer inaperçus au cours classique imagerie time-lapse en utilisant confocale microscopie.
Le principal avantage de ce test est la capacité de cellules d'image sur de longues périodes de temps. L'utilisation de grand champ 6 au lieu de confocale à balayage laser 7 microscopie est essentiel pour cela. La microscopie confocale a traditionnellement offert plusieurs avantages par rapport à champ large microscopie, y compris la capacité à prendre des sections optiques et d'éliminer de l'information sont directement axées 7, mais l'utilisation d'un trou d'épingle conduit à une perte de lumière au détecteur 8, à l'exclusion d'une quantité importante d'informations, et nécessitant des temps d'exposition plus longue. À grand champ microscopie, d'autre part, utilise illumination à fond plein et toute la lumière passant à travers l'objectif est envoyé au détecteur. Ceci, couplé avec un rendement quantique élevé refroidi dispositif à couplage de (CCD) assure l'imagerie avec un signal haute par rapport au bruit par rapport à la microscopie confocale 9, avec des temps d'exposition très rapides. Dans notre application, nous devons l'image pendant de longues périodes, record piles 3D et finalement résoudre les petits-près-diffraction des structures limitées. Bien que la microscopie confocale empêche l'extérieur de la focalisation de la lumière de jamais atteindre le détecteur, et donc de réduire de façon significative de fond dans épaisseur, à forte densité de marqués échantillons 7, 8, il ne réalise qu'une utilisable signal à bruit d'entrée de lumière avec beaucoup plus grande que large 10 microscopie en champ. Pour sparsel très sensible à la lumièreéchantillons y étiquetés comme nos électroporées tranches de la moelle épinière, par conséquent, la microscopie en champ large est plus performant que la microscopie confocale. Lorsqu'il est combiné avec la restauration d'images par déconvolution, qui élimine de l'information mise au point et améliore le contraste, à champ large est alors particulièrement bonne pour la détection de petits objets faibles 11. Bien que n'étant pas approprié pour toutes les expériences d'imagerie des tissus, cette approche a été très efficace pour notre application d'imagerie cellulaire en direct.
Le choix de la protéine fluorescente est un facteur important qui peut influer sur la survie des cellules. Nous constatons que les meilleurs résultats sont obtenus à partir de constructions qui utilisent la protéine fluorescente verte (GFP) 12 en tant que marqueur. Nous sommes en train d'évaluer une gamme de protéines fluorescentes rouges qui peuvent être efficacement utilisés en combinaison avec la GFP pour le canal de temps dual est caduque. Il existe une variété de ces protéines disponibles 13. Bien que de nombreuses protéines sont stables sous forme de fusions avec une protéine fluorescente, Certaines protéines peuvent être rendus instables par fusion, ce qui réduit la viabilité des cellules. Dans de tels cas, l'utilisation de constructions contenant un site d'entrée interne des ribosomes (IRES) est utile pour séparer la protéine d'intérêt de la protéine fluorescente.
Lorsque les tranches d'imagerie de la moelle épinière, des précautions doivent être prises pour minimiser l'exposition à la lumière fluorescente. Les oculaires du microscope doit être utilisé uniquement avec la lumière transmise (fond clair) pour localiser la position et les tranches. Images fluorescentes doivent toujours être acquis à l'aide du logiciel microscope en utilisant un minimum de temps d'exposition. Ceux-ci devraient être conservés dans la gamme de 5-50 ms et l'utilisation de filtres de densité neutre devrait être expérimenté. Bien que les temps d'exposition plus longs peuvent produire initialement des images plus claires, les effets phototoxiques de exposition à la lumière fluorescente peut conduire à la mort cellulaire. Les 5-10 premières um de tissu qui est la plus proche de la lamelle ne doit pas être représenté comme cette région contient des tissus qui peut avoir été endommagé pendant letranchage.
Bien que les exemples présentés ici sont des embryons au stade électroporés HH 10 et imagées 6-7 heures plus tard, cette méthode peut être utilisée pour les embryons à Son Altesse l'étape 18. Aux stades ultérieurs, la moelle épinière est beaucoup plus grande et est donc difficile de trancher la main. Dans ces cas, l'incorporation des embryons à faible point de fusion d'agarose et la coupe avec un vibratome peut donner de meilleurs résultats. Même si nous présentons cette analyse comme une méthode pour l'imagerie de cellules dans la moelle épinière en développement, cette approche a également été modifié pour d'autres images des tissus embryonnaires, y compris le sensorielle placodes 3.
Déclarations de divulgation
Pas de conflits d'intérêt déclarés.
matériels
| Name | Company | Catalog Number | Comments |
| Réactif | Entreprise | Numéro de catalogue | Commentaires |
| WillCo vaisselle | WillCo Wells | GWst-3522 | |
| poly-L-lysine | Sigma | P8970 | 0,1% dans du Tris |
| Le verre borosilicate capillaires | Harvard Instruments | GC100-10 | 1,0 mm de diamètre externe, 0.58mm diamètre intérieur |
| Électrodes (Genetrodes, dans ovo, en forme de L | BTX | 10-001850-01 | Or plaqué 5mm |
| ECM 830 carré générateur d'impulsions | BTX | 45-0002 | dans electroopration ovo |
| Fast Green | Sigma | F7258 | |
| Tapez collagène I de rat | Sigma | C3857 | |
| Milieu L-15 (en poudre) | Sigma | 2,76 g / L dans l'eau distillée pour solution 5x | |
| Le bicarbonate de sodium | Sigma | S8761 | Solution à 7,5% |
| Milieu Neurobasal | Invitrogen | 12348-017 | sans rouge de phénol |
| Glutamax | Invitrogen | A1286001 | Solution 100x |
| B-27 supplément | Invitrogen | 17504-044 | |
| La gentamicine | Invitrogen | 15710049 | 10 mg / ml |
| Du milieu L15 (liquide 1x) | Invitrogen | 11415-049 | |
| Microknife | Altomed | A10102 | 15 pente degré |
| Goupilles en acier inoxydable sans tête | Watkins et Doncaster | E6871 | Format A1 |
| Sylgard 184 élastomère | VWR | 634165S | |
| Système DeltaVision microscope de base | Applied Precision | ||
| Coolsnap HQ2 caméra CCD | Photometrics |
Références
- Ulloa, F., Briscoe, J. Morphogens and the Control of Cell Proliferation and Patterning in the Spinal Cord. Cell Cycle. 6, 2640-2649 (2007).
- Briscoe, J., Novitch, B. G. Regulatory pathways linking progenitor patterning, cell fates and neurogenesis in the ventral neural tube. Philosophical Transactions of the Royal Society B: Biological Sciences. 363, 57-70 (2008).
- Shiau, C., Das, R., Storey, K. An effective assay for high cellular resolution time-lapse imaging of sensory placode formation and morphogenesis. BMC Neuroscience. 12, 37 (2011).
- Wilcock, A. C., Swedlow, J. R., Storey, K. G. Mitotic spindle orientation distinguishes stem cell and terminal modes of neuron production in the early spinal cord. Development. 134, 1943-1954 (2007).
- Vilas-Boas, F., Fior, R., Swedlow, J. R., Storey, K. G., Henrique, D. A novel reporter of notch signalling indicates regulated and random notch activation during Vertebrate neurogenesis. BMC Biology. 9, 58 (2011).
- Herman, B. Fluorescence microscopy. Bios Scientific Publishers. , (1998).
- Conchello, J. -. A., Lichtman, J. W. Optical sectioning microscopy. Nat. Meth. 2, 920-931 (2005).
- Pawley, J. B., Pawley, J. B. . Fundamental Limits in Confocal Microscopy Handbook Of Biological Confocal. , (2006).
- Swedlow, J. R., Hu, K., Andrews, P. D., Roos, D. S., Murray, J. M. Measuring tubulin content in Toxoplasma gondii: A comparison of laser-scanning confocal and wide-field fluorescence microscopy. Proceedings of the National Academy of Sciences. 99, 2014-2019 (2002).
- Murray, J. M., Appleton, P. L., Swedlow, J. R., Waters, J. C. Evaluating performance in three-dimensional fluorescence microscopy. Journal of Microscopy. 228, 390-405 (2007).
- Biggs, D. S. C. 3D Deconvolution Microscopy. Current Protocols in Cytometry. , (2001).
- Chalfie, M., Tu, Y., Euskirchen, G., Ward, W., Prasher, D. Green fluorescent protein as a marker for gene expression. Science. 263, 802-805 (1994).
- Wiedenmann, J., Oswald, F., Nienhaus, G. U. Fluorescent proteins for live cell imaging: Opportunities, limitations, and challenges. IUBMB Life. 61, 1029-1042 (2009).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon