Method Article
Diagnóstico rápido basado en la reacción en cadena de la polimerasa cuantitativa (qPCR) de la infección por Helicobacter pylori y la resistencia a los antibióticos
* Estos autores han contribuido por igual
En este artículo
Resumen
El protocolo presenta un método no invasivo para el diagnóstico rápido de infecciones estomacales por Helicobacter pylori a través de la prueba de cadena y determina su resistencia a antibióticos a claritromicina y levofloxacino mediante reacción cuantitativa en cadena de la polimerasa (qPCR).
Resumen
Helicobacter pylori es un importante patógeno humano que infecta aproximadamente a la mitad de la población mundial y se está convirtiendo en una grave amenaza para la salud debido a su creciente resistencia a los antibióticos. Es el agente causal de la gastritis crónica activa, la enfermedad de úlcera péptica y el cáncer gástrico y ha sido clasificado como un carcinógeno del Grupo I por la Agencia Internacional para la Investigación del Cáncer. Por lo tanto, el diagnóstico rápido y preciso de H. pylori y la determinación de su resistencia a los antibióticos son importantes para la erradicación eficiente de este patógeno bacteriano. Actualmente, los métodos de diagnóstico de H. pylori incluyen principalmente la prueba de aliento con urea (UBT), la prueba de antígenos, la prueba de anticuerpos séricos, la gastroscopia, la prueba rápida de ureasa (RUT) y el cultivo bacteriano. Entre ellos, los tres primeros métodos de detección no son invasivos, lo que significa que son pruebas fáciles de realizar. Sin embargo, las bacterias no se pueden recuperar a través de estas técnicas; Por lo tanto, no se pueden realizar pruebas de resistencia a los medicamentos. Los últimos tres son exámenes invasivos, pero son costosos, requieren altas habilidades y tienen el potencial de causar daño a los pacientes. Por lo tanto, un método no invasivo, rápido y simultáneo para la detección de H. pylori y las pruebas de resistencia a los medicamentos es muy importante para erradicar eficientemente H. pylori en la práctica clínica . Este protocolo tiene como objetivo presentar un procedimiento específico que involucra la prueba de cuerdas en combinación con la reacción en cadena de la polimerasa cuantitativa (qPCR) para la detección rápida de la infección por H. pylori y la resistencia a los antibióticos. A diferencia de los cultivos bacterianos, este método permite un diagnóstico fácil, rápido y no invasivo del estado de infección por H. pylori y la resistencia a los medicamentos. Específicamente, utilizamos qPCR para detectar rea para la infección por H. pylori y mutaciones en los genes 23S rRNA y gyrA , que codifican resistencia contra claritromicina y levofloxacino, respectivamente. En comparación con las técnicas de cultivo utilizadas de forma rutinaria, este protocolo proporciona una técnica no invasiva, de bajo costo y que ahorra tiempo para detectar la infección por H. pylori y determinar su resistencia a los antibióticos mediante qPCR.
Introducción
H. pylori es una bacteria gramnegativa, en forma de espiral, altamente móvil, que vive principalmente en la región del píloro del estómago1. Es un patógeno común que infecta a casi el 50% de la población mundial2. La mayoría de las personas con infección por H. pylori no tienen manifestaciones clínicas, y la mayoría desarrolla diferentes enfermedades después de varios años de infección, incluyendo gastritis crónica, úlceras pépticas, úlceras gástricas y cáncer gástrico3. En varios estudios basados en diferentes poblaciones, se ha demostrado la eficacia de la eliminación de H. pylori para prevenir el cáncer de estómago y las lesiones precancerosas 4,5. Por lo tanto, el Centro Internacional de Investigaciones sobre el Cáncer de la Organización Mundial de la Salud (OMS) ha aconsejado la erradicación de H. pylori como medida preventiva6.
El uso de métodos no invasivos para identificar la infección por H. pylori es un componente clave del tratamiento para la mayoría de los individuos con dispepsia asintomática. La prueba de aliento con urea (UBT), la prueba de antígeno fecal de H. pylori (SAT) y las pruebas serológicas son técnicas no invasivas populares. Entre estos, el UBT es el procedimiento menos intrusivo y más preciso disponible. UBT utiliza ureasa, abundantemente presente en H. pylori, para hidrolizar urea marcada isotópicamente en amoníaco y dióxido de carbono (13C o 14C). Por el contrario, el ensayo inmunocromatográfico (ICA)7 es conveniente, simple y no invasivo para el muestreo. Sin embargo, la precisión de la prueba se ve afectada por varios factores, como la calidad de la muestra de heces, la temperatura y el intervalo entre la recolección de la muestra y la prueba. Otra prueba basada en la respuesta inmune es la prueba de anticuerpos séricos contra H. pylori , que detecta anticuerpos en el suero de un paciente. Sin embargo, esta prueba no es adecuada para el análisis posterior al tratamiento, ya que los anticuerpos permanecen mucho tiempo después de que las bacterias hayan sido eliminadas8. Otro inconveniente importante es que estos métodos solo diagnostican la infección por H. pylori y no permiten pruebas de resistencia a los medicamentos para guiar el tratamiento basado en la sensibilidad.
Para los métodos de prueba invasivos, el tejido de la biopsia gástrica debe tomarse mediante endoscopia y luego someterse a histología, la prueba rápida de ureasa y el cultivo bacteriano. Estos métodos de prueba también son muy limitados debido a varios factores. Actualmente, estas técnicas se limitan a pacientes ancianos, pacientes con alto riesgo de enfermedad precancerosa o maligna y pacientes que han fracasado en la terapia de primera línea para la enfermedad por reflujo gastroesofágico o infección por H. pylori 9. En segundo lugar, debido a las características de crecimiento únicas de H. pylori, la tasa de éxito del cultivo bacteriano solo alcanza el 50%10. Por lo tanto, los métodos de detección molecular ofrecen una nueva esperanza para superar las altas demandas de los métodos de detección invasivos y guiar el tratamiento basado en la sensibilidad. Entre los métodos de detección molecular, la PCR cuantitativa ha evolucionado enormemente en los últimos años. qPCR, a diferencia de la PCR tradicional, no requiere electroforesis en gel y cuantifica con precisión el ADN/ARN en muestras mediante la adición de cebadores y sondas en la etapa de recocido. Los kits de qPCR para la detección de la infección por H. pylori y la resistencia a los medicamentos ya están disponibles comercialmente. Sin embargo, cada método tiene sus limitaciones; Por lo tanto, el diagnóstico clínico y el tratamiento de un paciente deben considerarse junto con sus síntomas, signos, antecedentes, otras pruebas de laboratorio y respuesta al tratamiento.
Actualmente, el método principal para tratar las infecciones por H. pylori es tomar antibióticos, pero últimamente, cada vez es más difícil tratar estas infecciones debido al aumento de la resistencia a los antibióticos. Posteriormente, se ha observado una disminución significativa en la eficacia del tratamiento con H. pylori a nivel mundial, lo que hace que la erradicación de H. pylori sea un importante problema de salud pública11.
La claritromicina y la levofloxacina son los dos antibióticos de amplio espectro utilizados para tratar las infecciones causadas por H. pylori, pero varios estudios han reportado una resistencia generalizada contra estos dos medicamentos en aislados de H. pylori. A2143G, A2142G y A2142C son tres de las numerosas mutaciones puntuales encontradas en el gen 2.9 kb 23S rRNA que resultan en resistencia a la claritromicina al evitar que el macrólido se una. Al mismo tiempo, los loci de mutación del gen de resistencia a levofloxacino se localizan principalmente en los seis sitios de mutación (A260T, C261A, T261G, G271A, G271T, A272G) del gen gyrA 12. El descubrimiento de estos mecanismos de resistencia basados en mutaciones genéticas ha llevado a un cambio gradual en la detección de H. pylori a través de estudios basados en el cultivo a pruebas moleculares.
En general, existe una necesidad clínica urgente de un método de diagnóstico no invasivo, efectivo y simultáneo para la detección de infecciones por H. pylori y resistencia a los medicamentos. Adoptamos una prueba de cuerda combinada y un método de qPCR para superar las dificultades de muestreo y lograr el objetivo de la detección simultánea de la infección por H. pylori y la resistencia a los medicamentos utilizando diferentes sondas de cebador.
Protocolo
El presente estudio se realizó de conformidad con las consideraciones éticas establecidas por el comité ético del Hospital Popular Provincial de Guangdong, Universidad Médica del Sur, Guangzhou, China (Número de aprobación: KY-Q-2022-384-02). Los pacientes en el rango de edad de 18-60 años fueron incluidos en este estudio. Los pacientes que tomaron antibióticos, medicamentos chinos antibacterianos, medicamentos como inhibidores de la bomba de protones (IBP) o antagonistas de los receptoresH2 , etc., dentro de las 2 semanas anteriores a la prueba no se incluyeron en este estudio. Aquellos pacientes que habían recibido tratamiento contra H. pylori en los últimos 3 meses también fueron excluidos de este estudio. A las personas con problemas cardíacos, hepáticos, renales, neuropatía grave o enfermedad mental graves tampoco se les permitió participar en este estudio. No hubo mujeres embarazadas y madres lactantes incluidas en este estudio. Los detalles de los suministros (reactivos, productos químicos, equipos y software) utilizados en este estudio se dan en la Tabla de materiales.
1. Prueba de cadena para el muestreo de fluido gástrico
- Pídale al paciente que ayune durante la noche antes de la recolección de la muestra.
- Al día siguiente, abra el kit de prueba de cuerdas, tome la cápsula y mantenga el lazo al final de la cuerda.
- Pegue la cuerda en la mejilla del paciente y pídale que coloque la cápsula en la lengua cerca de la parte posterior de la garganta y que la trague con un sorbo de agua.
- Permita que la cápsula se disuelva en el estómago del paciente, exponiendo así la cuerda dentro de la cápsula para absorber el moco gástrico.
- Pídale a un asistente capacitado que saque la cuerda con cuidado 1 hora después de que el paciente tragó la cápsula.
- Corte el extremo inferior de la cuerda (40 cm) empapada en el líquido gástrico, colóquela en la solución de preservación de muestras de TSE (Tris/solución salina/EDTA) y luego envíela al laboratorio clínico a temperatura ambiente (RT) para la posterior detección de H. pylori y la determinación de los perfiles de resistencia a antibióticos mediante qPCR.
2. Extracción de ADN
- Coloque los tubos de recolección que transportan muestras en un estante de tubos de ensayo, debidamente etiquetados, y vórtice durante 10 s.
- Gire la placa de 32 pocillos (kit de extracción o purificación de ácidos nucleicos) varias veces para resuspender las perlas magnéticas. Después de un vórtice corto durante 10 s, retire con cuidado el sellado de papel de aluminio de la placa.
- Transfiera 200 μL de líquido gástrico de cada muestra y ADN de H. pylori como control positivo a los pocillos separados.
- Coloque la placa de 32 pocillos en la ranura de muestra correspondiente de la máquina extractora de ácidos nucleicos para la extracción automática de ADN.
- Después de la extracción y confirmación del ADN, iniciar las pruebas de resistencia a antibióticos para claritromicina y levofloxacino a través de qPCR en las muestras positivas.
- Coloque el exceso de líquido gástrico y las muestras de ADN extraídas a -20 °C para su almacenamiento a largo plazo y uso futuro.
- Realice todos estos pasos en un gabinete de bioseguridad para evitar cualquier contaminación.
3. qPCR para la detección de H. pylori y resistencia a antibióticos (claritromicina y levofloxacino)
- Realizar qPCR para la detección de H. pylori y las mutaciones sospechosas de resistencia a antibióticos en su genoma.
- Descongele la mezcla de reacción qPCR (kit de detección de ácido nucleico H. pylori ) en hielo y mézclela moviendo y girando para evitar cualquier pérdida de reactivo.
- Para cada muestra en una placa de 32 pocillos, mezclar 20 μL de mezcla de reacción por PCR (premezcla de reacción de genotipado [que contiene la enzima Taq, trifosfato de desoxirribonucleósido, tampón de reacción y cloruro de magnesio], con cebadores de ureA hacia adelante y hacia atrás, y una sonda de ureA), y 5 μL del ADN extraído.
- Ejecute la placa qPCR de 32 pocillos en la máquina qPCR. Programe el termociclador: la mezcla de reacción reaccionará primero secuencialmente a 42 °C y 95 °C (ambos durante un ciclo) durante 2 min cada uno; luego, 40 ciclos a 95 °C, desnaturalización durante 10 s, y recocido y extensión a 58 °C durante 45 s.
- Establezca la adquisición de la señal de fluorescencia en FAM (H. pylori) y la adquisición de datos en el período de extensión de amplificación. Una vez completada la reacción, guarde los datos para futuros análisis de resultados.
- Analice los datos utilizando un software específico para qPCR, ya que el instrumento seleccionará automáticamente los umbrales de referencia.
- Si el resultado de la prueba para H. pylori es positivo, la máquina iniciará automáticamente las pruebas de resistencia a los medicamentos en la muestra. Reemplace el kit de reactivos con reactivos de detección para mutaciones del gen 23S rRNA y del gen gyrA en el kit H . Pylori (qPCR).
NOTA: El kit de reactivos (reactivos de detección para mutaciones del gen 23S rRNA y del gen gyrA en Helicobacter pylori [qPCR]) incluye la mezcla de reacción de PCR (premezcla de reacción de genotipado [que contiene la enzima Taq, trifosfato de desoxirribonucleósido, tampón de reacción, cloruro de magnesio], cebadores y sondas). Todos los productos de control de calidad negativo son agua estéril y purificada. El producto de control de calidad positivo en el kit de detección de ácido nucleico de H. pylori son perlas estándar de H. pylori inactivadas (ATCC 43504). Los productos de control de calidad positivos fuertes y positivos débiles de H. pylori en el kit (reactivos de detección para el gen 23S rRNA y mutaciones del gen gyrA en H. pylori) son el ADN de la cepa inactivada de H. pylori que contiene el gen objetivo y el gen mutante. Del mismo modo, según la situación real, todos los estándares de diagnóstico, incluida la presencia de infección por H. pylori o resistencia a los medicamentos, se establecen en TC ≤ 35 y tienen una curva típica en forma de S. La concentración del control de calidad débil es de 1,0 x 103 copias/ml, mientras que la concentración del control de calidad fuerte es de 1,0 x 108 copias/ml.
Resultados
Detección de infección por H. pylori y resistencia a antibióticos en líquido estomacal mediante qPCR
Realizamos qPCR para la detección de la infección por H. pylori mediante la amplificación del gen ureA y determinamos su perfil de resistencia a antibióticos dirigiéndonos a mutaciones puntuales en el gen 23S rRNA y el gen gyrA (Tabla 1). Los valores de TC de control de calidad en los tres grupos de los experimentos de qPCR estaban dentro del rango recomendado, lo que indica que todas las muestras estaban en un estado normal en el momento del experimento y que los resultados de la prueba eran confiables. En este estudio, se seleccionaron cinco muestras con diferentes resultados de prueba (S1-S5) para caracterizar la confiabilidad del protocolo experimental. S1 representa una cepa representativa de H. pylori sin infección, mientras que las muestras de S2-S5 son aquellas infectadas con H. pylori con diferentes resultados de resistencia (Figura 1). Configuramos el sistema para que no realizara más pruebas de resistencia con las muestras no infectadas con H. pylori, por lo que la muestra S1 no ingresó a la prueba de resistencia después de que la prueba del sistema mostró un resultado negativo para H. pylori. En cuanto a las muestras positivas para la infección por H. pylori, los valores de TC S2 estaban todos dentro del rango de detección, lo que indica que la muestra era H. pylori positiva y mostró doble resistencia a claritromicina y levofloxacino, y se recomendó a los médicos que eligieran otros métodos de tratamiento a su discreción. Los valores de TC S3 estuvieron dentro del rango de detección para la infección por H. pylori y la prueba de resistencia a levofloxacino, mientras que no se detectaron valores de TC en la prueba de resistencia a claritromicina, lo que indica que la muestra S3 era de un paciente resistente a levofloxacino. Del mismo modo, el valor de TC de la muestra S4 estuvo dentro del rango de detección para la infección por H. pylori y la resistencia a la claritromicina, mientras que no se detectó ningún valor de TC para la resistencia a la levofloxacina, lo que indica que este paciente era resistente a la claritromicina, y se recomendó que tomaran levofloxacino para el tratamiento. Finalmente, la prueba de muestra S5 mostró valores de TC dentro del rango de detección solo para la detección de infección por H. pylori, lo que indica que este paciente era sensible a ambos antibióticos y podía ser tratado con cualquiera de los dos medicamentos. En comparación con el método de cultivo bacteriano, que también detecta la infección por H. pylori y la resistencia a los medicamentos, este método es seguro y eficaz para detectar la infección por H. pylori y la resistencia a los medicamentos sin causar daño al paciente y se puede utilizar para guiar al médico en la formulación de un plan de tratamiento adecuado.
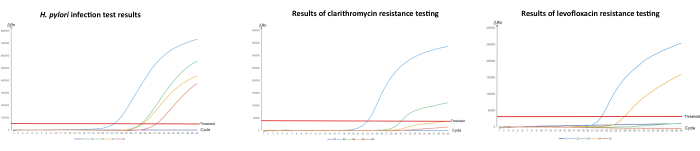
Figura 1: Detección de H. pylori y su resistencia a antibióticos en líquido estomacal por qPCR.
(A) Amplificación cuantitativa por PCR de la infección por H. pylori , (B) detección de resistencia a claritromicina y (C) detección de resistencia a levofloxacino. "S" significa muestra. "S1" es una muestra que resultó negativa para la infección por H. pylori y también se analizó la resistencia a los antibióticos; "S2" es una muestra infectada por H. pylori con resistencia tanto a claritromicina como a levofloxacino; "S3" también es una muestra positiva para H. pylori pero es susceptible a la claritromicina y resistente a la levofloxacina; "S4" también es una muestra positiva para H. pylori pero es resistente a la claritromicina y susceptible a la levofloxacina; "S5" es una muestra positiva para H. pylori pero es susceptible tanto a claritromicina como a levofloxacina. La concentración del control de calidad débil es de 1,0 x 103 copias/ml, mientras que la concentración del control de calidad fuerte es de 1,0 x 108 copias/ml. Haga clic aquí para ver una versión más grande de esta figura.
| Muestra | H. pylori | Claritromicina | Levofloxacino | |||
| +/- | CT | +/- | CT | +/- | CT | |
| S1 | - | U | - | U | - | U |
| S2 | + | 22.61 | + | 22.77 | + | 23 |
| S3 | + | 28.32 | - | U | + | 30.18 |
| S4 | + | 28.76 | + | 27.67 | - | U |
| S5 | + | 31.59 | - | U | - | U |
Tabla 1: Tabla que muestra los resultados de la qPCR de la detección de infección por H. pylori y resistencia a claritromicina y levofloxacino. Esta tabla presenta los resultados cualitativos para la infección por H. pylori , la detección de mutaciones en el gen 23S rRNA que muestran que el aislado es resistente a la claritromicina y la detección de mutaciones en el gen gyrA que muestran que el aislado es resistente a la levofloxacina. +/−, resultado cualitativo; +, resultado positivo; −, resultado negativo.
Discusión
La detección de H. pylori se puede realizar utilizando métodos invasivos y no invasivos13. Las técnicas invasivas comúnmente utilizadas, como la histopatología, la prueba rápida de ureasa, la reacción en cadena de la polimerasa (PCR) y el cultivo bacteriano requieren endoscopia y biopsia. Entre los procedimientos no invasivos se recomiendan pruebas serológicas, pruebas de aliento con urea y ensayos inmunoabsorbentes ligados a enzimas (ELISA)14. Si bien los métodos no invasivos son fáciles de realizar, económicos y más cómodos para los pacientes, el aislamiento y el cultivo de H. pylori para realizar ensayos adicionales, como la identificación de cepas y las pruebas de susceptibilidad a los antibióticos, requieren pruebas invasivas. Con el aumento actual de la resistencia a los antibióticos, los antibióticos utilizados anteriormente se están volviendo ineficaces y, por lo tanto, no pueden tratar la infección causada por aislados de H. pylori resistentes a los medicamentos. En este caso, puede ser necesario recuperar H. pylori para realizar pruebas de susceptibilidad a los antibióticos para seleccionar los antibióticos que probablemente eliminarán con éxito las bacterias resistentes.
Los ensayos de susceptibilidad basados en cultivos realizados rutinariamente, como el método de dilución en agar y la prueba del epsiómetro (prueba E) tienen varias limitaciones: requieren mucho tiempo, ya que H. pylori es una bacteria de crecimiento lento, costosa, basada en habilidades y requiere técnicas invasivas. Se pueden utilizar técnicas moleculares alternativamente diferentes, como las técnicas de hibridación fluorescente in situ (FISH) y la qPCR, para identificar varias mutaciones, como en el gen 23S rRNA, que codifica la resistencia contra la claritromicina15,16. Se seleccionaron tres sitios de mutación puntual (A2142G, A2143G, A2142C) del gen de resistencia al ARNr 23S de H. pylori y seis sitios de mutación puntual (A260T, C261A, T261G, G271A, G271T, A272G) del gen de resistencia a gyrA de antibióticos quinolonas para diseñar cebadores y sondas para determinar la resistencia contra claritromicina y levofloxacino, respectivamente.
La prueba de cuerdas, que se originó a partir de la ENTEO-TEST, utiliza una cápsula unida a una cuerda de nylon altamente absorbente, que se traga para recoger las secreciones gástricas17. Actualmente, las pruebas de cuerda se han utilizado para diagnosticar la tuberculosis18, para diferenciar Klebsiella pneumoniae (hvKp) altamente virulenta de Klebsiella pneumoniae19 tradicional, para identificar bacterias y levaduras Gram-positivas y Gram-negativas, y para diagnosticar H. pylori a partir del fluido gástrico20. En este estudio, combinamos la prueba de cuerda, una técnica mínimamente invasiva, con qPCR para la identificación de infecciones por H. pylori en una configuración clínica y realizamos pruebas de susceptibilidad dirigidas a mutaciones que codifican resistencia previamente informadas en el genoma de H. pylori. Elegimos el gen de la ureA porque es un gen de mantenimiento exclusivo de H. pylori sin riesgo potencial de reacción cruzada con otros organismos. Todos los aislados de H. pylori deben tener el gen de la ureA para sobrevivir en el estómago humano, y los experimentos de knock-out han demostrado que H. pylori sin el gen de la ureA no tiene la capacidad de colonizar en el estómago.
Para la qPCR, los estándares de control de calidad son extremadamente importantes. En la detección por qPCR de H. pylori, los requisitos de las normas de control de calidad son los siguientes: un producto de control de calidad negativo (sin aumento de la señal de fluorescencia en la vía de detección FAM, sin curva de amplificación típica de tipo S, valor de TC > 35,00 o sin señal obvia); un producto de control de calidad positivo (curva de crecimiento de la señal de fluorescencia de la vía de detección FAM que muestre una curva en forma de S, valor CT ≤ 35,00), y los requisitos anteriores deberán cumplirse simultáneamente; de lo contrario, la prueba se considerará no válida y deberá realizarse nuevamente. Además, para la detección por qPCR de la resistencia de H. pylori a antibióticos (claritromicina y levofloxacino), excepto para el valor de TC que distingue entre resultados positivos y negativos, que cambia a 30.00 para la detección de resistencia, los otros requisitos son los mismos que los anteriores. En particular, la información de secuencia sobre los cebadores y las sondas no está disponible en este documento porque involucra derechos de propiedad comercial.
Con el fin de seleccionar antibióticos más eficientes para cada paciente, es crucial estudiar la prevalencia local de H. pylori y su perfil de resistencia a los antibióticos debido al aumento de la resistencia a los antimicrobianos a nivel mundial y, además, debido a la variación en los patrones de resistencia entre varias áreas geográficas. La principal limitación de nuestro estudio es que el tamaño de la muestra fue muy pequeño y no cubrió diferentes regiones geográficas en China para mostrar la prevalencia de la infección por H. pylori y su perfil completo de resistencia a antibióticos, pero la técnica de prueba de cuerdas está bien desarrollada, y al combinarla con qPCR, logramos un diagnóstico simultáneo de H. pylori Detección de infecciones y farmacorresistencias. Estos resultados sugieren que este enfoque podría usarse a mayor escala en diferentes regiones del mundo.
Divulgaciones
Ninguno.
Agradecimientos
Este trabajo fue apoyado por el Proyecto Sanming de Medicina en Shenzhen (Subvención No. SZSM201510050) y la Fundación de Investigación Básica y Aplicada de Guangdong (Subvención Nº 2022A1515220023). Fundación de Investigación para Talentos Avanzados del Hospital Popular Provincial de Guandong (No. KJ012021097), y la Fundación Nacional de Ciencias Naturales de China (81871734, 82072380, 82272423). Los financiadores no tuvieron ningún papel en el diseño del estudio, la recopilación y el análisis de datos, la decisión de publicar o la preparación del manuscrito.
Materiales
| Name | Company | Catalog Number | Comments |
| 23S rRNA and gyrA gene point mutations detection kit (PCR-Fluorescence Probing) | Hongmed Infagen | Detection of Helicobacter pylori resistance to clarithromycin and levofloxacin | |
| ABI 7500 fluorescence quantitative PCR machine | Thermo Fisher Scientific | SEDA 20163220767 | Fluorescent quantitative PCR amplification |
| ABI 7500 software | Thermo Fisher Scientific | Data Analysis | |
| BSC-1500IIA2-X | BIOBASE | SEDA 20143222263 | Biosafety cabinet |
| DNA extraction kit | Daan Gene | ||
| E-Centrifuge | WEALTEC | Centrifuge the residual liquid off the wall of the tube. | |
| H. Pylori DNA detection kit (PCR-Fluorescence Probing) | Hongmed Infagen | Testing for H. pylori infection | |
| Stream SP96 automated nucleic acid extractor | Daan Gene | SEDA 20140104 | For DNA extraction |
| String test kit | Hongmed Infagen | It contains a capsule attached to a string, scissors, cotton swab, and sample preservation tube | |
| Ultra-low temperature freezers (DW-YL450) | MELING | SEDA 20172220091 | -20 °C for storing reagents |
| Vortex-5 | Kylin-bell | For mixing reagent |
Referencias
- Proença-Modena, J. L., Acrani, G. O., Brocchi, M. Helicobacter pylori: Phenotypes, genotypes and virulence genes. Future Microbiology. 4 (2), 223-240 (2009).
- Hussein, R. A., Al-Ouqaili, M. T. S., Majeed, Y. H. Association between alcohol consumption, cigarette smoking, and Helicobacter pylori infection in Iraqi patients submitted to gastrointestinal endoscopy. Journal of Emergency Medicine, Trauma and Acute Care. 2022 (6), 12(2022).
- Reshetnyak, V. I., Burmistrov, A. I., Maev, I. V. Helicobacter pylori: Commensal, symbiont or pathogen. World Journal of Gastroenterology. 27 (7), 545-560 (2021).
- Thrift, A. P., Wenker, T. N., El-Serag, H. B. Global burden of gastric cancer: Epidemiological trends, risk factors, screening and prevention. Nature Reviews Clinical Oncology. 20 (5), 338-349 (2023).
- Liou, J. M., et al. Screening and eradication of Helicobacter pylori for gastric cancer prevention: The Taipei global consensus. Gut. 69 (12), 2093-2112 (2020).
- IARC Helicobacter pylori Working Group. Helicobacter Pylori Eradication as A Strategy for Preventing Gastric Cancer. , International Agency for Research on Cancer. Lyon, France. (2014).
- Vaira, D., et al. The stool antigen test for detection of Helicobacter pylori after eradication therapy. Annals of Internal Medicine. 136 (4), 280-287 (2002).
- Laheij, R. J. F., Straatman, H., Jansen, J. B. M. J., Verbeek, A. L. M. Evaluation of commercially available Helicobacter pylori serology kits: A review. Journal of Clinical Microbiology. 36 (10), 2803-2809 (1998).
- Malfertheiner, P., et al. Management of Helicobacter pylori infection - The Maastricht IV/ Florence consensus report. Gut. 61 (5), 646-664 (2012).
- Peng, X., et al. Gastric juice-based real-time PCR for tailored Helicobacter Pylori treatment: A practical approach. International Journal of Medical Sciences. 14 (6), 595-601 (2017).
- Thung, I., et al. Review article: The global emergence of Helicobacter pylori antibiotic resistance. Alimentary Pharmacology and Therapeutics. 43 (4), 514-533 (2016).
- Zhang, Y., et al. Mutations in the antibiotic target genes related to clarithromycin, metronidazole and levofloxacin resistance in Helicobacter pylori strains from children in China. Infection and Drug Resistance. 13, 311-322 (2020).
- Hussein, R. A., Al-Ouqaili, M. T. S., Majeed, Y. H. Detection of Helicobacter pylori infection by invasive and noninvasive techniques in patients with gastrointestinal diseases from Iraq: A validation study. PLoS One. 16 (8), e0256393(2021).
- Chen, Q., et al. Advanced sensing strategies based on different types of biomarkers toward early diagnosis of H. pylori. Critical Reviews in Analytical Chemistry. , (2023).
- Hussein, R. A., Al-Ouqaili, M. T. S., Majeed, Y. H. Detection of clarithromycin resistance and 23SrRNA point mutations in clinical isolates of Helicobacter pylori isolates: Phenotypic and molecular methods. Saudi Journal of Biological Sciences. 29 (1), 513-520 (2022).
- Xuan, S. H., Wu, L. P., Zhou, Y. G., Xiao, M. B. Detection of clarithromycin-resistant Helicobacter pylori in clinical specimens by molecular methods: A review. Journal of Global Antimicrobial Resistance. 4, 35-41 (2016).
- Perez-Trallero, E., Montes, M., Alcorta, M., Zubillaga, P., Telleria, E. Non-endoscopic method to obtain Helicobacter pylori for culture. Lancet. 345 (8950), 622-623 (1995).
- DiNardo, A. R., et al. Use of string test and stool specimens to diagnose pulmonary tuberculosis. International Journal of Infectious Diseases. 41, 50-52 (2015).
- Li, G., et al. Identification of hypervirulent Klebsiella pneumoniae isolates using the string test in combination with Galleria mellonella infectivity. European Journal of Clinical Microbiology and Infectious Diseases. 39 (9), 1673-1679 (2020).
- Agbonlahor, D. E., Odugbemi, T. O., Udofia, P. O. Differentiation of gram-positive and gram-negative bacteria and yeasts using a modification of the "string" test. The American Journal of Medical Technology. 49 (3), 177-178 (1983).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados