Method Article
Clasificación del estado de activación de células madre neurales in vitro mediante autofluorescencia
En este artículo
Resumen
Este protocolo describe estrategias para identificar y enriquecer el estado celular en cultivos primarios de células madre neurales de ratón adulto mediante imágenes de autofluorescencia utilizando i) un microscopio confocal, ii) un clasificador de células activadas fluorescentes para realizar imágenes de intensidad, o iii) un microscopio multifotónico para realizar imágenes de fluorescencia de por vida.
Resumen
Las células madre neurales (NSC, por sus siglas en inglés) se dividen y producen neuronas recién nacidas en el cerebro adulto a través de un proceso llamado neurogénesis adulta. Las NSC adultas son principalmente inactivas, un estado celular reversible en el que han salido del ciclo celular (G0) pero siguen respondiendo al medio ambiente. En el primer paso de la neurogénesis adulta, las NSC inactivas (qNSC) reciben una señal y se activan, saliendo de la quiescencia y volviendo a entrar en el ciclo celular. Por lo tanto, la comprensión de los reguladores de la inactividad y la salida de la quiescencia de NSC es fundamental para futuras estrategias dirigidas a la neurogénesis adulta. Sin embargo, nuestra comprensión de la inactividad de las NSC está limitada por las limitaciones técnicas en la identificación de las NSC inactivas (qNSC) y las NSC activadas (aNSC). Este protocolo describe un nuevo enfoque para identificar y enriquecer las qNSCs y las aNSCs generadas en cultivos in vitro mediante la obtención de imágenes de la autofluorescencia de NSC. En primer lugar, este protocolo describe cómo utilizar un microscopio confocal para identificar marcadores autofluorescentes de qNSCs y aNSCs para clasificar el estado de activación de NSC utilizando la intensidad de la autofluorescencia. En segundo lugar, este protocolo describe cómo utilizar un clasificador de células activadas fluorescentes (FACS) para clasificar el estado de activación de NSC y enriquecer las muestras para qNSC o aNSC utilizando la intensidad de la autofluorescencia. En tercer lugar, este protocolo describe cómo utilizar un microscopio multifotónico para realizar imágenes de vida útil de fluorescencia (FLIM) con resolución de una sola célula, clasificar el estado de activación de NSC y rastrear la dinámica de la salida de reposo utilizando tanto intensidades de autofluorescencia como tiempos de vida de fluorescencia. Por lo tanto, este protocolo proporciona un conjunto de herramientas de resolución de células vivas, sin etiquetas y de una sola célula para estudiar la inactividad y la salida de la inactividad de NSC.
Introducción
Las NSC crean neuronas recién nacidas a lo largo de la vida en muchos organismos en un proceso conocido como neurogénesis adulta 1,2. Para producir neuronas recién nacidas, primero debe activarse una qNSC, entrando en el ciclo celular para expandir la población y producir progenitores neuronales 3,4,5,6. Aunque se sabe mucho sobre la inactividad de las NSC, nuestra capacidad para identificar completamente los impulsores y reguladores de la inactividad de las NSC está limitada por las limitaciones técnicas que existen para aislar e identificar las qNSC y su transición a la activación. Las imágenes de autofluorescencia han tenido éxito anteriormente en el estudio de los cambios en el estado celular en muchos tipos diferentes de células, como la microglía y las células T, al resolver la remodelación metabólica, que influye en las propiedades ópticas de los cofactores metabólicos autofluorescentes como el fosfato de dinucleótido de nicotinamida y adenina (NAD(P)H) y el dinucleótido de flavina adenina (FAD)7,8. Las NSC remodelan sustancialmente sus redes metabólicas a medida que experimentan una salida de quiescencia 9,10,11,12,13,14. Por lo tanto, para aprovechar estas diferencias, la autofluorescencia de NSC se utilizó recientemente para identificar y enriquecer el estado de activación de NSC mediante la detección de cambios en la autofluorescencia atribuidos a la remodelación metabólica que ocurre cuando las NSC salen de la quiescencia15. La autofluorescencia por imagen proporciona varias ventajas técnicas: i) no requiere la adición de marcadores exógenos, lo que puede afectar el comportamiento celular; ii) puede proporcionar datos de alta resolución de una sola célula sobre el estado de activación del NSC; y iii) no requiere la destrucción de la célula 7,16. Este protocolo describe tres estrategias para aprovechar la autofluorescencia de NSC para estudiar los estados de reposo y activación de las células NSC15.
Recientemente, se encontró que las NSC aisladas de ratones machos de 6 semanas de edad de la zona subgranular del hipocampo, cultivadas y puestas en quiescencia reversible in vitro 10,13,17,18,19,20,21, exhiben niveles aumentados de autofluorescencia punteada (PAF) que excitan entre 400-600 nm y emiten entre 500-700 nm. Esta señal era específica de las qNSC en comparación con las NSC cíclicasactivadas 15. La capacidad de separar visualmente estas dos poblaciones sin el uso de marcadores de anticuerpos o reporteros adicionales es útil para muchas preguntas experimentales sobre la naturaleza de las qNSC y las salidas de inactividad. Por lo tanto, en primer lugar, este protocolo describe estrategias para obtener imágenes de la PAF en qNSC utilizando un microscopio confocal, que se puede utilizar para identificar el estado de activación de NSC. En segundo lugar, este protocolo describe estrategias para detectar el PAF mediante la clasificación de células activadas por fluorescencia (FACS) y describe además cómo clasificar en función de esta señal para enriquecer qNSC o aNSC. Estas estrategias proporcionan una medida que se puede usar para agrupar y separar las NSC en función del estado de la célula.
Para desarrollar un método de mayor resolución para separar las NSC no solo en estados distintos, sino también a medida que pasan de la salida de quiescencia hacia la activación completa, se realizaron imágenes de fluorescencia de por vida (FLIM) utilizando un microscopio multifotónico para obtener imágenes de la autofluorescencia de NAD(P)H (denominado Canal 1) y la autofluorescencia verde (denominada Canal 2; que detecta tanto la autofluorescencia FAD como la PAF en las qNSC) junto con su intensidad. Este enfoque aprovecha el hecho de que las propiedades ópticas de las moléculas en la célula dependen de sus propiedades físicas16,22. Por ejemplo, el NAD(P) (NAD y el NADP son ópticamente indistinguibles, por lo que el NAD(P) se utiliza para referirse a ambas especies) no es autofluorescente en el estado oxidado, pero sí en su estado reducido (NAD(P)H)23. Además, las propiedades físicas adicionales de las moléculas autofluorescentes, como su estado de unión a las enzimas, se pueden extrapolar mediante la realización de imágenes de fluorescencia de por vida 7,22,24. Por ejemplo, el NAD(P)H tiene una vida útil de fluorescencia más corta cuando no está unido a una enzima22. Dado que las moléculas autofluorescentes como el NAD(P)H, que participa en cientos de reacciones metabólicas, son utilizadas de forma diferente por las células que progresan a través de diferentes estados o comportamientos celulares, estos cambios pueden detectarse y cuantificarse utilizando un microscopio multifotónico que detecta la vida útil de la autofluorescencia23. Junto con la abundancia, o intensidad, de la autofluorescencia, estas medidas proporcionan información multidimensional para separar las NSC en un estado celular u otro y a través de las transiciones dinámicas entre estados. En tercer lugar, este protocolo describe la realización, el análisis y la interpretación de las medidas de intensidad y FLIM de las señales de Canal 1 (NAD(P)H) y Canal 2 (PAF) utilizando un microscopio multifotónico. En resumen, este protocolo describe un conjunto de herramientas de células vivas y sin etiquetas para estudiar la inactividad de NSC que proporciona datos de alta resolución de una sola célula sobre el estado de NSC.
Protocolo
Todos los procedimientos de este protocolo están aprobados por el Comité Institucional de Cuidado y Uso de Animales (IACUC) de la Universidad de Wisconsin-Madison.
1. Uso de un microscopio confocal para obtener imágenes de PAF en qNSC y aNSC para identificar el estado de la célula NSC
- Genere qNSCs y aNSCs in vitro a partir de cultivos primarios de NSCs.
- Cultivo de NSCs purificadas a partir de cerebros de ratones adultos en un medio proliferativo para su expansión (Tabla 1)18,20,21. Por lo tanto, de forma predeterminada, los cultivos de NSC in vitro son aNSC.
- Para generar qNSC, utilice el siguiente protocolo para colocar aNSC en cristalería revestida y, a continuación, trátelas con medio qNSC para inducir la inactividad durante 3 días.
- Prepare la cristalería con un índice de refracción adecuado para los objetivos de inmersión en aceite para adherir NSC recubriendo primero con una solución de poli-L-ornitina (PLO; 10 mg/mL en agua para plástico, 50 mg/mL en agua para vidrio) suficiente para cubrir el fondo de la placa (para un pozo de cubeta de imágenes de1 cm 2 , use ~ 150 μL) durante 1 h a 37 °C en ddH2O.
- Retire la solución de PLO y lave 3 veces con solución salina tamponada con fosfato (PBS).
- Cubra con una solución de laminina (5 mg/mL) suficiente para cubrir el fondo del plato (para un pozo de cubeta de imágenes de 1 cm2 , use ~ 150 μL) en PBS e incube como mínimo durante 3 h a 37 °C, o durante la noche.
- Retire la solución de laminina, luego agregue un medio de cultivo suficiente para cubrir el fondo de la placa (para un pozo de cubeta de imágenes de 1 cm2 , use ~ 150 μL).
- Prepare las células para la tripsinización centrifugando las aNSC (pueden comenzar como monocapas o neuroesferas) a 120 x g durante 4 min a temperatura ambiente (RT, ~25 °C).
- Tripsinizar las aNSC peletizadas del paso anterior en 100 μL de mezcla de tripsina durante 5 min a 37 °C y luego enfriar la tripsinización con 200 μL de mezcla de inhibidores de tripsina (Tabla 1).
- Deje reposar los NSC durante 2 minutos, luego tritúrelos mecánicamente pipeteándolos hacia arriba y hacia abajo 10 veces.
- Agregue 5 mL de medio aNSC y haga girar las células a 120 x g durante 4 min.
- Vuelva a suspender las NSC en 1 mL de medio aNSC y coloque las células en una confluencia del 10%-20% en el medio aNSC. Por ejemplo, coloque ~5.000 células en un pocillo de una cubeta de imágenes de 1cm2 con un medio de 150 μL.
- Después de dejar pasar 24 horas para que las aNSC se adhieran a la superficie de la placa, retire el medio aNSC y reemplácelo con un medio qNSC suficiente para cubrir el fondo de la placa (para un pocillo de cubeta deimágenes de 1 cm 2, use ~ 150 μL; Tabla 1- como se describió anteriormente 10,13,17,18) en pozos en los que se inducirá la quiescencia.
- Cambie el medio aNSC y qNSC al menos una vez cada 2 días. Después de 3 días en medio qNSC, las NSC están inactivas y se pueden utilizar para experimentos de imagen.
- Utilice un microscopio confocal para obtener imágenes de qNSC y aNSC vivas in vitro.
NOTA: Cada microscopio tiene su software patentado; Por lo tanto, siga estos pasos realizando acciones análogas para configurar un microscopio específico. Este protocolo proporcionará instrucciones para trabajar en NIS-Elements.- Configure el microscopio confocal para la obtención de imágenes de autofluorescencia.
- Haga clic en Láser 405 y escriba en la ventana de emisión dentro del menú Láser 405 . Haga clic en TD para capturar una imagen de luz transmitida y establecer el valor HV para visualizar la muestra.
- Ajuste la potencia del láser a 3,5% y la ganancia a 75. Establezca Zoom en 2. Configure los parámetros de escaneo confocal, establezca Pixel Dwell en 0.5 y Resolution en 2048 x 2048 pixels.
- Enfoca las celdas bajo una lente de objetivo de inmersión en aceite de ~ 60x usando campo claro para enfocar.
- Cambie al modo de escaneo confocal haciendo clic en Eye Port, asegurándose de que la trayectoria de la luz vaya ahora al cabezal de escaneo y de que no haya ningún cubo de filtro en la ruta de la luz.
- El PAF está enriquecido en qNSCs y es ampliamente excitable y emitible (ver Figura 1). Elija la mejor configuración óptica en función de las capacidades del sistema utilizando la Figura 1 como guía. Para obtener la señal más fuerte y limpia, excite con un láser entre ~ 400-500 nm, recoja ~ 580-620 nm y use una lente de objetivo de inmersión en aceite de ~ 60x.
NOTA: Las imágenes de autofluorescencia requieren potencias láser más altas para excitarse que las que requerirían los experimentos de imágenes típicos. Si obtiene imágenes de otras moléculas autofluorescentes u otras etiquetas junto con PAF, diseñe cuidadosamente la configuración óptica para evitar el sangrado en los canales de autofluorescencia. - Adquiera imágenes de autofluorescencia en qNSCs y aNSCs. Haga clic en Escanear, luego enfoque en la imagen y, a continuación, haga clic en Capturar. Para obtener las mejores imágenes, recoja imágenes de un solo plano con el citoplasma de las células (basado en una autofluorescencia más tenue distribuida difusamente por toda la célula) enfocada para obtener una sección transversal representativa de cada célula.
- Utilice una configuración óptica idéntica (por ejemplo, la misma potencia láser) y muestras de imágenes el mismo día si desea comparar directamente diferentes imágenes.
- Las potencias exactas del láser y del detector dependerán de cada sistema específico. Comience usando potencias láser bajas y luego aumente lentamente las potencias hasta que la señal sea detectable sin saturarse.
- Configure el microscopio confocal para la obtención de imágenes de autofluorescencia.
- Analice PAF en qNSC en vivo y aNSC in vitro.
- Después de adquirir imágenes de autofluorescencia en qNSC o aNSC, cuantifique las imágenes abriendo un archivo en ImageJ25.
- Utilice la opción Procesar > filtros > desenfoque gaussiano (Sigma: 2) en la imagen de la autofluorescencia qNSC/aNSC para suavizar los píxeles irregulares.
- Utilice Imagen > Ajustar > Umbral para seleccionar los píxeles que representan PAF (eliminar los píxeles de fondo) y haga clic en Aplicar. Utilice los mismos parámetros de umbral para todas las imágenes que se compararán directamente.
- Utilice el proceso > la cuenca > binaria para separar los PAF en una proximidad espacial cercana.
- Usando el mouse de la computadora, dibuje una región de interés (ROI) alrededor de una célula, según lo determinado por la observación de una imagen de campo claro o mediante la observación de la autofluorescencia difusa presente de manera uniforme en todo el citoplasma de la célula.
NOTA: Por lo general, está bien excluir los procesos periféricos delgados e incluir el núcleo, ya que la autofluorescencia generalmente no está presente en estos lugares y, por lo tanto, no interrumpiría el análisis. - Utilice Analizar > analizar partículas (tamaño: 0,1-infinito μm; Circularidad: 0-1) con resumen marcado.
NOTA: ImageJ producirá una ventana de salida con datos sobre las partículas en la región de interés, lo que permite la tabulación del número de PAF en cada celda. Otras moléculas fluorescentes se pueden emparejar con la obtención de imágenes de PAF. Sin embargo, dado que la PAF es una señal relativamente tenue, es importante validar que otros fluoróforos no penetren en el canal de la PAF. Por lo general, los fluoróforos que absorben 600+ nm y emiten 650+ nm se pueden emparejar con imágenes PAF, como LipidSpot 610.
2. Uso de FACS para enriquecer el estado de activación de NSC en NSC cultivadas basadas en autofluorescencia
- Genere qNSC y aNSC como se describe en la sección 1.
- Ordene una mezcla de qNSC y aNSC en vivo en función de la autofluorescencia.
NOTA: A modo de ejemplo, este protocolo discutirá cómo enriquecer aNSC o qNSC en una muestra generada mezclando qNSC y aNSC en una proporción de 1:1.- Antes de la tripsinización y la clasificación celular, etiquete las células que progresan a través de la fase S agregando 10 μM de 5-etinil-2'-desoxiuridina (EdU; un análogo de timidina que se incorpora a las células que sintetizan ADN, como en la fase S del ciclo celular) al medio de cultivo celular durante 1 h.
- Tripsinizar las qNSC y las aNSC como se explica en los pasos 1.1.8, volver a suspender en 0,5 mL de tampón FACS (Tabla 1) y luego colocar en hielo.
NOTA: los qNSC se adhieren fuertemente al plato. A una confluencia relativamente mayor, las qNSC pueden disociarse mecánicamente de la placa. Sin embargo, si las qNSC se encuentran en una confluencia relativamente más baja o son difíciles de eliminar del plato mecánicamente mediante pipeteo, use tripsina para eliminarlas del plato.- Retire el medio de cultivo de las qNSC y agregue un 0,25% de tripsina suficiente para cubrir el fondo de la placa (para un pozo de cubeta de imágenes de 1cm2 , use ~ 150 μL), incubando durante 3 min a 37 °C.
- Después de la incubación, apague la reacción de tripsina agregando un medio correspondiente a cada tipo de célula igual al volumen de tripsina agregado. Disociar mecánicamente las células de la placa antes de la recolección y centrifugación.
- Usando una boquilla de ~130 μm, analice qNSC y aNSC usando un citómetro de flujo o FACS con condiciones ópticas basadas en las capacidades del instrumento y detecte PAF como se muestra en la Figura 1. Por ejemplo, use un láser de 405 nm para excitar la muestra y use un filtro de 580-620 nm para la detección.
- Recopile al menos 10.000 qNSC y aNSC singletes en un archivo de datos y, a continuación, utilícelos para diseñar puertas que recopilen un enriquecimiento de qNSC o aNSC. Por ejemplo, configure las puertas para recoger el 25 % superior o el 25 % inferior de las NSC en función de la intensidad de la autofluorescencia en la muestra mixta aNSC:qNSC (1:1).
- Después de configurar las compuertas para clasificar los singletes con autofluorescencia más brillante o más tenue como se describió anteriormente, clasifique las células en pocillos recubiertos de PLO, laminina llenos de medio aNSC (10.000 células/cm2, consulte el paso 1.1.3).
- Después de FACS, permita que las NSC permanezcan sentadas durante 3 horas en la incubadora. Fije las células tratándolas con un 4% de paraformaldehído suficiente para cubrir el fondo del plato (para un pozo de cubeta de imágenes de 1 cm2 , use ~ 150 μL) durante 15 min en RT.
NOTA: Para visualizar EdU en las células, lo más fácil es obtener un kit comercial para detectar EdU y seguir el protocolo recomendado por el fabricante. - En primer lugar, permeabilizar las células mediante tratamiento con tritón al 0,25% en PBS durante 15 min a RT.
- A continuación, trate las células con una solución de tinción de EdU en RT durante 30 min.
NOTA: La composición precisa de la solución de tinción de EdU variará según la fuente de los reactivos, pero aproximadamente consta de un tampón de reacción, un aditivo tampón de reacción, sulfato de cobre y una molécula modificada con alquino o azida. Consulte la Tabla de materiales para obtener un kit recomendado. - Después de la tinción para visualizar el EdU, lave las muestras 3 veces durante 10 min con PBS.
3. Uso de un microscopio multifotónico para detectar la autofluorescencia de los canales 1 y canales 2 y realizar FLIM en NSC in vitro para identificar las poblaciones y transiciones de estados celulares de NSC
NOTA: Cada microscopio tiene su software patentado; Por lo tanto, siga estos pasos realizando acciones análogas para configurar un microscopio específico. Este protocolo proporcionará instrucciones para trabajar en Prairie View.
- Genere qNSC y aNSC como se describe en la sección 1.
- Configure el microscopio multifotónico para obtener imágenes de fluorescencia durante toda su vida útil.
- Utilice una lente objetivo con un aumento de 40x o más (~1,15 NA).
- Utilice un microscopio multifotónico con los siguientes componentes, o equivalentes: un láser ultrarrápido sintonizable, un dicroico de paso largo de 720 nm, un tubo fotomultiplicador (PMT) de fosfuro de arsenio de galio (GaAsP), electrónica de conteo de fotón único correlacionada en el tiempo y un medidor de potencia analógico.
- Para obtener imágenes del canal 1, configure el láser sintonizable a 750 nm y utilice un cubo de filtro de emisión de 440/80 nm. Para obtener imágenes del canal 2, ajuste el láser a 890 nm y utilice un cubo de filtro de emisión de 550/100 nm.
NOTA: Apague las luces de la habitación antes de encender los PMT.
- Función de respuesta del instrumento
- Capture una función de respuesta del instrumento (IRF) mediante la obtención de imágenes de un cristal de urea triturado para tener en cuenta la respuesta temporal del instrumento en la desintegración medida.
NOTA: El IRF se convoluciona con el decaimiento para calcular con precisión la curva de ajuste del decaimiento. Para la adquisición, el láser se establece en 890 nm y se utiliza el cubo de filtro de emisión de 440/80 nm. El GaAsP PMT se establece en una ganancia de 800, y los pockels (potencia del láser) se establecen muy bajos inicialmente, generalmente 1. - Seleccione un campo de visión con urea cristalina y aumente ligeramente los pockels (hasta un máximo de 15).
- A continuación, adquiera una imagen utilizando los mismos parámetros que se utilizan para la obtención de imágenes de células: discriminador de fracción constante (CFD) 1 x 104 - 1 x 105, resolución de 256 x 256, tiempo de permanencia establecido en 4,8 μs, zoom óptico establecido en 1,5x y un tiempo de integración de 60 s.
- Capture una función de respuesta del instrumento (IRF) mediante la obtención de imágenes de un cristal de urea triturado para tener en cuenta la respuesta temporal del instrumento en la desintegración medida.
- Realice imágenes de fluorescencia de por vida e imágenes de intensidad para la autofluorescencia de canal 1 y canal 2 en qNSC y aNSC en vivo in vitro
- Encuentre un campo de visión para obtener imágenes: Utilice los oculares para encontrar un campo de visión adecuado y cambie la trayectoria de la luz a la muestra en el microscopio para permitir la obtención de imágenes.
NOTA: Primero se adquirirá una imagen del Canal 1, seguida de una imagen del Canal 2 del mismo campo de visión. - Configuración para la recolección de la imagen del canal 1: Haga clic en la pestaña Potencia/Ganancia y establezca la ganancia en el PMT en 800, haga clic en la pestaña Láser 2-P y seleccione 750 nm para el láser, y asegúrese de que se utilice el cubo de filtro adecuado (440/80 nm).
- Recopile la imagen del canal 1.
- Inicie el modo de vista previa yendo a la pestaña Encendido/Ganancia y configurando los Pockels en 30, luego vaya a la sección Escaneo de esa pantalla y haga clic en Escaneo en vivo.
- Aumente los Pockels para encontrar un buen campo de visión a baja potencia láser (por debajo de 3,6 mW). Una vez que se encuentra el campo de visión, ajuste los Pockels para que el medidor de potencia lea ~ 3.6 mW en la potencia después de la celda de Pokel, y el discriminador de fracción constante (CFD) en la pestaña Tasas de conteo mide 1 x 104 - 1 x 105.
- Vaya a la sección Escaneo y haga clic en Escaneo único una vez que se cumplan esos parámetros. Esto adquirirá una imagen de Canal 1 durante 60 s.
- Para capturar una imagen del Canal 2, haga clic en la pestaña Láser 2-P y seleccione 890 nm para el láser, cambie el cubo de filtro por el cubo de emisión del Canal 2 (550/100 nm).
- Inicie el modo de vista previa como se describe en 3.4.3, aumente los Pockels hasta que el medidor de potencia marque ~ 7 mW, y los recuentos de CFD sean una vez más 1 x 104 -1 x 105.
- Vaya a la sección Escaneo y haga clic en Escaneo único una vez que se cumplan esos parámetros. Esto adquirirá una imagen del Canal 2 durante 60 s.
- Adquirir más imágenes: después de adquirir una imagen de canal 1 y canal 2, busque un nuevo campo de visión y repita los pasos 3.4.2 a 3.4.4. Por lo general, la recopilación de 4-6 campos de visión para obtener una imagen de ~ 50-100 celdas es suficiente para una condición en un experimento.
NOTA: mCherry se puede utilizar como una etiqueta fluorescente adicional sin sangrado en los canales 1 y canal 2.
- Encuentre un campo de visión para obtener imágenes: Utilice los oculares para encontrar un campo de visión adecuado y cambie la trayectoria de la luz a la muestra en el microscopio para permitir la obtención de imágenes.
- Analice las imágenes fluorescentes de la vida útil.
- Genere matrices de decaimiento para imágenes de vida útil de fluorescencia en SPCImage siguiendo los pasos 3.5.2-3.1.14.
NOTA: Para obtener información adicional, consulte los manuales actualizados disponibles gratuitamente en línea26. - Abra SPCImage y abra la imagen IRF FLIM recopilada en la sección 3.3.
- Ajuste las barras negras verticales en el histograma para que se limiten a la curva de decaimiento IRF. En la barra de menú superior, haga clic en IRF > Copiar al portapapeles.
- Abra un fichero de imagen FLIM dentro del conjunto de datos a analizar, obtenido en la sección 3.4.
- En la barra de menú superior, haga clic en IRF > Pegar desde el portapapeles.
- En la parte inferior derecha del software, establezca Componentes en 2.
- En la barra de menú superior del software, haga clic en Opciones > modelo. A continuación, seleccione MLE para Método de ajuste, Agrupación espacial a cuadrado, Umbral a pico y, en Configuración, seleccione Decaimiento multiexponencial.
- En la parte inferior izquierda del software, aumente Bin hasta que el recuento de fotones sea de al menos 100 en píxeles citoplasmáticos representativos en el pico del recuento de fotones inmediatamente después de la excitación.
- En la parte inferior izquierda del software, establezca el umbral. Para el canal 1, utilice un umbral de "50". Para el canal 2, use un umbral de "0".
- En la barra de menú superior del software (este protocolo describe cómo operar la versión 8.3), haga clic en Calcular > matriz de descomposición.
NOTA: Asegúrese de que los valores de Chi2 sean ~0.8-1.2 en toda la imagen. Esto confirma que los datos serán robustos mediante la validación de modelos de decaimiento biexponencial. - En la barra de menú superior, haga clic en Archivo > Guardar.
- En la barra de menú superior del software, haga clic en Exportar > de archivos y exporte lo siguiente:
Valor codificado por colores, T1, T2, Chi2, intensidades de píxeles, A1[%], A2[%] e imagen codificada por colores (con leyenda)
NOTA: Ahora SPCImage está configurado para realizar un proceso por lotes, analizando todas las imágenes de un canal determinado (analice las imágenes FLIM del canal 1 juntas y luego repita comenzando en el paso 3.5.2 para las imágenes FLIM del canal 2). - Para realizar un proceso por lotes, haga clic en Calcular > procesamiento por lotes y seleccione las imágenes FLIM que se van a procesar.
- En la barra de menú superior, haga clic en Exportar > de archivos > Exportación por lotes y seleccione las matrices de decaimiento que desea exportar (generadas en el paso anterior).
- Utilice CellProfiler para crear máscaras citoplasmáticas siguiendo los pasos 3.5.16-3.5.24.
NOTA: Para hacer esto, los núcleos se colocarán manualmente alrededor de las imágenes de intensidad del Canal 1, donde el núcleo es negro y claramente visible. A continuación, una canalización de CellProfiler manual_segmentation.cpproj27 (Archivo complementario 1) producirá automáticamente una célula completa y una máscara citoplasmática, según las indicaciones de la máscara nuclear. Alternativamente, los núcleos se pueden marcar con mCherry y obtener imágenes junto con la autofluorescencia del canal 1 y el canal 2 para proporcionar la capacidad de automatizar la identificación de núcleos. Sin embargo, muchos tintes nucleares convencionales sangrarán espectralmente en los canales de autofluorescencia y, por lo tanto, son incompatibles con esta técnica. - En R Studio, use R_ASCtoTIFF.rmd27 (archivo complementario 2) para convertir los archivos X_photons.asc , donde X es el nombre de la imagen adquirida de la imagen del canal 1 establecida en archivos TIF.
- Abra Cell Profiler y abra manual_segmentation.cpproj.
- Cargue en el canal 1 los TIF de fotones creados en el paso 3.5.16 en el paso Imágenes en la parte superior de la canalización.
- En los 3 pasos inferiores de la canalización titulada SaveImages, establezca una ruta de acceso de guardado para las máscaras que generará CellProfiler.
- Haga clic en Iniciar modo de prueba en la parte inferior izquierda de CellProfiler.
- Haga clic en Ejecutar en la parte inferior izquierda de CellProfiler. Cuando CellProfiler llegue al paso IdentifyObjectsMmanualmente , aparecerá una ventana que muestra la imagen actual del Canal 1 que se está procesando.
- Haga clic en la tecla F del teclado y, a continuación, dibuje manualmente un trazo de los núcleos de una célula mientras mantiene pulsado el botón izquierdo del ratón. Después de trazar los núcleos, suelte el botón izquierdo del ratón. Repita este paso para todas las celdas de la imagen.
- Haga clic en Listo en la parte inferior derecha de la ventana emergente con la imagen de intensidad. El software ahora terminará la tubería y exportará máscaras citoplasmáticas, nucleares y de células totales.
- En la parte inferior izquierda, haga clic en Siguiente conjunto de imágenes y repita los pasos 3.5.21-3.5.23 para todas las imágenes TIF del canal 1.
- Utilice el script R (Integrate decay matrices and cytoplasmic masks.rmd) para integrar las matrices de desintegración generadas en los pasos 3.5.2-3.5.14 y las máscaras citoplasmáticas generadas en los pasos 3.5.15-3.5.24 para obtener variables de imágenes de fluorescencia promedio para el citoplasma de cada célula siguiendo los pasos 3.5.26-3.5.30
- Usando una hoja de cálculo (por ejemplo, Microsoft Excel), cree un documento .csv titulado test_key27 [Archivo complementario 3] para un ejemplo) con 3 columnas.
- Asigne un título a las columnas como Carpeta, NADH y FAD. Rellene la tabla para enumerar la ubicación de la carpeta en la carpeta y, a continuación, el prefijo del título de la imagen en NADH y FAD, respectivamente, para vincular cada imagen del canal 1 con cada imagen del canal 2 (consulte los datos de ejemplo: tabla 2).
- Genere matrices de decaimiento para imágenes de vida útil de fluorescencia en SPCImage siguiendo los pasos 3.5.2-3.1.14.
- En R Studio, abra Integrar matrices de desintegración y máscaras citoplasmáticas.rmd27(Archivo complementario 4).
- Establezca las siguientes rutas de archivo como anotadas en el script: Establecer directorio de trabajo, ubicación de archivo de canal 1, ubicación de archivo FAD, ubicación de archivo de máscara y ubicación y nombre de archivo de salida.
- Ejecute todo el script. Una vez completado con éxito el script, se generará un archivo de salida, incluidos los metadatos y las variables FLIM promedio.
- Realice el análisis utilizando la autofluorescencia FLIM data.rmd27 (Archivo complementario 5) o utilizando cualquier otro software de análisis.
Resultados
Imágenes de autofluorescencia confocal para separar el estado de la célula NSC (Figura 1)
Con el fin de utilizar la microscopía confocal para resolver el estado de activación de las NSC, se generaron qNSCs y aNSCs in vitro utilizando un medio de activación o un medio de quiescencia, como se describió anteriormente 10,13,17,18. Para detectar PAF en NSCs, se tomaron imágenes de qNSCs y aNSCs vivas utilizando la misma exposición en un microscopio confocal (Ex: 405 nm, Em: 580-620 nm). Las qNSC exhibieron un mayor número de PAF en comparación con las aNSC (Figura 1A,B). Este hallazgo ilustra cómo las propiedades de autofluorescencia se pueden utilizar como marcadores para identificar el estado celular de las qNSC y las aNSC.
Enriquecimiento FACS del estado de la célula NSC mediante autofluorescencia (Figura 2)
Para enriquecer el estado del ciclo celular utilizando FACS, se generaron qNSCs y aNSCs in vitro 10,13,17,18,19 como se describe en este protocolo y se pre-marcaron con EdU durante 1 h antes de la tripsinización y el análisis en el citómetro de flujo para marcar las células que progresan a través de la fase S. A continuación, las qNSC y las aNSC se analizaron mediante citometría de flujo, ya sea por separado o mezcladas en una proporción de 1 qNSC:1 aNSC (Figura 2). Se dibujaron puertas para enriquecer para las aNSC o qNSC de la población mixta, y luego se clasificaron las celdas en función de estas puertas. Después de la FACS, las células de cada muestra se colocaron en vidrio recubierto de PLO y laminina y se dejaron adherir a la placa durante 3 horas antes de fijarlas, teñirlas y analizarlas en busca de células %EdU+. Como era de esperar, las células clasificadas a partir de la puerta de alta autofluorescencia eran menos EdU+ que la muestra Mix, y las muestras clasificadas a través de la puerta de baja autofluorescencia eran más proliferativas que la muestra Mix (Figura 2C). Este hallazgo confirma la capacidad de enriquecer para el estado de activación de NSC a partir de una mezcla heterogénea de qNSCs y aNSCs utilizando FACS.
Imágenes de fluorescencia multifotónica de por vida para clasificar el estado de la célula NSC (Figura 3)
Las qNSCs y aNSCs se generaron in vitro y luego se obtuvieron imágenes utilizando un microscopio multifotónico para realizar FLIM en la autofluorescencia del Canal 1 (Ex: 750 nm (2P), Em: 360-520 nm) y el Canal 2 (Ex: 890 nm (2P), Em: 450-650 nm) (Figura 3A, Tabla 2). Las qNSC y las aNSC exhibieron perfiles autofluorescentes que fueron significativamente diferentes. Por ejemplo, las qNSC tenían una vida media de fluorescencia del canal 1 τm más alta, pero una α1 más baja en comparación con las aNSC. Para evaluar la capacidad de los datos de autofluorescencia de NSC FLIM para predecir el estado de activación de NSC, se generó un modelo de regresión logística con intensidad de Canal 1, α1, τ1, τ2 e intensidad de Canal 2, α1, τ1, τ2. Una curva del operador del receptor ilustra que estos datos son suficientes para crear un modelo casi perfecto (área bajo la curva = 0,963), que predice con precisión el estado de activación del NSC. En conjunto, estos datos ilustran la capacidad de FLIM y la autofluorescencia para clasificar el estado de la célula NSC.
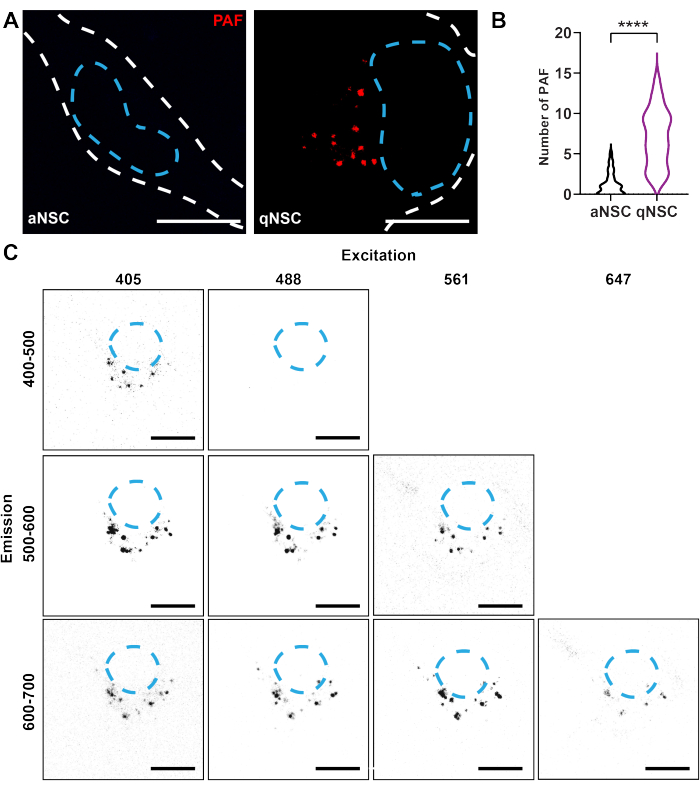
Figura 1: Las imágenes confocales resuelven los biomarcadores autofluorescentes del estado de activación de NSC. Las qNSC (púrpura) y las aNSC (negro) (A-B) se obtuvieron utilizando la misma exposición en un microscopio confocal (rojo; Ej. 405 nm, Em 580-620 nm) y se analizó el número de PAF (N = 3, prueba de Mann-Whitney, media ± DE). Las líneas discontinuas blancas denotan el borde de la celda y las líneas discontinuas azules denotan los núcleos. (C) Se obtuvieron imágenes de la autofluorescencia en el mismo qNSC utilizando varias condiciones de excitación y emisión, como se indica en la figura, con idéntica potencia y ganancia láser. Barras de escala: 10 μm. **** p < 0,0001. Haga clic aquí para ver una versión más grande de esta figura.
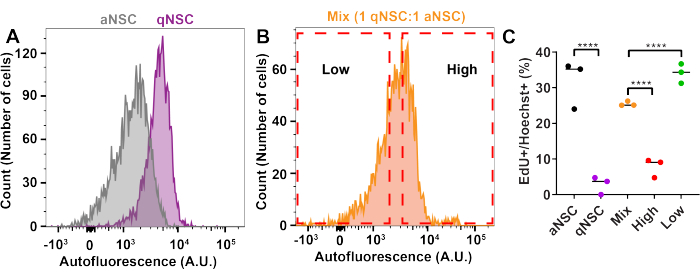
Figura 2: FACS puede enriquecerse para el estado de activación de NSC. (A-C) qNSCs, aNSCs o qNSCs: la mezcla de aNSCs (1:1) se trató con EdU durante 1 h, tripsinizada, y luego se analizó por citometría de flujo (Ex: 405 nm, Em: 580-620 nm). A continuación, las células de mezcla (1:1) se clasificaron por FACS para las células que tenían una autofluorescencia baja o alta, se platearon y se analizaron para la proliferación midiendo el porcentaje de células que eran EdU+. Las unidades arbitrarias se abrevian como "A.U." (N = 4, ANOVA de dos factores con prueba de Tukey post hoc, media ± DE). p < 0,0001. Haga clic aquí para ver una versión más grande de esta figura.
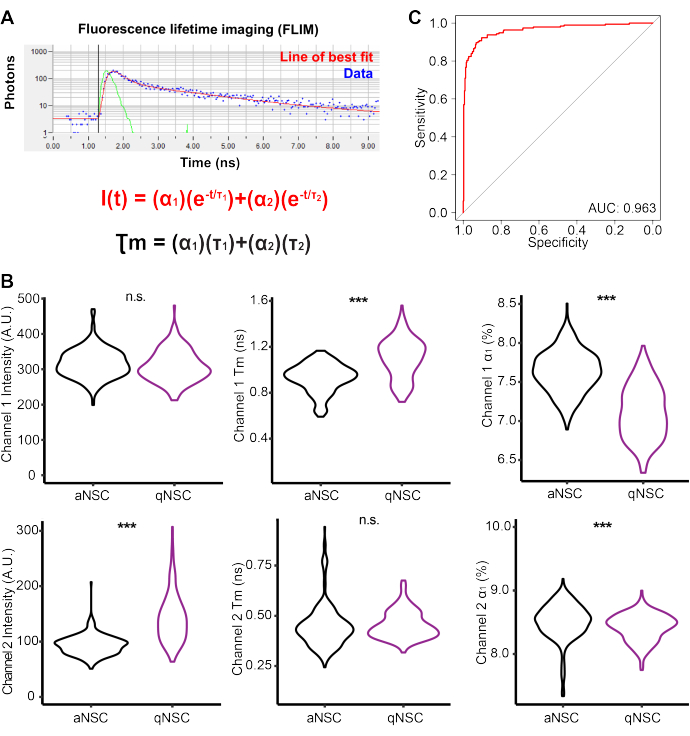
Figura 3: Las imágenes de fluorescencia multifotónica de por vida revelan biomarcadores del estado de activación de NSC. (A) Esquema que representa el análisis de ajuste de curva de los datos FLIM adquiridos. (B) Mediciones FLIM de canal 1 y canal 2, incluidos los valores de intensidad, vida media (Tm) y contribución fraccional (α1) para datos qNSC (púrpura) y aNSC (negro) (n = 501 celdas, regresión logística bilateral, modelo lineal generalizado). (C) Curva del operador del receptor que muestra un modelo de regresión logística generado utilizando la intensidad del canal 1, la intensidad del canal1, α 1, τ 1, τ2 y el canal 2, α1, τ1, τ2 para clasificar las NSC como aNSC o qNSC. P < 0,001. Haga clic aquí para ver una versión más grande de esta figura.
Tabla 1: Medios y soluciones. Recetas para todas las soluciones utilizadas en este protocolo. Haga clic aquí para descargar esta tabla.
Tabla 2: Datos de ejemplo. Datos representativos de qNSC y aNSC FLIM para la autofluorescencia de canal 1 y canal 2. Haga clic aquí para descargar esta tabla.
Archivo complementario 1: manual_segmentation.cpproj Haga clic aquí para descargar este archivo.
Archivo complementario 2: R_ASCtoTIFF.rmd Haga clic aquí para descargar este archivo.
Archivo complementario 3: test_key Haga clic aquí para descargar este archivo.
Archivo Suplementario 4: Integración de matrices de desintegración y máscaras citoplasmáticas.rmd Haga clic aquí para descargar este archivo.
Archivo suplementario 5: autofluorescencia FLIM data.rmd Haga clic aquí para descargar este archivo.
Discusión
Este protocolo describe una técnica de resolución de célula única, sin marcaje, no destructiva y de célula viva, que permite la clasificación del estado de la célula NSC in vitro a través de imágenes de señales autofluorescentes en NSC. Este enfoque detecta los cambios metabólicos que se producen durante la salida de la quiescencia de NSC, que influyen en las propiedades ópticas de los cofactores metabólicos, como el NAD(P)H, y ofrece muchas ventajas sobre las tecnologías existentes para estudiar la quiescencia de NSC. Por ejemplo, muchas técnicas convencionales para estudiar las qNSCs y las aNSCs, como el marcaje con marcadores del ciclo celular como el EdU, requieren la fijación de muestras. Los métodos que existen actualmente que son capaces de estudiar las NSC vivas se ven aún más limitados al requerir la introducción de marcadores exógenos, normalmente mediante la generación de transgenes fluorescentes que codifican proteínas. Estas herramientas son limitadas tanto en los recursos necesarios para generarlas como en muchas advertencias técnicas. Por ejemplo, las proteínas de filamento intermedio nestina y proteína ácida fibrilar glial (GFAP) se utilizan comúnmente como marcadores de qNSCs y aNSCs 12,13,18,28,29. Sin embargo, se sabe que la nestina y el GFAP se expresan diferencialmente entre qNSCs y aNSCs 12,13,18,28,29. Además, las imágenes de autofluorescencia proporcionan datos de alta resolución de una sola célula, que pueden desentrañar la heterogeneidad de una sola célula que se pierde o es complicada de interpretar en experimentos que culminan en un análisis masivo de una población de células.
Sin embargo, las imágenes de autofluorescencia también tienen varias limitaciones. Muchas de las señales autofluorescentes se pierden durante la fijación celular. Por lo tanto, las imágenes de autofluorescencia se limitan en gran medida al estudio de células vivas. Las imágenes de autofluorescencia también se basan en la falta de fluoróforos introducidos exógenamente, que pueden sangrar en los canales autofluorescentes. Aunque muchos fluoróforos pueden ser compatibles con las imágenes de autofluorescencia en situaciones específicas, no siempre es posible emparejar las imágenes de autofluorescencia con muchas herramientas existentes. Las imágenes de células vivas también pueden inducir fototoxicidad. Una iteración de imágenes utilizando este protocolo no induce suficiente fototoxicidad para reducir la viabilidad de NSC. Sin embargo, la repetición de imágenes de células en un curso de tiempo a intervalos más frecuentes muchas veces al día puede generar una fototoxicidad significativa y, por lo tanto, no es una estrategia viable para estudiar la quiescencia de NSC. Por último, aunque las imágenes de autofluorescencia se pueden utilizar para estudiar las NSC de ratones machos de 6 semanas de edad, ya sea del hipocampo o de los ventrículos laterales, no está claro hasta qué punto se puede utilizar esta técnica para estudiar las NSC de otras fuentes, edades u otros tipos de células madre.
El protocolo aquí descrito también supone la producción precisa de qNSCs y aNSCs in vitro utilizando protocolos previamente establecidos 10,13,17,18,19. Si reproducir los resultados de este protocolo es un desafío, asegúrese de que las qNSC y las aNSC se generen correctamente sondeando la presencia o ausencia de varios marcadores de qNSC y aNSC, como se describió anteriormente 10,13,17,18,19. En conjunto, las imágenes de autofluorescencia proporcionan un enfoque técnico novedoso para identificar qNSC y aNSC y estudiar las salidas de inactividad de NSC.
Divulgaciones
Los autores declaran no tener intereses contrapuestos.
Agradecimientos
Agradecemos al núcleo de citometría de flujo de la UW-Madison (P30 CA014520 y 1S10RR025483-01) y a los miembros del laboratorio de Moore y de la comunidad de la UW-Madison por sus aportes. Agradecemos a nuestras fuentes de financiamiento: NIH T32 T32GM008688 (a C.S.M.), Diana Jacobs Kalman Fellowship de AFAR (a C.S.M.), Wisconsin Graduate Fellowship (a C.S.M.), DP2 NIH New Innovator Award (1DP2OD025783, a D.L.M.), Vallee Scholar Award (a D.L.M.), NIH 1R56NS130450 (a D.L.M y M.C.S.), R01 CA185747 (a M.C.S.), R01 CA205101 (a M.C.S.), R01 CA211082 (a M.C.S.) y la Subvención No. CBET-1642287 (a M.C.S.).
Materiales
| Name | Company | Catalog Number | Comments |
| 40x Water objective lens | Nikon | MRD77410 | Objective lens used in multiphoton microscope in Part 3 |
| 8 well cuvette | Ibidi | 80826-90 | For imaging aNSCs/qNSCs |
| Analog power meter | Thorlabs | PM100A | Used in multiphoton microscope in Part 3 |
| Antibiotic-Antimycotic (100X) (PSF) | Thermo Fisher | 15240062 | Antibiotic for NSC media |
| B-27 | Invitrogen | 17504044 | Nutrient supplement for NSC media |
| BMP4 | Fisher Scientific | 5020BP010 | Factor for inducing quiescence |
| Bovine serum albumin | Sigma | A4919-25G | For making BMP4 |
| Chameleon ultrafast laser | Coherent | N/A | Laser used in multiphoton microscope in Part 3 |
| Confocal microscope | Nikon | C2 | Microscope used for Part 1 |
| DMEM/F-12 (without GlutaMAX) | Invitrogen | 11320033 | Base media for NSCs |
| DNAse | Sigma | D5025-15KU | Added to trypsin inhibitor |
| EdU assay kit | Invitrogen | C10337 | Proliferation assay for cell culture |
| EGF | PeproTech | AF-100-15-500UG | Growth factor for NSC media |
| FGF | PeproTech | 100-18B | Growth factor for NSC media |
| Fluorescent activated cell sorter | BD | FACSAria | Fluorescent Activated Cell Sorter used for Part 2 |
| Heparin | Sigma | H3149-50KU | Additive for NSC media |
| L-15 | Invitrogen | 21083027 | For preparing trypsin inhibitor solution |
| Laminin | Sigma | L2020-1MG | For coating glassware |
| Nikon TiE inverted microscope | Nikon | N/A | Microscope frame Used for Part 3 |
| PLO | Sigma | P3655-100MG | For coating glassware |
| SPC-150 Single photon counting electronics | Becker and Hickl | N/A | Used in multiphoton microscope in Part 3 |
| Trypsin (for trypsinizing pellets of aNSCs that were growing as spheres or monolayers) | Gibco | 15090046 | For trypsinizing neurospheres or adherent aNSCs |
| Trypsin (for trypsinizing qNSCs) | Gibco | 25200072 | For trypsinizing adherent qNSCs |
| Trypsin inhibitor | Sigma | T6522-100MG | For inhibiting trypsinization of aNSCs |
| Urea crystals | Sigma | U5128-5G | Used to collect an IRF |
| Versene | Thermo Fisher | 15040066 | For preparing trypsin |
Referencias
- Goncalves, J. T., Schafer, S. T., Gage, F. H. Adult neurogenesis in the hippocampus: From stem cells to behavior. Cell. 167 (4), 897-914 (2016).
- Kuhn, H. G., Dickinson-Anson, H., Gage, F. H. Neurogenesis in the dentate gyrus of the adult rat: age-related decrease of neuronal progenitor proliferation. J Neurosci. 16 (6), 2027-2033 (1996).
- Silva-Vargas, V., Maldonado-Soto, A. R., Mizrak, D., Codega, P., Doetsch, F. Age-dependent niche signals from the choroid plexus regulate adult neural stem cells. Cell Stem Cell. 19 (5), 643-652 (2016).
- Urban, N., Blomfield, I. M., Guillemot, F. Quiescence of adult mammalian neural stem cells: A highly regulated rest. Neuron. 104 (5), 834-848 (2019).
- Urban, N., et al. Return to quiescence of mouse neural stem cells by degradation of a proactivation protein. Science. 353 (6296), 292-295 (2016).
- Kalamakis, G., et al. Quiescence modulates stem cell maintenance and regenerative capacity in the aging brain. Cell. 176 (6), 1407-1419 (2019).
- Walsh, A. J., et al. Classification of T-cell activation via autofluorescence lifetime imaging. Nat Biomed Eng. 5 (1), 77-88 (2021).
- Sagar, M. A. K., et al. Microglia activation visualization via fluorescence lifetime imaging microscopy of intrinsically fluorescent metabolic cofactors. Neurophotonics. 7 (3), 035003 (2020).
- Knobloch, M., et al. Metabolic control of adult neural stem cell activity by Fasn-dependent lipogenesis. Nature. 493 (7431), 226-230 (2013).
- Knobloch, M., et al. A fatty acid oxidation-dependent metabolic shift regulates adult neural stem cell activity. Cell Rep. 20 (9), 2144-2155 (2017).
- Knobloch, M., et al. SPOT14-positive neural stem/progenitor cells in the hippocampus respond dynamically to neurogenic regulators. Stem Cell Reports. 3 (5), 735-742 (2014).
- Shin, J., et al. Single-cell RNA-Seq with waterfall reveals molecular cascades underlying adult neurogenesis. Cell Stem Cell. 17 (3), 360-372 (2015).
- Leeman, D. S., et al. Lysosome activation clears aggregates and enhances quiescent neural stem cell activation during aging. Science. 359 (6381), 1277-1283 (2018).
- Beckervordersandforth, R. Mitochondrial metabolism-mediated regulation of adult neurogenesis. Brain Plast. 3 (1), 73-87 (2017).
- Morrow, C. S., et al. Autofluorescence is a biomarker of neural stem cell activation state. Cell Stem Cell. , (2024).
- Kolenc, O. I., Quinn, K. P. Evaluating cell metabolism through autofluorescence imaging of NAD(P)H and FAD. Antioxid Redox Signal. 30 (6), 875-889 (2019).
- Mira, H., et al. Signaling through BMPR-IA regulates quiescence and long-term activity of neural stem cells in the adult hippocampus. Cell Stem Cell. 7 (1), 78-89 (2010).
- Morrow, C. S., et al. Vimentin coordinates protein turnover at the aggresome during neural stem cell quiescence exit. Cell Stem Cell. 26 (4), 558-568 (2020).
- Martynoga, B., et al. Epigenomic enhancer annotation reveals a key role for NFIX in neural stem cell quiescence. Genes Dev. 27 (16), 1769-1786 (2013).
- Bin Imtiaz, M. K., Jessberger, S. Isolation of adult mouse hippocampal neural stem cells for fluorescence loss in photobleaching assays. STAR Protoc. 2 (3), 100695 (2021).
- Guo, W., Patzlaff, N. E., Jobe, E. M., Zhao, X. Isolation of multipotent neural stem or progenitor cells from both the dentate gyrus and subventricular zone of a single adult mouse. Nat Protoc. 7 (11), 2005-2012 (2012).
- Lakowicz, J. R., Szmacinski, H., Nowaczyk, K., Johnson, M. L. Fluorescence lifetime imaging of free and protein-bound NADH. Proc Natl Acad Sci U S A. 89 (4), 1271-1275 (1992).
- Blacker, T. S., Duchen, M. R. Investigating mitochondrial redox state using NADH and NADPH autofluorescence. Free Radic Biol Med. 100, 53-65 (2016).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: fundamentals and advances in instrumentation, analysis, and applications. J Biomed Opt. 25 (7), 1-43 (2020).
- . ImageJ Available from: https://imagej.nih.gov/ij/ (2021)
- . Becker & Hichl Handbooks Available from: https://imagej.nih.gov/ij/ (2024)
- . GitHub Materials Available from: https://github.com/chrismorrow5/Neural-Stem-Cell-Autofluorescence-Analysis/ (2024)
- Codega, P., et al. Prospective identification and purification of quiescent adult neural stem cells from their in vivo niche. Neuron. 82 (3), 545-559 (2014).
- Llorens-Bobadilla, E., et al. Single-cell transcriptomics reveals a population of dormant neural stem cells that become activated upon brain injury. Cell Stem Cell. 17 (3), 329-340 (2015).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados