Method Article
Klassifizierung des Aktivierungszustands neuraler Stammzellen in vitro mittels Autofluoreszenz
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt Strategien zur Identifizierung und Anreicherung des Zellzustands in primären neuralen Stammzellkulturen adulter Mäuse durch Autofluoreszenzbildgebung unter Verwendung i) eines konfokalen Mikroskops, ii) eines fluoreszenzaktivierten Zellsortierers zur Durchführung einer Intensitätsbildgebung oder iii) eines Multiphotonenmikroskops zur Durchführung einer Fluoreszenzlebensdauerbildgebung.
Zusammenfassung
Neuronale Stammzellen (NSCs) teilen sich und produzieren neugeborene Neuronen im erwachsenen Gehirn durch einen Prozess, der als adulte Neurogenese bezeichnet wird. Adulte NSCs sind in erster Linie ruhig, ein reversibler Zellzustand, in dem sie den Zellzyklus (G0) verlassen haben, aber weiterhin auf die Umgebung reagieren. Im ersten Schritt der adulten Neurogenese empfangen ruhende NSCs (qNSCs) ein Signal und aktivieren sich, verlassen die Ruhephase und treten wieder in den Zellzyklus ein. Daher ist das Verständnis der Regulatoren der NSC-Ruhephase und des Ruheaustritts entscheidend für zukünftige Strategien, die auf die adulte Neurogenese abzielen. Unser Verständnis der NSC-Stille ist jedoch durch technische Einschränkungen bei der Identifizierung von ruhenden NSCs (qNSCs) und aktivierten NSCs (aNSCs) begrenzt. Dieses Protokoll beschreibt einen neuen Ansatz zur Identifizierung und Anreicherung von qNSCs und aNSCs, die in in vitro-Kulturen durch Bildgebung der NSC-Autofluoreszenz erzeugt wurden. Zunächst beschreibt dieses Protokoll, wie ein konfokales Mikroskop verwendet wird, um autofluoreszierende Marker von qNSCs und aNSCs zu identifizieren, um den NSC-Aktivierungszustand anhand der Autofluoreszenzintensität zu klassifizieren. Zweitens beschreibt dieses Protokoll, wie ein fluoreszierender aktivierter Zellsortierer (FACS) verwendet wird, um den NSC-Aktivierungszustand zu klassifizieren und Proben für qNSCs oder aNSCs mit Hilfe der Autofluoreszenzintensität anzureichern. Drittens beschreibt dieses Protokoll, wie ein Multiphotonenmikroskop verwendet wird, um eine Fluoreszenzlebensdauerbildgebung (FLIM) mit Einzelzellauflösung durchzuführen, den NSC-Aktivierungszustand zu klassifizieren und die Dynamik des Ruheaustritts sowohl anhand von Autofluoreszenzintensitäten als auch von Fluoreszenzlebensdauern zu verfolgen. Somit bietet dieses Protokoll ein Toolkit zur Lebendzell-, Markierungsfreiheit und Einzelzellauflösung für die Untersuchung der Stille und des Ruheaustritts von NSC.
Einleitung
NSCs bilden in vielen Organismen lebenslang neugeborene Neuronen in einem Prozess, der als adulte Neurogenese bezeichnet wird 1,2. Um neugeborene Neuronen zu produzieren, muss zunächst ein qNSC aktiviert werden, der in den Zellzyklus eintritt, um die Population zu vergrößern und neuronale Vorläuferzellenzu produzieren 3,4,5,6. Obwohl viel über die Stilllegung von NSCs bekannt ist, wird unsere Fähigkeit, die Treiber und Regulatoren der Stilllegung von NSCs vollständig zu identifizieren, durch technische Einschränkungen eingeschränkt, die bei der Isolierung und Identifizierung von qNSCs und ihrem Übergang zur Aktivierung bestehen. Die Autofluoreszenzbildgebung hat sich in der Vergangenheit als erfolgreich erwiesen, um Veränderungen des Zellzustands in vielen verschiedenen Zelltypen, wie Mikroglia und T-Zellen, zu untersuchen, indem sie metabolische Umbaumaßnahmen auflöste, die die optischen Eigenschaften von autofluoreszierenden metabolischen Cofaktoren wie Nicotinamidadenindinukleotidphosphat (NAD(P)H) und Flavinadenindinukleotid (FAD) beeinflussen7,8. NSCs bauen ihre metabolischen Netzwerke erheblich um, wenn sie den Ruheausgang 9,10,11,12,13,14 durchlaufen. Um diese Unterschiede zu nutzen, wurde die NSC-Autofluoreszenz kürzlich verwendet, um den NSC-Aktivierungszustand zu identifizieren und anzureichern, indem Verschiebungen in der Autofluoreszenz nachgewiesen wurden, die auf den metabolischen Umbau zurückzuführen sind, der auftritt, wenn NSCs die Ruhephaseverlassen 15. Die bildgebende Autofluoreszenz bietet mehrere technische Vorteile: i) Sie erfordert keine Zugabe von exogenen Markierungen, die das Zellverhalten beeinflussen können; ii) es kann hochauflösende Einzelzelldaten über den Aktivierungszustand von NSC liefern; und iii) es erfordert nicht die Zerstörung der Zelle 7,16. Dieses Protokoll skizziert drei Strategien zur Nutzung der NSC-Autofluoreszenz zur Untersuchung von NSC-Ruhezuständen und aktivierten Zellzuständen15.
Kürzlich wurde festgestellt, dass NSCs, die von 6 Wochen alten männlichen Mäusen aus der subgranularen Zone des Hippocampus isoliert und in vitro kultiviert und reversibel in den Ruhezustand versetzt wurden, 10,13,17,18,19,20,21 ein erhöhtes Maß an punktuierter Autofluoreszenz (PAF) aufwiesen, das zwischen 400 und 600 nm anregt und zwischen 500 und 700 nm emittiert. Dieses Signal war spezifisch für qNSCs im Vergleich zu aktivierten, zyklischen NSCs15. Die Möglichkeit, diese beiden Populationen visuell zu trennen, ohne zusätzliche Antikörpermarker oder Reporter zu verwenden, ist nützlich für viele experimentelle Fragen zur Natur von qNSCs und Quiescence-Ausgängen. Daher beschreibt dieses Protokoll zunächst Strategien zur Abbildung der PAF in qNSCs mit Hilfe eines konfokalen Mikroskops, das zur Identifizierung des NSC-Aktivierungszustands verwendet werden kann. Zweitens beschreibt dieses Protokoll Strategien zum Nachweis der PAF mittels fluoreszenzaktivierter Zellsortierung (FACS) und beschreibt weiterhin, wie auf der Grundlage dieses Signals sortiert werden kann, um qNSCs oder aNSCs anzureichern. Diese Strategien stellen eine Maßnahme dar, die verwendet werden kann, um NSCs basierend auf dem Zellstatus zu gruppieren und zu trennen.
Um eine höher aufgelöste Methode zur Trennung von NSCs nicht nur in unterschiedlichen Zuständen, sondern auch beim Übergang durch den Ruheaustritt zur vollständigen Aktivierung zu entwickeln, wurde mit einem Multiphotonenmikroskop eine Fluoreszenzlebensdauer-Bildgebung (FLIM) durchgeführt, um die Lebensdauer von NAD(P)H (genannt Kanal 1) und die grüne Autofluoreszenz (genannt Kanal 2; der sowohl FAD-Autofluoreszenz als auch PAF in qNSCs nachweist) Lebensdauern zusammen mit ihrer Intensität abzubilden. Dieser Ansatz macht sich die Tatsache zunutze, dass die optischen Eigenschaften von Molekülen in der Zelle von ihren physikalischen Eigenschaften abhängen16,22. Zum Beispiel ist NAD(P) (NAD und NADP sind optisch nicht unterscheidbar, und daher wird NAD(P) verwendet, um sich auf beide Spezies zu beziehen) im oxidierten Zustand nicht autofluoreszierend, sondern autofluoreszierend in seinem reduzierten Zustand (NAD(P)H)23. Darüber hinaus können zusätzliche physikalische Eigenschaften von autofluoreszierenden Molekülen, wie z. B. ihr Bindungsstatus an Enzyme, durch Durchführung einer Fluoreszenzlebensdauer-Bildgebung extrapoliert werden 7,22,24. Zum Beispiel hat NAD(P)H eine kürzere Fluoreszenzlebensdauer, wenn es nicht an ein Enzym22 gebunden ist. Da autofluoreszierende Moleküle wie NAD(P)H, das an Hunderten von Stoffwechselreaktionen beteiligt ist, von Zellen, die unterschiedliche Zustände oder Zellverhaltensweisen durchlaufen, unterschiedlich verwendet werden, können diese Verschiebungen mit einem Multiphotonenmikroskop nachgewiesen und quantifiziert werden, das die Autofluoreszenzlebensdauer detektiert23. Zusammen mit der Häufigkeit oder Intensität der Autofluoreszenz liefern diese Messungen mehrdimensionale Informationen, um NSCs in den einen oder anderen Zellzustand und durch die dynamischen Übergänge zwischen den Zuständen zu unterteilen. Drittens beschreibt dieses Protokoll die Durchführung, Analyse und Interpretation von FLIM- und Intensitätsmessungen von Signalen für Kanal 1 (NAD(P)H) und Kanal 2 (PAF) unter Verwendung eines Multiphotonenmikroskops. Zusammenfassend lässt sich sagen, dass dieses Protokoll ein lebenszellfreies, markierungsfreies Toolkit zur Untersuchung der NSC-Stille beschreibt, das hochauflösende Einzelzelldaten über den NSC-Zustand liefert.
Protokoll
Alle Verfahren in diesem Protokoll sind vom Institutional Animal Care and Use Committee (IACUC) an der University of Wisconsin-Madison genehmigt.
1. Verwendung eines konfokalen Mikroskops zur Abbildung von PAF in qNSCs und aNSCs zur Identifizierung des NSC-Zellzustands
- Generieren Sie qNSCs und aNSCs in vitro aus primären NSC-Kulturen.
- Kultur-NSCs, die aus adulten Mausgehirnen in einem proliferativen Medium zur Expansion gereinigt wurden (Tabelle 1)18,20,21. Daher sind in vitro NSC-Kulturen standardmäßig aNSCs.
- Um qNSCs zu erzeugen, verwenden Sie das folgende Protokoll, um aNSCs auf beschichtete Glaswaren aufzuplättieren und sie dann mit qNSC-Medium zu behandeln, um eine Ruhephase über 3 Tage zu induzieren.
- Bereiten Sie Glaswaren mit einem Brechungsindex vor, der für Ölimmersionsobjektive zum Anhaften von NSCs geeignet ist, indem Sie zuerst mit einer Poly-L-Ornithin-Lösung (PLO; 10 mg/mL in Wasser für Kunststoff, 50 mg/mL in Wasser für Glas) beschichten, die ausreicht, um den Boden der Schale zu bedecken (für eine 1 cm2 Abbildungsküvette verwenden Sie ~150 μL) für 1 h bei 37 °C in ddH2O.
- Entfernen Sie die PLO-Lösung und waschen Sie sie 3 Mal mit phosphatgepufferter Kochsalzlösung (PBS).
- Mit Lamininlösung (5 mg/ml) bestreichen, die ausreicht, um den Boden der Schale zu bedecken (für eine 1 cm2 Bildküvette verwenden, verwenden Sie ~150 μl) in PBS und mindestens 3 Stunden bei 37 °C oder über Nacht inkubieren.
- Entfernen Sie die Lamininlösung und fügen Sie dann ein Kulturmedium hinzu, das ausreicht, um den Boden der Schale zu bedecken (für eine 1 cm2 Bildgebungsküvette verwenden Sie ~150 μl).
- Bereiten Sie die Zellen für die Trypsinisierung vor, indem Sie aNSCs (sie können als Monoschichten oder Neurosphären beginnen) bei 120 x g für 4 min bei Raumtemperatur (RT, ~25 °C) zentrifugieren.
- Trypsinisieren Sie pelletierte aNSCs aus dem vorherigen Schritt 5 Minuten lang in 100 μl Trypsin-Mix bei 37 °C und löschen Sie dann die Trypsinisierung mit 200 μl Trypsin-Inhibitor-Mix (Tabelle 1).
- Lassen Sie die NSCs 2 Minuten lang ruhen und zerkleinern Sie sie dann mechanisch, indem Sie sie 10 Mal auf und ab pipettieren.
- Fügen Sie 5 mL aNSC-Medium hinzu und drehen Sie die Zellen 4 Minuten lang bei 120 x g .
- Resuspendieren Sie die NSCs in 1 mL aNSC-Medium und plattieren Sie die Zellen bei 10%-20% Konfluenz in aNSC-Medium. Platten Sie beispielsweise ~5.000 Zellen in eine Vertiefung einer 1 cm2 großen Bildgebungsküvettenvertiefung mit 150 μl Medium.
- Nachdem Sie 24 Stunden gewartet haben, bis aNSCs an der Oberfläche der Schale haften, entfernen Sie das aNSC-Medium und ersetzen Sie es durch qNSC-Medium, das ausreicht, um den Boden der Schale zu bedecken (für eine 1 cm2 Bildgebungsküvette verwenden Sie ~150 μL; Tabelle 1 - wie zuvor beschrieben 10,13,17,18) in Vertiefungen, in denen Ruhe induziert wird.
- Wechseln Sie aNSC und qNSC medium mindestens alle 2 Tage. Nach 3 Tagen im qNSC-Medium sind die NSCs ruhig und können für bildgebende Experimente verwendet werden.
- Verwenden Sie ein konfokales Mikroskop, um lebende qNSCs und aNSCs in vitro abzubilden.
HINWEIS: Jedes Mikroskop hat seine eigene Software; Befolgen Sie daher diese Schritte, indem Sie analoge Aktionen ausführen, um ein bestimmtes Mikroskop zu konfigurieren. Dieses Protokoll enthält Anweisungen für die Arbeit in NIS-Elements.- Konfigurieren Sie das konfokale Mikroskop für die Autofluoreszenzbildgebung.
- Klicken Sie auf 405 Laser und geben Sie den Text in das Emissionsfenster im Menü 405 Laser ein. Klicken Sie auf TD , um ein Durchlichtbild aufzunehmen, und stellen Sie den HV-Wert ein, um die Probe zu visualisieren.
- Stellen Sie die Laserleistung auf 3,5 % und die Verstärkung auf 75 % ein. Legen Sie den Zoom auf 2 fest. Richten Sie die konfokalen Scanparameter ein, legen Sie die Pixelverweildauer auf 0,5 und die Auflösung auf 2048 x 2048 Pixel fest.
- Bringen Sie die Zellen unter einer ~60-fachen Öl-Immersionsobjektivlinse mit Hellfeld in den Fokus.
- Wechseln Sie in den konfokalen Scanmodus, indem Sie auf Eye Port klicken und sicherstellen, dass der Strahlengang nun zum Scankopf geht und sich kein Filterwürfel im Strahlengang befindet.
- PAF ist mit qNSCs angereichert und weitgehend erregbar und emittierbar (siehe Abbildung 1). Wählen Sie die beste optische Konfiguration basierend auf den Fähigkeiten des Systems aus, wobei Sie sich an Abbildung 1 orientieren. Für das stärkste und sauberste Signal regen Sie mit einem Laser zwischen ~400-500 nm an, sammeln Sie ~580-620 nm und verwenden Sie eine ~60x Öl-Immersionsobjektivlinse.
HINWEIS: Die Autofluoreszenzbildgebung erfordert höhere Laserleistungen zur Anregung als bei typischen Bildgebungsexperimenten. Wenn Sie andere autofluoreszierende Moleküle oder andere Markierungen zusammen mit PAF abbilden, ist die optische Konfiguration sorgfältig zu gestalten, um ein Durchscheinen in Autofluoreszenzkanäle zu vermeiden. - Erfassen Sie Bilder der Autofluoreszenz in qNSCs und aNSCs. Klicken Sie auf Scannen, fokussieren Sie sich auf das Bild, und klicken Sie dann auf Erfassen. Die besten Bilder erhalten Sie, wenn Sie Bilder in einer einzelnen Ebene mit dem Zytoplasma von Zellen (basierend auf einer diffus in der Zelle verteilten Dimmer-Autofluoreszenz) im Fokus aufnehmen, um einen repräsentativen Querschnitt jeder Zelle zu erhalten.
- Verwenden Sie eine identische optische Konfiguration (z. B. gleiche Laserleistung) und Bildproben am selben Tag, wenn Sie verschiedene Bilder direkt vergleichen möchten.
- Die genaue Leistung des Lasers und des Detektors hängt vom jeweiligen System ab. Beginnen Sie mit niedrigen Laserleistungen und erhöhen Sie dann langsam die Leistungen, bis das Signal nachweisbar ist, ohne gesättigt zu sein.
- Konfigurieren Sie das konfokale Mikroskop für die Autofluoreszenzbildgebung.
- Analysieren Sie PAF in lebenden qNSCs und aNSCs in vitro.
- Nachdem Sie Autofluoreszenzbilder in qNSCs oder aNSCs aufgenommen haben, quantifizieren Sie die Bilder, indem Sie eine Datei in ImageJ25 öffnen.
- Verwenden Sie die Option Prozess > Filter > Gaußscher Weichzeichner (Sigma: 2) auf das Bild der qNSC/aNSC-Autofluoreszenz, um ungleichmäßige Pixel zu glätten.
- Verwenden Sie Bild> > Schwellenwert anpassen , um Pixel auszuwählen, die PAF darstellen (Hintergrundpixel entfernen), und klicken Sie auf Übernehmen. Verwenden Sie die gleichen Schwellenwertparameter für alle Bilder, die direkt verglichen werden.
- Verwenden Sie Process > Binary > Watershed , um PAFs in unmittelbarer räumlicher Nähe zu trennen.
- Zeichnen Sie mit der Computermaus eine Region of Interest (ROI) um eine Zelle, die entweder durch Betrachten eines Hellfeldbildes oder durch Betrachten der diffusen Autofluoreszenz bestimmt wird, die gleichmäßig im Zytoplasma der Zelle vorhanden ist.
HINWEIS: Es ist im Allgemeinen in Ordnung, dünne periphere Prozesse auszuschließen und den Zellkern einzubeziehen, da an diesen Stellen in der Regel keine Autofluoreszenz vorhanden ist und daher die Analyse nicht stören würde. - Verwenden Sie Analysieren > Partikel analysieren (Größe: 0,1 μm unendlich; Zirkularität: 0-1) mit aktivierter Zusammenfassung.
HINWEIS: ImageJ erzeugt dann ein Ausgabefenster mit Daten zu den Partikeln im interessierenden Bereich, das die tabellarische Darstellung der Anzahl der PAF in jeder Zelle ermöglicht. Andere fluoreszierende Moleküle können mit bildgebender PAF gepaart werden. Da PAF jedoch ein relativ schwaches Signal ist, ist es wichtig zu validieren, dass andere Fluorophore nicht in den PAF-Kanal einbluten. In der Regel können Fluorophore, die 600+ nm absorbieren und 650+ nm emittieren, mit bildgebenden PAF, wie z. B. LipidSpot 610, gepaart werden.
2. Verwendung von FACS zur Anreicherung des NSC-Aktivierungszustands in kultivierten NSCs auf der Grundlage von Autofluoreszenz
- Generieren Sie qNSCs und aNSCs, wie in Abschnitt 1 beschrieben.
- Sortieren Sie eine Mischung aus lebenden qNSCs und aNSCs basierend auf Autofluoreszenz.
HINWEIS: Als Beispiel wird in diesem Protokoll erläutert, wie aNSCs oder qNSCs in einer Probe angereichert werden, die durch Mischen von qNSCs und aNSCs im Verhältnis 1:1 erzeugt wird.- Vor der Trypsinisierung und Zellsortierung markieren Sie die Zellen, die die S-Phase durchlaufen, indem Sie dem Zellkulturmedium 10 μM 5-Ethinyl-2'-Desoxyuridin (EdU; ein Thymidinanalogon, das in Zellen eingebaut wird, die DNA synthetisieren, wie z. B. in der S-Phase des Zellzyklus) hinzufügen.
- Trypsinisieren Sie qNSCs und aNSCs wie in Schritt 1.1.8 beschrieben, resuspendieren Sie sie in 0,5 ml FACS-Puffer (Tabelle 1) und legen Sie sie dann auf Eis.
HINWEIS: qNSCs haften stark an der Schale. Bei relativ höherer Konfluenz können qNSCs mechanisch von der Schale dissoziiert werden. Wenn sich qNSCs jedoch bei einer relativ geringeren Konfluenz befinden oder es schwierig ist, sie mechanisch durch Pipettieren aus der Schale zu entfernen, verwenden Sie Trypsin, um sie aus der Schale zu entfernen.- Entfernen Sie das Nährmedium aus den qNSCs und fügen Sie 0,25 % Trypsin hinzu, das ausreicht, um den Boden der Schale zu bedecken (für eine Vertiefung mit einer Bildgebungsküvette von 1 cm2 verwenden Sie ~150 μl) und inkubieren Sie es 3 Minuten lang bei 37 °C.
- Nach der Inkubation wird die Trypsinreaktion durch Zugabe von Medium, das jedem Zelltyp entspricht, gleich dem Volumen des zugegebenen Trypsins gequält. Dissoziation der Zellen von der Schale vor der Entnahme und Zentrifugation.
- Mit einer ~130-μm-Düse können qNSCs und aNSCs mit einem Durchflusszytometer oder FACS unter optischen Bedingungen basierend auf den Fähigkeiten des Geräts analysiert und PAF nachgewiesen werden, wie in Abbildung 1 gezeigt. Verwenden Sie beispielsweise einen 405-nm-Laser, um die Probe anzuregen, und verwenden Sie einen 580-620-nm-Filter für die Detektion.
- Sammeln Sie mindestens 10.000 Singulett-qNSCs und aNSCs in einer Datendatei und verwenden Sie diese dann, um Gates zu entwerfen, um eine Anreicherung von qNSCs oder aNSCs zu sammeln. Legen Sie beispielsweise Gates fest, um die oberen 25 % oder unteren 25 % der NSCs basierend auf der Autofluoreszenzintensität in der gemischten Probe aNSC:qNSC (1:1) zu erfassen.
- Nach dem Setzen von Gates zum Sortieren von Einzellingen mit hellerer oder dunklerer Autofluoreszenz wie oben beschrieben, sortieren Sie die Zellen in PLO-, Laminin-beschichtete Vertiefungen, die mit aNSC-Medium (10.000 Zellen/cm2, siehe Schritt 1.1.3) gefüllt sind.
- Lassen Sie die NSCs nach dem FACS 3 Stunden im Inkubator sitzen. Fixieren Sie die Zellen, indem Sie sie mit 4% Paraformaldehyd behandeln, das ausreicht, um den Boden der Schale zu bedecken (für eine 1 cm2 Bildgebungsküvette verwenden Sie ~150 μl) für 15 Minuten bei RT.
HINWEIS: Um EdU in Zellen sichtbar zu machen, ist es am einfachsten, ein kommerzielles Kit zum Nachweis von EdU zu erwerben und das vom Hersteller empfohlene Protokoll zu befolgen. - Zuerst permeabilisieren Sie die Zellen durch Behandlung mit 0,25% Triton in PBS für 15 min bei RT.
- Behandeln Sie anschließend die Zellen 30 Minuten lang mit einer EdU-Färbelösung bei RT.
HINWEIS: Die genaue Zusammensetzung der EdU-Färbelösung hängt von der Quelle der Reagenzien ab, besteht jedoch grob aus einem Reaktionspuffer, einem Reaktionspufferzusatz, Kupfersulfat und einem Alkin- oder Azid-modifizierten Molekül. In der Materialtabelle finden Sie ein empfohlenes Kit. - Nach der Färbung zur Visualisierung von EdU waschen Sie die Proben 3 Mal für 10 Minuten mit PBS.
3. Verwendung eines Multiphotonenmikroskops zum Nachweis der Autofluoreszenz von Kanal 1 und Kanal 2 und Durchführung von FLIM an NSCs in vitro zur Identifizierung von NSC-Zellzustandspopulationen und -übergängen
HINWEIS: Jedes Mikroskop hat seine eigene Software; Befolgen Sie daher diese Schritte, indem Sie analoge Aktionen ausführen, um ein bestimmtes Mikroskop zu konfigurieren. Dieses Protokoll enthält Anweisungen für die Arbeit in Prairie View.
- Generieren Sie qNSCs und aNSCs, wie in Abschnitt 1 beschrieben.
- Richten Sie das Multiphotonenmikroskop für die Fluoreszenzlebensdauer-Bildgebung ein.
- Verwenden Sie ein Objektiv mit 40-facher oder größerer Vergrößerung (~1,15 NA).
- Verwenden Sie ein Multiphotonenmikroskop mit den folgenden oder gleichwertigen Komponenten: einem abstimmbaren Ultrakurzpulslaser, einem dichroitischen 720-nm-Langpassmikroskop, einer Galliumarsenidphosphid (GaAsP)-Photomultiplier-Röhre (PMT), einer zeitkorrelierten Einzelphotonen-Zählelektronik und einem analogen Leistungsmesser.
- Stellen Sie für die Bildgebung von Kanal 1 den abstimmbaren Laser auf 750 nm ein und verwenden Sie einen 440/80-nm-Emissionsfilterwürfel. Stellen Sie für die Bildgebung von Kanal 2 den Laser auf 890 nm ein und verwenden Sie einen 550/100-nm-Emissionsfilterwürfel.
HINWEIS: Schalten Sie die Raumbeleuchtung aus, bevor die PMTs eingeschaltet werden.
- Funktion "Ansprechverhalten des Geräts"
- Erfassen Sie eine Instrumentenreaktionsfunktion (IRF), indem Sie einen zerkleinerten Harnstoffkristall abbilden, um die zeitliche Reaktion des Instruments beim gemessenen Zerfall zu berücksichtigen.
HINWEIS: Die IRF wird mit dem Abklingen gefaltet, um die Abklinganpassungskurve genau zu berechnen. Für die Erfassung wird der Laser auf 890 nm eingestellt und der 440/80-nm-Emissionsfilterwürfel verwendet. Der GaAsP PMT ist auf eine Verstärkung von 800 eingestellt, und die Pockels (Laserleistung) werden anfangs sehr niedrig eingestellt, in der Regel 1. - Wählen Sie ein Sichtfeld mit kristallinem Harnstoff und erhöhen Sie die Pockeln leicht (bis zu einem Maximum von 15).
- Nehmen Sie als Nächstes ein Bild mit den gleichen Parametern auf, die für die Zellbildgebung verwendet werden: konstanter Fraktionsdiskriminator (CFD) 1 x 104 - 1 x 105, Auflösung 256 x 256, Verweilzeit auf 4,8 μs, optischer Zoom auf 1,5x und Integrationszeit von 60 s.
- Erfassen Sie eine Instrumentenreaktionsfunktion (IRF), indem Sie einen zerkleinerten Harnstoffkristall abbilden, um die zeitliche Reaktion des Instruments beim gemessenen Zerfall zu berücksichtigen.
- Durchführung von Fluoreszenzlebensdauer-Bildgebung und Intensitätsbildgebung für Autofluoreszenz von Kanal 1 und Kanal 2 an lebenden qNSCs und aNSCs in vitro
- Finden Sie ein Sichtfeld für das Bild: Verwenden Sie das Okular, um ein geeignetes Sichtfeld zu finden, und ändern Sie den Lichtweg zur Probe am Mikroskop, um die Bildgebung zu ermöglichen.
HINWEIS: Zuerst wird ein Bild von Kanal 1 erfasst, gefolgt von einem Bild von Kanal 2 mit demselben Sichtfeld. - Setup für die Erfassung des Kanal-1-Bildes: Klicken Sie auf die Registerkarte Power/Gain und stellen Sie die Verstärkung am PMT auf 800 ein, klicken Sie auf die Registerkarte 2-P-Laser und wählen Sie 750 nm für den Laser und stellen Sie sicher, dass der richtige Filterwürfel (440/80 nm) verwendet wird.
- Erfassen Sie das Bild von Kanal 1.
- Starten Sie den Vorschaumodus , indem Sie auf die Registerkarte Power/Gain gehen und die Pockels auf 30 einstellen, dann zum Scan-Bereich dieses Bildschirms gehen und auf Live-Scan klicken.
- Erhöhen Sie die Pockels , um ein gutes Sichtfeld bei geringer Laserleistung (unter 3,6 mW) zu finden. Sobald das Sichtfeld gefunden ist, stellen Sie die Pockels so ein, dass der Leistungsmesser ~3,6 mW für die Leistung nach der Pockel-Zelle anzeigt und der konstante Fraktionsdiskriminator (CFD) auf der Registerkarte "Count Rates " 1 x 104 - 1 x 105 misst.
- Gehen Sie zum Abschnitt Scannen und klicken Sie auf Einzelner Scan , sobald diese Parameter erfüllt sind. Dadurch wird ein Bild von Kanal 1 über einen Zeitraum von 60 s aufgenommen.
- Um ein Bild von Kanal 2 aufzunehmen, klicken Sie auf die Registerkarte 2-P-Laser und wählen Sie 890 nm für den Laser aus, tauschen Sie den Filterwürfel gegen den Emissionswürfel von Kanal 2 (550/100 nm) aus.
- Starten Sie den Vorschaumodus wie in 3.4.3 beschrieben, erhöhen Sie die Pockels , bis der Leistungsmesser ~7 mW anzeigt und die CFD-Zählungen wieder 1 x 104 -1 x 105 betragen.
- Gehen Sie zum Abschnitt Scannen und klicken Sie auf Einzelner Scan , sobald diese Parameter erfüllt sind. Dadurch wird ein Bild von Kanal 2 über eine Länge von 60 s aufgenommen.
- Weitere Bilder aufnehmen - Nachdem Sie ein Bild von Kanal 1 und Kanal 2 aufgenommen haben, suchen Sie ein neues Sichtfeld und wiederholen Sie die Schritte 3.4.2 - 3.4.4. In der Regel ist das Sammeln von 4-6 Sichtfeldern zur Abbildung von ~50-100 Zellen für eine Bedingung in einem Experiment ausreichend.
HINWEIS: mCherry kann als zusätzliche fluoreszierende Markierung verwendet werden, ohne in die Kanäle von Kanal 1 und Kanal 2 durchzuscheinen.
- Finden Sie ein Sichtfeld für das Bild: Verwenden Sie das Okular, um ein geeignetes Sichtfeld zu finden, und ändern Sie den Lichtweg zur Probe am Mikroskop, um die Bildgebung zu ermöglichen.
- Analysieren Sie die fluoreszierenden Lebensdauerbilder.
- Generieren Sie Zerfallsmatrizen für Bilder mit Fluoreszenzlebensdauer in SPCImage, indem Sie die Schritte 3.5.2-3.1.14 befolgen.
HINWEIS: Weitere Informationen finden Sie in den aktualisierten Handbüchern, die online frei verfügbar sind26. - Öffnen Sie SPCImage und öffnen Sie das in Abschnitt 3.3 gesammelte IRF-FLIM-Image.
- Passen Sie die vertikalen schwarzen Balken im Histogramm so an, dass sie auf die IRF-Abklingkurve beschränkt sind. Klicken Sie in der oberen Menüleiste auf IRF > In die Zwischenablage kopieren.
- Öffnen Sie eine FLIM-Bilddatei innerhalb des zu analysierenden Datensatzes, den Sie in Abschnitt 3.4 erhalten haben.
- Klicken Sie in der oberen Menüleiste auf IRF > Aus Zwischenablage einfügen.
- Legen Sie unten rechts in der Software Komponenten auf 2 fest.
- Klicken Sie in der oberen Menüleiste der Software auf Optionen > Modell. Wählen Sie dann MLE für Anpassungsmethode, Räumliches Binning in Quadrat, Schwellenwert bis Spitze aus, und wählen Sie unter Einstellungen die Option Multiexponentieller Zerfall aus.
- Erhöhen Sie Bin unten links in der Software, bis die Photonenzahl in repräsentativen zytoplasmatischen Pixeln am Höhepunkt der Photonenzahl unmittelbar nach der Anregung mindestens 100 beträgt.
- Legen Sie unten links in der Software den Schwellenwert fest. Verwenden Sie für Kanal 1 einen Schwellenwert von "50". Verwenden Sie für Kanal 2 einen Schwellenwert von "0".
- Klicken Sie in der oberen Menüleiste der Software (dieses Protokoll beschreibt die Bedienung der Version 8.3) auf Calculate > Decay Matrix.
HINWEIS: Stellen Sie sicher, dass die Chi2-Werte im gesamten Bild ~0,8-1,2 betragen. Dies bestätigt, dass die Daten robust sein werden, indem die biexponentielle Zerfallsmodellierung validiert wird. - Klicken Sie in der oberen Menüleiste auf Datei > Speichern.
- Klicken Sie in der oberen Menüleiste der Software auf Datei > Exportieren und exportieren Sie Folgendes:
Farbcodierter Wert, T1, T2, Chi2, Pixelintensitäten, A1[%], A2[%] und farbcodiertes Bild (mit Legende)
HINWEIS: Jetzt ist SPCImage so eingerichtet, dass es einen Stapelprozess durchführt, bei dem alle Bilder eines bestimmten Kanals analysiert werden (analysieren Sie die FLIM-Bilder von Kanal 1 zusammen und wiederholen Sie dann den Vorgang ab Schritt 3.5.2 für FLIM-Bilder von Kanal 2). - Um einen Stapelprozess durchzuführen, klicken Sie auf > Stapelverarbeitung berechnen und wählen Sie die zu verarbeitenden FLIM-Bilder aus.
- Klicken Sie in der oberen Menüleiste auf Datei > Export > Batch-Export und wählen Sie die zu exportierenden Decay-Matrizen aus (die im vorherigen Schritt generiert wurden).
- Verwenden Sie CellProfiler, um zytoplasmatische Masken zu erstellen, indem Sie die Schritte 3.5.16-3.5.24 befolgen.
HINWEIS: Zu diesem Zweck werden die Kerne manuell um die Intensitätsbilder von Kanal 1 herum angeordnet, wobei der Kern schwarz und deutlich sichtbar ist. Dann erzeugt eine CellProfiler-Pipeline manual_segmentation.cpproj27 (Ergänzende Datei 1) automatisch eine Ganzzell- und Zytoplasmamaske, wie von der Kernmaske angewiesen. Zellkerne können alternativ mit mCherry markiert und zusammen mit der Autofluoreszenz von Kanal 1 und Kanal 2 abgebildet werden, um die Identifizierung von Zellkernen automatisieren zu können. Viele herkömmliche Kernfarbstoffe bluten jedoch spektral in die Autofluoreszenzkanäle und sind daher mit dieser Technik nicht kompatibel. - Verwenden Sie in R Studio R_ASCtoTIFF.rmd27 (Ergänzende Datei 2), um die X_photons.asc-Dateien zu konvertieren, wobei X der Name des erfassten Bildes aus dem Kanal 1-Bildsatz in TIF-Dateien ist.
- Öffnen Sie Cell Profiler, und öffnen Sie manual_segmentation.cpproj.
- Laden Sie die Photonen-TIFs für Kanal 1, die in Schritt 3.5.16 im Schritt "Bilder" am Anfang der Pipeline erstellt wurden.
- Legen Sie in den unteren 3 Schritten in der Pipeline mit dem Titel "SaveImages" einen Speicherpfad für die Masken fest, die CellProfiler generiert.
- Klicken Sie unten links im CellProfiler auf Testmodus starten .
- Klicken Sie unten links in CellProfiler auf Ausführen . Wenn CellProfiler zum Schritt Identify ObjectsManually gelangt, wird ein Fenster angezeigt, in dem das aktuelle Bild von Kanal 1 angezeigt wird, das gerade verarbeitet wird.
- Klicken Sie auf die Taste F auf der Tastatur, und zeichnen Sie dann manuell eine Spur der Zellkerne, während Sie die linke Maustaste gedrückt halten. Nachdem Sie die Kerne verfolgt haben, lassen Sie die Linksklick-Taste auf der Maus los. Wiederholen Sie diesen Schritt für alle Zellen im Bild.
- Klicken Sie unten rechts im Popup-Fenster mit dem Intensitätsbild auf Fertig . Die Software wird nun die Pipeline abschließen und zytoplasmatische, nukleare und totale Zellmasken exportieren.
- Klicken Sie unten links auf Next Image Set und wiederholen Sie die Schritte 3.5.21-3.5.23 für alle Channel 1 TIF-Bilder.
- Verwenden Sie das R-Skript (Integrate decay matrices and cytoplasmic masks.rmd), um die in den Schritten 3.5.2 bis 3.5.14 generierten Zerfallsmatrizen und die in den Schritten 3.5.15 bis 3.5.24 generierten zytoplasmatischen Masken zu integrieren, um die durchschnittlichen Fluoreszenzlebensdauer-Bildgebungsvariablen für das Zytoplasma jeder Zelle zu erhalten, indem Sie die Schritte 3.5.26 bis 3.5.30 befolgen
- Erstellen Sie mithilfe einer Tabellenkalkulation (z. B. Microsoft Excel) ein .csv Dokument mit dem Titel test_key27 [Ergänzungsdatei 3] als Beispiel) mit 3 Spalten.
- Betiteln Sie die Spalten als Ordner, NADH und FAD. Füllen Sie die Tabelle aus, um den Ordnerspeicherort im Ordner aufzulisten, und geben Sie dann das Präfix des Bildtitels in NADH bzw. FAD an, um jedes Bild von Kanal 1 mit jedem Bild von Kanal 2 zu verknüpfen (siehe Beispieldaten - Tabelle 2).
- Generieren Sie Zerfallsmatrizen für Bilder mit Fluoreszenzlebensdauer in SPCImage, indem Sie die Schritte 3.5.2-3.1.14 befolgen.
- Öffnen Sie in R Studio Integrate decay matrices and cytoplasmic masks.rmd27(Ergänzende Datei 4).
- Legen Sie die folgenden Dateipfade fest, wie sie im Skript mit Anmerkungen versehen sind: Festlegen des Arbeitsverzeichnisses, des Dateispeicherorts von Kanal 1, des Speicherorts der FAD-Datei, des Speicherorts der Maskendatei sowie des Speicherorts und des Namens der Ausgabedatei.
- Führen Sie das gesamte Skript aus. Nach erfolgreichem Abschluss des Skripts wird eine Ausgabedatei generiert, die Metadaten und durchschnittliche FLIM-Variablen enthält.
- Führen Sie die Analyse mit der Autofluoreszenz-FLIM-Datei data.rmd27 (Ergänzende Datei 5) oder mit einer anderen Analysesoftware durch.
Ergebnisse
Konfokale Autofluoreszenzbildgebung zur Trennung des NSC-Zellzustands (Abbildung 1)
Um die konfokale Mikroskopie zur Auflösung des NSC-Aktivierungszustands zu verwenden, wurden qNSCs und aNSCs in vitro entweder unter Verwendung eines Aktivierungsmediums oder eines Ruhemediums erzeugt, wie zuvor beschrieben 10,13,17,18. Zum Nachweis von PAF in NSCs wurden lebende qNSCs und aNSCs mit der gleichen Belichtung auf einem konfokalen Mikroskop abgebildet (Bsp.: 405 nm, Em: 580-620 nm). qNSCs wiesen im Vergleich zu aNSCs eine höhere Anzahl von PAF auf (Abbildung 1A,B). Diese Erkenntnis zeigt, wie Autofluoreszenzeigenschaften als Marker verwendet werden können, um den Zellzustand von qNSCs und aNSCs zu identifizieren.
FACS-Anreicherung des NSC-Zellzustands mittels Autofluoreszenz (Abbildung 2)
Zur Anreicherung für den Zellzykluszustand mittels FACS wurden qNSCs und aNSCs in vitro 10,13,17,18,19 wie in diesem Protokoll beschrieben erzeugt und vor der Trypsinisierung und Analyse im Durchflusszytometer 1 h lang mit EdU vormarkiert, um Zellen zu markieren, die die S-Phase durchlaufen. qNSCs und aNSCs wurden dann mittels Durchflusszytometrie entweder getrennt oder gemischt in einem Verhältnis von 1 qNSC:1 aNSC analysiert (Abbildung 2). Es wurden Gates gezogen, um aNSCs oder qNSCs aus der gemischten Population anzureichern, und die Zellen wurden dann basierend auf diesen Gates sortiert. Nach der FACS wurden die Zellen in jeder Probe auf PLO- und lamininbeschichtete Glaswaren aufgebracht und 3 Stunden lang an der Schale haften, bevor sie fixiert, gefärbt und auf %EdU+-Zellen analysiert wurden. Erwartungsgemäß wiesen Zellen, die aus dem hohen Autofluoreszenz-Gate sortiert wurden, weniger EdU+ auf als die Mix-Probe, und Proben, die durch das niedrige Autofluoreszenz-Gate sortiert wurden, waren proliferativer als die Mix-Probe (Abbildung 2C). Dieser Befund bestätigt die Fähigkeit zur Anreicherung des NSC-Aktivierungszustands aus einer heterogenen Mischung von qNSCs und aNSCs unter Verwendung von FACS.
Multiphotonen-Fluoreszenzlebensdauer-Bildgebung zur Klassifizierung des NSC-Zellzustands (Abbildung 3)
qNSCs und aNSCs wurden in vitro erzeugt und dann mit einem Multiphotonenmikroskop abgebildet, um FLIM auf Kanal 1 Autofluoreszenz (Bsp.: 750 nm (2P), Em: 360-520 nm) und Kanal 2 (Bsp.: 890 nm (2P), Em: 450-650 nm) durchzuführen (Abbildung 3A, Tabelle 2). qNSCs und aNSCs wiesen autofluoreszierende Profile auf, die sich weitgehend signifikant unterschieden. Zum Beispiel wiesen qNSCs im Vergleich zu aNSCs eine höhere mittlere Lebensdauer der Fluoreszenz von Kanal 1τ m auf, aber eine niedrigere α1 . Um die Fähigkeit von NSC-FLIM-Autofluoreszenzdaten zur Vorhersage des NSC-Aktivierungszustands zu bewerten, wurde ein logistisches Regressionsmodell mit der Intensität von Kanal 1, α1, τ1,τ 2 und der Intensität von Kanal 2, α1, τ1, τ2 erstellt. Eine Empfängeroperatorkurve zeigt, dass diese Daten ausreichen, um ein nahezu perfektes Modell (Fläche unter der Kurve = 0,963) zu erstellen, das den NSC-Aktivierungszustand genau vorhersagt. Zusammengenommen veranschaulichen diese Daten die Fähigkeit von FLIM und Autofluoreszenz, zur Klassifizierung des NSC-Zellzustands verwendet zu werden.
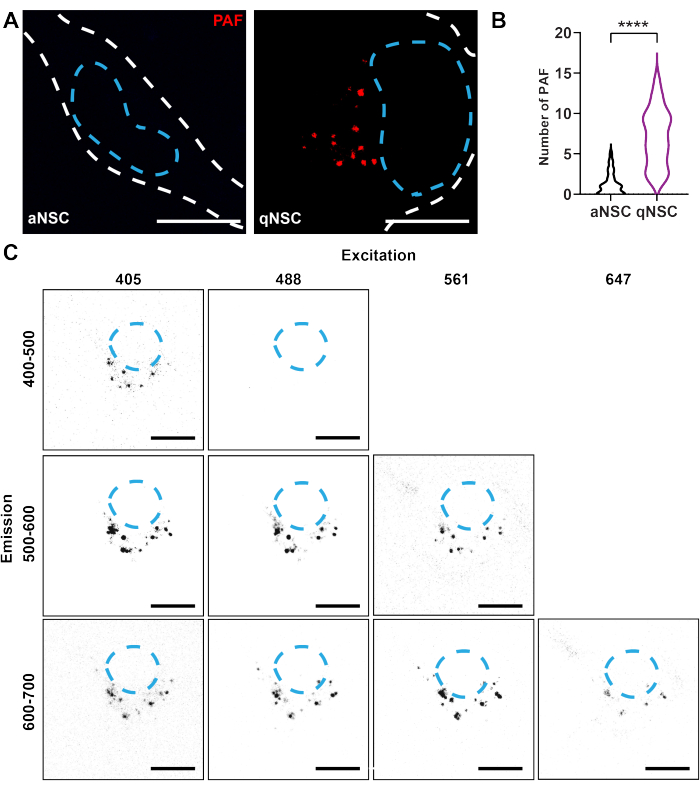
Abbildung 1: Die konfokale Bildgebung klärt autofluoreszierende Biomarker für den NSC-Aktivierungszustand auf. (A-B) qNSCs (violett) und aNSCs (schwarz) wurden mit der gleichen Belichtung auf einem konfokalen Mikroskop (rot; Bsp. 405 nm, Em 580-620 nm) abgebildet und auf die Anzahl der PAF analysiert (N = 3, Mann-Whitney-Test, mittlere ± SD). Weiße gestrichelte Linien kennzeichnen den Rand der Zelle, und blaue gestrichelte Linien kennzeichnen Zellkerne. (C) Die Autofluoreszenz im selben qNSC wurde unter verschiedenen Anregungs- und Emissionsbedingungen, wie in der Abbildung gezeigt, mit identischer Laserleistung und -verstärkung abgebildet. Maßstab: 10 μm. **** p < 0,0001. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
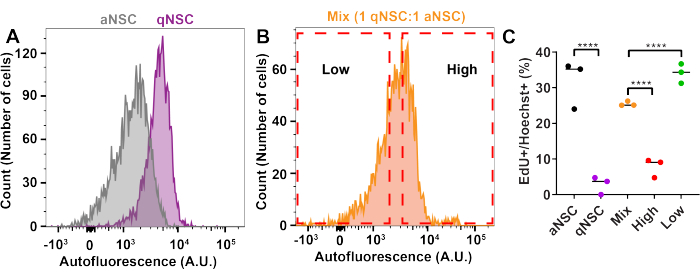
Abbildung 2: FACS kann für den NSC-Aktivierungszustand anreichern. (A-C) qNSCs, aNSCs oder a qNSCs: aNSCs Mix (1:1) wurden 1 h lang mit EdU behandelt, trypsinisiert und anschließend mittels Durchflusszytometrie analysiert (Bsp.: 405 nm, Em: 580-620 nm). Die Mischzellen (1:1) wurden dann nach FACS nach Zellen sortiert, die entweder eine niedrige oder eine hohe Autofluoreszenz aufwiesen, plattiert und auf Proliferation analysiert, indem der Prozentsatz der Zellen gemessen wurde, die EdU+ waren. Arbitrary units wird mit "A.U." abgekürzt. (N = 4, bidirektionale ANOVA mit post-hoc-Tukey-Test, Mittelwert ± SD). p < 0,0001. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
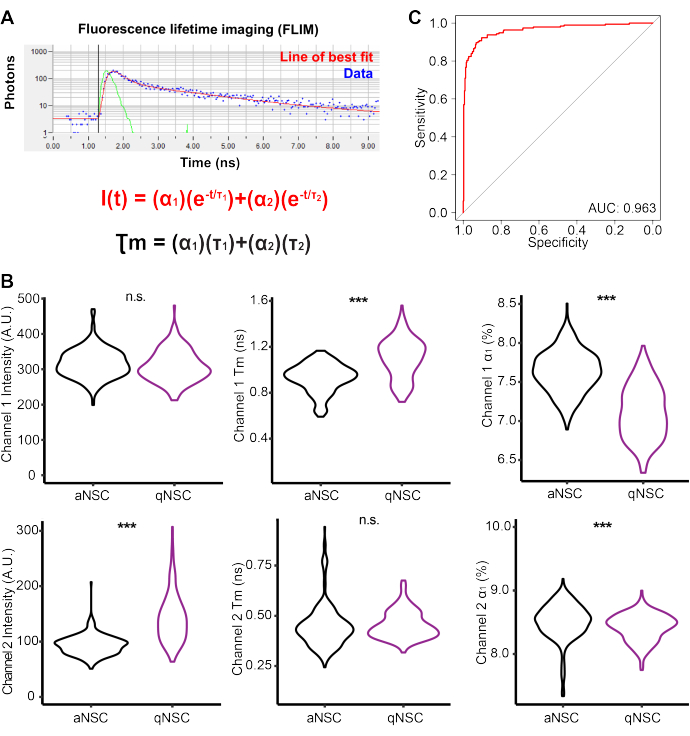
Abbildung 3: Die Bildgebung der Multiphotonen-Fluoreszenzlebensdauer zeigt Biomarker für den NSC-Aktivierungszustand. (A) Schematische Darstellung der Kurvenanpassungsanalyse der erfassten FLIM-Daten. (B) FLIM-Messungen von Kanal 1 und Kanal 2, einschließlich der Werte für Intensität, mittlere Lebensdauer (Tm) und fraktionellen Beitrag (α1) für qNSC- (violett) und aNSC-Daten (schwarz) (n = 501 Zellen, beidseitige logistische Regression, verallgemeinertes lineares Modell). (C) Empfängeroperatorkurve, die ein logistisches Regressionsmodell zeigt, das unter Verwendung der Intensität von Kanal 1, α1, τ1, τ2 und der Intensität von Kanal 2, α1, τ1, τ2 generiert wurde, um NSCs als aNSCs oder qNSCs zu klassifizieren. p < 0,001. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Tabelle 1: Medien und Lösungen. Rezepte für alle Lösungen, die in diesem Protokoll verwendet werden. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 2: Beispieldaten. Repräsentative qNSC- und aNSC-FLIM-Daten für die Autofluoreszenz von Kanal 1 und Kanal 2. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Ergänzende Datei 1: manual_segmentation.cpproj Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 2: R_ASCtoTIFF.rmd Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 3: test_key Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 4: Zerfallsmatrizen und zytoplasmatische Masken integrieren.rmd Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 5: Autofluoreszenz FLIM data.rmd Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Dieses Protokoll beschreibt eine lebende, markierungsfreie, zerstörungsfreie Einzelzellauflösungstechnik, die die Klassifizierung des NSC-Zellzustands in vitro durch Bildgebung von Autofluoreszenzsignalen in NSCs ermöglicht. Dieser Ansatz detektiert metabolische Verschiebungen, die während des Austritts der NSC-Ruhephase auftreten, die die optischen Eigenschaften von metabolischen Cofaktoren, wie z.B. NAD(P)H, beeinflussen, und bietet viele Vorteile gegenüber bestehenden Technologien zur Untersuchung der NSC-Ruhe. Beispielsweise erfordern viele herkömmliche Techniken zur Untersuchung von qNSCs und aNSCs, wie z. B. die Markierung mit Zellzyklusmarkern wie EdU, die Fixierung von Proben. Derzeit existierende Methoden, die in der Lage sind, lebende NSCs zu untersuchen, sind weiter eingeschränkt, da die Einführung exogener Markierungen erforderlich ist, typischerweise durch die Erzeugung von fluoreszierenden Protein-kodierenden Transgenen. Diese Tools sind sowohl in Bezug auf die Ressourcen, die für ihre Generierung erforderlich sind, als auch in vielen technischen Vorbehalten begrenzt. Zum Beispiel werden die intermediären Filamentproteine Nestin und Glial Fibrillary Acidic Protein (GFAP) häufig als Marker für qNSCs und aNSCsverwendet 12,13,18,28,29. Es ist jedoch bekannt, dass Nestin und GFAP zwischen qNSCs und aNSCs unterschiedlich exprimiert werden 12,13,18,28,29. Darüber hinaus liefert die Autofluoreszenzbildgebung hochauflösende Einzelzelldaten, die die Heterogenität einzelner Zellen enträtseln können, die in Experimenten, die in einer Massenanalyse einer Zellpopulation gipfeln, verloren geht oder nur schwer zu interpretieren ist.
Die Autofluoreszenzbildgebung hat jedoch auch einige Einschränkungen. Viele der autofluoreszierenden Signale gehen bei der Zellfixierung verloren. Daher ist die Autofluoreszenzbildgebung weitgehend auf die Untersuchung lebender Zellen beschränkt. Die Autofluoreszenzbildgebung beruht auch auf einem Mangel an exogen eingeführten Fluorophoren, die in autofluoreszierende Kanäle bluten können. Obwohl viele Fluorophore in bestimmten Situationen mit der Autofluoreszenzbildgebung kompatibel sein können, ist die Kombination der Autofluoreszenzbildgebung mit vielen vorhandenen Werkzeugen nicht immer möglich. Die Bildgebung lebender Zellen kann auch Phototoxizität induzieren. Eine Iteration der Bildgebung mit diesem Protokoll induziert keine ausreichende Phototoxizität, um die Lebensfähigkeit von NSC zu verringern. Die wiederholte Bildgebung von Zellen in einem Zeitverlauf in häufigeren Abständen viele Male pro Tag kann jedoch zu einer signifikanten Phototoxizität führen und ist daher keine praktikable Strategie zur Untersuchung der NSC-Ruhe. Obwohl die Autofluoreszenzbildgebung verwendet werden kann, um NSCs von 6 Wochen alten männlichen Mäusen entweder aus dem Hippocampus oder den lateralen Ventrikeln zu untersuchen, ist unklar, wie breit diese Technik verwendet werden kann, um NSCs aus anderen Quellen, Altersgruppen oder anderen Stammzelltypen zu untersuchen.
Das hier beschriebene Protokoll geht auch von einer genauen Herstellung von qNSCs und aNSCs in vitro unter Verwendung der zuvor etablierten Protokolle 10,13,17,18,19 aus. Wenn die Reproduktion der Ergebnisse dieses Protokolls eine Herausforderung darstellt, stellen Sie sicher, dass qNSCs und aNSCs ordnungsgemäß generiert werden, indem Sie nach dem Vorhandensein oder Fehlen verschiedener Marker von qNSCs und aNSCs suchen, wie zuvor beschrieben 10,13,17,18,19. Zusammengenommen bietet die Autofluoreszenzbildgebung einen neuartigen technischen Ansatz zur Identifizierung von qNSCs und aNSCs und zur Untersuchung von NSC-Ruheausgängen.
Offenlegungen
Die Autoren erklären, dass keine konkurrierenden Interessen bestehen.
Danksagungen
Wir danken dem UW-Madison-Durchflusszytometrie-Kern (P30 CA014520 und 1S10RR025483-01) sowie den Mitgliedern des Moore-Labors und der UW-Madison-Community für ihren Beitrag. Wir danken unseren Finanzierungsquellen: NIH T32 T32GM008688 (an C.S.M.), Diana Jacobs Kalman Fellowship von AFAR (an C.S.M.), Wisconsin Graduate Fellowship (an C.S.M.), DP2 NIH New Innovator Award (1DP2OD025783, an D.L.M.), Vallee Scholar Award (an D.L.M.), NIH 1R56NS130450 (an D.L.M und M.C.S.), R01 CA185747 (an M.C.S.), R01 CA205101 (an M.C.S.), R01 CA211082 (an M.C.S.) und die National Science Foundation Grant No. CBET-1642287 (zu M.C.S.).
Materialien
| Name | Company | Catalog Number | Comments |
| 40x Water objective lens | Nikon | MRD77410 | Objective lens used in multiphoton microscope in Part 3 |
| 8 well cuvette | Ibidi | 80826-90 | For imaging aNSCs/qNSCs |
| Analog power meter | Thorlabs | PM100A | Used in multiphoton microscope in Part 3 |
| Antibiotic-Antimycotic (100X) (PSF) | Thermo Fisher | 15240062 | Antibiotic for NSC media |
| B-27 | Invitrogen | 17504044 | Nutrient supplement for NSC media |
| BMP4 | Fisher Scientific | 5020BP010 | Factor for inducing quiescence |
| Bovine serum albumin | Sigma | A4919-25G | For making BMP4 |
| Chameleon ultrafast laser | Coherent | N/A | Laser used in multiphoton microscope in Part 3 |
| Confocal microscope | Nikon | C2 | Microscope used for Part 1 |
| DMEM/F-12 (without GlutaMAX) | Invitrogen | 11320033 | Base media for NSCs |
| DNAse | Sigma | D5025-15KU | Added to trypsin inhibitor |
| EdU assay kit | Invitrogen | C10337 | Proliferation assay for cell culture |
| EGF | PeproTech | AF-100-15-500UG | Growth factor for NSC media |
| FGF | PeproTech | 100-18B | Growth factor for NSC media |
| Fluorescent activated cell sorter | BD | FACSAria | Fluorescent Activated Cell Sorter used for Part 2 |
| Heparin | Sigma | H3149-50KU | Additive for NSC media |
| L-15 | Invitrogen | 21083027 | For preparing trypsin inhibitor solution |
| Laminin | Sigma | L2020-1MG | For coating glassware |
| Nikon TiE inverted microscope | Nikon | N/A | Microscope frame Used for Part 3 |
| PLO | Sigma | P3655-100MG | For coating glassware |
| SPC-150 Single photon counting electronics | Becker and Hickl | N/A | Used in multiphoton microscope in Part 3 |
| Trypsin (for trypsinizing pellets of aNSCs that were growing as spheres or monolayers) | Gibco | 15090046 | For trypsinizing neurospheres or adherent aNSCs |
| Trypsin (for trypsinizing qNSCs) | Gibco | 25200072 | For trypsinizing adherent qNSCs |
| Trypsin inhibitor | Sigma | T6522-100MG | For inhibiting trypsinization of aNSCs |
| Urea crystals | Sigma | U5128-5G | Used to collect an IRF |
| Versene | Thermo Fisher | 15040066 | For preparing trypsin |
Referenzen
- Goncalves, J. T., Schafer, S. T., Gage, F. H. Adult neurogenesis in the hippocampus: From stem cells to behavior. Cell. 167 (4), 897-914 (2016).
- Kuhn, H. G., Dickinson-Anson, H., Gage, F. H. Neurogenesis in the dentate gyrus of the adult rat: age-related decrease of neuronal progenitor proliferation. J Neurosci. 16 (6), 2027-2033 (1996).
- Silva-Vargas, V., Maldonado-Soto, A. R., Mizrak, D., Codega, P., Doetsch, F. Age-dependent niche signals from the choroid plexus regulate adult neural stem cells. Cell Stem Cell. 19 (5), 643-652 (2016).
- Urban, N., Blomfield, I. M., Guillemot, F. Quiescence of adult mammalian neural stem cells: A highly regulated rest. Neuron. 104 (5), 834-848 (2019).
- Urban, N., et al. Return to quiescence of mouse neural stem cells by degradation of a proactivation protein. Science. 353 (6296), 292-295 (2016).
- Kalamakis, G., et al. Quiescence modulates stem cell maintenance and regenerative capacity in the aging brain. Cell. 176 (6), 1407-1419 (2019).
- Walsh, A. J., et al. Classification of T-cell activation via autofluorescence lifetime imaging. Nat Biomed Eng. 5 (1), 77-88 (2021).
- Sagar, M. A. K., et al. Microglia activation visualization via fluorescence lifetime imaging microscopy of intrinsically fluorescent metabolic cofactors. Neurophotonics. 7 (3), 035003 (2020).
- Knobloch, M., et al. Metabolic control of adult neural stem cell activity by Fasn-dependent lipogenesis. Nature. 493 (7431), 226-230 (2013).
- Knobloch, M., et al. A fatty acid oxidation-dependent metabolic shift regulates adult neural stem cell activity. Cell Rep. 20 (9), 2144-2155 (2017).
- Knobloch, M., et al. SPOT14-positive neural stem/progenitor cells in the hippocampus respond dynamically to neurogenic regulators. Stem Cell Reports. 3 (5), 735-742 (2014).
- Shin, J., et al. Single-cell RNA-Seq with waterfall reveals molecular cascades underlying adult neurogenesis. Cell Stem Cell. 17 (3), 360-372 (2015).
- Leeman, D. S., et al. Lysosome activation clears aggregates and enhances quiescent neural stem cell activation during aging. Science. 359 (6381), 1277-1283 (2018).
- Beckervordersandforth, R. Mitochondrial metabolism-mediated regulation of adult neurogenesis. Brain Plast. 3 (1), 73-87 (2017).
- Morrow, C. S., et al. Autofluorescence is a biomarker of neural stem cell activation state. Cell Stem Cell. , (2024).
- Kolenc, O. I., Quinn, K. P. Evaluating cell metabolism through autofluorescence imaging of NAD(P)H and FAD. Antioxid Redox Signal. 30 (6), 875-889 (2019).
- Mira, H., et al. Signaling through BMPR-IA regulates quiescence and long-term activity of neural stem cells in the adult hippocampus. Cell Stem Cell. 7 (1), 78-89 (2010).
- Morrow, C. S., et al. Vimentin coordinates protein turnover at the aggresome during neural stem cell quiescence exit. Cell Stem Cell. 26 (4), 558-568 (2020).
- Martynoga, B., et al. Epigenomic enhancer annotation reveals a key role for NFIX in neural stem cell quiescence. Genes Dev. 27 (16), 1769-1786 (2013).
- Bin Imtiaz, M. K., Jessberger, S. Isolation of adult mouse hippocampal neural stem cells for fluorescence loss in photobleaching assays. STAR Protoc. 2 (3), 100695 (2021).
- Guo, W., Patzlaff, N. E., Jobe, E. M., Zhao, X. Isolation of multipotent neural stem or progenitor cells from both the dentate gyrus and subventricular zone of a single adult mouse. Nat Protoc. 7 (11), 2005-2012 (2012).
- Lakowicz, J. R., Szmacinski, H., Nowaczyk, K., Johnson, M. L. Fluorescence lifetime imaging of free and protein-bound NADH. Proc Natl Acad Sci U S A. 89 (4), 1271-1275 (1992).
- Blacker, T. S., Duchen, M. R. Investigating mitochondrial redox state using NADH and NADPH autofluorescence. Free Radic Biol Med. 100, 53-65 (2016).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: fundamentals and advances in instrumentation, analysis, and applications. J Biomed Opt. 25 (7), 1-43 (2020).
- . ImageJ Available from: https://imagej.nih.gov/ij/ (2021)
- . Becker & Hichl Handbooks Available from: https://imagej.nih.gov/ij/ (2024)
- . GitHub Materials Available from: https://github.com/chrismorrow5/Neural-Stem-Cell-Autofluorescence-Analysis/ (2024)
- Codega, P., et al. Prospective identification and purification of quiescent adult neural stem cells from their in vivo niche. Neuron. 82 (3), 545-559 (2014).
- Llorens-Bobadilla, E., et al. Single-cell transcriptomics reveals a population of dormant neural stem cells that become activated upon brain injury. Cell Stem Cell. 17 (3), 329-340 (2015).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten