Method Article
Sondas de tensión de ADN para mapear las fuerzas transitorias del receptor de piconewton por las células inmunes
En este artículo
Resumen
Este documento describe un protocolo detallado para usar sondas de tensión basadas en ADN para obtener imágenes de las fuerzas receptoras aplicadas por las células inmunes. Este enfoque puede mapear las fuerzas del receptor >4.7pN en tiempo real y puede integrar fuerzas a lo largo del tiempo.
Resumen
Las fuerzas mecánicas transmitidas en la unión entre dos células vecinas y en la unión entre las células y la matriz extracelular son críticas para regular muchos procesos que van desde el desarrollo hasta la inmunología. Por lo tanto, desarrollar las herramientas para estudiar estas fuerzas a escala molecular es fundamental. Nuestro grupo desarrolló un conjunto de sensores de tensión molecular para cuantificar y visualizar las fuerzas generadas por las células y transmitidas a ligandos específicos. La clase más sensible de sensores de tensión molecular se compone de horquillas de bucle madre-bucle de ácido nucleico. Estos sensores utilizan pares fluoróforo-apagador para informar sobre la extensión mecánica y el despliegue de horquillas de ADN bajo fuerza. Un desafío con los sensores de tensión de horquilla de ADN es que son reversibles con un rápido repliegue de horquilla al finalizar la tensión y, por lo tanto, las fuerzas transitorias son difíciles de registrar. En este artículo, describimos los protocolos para preparar sensores de tensión de ADN que pueden "bloquearse" y evitar que se vuelvan a plegar para permitir el "almacenamiento" de información mecánica. Esto permite el registro de fuerzas piconewton altamente transitorias, que posteriormente pueden ser "borradas" mediante la adición de ácidos nucleicos complementarios que eliminan la cerradura. Esta capacidad de alternar entre el mapeo de tensión en tiempo real y el almacenamiento de información mecánica revela fuerzas débiles, de corta duración y menos abundantes, que son comúnmente empleadas por las células T como parte de sus funciones inmunes.
Introducción
Las células inmunes se defienden contra patógenos y células cancerosas rastreando y escaneando continuamente las superficies de las células diana en busca de antígenos, tachonando su superficie 1,2. El reconocimiento de antígenos se inicia tras la unión entre el receptor de células T (TCR) y el complejo MHC del complejo de histocompatibilidad péptido-mayor (pMHC) expresado en la superficie de las células diana. Debido a que el reconocimiento TCR-pMHC ocurre en la unión entre dos células móviles, durante mucho tiempo se ha sospechado que experimenta fuerzas mecánicas. Además, esto condujo al modelo mecanosensor de activación TCR, que sugiere que las fuerzas TCR contribuyen a su función 3,4. Para comprender cuándo, dónde y cómo las fuerzas mecánicas contribuyen a la función de las células T, es imperativo desarrollar herramientas para visualizar las fuerzas moleculares transmitidas por las células T. Tradicionalmente, métodos como la microscopía de fuerza de tracción (TFM) y las matrices de micropilares se utilizan para investigar las fuerzas celulares 5,6. Sin embargo, la sensibilidad a la fuerza de las matrices TFM y micropilares está en la escala nanonewton (nN) y, por lo tanto, a menudo es insuficiente para estudiar las fuerzas moleculares de piconewton (pN) transmitidas por los receptores celulares7. Para mejorar la fuerza y la resolución espacial para la detección, nuestro laboratorio fue pionero en el desarrollo de sondas de tensión molecular, que inicialmente se sintetizaron utilizando polímeros de polietilenglicol (PEG)7. Las sondas de tensión molecular están compuestas por un "resorte" molecular extensible (PEG, proteína, ADN) flanqueado por un fluoróforo y un quencher y están anclados en una superficie. Las fuerzas aplicadas al extremo de la sonda conducen a su extensión, separando el fluoróforo y el exprimidor, generando así una fuerte señal de fluorescencia (Figura 1A)8,9,10.
Durante la última década hemos desarrollado una biblioteca de diferentes clases de sondas de tensión molecular con elementos de resorte hechos de ácidos nucleicos11, proteínas10 y polímeros8. Entre estos, las sondas de tensión basadas en ADN proporcionan la mayor relación señal / ruido y la mayor sensibilidad de fuerza, que se sintoniza fácilmente desde unos pocos pN hasta ~ 20 pN11. Hemos utilizado estas sondas de tensión de ADN en tiempo real para estudiar las fuerzas moleculares generadas por muchos tipos de células diversas, incluidos fibroblastos, células cancerosas, plaquetas y células inmunes11,12,13. Este manuscrito describirá protocolos para sintetizar y ensamblar sondas de tensión de ADN en una superficie para mapear las fuerzas del receptor molecular con resolución de fuerza pN utilizando un microscopio de fluorescencia convencional. Si bien el procedimiento actual incluye modificaciones químicas al ácido nucleico para introducir el reportero fluorescente (Figura 1B), es importante tener en cuenta que muchos de los pasos de modificación y purificación pueden subcontratarse a compañías personalizadas de síntesis de ADN. Por lo tanto, la tecnología de sondas de tensión de ADN es fácil y accesible para las comunidades más amplias de biología celular y mecanobiología.
Brevemente, para ensamblar sensores de tensión de ADN, una horquilla de ADN se hibrida con una hebra de ligando fluorescente en un brazo y una hebra de anclaje de quencher en el otro brazo y luego se inmoviliza en un sustrato de vidrio (Figura 1C, tensión en tiempo real). En ausencia de fuerza mecánica, la horquilla se cierra y, por lo tanto, la fluorescencia se apaga. Sin embargo, cuando la fuerza mecánica aplicada es mayor que F1/2 (la fuerza en equilibrio que conduce a una probabilidad del 50% de despliegue), la horquilla se derrite mecánicamente y se genera una señal fluorescente.
Sobre la base del sensor de tensión de ADN en tiempo real, también describimos protocolos para mapear las fuerzas acumuladas, lo que es particularmente útil para estudiar las interacciones entre los receptores en las células inmunes y su ligando natural. Esto se debe a que los receptores inmunes a menudo muestran enlaces de corta duración 3,14. Las fuerzas acumuladas se visualizan utilizando una hebra de "bloqueo" que se une preferentemente a horquillas de ADN abiertas y permite el almacenamiento de señales de fluorescencia asociadas con eventos de tracción mecánica (Figura 1C, tensión bloqueada). La hebra de bloqueo está diseñada para unir un sitio de unión críptico que se expone al derretir mecánicamente la horquilla inducida mecánicamente y bloquear la horquilla en estado abierto bloqueando el plegado de la horquilla, almacenando así la señal de tensión y generando un mapa de tensión acumulada. Además, la hebra de bloqueo está diseñada con un punto de apoyo de ocho nucleótidos, que permite una reacción de desplazamiento de la hebra mediada por la puntera con su complemento completo, la hebra de "desbloqueo". Con la adición de la hebra de desbloqueo, la hebra de bloqueo atada se quita de la construcción de horquilla, borrando la señal de tensión almacenada y restableciendo la horquilla al estado en tiempo real.
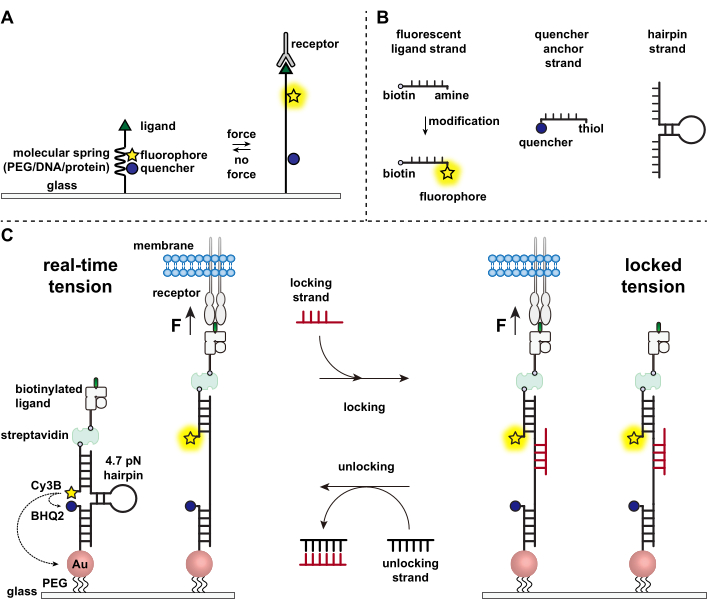
Figura 1: Esquema de las sondas de tensión molecular de última generación. (A) Diseño general de sondas de tensión molecular en tiempo real, (B) hebras para la construcción de la sonda de tensión basada en ADN, y (C) sondas de tensión basadas en ADN diseñadas y su alternancia entre el estado en tiempo real y el estado bloqueado. Haga clic aquí para ver una versión más grande de esta figura.
El protocolo principal consta de cuatro secciones principales: preparación de oligonucleótidos, preparación de superficies, imágenes y análisis de datos. Este protocolo ha sido demostrado con éxito por nuestro laboratorio y otros en células T OT-1 CD8 + ingenuas y activadas, células OT-II CD4 +, así como hibridomas, y se puede aplicar para interrogar diferentes receptores de células inmunes, incluidos el receptor de células T, el receptor de muerte celular programada (PD1) y las fuerzas del antígeno 1 asociado a la función de los linfocitos (LFA-1). Las células T OT-1 CD8 + no naïve se utilizan como una línea celular de ejemplo en este documento.
Protocolo
Los ratones transgénicos OT-1 se encuentran en la División de Recursos Animales de la Universidad de Emory. Todos los experimentos fueron aprobados y realizados bajo el protocolo del Comité Institucional de Cuidado y Uso de Animales (IACUC).
1. Preparación de oligonucleótidos
- Disuelva el ADN de la hebra de ligando en agua (resistividad de 18,2 MΩ, utilizada en todo el protocolo). Vortex y gire hacia abajo la solución con una centrífuga de mesa. Ajuste el volumen de agua de tal manera que la concentración final sea de 1 mM. Validar la concentración mediante el uso de un espectrofotómetro de nanogotas para medir la absorbancia a 260 nm y determinar la concentración final en función del coeficiente de extinción del oligonucleótido.
NOTA: La hebra del ligando tiene una modificación en cada extremo, 5' amina y 3' biotina, para conjugarse con el fluoróforo y presentar el ligando biotinilado. El grupo amina en la hebra del ligando debe conjugarse con un fluoróforo. El colorante Cy3B se utiliza para esta conjugación debido a su alto brillo y fotoestabilidad, pero generalmente no se ofrece comercialmente y requiere conjugación interna. En consecuencia, la siguiente sección describe la conjugación entre aminas y colorantes éster NHS. Para los usuarios finales que no tienen acceso a instalaciones o recursos para la modificación de ácidos nucleicos, los ácidos nucleicos modificados se pueden comprar a proveedores de síntesis de ADN personalizados que ofrecen tintes brillantes y fotoestables, como la familia de tintes Alexa y Atto. - Prepare soluciones 10x PBS y 1 M NaHCO3 . Mezclar 10 μL de la solución de ligando de amina de 1 mM (10 nmol) con 10 μL de 10x PBS, 10 μL de 1 M NaHCO3 y 60 μL deH2O. Disolver 50 μg de éster Cy3B NHS en 10 μL de DMSO inmediatamente antes de usar y añadir a la mezcla para obtener un volumen de reacción total de 100 μL. Añadir Cy3B NHS éster al final. Dejar reaccionar a temperatura ambiente durante 1 h o 4 °C durante la noche.
- Prepare la hebra de bloqueo Atto647N conjugando la hebra de bloqueo de amina con el éster Atto647N NHS. Prepare 10x PBS y 1 M NaHCO3 solución. Mezclar 10 μL de la solución de cadena bloqueante de amina de 1 mM (10 nmol) con 10 μL de 10x PBS, 10 μL de 1 M NaHCO3 y 60 μL deH2O. Disolver 50 μg de éster Atto647N NHS en 10 μL de DMSO inmediatamente antes de su uso y añadir a la mezcla para obtener un volumen de reacción total de 100 μL. Añadir Atto647N NHS éster al final. Dejar reaccionar a temperatura ambiente durante 1 h o 4 °C durante la noche.
- Después de las reacciones, elimine los subproductos, el exceso de colorante y las sales mediante filtración de gel desalinizante P2. Diluir la mezcla de reacción conH2Ohasta un volumen total de 300 μL, que sea apropiado para la siguiente etapa de purificación por HPLC. Agregue 650 μL de gel P2 hidratado a un dispositivo centrífugo y gire hacia abajo a 18,000 x g durante 1 minuto. Retire el líquido en la parte inferior del dispositivo, agregue la mezcla de reacción a la columna que contiene gel P2, gire hacia abajo a 18,000 x g durante 1 minuto y recoja la mezcla de reacción en la parte inferior del dispositivo.
NOTA: P2 gel debe hidratarse al menos 4 h antes de usar conH2O. - Purificar la mezcla de reacción desalinizada con HPLC utilizando una columna C18 designada para la purificación de oligonucleótidos, con disolvente A: 0,1 M TEAA enH2Oy B:ACN como fase móvil para una elución de gradiente lineal 10-100% B durante 50 min a un caudal de 0,5 mL/min. Inyectar la mezcla de reacción desalinizada en HPLC de fase inversa con un bucle de inyección de 500 μL para su purificación. Recoja el producto que tiene un pico de absorbancia para el ADN (260 nm) y un pico de absorbancia para el fluoróforo (560 nm para Cy3B y 647 nm para Atto647N) y séquelos en un concentrador centrífugo de vacío durante la noche (véase la figura 2A).
- Reconstituir el producto oligocolorante seco en 100 μL de agua. Determine la concentración de la hebra de ligando Cy3B y la hebra de bloqueo Atto647N con el espectrofotómetro de nanogota. Asegúrese de que la proporción de etiquetado del tinte sea cercana a 1: 1. Corrija la absorbancia de 260 nm del colorante si es necesario al determinar la concentración de oligonucleótidos.
- Validar el producto purificado con MALDI-TOF-MS utilizando 3-HPA como sustrato en 50% ACN/H2Ocon 0,1% TFA y 5 mg/ml de citrato de amonio utilizando 0,5 μL del producto a 1-5 μM para la preparación de muestras MALDI-TOF-MS. Un ejemplo de espectro de masas se puede encontrar en la Figura 2B.
- Disuelva la horquilla y la hebra de anclaje en agua y asegúrese de que la concentración de las soluciones madre esté entre 50 y 100 μM.
NOTA: La horquilla no está modificada y se puede sintetizar directamente de forma personalizada de un proveedor. La hebra de anclaje tiene un grupo de anclaje de tiol y un quencher BHQ2 y se puede sintetizar directamente de forma personalizada de un proveedor. - Alícuota todos los oligonucleótidos. Para uso y almacenamiento a corto plazo, almacenar estos oligonucleótidos a 4 °C. Para el almacenamiento a largo plazo, congelarlos y mantenerlos a -20 °C. En este punto, todos los oligonucleótidos están listos para el ensamblaje de la sonda de tensión de ADN.
NOTA: Los ciclos repetidos de congelación-descongelación no son problemáticos para los oligonucleótidos.
2. Preparación de la superficie
NOTA: La preparación de sustratos de sonda de tensión de horquilla de ADN lleva dos días. La sonda de tensión de horquilla de ADN se funcionalizará en cubreobjetos de vidrio.
- Día 1
- Coloque los cubreobjetos de 25 mm en una rejilla de politetrafluoroetileno en un vaso de precipitados de 50 ml. Cada bastidor puede contener hasta 8 cubreobjetos. Enjuague los cubreobjetos sumergiéndolos en agua tres veces.
- Agregue 40 ml de una solución de etanol mezclado con agua al vaso de precipitados que contiene la rejilla y los cubreobjetos, y selle el vaso de precipitados con una película de parafina.
- Sonicar el vaso de precipitados durante 15 minutos en un limpiador de ultrasonidos (frecuencia de funcionamiento de 35 KHz) para limpiar los cubreobjetos. Después de la sonicación, deseche el líquido y enjuague el vaso de precipitados con la rejilla y cubra con agua al menos 6 veces para eliminar cualquier solvente orgánico restante.
- Prepare una solución fresca de piraña mezclando ácido sulfúrico y peróxido de hidrógeno en una proporción de 3: 1. Para hacer 40 ml de solución de piraña , agregue 30 ml de ácido sulfúrico a un vaso de precipitados limpio de 50 ml primero y luego agregue lentamente 10 ml deH2O2. La solución de piraña se calentará rápidamente y burbujeará al agregar elH2O2. Mezclar suavemente la piraña con el extremo de una pipeta de vidrio.
- A continuación, transfiera la rejilla que sujeta los cubreobjetos al vaso de precipitados que contiene una solución de piraña suavemente mezclada para grabado (Figura 3A). Deje que la solución de piraña hidroxile y limpie los cubreobjetos durante 30 minutos a temperatura ambiente. Después del grabado de piraña, transfiera la rejilla con pinzas de acero o politetrafluoroetileno a un vaso de precipitados limpio de 50 ml con agua y enjuague nuevamente con agua al menos 6 veces.
PRECAUCIÓN: Grandes cantidades de sustancias orgánicas podrían reaccionar vigorosamente con la solución de piraña y pueden causar una explosión. Tenga cuidado y siempre trabaje con la solución Piranha en una campana extractora. Asegúrese de usar una bata de laboratorio, guantes y gafas de seguridad. Nunca guarde la solución fresca de piraña en un recipiente sellado.
NOTA: La proporción de peróxido de hidrógeno a ácido sulfúrico debe mantenerse por debajo de 1: 2 (v: v) y nunca debe exceder 1: 1. Al sumergir la rejilla con cubreobjetos en solución Piranha, colóquelos en la solución lenta y cuidadosamente. No deseche la solución inmediatamente después del grabado, ya que todavía está activa y caliente. Déjelo en el vaso de precipitados durante la noche antes de verterlo en el recipiente de desechos ácidos. - Sumerja la rejilla que sostiene los cubreobjetos en un vaso de precipitados de 50 ml con 40 ml de etanol para eliminar el agua. Deseche el etanol y repita 3 veces para asegurarse de que el agua haya sido eliminada.
- Luego sumerja la rejilla en aminopropil trietoxisilano al 3% (APTES) (v / v) en 40 ml de etanol para reaccionar con el -OH en los cubreobjetos durante 1 h a temperatura ambiente (Figura 3B).
NOTA: El etanol puede ser reemplazado por acetona. - Enjuague las superficies 6 veces sumergiéndolas en 40 ml de etanol, luego séquelas en el horno a 80 °C durante 20 minutos. Después del enfriamiento, guarde los cubreobjetos modificados con aminas secas a -20 °C para su uso futuro (hasta 6 meses).
- Cubra la parte inferior interna de las placas de Petri de plástico de 10 cm de diámetro con película de parafina. La película de parafina evita que los cubreobjetos se deslicen dentro de la placa de Petri y ayuda a mantener la solución para los próximos pasos de funcionalización en los cubreobjetos. Coloque los cubreobjetos modificados con aminas enfriadas en las placas de Petri. El lado a funcionalizar debe estar mirando hacia arriba.
- Para modificar los grupos amina en los cubreobjetos, añadir 300 μL de 0,5% p/v de ácido lipoico PEG NHS (LA-PEG-SC) y 2,5% p/v mPEG NHS (mPEG-SC) en 0,1 M NaHCO3 en cada cubreobjetos e incubar durante 1 h a temperatura ambiente (Figura 3C). Por cada cubreobjetos de 25 mm, pesar 1,5 mg de LA-PEG-SC y 7,5 mg de mPEG-SC. Disuelva los reactivos NHS inmediatamente antes de agregarlos a las superficies, ya que tienen una vida media corta (~ 10 min) en solución acuosa a temperatura ambiente. Después de la reacción, enjuague las superficies 3 veces con agua.
NOTA: La reacción del NHS se puede realizar a 4 °C durante la noche. Los reactivos del NHS tienen una vida media más larga antes de la hidrólisis a 4 °C, que es alrededor de 4-6 h. Esto dará como resultado un procedimiento de preparación de superficie de tres días. - Añadir 100 μL de NaHCO3 0,1 M que contenga 1 mg/ml de acetato de sulfo-NHS a un conjunto de cubreobjetos "sándwich" (dos cubreobjetos uno frente al otro con tampón de reacción en el medio). Permita que la pasivación ocurra durante al menos 30 minutos. Para ahorrar reactivo, este paso podría realizarse con 50 μL de acetato de sulfo-NHS de 1 mg/ml. Enjuague con agua tres veces después de la pasivación.
- Agregue 0.5 ml de nanopartículas de oro (AuNP, 8.8 nm, ácido tánico, 0.05 mg / ml) a cada cubreobjetos e incube durante 30 minutos a temperatura ambiente (Figura 3D). Para guardar el reactivo, este paso se puede hacer intercalando dos cubreobjetos también. Asegúrese de que no haya sales presentes en el sistema de los pasos anteriores para evitar la agregación de nanopartículas de oro. No deje que los cubreobjetos se sequen después de este paso.
- Mientras tanto, la horquilla prehibridada de 4.7 pN, la hebra de ligando Cy3B y la hebra de anclaje BHQ2 que forman las sondas de tensión de ADN se construyen en una proporción de 1.1: 1: 1 en 1 M NaCl a 300 nM en un tubo de PCR. Analice las hebras calentando la solución hasta 95 °C durante 5 min, luego enfríe gradualmente disminuyendo la temperatura a 20 °C durante 30 min en un termociclador.
- Enjuague los cubreobjetos con agua tres veces después de 30 minutos de incubación con nanopartículas de oro. Agregue una hebra de anclaje BHQ2 adicional (de 100 μM) a la solución de ADN recocido para hacer la relación entre la hebra de anclaje BHQ2 y la hebra de ligando Cy3B 10: 1. En este punto, la solución de ADN debe contener 300 nM de construcción de sonda de tensión y 2,7 μM de hebra BHQ2. Agregue 100 μL por dos cubreobjetos para hacer el "sándwich" (Figura 3E).
- Coloque cuidadosamente una bola de tejido de laboratorio húmeda en la placa de Petri (lejos de cubreobjetos) y selle el plato con una película de parafina para evitar que la solución se seque. Cubrir el plato con papel de aluminio e incubar a 4 °C durante la noche.
- Día 2
- Lave el exceso de sondas de los cubreobjetos con 1x PBS. Verifique la calidad de la superficie de la sonda de tensión de ADN bajo un microscopio epifluorescente.
- Preparar 40 μg/ml de estreptavidina en 1x PBS e incubar en cubreobjetos durante 30 min a temperatura ambiente (Figura 3F). Por lo general, 100 μL es suficiente para un cubreobjetos de 25 mm. Enjuague con PBS 3 veces después de la incubación para eliminar la cantidad excesiva de estreptavidina.
- Preparar 40 μg/ml de anticuerpo/ligando biotinilado en 1x PBS. Añadir 50-100 μL por sándwich e incubar durante 30 min a temperatura ambiente (Figura 3G). Enjuague con PBS tres veces después de la incubación para eliminar la cantidad excesiva de anticuerpo/ligando biotinilado.
- Ensamble cuidadosamente las cámaras de imágenes limpias con superficies. Las superficies se pueden agrietar fácilmente al apretar las cámaras. Agregue 0.5-1 ml de solución salina balanceada de Hank (HBSS) a las cámaras de imágenes y manténgalas listas para obtener imágenes con células (Figura 3H).
3. Obtención de imágenes de las fuerzas del receptor celular
- Preparar las células inmunes de interés en HBSS a 1-2 x 106 células/ml.
NOTA: Las células OT-1 CD8+ naïve se utilizan como ejemplo en este documento. Purificar las células T no infectadas con CD8+ OT-1 del bazo de ratones sacrificados utilizando el kit de aislamiento de células T CD8+ de ratón MACS con un separador MACS siguiendo las instrucciones del fabricante. Aísle y enriquezca las células T CD8 + eliminando cualquier célula T no CD8 + a la que se unieron los anticuerpos que agotan magnéticamente. Resuspenda las células T purificadas OT-1 CD8 + no tratadas con tratamiento previo en HBSS a 2 x 106 células/ml y manténgalas en hielo antes de usarlas. - Verifique la calidad de la superficie de la sonda de tensión de horquilla de ADN bajo un microscopio de fluorescencia (objetivo 100x) para el control de calidad antes de agregar ligandos o células de recubrimiento. Obtener una imagen y cuantificar la intensidad de fondo promedio en el canal Cy3B de una superficie de sonda de tensión de horquilla de ADN desde al menos 5 posiciones diferentes y 3 réplicas. Mantenga las condiciones de adquisición de imágenes consistentes para que este valor pueda usarse como un marcador confiable de la calidad de la superficie y la densidad de la sonda (Figura 4C).
NOTA: Cuantificar el número de hebras de ADN por nanopartícula de oro y el número de nanopartículas de oro por μm2 las primeras veces de preparación de la superficie de acuerdo con la literatura12, que puede usarse como otro marcador confiable de la calidad de la superficie. - Placa ~4 x 10 4 - 10 x 104 celdas en cada cubreobjetos funcionalizados de la sonda de tensión de ADN y permite que se unan y se extiendan durante ~15 min a temperatura ambiente.
- A medida que las células se colocan en las sondas de tensión de horquilla de ADN y comienzan a propagarse, obtenga una imagen de las señales de fluorescencia que se generan en el canal Cy3B con el objetivo 100x (Figura 3I).
- Después de que las células comiencen a producir una señal de tensión en tiempo real en la superficie de la sonda de tensión de horquilla de ADN en el canal Cy3B, adquiera imágenes en los canales Cy3B y Atto647N (la microscopía TIRF proporciona una mejor relación señal-ruido que la epifluorescencia). Posteriormente, agregue la hebra Atto647N a las cámaras de imagen a una concentración final de 200 nM para la hibridación mecánicamente selectiva.
- Después de 10 minutos de incubación, retire rápida y suavemente el tampón que contiene la hebra de bloqueo fluorescente Atto647N y reemplácela con sales frescas balanceadas de Hank. Imagen en los canales Cy3B y Atto647N de nuevo y determinar el coeficiente de correlación de Pearson con el software Fiji15.
- En el momento de interés para la investigación, introducir una hebra de bloqueo no fluorescente a las células en la cámara de imágenes para almacenar la señal de tensión. Preparar el material de bloqueo de la hebra (100 μM) y añadir a las células a una concentración final de 1 μM. Pipetear suavemente para mezclar. La duración del bloqueo puede variar, pero 10 minutos es el tiempo recomendado.
- Adquiera películas de lapso de tiempo o imágenes de punto final en epifluorescencia tanto para mapeo de tensión cualitativo como para análisis cuantitativo según sea necesario (Figura 3J y Figura 5).
NOTA: Si se desea medir la tensión en varios puntos de tiempo, inicie el borrado de las señales de tensión almacenadas mediante la adición de una hebra de desbloqueo. Para evitar el enjuague excesivo, se utiliza una mayor concentración final de hebra de desbloqueo a 2 μM para iniciar una reacción de desplazamiento de la hebra mediada por la puntera con la hebra de bloqueo durante 3 minutos, lo que borra las señales almacenadas (Figura 3J). Enjuague suavemente el exceso de oligonucleótidos con HBSS. La superficie de la sonda de tensión de horquilla de ADN y las células están listas para otra ronda de almacenamiento y mapeo de tensión. El desbloqueo de las señales de tensión no es necesario si solo un punto de tiempo es de interés en el estudio.
4. Análisis de datos
NOTA: El análisis de imágenes se realiza utilizando el software Fiji, y el análisis cuantitativo se realiza utilizando el software de análisis.
- Corrija cualquier desviación durante la adquisición de imágenes con el comando Corregir desviación 3D en Registro en el menú Plugins .
- Elimine el fondo de la cámara de la imagen con el comando Restar en el menú Proceso .
- Determine el coeficiente de correlación de Pearson con la función Colocalización en el menú Analizar .
- Promediar y restar el fondo de fluorescencia producido por las sondas no abiertas de tres regiones de fondo locales diferentes. Dibuje ROI de celdas en imágenes sustraídas al fondo o imágenes RICM (microscopía de contraste de interferencia de reflexión) con la herramienta Image J Freehand Selections . Mida cualquier métrica de interés de los ROI, por ejemplo, la intensidad de fluorescencia integrada y la ocupación de la tensión utilizando la herramienta Medir en el menú Analizar (Figura 6).
- Exporte las mediciones para el análisis cuantitativo con el software de análisis.
- Trazar los datos con cualquier software de análisis.
Resultados
Aquí mostramos imágenes representativas del control de calidad de la superficie (Figura 4). Una superficie de alta calidad debe tener un fondo limpio en el canal RICM (Figura 4B) y una intensidad de fluorescencia uniforme en el canal Cy3B (Figura 4C). Con el mismo equipo de imágenes y condiciones idénticas de adquisición de imágenes de fluorescencia, la intensidad de fluorescencia de fondo debe ser consistente y reproducible cada vez que se realicen experimentos con sondas de ADN. Recomendamos usarlo como marcador para el control de calidad de la superficie antes de cada conjunto de experimentos individuales.
En este trabajo mostramos algunos datos representativos (Figura 5) con células CD8+ no tratadas con OT-1 como tipo de célula modelo, que reconocen específicamente la ovoalbúmina de pollo. El anticuerpo CD3ε se incluye como control positivo para el sistema y el antígeno afín pMHC N4 (SIINFEKL) se utiliza como ejemplo. Las células se colocaron en sustratos que se funcionalizaron con sondas de horquilla de ADN de 4,7 pN que presentaban antiCD3ε o pMHC N4. Después de 30 min, se capturó la dispersión celular en RICM y la tensión en tiempo real en el canal Cy3B, y la hebra de bloqueo se introdujo a una concentración final de 1 μM. El lapso de tiempo de bloqueo de tensión mostró que las señales de tensión TCR-antiCD3ε y TCR-pMHC se amplificaron debido al bloqueo de horquilla y la acumulación de señal. Además, la interacción TCR-pMHC N4 mostró una señal de fuerza débil en tiempo real (0 min), probablemente debido a su corta vida útil de enlace. La hebra de bloqueo registró estos eventos de tracción mecánica de corta duración, lo que facilita el análisis cuantitativo. Además, el bloqueo reveló el patrón de la fuerza TCR-pMHC a medida que se acumula, que era bastante distinto del control antiCD3ε y se parecía al patrón de "ojo de buey" de las sinapsis inmunes. La cuantificación de la señal de fluorescencia se describe en el paso 4.4. Por lo general, utilizamos la ocupación de la tensión (definida como el porcentaje de área que tiene señal de tensión sobre el área de contacto) y la intensidad de fluorescencia integrada de cada celda como métrica para evaluar la tensión acumulada que es > 4.7 pN.

Figura 2: Ejemplos de preparación de oligonucleótidos. (A) Cromatograma HPLC de purificación de la hebra del ligando Cy3B; (B) espectros MALDI-TOF-MS del producto; (C) Tabla de la masa calculada y los picos m/z encontrados del material de partida y el oligonucleótido marcado. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Funcionalización de los sustratos de la sonda de tensión de ADN y procedimientos experimentales. Los pasos se describen en el protocolo correspondiente. Haga clic aquí para ver una versión más grande de esta figura.
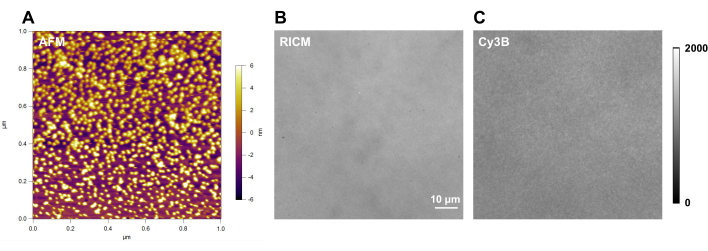
Figura 4: Ejemplo de superficie de sonda de tensión de ADN con buena calidad. (A) caracterización por microscopía de fuerza atómica (AFM) de la superficie de la sonda de tensión de ADN; e imágenes de microscopía en bruto de una superficie de sonda de tensión de ADN en (B) RICM y (C) canal Cy3B. Haga clic aquí para ver una versión más grande de esta figura.
Figura 5: Ejemplo de imágenes de microscopía en bruto de un experimento exitoso. Las imágenes muestran células CD8+ naïve OT-1 que producen fuerzas TCR contra (A) antiCD3ε y (B) pMHC N4. Barra de escala = 5 μm. Haga clic aquí para ver una versión más grande de esta figura.

Figura 6: Ejemplos de procesamiento de datos y análisis cuantitativo. Haga clic aquí para ver una versión más grande de esta figura.
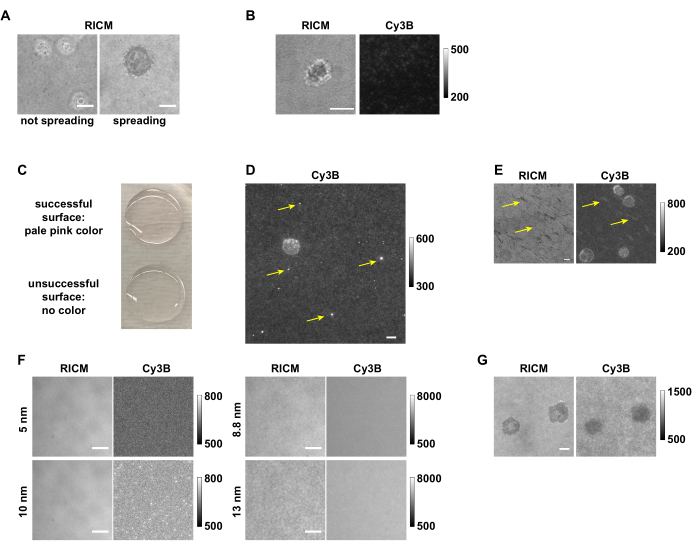
Figura 7: Ejemplos de superficies exitosas y no exitosas y mapeo de tensión. (A) Células que no se diseminan en comparación con las células que se diseminan en las superficies pMHC N4 en RICM. (B) Célula OT-1 que se diseminó pero no generó ninguna señal de tensión en una superficie antiCD3ε con baja densidad de sonda de tensión de ADN. (C) Imagen que muestra que la superficie exitosa es de color rosa pálido, y la superficie fallida es incolora. (D) Imagen en el canal Cy3B que muestra los agregados fluorescentes de un antiguo stock de ADN. (E) El patrón en el canal RICM y el canal Cy3B que probablemente se deba a un lavado insuficiente o al secado accidental de la superficie entre pasos. (F) Imágenes en los canales RICM y Cy3B que muestran el efecto del uso de NPs Au de diferentes tamaños. (G) Imágenes RICM y Cy3B de células OT-1 que no ejercieron señales de tensión, sino que agotaron el ADN. Barra de escala = 5 μm. Tenga en cuenta que los datos sin procesar que se muestran aquí fueron recopilados por diferentes miembros del grupo con diferentes configuraciones de adquisición de imágenes de 3 microscopios, por lo que tienen diferentes niveles de fondo de fluorescencia. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Con los procedimientos detallados proporcionados aquí, se pueden preparar sustratos de sonda de tensión de horquilla de ADN para mapear y cuantificar la tensión del receptor producida por las células inmunes. Cuando las células se colocan en el sustrato de la sonda de tensión de horquilla de ADN, aterrizan, se unen y se propagan a medida que los receptores detectan los ligandos tanto química como mecánicamente, el último de los cuales es detectado por nuestras sondas. Sin embargo, en algunos casos las células pueden no propagarse (Figura 7A) o no producen señal de tensión. Esto es a menudo una consecuencia de problemas químicos de superficie y requiere solución de problemas (Cuadro 1). La baja densidad de ligandos es uno de los problemas más comunes que resulta en una falla de propagación celular. Esto podría ser el resultado de múltiples factores, como ligandos degradados, cadenas de ADN degradadas, reactivos hidrolizados como APTES y / o LA-PEG-NHS, nanopartículas de oro agregadas o limpieza insuficiente del cubreobjetos de vidrio. Al comparar la intensidad de fluorescencia de fondo del sustrato en experimentos exitosos y fallidos (Figura 7B), se puede evaluar rápidamente el origen del problema. Por ejemplo, las superficies con una fuerte señal de fluorescencia de las mediciones de fondo indican que la inmovilización de las sondas de ADN fue exitosa. La baja intensidad de fondo sugiere problemas en los pasos de preparación previos a la adición de la estreptavidina y el ligando biotinilado. Si el problema es causado por el ligando, su calidad se puede verificar recubriéndolo en un portaobjetos de vidrio o pocillos en una placa de 96 pocillos a 10 μg / ml para probar la adhesión celular. Esta es una prueba funcional útil de la actividad del ligando para ligandos pMHC y anticuerpos anti-CD3. Al observar el cambio de color (rosa pálido) de los cubreobjetos funcionalizados después de la etapa de incubación Au NP, se puede evaluar si el problema es causado por la degradación del ADN o los pasos previos a la incubación con ADN (Figura 7C). La calidad de la cadena de ADN se puede validar con HPLC y MALDI-TOF-MS, en la que una sola muestra de ADN mostrará múltiples picos si se degrada. La calidad de los NP Au se puede confirmar comparando los espectros UV-Vis y las imágenes TEM con la caracterización proporcionada por el fabricante. Si se valida la calidad del ligando, las cadenas de ADN y Au NP y estos reactivos no causan el problema de la superficie, recomendamos reemplazar los reactivos que incluyen APTES, LA-PEG-NHS, ácido sulfúrico y H.2O2 para la preparación futura de la superficie en lugar de perder tiempo para localizar con precisión la causa del problema de química de la superficie. Excepto por el problema de baja densidad que causará una falla importante del sustrato de la sonda de ADN, hay algunos problemas menores que podrían afectar la calidad de los datos. Dado que la preparación de la superficie tiene múltiples pasos de incubación, los tampones utilizados durante la preparación de la superficie deben hacerse frescos, filtrarse y mantenerse a 4 ° C para su almacenamiento para evitar la contaminación. Si una superficie está contaminada, será visible en el canal RICM y puede dar lugar a un fondo de fluorescencia extraño. Además de la contaminación, los agregados pueden formarse en la solución de ADN con el tiempo, lo que puede dar lugar a puntos brillantes en el canal Cy3B (Figura 7D). Por lo general, unos pocos agregados no afectarán la calidad de los datos; Sin embargo, si una célula aterriza en regiones con tales defectos, el análisis de datos es menos confiable. Ocasionalmente, se pueden observar patrones irregulares que afectan el mapeo de tensión tanto en los canales RICM como en los Cy3B, que podrían provenir de un lavado insuficiente después del paso APTES, o un secado accidental de la superficie durante los pasos después de que las partículas de oro se funcionalizan en la superficie (Figura 7E). Estos patrones pueden afectar el mapeo de tensión dependiendo de su gravedad. Para ahorrar reactivos relativamente valiosos, como anticuerpos o pMHC, recomendamos comprobar siempre la calidad de la superficie (ver 2.2.1 y 3.3) antes de la funcionalización de las sondas de tensión de horquilla de ADN con anticuerpo/ligando. Aunque este protocolo de preparación de superficies ha sido robusto y confiable en nuestras manos, tenga en cuenta que no siempre tolera muy bien las alteraciones en ciertos pasos. Por ejemplo, cuando se utiliza grabado base y plasma en lugar de grabado de piraña, es muy probable que la densidad de la sonda disminuya y haya más defectos superficiales. Las alteraciones del tamaño de Au NP y los reactivos de tapado probablemente afectarán dramáticamente los resultados de funcionalización de la superficie (Figura 7F). Por ejemplo, hemos encontrado que los NP de Au de 5 y 10 nm no se unen muy bien a las superficies modificadas con ácido lipoico, lo que resulta en un ADN mínimo después de la preparación de la superficie. Sin embargo, las NP Au de 8,8 nm y 13 nm pueden unirse a las superficies de ácido lipoico a una densidad mucho más alta, lo que se ha observado en RICM (rugosidad) y canal Cy3B (intensidad de fluorescencia).
Una vez que se preparan las superficies de la sonda de tensión de horquilla de ADN de buena calidad, se pueden realizar experimentos con células de interés y adquirir datos con un microscopio de fluorescencia. La tensión en tiempo real se puede capturar con un microscopio de fluorescencia ordinario equipado con un objetivo de alta NA (apertura numérica) y EMCCD. Sin embargo, tenga en cuenta que hemos observado que las células T primarias ocasionalmente muestran artefactos en las superficies de la sonda de tensión de ADN debido a las condiciones particulares de ese ratón. Como anécdota, es más probable que tales artefactos ocurran con ratones más viejos. Por ejemplo, hemos observado un agotamiento negativo de las señales de fluorescencia en lugar de señales de fluorescencia activadas (Figura 7G). En este caso, termine el experimento y rehaga con un nuevo lote de células. Antes de utilizar una hebra de bloqueo no fluorescente para el análisis cuantitativo de las fuerzas del receptor, el almacenamiento y borrado de la señal de tensión debe confirmarse con una hebra de bloqueo fluorescente Atto647N. Después del bloqueo inicial durante 10 minutos, la señal Atto647N debe estar fuertemente colocalizada con la señal Cy3B, ya que refleja el historial de tensión durante esta incubación. Como la señal Cy3B refleja no solo el historial de tensión sino también las fuerzas en tiempo real después de eliminar la cadena de bloqueo, es posible que un pequeño subconjunto de la señal Cy3B no esté acompañado por la señal Atto647N (Figura 3J). Para un análisis más cuantitativo de la tensión, recomendamos utilizar una hebra de bloqueo no fluorescente que eliminaría la transferencia potencial de energía entre el Cy3B y el Atto647N, así como evitar el exceso de lavados entre las adquisiciones de imágenes. Tenga en cuenta que la adición de la hebra de bloqueo inevitablemente causará un ligero aumento del fondo de fluorescencia, ya que la termodinámica impulsará una hibridación menor entre la horquilla y la hebra de bloqueo, que se puede restar antes de la cuantificación. Se puede cuantificar la intensidad integrada de cada célula después del procedimiento de bloqueo para estudiar la fuerza generada por un determinado receptor. Además, la cinética de bloqueo probablemente refleja el 2D k on y koff de un receptor a su ligando.
| Problema | Posible razón | Solución |
| Las superficies no son rosadas después de la incubación de AuNP | Grabado insuficiente | Incluya un paso de grabado base después del grabado de pirañas. Cubreobjetos de grabado en 0,5 M KOH en baño de hielo durante 1 h con sonicación. |
| APTES es hidrolizado | Usar el nuevo APTES | |
| Los reactivos del NHS se hidrolizan | Utilice nuevos reactivos PEG-NHS | |
| Los reactivos PEG-NHS se hidrolizan antes de añadirse a las superficies | Trabaja más rápido | |
| Au NP ha agregado | Usar el nuevo Au NP | |
| Residuos de agua en los cubreobjetos diluidos en el PN Au | Asegúrese de que no haya o haya poco residuo de agua antes de agregar Au NP | |
| Las células no se propagan en la superficie | Baja densidad de sonda de ADN | Si las superficies no son tan rosadas, solucione los problemas descritos anteriormente. Si la funcionalización de Au NP es normal, sintetizar y usar ADN nuevo |
| Baja densidad de ligandos | Usar nuevos reactivos de estreptavidina y ligando | |
| Las células no se propagan sobre el ligando | Verifique la propagación celular en una superficie recubierta de ligando, si las células aún no se diseminan, incluya moléculas adheridas como se describe en ref.12. | |
| Las células se diseminan bien, pero no se observa ninguna señal de tensión o solo una señal de tensión muy débil incluso con control de anticuerpos | La interacción receptor-ligando no tiene transmisión forzada | NA |
| La fuerza es de corta duración o las señales de fuerza son escasas | Usar oligonucleótido de bloqueo | |
| La superficie no se pasiva bien | Aumentar el conc. de acetato de sulfo-NHS, hasta 10 mg / ml | |
| No se observa ninguna señal de bloqueo en Atto647N | El oligonucleótido se ha degradado | Consulte con MALDI-TOF-MS |
| La señal de bloqueo es antilocalizada con sonda en tiempo real | La fuerza del receptor es mayor que el rango de trabajo del nucleótido de bloqueo | Utilice la sonda de horquilla en tiempo real de 19 pN de la ref. 12 como se describe. |
Tabla 1: Resolución de problemas con el sistema de sonda de tensión basado en ADN.
La aplicación del almacenamiento mecánico de información es particularmente útil para mapear las fuerzas receptoras generadas por las células inmunes, que a menudo son transitorias y menos abundantes. Según lo determinado por las mediciones de espectroscopia de una sola molécula, la vida útil máxima del enlace se observa con la aplicación de ~ 10 pN de fuerza con una vida útil que varía de menos de 100 milisegundos a varios segundos de duración. Estos enlaces de corta duración son difíciles de capturar con imágenes convencionales de sonda de tensión de horquillade ADN 15. Otro uso de este sistema de almacenamiento mecánico es cuando la densidad del receptor de interés es relativamente baja en la superficie celular16, lo que resulta en una señal dispersa que a menudo es difícil de distinguir del fondo con las sondas de tensión de horquilla de ADN convencionales. Tales fuerzas receptoras de baja densidad pueden revelarse mediante el mapeo de tensión acumulada con esta estrategia. Tenga en cuenta que limitamos específicamente la aplicación de esta estrategia a las células inmunes, ya que se sabe que las fuerzas presentes en las células inmunes están en el régimen inferior (TCR < 19 pN) en comparación con las integrinas en fibroblastos o plaquetas (hasta o más de 56 pN)12,13,15. La razón es que la constante de velocidad de hibridación khyb no se ve afectada cuando la carga es inferior a 20 pN según las mediciones de pinzas ópticas, que está en el rango de 106 M-1 S-1. Además, se predice que el k hyb bajo fuerzas < 20 pN será ligeramente mayor que en khyb a fuerza cero, ya que la fuerza débil ayuda a alinear la hebra para que la hebra complementaria se hibrida con17. Por otro lado, se espera que fuerzas mayores dificulten la hibridación, ya que el koff aumenta y crea una barrera para que ocurra la hibridación18. Para una cerradura de 15-17 nucleótidos de largo, estimamos que una fuerza alrededor de 27.8 a 30.8 pN dificultará la hibridación. Dadas estas limitaciones, sugerimos aplicar exclusivamente este método en investigaciones de las fuerzas del receptor de células inmunes. Además, es probable que la presencia de la hebra de bloqueo incline el paisaje de energía para el despliegue de horquillas, lo que podría disminuir ligeramente el F1/2 efectivo.
Divulgaciones
Los autores declaran no tener ningún conflicto de intereses.
Agradecimientos
Este trabajo fue apoyado por las subvenciones R01GM131099 de los NIH, NIH R01GM124472 y NSF CAREER 1350829. Agradecemos a la Instalación de Tetrámeros de los NIH por los ligandos pMHC. Este estudio fue apoyado, en parte, por el Emory Comprehensive Glycomics Core.
Materiales
| Name | Company | Catalog Number | Comments |
| 3-hydroxypicolinic acid (3-HPA) | Sigma | 56197 | maldi-TOF-MS matrix |
| mPEG-SC | Biochempeg | MF001023-2K | surface prep |
| (3-Aminopropyl)triethoxysilane | Acros | AC430941000 | surface prep |
| 10x Red blood cell lysis buffer | Biolegend | 00-4333-57 | buffer |
| 8.8 nm gold nanoparticles, tannic acid | Nanocomposix | customized order | surface prep |
| Atto647N NHS ester | Sigma | 18373-1MG-F | fluorophore, oligo prep |
| Attofluor Cell Chamber, for microscopy | Thermo Fisher Scientific | A7816 | imaging |
| BD Syringes only with Luer-Lok | BD bioscience | 309657 | cells |
| biotinylated anti-mouse CD3e | ebioscience | 13-0031-82 | antibody/ligand |
| Biotinylated pMHC ovalbumin (SIINFEKL) | NIH Tetramer Core Facility at Emory University | NA | antibody/ligand |
| bovine serum albumin | Sigma | 735078001 | block non-specific interactions |
| Cell strainers | Biologix | 15-1100 | cells |
| Coverslip Mini-Rack, teflon | Thermo Fisher Scientific | C14784 | surface prep |
| Cy3B NHS ester | GE Healthcare | PA63101 | fluorophore, oligo prep |
| Dulbecco's phosphate-buffered saline (DPBS) | Corning | 21-031-CM | buffer |
| ethanol | Sigma | 459836 | surface prep |
| Hank’s balanced salts (HBSS) | Sigma | H8264 | buffer |
| hydrogen peroxide | Sigma | H1009 | surface prep |
| LA-PEG-SC | Biochempeg | HE039023-3.4K | surface prep |
| Midi MACS (LS) startup kit | Miltenyi Biotec | 130-042-301 | cells |
| mouse CD8+ T cell isolation kit | Miltenyi Biotec | 130-104-075 | cells |
| Nanosep MF centrifugal devices | Pall laboratory | ODM02C35 | oligo prep |
| No. 2 round glass coverslips | VWR | 48382-085 | surface prep |
| NTA-SAM | Dojindo Molecular Technologies | N475-10 | surface prep |
| P2 gel | Bio-rad | 1504118 | oligo prep |
| sufuric acid | EMD Millipore Corporation | SX1244-6 | surface prep |
| Sulfo-NHS acetate | Thermo Fisher Scientific | 26777 | surface prep |
| Equipment | |||
| Agilent AdvanceBio Oligonucleotide C18 column, 4.6 x 150 mm, 2.7 μm | 653950-702 | oligonucleotide preparation | |
| Barnstead Nanopure water purifying system | Thermo Fisher | water | |
| CFI Apo 100× NA 1.49 objective | Nikon | Microscopy | |
| Cy5 cube | CHROMA | Microscopy | |
| evolve electron multiplying charge coupled device (EMCCD) | Photometrics | Microscopy | |
| High-performance liquid chromatography | Agilent 1100 | oligonucleotide preparation | |
| Intensilight epifluorescence source | Nikon | Microscopy | |
| Matrix-assisted laser desorption/ionization time-of-flight mass spectrometer (MALDI-TOF-MS) | Voyager STR | oligonucleotide preparation | |
| Nanodrop 2000 UV-Vis Spectrophotometer | Thermo Fisher | oligonucleotide preparation | |
| Nikon Eclipse Ti inverted microscope | Nikon | Microscopy | |
| Nikon Perfect Focus System | Nikon | Microscopy | |
| NIS Elements software | Nikon | Microscopy | |
| quad band TIRF 405/488/561/647 cube | CHROMA | Microscopy | |
| RICM cube | CHROMA | Microscopy | |
| TIRF launcher with 488 nm (50 mW), 561 nm (50 mW), and 640 nm | Coherent | Microscopy | |
| TRITC cube | CHROMA | Microscopy | |
| oligo name | 5' modification / 3' modification | sequence (5' to 3') | Use |
| 15mer amine locking strand | 5' modification: no modification 3' modification: /3AmMO/ | AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer Atto647N locking strand | 5' modification: Atto647N 3' modification: /3AmMO/ | AAA AAA CAT TTA TAC CCT ACC TA | locking real-time tension signal |
| 15mer non-fluoresccent locking strand | 5' modification: no modification 3' modification: no modification | A AAA AAC ATT TAT AC | locking real-time tension signal for quantitative analysis |
| 4.7 pN hairpin strand | 5' modification: no modification 3' modification: no modification | GTGAAATACCGCACAGATGCGT TTGTATAAATGTTTTTTTCATTTAT ACTTTAAGAGCGCCACGTAGCC CAGC | hairpin probe |
| amine ligand strand | 5' modification: /5AmMC6/ 3' modification: /3Bio/ | CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| BHQ2 anchor strand | 5' modification: /5ThiolMC6-D/ 3' modification: /3BHQ_2/ | TTTGCTGGGCTACGTGGCGCTCTT | hairpin probe |
| Cy3B ligand strand | 5' modification: Cy3B 3' modification: /3Bio/ | CGCATCTGTGCG GTA TTT CAC TTT | hairpin probe |
| unlocking strand | 5' modification: no modification 3' modification: no modification | TAG GTA GGG TAT AAA TGT TTT TTT C | unlocking accumulated tension signal |
Referencias
- Dustin, M. L. T-cell activation through immunological synapses and kinapses. Immunological Reviews. 221 (1), 77-89 (2008).
- Spillane, K. M., Tolar, P. B cell antigen extraction is regulated by physical properties of antigen-presenting cells. Journal of Cell Biology. 216 (1), 217-230 (2017).
- Feng, Y., et al. Mechanosensing drives acuity of αβ T-cell recognition. Proceedings of the National Academy of Sciences. 114 (39), 8204-8213 (2017).
- Hong, J., et al. A TCR mechanotransduction signaling loop induces negative selection in the thymus. Nature Immunology. 19 (12), 1379-1390 (2018).
- Basu, R., et al. Cytotoxic T cells use mechanical force to potentiate target cell killing. Cell. 165 (1), 100-110 (2016).
- Bashour, K. T., et al. CD28 and CD3 have complementary roles in T-cell traction forces. Proceedings of the National Academy of Sciences. 111 (6), 2241-2246 (2014).
- Ma, V. P. Y., Salaita, K. DNA nanotechnology as an emerging tool to study mechanotransduction in living systems. Small. 15 (26), 1900961 (2019).
- Liu, Y., Yehl, K., Narui, Y., Salaita, K. Tension sensing nanoparticles for mechano-imaging at the living/nonliving interface. Journal of the American Chemical Society. 135 (14), 5320-5323 (2013).
- Glazier, R., et al. DNA mechanotechnology reveals that integrin receptors apply pN forces in podosomes on fluid substrates. Nature Communications. 10 (1), 1-13 (2019).
- Galior, K., Liu, Y., Yehl, K., Vivek, S., Salaita, K. Titin-based nanoparticle tension sensors map high-magnitude integrin forces within focal adhesions. Nano Letters. 16 (1), 341-348 (2016).
- Zhang, Y., Ge, C., Zhu, C., Salaita, K. DNA-based digital tension probes reveal integrin forces during early cell adhesion. Nature Communications. 5, 5167 (2014).
- Liu, Y., et al. DNA-based nanoparticle tension sensors reveal that T-cell receptors transmit defined pN forces to their antigens for enhanced fidelity. Proceedings of the National Academy of Sciences. 113 (20), 5610-5615 (2016).
- Zhang, Y., et al. Platelet integrins exhibit anisotropic mechanosensing and harness piconewton forces to mediate platelet aggregation. Proceedings of the National Academy of Sciences. 115 (2), 325-330 (2018).
- Huang, J., et al. The kinetics of two-dimensional TCR and pMHC interactions determine T-cell responsiveness. Nature. 464 (7290), 932-936 (2010).
- Ma, R., et al. DNA probes that store mechanical information reveal transient piconewton forces applied by T cells. Proceedings of the National Academy of Sciences. 116 (34), 16949-16954 (2019).
- Hui, E., et al. T cell costimulatory receptor CD28 is a primary target for PD-1-mediated inhibition. Science. 355 (6332), 1428-1433 (2017).
- Whitley, K. D., Comstock, M. J., Chemla, Y. R. Elasticity of the transition state for oligonucleotide hybridization. Nucleic Acids Research. 45 (2), 547-555 (2016).
- Brockman, J. M., et al. Live-cell super-resolved PAINT imaging of piconewton cellular traction forces. Nature Methods. 17 (10), 1018-1024 (2020).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados