Method Article
Fluorescencia microscopia de Selección y secuenciación de próxima generación: Herramientas útiles para la identificación de genes implicados en la integridad de los Orgánulos
En este artículo
Resumen
Una misión fundamental de la biología celular es definir los mecanismos que subyacen a la identidad de los orgánulos que hacen que las células eucariotas. Aquí proponemos un método para identificar los genes responsables de la integridad morfológica y funcional de los orgánulos de las plantas mediante microscopía de fluorescencia y la próxima generación de herramientas de secuenciación.
Resumen
Este protocolo describe un microscopio de fluorescencia basada en el cribado de las plántulas de Arabidopsis y se describe cómo asignar las mutaciones recesivas que alteran la distribución subcelular de un marcador específico etiquetado fluorescente en la vía secretora. Arabidopsis es un poderoso modelo biológico para estudios genéticos debido a su tamaño del genoma, tiempo de generación y conservación de los mecanismos moleculares entre reinos. La matriz de genotipado como una aproximación al mapa de la mutación en alternativa al método tradicional, basado en marcadores moleculares, es ventajoso porque es relativamente más rápido y puede permitir la asignación de varios mutantes en un marco de tiempo muy corto. Este método permite la identificación de proteínas que pueden influir en la integridad de cualquier orgánulo en las plantas. Aquí, como un ejemplo, proponemos una pantalla para los genes del mapa importantes para la integridad del retículo endoplasmático (ER). Nuestro enfoque, sin embargo, se puede extender fácilmente a otros orgánulos de las células vegetales(Véase por ejemplo 1,2), y por lo tanto representa un paso importante hacia la comprensión de las bases moleculares que rigen en otras estructuras subcelulares.
Protocolo
1. EMS Tratamiento
Semillas de Arabidopsis thaliana se mutagénesis utilizando como agente mutágeno de metano sulfonato de etilo (EMS) de 3,4, lo que induce en el genoma de C a T cambios resultantes en la C / G a T / A mutaciones 5-7.
- Pesar 0,8 g de semillas de Arabidopsis (aproximadamente 40.000 semillas) que llevan el marcador fluorescente orgánulo (específicamente, en este estudio ssGFPHDEL (secuencia señal-GFP-HDEL tetrapéptido) ha sido utilizado como un marcador ER).
- Transferencia de las semillas en un tubo Falcon de 50 ml, a continuación, añadir 25 ml de agua destilada.
- Añadir 0,2% (v / v) sulfonato de etilo metano.
- Incubar durante 16 horas en la oscilante batidora a velocidad baja.
- Aspirar el líquido y desecharlo en un matraz que contenía 1,0 M de NaOH para inactivar el SME.
- Añadir 25 ml de agua al tubo Falcon que contiene las semillas, muy cerca, e invertir 5 veces para lavar las semillas, espere hasta que todas las semillas que se han asentado, y aspirar el agua y desechar en el Na 1,0 M OH matraz.
- Repetir la etapa de lavado hasta 10 veces.
- Después del lavado final, volver a suspender las semillas en una cantidad mínima de agua.
- Proceda a la esterilización de semillas añadir 25 ml de lejía al 10%, agitar con fuerza durante 30 s. Que las semillas se depositan en el fondo, a continuación, verter el cloro y enjuagar con 25 ml de agua estéril. Vierta el agua estéril y añadir 25 ml de etanol al 70%. Agitar los tubos durante 30 s. Que las semillas se depositan en el fondo. Retirar el etanol y el enjuague con 25 ml de agua estéril. Repetir dos veces el lavado con agua estéril, y luego verter las semillas en una placa Petri de plástico que contiene un papel de filtro 3 MM y dejar secar bajo el capó. Almacenar a 4 ° C durante 2 días.
- Placa de las semillas en ½ MS Phytagel (concentración media de medio Murashige y Skoog), de 150 mm placa de Petri (aproximadamente 250-300 semillas para la placa).
- Crecer semillas M1 en la placa durante 2 semanas, para luego trasplantarlas en el suelo.
- Recoger las semillas de plantas individuales M2 M1 M2 para generar las líneas (1000 líneas independientes).
En esta sección se describe la observación de las plantas de semillero con un microscopio confocal o de fluorescencia como se describió anteriormente 8.
- Sesenta semillas de cada línea M2 son cultivadas por 7 a 10 días en cajas Petri de plástico de ½ MS Phytagel, en el mismo plato también se cultivan plantas EMS-5 sin tratamiento (control).
- De cinco a diez cotiledones están montados con la cara abaxial hacia el objetivo (40X) en un portaobjetos de microscopio y encerrados con un cubreobjetos.
- Cada cotiledón se observa, desde la cortical a la región media, bajo fluorescencia para cualquier alteración de la distribución subcelular del marcador orgánulo.
- Plantas positivos se trasplantan en la tierra y sus semillas se recogen y se proyectó de nuevo para confirmar el fenotipo mutante en la generación M3.
- Quite los contaminantes a través de mutaciones de fondo retrocruzamiento al menos tres veces a un genoma de tipo salvaje, posiblemente, containing el deseado marcador fluorescente orgánulo.
- De la planta madre, el uso de tijeras o pinzas finas retire silicuas maduras y flores abiertas.
- Eliminar los brotes que son demasiado pequeños del meristema.
- Inserte la punta de un par de pinzas entre los pétalos y los sépalos de abrir un capullo de la flor y eliminar todas las anteras.
- Usando un par de pinzas tome una flor madura abierta de la planta padre y frote las anteras sobre el estigma de la planta de castrado.
3. Mapeo
En esta sección se describe esencialmente la forma de asignar una mutación recesiva con un protocolo modificado de Borevitz 9 que es más rápido si se compara con los métodos de mapeo tradicionales 10,11. Este enfoque utilizar matrices de alta densidad de oligonucleótidos con la capacidad de detectar polimorfismos numerosas sola característica (SFP) en un solo ensayo 12. Uso de la Arabidopsis Affymetrix GeneChip ATH1 matriz es posibleanalizar los aproximadamente 24.000 genes. Un grupo de individuos F 2 que muestran que la mutación se compara con una piscina de tipo salvaje F 2 plantas recolectadas dentro de la misma población segregante. Entonces, la mutación se asignan en la región donde se enriquece la piscina mutante para los alelos mutantes genotipo y por consiguiente en la misma región de la piscina de tipo salvaje resultará enriquecido para los alelos matrices de tipo salvaje 13.
- El ADN genómico (3 g) se extrae de la homocigotos Columbia mutante (M3), utilizando una Qiagen DNeasy Plant Mini Kit y se somete a Illumina Genoma Analyzer II (GA II) 14 secuenciación.
- El mutante homocigótico Columbia (M3) se cruza con Landsberg erecta para generar una población de mapeo.
- La asignación se realiza en 70 hasta 100 individuos F2 que muestran el fenotipo aberrante y el mismo número de plantas F2 con un fenotipo de tipo salvaje.
- Recoger de cada planta de un disco de la hoja (0,60 mm) utilizando un punzón. Ladisco de la hoja debe ser recogida de las hojas de la misma edad para asegurarse de que tienen cantidades similares de ADN. Las muestras pueden ser procesadas para la extracción genómico por separado o en grupos. En este caso, es posible recoger muestras 5-10 para cada tubo Eppendorf de modo que al final tendrá menos tubos eppendorf de ADN genómico que tiene que ser extraído.
- MasterPure Planta Hoja de purificación de ADN kit (Epicentro) se utiliza para extraer el ADN genómico. El ADN genómico obtenido de cada muestra se cuantifica con un NanoDrop.
- La misma cantidad de ADN genómico de cada muestra se pone entonces juntos hasta un total de 300 ng (~ 30 l) de ADN de la planta; añadir 60 l solución 2.5X cebadores al azar [125 mM Tris-HCl (pH 6,8), 12,5 mM MgCl2, 25 mM 2-mercaptoetanol, 750 ug / ml cebadores oligodesoxirribonucleótidos (octámeros aleatorios)] (Bioprime kit) y 42 l de agua (volumen final de 132 l).
- Desnaturalizan las autoridades nacionales designadas a> 95 ° C durante 5 a 10 min.
- Dejar enfriar en hielo.
- Para cada denaturojo ADN añadir 15 l de mezcla dNTP 10X con biotina dCTP [1 mM biotina-14-dCTP, 1 mM dCTP, 2 mM de dATP, 2 mM de dGTP, 2 mM dTTP en 10 mM Tris-HCl (pH 7,5), 1 mM Na2EDTA] (Bioprime kit), y completar con la polimerasa Klenow 3 l (kit Bioprime).
- Incubar toda la noche a 25 ° C.
- Al día siguiente, añadir 15 l 3 M NaOAc y 400 mL en frío 100% de etanol y mezclar.
- Incubar a -80 ° C durante 1 hora, luego girar a 20.500 xg durante 15 minutos, retire el sobrenadante y se lava con 500 l de EtOH 75% frío.
- Gira a 20.500 xg durante 10 minutos.
- Gránulos secos de ADN a 37 ° C durante 10 min y resuspende en 100 l de agua.
- Utilizar 5 l para comprobar el rendimiento y calidad en un gel (Figura 2).
- Enviar 95 l de etiquetado de reacción para el tipo salvaje y mutante de Arabidopsis hibridación GeneChip ATH1 del Genoma de la matriz.
- Los arreglos son escaneados y los archivos. CEL obtenidos son analizados utilizando el software R ( http://www.r-project.org /).
- Instalar el software R, a continuación, abra el programa y pegar la siguiente cadena:
fuente (" http://bioconductor.org/biocLite.R ")
biocLite ()
A continuación, pulse la tecla, este regreso va a instalar los paquetes de Bioconductor estándar ( http://www.bioconductor.org ). - En una nueva carpeta en el escritorio, descarga desde el siguiente sitio web ( http://www.naturalvariation.org/methods ) los archivos:
readcel.R
SFP.R
Map.R
ath1V5.RData
ColLerCEL.zip
El ColLerCEL.zip archivo debe ser descomprimido y pegar los archivos resultantes en la nueva carpeta. - Cambiar el nombre de los datos obtenidos a partir de su experimento GeneChip en wildtype.CEL y mutant.CEL.
- Copie ahora los datos de GeneChip (wildtype.CEL ymutant.CEL) en la carpeta.
- Abierto I, a) para Mac: haga clic en "Varios", y luego haga clic en "Cambiar directorio de trabajo." b) para PC: haga clic en "Archivo" y luego "Cambiar directorio"
- a) para Mac: Seleccione la carpeta que contiene los archivos. Haga clic en "Espacio de trabajo", "archivo de espacio de trabajo de carga" y seleccione ath1V5.RData, b) para PC: Seleccione la carpeta que contiene los archivos anteriores, haga clic en "Archivo", luego "espacio de trabajo de carga" y seleccione ath1V5.RData.
- ReadCEL.R abierto el Bloc de notas y copiar todo el texto de R.
- SFP.R abierto el Bloc de notas y copiar todo el texto de R.
- Map.R abierto el Bloc de notas y copiar el texto completo de R.
- En la ventana de la consola, verá el siguiente mensaje:
Cromosoma X limita Mb yz
Genes de la búsqueda en TAIR pegar en este enlace
3 http://arabidopsis.org/servlets/sv?action=download&chr=x&start=yY fin = z - Copia y pega el enlace en su navegador de Internet. Aparecerá una ventana y pedir para abrir o guardar el archivo, debe seleccionar guardar. Elegir un nombre de archivo y adjuntar la extensión ". Xls", a continuación, haga clic en Guardar.
- Abra el archivo ". Xls", donde usted puede encontrar la coordenada para el mutante (el resultado se representa gráficamente en la Figura 3).
- Dentro del área asignada en el Illumina montado lee el genoma mutante identificar las transiciones específicas del SME 4 entre los polimorfismos de nucleótido único. Complementar el fenotipo mutante de la transformación con el gen de tipo salvaje (s), utilizando procedimientos estándar 15.
4. Los resultados representativos
La Figura 1 muestra el método utilizado para la identificación de un mutante de la ruta secretora mediante cribado microscopía confocal. La figura 2 muestra una preparación típica de ADN marcado genómico para la hibridación matriz. En la figura 3, un resultado típico se esperaba después de analizar los datos obtenidos a partir de la matriz GeneChip genoma de Arabidopsis ATH1 se presenta.

Figura 1. Plantas transgénicas de Arabidopsis que expresan ssGFPHDEL (ER marcador) (1) fueron cultivadas para producir suficientes semillas para EMS tratamiento (2). Las semillas tratadas con EMS se sembraron entonces para generar plantas M1 (3). Cada planta M1 representa una línea diferente y las semillas se obtuvieron por separado de cada uno de ellos. Semillas se sembraron en M2 ½ sustrato MS (4) y luego examinados para detectar defectos en la morfología de ER por microscopía confocal (5). Durante la proyección hemos encontrado plantas que conserva la morfología de tipo salvaje ER (6) y las plantas que muestran defectuosos fenotipos ER (7). Estas plantas se cultivaron para obtener la generación M3 y para confirmar el fenotipo (8). ADN genómico de la planta M3 fue utilizado para Solexa Illumina secuenciación (a). La misma plantase utilizó también para cruces con Ler en peso para obtener la población de mapeo F2 (b).
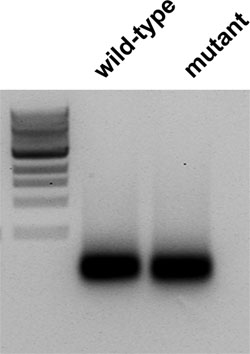
Figura 2. Reacciones Bioprime etiquetado aleatorios (5 l de 100 l) se cargaron en gel de agarosa al 1%. Lane, una es de un grupo de plantas silvestres F2, y el carril b es de un grupo de mutantes plantas F2. (Marcador es 1 Kb de ADN escalera N3232, de la NEB).

Figura 3. La figura representa un ejemplo de asignación de Col-0 mutación usando Arabidopsis GeneChip ATH1 Genoma variedad de hibridación. La mutación en este ejemplo se encuentra en el cromosoma 1 delimitado por barras verticales. Las barras horizontales representan los umbrales de detección.
Discusión
Aquí se describe una confocal de microscopía basada en el cribado para la identificación de mutantes endomembrane. Este enfoque se puede extender fácilmente a otros orgánulos de la célula para que determinados marcadores de proteínas fluorescentes están disponibles. La pantalla se basa en la identificación de mutantes que muestran una distribución aberrante del marcador fluorescente o bien en el orgánulo meta o para orgánulos que no se supone que contienen el marcador. Respectivamente, estos mutantes representan poblaciones en las que se ve comprometida ya sea la capacidad del orgánulo a subcompartimentalize el marcador o mutantes que no pueden trasladar correctamente el marcador entre orgánulos. Durante la investigación nos dimos cuenta de que algunos mutantes mostraron un fenotipo en las etapas tempranas del desarrollo tan pronto como sea 7 días después de la germinación, sin embargo, otros mostraron el fenotipo sólo en una etapa posterior del desarrollo. Si bien esto puede estar vinculado a una serie de razones, incluyendo la expresión desarrollo dependiente de la mutadoalelo (s), esto sugiere que las plantas deben ser examinados en diferentes etapas de crecimiento (por lo menos 7 a 14 días) para garantizar que los mutantes potencialmente interesantes, no se descartan.
El enfoque descrito en este protocolo nos permite mapear un gen en un tiempo relativamente corto en comparación con el método de asignación clásica. De hecho, recogiendo un número suficiente de individuos de la población F2 utilizado para la matriz GeneChip requiere una cantidad de tiempo relativamente pequeña en comparación con las plantas F2 requeridos para la clásica fina mapeo procedimiento. Sin embargo, hay pasos críticos que deben ser observadas. La selección de muestras de población F2 que muestran o no mostrando el fenotipo debe llevarse a cabo cuidadosamente, puesto que la adición de incluso un pequeño número de plantas con el fenotipo equivocada por error puede introducir errores en el resultado final. Esto es mayormente relacionado con el hecho de que la población F2 utiliza para la asignación áspera es muy pequeña. Por esta razón, la selección de plantas que eitla muestran o no muestran el fenotipo debe llevarse a cabo cada vez que en el mismo día después de la germinación. Esto es porque, como ya se mencionó anteriormente, la aparición del fenotipo puede estar relacionado con el crecimiento. Otro punto importante a tener en cuenta es prestar atención a la próxima generación postalignment de datos, que es generado por los programas que nos dan información importante como el porcentaje de lecturas que contiene la variante. El valor teórico esperado para una variante heterocigótica es de alrededor de 50%, esto significa que el 50% de las secuencias que contienen la variante mientras que para una variante homocigótica, es alrededor de 100%. Por desgracia, la situación después de un postalignment puede ser medida a partir del valor teórico, de hecho, el porcentaje de lecturas que contiene la variante de heterocigotos puede variar desde el 20% a 80, mientras 60 a 100% de homocigotos 16. Por lo tanto, para no perderse los principales polimorfismos de nucleótido único (SNP), es importante no descartar mutantes con un porcentaje inferior al 100% de la reanuncios de la variante.
Divulgaciones
No tenemos nada que revelar.
Agradecimientos
Reconocemos el apoyo de la Ciencias Químicas, Geociencias y Ciencias Biológicas División de la Oficina de Ciencias Básicas de Energía, Oficina de Ciencia, EE.UU. Departamento de Energía (número de adjudicación DE-FG02-91ER20021) y la National Science Foundation (MCB 0948584) (FB). Estamos muy agradecidos a la Sra. Karen Bird para la edición del manuscrito.
Materiales
| Name | Company | Catalog Number | Comments |
| Nombre del reactivo | Empresa | Número de catálogo | |
| Etilmetano sulfonato | Sigma | M0880 | |
| NaOH | JT Baker | 3722-05 | |
| Murashige Skoog basal media w vitaminas Gamborg | Phyto Technolog Laboratorie | M404 | |
| Phytagel | Sigma | P8169-1Kg | |
| RNeasy mini kit | Qiagen | 74104 | |
| Maestro de la hoja pura de plantas de purificación de ADN kit | Epicentro | MPP92100 | |
| ADN Bioprime sistema de etiquetado | Invitrogen | 18094-011 | |
| El alcohol 200 prueba | Decan laboratorios inc. | 2716 | |
| NaOAc | JT Baker | ||
| Chip de genes de Arabidopsis ATH1amplia del genoma | Affymetrix | 900385 | |
| Tubos Falcon de 50 ml | Corning | 430290 | |
| Tubos Eppendorf de 1,5 ml | |||
| De papel de filtro de 90mm | Whatman | 1001090 | |
| Balanza Analítica | Mettler Toledo AB54-S | na | |
| Oscilante (de onda) agitador | Heidolph POLYMAX 1040 | na | |
| Centrifugar | Eppendorf 5417-R | na |
Referencias
- Marti, L. A missense mutation in the vacuolar protein GOLD36 causes organizational defects in the ER and aberrant protein trafficking in the plant secretory pathway. The Plant journal : for cell and molecular biology. 63, 901-913 (2010).
- Stefano, G., Renna, L., Moss, T., McNew, J., Brandizzi, F. Arabidopsis the spatial and dynamic organization of the endoplasmic reticulum and Golgi apparatus is influenced by the integrity of the c-terminal domain of RHD3, a non-essential GTPase. The Plant Journal. , Forthcoming (2011).
- Kim, Y., Schumaker, K. S., Zhu, J. K. EMS mutagenesis of Arabidopsis. Methods in molecular biology. 323, 101-103 (2006).
- Maple, J., Moller, S. G. Mutagenesis in Arabidopsis. Methods in molecular biology. 362, 197-206 (2007).
- Greene, E. A. Spectrum of chemically induced mutations from a large-scale reverse-genetic screen in Arabidopsis. Genetics. 164, 731-740 (2003).
- Krieg, D. R. Ethyl methanesulfonate-induced reversion of bacteriophage T4rII mutants. Genetics. 48, 561-580 (1963).
- Kovalchuk, I., Kovalchuk, O., Hohn, B. Genome-wide variation of the somatic mutation frequency in transgenic plants. The EMBO journal. 19, 4431-4438 (2000).
- Boulaflous, A., Faso, C., Brandizzi, F. Deciphering the Golgi apparatus: from imaging to genes. Traffic. 9, 1613-1617 (2008).
- Borevitz, J. Genotyping and mapping with high-density oligonucleotide arrays. Methods in molecular biology. 323, 137-145 (2006).
- Konieczny, A., Ausubel, F. M. A procedure for mapping Arabidopsis mutations using co-dominant ecotype-specific PCR-based markers. The Plant journal : for cell and molecular biology. 4, 403-410 (1993).
- Bell, C. J., Ecker, J. R. Assignment of 30 microsatellite loci to the linkage map of Arabidopsis. Genomics. 19, 137-144 (1994).
- Hazen, S. P., Kay, S. A. Gene arrays are not just for measuring gene expression. Trends in Plant Science. 8, 413-416 (2003).
- Hazen, S. P. Rapid array mapping of circadian clock and developmental mutations in Arabidopsis. Plant Physiology. 138, 990-997 (2005).
- Bentley, D. R. Accurate whole human genome sequencing using reversible terminator chemistry. Nature. 456, 53-59 (2008).
- Weigel, D., Glazebrook, J. Arabidopsis. A Laboratory Manual. , Cold Spring Harbor Laboratory Press. Cold Spring Harbor, New York. (2002).
- Voelkerding, K. V., Dames, S., Durtschi, J. D. Next generation sequencing for clinical diagnostics-principles and application to targeted resequencing for hypertrophic cardiomyopathy: a paper from the 2009 William Beaumont Hospital Symposium on Molecular Pathology. J. Mol. Diagn. 12, 539-551 (2010).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados