Method Article
Fluoreszenz-Mikroskopie Screening und Next-Generation-Sequencing: Nützliche Tools für die Identifizierung von Genen in den Organellen Integrität beteiligt
In diesem Artikel
Zusammenfassung
Eine grundlegende Aufgabe in der Zellbiologie ist es, die Mechanismen, die die Identität der Organellen, die eukaryotischen Zellen machen zu Grunde liegen zu definieren. Hier schlagen wir eine Methode, um die Gene verantwortlich für die morphologische und funktionelle Integrität der Anlage Organellen mittels Fluoreszenzmikroskopie und Next-Generation-Sequencing-Tools zu identifizieren.
Zusammenfassung
Dieses Protokoll beschreibt ein Fluoreszenzmikroskop-basierte Screening von Arabidopsis-Keimlinge und beschreibt, wie rezessive Mutationen, die die subzelluläre Verteilung eines spezifischen fluoreszierenden Marker markiert in den sekretorischen Weg verändern zu kartieren. Arabidopsis ist ein leistungsfähiges biologisches Modell für genetische Studien wegen seiner Genomgröße, Generationszeit, und die Erhaltung der molekularen Mechanismen unter Königreiche. Die Array-Genotypisierung als ein Ansatz, um die Mutation in Alternative zu den traditionellen Verfahren auf der Basis molekularer Marker zugeordnet ist vorteilhaft, weil es relativ schneller ist und kann die Zuordnung von mehreren Mutanten in wirklich kurzer Zeit ermöglichen. Dieses Verfahren ermöglicht die Identifizierung von Proteinen, die die Integrität einer Organelle in Pflanzen beeinflussen können. Hier, als Beispiel, schlagen wir einen Bildschirm zur Karte Gene, die für die Integrität des endoplasmatischen Retikulum (ER). Unser Ansatz kann aber leicht auf andere Pflanzen Zellorganellen erweitert werden(Siehe zum Beispiel 1,2), und stellt daher einen wichtigen Schritt zum Verständnis der molekularen Grundlagen für die anderen subzellulären Strukturen.
Protokoll
1. EMS-Behandlung
Arabidopsis thaliana Samen werden mit mutagenisiert als Mutagen Ethylmethansulfonat Mittel (EMS) 3,4, die in das Genom C-zu-T-Änderungen, die sich in C / G, T / A-Mutationen 5-7 induziert.
- 0,8 g wiegen Arabidopsis Samen (~ 40.000 Samen), die den fluoreszierenden Marker Organellen (insbesondere die in dieser Studie ssGFPHDEL (Signalsequenz-GFP-Tetrapeptid HDEL) wurde als ein ER-Marker verwendet wurden).
- Übertragen Sie die Samen in einem 50-ml-Falcon-Röhrchen, dann 25 ml destilliertem Wasser.
- 0,2% (v / v) Ethylmethansulfonat.
- Inkubieren Sie für 16 Stunden auf Taumelmischer bei niedrigen Drehzahlen.
- Absaugen der Flüssigkeit und verwerfen in einen Kolben mit 1,0 M NaOH, um den EMS zu inaktivieren.
- 25 ml Wasser auf das Falcon-Röhrchen mit den Samen, in der Nähe, und 5 Mal umdrehen, um die Samen zu waschen; warten, bis alle Samen niedergelassen haben, dann saugen das Wasser und in der 1,0 M Na verwerfen OH-Kolben.
- Wiederholen der Wasch-Schritt bis zu 10 mal.
- Nach dem letzten Waschen, erneut zu suspendieren die Samen in einer minimalen Menge Wasser.
- Gehen Sie zur Sterilisation Samen Zugabe von 25 ml 10% Bleichmittel, kräftig schütteln, 30 s. Lassen Samen zu Boden absetzen, dann abgießen Bleichmittel und spülen mit 25 ml sterilem Wasser. Abgießen, sterilem Wasser und 25 ml 70% Ethanol. Schütteln Röhrchen für 30 s. Lassen Samen zu Boden absetzen. Abgießen und Ethanol spülen mit 25 ml sterilem Wasser. Wiederholen Sie die Wäsche zweimal mit sterilem Wasser, dann gießen Samen auf einer Kunststoff-Petrischale mit 3 MM Filterpapier und lassen unter der Haube trocknen. Lagerung bei 4 ° C für 2 Tage.
- Tafel die Samen auf S MS Phytagel (halbe Konzentration von Murashige und Skoog-Medium), 150-mm-Petrischale (~ 250-300 Samen für die Platte).
- Wachsen M1 Samen auf Platte für 2 Wochen, dann in den Boden zu verpflanzen.
- Sammle M2 Samen von einzelnen Pflanzen M1 bis M2 Linien (1000 unabhängige Linien) zu generieren.
In diesem Abschnitt beschreiben wir die Beobachtung von Sämlingen mit einem konfokalen Fluoreszenzmikroskop oder wie oben beschrieben 8.
- Sechzig Samen aus jeder Linie M2 sind für 7 bis 10 Tagen auf Kunststoff-Petrischalen, ½ MS Phytagel gewachsen, auf der gleichen Platte sind auch 5 Sämlinge gewachsen EMS-unbehandelten (Kontrolle).
- Fünf bis zehn Cotyledonen mit der unterseitigen Seite in Richtung der Linse (40X) auf einem Objektträger montiert und mit einem Deckglas eingeschlossen.
- Jedes Keimblatt beobachtet wird, von der kortikalen zu medialen Bereich, unter Fluoreszenz für jede veränderte subzelluläre Verteilung der Organellen-Marker.
- Positiv Pflanzen werden in Erde und ihre Samen werden gesammelt und erneut, um den mutierten Phänotyp in der M3-Generation bestätigen gescreent.
- Entfernen Sie die verunreinigende Hintergrund Mutationen durch Rückkreuzung mindestens dreimal zu einem Wildtyp-Genom gegebenenfalls COntaining die gewünschte Organelle fluoreszierenden Marker.
- Von der Mutterpflanze, mit einer feinen Schere oder Zange entfernen reifen Schoten und offenen Blüten.
- Entfernen Sie die Knospen, die zu klein sind aus dem Meristem.
- Legen Sie die Spitze von einem Pinzette zwischen Kelch-und Blütenblätter auf eine Knospe öffnen und entfernen Sie alle Antheren.
- Mit einem Zange nehmen eine offene reifen Blume aus dem Vater-Anlage und reiben Sie die Antheren auf die Narbe der Pflanze entmannt.
3. Mapping
Dieser Abschnitt beschreibt, wie man Wesentlichen eine rezessive Mutation unter Verwendung eines modifizierten Protokolls von Borevitz 9 schneller, wenn die traditionellen Mapping-Verfahren im Vergleich 10,11 abzubilden. Dieser Ansatz verwenden high-density Oligonukleotid-Arrays mit der Fähigkeit, zahlreiche Einzel-Merkmal-Polymorphismen (SFP) in einem einzigen Assay 12 zu erfassen. Mit dem Affymetrix GeneChip Arabidopsis ATH1 Array ist möglich,analysieren rund 24.000 Gene. Ein Pool von F 2-Individuen, welche die Mutation wird auf einen Pool von Wildtyp-F 2-Pflanzen innerhalb der gleichen Trennung Bevölkerung gesammelt verglichen. Dann wird die Mutation in dem Bereich, wo die Mutanten-Pool für mutierten Genotyp Allele angereichert zugeordnet wird, und folglich in der gleichen Region der Wildtyp-Pool wird führen angereichert für die Wildtyp-Eltern-Allele 13.
- Genomische DNA (3 ug) von der Columbia homozygoten mutanten (M3) unter Verwendung eines Qiagen DNeasy Plant Mini Kit extrahiert und für Illumina Genome Analyzer II (GA II) 14 Sequenzierung unterzogen.
- Der homozygote Mutante Columbia (M3) mit Landsberg erecta gekreuzt, um eine Zuordnung Bevölkerung zu schaffen.
- Die Darstellung wird auf 70 bis 100 F2 Individuen mit dem aberranten Phänotyp und die gleiche Anzahl von F 2-Pflanzen mit einem Wildtyp-Phänotyp durchgeführt.
- Sammle von jeder Pflanze ein Blatt Disc (0,60 mm) mit einem Locher. DieBlattscheiben sollte aus den Blättern des gleichen Alters gesammelt werden, um sicher sein, ähnliche Mengen an DNA zu haben. Die Proben können für die genomische Extraktion einzeln oder in Gruppen bearbeitet werden. In diesem Fall ist es möglich, zu 5-10 Proben für jede Eppendorf-Röhrchen zu sammeln, um am Ende wird es weniger Eppendorf-Röhrchen aus dem genomischen DNA extrahiert werden muss haben.
- MasterPure Pflanze Blatt DNA Purification Kit (Epicentre) verwendet wird, um die genomische DNA zu extrahieren. Die genomische DNA von jeder Probe erhalten wird mit einem Nano-Drop quantifiziert.
- Die gleiche Menge an genomischer DNA aus jeder Probe wird dann zusammen bis zu einem Gesamtbetrag von 300 ng (~ 30 ul) von Pflanzen-DNA; mit 60 ul 2,5 X zufälliger Primer-Lösung [125 mM Tris-HCl (pH 6,8), 12,5 mM MgCl2, 25 mM 2-Mercaptoethanol, 750 ug / ml Oligodesoxyribonukleotid Primer (random Octamere)] (BioPrime Kit) und 42 ul Wasser (Endvolumen 132 ul).
- Denaturieren DNAs bei> 95 ° C für 5 bis 10 min.
- Auf Eis abgekühlt.
- Zu jedem Denaturierungroten DNA hinzuzufügen 15 ul 10X dNTPs Mischung mit Biotin dCTP [1 mM Biotin-14-dCTP, 1 mM dCTP, 2 mM dATP, 2 mM dGTP, dTTP 2 mM in 10 mM Tris-HCl (pH 7,5), 1 mM Na2EDTA] (BioPrime Kit), und mit 3 ul Klenow-Polymerase (BioPrime Kit) abzuschließen.
- Inkubieren über Nacht bei 25 ° C.
- Am folgenden Tag, fügen Sie 15 ul 3 M NaOAc und 400 ul kaltem 100% EtOH und mischt.
- Inkubation bei -80 ° C für 1 Stunde, dann bei 20.500 xg für 15 min zu spinnen, zu entfernen Überstand und wäscht mit 500 ul kaltem 75% EtOH.
- Zentrifugieren bei 20.500 xg für 10 min.
- Chemische DNA-Pellets bei 37 ° C für 10 min und Resuspension in 100 ul Wasser.
- Verwenden Sie 5 ul auf Ertrag und Qualität auf einem Gel (Abbildung 2) zu überprüfen.
- Senden Sie 95 ul der Kennzeichnung Reaktion für die Wildtyp und Mutante für GeneChip Arabidopsis ATH1 Genome Array-Hybridisierung.
- Die Arrays werden gescannt und die. CEL-Dateien erhalten werden mittels R-Software ( http://www.r-project.org /).
- Installieren Sie R-Software, dann öffnen Sie das Programm und fügen Sie die folgende Zeichenfolge ein:
Quelle (" http://bioconductor.org/biocLite.R ")
biocLite ()
Drücken Sie dann die Return-Taste-das wird die Standard-Pakete Bioconductor (installieren http://www.bioconductor.org ). - In einem neuen Ordner auf Ihrem Desktop, von der folgenden Website (Download http://www.naturalvariation.org/methods ) die Dateien:
readcel.R
SFP.R
Map.R
ath1V5.RData
ColLerCEL.zip
Die Datei ColLerCEL.zip sollten ausgepackt und die resultierenden Dateien in Ihrem neuen Ordner eingefügt. - Benennen Sie die Daten aus Ihrer GeneChip Experiment in wildtype.CEL und mutant.CEL erhalten.
- Kopieren Sie nun Ihre Daten GeneChip (wildtype.CEL undmutant.CEL) in Ihrem Ordner.
- Open-R, a) für Mac: Klicken Sie auf "Misc", und klicken Sie dann auf "Change Arbeitsverzeichnis." b) für PC: Klicken Sie auf "Datei" dann "Verzeichnis wechseln"
- a) für Mac: Wählen Sie den Ordner mit den Dateien. Klicken Sie auf "Workspace", "Load Workspace-Datei" und wählen Sie ath1V5.RData, b) für PC: Wählen Sie den Ordner mit den Dateien, die oben, klicken Sie auf "Datei", dann "Load Workspace" und wählen Sie ath1V5.RData.
- Offene readCEL.R mit dem Editor und kopiere den gesamten Text zu R.
- Offene SFP.R mit dem Editor und kopiere den gesamten Text zu R.
- Offene Map.R mit dem Editor und kopiere den gesamten Text zu R.
- In der Console-Fenster sehen Sie die folgende Meldung:
Chromosom X begrenzt Mb yz
Suche Gene bei TAIR in diesem Link fügen
3 http://arabidopsis.org/servlets/sv?action=download&chr=x&start=y& End = z - Kopieren und fügen Sie den Link in Ihren Internet-Browser. Ein Fenster erscheint und fragen Sie zu öffnen oder speichern Sie die Datei, und Sie sollten wählen Sie Speichern. Wählen Sie einen Dateinamen ein und befestigen Sie die Erweiterung ". Xls", klicken Sie auf Speichern.
- Öffnen Sie die Datei ". Xls", wo Sie die Koordinaten für Ihr Mutante (das Ergebnis wird grafisch in Abbildung 3 dargestellt) finden können.
- Innerhalb des kartierten Gebietes im zusammengebauten Illumina liest der Mutantengenom identifizieren spezifische EMS Übergänge zwischen den vier Einzel-Nukleotid-Polymorphismen. Ergänzen das Mutantenphänotyps durch Transformation mit Wildtyp-Gen (e) unter Verwendung von Standardverfahren 15.
4. Repräsentative Ergebnisse
1 zeigt den Ansatz zur Identifizierung einer Mutante von den sekretorischen Weg mittels konfokaler Mikroskopie Screening verwendet. 2 zeigt eine typische Herstellung der markierten genomischen DNA für Array-Hybridisierung. In Abbildung 3 ist ein typisches Ergebnis nach Analyse der Daten aus dem GeneChip Arabidopsis Genome Array erhalten ATH1 präsentiert wird erwartet.

Abbildung 1. Arabidopsis transgene Pflanzen, die ssGFPHDEL (ER-Marker) (1) wurden gezüchtet, um genügend Saatgut für die EMS-Behandlung (2) zu produzieren. Samen mit EMS behandelt wurden dann gesät, um M1 Pflanzen (3) zu generieren. Jeder M1 Pflanze stellt eine andere Linie, und Samen wurden getrennt von jeder von ihnen gesammelt. M2-Samen wurden auf S MS Substrat (4) plattiert wird und dann für Mängel ER Morphologie durch konfokale Mikroskopie (5) abgeschirmt ist. Während der Screening fanden wir, dass Pflanzen konserviert die Wildtyp-ER Morphologie (6) und Pflanzen, die defekte ER Phänotypen (7) zu zeigen. Diese Pflanzen wurden gezüchtet, um den M3-Generation zu erhalten und den Phänotyp (8) zu bestätigen. Genomische DNA von M3-Anlage wurde für Solexa Illumina Sequenzierung (a) verwendet wird. Die gleiche Pflanzewurde auch für Kreuzungen mit Ler-wt verwendet, um den F2-Mapping Bevölkerung (b) zu erhalten.
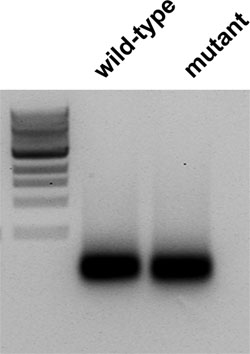
Abbildung 2. BioPrime zufällig Markierungsreaktionen (5 ul 100 ul) wurden auf Agarosegel 1% geladen. Spur a ist aus einem Pool von Wildtyp-F2-Pflanzen, und die Spur b aus einem Pool von Mutante F2-Pflanzen. (Markierung ist 1 kb DNA-Leiter-N3232, von NEB).

Abbildung 3. Die Abbildung stellt ein Beispiel der Abbildung von Col-0-Mutation mit GeneChip Arabidopsis ATH1 Genome Array-Hybridisierung. Die Mutation in diesem Beispiel befindet sich auf Chromosom 1, durch vertikale Balken befindet. Die horizontalen Balken stellen die Schwellenwerte für die Erkennung.
Diskussion
Hier beschreibt eine konfokale Mikroskopie-Screening zur Identifizierung von Mutanten Endomembransystem. Dies kann leicht zu einer anderen Organellen der Zelle, für die spezifische fluoreszierende Protein-Marker zur Verfügung verlängert werden. Der Bildschirm wird auf die Identifizierung von Mutanten, die eine anomale Verteilung der fluoreszierenden Marker, entweder in der Zielorganelle oder Organellen, die nicht dazu bestimmt sind, um den Marker enthalten, die zeigen. Jeweils stellen diese Mutanten untersucht, bei denen entweder die Fähigkeit des Organelle, um den Marker subcompartimentalize verletzt oder Mutanten, die nicht korrekt translozieren die Marker unter Organellen. Während der Vorführung haben wir festgestellt, dass einige Mutanten einen Phänotyp zeigte in frühen Entwicklungsstadien so früh wie 7 Tage nach der Keimung, jedoch zeigte anderem den Phänotyp erst in einem späteren Stadium der Entwicklung. Während dies kann zu einer Reihe von Gründen, einschließlich der Entwicklung-abhängige Expression des mutierten verknüpft werdenAllel (s) legt dies nahe, dass Pflanzen in unterschiedlichen Entwicklungsstadien geprüft werden sollte (mindestens 7 bis 14 Tage) zu garantieren, dass potentiell interessante Mutanten nicht verworfen werden.
Das in dieser beschriebenen Protokoll ermöglicht es uns, ein Gen in einer relativ kurzen Zeit-Karte gegenüber dem klassischen Mapping-Verfahren. In der Tat sammelt eine ausreichende Anzahl von Einzelpersonen in der F2-Population für die GeneChip Array verwendet wird, erfordert eine relativ geringe Menge im Vergleich zu F 2-Pflanzen für die klassische Feinkartierung Verfahren benötigt werden. Es gibt jedoch wichtige Schritte, die beachtet werden sollte. Auswahl der F2-Population Proben ersichtlich ist oder nicht, die den Phänotyp sollte vorsichtig erfolgen, da die Zugabe von sogar einer kleinen Anzahl von Pflanzen mit der falschen Phänotyp, der durch Fehler befördert werden Fehler in das Endergebnis einzuführen. Dies ist vor allem auf die Tatsache, dass die F2-Population für die Grobkartierung sehr klein ist verbunden. Aus diesem Grund ETI Die Auswahl von Pflanzen,ihr zu zeigen oder nicht zeigen, der Phänotyp jedes Mal sollte am selben Tag nach der Keimung durchgeführt werden. Dies liegt daran, wie oben erwähnt, das Auftreten des Phänotyps zu Wachstum verbunden sein können. Ein weiterer wichtiger Punkt ist uns genau anschauen, die nächste Generation postalignment Daten, die von Programmen, die uns wichtige Informationen, wie der Prozentsatz der liest, die die Variante erzeugt wird. Der theoretische Wert für eine heterozygote Variante erwartet, etwa 50%, bedeutet dies, dass 50% der Sequenzen würde die Variante enthalten, während für eine homozygote Variante ist etwa 100%. Leider ist die Situation nach einer postalignment können weit von den theoretischen Wert, nämlich der Anteil der Aufrufe, die die Variante für heterozygote aus dem 20 bis 80%, während 60 bis 100% für homozygote 16 variieren. Deshalb, um den Schlüssel nicht zu Einzel-Nukleotid-Polymorphismen (SNP) verpassen, ist es wichtig, nicht zu Mutanten mit weniger als 100% Prozentsatz der Wiederverwendung ablegenAnzeigen für die Variante.
Offenlegungen
Wir haben nichts zu offenbaren.
Danksagungen
Wir danken für die Unterstützung von der Chemical Sciences, Geosciences and Biosciences Division, Office of Basic Energy Sciences, Office of Science, US Department of Energy (Vergabe-Nummer DE-FG02-91ER20021) und der National Science Foundation (MCB 0948584) (FB). Wir sind dankbar, dass Frau Karen Vogel für die Bearbeitung des Manuskripts.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenzes | Firma | Katalog-Nummer | |
| Ethylmethansulfonat | Sigma | M0880 | |
| NaOH | JT Baker | 3722-05 | |
| Murashige Skoog Basalmedium w Gamborg Vitamine | Phyto technolog laboratorie | M404 | |
| Phytagel | Sigma | P8169-1 kg | |
| RNeasy Mini Kit | Qiagen | 74104 | |
| Meister reinen Pflanzenblatt DNA Purification Kit | Epicentre | MPP92100 | |
| BioPrime DNA-Kennzeichnungssystem | Invitrogen | 18094-011 | |
| Alcohol 200 Proof | Dekan Laboratories Inc. | 2716 | |
| NaOAc | JT Baker | ||
| Gen-Chip-Arabidopsis ATH1Genom Array | Affymetrix | 900385 | |
| Falcon-Röhrchen 50 ml | Corning | 430290 | |
| Eppendorf-Röhrchen 1,5 ml | |||
| Filterpapier 90mm | Whatman | 1001090 | |
| Analysenwaage | Mettler Toledo AB54-S | na | |
| Taumelscheibe (Welle) Schüttler | Heidolph Polymax 1040 | na | |
| Zentrifugieren | Eppendorf 5417-R | na |
Referenzen
- Marti, L. A missense mutation in the vacuolar protein GOLD36 causes organizational defects in the ER and aberrant protein trafficking in the plant secretory pathway. The Plant journal : for cell and molecular biology. 63, 901-913 (2010).
- Stefano, G., Renna, L., Moss, T., McNew, J., Brandizzi, F. Arabidopsis the spatial and dynamic organization of the endoplasmic reticulum and Golgi apparatus is influenced by the integrity of the c-terminal domain of RHD3, a non-essential GTPase. The Plant Journal. , Forthcoming (2011).
- Kim, Y., Schumaker, K. S., Zhu, J. K. EMS mutagenesis of Arabidopsis. Methods in molecular biology. 323, 101-103 (2006).
- Maple, J., Moller, S. G. Mutagenesis in Arabidopsis. Methods in molecular biology. 362, 197-206 (2007).
- Greene, E. A. Spectrum of chemically induced mutations from a large-scale reverse-genetic screen in Arabidopsis. Genetics. 164, 731-740 (2003).
- Krieg, D. R. Ethyl methanesulfonate-induced reversion of bacteriophage T4rII mutants. Genetics. 48, 561-580 (1963).
- Kovalchuk, I., Kovalchuk, O., Hohn, B. Genome-wide variation of the somatic mutation frequency in transgenic plants. The EMBO journal. 19, 4431-4438 (2000).
- Boulaflous, A., Faso, C., Brandizzi, F. Deciphering the Golgi apparatus: from imaging to genes. Traffic. 9, 1613-1617 (2008).
- Borevitz, J. Genotyping and mapping with high-density oligonucleotide arrays. Methods in molecular biology. 323, 137-145 (2006).
- Konieczny, A., Ausubel, F. M. A procedure for mapping Arabidopsis mutations using co-dominant ecotype-specific PCR-based markers. The Plant journal : for cell and molecular biology. 4, 403-410 (1993).
- Bell, C. J., Ecker, J. R. Assignment of 30 microsatellite loci to the linkage map of Arabidopsis. Genomics. 19, 137-144 (1994).
- Hazen, S. P., Kay, S. A. Gene arrays are not just for measuring gene expression. Trends in Plant Science. 8, 413-416 (2003).
- Hazen, S. P. Rapid array mapping of circadian clock and developmental mutations in Arabidopsis. Plant Physiology. 138, 990-997 (2005).
- Bentley, D. R. Accurate whole human genome sequencing using reversible terminator chemistry. Nature. 456, 53-59 (2008).
- Weigel, D., Glazebrook, J. Arabidopsis. A Laboratory Manual. , Cold Spring Harbor Laboratory Press. Cold Spring Harbor, New York. (2002).
- Voelkerding, K. V., Dames, S., Durtschi, J. D. Next generation sequencing for clinical diagnostics-principles and application to targeted resequencing for hypertrophic cardiomyopathy: a paper from the 2009 William Beaumont Hospital Symposium on Molecular Pathology. J. Mol. Diagn. 12, 539-551 (2010).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten