Method Article
Umfassender Ansatz für die mikrobielle Isolierung aus Hidradenitis suppurativa-Tunneln
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Wir stellen eine Methode vor, mit der anspruchsvolle und anaerobe Organismen erfolgreich aus kutanen Sinusbahnen (Tunneln) in Geweben isoliert werden können, die von Patienten mit Hidradenitis Suppurativa exzidiert wurden.
Zusammenfassung
Hidradenitis Suppurativa (HS) ist eine schwächende Erkrankung, die durch schmerzhafte Knötchen und Abszesse gekennzeichnet ist, die zu den Nebenhöhlen (Tunneln) innerhalb der Hautschichten fortschreiten und erhebliche Beschwerden, übelriechenden Ausfluss, Entstellungen, Kontrakturen und Narbenbildung verursachen, die die Lebensqualität stark beeinträchtigen. HS wird mit Veränderungen des Hautmikrobioms in Verbindung gebracht, die sich auf die Immunregulation und die Abwehr der Haut gegen schädliche Bakterien auswirken. Trotz seiner Prävalenz ist der Beitrag des HS-Mikrobioms zur Krankheitspathologie und das begrenzte Ansprechen auf die Behandlung weitgehend unbekannt. Bisher haben mehrere 16S rRNA-Sequenzierungsstudien an HS-Gewebe nur eine Granularität auf Gattungsebene erreicht und eine Zunahme gramnegativer Anaerobier und eine Abnahme der Hautkommensalen festgestellt. Ein tieferes Verständnis der mikrobiellen Dysbiose bei Personen mit HS ist für die Optimierung von Behandlungsstrategien unerlässlich. Dies erfordert einen zweigleisigen Ansatz zur Bewertung des HS-Mikrobioms, einschließlich der Isolierung von Bakterienarten, die in translationalen Studien, die sich auf Hauterkrankungen konzentrieren, oft zu wenig genutzt werden. Die Isolierung einzelner Mikroorganismen aus HS-Gewebe ist entscheidend für die Aufklärung der Rolle von Bakterien in der HS-Pathogenese. Hier stellen wir reproduzierbare Methoden zur erfolgreichen Isolierung anaerober Krankheitserreger aus HS-Tunnelgewebe vor und stellen damit den ersten und wichtigsten Schritt zum Verständnis der bakteriellen Rolle bei HS dar. Diese Methode ebnet den Weg für die gezielte Erforschung der mikrobiellen Beiträge zur HS und für die Entwicklung effektiverer, personalisierter Behandlungsstrategien, die die komplexe mikrobielle Belastung durch diese chronische Erkrankung adressieren.
Einleitung
Hidradenitis Suppurativa (HS) ist eine häufige dermatologische Erkrankung, die durch Knötchen und Abszesse gekennzeichnet ist, die zu den Nebenhöhlen (Tunneln) fortschreiten, die sich in den dermalen und subkutanen Schichten der Haut bilden, erhebliche Schmerzen verursachen, eitrigen Ausfluss, Entstellungen und schwächende psychosoziale Folgen verursachen 1,2. HS betrifft überproportional Frauen und Personen mit farbiger Haut, die typischerweise in der späten Jugend oder im frühen Erwachsenenalter auftreten2. Die schweren körperlichen Symptome der Erkrankung werden durch ihre tiefgreifenden psychosozialen Auswirkungen wie Depressionen, Angstzustände und soziale Isolation verstärkt, die die Lebensqualität stark beeinträchtigen können3. HS-Tunnel, die im fortgeschrittenen Krankheitsstadium gebildet werden, verringern die Wahrscheinlichkeit, dass die Patienten auf die derzeit zugelassene biologische Therapie ansprechen, erheblich, und die chirurgische Exzision bleibt der einzige Behandlungsansatz 4,5.
Mehrere Studien haben das Mikrobiom im Zusammenhang mit HS-Tunneln charakterisiert und eine Prävalenz anaerober Krankheitserreger und eine verminderte Abundanz von kutanen Kommensalen gezeigt 6,7,8, wobei neuere Studien unter anderem Porphyromonas spp. (Typ I) und Prevotella spp. (IV) in Tunneln identifizierten9. Eine andere Studie fand eine Variation des HS-Mikrobioms durch die Tiefe der Gewebeprobenahme, was die Einzigartigkeit der mikrobiellen Zusammensetzung im Tunnelgewebe bestätigt10. Darüber hinaus wurde gezeigt, dass die Dysbiose die Immunantwort bei HS beeinflusst, was die Rolle von Mikroben bei der Krankheitspathogenese weiter impliziert 11,12,13,14. Obwohl eine verlängerte systemische Antibiotikabehandlung mit Clindamycin, Rifampicin und Tetracyclinen im Einsatz ist und eine Verringerung der Anzahl der Drainagetunnel bei betroffenen Patienten gezeigt hat15,16, sind die Daten zu antibiotikaresistenten anaeroben Erregern bei HS nach wie vor unbekannt. Bisher wurde nicht über die Isolierung von Bakterienstämmen aus HS-Tunneln berichtet, was Studien zu neuartigen antimikrobiellen Behandlungen und eine eingehende Bewertung des Beitrags von Krankheitserregern zur Krankheitspathogenese von HS einschränkt. Eine Standardisierung der Methoden zur Isolierung von Krankheitserregern würde nicht nur echte Einblicke in die bakterielle Rolle in der Pathogenese von HS ermöglichen, sondern auch eine Translation in Richtung gezielterer und verbesserter Interventionen ermöglichen.
Hier stellen wir eine Methode vor, mit der Mikroorganismen erfolgreich aus HS-Tunnelgewebe isoliert werden können. Die Isolierung einzelner Bakterienarten ist ein entscheidender erster Schritt, um ihre Rolle in der HS-Pathologie zu verstehen. Diese Methode ebnet den Weg für die gezielte Erforschung des mikrobiellen Beitrags zur HS und für die Entwicklung effektiverer, personalisierter Behandlungsstrategien, die sich mit bakteriellen Erregern bei dieser chronischen Erkrankung befassen.
Protokoll
Dieses Protokoll wurde vom Institutional Review Board (IRB) an der Universität von Miami genehmigt (Protokoll #20200187). Die Einwilligungserklärung wird von Patienten (n = 18, mittleres Alter ± Standardabweichung = 30,9 ± 9,4, 12 Frauen, 6 Männer), bei denen vor dem Eingriff HS-Tunnel diagnostiziert wurden, und/oder ihrem/ihren Erziehungsberechtigten(n) nach vorheriger Diskussion der Forschung eingeholt, um Bedenken oder Fragen auszuräumen.
1. Entnahme von Patientenproben
- Vor dem Umgang mit herausgeschnittenem Gewebe sollten strenge sterile Vorsichtsmaßnahmen getroffen werden, um das Risiko einer Kontamination zu minimieren. Die Person, die mit dem Gewebe umgeht, sollte einen Laborkittel, eine Gesichtsmaske und eine OP-Kappe tragen und sterile Handschuhe und sterile Instrumente verwenden, um das Gewebe zu handhaben.
- Zur Sterilitätskontrolle tauchen Sie eine vorautoklavierte Pinzette in das anaerobe Transportfläschchen, bevor Sie das Gewebe verarbeiten (Materialtabelle).
- Sammeln Sie Hautgewebe, das mit einem Skalpell oder CO2 -Laser chirurgisch reseziert wurde, aus betroffenen Bereichen von Patienten, bei denen HS-Tunnel diagnostiziert wurden, und legen Sie es sofort mit einer sterilen Pinzette in eine sterile Petrischale zur weiteren Verarbeitung.
- Identifizieren Sie im Operationszentrum oder in der Ambulanz Tunnel im exzidierten Gewebe, indem Sie die Haut mit einer präautoklavierten Pinzette untersuchen, wie in Abbildung 1A gezeigt.
- Entnehmen Sie unmittelbar nach der Gewebeexzision 6-mm-Stanzbiopsien durch die Vollhaut des identifizierten Tunnels und tauchen Sie sie in das halbfeste gepufferte Medium mit Reduktionsmitteln in die Glasfläschchen des anaeroben Systems ein, um das Überleben anaerober Bakterienarten für die weitere Analyse zu optimieren.
- Transportieren Sie das Tunnelgewebe in den anaeroben Fläschchen auf Eis ins Labor.
- Führen Sie die Beispieldokumentation wie unten beschrieben durch.
- Sammeln Sie am Tag des Eingriffs Patienteninformationen, einschließlich demografischer Daten, Alter, selbstberichteter ethnischer Zugehörigkeit/Rasse und aktueller Medikamente, ohne persönliche Identifikatoren. Die Körperstellen des resezierten Gewebes begleiten ein Körperdiagramm, da HS-Tunnel an mehreren Körperstellen auftreten können. Machen Sie vor und nach der Gewebeaufbereitung Fotos von exzidiertem Gewebe von beiden Seiten der Probe und dokumentieren Sie den Ort der Biopsie, die aus einem Tunnel erzeugt wurde. Bei der Ankunft im Labor protokollieren Sie die codierten Gewebeproben ohne persönliche Identifikatoren in einer elektronischen, sicheren Datenbank.
2. HS-Hautverarbeitung
- Einrichten und Plattieren
- Tragen Sie einen Laborkittel und Handschuhe und binden Sie die Haare, um eine Kontamination zu vermeiden. Vorwärmen von Blutagarplatten, einschließlich Laked Brucella Blutagar mit Kanamycin und Vancomycin (LKV) Agar, Trypticase Soja Agar (TSA II) 5% Schafblut (SB) Agar, Brain Heart Infusion (BHI) und Phenylethylalkohol Agar (PEA) mit 5% SB Agar auf 37 °C in einem Inkubator für 10 Minuten. Reinigen Sie die Biosicherheitswerkbank mit 70% Ethanol, bevor Sie das Gewebe verarbeiten.
- Sobald die Agarplatten erwärmt sind, beschriften Sie den Boden oder die Seite der Platte mit dem codierten Probennamen und dem Datum. Legen Sie autoklavierte Pinzetten, Skalpelle, vorsterilisierte Impfösen und Agarplatten in die Biosicherheitswerkbank.
- Entnehmen Sie die 6-mm-Stanzbiopsie mit einer sterilen Pinzette aus dem anaeroben Transportfläschchen, indem Sie die Pinzette in das Medium eintauchen. Schneiden Sie das Gewebe mit einem sterilen Skalpell schnell in kleine Stücke von jeweils ca. 1 mm2 . Das Gewebe in ein steriles Mikrozentrifugenröhrchen mit 500 μl verstärktem Clostridienmedium (RCM) geben und kurz vortexen.
HINWEIS: Die Homogenisierung wurde nicht verwendet, da die Proben über einen längeren Zeitraum der aeroben Umgebung ausgesetzt gewesen wären, was zu einer zusätzlichen Belüftung während des Prozesses und einem möglichen Verlust anaerober Bakterien hätte führen können. - Führen Sie mit einer neuen sterilen Schleife wiederholte Quadrantenstreifen der Gewebesuspension, die bakterielle Organismen enthält, auf die TSA II 5 % SB-, LKV-, PEA- mit 5 % SB- und BHI-Agarplatten durch.
- Stellen Sie unter Verwendung der restlichen gehackten Gewebesuspension Glycerinbrühen her, indem Sie 20 % v/v steriles Glycerin und 80 % v/v Gewebesuspension in ein Kryoröhrchen geben. Lagern Sie die Glycerinvorräte bei 80 °C. Bei eingeschränkter Lebensfähigkeit isolierter Bakterien können Gewebebestände verwendet werden, um die Isolierung zu wiederholen.
- Stellen Sie alle Platten in den Inkubator bei 37 °C in eine anaerobe Kammer, die mit einer CO2 -Packung umschlossen ist. Überprüfen Sie die Platten alle 2-3 Tage und erwarten Sie das Wachstum der Anaerobier 7-14 Tage nach der Beschichtung. Dokumentieren Sie die Morphologien der Kolonie, indem Sie Platten fotografieren.
- Gewebebanken
- Verwenden Sie eine 8-mm-Stanzbiopsie, um zusätzliche Hautproben in voller Dicke durch den Tunnel und außerhalb des Tunnels für die FFPE-Probe (Fixed Paraffin Embedded) und die histologische Bewertung zu entnehmen, da eine größere Gewebeprobe mit größerer Wahrscheinlichkeit den gesamten Tunnel erfasst.
- Bewahren Sie zusätzliche 6-mm-Stanzbiopsien für DNA (Snap Frozen), RNA (später in RNA) und Proteinisolierung (Snap Frozen) auf.
- Erhaltung der Kolonie
- Sobald die Kolonien auf den Originalplatten beobachtet sind, fotografieren Sie die Platten und notieren Sie sich die ausgeprägte Morphologie der Kolonie17. Im Allgemeinen sollten Sie mit 10-15 verschiedenen Koloniemorphologien rechnen (siehe Abbildung 1).
- Bereiten Sie vorgewärmte LKV-, BHI-, PEA-Platten mit 5 % SB und TSA II 5 % SB-Agarplatten vor (Materialtabelle). Beschriften Sie jedes Plattenetikett mit der Proben-ID, dem Ort der Biopsie und dem Datum.
- Nehmen Sie mit einer sterilen Pipettenspitze eine einzelne Kolonie auf und streichen Sie sie in einer Zickzackbewegung auf dieselbe Art von Platte, auf der sie nachgewiesen wurde. Verwenden Sie dann dieselbe Pipettenspitze mit der Kolonie als Vorlage für eine 16s-rDNA-PCR-Amplifikation und führen Sie die folgenden Schritte aus.
- Schritt 2.3.3 wird mit den Kolonien, die aus allen Arten von Agarplatten ausgewählt wurden, mit einer neuen sterilen Pipettenspitze und einer anderen singulären Kolonie mit der von der ursprünglichen Platte abweichenden Morphologie wiederholt. Auch hier gilt: Verwenden Sie unmittelbar nach dem Streifen die Pipettenspitze mit der verbleibenden Kolonie, um sie in eine spezielle PCR-Plattenvertiefung zu tauchen, da dies eine Vorlage für die PCR-Amplifikation bietet.
- Die Schritte 2.3.3 bis 2.3.4 werden für jede einzelne Kolonie wiederholt, bis alle Kolonien mit spezifischen Morphologien in allen vier Arten von Agarplatten gestreift und die entsprechenden PCR-Vertiefungen gleichzeitig inokuliert wurden.
- Lagern Sie die neu plattierten Agarplatten im Inkubator bei 37 °C in einer anaeroben Kammer, die mit einer CO2 -Packung umschlossen ist. Sobald die Bienenvölker gewachsen sind, stellen Sie die Reinheit der isolierten Kolonie sicher, bevor Sie die Bakterienbestände aufbewahren (siehe unten). Sind die plattierten Kolonien nicht gleichmäßig, so ist Schritt 2.3.3 zu wiederholen. Führen Sie den Neubeschichtungsprozess je nach Kolonenwachstum alle 4-5 Tage durch, bis die Völker einheitlich aussehen.
- Identifizierung von Bakterienarten durch 16s-rDNA-Kolonie-Amplikon-Sequenzierung
- Bereiten Sie eine PCR-Platte mit einem Mastermix vor, der aus 2,5 μl 10 μM 16S rDNA V1-V3 vorwärts (5' AGAGTTTGATTCCTGGCTCAG-3') und umgekehrten Primern (5' AGAGTTTGATTCCTGGCTCAG-3'; 10 μM), 12,5 μl 2x Master Mix Polymerase und 10 μl mikrobiellem DNA-freiem Wasser pro Vertiefung für ein Gesamtvolumen von 25 μl pro Kolonie besteht.
- Berechnen Sie das Volumen eines PCR-Mastermixes, das für die Amplifikation aus allen ausgewählten Kolonien, der Negativkontrolle (kein Template) und der Positivkontrolle erforderlich ist - jeder Laborstamm kann verwendet werden; Wir verwenden häufig Staphylococcus aureus USA300.
- Bringen Sie eine PCR-Platte zur Biosicherheitswerkbank und decken Sie die Vertiefungen ab, um eine Kontamination zu vermeiden. Verwenden Sie für die Beschichtung eine einzelne Kolonie aus einer sterilen Pipettenspitze. Suspendieren Sie die Kolonie sofort wieder durch rigoroses Schwenken in die dafür vorgesehene Vertiefung mit dem vorgefüllten Mastermix. Verwenden Sie dieselbe Pipettenspitze sowohl für die Subkultivierung als auch für die Probenahme für die PCR.
- Schritt 2.4.3 ist für alle singulären HS-Kolonien und die Positivkontrolle zu wiederholen.
- Führen Sie die PCR mit dem folgenden Protokoll durch: 95 °C für 5 Minuten für die anfängliche Denaturierung, die zur Lyse der ausgewählten Kolonie erforderlich ist, gefolgt von 40 Zyklen mit 95 °C für 15 s, 54 °C für 1 Minute, die Verwendung des abschließenden 4 °C-Zyklus ist optional. Ein Thermocycler kann auch mit den gleichen Programmeinstellungen verwendet werden.
- Führen Sie eine Gelelektrophorese mit amplifizierten qPCR-Produkten durch, um die Größe des Amplikons zu überprüfen, die voraussichtlich 311 bp betragen wird (Abbildung 2). Reinigen Sie erfolgreich ein amplifiziertes 16s-rDNA-Fragment mit dem PCR-Aufreinigungskit gemäß den Anweisungen des Herstellers.
- Senden Sie das gereinigte PCR-Produkt für die ribosomale 16S-RNA-Sanger-Sequenzierung mit 16S rDNA V1-V3-Forward- und Reverse-Primern.
- Bakterien-Vorräte
HINWEIS: Die anspruchsvollen Anaerobier sind dafür bekannt, dass sie auf den Agarplatten nur begrenzt lebensfähig sind.- Um die Konservierung des Stammes vor dem Erhalt von Sequenzierungsergebnissen zu gewährleisten, die die Artidentität bestätigen, Agarplatten aus einer einzigen Kolonie zu generieren und die Reinheit zu bestätigen, um Bestände zu erzeugen. Generieren Sie Vorräte direkt aus der Agarplatte, parallel zur Optimierung der Wachstumsbedingungen in den flüssigen Medien.
- Um den Fond aus der in Schritt 2.3 erzeugten Platte vorzubereiten, verwenden Sie eine sterile 6-mm-Impfschlaufe, um so viele gleichmäßige Kolonien wie möglich zu entnehmen. Anschließend werden die Bakterien in 800 μl RCM mit 20 % sterilem Glycerin in Kryoröhrchen kräftig resuspendiert und bei -80 °C gelagert, um einen Bakterienvorrat zu bilden.
HINWEIS: Dieser Schritt gewährleistet die rechtzeitige Konservierung anspruchsvoller Krankheitserreger, bevor die Charakterisierung durch Sequenzierungsdaten erfolgt. - Optimierung des Wachstums der Flüssigkultur und der Generierung von Stamm aus einer einzigen Kolonie, sobald die Klassifizierung des Stammes bestätigt ist.
Ergebnisse
In dieser Studie beschreiben wir ein Protokoll zur Isolierung und Charakterisierung von anaeroben Bakterien aus HS-Tunneln. Dieses Protokoll ist neuartig und zeichnet sich dadurch aus, dass es das Potenzial bietet, die Funktion und Virulenz dieser Bakterien unter Verwendung von in vitro-, ex vivo- und in vivo-Hautinfektionsmodellen zu testen, um unser Verständnis des mikrobiellen Beitrags zur Pathogenese von HS zu verbessern. Zunächst identifizierten wir die Tunnel aus resezierter Haut, indem wir sie mit einer sterilen Pinzette untersuchten (Abbildung 1A). Wir haben Tunnel von Knötchen und Abszessen unterschieden, da letztere sich nicht unter der Hautoberfläche verbinden. Nehmen Sie 6-mm-Stanzbiopsien in voller Dicke durch Tunnel und konservieren Sie sie unter anaeroben Bedingungen für Bakterienkulturen, wobei LKV-Agar zur vorwiegenden Selektion von gramnegativen Bakterien wie Porphyromonas sp. und Prevotella sp. verwendet wird. TSA II 5% SB-Agarplatten ermöglichen das Wachstum sowohl gramnegativer als auch grampositiver Bakterien, einschließlich Peptoniphilus sp. und Streptococcus sp. Die resultierenden Kolonien unterscheiden sich in ihrer Morphologie, einschließlich verschiedener Farben (z. B. schwarz, gelb, weiß usw.), Größen und Transparenzen, mit oder ohne Hämolysezone (Abbildung 1B). Kolonien, die innerhalb von 3 Tagen wachsen, stellen mit geringerer Wahrscheinlichkeit fakultative Anaerobier dar und umfassen in der Regel Staphylococcus - und Corynebacterium-Arten . Anaerobier können nach 7-14 Tagen Wachstum auftreten und in der Regel sehr kleine Kolonien aufweisen, die entweder pigmentiert oder durchscheinend sind. Daher können Kolonien, die früh nach der anfänglichen Plattierung innerhalb von 2-4 Tagen auftreten, charakterisiert und erhalten werden, aber dies sind in der Regel Arten, die nicht repräsentativ für das tunnelspezifische Mikrobiom sind und häufig aus der oberflächlichen Haut und den vom Tunnel getrennten Knötchen isoliert werden. Zu den repräsentativen obligatorischen und fakultativen anaeroben Spezies, die mittels Amplikon-Sequenzierung isoliert und klassifiziert wurden, gehörten Prevotella sp, Peptoniphilus sp, Porphyromonas sp, Streptococcus anginosus, Staphylococcus lugdunensis, Parvimonas micra, Proteus mirabillis und Actinotignum schaalii. Die Isolierung von Bakterien aus der HS-Haut, ohne Tunnelstrukturen (außerhalb des Tunnels), ist ebenfalls von Bedeutung, da sie Unterschiede in beiden Gewebequellen lokalisieren und die Selektion tunnelspezifischer Bakterien erleichtern kann. Wichtig ist, dass die Sterilitätskontrolle des Verfahrens unerlässlich ist und kein Wachstum hervorruft.
Die Histologie diente auch zur Verifizierung klinisch identifizierter Tunnel, da die HS-Morphologie komplex und auch innerhalb einzelner Proben sehr variabel ist18. Die Tunnelhistologie zeigte eine variable Epithelisierung im Lumen, die, wenn vorhanden, dem darüber liegenden Oberflächenepithel ähnelte, was mit der zuvor beschriebenen Literaturübereinstimmt 12 (Abbildung 3). Keratinreiches Material wurde auch im Lumen gefunden, das als Oberfläche für die bakterielle Anheftung dienen kann, zusammen mit dichtem Entzündungsinfiltrat, das eine robuste Entzündung widerspiegelt, ein Kennzeichen der HS-Tunnel12. In einigen Proben war das Tunnelepithel auch grob sichtbar und zeigte sich als Flecken aus verdicktem Gewebe entlang des Lumens. Die Entnahme von mehreren 8 mm Stempeln in voller Dicke war machbar und lieferte eine repräsentative histologische Auswertung, da sie die Erfassung des gesamten Tunnels und der umliegenden Strukturen ermöglichte (Abbildung 3).

Abbildung 1: Isolierung von mikrobiellen Stämmen aus HS-Tunneln. (A) Ein Tunnel wurde in der Hautprobe durch Sondierung mit einer sterilen Pinzette identifiziert. (B) Es sind repräsentative Blutagarplatten dargestellt, die aus dem Tunnelgewebe gestreift sind, das in einem anaeroben Transportfläschchen transportiert wird. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Repräsentative 16S rDNA-Amplikon-Gelelektrophorese. Die Proben 1-6 repräsentieren 16S rDNA-PCR-Produkte aus unterschiedlichen Bakterienkolonien, die mittels Gelelektrophorese analysiert wurden, wobei jede Spur einer einzigartigen Kolonie entspricht. Die Negativkontrolle enthält keine DNA, was die Spezifität der PCR-Reaktion bestätigt. Das Vorhandensein spezifischer 16S rDNA-Banden deutet auf eine erfolgreiche Amplifikation aus den Bakterienkolonien hin. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
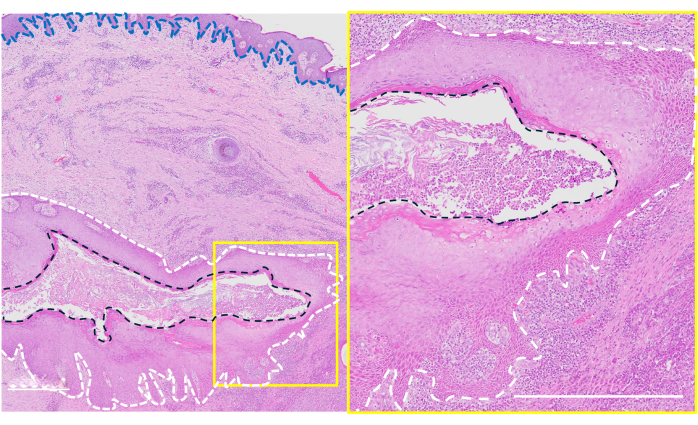
Abbildung 3: Repräsentative Histologie mit epithelialisiertem Tunnel. Die schwarze gestrichelte Linie umreißt das Tunnellumen; die weiße gestrichelte Linie umreißt das Tunnelepithel; Die blaue Linie zeigt die Basalmembran zwischen der oberflächlichen Hautepidermis und der Dermis. Maßstabsleiste = 500 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
In dieser Studie stellen wir eine neuartige Methode zur Isolierung und Aufrechterhaltung von Bakterien vor, die HS-Tunnel besiedeln. Diese reproduzierbare Methode wird nicht nur eine detaillierte Charakterisierung der in diesen pathologischen Strukturen vorhandenen Stämme ermöglichen, sondern auch nachfolgende funktionelle Studien zur Erforschung der Rolle spezifischer Mikroben in der Pathogenese von HS. Zu den kritischen Schritten des Protokolls gehören die Gewebeentnahme und der Transport in anaeroben Medien, was die Konservierung lebensfähiger, anspruchsvoller Mikroorganismen aus HS-Tunneln erleichtert. Wie bei jeder Studie gibt es nach wie vor Einschränkungen, einschließlich derjenigen, die mit chirurgischen Eingriffen verbunden sind, wie z. B. die Verwendung von Hautdesinfektionsmitteln und Lokalanästhesie, die sich auf das Mikrobiom des Gewebes auswirken können, ebenso wie die verschiedenen Umwelteinflüsse der Patienten, hygienische Praktiken, Medikamente, die Schwere der Erkrankung und Komorbiditäten19. In der Erkenntnis, dass die während des Laserverfahrens erzeugte Wärme die mikrobielle Lebensfähigkeit beeinträchtigen könnte, wurden Bakterien aus HS-Gewebe außerhalb der Exzisionsstelle isoliert, um die potenziellen Auswirkungen auf die Zusammensetzung des Mikrobioms zu vermeiden. Patienten mit HS werden auch häufig mit topischen und/oder systemischen Antibiotika sowie immunsuppressiven Medikamenten behandelt, die die Hautbesiedlung beeinflussen können20. Diese Faktoren machen eine Standardisierung bei der Probenentnahme und -verarbeitung18 umso wichtiger. Wir erkennen auch, dass ein bestimmter Prozentsatz der Bakterien nicht kultivierbar ist. Die Protokolle sind jedoch so optimiert, dass sie die repräsentativsten Spezies isolieren, die in HS-Mikrobiomstudien identifiziert wurden. Unsere Ergebnisse stimmen mit den früheren Mikrobiomstudien an HS-Tunnelnüberein 6,7,8, da zu den in dieser Studie isolierten Bakterien aus HS-Tunneln Prevotella sp, Peptoniphilus sp, Porphyromonas sp, Staphylococcus lugdunensis, Streptococcus anginosus, Parvimonas micra, Proteus mirabillis und Actinotignum schaalii gehörten. Darüber hinaus könnte Gewebe, das für die DNA-Isolierung konserviert wurde, zur Validierung durch Mikrobiomstudien verwendet werden, entweder durch Metagenom- oder Amplikon-Sequenzierungsansatz. Mit diesem standardisierten Ansatz hoffen wir auch, die Gewebeentnahme und Reproduzierbarkeit zukünftiger Mikrobiomstudien bei HS zu verbessern.
Die beschriebene Methode schafft die Voraussetzung, um unser Verständnis der Beiträge von HS-Erregern zu verschiedenen Aspekten der Krankheitspathogenese, einschließlich Wirt-Pathogen-Interaktionsstudien, zu erweitern. Neuere Studien haben die Reaktion humaner Keratinozyten auf kommerziell erhältliche ATCC-Stämme von vorherrschenden Spezies, die in HS12,13 identifiziert wurden, charakterisiert. Die verwendeten ATCC-Stämme wurden jedoch aus Sputum, zerviko-fazialen Läsionen und Empyemen isoliert und nicht aus HS-Gewebe, was den Bedarf an zuverlässigeren HS-Modellen unterstreicht, die den menschlichen Zustand nachahmen, insbesondere solche, die HS-Tunnel repräsentieren21. Diese Methode ermöglicht auch die Einlagerung von Bakterienstämmen, die aus HS-Tunneln isoliert wurden, was das Potenzial hat, in größerem Maßstab funktionelle Tests zu ermöglichen und die Rolle der Erreger bei der Entwicklung und dem Fortschreiten von HS-Tunneln aufzuklären. Wichtig ist, dass die Isolierung von Tunnelerregern auch eine weitere Bewertung der antimikrobiellen Empfindlichkeit und der Prävalenz von Antibiotikaresistenzen ermöglicht, die im Zusammenhang mit dem häufigen Einsatz von topischen und systemischen Antibiotika wie Doxycyclin und Clindamycin zur Behandlung von Krankheitsschüben bekannt sind22. Tunnelpathogene können auch zu einem unterschiedlichen Ansprechen der Patienten auf die biologische Therapie beitragen14. Daher besteht ein großes translationales Potenzial für Studien mit HS-Tunnel-Erregern, einschließlich der Optimierung der Therapieauswahl und der Entwicklung erregerspezifischer Behandlungen, um den Antibiotikaeinsatz zu verringern und dadurch das Risiko von Antibiotikaresistenzen zu verringern. Darüber hinaus kann die Analyse der Zusammensetzung des Mikrobioms in HS Verbesserungen der diagnostischen und prognostischen Instrumente zur Beurteilung des Krankheitsverlaufs und der Rezidivrisiken ermöglichen. Zusammenfassend haben wir eine praktische und reproduzierbare Methode zur Isolierung und Konservierung von Bakterien aus HS-Tunneln für weitere Charakterisierungen und funktionelle Studien vorgestellt.
Offenlegungen
Die Autoren berichten von keinem Interessenkonflikt.
Danksagungen
Diese Arbeit wurde von R01AR083385 (IP, MTC, HLT), P50MD017347 (TG, IP, HLT) und dem Danby Research Grant (TG) der HS Foundation unterstützt. Diese Arbeit wurde zusätzlich durch den NIH-Zuschuss 1S1OD023579-01 für das VS120 Slide Scanner Haus an der Miller School of Medicine der University of Miami Miller School of Medicine Analytical Imaging Core Facility unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 6mm punch biospy | INTEGRA | 33-36 | Other suppliers can be used |
| 8mm punch biospy | INTEGRA | 33-37 | Other suppliers can be used |
| Agarose | Sigma Aldrich | A9539 | Other suppliers can be used |
| Anaerobic Chambers | BD | 260672 | |
| Anaerobic Transport Media | Anaerobic Systems | AS-911 | |
| Brain heart Infusion Agar | Anaerobic Systems | AS-6426 | |
| CO2 gaspak | BD | 260678 | |
| Difco Reinforced Clostridial Medium | BD | 218081 | |
| Glycerol | SIGMA | G5516-1L | Other suppliers can be used |
| Hard shell PCR plates | BIO-RAD | HSP9601 | Other suppliers can be used |
| Incubator | VWR | Symphony | Any callibrated incubator can be used |
| Inoculation loops | VWR | 76544-926 | Other suppliers can be used |
| LKV agar | HARDY Diagnostics | A60 | |
| Microbial DNA-Free Water | Qiagen | 338132 | |
| Nunc CryoTube | Thermo scientific | 377267 | Other suppliers can be used |
| PCR (CFX Connect Real Time System) | BIO-RAD | CFX Connect Optics Module | Regular Themocycler can be used |
| PEA agar | HARDY Diagnostics | A93 | |
| Q5 High Fidelity 2X Master Mix | BioLabs | M0492S | |
| QIAquick PCR Purification Kit | QIAGEN | 28104 | |
| Reinforced clostridia media | BD | 218081 | |
| Thin Forceps | Millipore Sigma | F4017 | Other suppliers can be used |
| Trypticase Soy Agar (TSA II) with 5% sheep blood | Thermo scientific | 221261 |
Referenzen
- Gonzalez-Lopez, M. A. Hidradenitis suppurativa. Med Clin. 162 (4), 182-189 (2024).
- Revankar, R., Murrell, D. F., Murase, J. E. Shedding light on the impact of hidradenitis suppurativa on women and their families: A focus of the international journal of women's dermatology. Int J Womens Dermatol. 7 (5Part B), 661-663 (2021).
- Gooderham, M., Papp, K. The psychosocial impact of hidradenitis suppurativa. J Am Acad Dermatol. 73 (5 Suppl 1), S19-S22 (2015).
- Frew, J. W., et al. Clinical response rates, placebo response rates, and significantly associated covariates are dependent on choice of outcome measure in hidradenitis suppurativa: A post hoc analysis of pioneer 1 and 2 individual patient data. J Am Acad Dermatol. 82 (5), 1150-1157 (2020).
- Bechara, F. G., et al. Efficacy and safety of adalimumab in conjunction with surgery in moderate to severe hidradenitis suppurativa: The sharps randomized clinical trial. JAMA Surg. 156 (11), 1001-1009 (2021).
- Williams, S. C., Frew, J. W., Krueger, J. G. A systematic review and critical appraisal of metagenomic and culture studies in hidradenitis suppurativa. Exp Dermatol. 30 (10), 1388-1397 (2021).
- Guet-Revillet, H., et al. The microbiological landscape of anaerobic infections in hidradenitis suppurativa: A prospective metagenomic study. Clin Infect Dis. 65 (2), 282-291 (2017).
- Riverain-Gillet, E., et al. The surface microbiome of clinically unaffected skinfolds in hidradenitis suppurativa: A cross-sectional culture-based and 16s rrna gene amplicon sequencing study in 60 patients. J Invest Dermatol. 140 (9), 1847-1855.e6 (2020).
- Ring, H. C., et al. The microbiome of tunnels in hidradenitis suppurativa patients. J Eur Acad Dermatol Venereol. 33 (9), 1775-1780 (2019).
- Pardo, L. M., et al. Bacterial microbiota composition in hidradenitis suppurativa differs per skin layer. J Invest Dermatol. 144 (2), 426-430.e5 (2024).
- Jiang, S. W., Whitley, M. J., Mariottoni, P., Jaleel, T., Macleod, A. S. Hidradenitis suppurativa: Host-microbe and immune pathogenesis underlie important future directions. JID Innov. 1 (1), 100001(2021).
- Navrazhina, K., et al. Epithelialized tunnels are a source of inflammation in hidradenitis suppurativa. J Allergy Clin Immunol. 147 (6), 2213-2224 (2021).
- Williams, S. C., et al. Gram-negative anaerobes elicit a robust keratinocytes immune response with potential insights into hs pathogenesis. Exp Dermatol. 33 (5), e15087(2024).
- Chopra, D., et al. Innate immunity and microbial dysbiosis in hidradenitis suppurativa - vicious cycle of chronic inflammation. Front Immunol. 13, 960488(2022).
- Van Straalen, K. R., et al. External validation of the ihs4-55 in a european antibiotic-treated hidradenitis suppurativa cohort. Dermatology. 239 (3), 362-367 (2023).
- Molinelli, E., et al. Systemic antibiotic therapy in hidradenitis suppurativa: A review on treatment landscape and current issues. Antibiotics. 12 (6), 978(2023).
- Andersson, T., Lood, R. 16S rRNA sequencing: A PCR-based technique to identify bacterial species. JoVE Sci Edu Database Microbiol. , e10510(2023).
- Nebo, I. D., Frew, J. W., Gudjonsson, J. E., Petukhova, L. Tissue comparability and bias in hidradenitis suppurativa transcriptomic studies. Proc Natl Acad Sci. 121 (23), e2404503121(2024).
- Naik, H. B., Piguet, V. Standardizing hidradenitis suppurativa skin microbiome research: The methods matter. J Invest Dermatol. 140 (9), 1688-1690 (2020).
- Ocker, L., Abu Rached, N., Seifert, C., Scheel, C., Bechara, F. C. Current medical and surgical treatment of hidradenitis suppurativa-a comprehensive review. J Clin Med. 11 (23), 7240(2022).
- Zouboulis, C. C., Hou, X., Von Waldthausen, H., Zouboulis, K. C., Hossini, A. M. Hs 3d-seboskin model enables the preclinical exploration of therapeutic candidates for hidradenitis suppurativa/acne inversa. Pharmaceutics. 15 (2), 619(2023).
- Koumaki, D., et al. Antimicrobial resistance trends in hidradenitis suppurativa lesions. J Clin Med. 13 (14), 4246(2024).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten