Method Article
Ein schneller, einfacher Workflow zur Quantifizierung externer adulter Drosophila-Strukturen
In diesem Artikel
Zusammenfassung
Hier stellen wir einen schnellen, kostengünstigen Workflow für die hochauflösende Bildgebung von erwachsenen Drosophila-Augen vor, um Musterbildung und Wachstumsdefekte zu quantifizieren. Wir beschreiben unser Protokoll für die Probenvorbereitung durch Punktmontage, hochauflösende Bildaufnahme und Bildanalyse.
Zusammenfassung
Das Drosophila-Facettenauge ist ein präzise gemustertes Gewebe, das molekulare Mechanismen und biologische Prozesse aufgedeckt hat, die die Morphogenese vorantreiben. Es handelt sich um eine einfache Struktur von sich wiederholenden Einheitsaugen, die als Ommatidien bezeichnet werden und zur Charakterisierung genetischer Interaktionen und Genfunktionen verwendet wird. Mutationen, die die Augenarchitektur beeinflussen, können leicht erkannt und analysiert werden. Daher wird dieses System häufig in unterversorgten Institutionen eingesetzt. Eine weitere phänotypische Analyse umfasst häufig ein Rasterelektronenmikroskop (REM), um Bilder mit hoher Vergrößerung zu erzeugen, die für die quantitative Analyse geeignet sind. REMs sind jedoch teuer und erfordern teure Reagenzien. Die Probenvorbereitung erstreckt sich über Tage; Und oft benötigen sie Vollzeitpersonal für die Probenvorbereitung und die Wartung der Instrumente. Dies schränkt ihren Nutzen bei unterfinanzierten Institutionen oder bei Haushaltskürzungen ein. In der Entomologie ist der Einsatz hochauflösender digitaler Bildgebungstechnologie eine gängige Praxis zur Identifizierung und Charakterisierung von Arten. In diesem Artikel wird eine Methode beschrieben, die Strategien kombiniert und eine hochauflösende digitale Bildgebung von adulten Drosophila-Strukturen und eine quantitative Analyse mit der offenen Software ImageJ ermöglicht. Der Workflow ist eine schnelle und studierendenfreundliche Alternative, die die Einschränkungen unterfinanzierter und unterfinanzierter Forschungseinrichtungen mit einem kostengünstigen und schnellen Ansatz für die quantitative phänotypische Analyse behebt.
Einleitung
Drosophila melanogaster ist ein leistungsfähiger genetischer Modellorganismus, der seit Jahrzehnten zur Aufklärung molekularer Signalwege und zellulärer Verhaltensweisen eingesetzt wird. Viele der evolutionär konservierten Signalwege, die für die multizelluläre Entwicklung essentiell sind, wurden erstmals in Drosophila identifiziert und ihr Wirkmechanismus definiert. Etwa 65-75% aller humanen krankheitsassoziierten Gene haben Orthologe in Drosophila 1,2. Das erwachsene Drosophila-Auge ist ein wichtiges Modell, das unvoreingenommene genetische Screenings ermöglicht hat, die die Entdeckung wichtiger konservierter Gene erleichtern, die an menschlichen Krankheiten beteiligt sind, einschließlich Krebs 3,4, Neurodegeneration5 und Stoffwechselstörungen6.
Das Drosophila-Auge besteht aus ~800 Augeneinheiten, die als Ommatidien bezeichnet werden und präzise in einem sechseckigen Muster über die Oberfläche des erwachsenen Auges angeordnet sind7. Jedes Ommatidium besteht aus acht Photorezeptorneuronen, die eine unterschiedliche Position innerhalb eines asymmetrischen Trapezes einnehmen. Diese werden von vier nicht-neuralen Zapfenzellen und zwei primären Pigmentzellen unterstützt, die Linsen und Pseudokegel sezernieren, um Licht auf die lichtempfindlichen Rhabdomeren der Photorezeptorneuronen zu fokussieren. Benachbarte Ommatidien sind durch eine einzige Reihe interommatidialer Gitterzellen getrennt, die aus sekundären Pigmentzellen, tertiären Pigmentzellen und mechanosensorischen Borstenkomplexen bestehen 8,9,10.
Störungen in der Augenentwicklung sind bei erwachsenen Augen als Zunahme oder Verminderung der Augengröße, abnormale Häufigkeit oder Struktur von Linsen oder Borsten oder als "raues Auge" sichtbar, bei dem die normalerweise invariante sechseckige Musterung gestört ist, so dass eine Reihe von Ommatidien nicht mehr über die Oberfläche des Auges verfolgt werden kann. Diese Phänotypen können mit Hilfe von Präpariermikroskopen auf der Ebene des groben Gewebes bewertet werden. Die detaillierte Analyse von Phänotypen umfasst traditionell die Rasterelektronenmikroskopie, gefolgt von einer quantitativen Bildanalyse11. Die Rasterelektronenmikroskopie erfordert jedoch teure Instrumente, teure Reagenzien, eine tagelange Probenvorbereitung und oft Vollzeitpersonal für die Wartung.

Abbildung 1: Arbeitsablauf für die Bildgebung adulter Drosophila-Strukturen . (A) Adulte Drosophila in 70 % Ethanol sammeln und fixieren. (B) Bereiten Sie die Proben für die Bildgebung vor, indem Sie sie punktuell montieren und an Stiften befestigen. (C) Erfassen Sie hochauflösende Bilder durch Fokus-Stacking und Integration. (D) Quantifizieren Sie Bilder mit FIJI. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
In diesem Artikel wird ein Arbeitsablauf vorgestellt, der relativ kostengünstig ist, eine kurze Probenvorbereitungszeit hat, leicht auf einem 3-Fuß-Labortisch eingerichtet werden kann, keine gefährlichen Materialien erfordert und eine langlebige Ergänzung für Drosophila-Forschungslabore sein könnte (Abbildung 1). Die Punktbefestigung ist eine entomologische Technik, die zur Lufttrocknung und Konservierung von kleinen, weichen Insekten wie Drosophila12 verwendet wird. Bei dieser Methode werden Mikroskopobjektive mit hochauflösenden DSLR-Kameras kombiniert, um effektive Vergrößerungen von 10x bis 1.000x zu erzielen. Die begrenzte Schärfentiefe, die der Makrofotografie eigen ist, wird durch Focus Stacking überwunden: Das Zusammenfügen einer Reihe von Bildern, wobei sich die Brennebene durch das interessierende Exemplar bewegt13. Diese Methode liefert hochauflösende Bilder, die sich für die Quantifizierung von Phänotypen eignen und leicht für andere interessante Strukturen wie Flügel, Bein, Thorax und Abdomen angepasst werden könnten. Für den Bildanalyse-Workflow wird das kostenlose Bildanalyseprogramm FIJI (NIH ImageJ) verwendet. Diese Methodik macht die Probenvorbereitung, hochauflösende Bildgebung und Analyse für Studenten und Wissenschaftler an unterversorgten Institutionen zugänglich.
Protokoll
1. Entnahme und Fixierung adulter Drosophila
- Setzen Sie Drosophila-Kreuzungen ein oder wählen Sie Stämme aus und legen Sie sie in Fläschchen mit Fliegenfutter. Inkubieren Sie die Fläschchen bei der gewünschten Temperatur (in der Regel 25 °C), bis sich die Fliegen entwickelt haben und die erwachsenen Tiere sich einschließen (~10-14 Tage bei 25 °C).
- Betäuben Sie die Fliegen mit CO2 und legen Sie sie auf ein CO2 -Pad.
- Sortieren Sie die Fliegen mit einer Feder und wählen Sie die Individuen mit dem gewünschten Phänotyp (z.B. gerade Flügel) aus. Stellen Sie einen Federfliegensortierer her, indem Sie eine Gänsefeder so zuschneiden, dass sie in das konische Ende einer serologischen 1-ml-Pipette passt.
- Bereiten Sie ein 1,7-ml-Mikrozentrifugenröhrchen mit 1 ml 70%igem Ethanol vor. Legen Sie die ausgewählten Fliegen in das Mikrozentrifugenröhrchen und legen Sie sie auf Eis. Lagern Sie die Mikrozentrifugenröhrchen über Nacht bei 4 °C (Abbildung 2A).
HINWEIS: Bewahren Sie Fliegen nicht länger als 24 Stunden in Ethanol auf. Die langfristige Lagerung von Fliegen in 70%igem Ethanol führt zum Verlust von Augen- und Körperpigmenten.
2. Probenvorbereitung durch Punktmontage
HINWEIS: Drosophila sind Insekten mit weichem Körper, die spröde werden und an der Luft zusammenfallen; Daher erfordert dieses Protokoll, dass die Proben am selben Tag abgebildet werden, an dem sie montiert werden. Arbeiten Sie in kleinen Sets von ~5 Fliegen auf einmal, um Probenverluste zu vermeiden. Erhöhen Sie die Anzahl der Proben in einem Satz basierend auf der Effizienz. Proben, die mehr Zeit vor der Bildgebung benötigen, können durch eine zunehmende Konzentrationsreihe von Hexamethyldisilazan (HMDS) dehydriert werden14.
- Schneiden Sie kleine dreieckige Spitzen (7,1 mm x 2,7 mm) aus 65 lb Archivkarton mit einem speziellen Punktlocher aus. Bereiten Sie Punkte vor, indem Sie die Spitze (schmalste 25%) mit der Dumont #5 Feinpinzette in einen 90°-Winkel biegen (Abbildung 2B).
- Entfernen Sie mit der Dumont #5 Feinpinzette die Fliegen aus den Mikrozentrifugenröhrchen (Schritt 1.4). Tupfen Sie die Fliegen vorsichtig mit fusselfreiem Laborgewebe ab, um überschüssiges Ethanol zu entfernen. Positionieren Sie jede Fliege auf der linken Seite auf einer Karteikarte unter einem Präpariermikroskop.
HINWEIS: Entfernen Sie die Fliegen aus der Röhre, indem Sie sich an einer anatomischen Struktur festhalten, die nicht der interessierende Bereich ist - bei der Abbildung des Kopfes halten wir die Proben am Flügel oder Bein. Halten Sie die Proben nicht am Bauch, da diese Struktur verwendet wird, um die Fliege auf die Kartenspitze zu kleben. - Bereiten Sie den Hautkleber vor und stellen Sie seine Konsistenz auf die gewünschte Viskosität ein. Mischen Sie 1-2 Tropfen Hautkleber mit 1-2 Tropfen deionisiertem (DI) Wasser und mischen Sie es mit einer Transferpipette auf einer Karteikarte. Nehmen Sie eine vorbereitete Kartenspitze am breiten Ende mit einer Pinzette auf und geben Sie eine kleine Menge des verdünnten Klebers auf die gebogene Spitze der Spitze, indem Sie ihn in die Leim-Wasser-Mischung tupfen (Abbildung 2C).
HINWEIS: Der Kleber sollte streichbar, aber nicht flüssig sein. - Bringen Sie die gebogene Spitze der Spitze auf die vordere Seite des rechten Abdomens um die Bauchsegmente 2-3 an (Abbildung 2C). Bevor der Kleber trocknet, nehmen Sie leichte Anpassungen am Hosenschlitz vor, so dass die vorder-hintere Achse des Fliegens senkrecht zur gebogenen Spitze der Spitze steht.
- Stecken Sie einen Befestigungsstift Nr. 3 in das breite Ende der Kartenspitze (Abbildung 2D) und befestigen Sie ihn an einem Insektenstiftblock (Abbildung 2E). Beschriften Sie jeden Pin oder jede Pin-Reihe mit dem entsprechenden Genotyp.

Abbildung 2: Probenvorbereitung. (A) Adulte Drosophila werden nach phänotypischen Markern sortiert und in markierten Mikrozentrifugenröhrchen mit 70 % Ethanol auf Eis gesammelt. Die Fliegen werden bei 4° C über Nacht gelagert. (B) Papierkartenspitzen werden vorbereitet, indem das schmale Ende mit einer Pinzette #5 um 90° vom Rest der Karte gebogen wird. (C) Fliegen werden aus den Röhren geborgen und kurz an der Luft trocknen gelassen. Hautkleber wird auf das kleine, gefaltete Ende der vorbereiteten Kartenspitze aufgetragen und an den Bauchsegmenten 2-3 auf die adulte Fliege geklebt. (D) Die Proben werden mit einem Identifikationsetikett auf einen Insektenstift #3 aus Edelstahl montiert. (E) Gesteckte Proben werden auf einer Probenplatine gelagert, bis sie für die Bildaufnahme bereit sind. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
3. Hochauflösende Fokus-Stacking-Bildaufnahme
- Erfassen Sie hochauflösende Fotos von Fliegenaugen mit einem zusammengebauten und individuell angepassten Focus-Stacking-Bildgebungssystem.
- Nehmen Sie Fotos mit einem DSLR-Kameragehäuse mit einem 70-200-mm-Teleobjektiv auf, das über einen 77-mm-Objektivadapter mit einem 20-fach-Apo-Mikroskopobjektiv verbunden ist.
- Stellen Sie sicher, dass die Probe durch einen Diffusor mit einem Blitz beleuchtet wird (Abbildung 3).
- Steuern Sie die Z-Positionierung mit einem Stackshot-Controller und einer Makroschiene.
- Verbinden Sie die Kamera, den Blitz und den motorisierten Tisch mit einem robusten Stativ aus eloxiertem Aluminium.
- Positionieren Sie jede punktgebundene Probe auf einem universellen Tisch-Gimbal, wobei der Kopf so ausgerichtet ist, dass das Auge zur Linse zeigt. Passen Sie die Kopfposition an, indem Sie den Kopf vorsichtig mit der Pinzette bewegen.
ACHTUNG: Große und schnelle Anpassungen können zu einer versehentlichen Enthauptung führen. - Wenn die Kamera an einen Laptop angeschlossen ist, passen Sie die Aufnahmeeinstellungen in der Software an. Fotografieren Sie Proben bei 20-facher Vergrößerung mit diesen Einstellungen: Blitzleistung 1/32, Verschlusszeit 1/200, Blende F2,8 und ISO 400. Stellen Sie sicher, dass alle Autofokus- und Bildstabilisierungsfunktionen deaktiviert sind.
HINWEIS: Diese Einstellungen sorgen für ein ausgewogenes Verhältnis zwischen optimaler Blitzbeleuchtung, Verschlusszeit und Schärfentiefe. Sie müssten für andere Vergrößerungen und/oder Linsenkombinationen angepasst werden. - Legen Sie den Speicherort für den resultierenden Bildstapel (10-50 Bilder) im gewünschten Dateiordner fest. Stellen Sie sicher, dass genügend Speicherkapazität für die Bilder vorhanden ist (~8,5 MB pro Bild).
- Passen Sie die Einstellungen für den Fokusstapel an der Stackshot-Steuereinheit im Auto-Distance-Modus an.
- Stellen Sie die Schrittweite auf 5 μm ein und berechnen Sie die Anzahl der Schritte , indem Sie die Start - und Stopppositionen des Fokusstapels festlegen.
- Betrachten Sie die Probe im LiveView-Modus und mit der Kamera im Auto-Shoot-Modus , um die Start- und Stopppositionen zu identifizieren.
- Bewegen Sie die Schiene so, dass der nächstgelegene Teil der Probe scharf ist ( Startposition einstellen), und bewegen Sie sich dann dorthin, wo das am weitesten entfernte interessierende Merkmal scharf ist ( Endposition einstellen).
- Versetzen Sie die Kamera wieder in den manuellen Aufnahmemodus und starten Sie die Bildaufnahme über die Stackshot-Steuereinheit.
HINWEIS: Die Bildaufnahmezeit hängt von der Größe der Probe ab. Je größer die Schärfentiefe, die für große Proben erforderlich ist, desto mehr Schichten werden in den Bildstapel aufgenommen, was die Gesamtaufnahmezeit verlängert. - Öffnen Sie Dateien in der referenzierten Focus-Stacking-Software. Generieren Sie ein gestapeltes Bild, indem Sie auf Stapeln | Ausrichten & Stapeln aller (PMax).
- Speichern Sie das endgültige Bild als .tif Datei auf der Festplatte des Computers, indem Sie auf Datei | Ausgabebild speichern.
HINWEIS: Abhängig von der Auflösung der gestapelten Image-Datei und der Anzahl der abgebildeten Exemplare können große externe Festplatten (1 TB) für die Image-Sicherung erforderlich sein. Bei diesem Protokoll sind gestapelte Bilder jeweils ca. 100 MB groß, bevor sie komprimiert werden.

Abbildung 3: Bildaufnahme. (A) Bildgebende Vorrichtung mit Teilen mit der folgenden Bezeichnung: a) Gehäuse der DSLR-Kamera; b) Teleobjektiv; c) 20x Apo Mikroskop Objektiv und Adapter; d) Blitz; e) Linsen- und Kuppeldiffusoren; f) Stackshot-Controller, Makroschiene und Drehtisch; g) Universelles Bühnen-Gimbal; h) Stativ. (B) Bildgebendes Gerät mit eingesetztem Lichtdiffusor. (C) Nahaufnahme einer montierten Probe in Position für die Bildgebung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
4. FIJI-Analyse-Workflow zur Berechnung der Augenpartie bei Erwachsenen
- Für die Bildanalyse laden Sie die FIJI-Software15 von der ImageJ.net-Website herunter.
- Wählen Sie Bilder für die Analyse aus, bei denen das Auge zentriert und ausgerichtet ist, mit angemessener Beleuchtung und minimaler peripherer Unschärfe, was auf eine korrekte Ausrichtung mit der Kamera hinweist.
- Kalibrieren Sie den Bildmaßstab.
- Laden Sie das Bild der Maßstabsleiste für eine 20-fache Vergrößerung herunter, die 500 μm entspricht. Alternativ können Sie zum Zeitpunkt der Bildaufnahme ein Lineal mit denselben Einstellungen fotografieren. Öffnen Sie das Bild in der FIJI-Software.
- Messen Sie die Länge der Maßstabsleiste (Abbildung 4A). Verwenden Sie das Werkzeug "Gerade Linie ", um die Linie genau nachzuzeichnen. Klicken Sie auf Analysieren | Messen. Dieser Pixelabstand entspricht 500 μm (Abbildung 4B).
- Berechnen Sie Pixel pro Mikrometer. Verwenden Sie diese Option, um Pixelmessungen in Mikrometermessungen umzuwandeln.
- Öffnen Sie die gestapelte Bilddatei in FIJI (Abbildung 4C).
- Wählen Sie Lupe aus der Symbolleiste, um den Fokusbereich zu vergrößern. Versuchen Sie, den Bildschirm mit dem Auge und der unmittelbar umgebenden Nagelhaut des Kopfes auszufüllen (Abbildung 4D).
- Wählen Sie Freihandwerkzeug aus der Symbolleiste aus. Umreißen Sie den Netzhautbereich so genau wie möglich und folgen Sie dabei den Konturen der äußersten Reihe von Ommatidien (Abbildung 4E). Um einen Teil der Auswahl zu entfernen, halten Sie die Optionstaste gedrückt und wählen Sie die Pixel aus, die Sie entfernen möchten. Um der Auswahl etwas hinzuzufügen, halten Sie die Wahltaste und die Umschalttaste gedrückt und wählen Sie die Pixel aus, die Sie hinzufügen möchten.
- Um die Fläche zu berechnen, wählen Sie Analysieren | Messen Sie über das obere Menü (Abbildung 4F). Es erscheint ein neues Fenster mit den Parametern Fläche, Mittelwert, Minimum und Maximum . Kopieren Sie diese Daten und fügen Sie sie in eine Tabelle ein, um sie zu dokumentieren und von Pixeln in Mikrometermessungen umzuwandeln.
- Führen Sie entsprechende statistische Analysen durch.
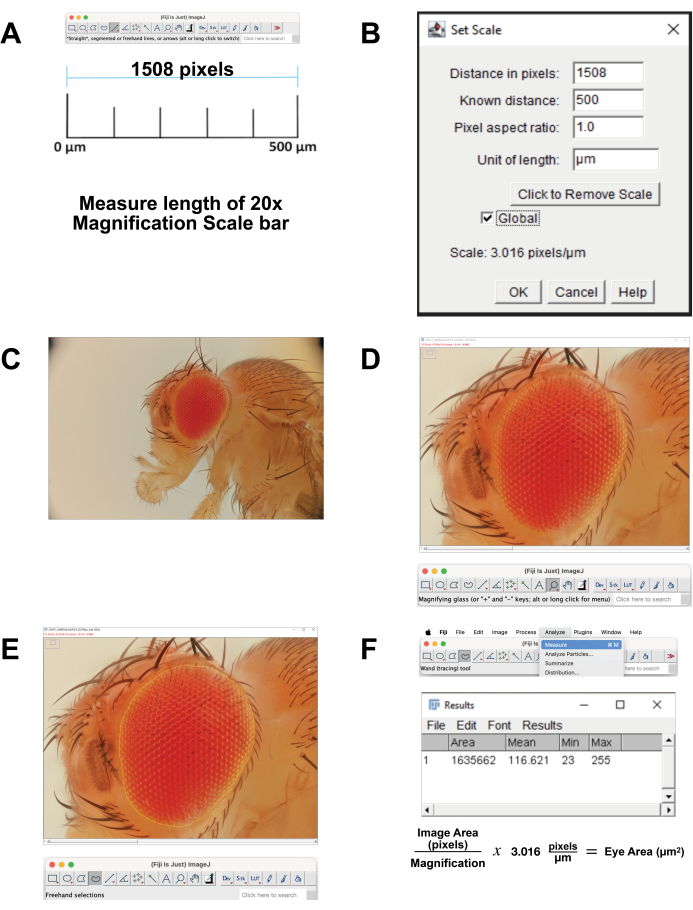
Abbildung 4: Bildanalyse in Fidschi. (A) Skalieren Sie das Originalbild. Laden Sie das Kalibrierbild herunter und messen Sie die Länge des 500-μm-Stabs. (B) Passen Sie die Skalierung mit der Funktion "Skalierung einstellen " an. (C) Öffnen Sie das gestapelte Bild. (D) Vergrößern Sie das Bild so, dass das Auge zentriert und nahezu im Vollbildmodus angezeigt wird. (E) Verwenden Sie das Freihandauswahl-Werkzeug , um das Auge an der Grenze zwischen der äußersten Reihe der Ommatidien und der umgebenden Nagelhaut zu umranden. (F) Messen Sie die Fläche innerhalb des ausgewählten Bereichs wird berechnet, indem Sie auf Analysieren | Messen | Fläche. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergebnisse
Das Drosophila-Auge ist ein hervorragendes Modellsystem für die Untersuchung von Gewebemustern, Wachstumskontrolle und Zelltod. Wir haben kürzlich eine Studie veröffentlicht, in der untersucht wurde, wie der intrazelluläre pH-Wert (pHi) das Gewebewachstum beeinflusst. Zunächst etablierten wir ein genetisches System, bei dem die Überexpression des Natrium-Protonen-Austauschers DNhe2 (das Ortholog des Säugetier-NHE1) im sich entwickelnden Auge zu Musterungsdefekten und erhöhter Proliferation führt16. Eine erhöhte Proliferation mit höherem pHi wird bei allen Spezies von Hefen bis hin zu Säugetieren beobachtet17. Wir beobachteten jedoch auch, dass das erwachsene Auge bei erhöhtem pHi im Vergleich zu den Kontrollen kleiner erschien.
Eine quantitative Analyse der adulten Augenpartie wurde durchgeführt, um zu bestimmen, wie eine erhöhte DNhe2-Expression die Gewebegröße im Drosophila-Auge verringert. Wir haben zunächst Bilder mit niedriger Auflösung mit einem Okulareinsatzmikroskop aufgenommen (Abbildung 5A), aber diese waren von unzureichender Qualität, um quantitative Messungen durchzuführen. Insbesondere werden die Augenränder nicht scharf gestellt, was eine genaue Messung der Augenpartie verhindert.
Wir maßen die Fläche jedes Auges in hochauflösenden Bildern von erwachsenen Drosophila-Augen (Abbildung 5B). GMR>DNhe2-exprimierende Fliegen hatten signifikant kleinere Augen (Mittelwert 74,28μm 2) als die genetische Hintergrundkontrolle mit1118 Fliegen (Mittelwert 125,83 μm2) oder die Treiberkontrolle GMRGAL4 heterozygote Fliegen (Mittelwert 142,49 μm2), was einer Flächenreduzierung von 41 % bzw. 48 % entspricht (Abbildung 5C). Diese Abnahme der Augengröße trotz erhöhter Proliferation könnte auf einen erhöhten Zelltod zurückgeführt werden.
Bekannte Autophagie-Gene, einschließlich Atg1, das die Phagophorbildung induziert, wurden in genetischen Interaktionsstudien verwendet (Abbildung 5). Die Auswirkungen auf die Augengröße bei Erwachsenen wurden mit hochauflösender Fotografie und Flächenanalyse quantifiziert (Abbildung 5B). Die Augengröße war bei GMR>DNhe2-Fliegen, die für Atg13 heterozygot waren, im Vergleich zu GMR>DNhe2 allein wiederhergestellt (129,9μm 2 vs. 74,28 μm2) und war ähnlich wie bei Kontrollen (Abbildung 5C). In Kombination mit der molekularen Markeranalyse unterstützen unsere Ergebnisse einen erhöhten autophagischen Zelltod bei höherem pHi.

Abbildung 5: Rettung einer verminderten Netzhautgröße in Verbindung mit einem erhöhten intrazellulären pH-Wert mit verminderter Autophagie. (A) Niedrig aufgelöste Bilder von erwachsenen Fliegenaugen, die mit einem Okulareinsatzmikroskop aufgenommen wurden. (B) Hochauflösende Bilder von adulten Fliegenaugen, die zur Quantifizierung der Augengröße für die folgenden Genotypen verwendet wurden: w1118; GMRGAL4; GMR>DNhe2; GMRGAL4; Atg13; GMR>DNhe2; Atg13. (C) Die durchschnittlich gemessenen Augengrößen bei Erwachsenen waren: w 1118 (125,83 μm2, N = 7); GMRGAL4 (142,49 μm2, N = 9) und GMR>DNhe2 (74,28 μm2, N = 6), GMRGAL4; Atg13 (146,4 μm2, N = 15); GMR>DNhe2; Atg13 (129,9 μm2, N = 19) und wird durch die Darstellung von Medianwerten mit Interquartilsabständen angezeigt. Die statistische Signifikanz wurde durch ungepaarte t-Tests mit Welch-Korrektur bestimmt. Die Daten in B und C wurden zuvor veröffentlicht18. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Unsere Schlussfolgerungen werden durch die Probenvorbereitung, die hochauflösende Bildgebung und die hier beschriebene Analyse untermauert. Diese Methoden ermöglichten eine präzise und quantitative Beschreibung der Größe adulter Drosophila-Augen mit verschiedenen Genotypen. Wir verwenden diesen Ansatz weiterhin, um andere genetische Interaktionen zu bewerten, die die Mechanismen der pH-abhängigen Wachstumskontrolle aufklären werden.
Diskussion
Hier beschreiben wir eine Methode zur Probenvorbereitung, hochauflösenden Bildgebung und Analyse von adulten Drosophila-Strukturen . Das Drosophila-Auge ist ein genetisch manipulierbares Modellsystem, das wichtige Einblicke in die molekularen Mechanismen von Krankheiten wie Krebs19, Neurodegeneration20 und Stoffwechselerkrankungen21 geliefert hat. Insbesondere werden "Avatare" von Krebspatienten generiert, bei denen transgene Drosophila mit onkogenen Mutationen erzeugt werden, die für das Hochdurchsatz-Wirkstoffscreening verwendet werden können22. Obwohl wir uns auf das erwachsene Auge konzentrieren, kann dieses Protokoll angepasst werden, um andere interessante Strukturen wie Flügel, Bein, Thorax und Abdomen zu analysieren.
Feinmotorische Fähigkeiten sind bei mehreren kritischen Schritten dieses Protokolls erforderlich. Die Probenvorbereitung der adulten Drosophila durch Punktmontage erfordert Geduld und Übung. Wir raten Anfängern, das Point-Mounten zu üben, bevor sie seltene oder wertvolle Proben vorbereiten. Es gibt mehrere kritische Schritte, die das Üben der Feinmotorik und sorgfältige Aufmerksamkeit erfordern.
Die ersten kritischen Schritte im Protokoll sind die Vorbereitung der Kartenpunkte und das Auftragen des Klebers, um den Kartenpunkt auf den Bauch zu kleben (Protokollschritt 2.4-2.5). Es ist wichtig, genau die richtige Menge Klebstoff aufzutragen, da die anatomischen Zielstrukturen (z. B. der Kopf) mit Klebstoff bedeckt sein können, wenn zu viel Klebstoff aufgetragen wird oder nicht die richtige Konsistenz vorliegt. Die Probe muss ebenfalls in einer geeigneten Position verklebt werden, wobei die vorder-hintere Achse senkrecht zum Befestigungsstift steht. Der zweite kritische Schritt ist die Positionierung des Kopfes für die hochauflösende Bildgebung (Protokollschritt 3.2). Entscheidend ist, dass der Kopf so ausgerichtet ist, dass das Auge auf die Kameralinse gerichtet ist. Um den Kopf zu verstellen, werden kleine Anpassungen mit einer Pinzette vorgenommen. Sanfte und schrittweise Bewegungen sind entscheidend, da es bei großen oder kräftigen Anpassungen zu einer versehentlichen Enthauptung kommen kann. Ein zusätzlicher Schritt, bei dem Feinmotorik erforderlich ist, ist die Verwendung des Freihand-Auswahlwerkzeugs aus der FIJI-Symbolleiste (Protokollschritt 4.6). Hier ist es entscheidend, so nah wie möglich an der äußeren Reihe der Ommatidien des Auges zu verfolgen. Diese Präzision ist der Schlüssel für rigorose und reproduzierbare Flächenmessungen des Auges (Abbildung 4E).
Bildgebende Verfahren wie die Rasterelektronenmikroskopie (REM) sind nach wie vor der primäre Ansatz, um äußere Strukturen sichtbar zu machen und zu analysieren. REM-Protokolle erfordern jedoch, dass der Benutzer mit gefährlichen Chemikalien umgeht und mehrere Instrumente bedient, darunter Trockner an kritischen Punkten, Sputterbeschichtungsgeräte und das REM selbst. Die Probenvorbereitung dauert mehrere Tage und erfordert eine intensive Schulung und Geschicklichkeit. Eine große Herausforderung für akademische Einrichtungen sind die finanziellen Investitionen und das Engagement für die Wartung des Instruments, und unserer Erfahrung nach wird Personal mit Erfahrung in der Wartung und dem Betrieb dieser Geräte in der Regel nicht auf institutioneller Ebene unterstützt. Unser Protokoll senkt die Kosten und eliminiert die Exposition gegenüber gefährlichen Chemikalien auf Kosten der Bildgebung mit geringerer Auflösung im Vergleich zu REM.
Das in dieser Studie beschriebene System war ein vormontiertes Paket, aber sein modularer Charakter ermöglicht es, die Kosten zu senken, indem einzelne Komponenten durch selbst hergestellte Geräte (z. B. Probentische, Blitzdiffusoren), alternative Marken oder Modelle oder Geräte, die möglicherweise bereits im Labor vorhanden sind, ersetzt werden. Während der Anschluss von Mikroskopobjektiven an Makrokameras bei Hobbyfotografen und professionellen Fotografen immer beliebter wird, kann bei verschiedenen Kombinationen von Objektiven und Objektiven ein gewisses Maß an Versuch und Irrtum erforderlich sein. Um das hier beschriebene System nachzubilden, muss ein unendlich korrigiertes Mikroskopobjektiv mit einem entsprechend gewindeförmigen Adapter an eine Tubuslinse (hier Teleobjektiv) angeschlossen werden. Eine weitere Überlegung beim Austausch von Komponenten besteht darin, sicherzustellen, dass der Arbeitsabstand des Objektivs mit den Abmessungen der Kamerahalterung, der Probenhalterung und der Makrofokussierschiene kompatibel ist. Schließlich ist die hier ausgewählte Focus-Stacking-Software kommerziell, aber es gibt viele kostengünstige oder kostenlose Focus-Stacking-Lösungen für die Nachbearbeitung, einschließlich ImageJ-Plugins.
Während dieses Protokoll Übung erfordert, um die Techniken zur Probenhandhabung zu beherrschen, ist es für die Erfassung und Analyse hochauflösender Bilder für eine detaillierte phänotypische Analyse konzipiert. Die hier vorgestellten Verfahren stellen eine schnelle und kostengünstige Alternative zu herkömmlichen bildgebenden Verfahren dar. Durch die Verwendung von Freeware-basierten digitalen Bildgebungstechniken für die quantitative Analyse ist unsere Methodik zugänglich und erfordert keine kostspieligen Softwarekäufe oder Updates. Die Bildgebung erfordert einige Kosten und Mühe, sollte aber nach der Etablierung eine langlebige Ergänzung für Drosophila-Labore sein. Diese Methode liefert hochauflösende Bilder, die sich für die Quantifizierung von Phänotypen eignen und leicht für andere interessante Strukturen wie Flügel, Bein, Thorax und Abdomen angepasst werden könnten. Diese Methodik macht die Probenvorbereitung, hochauflösende Bildgebung und Analyse für Studenten und Wissenschaftler an unterversorgten Institutionen zugänglich.
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Die Autoren danken den Mitgliedern des Grillo-Hill pHly-Labors für die Gespräche und die Unterstützung. Wir danken Tim Andriese, Randy Kirschner, Kitty (Ngoc-Huong) Nguyen, Marco Parent, Jonny Shaloub und Librado Veliz für die hervorragende technische Unterstützung. Diese Arbeit wurde durch NIH SC3GM132049 und 1R16GM153640 Awards (BKGH), einen CSU Biotechnology Faculty-Student Research Award (LM und BKGH) und Startkapital des College of Science und des Department of Biological Sciences an der San José State University (FJL) unterstützt. Besondere Erwähnung verdient Bernd Becker für seinen Einfallsreichtum und seine Unterstützung während dieses Prozesses. Wir danken der BioIcons (https://bioicons.com/)-Community für die Bereitstellung hochwertiger Symbole für unsere Abbildungen und insbesondere Serviere für das Pipettensymbol und DBCLS für die in Abbildung 1 und Abbildung 2 verwendeten Drosophila-, Pinzetten- und Desktop-Elektronenmikroskop-Symbole, die unter CC-BY 4.0 Unported lizenziert sind. Wir danken auch der SciDraw (https://scidraw.io/) Community für die Bereitstellung hochwertiger Icons für unsere Figuren, insbesondere Diogo Losch De Oliveira (doi.org/10.5281/zenodo.3925953), die unter der Creative Commons 4.0 Lizenz (CC-BY) lizenziert sind.
Materialien
| Name | Company | Catalog Number | Comments |
| 1 mL serological pipette | ThermoFisher Scientific | 170353N | |
| 1.7 mL microcentrifuge tubes | Genesee Scientific | 24-282LR | |
| 20x Apo Microscope Objective | Mitutoyo Corp. | 378-804-3 | |
| Archival 65 lb cardstock | Neenah, Inc. | 91901 | |
| Canon EF 70-200 mm USM II telephoto lens | Canon | 3044C002 | |
| Canon EOS 6D Mark II DSLR Camera Body | Canon | 1897C002 | |
| Diffuser Dome | Macroscopic Solutions | PA-DIF-GIM-SM | |
| Diffuser for Mitutoyo M Plan APO Objectives | Macroscopic Solutions | mitutoyo-diffusers | |
| Drosophila vials and plugs | Genesee Scientific | 32-117BF | |
| Dumont #5 fine-tip forceps | Fisher Scientific | NC9889584 | |
| Goose feathers | Amazon | B01CMMJI6U | |
| Heavy-Duty Anodized Aluminum Tripod | Really Right Stuff, LLC | TFA-32G | |
| Kimwipes | Fisher Scientific | 06-666A | lint-free lab tissue |
| Levenhuk M1000 Plus Digital Camera | Levenhuk | 70358 | |
| No. 3 mounting pin | Indigo Instruments | 33414-3 | |
| Nutri-Fly Bloomington Drosophila media | Genesee Scientific | 66-113 | fly food |
| Point-Punch | M.C. Mieth Manufacturing, Inc. | 448Detail | |
| Screwknob Clamp | Really Right Stuff, LLC | SK-Clamp | For attaching the macro rail to the tripod |
| Stackshot Controller and Macro Rail | Cognisys Inc. | ST3X_100_BUNDLE | |
| Step-down Ring Adapter | RAF Camera | 763461174207 | Lens adapter to connect the microscope objective to the camera lens |
| Titebond Glue | Franklin International | 5013 | |
| Yongnuo YN-24-EX Macro Twin Lite Flash | Shenzhen Yongnuo Photographic Equipment Co. | YN-24EX | |
| Software | |||
| Canon EOS Utility (v. 3.16.1). | Canon | acquisition software | |
| FIJI | National Institutes of Health | Fiji is released as open source under the GNU General Public License. FIJI Version 2.14.0/1.54f | |
| GraphPad Prism | GraphPad Software, Boston, Massachusetts USA | Prism Version 10.3.1 | |
| Zerene Stacker (v.1.04) | Zerene Systems, LLC | Focus Stacking Software |
Referenzen
- Rubin, G. M., et al. Comparative genomics of the eukaryotes. Science. 287 (5461), 2204-2215 (2000).
- Ugur, B., Chen, K., Bellen, H. J. Drosophila tools and assays for the study of human diseases. Dis Model Mech. 9 (3), 235-244 (2016).
- Hodgson, J. A., Parvy, J. -. P., Yu, Y., Vidal, M., Cordero, J. B. Drosophila larval models of invasive tumorigenesis for in vivo studies on tumour/peripheral host tssue interactions during cancer cachexia. Int J Mol Sci. 22 (15), 8317 (2021).
- Lam Wong, K. K., Verheyen, E. M. Metabolic reprogramming in cancer: mechanistic insights from Drosophila. Dis Model Mech. 14 (7), dmm048934 (2021).
- Bonini, N. M. A perspective on Drosophila genetics and its insight into human neurodegenerative disease. Front Mol Biosci. 9, e1060796 (2022).
- Drummond-Barbosa, D., Tennessen, J. M. Reclaiming Warburg: using developmental biology to gain insight into human metabolic diseases. Development. 147 (11), dev189340 (2020).
- Ready, D. F., Hanson, T. E., Benzer, S. Development of the Drosophila retina, a neurocrystalline lattice. Dev Biol. 53 (2), 217-240 (1976).
- Johnson, R. I. Hexagonal patterning of the Drosophila eye. Dev Biol. 478, 173-182 (2021).
- Weasner, B. P., Kumar, J. P. The early history of the eye-antennal disc of Drosophila melanogaster. Genetics. 221 (1), iyac041 (2022).
- Pichaud, F., Casares, F. Shaping an optical dome: The size and shape of the insect compound eye. Semin Cell Dev Biol. 130, 37-44 (2022).
- Oster, I. I., Crang, R. E. Scanning electron microscopy of Drosophila mutant and wild type eyes. Trans Am Microsc Soc. 91 (4), 600-602 (1972).
- Gibb, T. J., Oseto, C. . Insect collection and identification: Techniques for the field and laboratory. , (2019).
- Mertens, J., Roie, M. V., Merckx, J., Dekoninck, W. The use of low cost compact cameras with focus stacking functionality in entomological digitization projects. ZooKeys. 712, 141-154 (2017).
- Brown, B. V. A further chemical alternative to critical-point-drying for preparing small (or large) flies. Fly Times. 11, 10 (1993).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat Method. 9 (7), 676-682 (2012).
- Grillo-Hill, B. K., Choi, C., Jimenez-Vidal, M., Barber, D. L. Increased H+ efflux is sufficient to induce dysplasia and necessary for viability with oncogene expression. eLife. 4, e03270 (2015).
- White, K. A., Grillo-Hill, B. K., Barber, D. L. Cancer cell behaviors mediated by dysregulated pH dynamics at a glance. J Cell Sci. 130 (4), 663-669 (2017).
- Peralta, J., et al. Drosophila Nhe2 overexpression induces autophagic cell death. Mol Biol Cell. 35 (7), br13 (2024).
- Munnik, C., Xaba, M. P., Malindisa, S. T., Russell, B. L., Sooklal, S. A. Drosophila melanogaster: A platform for anticancer drug discovery and personalized therapies. Front Genet. 13, 949241 (2022).
- Nitta, Y., Sugie, A. Studies of neurodegenerative diseases using Drosophila and the development of novel approaches for their analysis. Fly. 16 (1), 275-298 (2022).
- Pletcher, R. C., et al. A genetic screen using the Drosophila melanogaster TRiP RNAi collection to identify metabolic enzymes required for eye development. G3: Genes|Genomes|Genetics. 9 (7), 2061-2070 (2019).
- Bangi, E., et al. A personalized platform identifies trametinib plus zoledronate for a patient with KRAS-mutant metastatic colorectal cancer. Sci Adv. 5 (5), eaav6528 (2019).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten