Method Article
Isolierung von Mitochondrien für die mitochondriale Superkomplexanalyse aus kleinen Gewebe- und Zellkulturproben
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt eine Technik zur Analyse von respiratorischen Superkomplexen, wenn nur geringe Mengen an Proben zur Verfügung stehen.
Zusammenfassung
In den letzten Jahrzehnten haben die gesammelten Beweise für die Existenz von respiratorischen Superkomplexen (SCs) unser Verständnis der Organisation der mitochondrialen Elektronentransportkette verändert, was zu dem Vorschlag des "Plastizitätsmodells" führte. Dieses Modell postuliert die Koexistenz unterschiedlicher Anteile von SCs und Komplexen in Abhängigkeit vom Gewebe bzw. dem zellulären Stoffwechselstatus. Die dynamische Natur der Anordnung in SCs würde es den Zellen ermöglichen, die Nutzung der verfügbaren Brennstoffe und die Effizienz des Elektronentransfers zu optimieren, die Erzeugung reaktiver Sauerstoffspezies zu minimieren und die Fähigkeit der Zellen zur Anpassung an Umweltveränderungen zu begünstigen.
In jüngerer Zeit wurden Anomalien in der SC-Assemblierung bei verschiedenen Krankheiten wie neurodegenerativen Erkrankungen (Alzheimer und Parkinson), Barth-Syndrom, Leigh-Syndrom oder Krebs berichtet. Die Rolle von Veränderungen der SC-Assemblierung bei der Krankheitsprogression muss noch bestätigt werden. Dennoch ist die Verfügbarkeit ausreichender Mengen an Proben zur Bestimmung des SC-Montagestatus oft eine Herausforderung. Dies geschieht bei Biopsie- oder Gewebeproben, die klein sind oder für mehrere Analysen geteilt werden müssen, bei Zellkulturen, die ein langsames Wachstum aufweisen oder aus mikrofluidischen Geräten stammen, bei einigen Primärkulturen oder seltenen Zellen, oder wenn die Wirkung bestimmter kostspieliger Behandlungen analysiert werden muss (mit Nanopartikeln, sehr teuren Verbindungen usw.). In diesen Fällen ist eine effiziente und einfach anzuwendende Methode erforderlich. In dieser Arbeit wird ein Verfahren vorgestellt, das geeignet ist, angereicherte mitochondriale Fraktionen aus kleinen Mengen von Zellen oder Geweben zu erhalten, um die Struktur und Funktion mitochondrialer SCs durch native Elektrophorese zu analysieren, gefolgt von In-Gel-Aktivitätsassays oder Western Blot.
Einleitung
Superkomplexe (SCs) sind supramolekulare Assoziationen zwischen einzelnen Komplexen der Atmungskette 1,2. Seit der erstmaligen Identifizierung von SCs und der Beschreibung ihrer Zusammensetzung durch die Gruppe von Schägger 2,3, die später von anderen Gruppen bestätigt wurde, wurde festgestellt, dass sie die respiratorischen Komplexe I, III und IV (CI, CIII bzw. CIV) in unterschiedlichen Stöchiometrien enthalten. Es können zwei Hauptpopulationen von SC definiert werden: solche, die CI enthalten (und entweder CIII allein oder CIII und CIV) und mit sehr hohem Molekulargewicht (MW, beginnend mit ~1,5 MDa für die kleinere SC: CI + CIII2) und solche, die CIII und CIV, aber nicht CI enthalten, mit viel kleinerer Größe (wie CIII2 + CIV mit ~680 kDa). Diese SCs koexistieren in der inneren Mitochondrienmembran mit freien Komplexen, ebenfalls in unterschiedlichen Anteilen. Während CI also hauptsächlich in seinen assoziierten Formen vorkommt (d. h. in SCs: ~80 % im Rinderherz und mehr als 90 % in vielen menschlichen Zelltypen)3, ist CIV in seiner freien Form sehr häufig (mehr als 80 % im Rinderherzen), wobei CIII eine ausgewogenere Verteilung aufweist (~40 % in seiner häufigeren freien Form, als Dimer, im Rinderherz).
Während ihre Existenz heute allgemein anerkannt ist, wird ihre genaue Rolle immer noch diskutiert 4,5,6,7,8,9,10. Nach dem Plastizitätsmodell können je nach Zelltyp oder Stoffwechselstatus unterschiedliche Anteile an SCs und einzelnen Komplexen existieren 1,7,11. Dieser dynamische Charakter der Anordnung würde es den Zellen ermöglichen, den Verbrauch verfügbarer Brennstoffe und die Effizienz des oxidativen Phosphorylierungssystems (OXPHOS) als Reaktion auf Umweltveränderungen zu regulieren 4,5,7. SCs könnten auch dazu beitragen, die Erzeugungsrate der reaktiven Sauerstoffspezies zu kontrollieren und an der Stabilisierung und dem Umsatz einzelner Komplexeteilzunehmen 4,12,13,14. Modifikationen des SC-Assemblierungsstatus wurden im Zusammenhang mit verschiedenen physiologischen und pathologischen Situationen15,16 und mit dem Alterungsprozess17 beschrieben.
So wurden Veränderungen in den SC-Mustern in Hefe in Abhängigkeit von der für das Wachstum verwendeten Kohlenstoffquellebeschrieben 2 und in kultivierten Säugetierzellen, wenn Glukose durch Galaktose substituiert wird4. Modifikationen wurden auch in der Leber von Mäusen nach dem Fasten berichtet8 und in Astrozyten, wenn die mitochondriale Fettsäureoxidation blockiert ist18. Darüber hinaus wurde eine Abnahme oder Veränderung von SCs und OXPHOS bei Barth-Syndrom19, Herzinsuffizienz20, mehreren metabolischen21 und neurologischen 22,23,24 Erkrankungen und verschiedenen Tumoren 25,26,27,28 festgestellt. Ob diese Veränderungen in der SC-Assemblierung und -Spiegel eine primäre Ursache oder sekundäre Effekte in diesen pathologischen Situationen darstellen, wird noch untersucht15,16. Unterschiedliche Methoden können Aufschluss über den Aufbau und die Funktion von SCs geben; Dazu gehören Aktivitätsmessungen 8,29, Ultrastrukturanalyse30,31 und Proteomik32,33. Eine nützliche Alternative, die zunehmend eingesetzt wird und den Ausgangspunkt für einige der zuvor genannten Methoden bildet, ist die direkte Bestimmung des SC-Assemblierungsstatus mittels Blue Native (BN) Elektrophorese, die zu diesem Zweck von Schäggers Gruppe34,35 entwickelt wurde.
Dieser Ansatz erfordert reproduzierbare und effiziente Verfahren zur Gewinnung und Solubilisierung von Mitochondrienmembranen und kann durch andere Techniken wie In-Gel-Aktivitätsanalyse (IGA), Second-Dimension-Elektrophorese und Western Blot (WB) ergänzt werden. Eine Einschränkung in den Untersuchungen zur SC-Dynamik mittels BN-Elektrophorese kann die Menge der Ausgangszellen oder Gewebeproben sein. Wir stellen eine Reihe von Protokollen für die Analyse der SC-Assemblierung und -Funktion vor, die von den Methoden der Schägger-Gruppe adaptiert wurden und auf frische oder gefrorene Zell- oder Gewebeproben ab nur 20 mg Gewebe angewendet werden können.
Protokoll
HINWEIS: Die Zusammensetzung aller Nährmedien und Puffer ist in Tabelle 1 angegeben, und Einzelheiten zu allen in diesem Protokoll verwendeten Materialien und Reagenzien sind in der Materialtabelle aufgeführt.
1. Isolierung von Mitochondrien aus Zellkulturen
HINWEIS: Das Mindestvolumen der untersuchten Zellen betrug ~30-50 μl gepackte Zellen (Schritt 1.4). Dies kann je nach Zelltyp ungefähr zwei oder drei 100-mm-Zellkulturplatten oder einer 150-mm-Platte bei 80-90% der Konfluenz entsprechen (zwischen 5 × 10,6 und 107 Zellen in L929-abgeleiteten Zellen oder MDA-MB-468). Ein effizienter Zellbruch ist ein kritischer Schritt.
- Züchten Sie adhärente Zellen auf ca. 80% Konfluenz oder suspendierte Zellen auf eine ausreichende Dichte.
- Ernte der Zellen durch Zentrifugation bei 500 × g für 5 min (direkt aus der Zellsuspension bei nicht adhärenten Zellen oder nach Standardtrypsinisierung mit einer Trypsin-EDTA-Lösung von 0,05 % Trypsin und 0,02 % EDTA in 1x PBS bei adhärenten Zellen).
- Die Zellen 2x mit kaltem 1x PBS waschen und durch Zentrifugation bei 500 × g für 5 min sedimentieren.
HINWEIS: Ab diesem Zeitpunkt müssen alle Schritte bei 4 °C durchgeführt werden. Daher müssen alle Reagenzien kalt sein und die Röhrchen mit Zellen oder Mitochondrien müssen auf Eis aufbewahrt werden. - Nach der zweiten Zentrifugation wird der Überstand verworfen, das Zellpelletvolumen (cpv) geschätzt und die Zellen mindestens 15 Minuten lang bei -80 °C eingefroren, um das Aufbrechen der Membran in Schritt 1.8 zu erleichtern.
HINWEIS: Das Protokoll kann zu diesem Zeitpunkt gestoppt werden und die Zellen können wochenlang bei -80 °C gelagert werden. In der Regel sind 10- oder 15-ml-Messröhrchen mit konischem Boden geeignet. - Lassen Sie die Zellen langsam auf Eis auftauen.
- Resuspendieren Sie das gepackte Zellpellet in einem Volumen hypotonischen Puffers, das dem 7-fachen des cpv entspricht (10 mM MOPS, 83 mM Saccharose, pH 7,2) (z. B. 100 μl gepackte Zellen in 700 μl Puffer).
HINWEIS: Die Lautstärke muss ausreichend sein, um effiziente Pops zu erzielen (siehe HINWEIS in Schritt 1.8). - Übertragen Sie die Zellsuspension in einen Potter-Dounce-Homogenisator der entsprechenden Größe und lassen Sie die Zellen quellen, indem Sie sie 2 Minuten lang in Eis inkubieren. Für ein Volumen von 700-800 μl Zellsuspension ist beispielsweise ein Homogenisator mit einer Kapazität von 1 ml ausreichend.
- Brechen Sie die Zellmembranen, indem Sie acht bis zehn Hübe im Homogenisator ausführen, der mit einem motorbetriebenen Teflonstößel gekoppelt ist, der sich mit 600 U/min dreht.
HINWEIS: Bei den Strichen ist es wichtig, ein Vakuum zu erzeugen, das die Effizienz des Zellbruchs zusammen mit den hypotonischen und gefrierenden Aktionen zum Anschwellen bzw. Zerbrechen der Membranen erhöht. Wenn es gut gemacht ist, ist beim Herunterziehen des Homogenisators bei jedem Hub ein "Knall"-Geräusch zu hören, das mit einer schnellen Bewegung ausgeführt wird. - Geben Sie das gleiche Volumen an hypertonischem Puffer in die Zellsuspension (7x das cpv) (30 mM MOPS, 250 mM Saccharose, pH 7,2), um eine isotonische Umgebung zu erzeugen.
- Das Homogenat in ein 10-15-ml-Röhrchen überführen und in einem festen Rotor bei 1.000 × g für 5 min bei 4 °C zentrifugieren.
HINWEIS: Zu diesem Zweck kann das Originalröhrchen mit den ungebrochenen Zellen verwendet werden. - Übertragen Sie den Überstand, der die mitochondriale Fraktion enthält, in 1,5-ml-Polypropylenröhrchen.
- OPTIONAL: Um die Ausbeute an Mitochondrien zu erhöhen, resuspendieren Sie das Pellet aus Schritt 1.10 in 7x das Zellpelletvolumen von Puffer A (10 mM Tris, 1 mM EDTA, 0,32 M Saccharose, pH 7,4), indem Sie auf und ab pipettieren und bei 1.000 × g für 5 min bei 4 °C zentrifugieren. Kombinieren Sie den neuen Überstand mit dem vorherigen, bevor Sie mit Schritt 1.13 fortfahren.
- In einer Mikrofuge bei 16.000 × g für 2 min bei 4 °C zentrifugieren, um die mitochondriale Rohfraktion zu sammeln.
- Den Überstand verwerfen und jedes mit Mitochondrien angereicherte Pellet mit 0,5 ml Puffer A resuspendieren, den Inhalt von zwei Röhrchen in einem zusammenfassen und unter den gleichen Bedingungen wie in Schritt 1.13 beschrieben zentrifugieren. Wiederholen Sie den gleichen Vorgang, bis sich das gesamte Material in nur einer Tube befindet.
- Verwerfen Sie den Überstand, resuspendieren Sie das endgültige Pellet mit 300 μl Puffer A und quantifizieren Sie die mitochondriale Proteinkonzentration mit dem Bradford-Assay. Zentrifugieren Sie erneut wie zuvor und fahren Sie mit Abschnitt 3 fort.
2. Isolierung von Mitochondrien aus kleinen Mengen tierischen Gewebes
HINWEIS: Die Mindestmenge an Gewebe, um dieses Protokoll mit einer gewissen Sicherheit anzuwenden, hängt vom Zelltyp und seiner mitochondrialen Häufigkeit ab, kann jedoch in den meisten Fällen ~20-30 mg Gewebe betragen. Bei den Gewebeproben kann es sich um frisches Material oder gefrorene Proben handeln. Im letzteren Fall lassen Sie die Proben in einem auf Eis gelegten Homogenisierungspuffer auftauen, bevor Sie mit dem Verfahren beginnen.
- Wiegen oder schätzen Sie so genau wie möglich die Menge (mg) des Gewebes.
- Schneiden Sie das Taschentuch mit einer Schere ab und waschen Sie 3-4 Mal im Homogenisierungspuffer mit Hilfe eines Siebes, wobei darauf zu achten ist, dass die kleineren Stücke nicht verloren gehen.
HINWEIS: Dieser Schritt ist in Geweben wie Skelettmuskeln oder Herzen wichtiger als im Gehirn oder in weicheren Geweben, deren Zellen leicht direkt durch den Homogenisierungsschritt disaggregiert werden können. - Fügen Sie frisches Homogenisierungsmedium hinzu (4 ml pro Gramm Leber, 10 ml pro Gramm Herz, braunes Fettgewebe (BAT) oder Muskel und 5 ml pro Gramm Gehirn oder Niere; siehe spezifische Pufferzusammensetzung für jeden Fall in Tabelle 1).
- Übertragen Sie die Gewebestücke mit Puffer in den Homogenisator und befolgen Sie je nach ausgewähltem Gewebe das optimale Homogenisierungs- und Mitochondrienisolationsprotokoll.
- Isolierung von Mitochondrien aus Leber, Milz und Niere
- Homogenisieren Sie mit vier bis sechs Auf- und Abschlägen im Elvehjem-Potter mit einem motorbetriebenen Teflonstößel bei 600 U/min.
- Übertragen Sie das homogenisierte Gewebe in ein steriles Zentrifugenröhrchen. Im Schwingrotor bei 1.000 × g für 5 min bei 4 °C zentrifugieren.
- Füllen Sie 1,5 mL Polypropylen-Röhrchen mit dem im vorherigen Schritt erhaltenen Überstand. 2 min bei 16.000 × g in einer Mikrofuge bei 4 °C zentrifugieren und im Folgenden wie zuvor beschrieben (Schritte 1.13 bis 1.15) verfahren
- Isolierung von Mitochondrien aus Herz- und Muskelproben
- Homogenisieren Sie mit sechs bis acht Hüben im Elvehjem-Potter mit einem motorbetriebenen Teflonstößel bei 600 U/min. Übertragen Sie das homogenisierte Gewebe in ein steriles Zentrifugenröhrchen.
- Im Schwingrotor bei 1.000 × g für 5 min bei 4 °C zentrifugieren. Gießen Sie den Überstand in saubere 1,5-ml-Polypropylenröhrchen.
HINWEIS: Eine zweite Homogenisierung des Pellets, die in Schritt 2.6.2 erhalten wurde, kann mit 4-5 zusätzlichen Hüben und 1/2 Volumen Homogenisierungspuffer durchgeführt werden, um die mitochondriale Ausbeute zu erhöhen. Der neue Überstand (nach erneuter Zentrifugation bei 1.000 × g für 5 min bei 4 °C) kann mit dem vorherigen kombiniert werden, bevor mit Schritt 2.6.3 fortgefahren wird. Eine Alternative zur Erhöhung der Rohmitochondrienausbeute besteht darin, Herz- und Muskelgewebeproben vor der Homogenisierung in Trypsinlösung zu dissoziieren36,37. In diesem Fall muss das Trypsin effizient inaktiviert/entfernt werden, bevor die Mitochondrienmembranen mit Digitonin solubilisiert werden (Schritt 3.2). - Bei 16.000 × g in einer Mikrofuge für 2 min bei 4 °C zentrifugieren, um die rohe mitochondriale Fraktion zu erhalten.
- Der Überstand wird verworfen und jedes mit Mitochondrien angereicherte Pellet mit 0,5 ml Puffer AT resuspendiert, wobei der Inhalt von zwei Röhrchen in einem kombiniert wird, und unter den gleichen Bedingungen wie in Schritt 2.6.3 beschrieben zentrifugiert. Wiederholen Sie den gleichen Vorgang, bis sich das gesamte Material in nur einer Tube befindet.
- Verwerfen Sie den Überstand, resuspendieren Sie das endgültige Pellet mit 300 μl Puffer A (kein BSA) und quantifizieren Sie die mitochondriale Proteinkonzentration mit dem Bradford-Assay. Zentrifugieren Sie erneut wie zuvor und fahren Sie mit Abschnitt 3 fort.
- Isolierung der Mitochondrien aus dem Gehirn
- Homogenisieren Sie die Teile des Gehirns mit 10-15 Hüben mit einem Dounce-Glashomogenisator mit einem manuell angetriebenen Glasstößel. Übertragen Sie das homogenisierte Gewebe in ein steriles Zentrifugenröhrchen.
- Im Schwingrotor bei 1.000 × g für 5 min bei 4 °C zentrifugieren.
- Sammeln Sie den Überstand in einem sauberen Zentrifugenröhrchen.
- Das im vorherigen Zentrifugationsschritt erhaltene Pellet wird in demselben Volumen des Mediums AT, das bei der ersten Homogenisierung verwendet wurde, resuspendiert. Homogenisieren Sie das Pellet erneut, indem Sie den in Schritt 2.7.1 beschriebenen Vorgang in 5-10 Durchgängen wiederholen und die Suspension in einem Schwingrotor bei 1.000 × g für 5 min bei 4 °C zentrifugieren.
- Den Überstand entfernen und in das in Schritt 2.7.3 vorbereitete Röhrchen geben. Bei 10.000 × g in einem Festwinkelrotor für 10 min bei 4 °C zentrifugieren, um die rohe mitochondriale Fraktion zu erhalten.
- Der Überstand wird verworfen und das Pellet auf 1/2 des ursprünglichen Homogenisierungsvolumens (Schritt 2.7.1) des Mediums AT resuspendiert. Die mitochondriale Suspension in saubere 1,5-ml-Polypropylenröhrchen verteilen und bei 16.000 × g in einer Mikrofuge für 2 min bei 4 °C zentrifugieren.
- Der Überstand wird verworfen und jedes mit Mitochondrien angereicherte Pellet mit 0,5 ml Puffer AT resuspendiert, wobei der Inhalt von zwei Röhrchen in einem Gefäß kombiniert und unter den gleichen Bedingungen wie in Schritt 2.7.6 beschrieben zentrifugiert wird. Wiederholen Sie den gleichen Vorgang, bis sich das gesamte Material in nur einer Tube befindet.
- Verwerfen Sie den Überstand, resuspendieren Sie das endgültige Pellet mit 300 μl Puffer A (kein BSA) und quantifizieren Sie die mitochondriale Proteinkonzentration mit dem Bradford-Assay. Zentrifugieren Sie erneut wie zuvor und fahren Sie mit Abschnitt 3 fort.
- Isolierung von Mitochondrien aus der BVT
- Homogenisieren Sie mit acht bis zehn Auf- und Abschlägen im Elvehjem-Potter mit einem motorbetriebenen Teflonstößel bei 600 U/min. Übertragen Sie das homogenisierte Gewebe in ein steriles Zentrifugenröhrchen.
- Im Schwingrotor bei 1.000 × g für 5 min bei 4 °C zentrifugieren.
- Füllen Sie 1,5 mL Polypropylen-Röhrchen mit dem im vorherigen Schritt erhaltenen Überstand und vermeiden Sie dabei die obere Fettschicht.
HINWEIS: Eine zweite Homogenisierung des Pellets, die in Schritt 2.8.2 erhalten wurde, kann durchgeführt werden, um die mitochondriale Ausbeute zu erhöhen. Der neue Überstand (nach erneuter Zentrifugation bei 1.000 × g für 5 min bei 4 °C) kann mit dem vorherigen kombiniert werden, bevor mit Schritt 2.8.4 fortgefahren wird. - 2 min bei 16.000 × g für 2 min in einer Mikrofuge bei 4 °C zentrifugieren.
- Der Überstand wird verworfen und jedes mit Mitochondrien angereicherte Pellet mit 0,5 ml Puffer AT2 resuspendiert, wobei der Inhalt von zwei Röhrchen in einem Gefäß kombiniert wird, und unter den gleichen Bedingungen wie in Schritt 2.8.4 beschrieben zentrifugiert. Wiederholen Sie den gleichen Vorgang, bis sich das gesamte Material in nur einer Tube befindet.
- Verwerfen Sie den Überstand, resuspendieren Sie das endgültige Pellet mit 300 μl Puffer A (kein BSA) und quantifizieren Sie die mitochondriale Proteinkonzentration mit dem Bradford-Assay. Zentrifugieren Sie erneut wie zuvor und fahren Sie mit Abschnitt 3 fort.
3. Vorbereitung der Proben für die Blue Native-Analyse
- Resuspendieren Sie die mitochondrialen Fraktionen (aus Säugetierzellkulturen oder -geweben) in BN-Probenpuffer (50 mM NaCl, 50 mM Imidazol, 5 mM Aminocapronsäure, 4 mM PMSF), um eine Proteinkonzentration von etwa 10 mg/ml zu erhalten. In Tabelle 2 sind die erwarteten Erträge für die verschiedenen Arten von Proben aufgeführt.
- Solubilisieren Sie die mitochondrialen Membranen durch Zugabe von Digitonin (10%ige Stammlösung), um ein Verhältnis von 4 g Digitonin zu g mitochondrialem Protein zu erhalten (4 μl Digitonin 10 % Stamm für 10 μl der in Schritt 3.1 hergestellten mitochondrialen Suspension).
HINWEIS: Das Waschmittel-Protein-Verhältnis (g/g) muss für jede Art von Probe optimiert werden, um reproduzierbare Ergebnisse zu erhalten. Es wurde festgestellt, dass 2-8 g Digitonin/g mitochondriales Protein für die meisten Gewebe optimal sind 34,35. - Mischen Sie, indem Sie vorsichtig auf und ab pipettieren. Inkubieren Sie die Proben 5 Minuten lang auf Eis.
HINWEIS: Nach diesem Schritt sollte die mitochondriale Suspension klarer werden (Verschiebung von undurchsichtig zu durchscheinend); Andernfalls könnte dies darauf hindeuten, dass die Menge des Detergens für eine korrekte Membransolubilisierung nicht ausreicht. - Zentrifugieren Sie bei voller Geschwindigkeit in einer Mikrofuge (ca. 20.000 × g ) für 25 min bei 4 °C, um unlösliches Material zu entfernen.
- Sammeln Sie den Überstand in einer frischen Tube. Dem Überstand wird ein Volumen von 5 % G-250 (Coomassie Blue G-250 5 % in 0,75 M Aminocapronsäure) zugegeben, das 1/3 des ursprünglichen Resuspensionsvolumens entspricht (Schritt 3.1; dies entspricht einem Endanteil von 1,6 g Coomassie/g Protein) und durch Pipettieren mischen.
- Vor dem Laden auf das Gel auf Eis legen oder Aliquote bei -80 °C einfrieren.
4. Blue Native Gelelektrophorese
HINWEIS: Die Elektrophorese wird im Kühlraum (4-8 °C) durchgeführt. Wenn keine Kühlraumeinrichtung vorhanden ist, kann ein Kühlblock in den Elektrophoresetank eingeführt werden. Es werden handelsübliche 3-13%ige native Polyacrylamid-Gele29 verwendet, es können aber auch hausgemachte Gele mit den gewünschten Gradientenkonzentrationen verwendet werden38,39.
- Die obere und untere Kammer werden mit den Kaltkathodenpuffern A bzw. Anodenpuffern beladen. Alternativ können Sie die Proben in die Vertiefungen laden, bevor Sie die obere Kammer vorsichtig mit Kathodenpuffer A füllen.
HINWEIS: In unserem Labor werden kommerzielle Elektrophoresepuffer verwendet, die jedoch wie in Tabelle 1 angegeben hergestellt werden können. - Laden Sie zwischen 30 und 100 μg mitochondriales Protein.
HINWEIS: In der Regel werden 30-50 μg Protein (für WB) oder 40-100 μg Protein (für IGA) pro Well in ein 10-Lane-Gel (0,5 x 0,15 cm Wells) geladen. Kathoden-A-Puffer enthält Coomassie Blue G-250 und hat eine intensive blaue Farbe, die es schwierig macht, die Gelvertiefungen für die Probenbeladung zu sehen. Es ist praktisch, die Mitte jeder Vertiefung zu markieren, z. B. mit einem roten Marker, um das Platzieren der Pipettenspitze während dieses Schritts zu erleichtern. Native Molekulargewichtsmarker können bei der Einrichtung der BN-PAGE-Technik nützlich sein, um zu bestätigen, dass die Gradientengele richtig gebildet sind und dass das Muster der Komplexe und SCs das richtige ist. - Laufen Sie 25-30 min lang bei 80-100 V und dann bei 160-180 V, wobei der Strom auf 12 mA/Gel begrenzt wird, bis der Farbstoff den Boden des Gels erreicht (insgesamt ~125-165 min).
- Wenn In-Gel-Aktivitätsassays (IGAs) durchgeführt werden sollen, ersetzen Sie den Kathodenpuffer A durch den Kathodenpuffer B, wenn sich die Farbstofffront in der Mitte des Gels befindet (~1 h nach Beginn des Laufs).
HINWEIS: Obwohl die Gele ohne Färbung direkt nach dem Lauf dokumentiert werden können, da einige Banden aufgrund ihrer Bindung an den G-250-Farbstoff sichtbar sind (hauptsächlich Komplex V, der als "interner" Maker mit einem MW von ~600 kDa angesehen werden kann), sind SCs und einzelne Komplexe in der Regel ohne Färbung oder IGA-Assays nicht sichtbar. Die Gele, die in IGA-Assays oder für WB verwendet werden sollen, sollten nicht gefärbt oder fixiert werden. - Zerlegen Sie das Gel aus der Kassette und fahren Sie mit der Coomassie-Blau-Färbung (Abschnitt 5), der IGA-Analyse (Abschnitt 6) oder der WB-Immundetektion (Abschnitt 7) fort.
5. Gel-Färbung
- Das Gel 10-15 min bei Raumtemperatur (RT) mit Coomassie-Blau-Farbstoffen färben (Coomassie-Farbstoff R-250 mit 0,25 % in 40 % Methanol, 10 % Essigsäure; 10-15 min färben).
- Mit mehreren Wäschen (typischerweise 4-5 x 15 min) in 40 % Methanol plus 10 % Essigsäure bei RT entfärben.
- Dokumentieren Sie das Gel.
6. In Gelaktivitäts-Assays (IGA)
- Bereiten Sie IGA-Lösungen für die Analyse der verschiedenen Atemwegskomplexe vor dem Ende der Elektrophorese vor und halten Sie sie im Dunkeln (z. B. durch Verwendung dunkler Kunststoffboxen oder Abdecken mit Aluminiumfolie). Die Zusammensetzung der einzelnen Puffer ist in Tabelle 1 aufgeführt.
- Legen Sie das Gel oder die zu verwendenden Bahnen in eine möglichst kleine Plastikbox, um es unterzubringen.
- Fügen Sie genügend Volumen der entsprechenden Lösung hinzu, um das Gel zu bedecken (normalerweise 5 oder 10 ml für 5 bzw. 10 Gelspuren) und inkubieren Sie bei RT unter leichtem Schütteln (60-80 U/min) und vor Licht geschützt.
HINWEIS: Die Inkubationszeit hängt von dem zu analysierenden Komplex sowie von der Art und Menge der geladenen Probe ab. CI und CIV sind am einfachsten und schnellsten zu Ergebnissen. So beginnt die CI-Aktivität in der Regel nach einigen Minuten sichtbar zu werden, während CII und CIV ~30 Minuten benötigen, um beobachtet zu werden, und CV ~1,5-2 h. Die Reaktion kann in allen Fällen stundenlang fortgesetzt werden, und die Geschwindigkeit der Signalverstärkung kann reduziert werden, indem die Inkubation in einen kalten Raum (4-6 °C) verlegt wird, z. B. für eine Inkubation über Nacht (ON). Die Ciii-Aktivität funktioniert nur für die klare native Elektrophorese (CN), nicht für BN34. - Wenn sich die entsprechenden Banden gebildet haben, stoppen Sie die Reaktionen, indem Sie die Testlösungen entfernen, 2x mit destilliertem Wasser waschen und das Gel mit 40 % Methanol plus 10 % Essigsäure (mit Ausnahme von CV, das nur mit Methanol fixiert ist) für 30 Minuten fixieren.
- Um die CIV-Aktivität nach CI-Nachweis durch IGA zu analysieren, waschen Sie das Gel 2x mit destilliertem Wasser, dokumentieren Sie die CI-Aktivität, inkubieren Sie das Gel (ohne es zu fixieren!) 2x für 30 min mit 50 mM Kaliumphosphatpuffer (pH 7,2) und dann mit dem vollständigen CIV-Reaktionspuffer, bis die komplexen IV-Banden erscheinen (normalerweise durch Inkubation ON bei RT oder 4 °C).
- Dokumentieren Sie die Gele.
HINWEIS: Da die entwickelten Banden bei der Dokumentation des Gels weiß sind, legen Sie es bei der Dokumentation auf einen dunklen Hintergrund, so dass die klaren Banden sichtbar sind.
7. Westliche Blot-Analyse
- Transferpuffer gemäß Tabelle 1 vorbereiten und bis zum Ende der Elektrophorese bei 4 °C halten.
- Legen Sie das Gel in eine Schale und fügen Sie den Transferpuffer hinzu. Inkubieren Sie 10-15 Minuten bei RT.
- Schneiden Sie ein Stück PVDF-Membran in der gleichen Größe wie das Gel ab und aktivieren Sie es 10 s lang unter Rühren in Methanol. Waschen Sie mehrmals mit destilliertem Wasser und fügen Sie Transferpuffer hinzu. Inkubieren Sie bei RT für 10-15 Minuten unter leichtem Schütteln.
- Bereiten Sie das Transfersandwich von unten nach oben vor und vermeiden Sie dabei Blasen zwischen dem Gel und der Membran in der folgenden Reihenfolge: schwarze Seite der Kassette - Schwamm - Löschpapier - Gel-Membran - Löschpapier - Schwamm - klare Seite der Kassette.
- Schließen und verriegeln Sie die Kassette und legen Sie das Sandwich in der richtigen Ausrichtung für den Transfer in den Transferbehälter (schwarze Seite zum Minuspol).
- Füllen Sie den Transferbehälter mit dem Transferpuffer, fügen Sie einen Magnetrührer hinzu, um den Puffer zu bewegen, und schließen Sie die Stromversorgung bei 80 V für 2 h oder bei 100 V für 1 h an. Führen Sie den Transfer zwischen 4 und 8 °C (z. B. im Kühlraum) und mit einem Kühlblock im Transfersystem durch.
- Entnehmen Sie die Membran und fahren Sie mit einem Standard-Western-Blot-Protokoll fort, wobei spezifische Antikörper für die verschiedenen zu detektierenden Atmungskomplexe verwendetwerden 29.
Ergebnisse
Die Ausbeuten an Mitochondrien, die nach den oben beschriebenen Protokollen erhalten werden, variieren in Abhängigkeit von mehreren Faktoren, wie z. B. der Zelllinie oder dem Gewebetyp, der Art der Proben (d. h. ob frisches oder gefrorenes Gewebe verwendet wird) oder der Effizienz des Homogenisierungsprozesses. Die erwarteten Erträge an Mitochondrien aus verschiedenen Zelllinien und Geweben sind in Tabelle 2 aufgeführt. Sobald die mitochondrialen Fraktionen erhalten wurden, ist der nächste Schritt die Analyse des respiratorischen SCs-Musters, die nach der Solubilisierung der mitochondrialen Rohprobe und der elektrophoretischen Trennung durch BN-PAGE durchgeführt wird, gefolgt von einer IGA-Analyse oder WB-Immundetektion. Abbildung 1 zeigt das Aussehen der ungefärbten Gelbahnen unmittelbar nach dem Lauf und das Muster der Banden nach der Standard-Coomassie-Färbung einer kultivierten Zelllinie und einer mitochondrialen Leberprobe der Maus.
In Abbildung 2A,B sind deutliche Unterschiede zwischen den SC-Assemblierungsmustern in humanen und Mauszellen nach Verwendung von IGA-Assays zu sehen. So wird der freie Komplex I in Mauszellen beobachtet, während er in menschlichen Mitochondrien nicht nachweisbar ist. Das komplexe IV-IGA-Muster ist in beiden menschlichen Zelllinien sehr ähnlich (Abbildung 2B), weist jedoch Unterschiede zwischen den beiden in Abbildung 2A analysierten Mauszelllinien auf. Diese Unterschiede sind darauf zurückzuführen, dass BL6-Zellen eine mutierte Variante von SCAF18 exprimieren.
Die SC-Muster, die mit dem hier beschriebenen Protokoll für die mitochondriale Isolierung für die Arbeit mit kleinen Proben erhalten wurden, werden in Bezug auf die "konventionellen" Protokolle, die für größere Proben verwendet werden, beibehalten. Dies ist in Abbildung 2C zu sehen, indem die Spuren 1 und 6 (nach mitochondrialer Isolierung unter Verwendung eines konventionellen Protokolls aus 3 g Leber und 1,1 g BVT gewonnen) mit den Spuren 2 und 5 (erhalten unter Verwendung der hier vorgestellten Protokolle aus etwa 0,1 g beider Gewebe) verglichen werden. Wie im Plastizitätsmodell vorgeschlagen, variiert der relative Anteil von respiratorischen Komplexen und SCs je nach Zelltyp und Stoffwechselzustand. Wie in Abbildung 2C,D gezeigt, zeigen die verschiedenen Gewebe unterschiedliche SC-Assemblierungsmuster. So weisen die Mitochondrien des Gehirns im Vergleich zu den anderen Geweben einen sehr niedrigen Gehalt an freiem Komplex I und einen höheren Anteil an SCs auf, und BAT zeichnet sich durch niedrige CV-Spiegel (Abbildung 2A) und hohe Mengen an SC III + IV (Abbildung 2D) aus. Herzmitochondrien weisen ein hohes Maß an SCs und CV auf. Die CV-Assemblierungsmuster sind zwischen der kultivierten Zelllinie und einer mitochondrialen Leberprobe der Maus sehr ähnlich (Abbildung 2E).
Die SC-Assemblierung kann auch mittels WB analysiert werden, wie in Abbildung 3 für zwei Proben aus Milzkontroll- und Tumorzellen gezeigt. In diesem Fall reichen in der Regel 30-50 μg mitochondriales Protein aus, um die verschiedenen Komplexe und SCs mit den spezifischen Antikörpern nachzuweisen. Abbildung 4 zeigt einige der wichtigsten Fallstricke, die bei der Anwendung dieser Protokolle auftreten können. Falsche Gelgradienten können zu abnormalen und unerwünschten Migrationsmustern führen, wie in Abbildung 4A, Spur 1, wo die SCs aufgrund einer überdurchschnittlich hohen Acrylamidkonzentration im Gel in der Nähe der Vertiefung im oberen Teil der Spur verbleiben. Die Inkubation der Proben, auch für relativ kurze Zeiträume, bei hohen Temperaturen (hier 37 °C und 40 °C für 10 min) führt zu einer Degradation von SCs und CI (Abbildung 4A , Fahrspuren 2-4). Ebenso ist das Waschmittel-Protein-Verhältnis entscheidend für gute Ergebnisse und eine angemessene Gelauflösung. Unterhalb eines Verhältnisses von 2 g Digitonin/g Protein wird die Bandenauflösung progressiv reduziert und bei der niedrigeren Konzentration (0,5 g/g) ist nur ein Abstrich im Bereich sichtbar, der den SCs entspricht (Abbildung 4B).

Abbildung 1: Muster der mitochondrialen Komplexe und Superkomplexe nach BNGE-Trennung von Digitonin-permeabilisierten Mitochondrien aus einer Kulturzelllinie (L929Balbc) und Mausleber. Die Spuren auf der linken Seite (1 und 2) stellen das Aussehen des ungefärbten Gels kurz nach dem Lauf dar, bei dem nur CV nachweisbar ist, während die Spuren auf der rechten Seite (3 und 4) das Muster der Banden nach der Coomassie-Färbung zeigen. Abkürzungen: BNGE = Blue Native Gelelektrophorese; CV = Komplex V; SCs = Superkomplexe. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
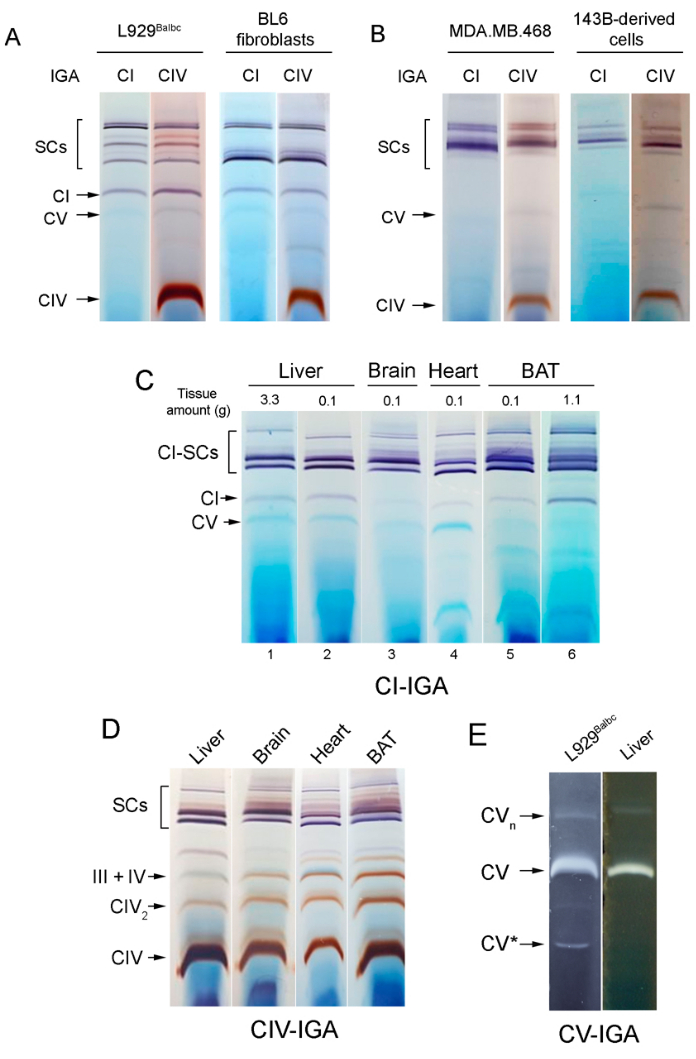
Abbildung 2: IGA-Analyse von Mitochondrien, die aus verschiedenen Zelllinien und Mausgeweben isoliert wurden. (A,B) In-Gel-Aktivität der angegebenen Komplexe in Mitochondrien, die aus (A) Maus- oder (B) humanen Zelllinien isoliert wurden. L929Balbc ist eine transmitochondriale Zelllinie, die in unserem Labor durch Übertragung von Mitochondrien aus Balb/cJ-Mausplättchen auf ρ°L929neo-Zellen erzeugt wird, wie zuvor beschrieben40,41. BL6-Fibroblasten sind eine immortalisierte Zelllinie, die in unserem Labor aus einer Ohrbiopsie einer BL6-Maus gewonnen wurde. (C) CI-IGA-Muster aus Mitochondrien von Mäusen, die entweder mit einer konventionellen Methode beginnend mit 3 g Leber (Spur 1) und mit 1,1 g BAT (Spur 6) oder mit dem hier vorgestellten Protokoll isoliert wurden und in allen Fällen mit etwa 0,1 g Gewebe beginnen (Spur 2, Leber; Spur 3, Herz; Spur 4, Gehirn und Spur 5 BAT). (D) CIV-IGA-Muster (durchgeführt nach CI-IGA, gezeigt in Panel 2C, Spuren 2-5) in Leber, Herz, Gehirn und BAT-Mitochondrien von Mäusen. (E) CV-IGA-Aktivität, analysiert in kultivierten Zellen und Lebern von Mäusen. Etwa 60-75 μg mitochondriales Protein pro Spur wurden geladen. Abkürzungen: IGA = In-Gel-Aktivität; SCs = Superkomplexe; CI = Komplex I; BAT = braunes Fettgewebe. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
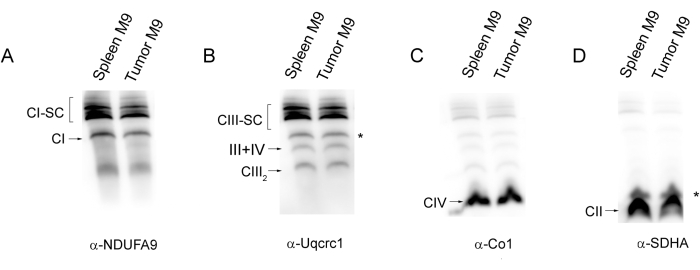
Abbildung 3: WB-Immundetektion der verschiedenen mitochondrialen Komplexe und Superkomplexe in Milzproben. Nach der Blue Native PAGE-Trennung von mitochondrialen Komplexen und SCs, die aus Milzkontroll- (Milz M9) und Tumorproben (Tumor M9) gewonnen wurden, wurden sie auf eine PVDF-Membran übertragen und sequentiell mit Antikörpern hybridisiert, die die angegebenen CI-, CIII-, CIV- und CII-Untereinheiten erkennen (Panels A-D). Pro Spur wurden etwa 50 μg mitochondriales Protein geladen. Sternchen zeigen das Signal an, das von einem früheren Western Blot übrig geblieben ist. Abkürzungen: SC = Superkomplex; CI = Komplex I; CIII = Komplex III; CIV = Komplex IV; CII = Komplex II. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Fallstricke bei der SC-Analyse durch Blue-Native PAGE. CI IGA-Analyse von Mitochondrien, die aus (A) frischer Mausleber und (B) gefrorener Rattenleber unter verschiedenen Solubilisierungsbedingungen isoliert wurden. (A) Spur 1, falsche Gelgradientenkonzentration (8-13% statt der normalen 3-13%). Bahnen 2-4: Die Proben wurden vor dem Laden 10 Minuten lang auf Eis oder bei 37 °C bzw. 40 °C inkubiert. (B) Muster nach IGA für CI von Mitochondrien, die aus gefrorener Rattenleber unter Verwendung unterschiedlicher Digitonin/Protein-Verhältnisse für die Solubilisierung gewonnen wurden. Abkürzungen: SC = Superkomplex; PAGE = Elektrophorese des Polyacrylamid-Gels; CI = Komplex I; IGA = In-Gel-Aktivität. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Mittel | Zusammensetzung | ||
| Hypotonischer Puffer | 10 mM MOPS, 83 mM Saccharose, pH 7,2 | ||
| Hypertonischer Puffer | 30 mM MOPS, 250 mM Saccharose, pH 7,2 | ||
| Homogenisierungs-Puffer A | 10 mM Tris, 1 mM EDTA, 0,32 M Saccharose, pH 7,4 | ||
| Homogenisierungspuffer AT | 225 mM Mannitol, 1 mM EGTA, 75 mM Saccharose, 0,01 % BSA pH 7,4 | ||
| Homogenisierungspuffer AT2 | 225 mM Mannitol, 1 mM EGTA, 75 mM Saccharose, 0,02% BSA pH 7,4 | ||
| BN Probenpuffer | 50 mM NaCl, 50 mM Imidazol, 2 mM Aminocapronsäure, 1 mM EDTA pH 7,0 | ||
| Digitonin-Lösung | 10% Digitonin in 50 mM NaCl, 50 mM Imidazol, 5 mM Aminocapronsäure, 4 mM PMSF | ||
| BN-Ladepuffer | 5% Coomassie Blue G in 0,75 M Aminocapronsäure | ||
| BN Kathodenpuffer A | 50 mM Tricin, 15 mM Bis-Tris pH = 7,0 (4 °C), 0,02 % G-250 (Coomassie Brillantblau G-250) | ||
| BN Kathodenpuffer B | 50 mM Tricin, 15 mM Bis-Tris pH=7,0 (4 °C), 0,002% G-250 | ||
| BN-Anodenpuffer | 50 mM Bis-Tris, pH=7,0 | ||
| Komplex I IGA-Substrat | 5 mM Tris-HCl pH 7,4, 0,1 mg/mL NADH, 2,5 mg/mL Nitroblue Tetrazolium (NBT) | ||
| Komplex II IGA-Substrat | 50 mM Kaliumphosphatpuffer pH 7,4, 20 mM Natriumsuccinat, 0,2 mM Phenazinmethosulfat (PMS), 2,5 mg/mL NBT | ||
| Komplex III IGA-Substrat | 50 mM Kaliumphosphatpuffer pH 7,2, 0,05 % Diaminobenzidin (DAB) | ||
| Komplexes IV IGA-Substrat | Gleiche Lösung wie CIII plus 50 μM Cytochrom c | ||
| Komplexes V IGA-Substrat | 35 mM Tris, 270 mM Glycin pH 8,3, 14 mM MgSO4, 0,2% Pb(NO3)2, 8 mM ATP | ||
| Lösung zum Färben | Coomassie-Farbstoff R-250 mit 0,25 % in 40 % Methanol und 10 % Essigsäure | ||
| Entfärbungs-/Fixierlösung | 40% Methanol, 10% Essigsäure | ||
| Puffer übertragen | 48 mM Tris, 39 mM Glycin, 20% Methanol | ||
Tabelle 1: Puffer und Medienzusammensetzung.
| Zelltyp/Gewebe | Ausgangsbetrag | Erwartete Ausbeute (μg mitochondriales Protein) | Anzahl der Bahnen (50-60 μg/Bahn) |
| Kultivierte Zellen | |||
| MDA-MB-468 | 100 μl vpc | 300-400 μg | 6-9 |
| 143B-abgeleitet | 100 μl vpc | 200-350 μg | 4-7 |
| Fibroblasten der Maus | 100 μl vpc | 150-200 μg | 3-5 |
| Herz | 100 mg | 400-500 μg | 10-12* |
| Skelettmuskulatur | 100 mg | 300-400 μg | 8-10* |
| Leber | 100 mg | 400-500 μg | 8-10 |
| Milz | 50 mg | 150-250 μg | 3-5 |
| FLEDERMAUS | 100 mg | 200-300 μg | 4-6 |
| Gehirn | 100 mg | 250-300 μg | 5-8 |
Tabelle 2: Erwartete Erträge von Mitochondrien aus verschiedenen Zelltypen und Mausgeweben. Die mitochondrialen Fraktionen, die aus dem Herz- oder Skelettmuskel gewonnen werden, sind reiner als die aus anderen Geweben gewonnenen. So können kleinere Mengen in die Gele geladen werden (*).
Diskussion
Die methodischen Anpassungen, die in den hier beschriebenen Protokollen eingeführt wurden, zielen darauf ab, Verluste zu vermeiden und die Ausbeute zu erhöhen, während die Aktivitäten des mitochondrialen Komplexes erhalten bleiben (was entscheidend ist, wenn die Verfügbarkeit ausreichender Probenmengen beeinträchtigt ist) und das erwartete Muster von SCs des Gewebes oder der Zelllinie reproduziert wird (siehe Abbildung 2C). Zu diesem Zweck und da eine hohe mitochondriale Reinheit nicht erforderlich ist, um die SCs richtig zu detektieren, wurden die Anzahl der Schritte, Zeiten und Volumina reduziert, wann immer dies möglich war.
So werden nach der Homogenisierung und Entfernung von Zellkernen und ungebrochenen Zellen durch Zentrifugation mit niedriger Geschwindigkeit die Überstände, die die Mitochondrien enthalten, in 1,5 mL Polypropylen-Röhrchen überführt, und die nachfolgenden Zentrifugationen zur Gewinnung der rohen mitochondrialen Fraktionen werden in einer Mikrofuge durchgeführt, indem die Geschwindigkeit erhöht und die Zeiten und Volumina im Vergleich zu herkömmlichen Protokollen reduziert werden. Dies ermöglicht die Verarbeitung mehrerer Proben in relativ kurzer Zeit: Eine genaue Schätzung ist, dass 10 Proben in ~2-2,5 h verarbeitet werden können (vom Beginn der Homogenisierung (Protokollschritt 1.6. oder gleichwertig für Gewebe) bis zur Gewinnung der SCs-Fraktion, die bereit ist, in ein Gel geladen zu werden (Protokollschritt 3.7) und minimiert die Verluste bei gleichbleibender Aktivität. Eine weitere Änderung gegenüber den ursprünglichen Protokollen der Schägger-Gruppe besteht darin, dass nach der Membransolubilisierung zur Gewinnung der SCs-Fraktion ein Ultrazentrifugationsschritt durch eine einfache Mikrofugenzentrifugation ersetzt wird (Protokollschritt 3.4).
Es gibt einige kritische Schritte in diesem Verfahren. Erstens, die Effizienz des Zellbruchs, insbesondere bei kultivierten Zellen (siehe Anmerkung nach Protokollschritt 1.8) und insbesondere bei kleinen Zellen. Gewebe und Zelllinien weisen einen unterschiedlichen Schwierigkeitsgrad beim Zellbruch auf: Im Herzen und im Muskel ist dieser Schritt beispielsweise in der Regel weniger effizient als in der Leber oder Milz, aber er wird in der Ausbeute kompensiert, da erstere einen höheren Mitochondriengehalt haben. Eine Alternative zur Verbesserung der mitochondrialen Ausbeute bei Isolierung aus Muskeln oder Herzen wäre die Behandlung von Proben mit Trypsin vor der Homogenisierung. Das Gehirn ist ein weiteres Gewebe, das aufgrund der manuellen Homogenisierung, die milder und weniger effizient ist, und wahrscheinlich auch aufgrund der höheren Variation der Zelltypen in diesem Gewebe, wie bereits berichtet wurde, eine geringe Effizienz beim Zellbruch aufweist 42. Dies kann teilweise durch einen zweiten Homogenisierungsschritt kompensiert werden, wie in unserem Protokoll vorgeschlagen (Schritt 2.7.4). Auf der anderen Seite, wenn der Zellbruch übermäßig ist, könnte die endgültige Probe, die SCs enthält, mit nuklearen DNA-Fragmenten kontaminiert sein, die den elektrophoretischen Ablauf beeinträchtigen können. Dieses Problem zeigt sich in einer Erhöhung der Viskosität der Probe bei Zugabe von Digitonin zur Solubilisierung der Mitochondrienmembranen und kann durch Behandlung mit DNAse I gelöst werden.
Zweitens, wenn das Detergenz-zu-Protein-Verhältnis zu niedrig ist, würde dies zu einer weniger effizienten Membransolubilisierung und der Möglichkeit der Aggregatbildung führen. Dies könnte sich in den Gelen als schlechte Auflösung in hochmolekularen SCs zeigen (siehe Abbildung 4) oder als CIV, das mehrere Banden ergibt, die den Oligomeren43 entsprechen. Ist das Verhältnis zu hoch, kann dies zu einer Disaggregation von SCs (hauptsächlich CI-SCs) führen. Drittens ist es wichtig, das Erhitzen der Proben während des gesamten Verfahrens zu vermeiden (siehe Abbildung 4) und wiederholte Zyklen des Einfrierens und Auftauens zu vermeiden, da dies den Status der SC-Assemblierung beeinträchtigen kann.
In Tabelle 2 ist eine Zusammenfassung der erwarteten Erträge für verschiedene Zelltypen und Gewebeproben dargestellt. Die Werte sind ungefähre Angaben und hängen von der Effizienz des Zellbruchs und der Art der Quelle ab. Bei Geweben führt das Einfrieren des Ausgangsmaterials tendenziell zu einer höheren Ausbeute als bei frischen Proben. Es wird eine Abschätzung der Anzahl der Lanes (unter Berücksichtigung einer Belastung zwischen 50 und 60 μg mitochondrialem Protein pro Lane) vorgestellt, die geladen werden können.
Die hier vorgestellten Protokolle decken eine Vielzahl von Geweben und Zelltypen mit geringen Variationen ab, sie können mit frischen oder gefrorenen Proben verwendet werden und erfordern eine einfache Ausrüstung und relativ niedrige Kosten, ohne dass teure Aufreinigungskits oder Gradienten verwendet werden müssen. Sie benötigen jedoch möglicherweise einige Anpassungen, um mit bestimmten Zelltypen oder Proben verwendet zu werden, sowie wenn spezifische mitochondriale Populationen, wie z. B. synaptische Mitochondrien des Gehirns oder subsarkolemmale Mitochondrien der Muskeln, zum Beispiel, Gegenstand der Analyse sind.
Da die genaue physiologische Rolle sowie die mögliche Beteiligung von SCs an pathologischen Situationen noch definiert werden müssen, ist die Analyse ihrer Dynamik und der Faktoren, die ihre Assemblierung, Stabilität und Funktion beeinflussen, erforderlich. Unsere Protokolle können wertvolle Werkzeuge darstellen, die es ermöglichen, reproduzierbare Ergebnisse an den SCs-Mustern mit kleinen Probengrößen und aus einer Vielzahl von Zelltypen und Geweben zu erhalten, insbesondere wenn mehrere experimentelle Bedingungen verglichen werden müssen29.
Offenlegungen
Die Autoren erklären, dass keine Interessenkonflikte bestehen.
Danksagungen
Diese Arbeit wurde durch die Fördernummer "PGC2018-095795-B-I00" des Ministerio de Ciencia e Innovación (https://ciencia.sede.gob.es/) und durch die Fördernummer "Grupo de Referencia: E35_17R" und die Fördernummer "LMP220_21" der Diputación General de Aragón (DGA) (https://www.aragon.es/) an PF-S und RM-L unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Acetic acid | PanReac | 131008 | |
| Aminocaproic acid | Fluka Analytical | 7260 | |
| ATP | Sigma-Aldrich | A2383 | |
| Bis Tris | Acrons Organics | 327721000 | |
| Bradford assay | Biorad | 5000002 | |
| Coomassie Blue G-250 | Serva | 17524 | |
| Coomassie Blue R-250 | Merck | 1125530025 | |
| Cytochrome c | Sigma-Aldrich | C2506 | |
| Diamino benzidine (DAB) | Sigma-Aldrich | D5637 | |
| Digitonin | Sigma-Aldrich | D5628 | |
| EDTA | PanReac | 131669 | |
| EGTA | Sigma-Aldrich | E3889 | |
| Fatty acids free BSA | Roche | 10775835001 | |
| Glycine | PanReac | A1067 | |
| Homogenizer Teflon pestle | Deltalab | 196102 | |
| Imidazole | Sigma-Aldrich | I2399 | |
| K2HPO4 | PanReac | 121512 | |
| KH2PO4 | PanReac | 121509 | |
| Mannitol | Sigma-Aldrich | M4125 | |
| Methanol | Labkem | MTOL-P0P | |
| MgSO4 | PanReac | 131404 | |
| Mini Trans-Blot Cell | BioRad | 1703930 | |
| MOPS | Sigma-Aldrich | M1254 | |
| MTCO1 Monoclonal Antibody | Invitrogen | 459600 | |
| NaCl | Sigma-Aldrich | S9888 | |
| NADH | Roche | 10107735001 | |
| NativePAGE 3 to 12% Mini Protein Gels | Invitrogen | BN1001BOX | |
| NativePAGE Cathode Buffer Additive (20x) | Invitrogen | BN2002 | |
| NativePAGE Running Buffer (20x) | Invitrogen | BN2001 | |
| NDUFA9 Monoclonal Antibody | Invitrogen | 459100 | |
| Nitroblue tetrazolium salt (NBT) | Sigma-Aldrich | N6876 | |
| Pb(NO3)2 | Sigma-Aldrich | 228621 | |
| PDVF Membrane | Amersham | 10600023 | |
| Phenazine methasulfate (PMS) | Sigma-Aldrich | P9625 | |
| Pierce ECL Substrate | Thermo Scientific | 32106 | |
| PMSF | Merck | PMSF-RO | |
| SDHA Monoclonal Antibody | Invitrogen | 459200 | |
| Sodium succinate | Sigma-Aldrich | S2378 | |
| Streptomycin/penicillin | PAN biotech | P06-07100 | |
| Sucrose | Sigma-Aldrich | S3089 | |
| Tris | PanReac | A2264 | |
| UQCRC1 Monoclonal Antibody | Invitrogen | 459140 | |
| XCell SureLock Mini-Cell | Invitrogen | EI0001 |
Referenzen
- Acin-Perez, R., Fernandez-Silva, P., Peleato, M. L., Perez-Martos, A., Enriquez, J. A. Respiratory active mitochondrial supercomplexes. Mol Cell. 32 (4), 529-539 (2008).
- Schagger, H., Pfeiffer, K. Supercomplexes in the respiratory chains of yeast and mammalian mitochondria. EMBO J. 19 (8), 1777-1783 (2000).
- Schagger, H., Pfeiffer, K. The ratio of oxidative phosphorylation complexes I-V in bovine heart mitochondria and the composition of respiratory chain supercomplexes. J Biol Chem. 276 (41), 37861-37867 (2001).
- Acin-Perez, R., Enriquez, J. A. The function of the respiratory supercomplexes: the plasticity model. Biochim Biophys Acta. 1837 (4), 444-450 (2014).
- Cogliati, S., Cabrera-Alarcon, J. L., Enriquez, J. A. Regulation and functional role of the electron transport chain supercomplexes. Biochem Soc Trans. 49 (6), 2655-2668 (2021).
- Genova, M. L., Lenaz, G. Functional role of mitochondrial respiratory supercomplexes. Biochim Biophys Acta. 1837 (4), 427-443 (2014).
- Kohler, A., Barrientos, A., Fontanesi, F., Ott, M. The functional significance of mitochondrial respiratory chain supercomplexes. EMBO Rep. 24 (11), e57092 (2023).
- Lapuente-Brun, E., et al. Supercomplex assembly determines electron flux in the mitochondrial electron transport chain. Science. 340 (6140), 1567-1570 (2013).
- Milenkovic, D., et al. Preserved respiratory chain capacity and physiology in mice with profoundly reduced levels of mitochondrial respirasomes. Cell Metab. 35 (10), 1799-1813 (2023).
- Vercellino, I., Sazanov, L. A. The assembly, regulation and function of the mitochondrial respiratory chain. Nat Rev Mol Cell Biol. 23 (2), 141-161 (2022).
- Moreno-Loshuertos, R., Fernández-Silva, P., Ostojic, S. . Clinical Bioenergetics. , 3-60 (2021).
- Fernandez-Vizarra, E., Ugalde, C. Cooperative assembly of the mitochondrial respiratory chain. Trends Biochem Sci. 47 (12), 999-1008 (2022).
- Javadov, S., Jang, S., Chapa-Dubocq, X. R., Khuchua, Z., Camara, A. K. S. Mitochondrial respiratory supercomplexes in mammalian cells: structural versus functional role. Journal of Molecular Medicine. 99 (1), 57-73 (2021).
- Lopez-Fabuel, I., et al. Complex I assembly into supercomplexes determines differential mitochondrial ROS production in neurons and astrocytes. Proc Natl Acad Sci U S A. 113 (46), 13063-13068 (2016).
- Mukherjee, S., Ghosh, A. Molecular mechanism of mitochondrial respiratory chain assembly and its relation to mitochondrial diseases. Mitochondrion. 53, 1-20 (2020).
- Nesci, S., et al. Molecular and supramolecular structure of the mitochondrial oxidative phosphorylation system: implications for pathology. Life (Basel). 11 (3), 242 (2021).
- Frenzel, M., Rommelspacher, H., Sugawa, M. D., Dencher, N. A. Ageing alters the supramolecular architecture of OxPhos complexes in rat brain cortex. Exp Gerontol. 45 (7-8), 563-572 (2010).
- Morant-Ferrando, B., et al. Fatty acid oxidation organizes mitochondrial supercomplexes to sustain astrocytic ROS and cognition. Nat Metab. 5 (8), 1290-1302 (2023).
- McKenzie, M., Lazarou, M., Thorburn, D. R., Ryan, M. T. Mitochondrial respiratory chain supercomplexes are destabilized in Barth Syndrome patients. J Mol Biol. 361 (3), 462-469 (2006).
- Rosca, M. G., et al. Cardiac mitochondria in heart failure: decrease in respirasomes and oxidative phosphorylation. Cardiovasc Res. 80 (1), 30-39 (2008).
- Ramirez-Camacho, I., Garcia-Nino, W. R., Flores-Garcia, M., Pedraza-Chaverri, J., Zazueta, C. Alteration of mitochondrial supercomplexes assembly in metabolic diseases. Biochim Biophys Acta Mol Basis Dis. 1866 (12), 165935 (2020).
- Gonzalez-Rodriguez, P., et al. Disruption of mitochondrial complex I induces progressive parkinsonism. Nature. 599 (7886), 650-656 (2021).
- Novack, G. V., Galeano, P., Castano, E. M., Morelli, L. Mitochondrial supercomplexes: physiological organization and dysregulation in age-related neurodegenerative disorders. Front Endocrinol (Lausanne). 11, 600 (2020).
- Ramirez-Camacho, I., Flores-Herrera, O., Zazueta, C. The relevance of the supramolecular arrangements of the respiratory chain complexes in human diseases and aging. Mitochondrion. 47, 266-272 (2019).
- Hollinshead, K. E. R., et al. Respiratory Supercomplexes Promote Mitochondrial Efficiency and Growth in Severely Hypoxic Pancreatic Cancer. Cell Rep. 33 (1), 108231 (2020).
- Ikeda, K., et al. Mitochondrial supercomplex assembly promotes breast and endometrial tumorigenesis by metabolic alterations and enhanced hypoxia tolerance. Nat Commun. 10 (1), 4108 (2019).
- Kamada, S., Takeiwa, T., Ikeda, K., Horie, K., Inoue, S. Emerging roles of COX7RP and mitochondrial oxidative phosphorylation in breast cancer. Front Cell Dev Biol. 10, 717881 (2022).
- Marco-Brualla, J., et al. Mutations in the ND2 subunit of mitochondrial complex I are sufficient to confer increased tumorigenic and metastatic potential to cancer cells. Cancers (Basel). 11 (7), 1027 (2019).
- Moreno-Loshuertos, R., et al. How hot can mitochondria be? Incubation at temperatures above 43 degrees C induces the degradation of respiratory complexes and supercomplexes in intact cells and isolated mitochondria. Mitochondrion. 69, 83-94 (2023).
- Vonck, J., Schafer, E. Supramolecular organization of protein complexes in the mitochondrial inner membrane. Biochim Biophys Acta. 1793 (1), 117-124 (2009).
- Althoff, T., Mills, D. J., Popot, J. L., Kuhlbrandt, W. Arrangement of electron transport chain components in bovine mitochondrial supercomplex I1III2IV1. EMBO J. 30 (22), 4652-4664 (2011).
- Cogliati, S., et al. Mechanism of super-assembly of respiratory complexes III and IV. Nature. 539 (7630), 579-582 (2016).
- Gonzalez-Franquesa, A., et al. Mass-spectrometry-based proteomics reveals mitochondrial supercomplexome plasticity. Cell Rep. 35 (8), 109180 (2021).
- Wittig, I., Schagger, H. Features and applications of blue-native and clear-native electrophoresis. Proteomics. 8 (19), 3974-3990 (2008).
- Wittig, I., Schagger, H. Native electrophoretic techniques to identify protein-protein interactions. Proteomics. 9 (23), 5214-5223 (2009).
- Garcia-Cazarin, M. L., Snider, N. N., Andrade, F. H. Mitochondrial isolation from skeletal muscle. J Vis Exp. (49), e2452 (2011).
- Lai, N., et al. Isolation of mitochondrial subpopulations from skeletal muscle: Optimizing recovery and preserving integrity. Acta Physiol (Oxf). 225 (2), e13182 (2019).
- Schagger, H. Native electrophoresis for isolation of mitochondrial oxidative phosphorylation protein complexes. Methods Enzymol. 260, 190-202 (1995).
- Wittig, I., Braun, H. P., Schagger, H. Blue native PAGE. Nat Protoc. 1 (1), 418-428 (2006).
- Chomyn, A., et al. Platelet-mediated transformation of mtDNA-less human cells: analysis of phenotypic variability among clones from normal individuals--and complementation behavior of the tRNALys mutation causing myoclonic epilepsy and ragged red fibers. Am J Hum Genet. 54 (6), 966-974 (1994).
- Moreno-Loshuertos, R., et al. Differences in reactive oxygen species production explain the phenotypes associated with common mouse mitochondrial DNA variants. Nat Genet. 38 (11), 1261-1268 (2006).
- Fernández-Vizarra, E., Fernández-Silva, P., Enríquez, J. A., Celis, J. E. . Cell Biology (Third Edition). , 69-77 (2006).
- Cogliati, S., Herranz, F., Ruiz-Cabello, J., Enríquez, J. A. Digitonin concentration is determinant for mitochondrial supercomplexes analysis by BlueNative page. Biochim Biophys Acta Bioenerg. 1862 (1), 148332 (2021).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten