Method Article
Ein auf Durchflusszytometrie basierender Zelloberflächenproteinbindungsassay zur Beurteilung der Selektivität und Spezifität eines Antikrebs-Aptamers
In diesem Artikel
Zusammenfassung
Ein notwendiger Schritt in der Entwicklung von Antikrebs-Aptamern besteht darin, seine Bindung an das Ziel zu testen. Wir demonstrieren einen durchflusszytometrischen Assay zur Untersuchung dieser Bindung und betonen die Bedeutung der Einbeziehung eines Negativkontrollaptamers und Krebszellen, die für dieses bestimmte Protein positiv oder negativ sind.
Zusammenfassung
Eine zentrale Herausforderung bei der Entwicklung eines Antikrebs-Aptamers besteht darin, die Selektivität und Spezifität des entwickelten Aptamers für das Zielprotein effizient zu bestimmen. Aufgrund seiner zahlreichen Vorteile gegenüber monoklonalen Antikörpern hat die Aptamer-Entwicklung unter Krebsforschern enorme Popularität erlangt. Die systematische Evolution von Liganden durch exponentielle Anreicherung (SELEX) ist die gebräuchlichste Methode zur Entwicklung von Aptameren, die spezifisch für Proteine von Interesse sind. Nach SELEX beschleunigt ein schneller und effizienter Bindungsassay den Identifizierungsprozess und bestätigt die Selektivität und Spezifität des Aptamers.
Dieser Artikel erläutert einen schrittweisen durchflusszytometrischen Bindungsassay eines Aptamers, das spezifisch für epitheliale zelluläre Adhäsionsmoleküle (EpCAM) ist. Das Transmembran-Glykoprotein EpCAM wird in den meisten Karzinomen überexprimiert und spielt eine Rolle bei der Initiierung, Progression und Metastasierung von Krebs. Daher ist es ein wertvoller Kandidat für die gezielte Medikamentenabgabe an Tumore. Um die Selektivität und Spezifität des Aptamers gegenüber der membrangebundenen EpCAM zu bewerten, werden EpCAM-positive und -negative Zellen benötigt. Zusätzlich wird ein unverbindlicher EpCAM-Aptamer mit ähnlicher Länge und 2-dimensionaler (2D) Struktur wie der EpCAM-bindende Aptamer benötigt. Der Bindungsassay umfasst verschiedene Puffer (Blockierungspuffer, Waschpuffer, Inkubationspuffer und FACS-Puffer) und Inkubationsschritte.
Das Aptamer wird mit den Zelllinien inkubiert. Nach den Inkubations- und Waschschritten werden die Zellen mit einem empfindlichen Durchflusszytometrie-Assay bewertet. Die Analyse der Ergebnisse zeigt die Bindung des EpCAM-spezifischen Aptamers an EpCAM-positive Zellen und nicht an die EpCAM-negativen Zellen. In EpCAM-positiven Zellen wird dies als Bandverschiebung in der Bindung des EpCAM-Aptamers nach rechts im Vergleich zur unverbindlichen Aptamerkontrolle dargestellt. In EpCAM-negativen Zellen überlappen sich die entsprechenden Banden von EpCAM-bindenden und -nicht-bindenden Aptameren. Dies zeigt die Selektivität und Spezifität des EpCAM-Aptamers. Während sich dieses Protokoll auf den EpCAM-Aptamer konzentriert, ist das Protokoll auf andere veröffentlichte Aptamere anwendbar.
Einleitung
Krebs ist nach wie vor eine der häufigsten Todesursachen weltweit1. Trotz der deutlichen Verbesserung der Krebsbehandlung in den letzten Jahrzehnten ist die Entwicklung von Krebsmedikamenten immer noch ein viel diskutiertes Thema. Dies liegt daran, dass die Chemotherapie als Hauptstütze der Krebsbehandlung von schwerwiegenden Nebenwirkungen begleitet wird, die die Einhaltung der Behandlung durch den Patienten einschränken. Darüber hinaus hat die durch Chemotherapie induzierte Krebsresistenz ihre Anwendung als einzige Wahl der medizinischen Intervention eingeschränkt. Die Anwendung von monoklonalen Antikörpern (mAbs) führte zu einem verbesserten Ansprechen aufKrebsbehandlungen 2. Der Grund für die Verwendung von mAbs bestand darin, die Wirksamkeit von Chemotherapeutika zu verbessern und ihre Nebenwirkungen zu minimieren. Die Verabreichung von mAbs wurde jedoch auch zu einer Herausforderung. Dies lag nicht nur an den mAb-induzierten immunologischen Reaktionen, sondern auch an den tierabhängigen und teuren Produktionskosten und schwierigen Lagerbedingungen3. Die Einführung von Aptameren in den 1990er Jahren4 weckte neue Hoffnungen in der Krebsbehandlung, da die Anwendung von Aptameren die mit mAbs verbundenen Herausforderungen angehen könnte.
Aptamere sind kurze Nukleinsäuresequenzen, die spezifisch für ein bestimmtes Ziel hergestellt werden. Die systematische Evolution von Liganden durch exponentielle Anreicherung (SELEX) ist eine gängige Methode in der Aptamerproduktion. In SELEX wird das interessierende Protein mit einer Bibliothek zufälliger Nukleotidsequenzen inkubiert, und durch eine Reihe von iterativen Zyklen wird das für dieses Protein spezifische Aptamer gereinigt. Aptamere haben eine ähnliche Zielselektivität und Spezifität wie mAbs, und daher zeigt die Medikamentenentwicklung in diesem Bereich vielversprechende zukünftige Anwendungen. Aptamere spezifisch für Krebsbiomarker könnten als Einzelmedikamente und Krebsdiagnosewerkzeuge eingesetzt werden 5,6,7. Aufgrund ihrer nanoskaligen Struktur könnten diese Aptamere auch als Wirkstoffträger fungieren, um zytotoxische Wirkstoffe spezifisch an den Tumor abzugeben8. Dies würde die Wirksamkeit der gezielten Medikamentenabgabe erhöhen und chemotherapieassoziierte, Off-Target-Nebenwirkungen verringern. Darüber hinaus haben diese Nanomedikamente eine hohe Gewebepenetration, was sie zu einem wünschenswerten Kandidaten für die Verabreichung und Behandlung von Medikamenten in tiefen Tumoren macht. Aptamere können auch so konzipiert werden, dass sie auf die Transporter abzielen, die auf der Blut-Hirn-Schranke (BBB) exprimiert werden, um die Medikamentenabgabe an Hirntumoren zu verbessern9. Ein gutes Beispiel für ein solches Aptamer sind bifunktionelle Aptamere, die auf den Transferrinrezeptor (TfR)10 abzielen, um die Wirkstoffabgabe über die BHS zu verbessern, und eine zytotoxische Wirkstoffnutzlast an Tumorzellen abgeben11.
Trotz aller Vorteile von Aptameren hat die Medikamentenentwicklung auf diesem Gebiet noch kein vermarktetes, erfolgreiches Krebsmedikament hervorgebracht. Ein Grund dafür könnte das Fehlen standardisierter und reproduzierbarer Methoden sein, denen Forscher weltweit folgen könnten. In dieser Arbeit wird ein Schritt-für-Schritt-Protokoll eines Aptamers demonstriert, das an ein natives Protein bindet, das auf der Zelloberfläche exprimiert wird. Dieses Protokoll ist ein Voraussetzung für die präklinische Bewertung von Antikrebs-Aptameren. Der Assay wird durchgeführt, um die Selektivität und Spezifität des gereinigten Aptamers aus SELEX oder einer veröffentlichten Aptamersequenz zur Bestätigung der Selektivität und Spezifität nachzuweisen. Dieser durchflusszytometrische Assay ist ein schneller, zuverlässiger und empfindlicher Assay, der die Selektivität und Spezifität des Aptamers genau zeigt, wobei das Aptamer gegen Proteine auf der Zelloberflächegetestet wird 12,13,14. Diese Methode wird anhand der Bindung eines für EpCAM spezifischen Aptamers demonstriert, das in diesem Paper15 gezeigt wird. EpCAM spielt als Transmembran-Glykoprotein eine Rolle bei der Signalisierung, Progression, Migration und Metastasierung von Tumorzellen16,17. Um die Selektivität und Spezifität dieses Aptamers zu zeigen, wurden EpCAM-positive und -negative Krebszellen verwendet. Das zuvor entwickelte EpCAM-spezifische Aptamer TEPP (5′-GC GCG GTAC CGC GC TA ACG GA GGTTGCG TCC GT-3′) und ein Negativkontrollaptamer, TENN (5′-GC GCG TGCA CGC GC TA ACG GA TTCCTTT TCC GT-3), wurden als EpCAM-bindende bzw. -unverbindliche Aptamere verwendet,10. Das 3'-Ende von TEPP und TENN wurde mit einem TYE665-Fluorophor markiert.
TEPP ist ein bifunktionaler Aptamer, der von einem Ende auf EpCAM und auf der anderen Seite auf TfR abzielt. Dies hat TEPP zu einem geeigneten Kandidaten für die Medikamentenabgabe bei EpCAM+ -Hirntumoren gemacht. Mit seinem TfR-spezifischen Ende durchquert TEPP die Blut-Hirn-Schranke und findet mit dem EpCAM-spezifischen Ende den Tumor und liefert seine Fracht (z. B. zytotoxische Medikamente) an den Tumor. TENN hat eine ähnliche Länge und 2D-Struktur wie TEPP, hat aber keine Affinität zur EpCAM oder TfR und ist daher ein geeigneter Negativkontrollaptamer. Unter Verwendung von TEPP und TENN wird in diesem Artikel gezeigt, wie die Bindung eines Aptamers an das Zielprotein mittels Durchflusszytometrie getestet wird. Dieses Protokoll gilt für die Entwicklung zellspezifischer Aptamere. Sie ist auch auf weitere Komplementär- und Bestätigungsanalysen der in der Literatur verfügbaren Aptamersequenzen anwendbar. Das Protokoll kann auch von Aptamer-Neulingen verwendet werden, die ein zuvor veröffentlichtes Aptamer für ihre Forschungs- und Entwicklungszwecke (F & E) verwenden möchten. In dieser Arbeit werden zwei in der Literatur verfügbare Aptamersequenzen untersucht.
Protokoll
HINWEIS: Tragen Sie vor Beginn des Experiments persönliche Schutzausrüstung, einschließlich Laborkittel, Handschuhe und Schutzbrille. Weitere Informationen zu Materialien, Reagenzien, Geräten und Software, die in diesem Protokoll verwendet werden, finden Sie in der Materialtabelle .
1. Für den Assay erforderliche Puffer
- Bereiten Sie die für dieses Experiment erforderlichen Puffer - den für die Aptamerfaltung erforderlichen SELEX-Puffer, den Blocking Buffer (BB), den Wash Buffer (WB) und den Binding Buffer (BiB) (Tabelle 1) - frisch am Tag des Versuchs vor und bewahren Sie sie auf Eis oder bei 4 °C auf.
HINWEIS: Jeder Aptamer benötigt eine einzigartige Faltbedingung. Dazu gehören der SELEX-Puffer und die Falttemperaturbedingungen. Es sollte darauf geachtet werden, dass die Methoden aus der Originalarbeit, die das Aptamer10 beschreibt, vollständig repliziert werden. In diesem Experiment werden alle Puffer in Dulbeccos phosphatgepufferter Kochsalzlösung (DPBS) hergestellt. Das in jedem Experiment erforderliche Puffervolumen hängt von der Anzahl der Zelllinien, der Anzahl der Replikate und der Anzahl der getesteten Aptamerkonzentrationen ab.
| Zutaten | Erforderliches Volumen | ||
| Artikel | Konzentration | ||
| SELEX Puffer | MgCl2 | 5 mM | 50 μL pro Probe + 10% Pipettierfehler |
| Blockieren des Puffers | MgCl2 | 5 mM | 500 μL pro Zelllinie |
| BSA a | 1 mg/ml | ||
| tRNA b | 0,1 mg/ml | ||
| FBS c | 10% (v/v) | ||
| Waschpuffer | MgCl2 | 5 mM | 1 mL für die erste Wäsche + 100 μL pro Testprobe + 10% Pipettierfehler |
| Bindungspuffer | MgCl2 | 5 mM | 50 μL pro Probe + 10% Pipettierfehler |
| BSA | 2 mg/ml | ||
| trna | 0,2 mg/ml | ||
| FBS | 20% (v/v) | ||
Tabelle 1: Erforderliche Puffer für den Bindungsassay. ein Rinderserumalbumin, bTransfer Ribonukleinsäure, cFötales Rinderserum.
2. Herstellung von Aptameren
HINWEIS: Die im Assay verwendeten Aptamere sind mit einem Fluoreszenzreportermolekül markiert und daher sollte darauf geachtet werden, sie vor Licht zu schützen.
- Vor dem Experiment einen 100 μM großen Vorrat (Bestand A) von Test- und Kontrollaptameren unter Verwendung von pyrogen- und RNase-freiem Reinstwasser vorbereiten (Abbildung 1).
HINWEIS: Zur Langzeitkonservierung sollte Vorrat A im Gefrierschrank bei -20 °C aufbewahrt werden. - Brühe B wird als Arbeitskonzentration von Aptameren hergestellt, indem Material A mit SELEX-Puffer verdünnt wird (Tabelle 1). Um dieses Protokoll zu befolgen, verdünnen Sie Bestand A auf eine 1.000 nM-Aktie, um Vorrat B vorzubereiten (Abbildung 1).
- Um das Aptamer für die Bildung der 3-dimensionalen (3D) Struktur bereit zu machen, verdünnen Sie in einem 250-μL-Röhrchen Brühe B mit SELEX-Puffer, um das erforderliche Volumen und die erforderliche Konzentration des Aptamers für die Faltung vorzubereiten.
HINWEIS: Die gefalteten Aptamere werden einem gleichen Zellvolumen ausgesetzt. Daher sollte die Konzentration des Aptamers, das zum Falten eingestellt ist, 2x konzentrierter sein als die gewünschte Endkonzentration. Verwenden Sie Gleichung (1), um die erforderlichen Volumina und Konzentrationen zu berechnen. Denken Sie daran, ein zusätzliches Volumen von 10% für den Pipettierfehler zu berücksichtigen.
Konzentrationsbestand A × Mengenbestand A = Konzentrationsbestand B × Mengenbestand B (1)

Abbildung 1: Ein Diagramm mit den Schritten bei der Herstellung von Aptameren. 1 Bestand 1wird zur Langzeitkonservierung bei -20 °C gelagert. arabische Ziffer Arbeitskonzentrationen werden im SELEX-Puffer aufbereitet und nicht gespeichert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
3. Erhaltung von Krebszellen
HINWEIS: Stellen Sie vor Beginn der Studie sicher, dass die Zellen ihre frühen Passagezahlen erreicht haben, ihre typischen morphologischen Merkmale aufweisen und frei von Mykoplasmen sind. Um die Selektivität und Spezifität des Aptamers zu testen, sind idealerweise Zelllinien erforderlich, die hohe, moderate und niedrige/negative Expressionoren des interessierenden Proteins sind.
- Die Zellen werden unter Verwendung geeigneter Kulturbedingungen in einem T75-Kulturkolben ausgesät. Züchten Sie sie in einem 5% CO2 befeuchteten Inkubator bei 37 °C.
HINWEIS: In dieser Studie wurde Dulbeccos modifiziertes Adlermedium (DMEM), ergänzt mit 10% fetalem Rinderserum (vollständiges Medium), verwendet. - Wenn die Zellen ~ 80% Konfluenz erreicht haben, geben Sie sie in einen neuen Kolben mit frischem vollständigem Medium.
HINWEIS: Abhängig vom interessierenden Protein und der Zelllinie könnte eine 80%ige Konfluenz eine geeignete Zellpopulation für den Bindungstest liefern. Für die Zelllinien in diesem Experiment, MDA-MB-231 und HEK 293T, ist eine 80%ige Konfluenz geeignet. Fahren Sie in diesem Stadium mit Abschnitt 4, dem verbindlichen Assay, fort. Überprüfen Sie immer die Expression des interessierenden Proteins mit mAbs, die für dieses Protein spezifisch sind.
4. Verbindlicher Assay
HINWEIS: Abbildung 2 fasst die Schritte zusammen, die im Bindungsassay in adhärenten Zellen erforderlich sind.
- In einer Biosicherheitswerkbank der Klasse II werden die Zellen jedes Kolbens wie folgt in Röhrchen gesammelt:
- Sammeln und entsorgen Sie die Medien im Kolben, fügen Sie 2 ml PBS hinzu, verteilen Sie es auf die Zellen und sammeln und entsorgen Sie dann das PBS. Wiederholen Sie diesen Schritt noch zweimal, um alle Spuren von Medien zu entfernen, die Trypsin deaktivieren können. 1 ml 0,25% Trypsin/EDTA in jeden Kolben geben und 5-10 min bei 37 °C inkubieren.
- Fügen Sie 1 ml komplettes Medium zu den Zellen hinzu und pipetten Sie die Zellen nach oben und unten, um eine einzellige Suspension herzustellen. Die Zellen in ein geeignetes Röhrchen pipettieren und bei 200 × g für 5 min zentrifugieren.
HINWEIS: Bei nicht adhärenten Zellen sammeln Sie die Zellen in einem Röhrchen, zentrifugieren (200 × g, 5 min) und fahren Sie mit Schritt 4.1.3 fort. - Verwerfen Sie den Überstand und resuspendieren Sie die Zellen in 1 ml frischem Medium. Zählen Sie die Zellen mit Trypanblau-Färbung, indem Sie ein bestimmtes Volumen der Zellsuspension mit Trypanblau verdünnen. Verteilen Sie ~ 15 μL der Mischung zwischen einem Hämozytometer und einem Deckglas. Zählen Sie die Zellen wie zuvor beschrieben18 mit Gleichung (2):
 (2)
(2)
HINWEIS: Verwenden Sie das minimal mögliche Volumen der Zellsuspension und notieren Sie sich den Verdünnungsfaktor. Zum Beispiel ergibt das Mischen gleicher Volumina der Zellsuspension und 0,04% Trypanblau einen Verdünnungsfaktor von 2. Stellen Sie eine hohe Lebensfähigkeit (lebende Zellen / Gesamtzellen × 100) von ~ 90% für die meisten adhärenten Zelllinien sicher, bevor Sie fortfahren. Tote Zellen nehmen unspezifisch Aptamere auf und verändern die Ergebnisse19. Es ist möglich, andere Zellzähltechniken zu verwenden, z. B. die Verwendung eines Zellzählers. - Sammeln Sie die erforderliche Anzahl von Zellen und stellen Sie sicher, dass Sie 10 × 104 Zellen pro Testprobe haben. Berücksichtigen Sie ein zusätzliches Volumen von 10% für den Pipettierfehler.
HINWEIS: Es ist wichtig, zwischen Experimenten und Replikationen immer die gleiche Zellzahl beizubehalten. - Die Zellen werden bei 37 °C für 2 h inkubiert, um die Stabilisierung des interessierenden Proteins auf der Zellmembran nach enzymatischer Ablösung zu ermöglichen.
HINWEIS: Diese Inkubationszeit kann je nach interessierendem Protein variieren.
- Während dieser 2-stündigen Inkubation:
- Stellen Sie die Temperatur der Zentrifuge auf 4 °C ein. Lassen Sie tRNA und Aptamervorräte bei Raumtemperatur oder auf Eis auftauen. Um das Fluoreszenzreportermolekül zu schützen, schützen Sie die Aptamerröhren vor Licht.
HINWEIS: Die Rolle der tRNA besteht darin, die Nukleinsäurebindungsstellen zu blockieren. - Bereiten Sie den SELEX-Puffer, BB, WB und BiB vor (siehe Abschnitt 1) und halten Sie sie alle auf Eis oder bei 4 °C. Stellen Sie die Thermocycler-Maschine auf einen leeren Zyklus. Legen Sie eine 96-Well-schwarze Platte und Durchflusszytometrieröhrchen auf Eis.
HINWEIS: Das Einstellen des Thermocyclers auf einen leeren Zyklus bereitet das Kühl- und Heizsystem vor und hilft, reproduzierbare Ergebnisse zu erzielen.
- Stellen Sie die Temperatur der Zentrifuge auf 4 °C ein. Lassen Sie tRNA und Aptamervorräte bei Raumtemperatur oder auf Eis auftauen. Um das Fluoreszenzreportermolekül zu schützen, schützen Sie die Aptamerröhren vor Licht.
- Nach der 2-stündigen Inkubation die Zellen bei 500 × g für 5 min zentrifugieren. Verwerfen Sie den Überstand und resuspendieren Sie die Zellen in 500 μL BB. Inkubieren Sie die Zellen bei 4 °C für 30 min mit intermittierendem Mischen.
- Führen Sie während dieser 30-minütigen Inkubation die Aptamerfaltung wie folgt durch:
- Bilden Sie die 2x-Konzentrationen der Aptamere (siehe Abschnitt 2) und mischen und inkubieren Sie dann die Aptamere in der Thermocycler-Maschine entsprechend den erforderlichen Faltbedingungen. Verwenden Sie für dieses EpCAM-Aptamer die folgenden Faltbedingungen von 95 °C, 5 min, gefolgt von 22 °C, 10 min, und 37 °C, 15 min.
HINWEIS: Fügen Sie immer eine Negativkontrolle hinzu (z. B. SELEX-Puffer ohne Aptamere).
- Bilden Sie die 2x-Konzentrationen der Aptamere (siehe Abschnitt 2) und mischen und inkubieren Sie dann die Aptamere in der Thermocycler-Maschine entsprechend den erforderlichen Faltbedingungen. Verwenden Sie für dieses EpCAM-Aptamer die folgenden Faltbedingungen von 95 °C, 5 min, gefolgt von 22 °C, 10 min, und 37 °C, 15 min.
- Nach der 30-minütigen Inkubation die Zellen zentrifugieren (500 × g, 5 min, 4 °C), den Überstand entfernen, 1 ml WB hinzufügen und die Zellen erneut zentrifugieren (500 × g, 5 min, 4 °C). Entfernen Sie den Überstand und resuspendieren Sie die Zellen in einem geeigneten Volumen des BiB.
- Pipettieren Sie 50 μL der resuspendierten Zellen in jede Vertiefung einer eiskalten, 96-fach schwarzen Platte. Halten Sie die Zellen auf Eis, um die Internalisierung des interessierenden Proteins zu hemmen.
- 50 μL der Aptamere auf ein 50 μL Zellvolumen pipettieren, mischen und bei Dunkelheit bei 4 °C 30 min inkubieren. Zentrifugieren Sie die Platte bei 500 × g, 5 min, 4 °C und entfernen Sie vorsichtig den Überstand.
- Das Pellet vorsichtig in WB resuspendieren und 5 min bei 500 × g zentrifugieren. Wiederholen Sie den Waschschritt (4.7) 2x und resuspendieren Sie ihn in 100 μL WB für die durchflusszytometrische Analyse.
HINWEIS: In Abbildung 3 finden Sie ein Diagramm der Interaktionen zwischen Aptameren und Zellen.

Abbildung 2: Ein Diagramm, das die Schritte zur Durchführung eines Aptamer-Protein-bindenden Assays darstellt. Abkürzungen: SELEX = Systematic Evolution of Ligands by EXponential Enrichment; BB = Sperrpuffer; WB = Waschpuffer; BiB = Bindungspuffer. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 3: Ein Diagramm, das die verschiedenen Zelltypen und Aptamere zeigt, die für die Durchführung des Aptamerbindungstests erforderlich sind. Abkürzung: EpCAM = epitheliales zelluläres Adhäsionsmolekül. Diese Figur wurde mit Biorender.com erstellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
5. Durchflusszytometrie und Datenanalyse
HINWEIS: Stellen Sie vor dem Einschalten des Durchflusszytometers sicher, dass sich in den Membranfiltereinheiten keine "Blasen" für die Abschaltlösung, die Reinigungslösung und die Mantelflüssigkeit (0,9% NaCl) befinden. "Ausbluten" Blasen, wenn sich Blasen in den Kapseln befinden. Stellen Sie sicher, dass der Abfallbehälter leer ist und die Behälter mit Mantelflüssigkeit, Wasser und 1% Bleichmittel in Reinstwasser voll sind.
- Schalten Sie das Durchflusszytometer und dann den Computer ein.
HINWEIS: Die hier erläuterten Details zum Betrieb des Durchflusszytometers sind spezifisch für die im Video demonstrierte Maschine und Software (siehe Materialtabelle). Andere Software würde eine entsprechende Schulung erfordern. - Öffnen Sie die Durchflusszytometrie-Analysesoftware, melden Sie sich beim Programm an, und führen Sie auf der Registerkarte Zytometer Fluidics Start aus.
- Um ein neues Experiment zu erstellen, klicken Sie auf der Registerkarte Experiment auf Neuer Ordner, und benennen Sie den Ordner/das Experiment entsprechend.
- Klicken Sie auf Neuer Ordner, um ihn hervorzuheben, klicken Sie dann erneut auf der Registerkarte Experiment auf Neues Experiment und benennen Sie das Experiment entsprechend.
- Um die erste Probe/Probe hinzuzufügen, klicken Sie auf der Registerkarte Experiment auf Neue Probe und benennen Sie diese Probe entsprechend (Name der Zelllinie/Kontrollprobe/Experimentprobe).
- Um eine Röhrchenprobe hinzuzufügen, markieren Sie die Probe (Gruppe), und klicken Sie auf der Registerkarte Experiment auf Neues Röhrchen. Fügen Sie die entsprechende Anzahl von Röhren und den Namen hinzu.
- Um die erforderlichen Diagramme vorzubereiten, öffnen Sie auf der Registerkarte Arbeitsblatt ein neues Arbeitsblatt . Sobald das neue Arbeitsblattfenster angezeigt wird, öffnen Sie Folgendes über den Arbeitsblattbildschirm (bewegen Sie den Mauszeiger über das Logo/die Bilder, um die Namen zu finden):
- Bereiten Sie ein Punkt-Blot-Diagramm der Vorwärtsstreuung (FSC) im Vergleich zur Seitenstreuung (SCC) vor, um die gewünschte Grundgesamtheit auszuwählen. Definieren Sie das erste Gatter, indem Sie die gewünschte Grundgesamtheit (P1) in einem Vorwärts- und Seitenstreudichtediagramm identifizieren und auswählen. Schließen Sie die Trümmer aus, die die Population mit dem niedrigsten Vorwärtsstreusignal darstellen.
HINWEIS: Der FSC-Parameter erkennt Zellen oder Ereignisse basierend auf ihrer Größe und der SCC unterscheidet sie anhand ihrer Granularität20. - Bereiten Sie ein Punkt-Blot-Diagramm der FSC-Fläche (FSC-A) im Vergleich zur FSC-Höhe (FSC-H) vor, um die Einzelzellpopulation auszuwählen. Definieren Sie das zweite Tor, indem Sie Dublettenzellpopulationen ausschließen, da Dublettenzellen die Ergebnisse und Schlussfolgerungen erheblich beeinflussen. Schließen Sie Dubletten aus, indem Sie FSC-H- und FSC-A-Dichtediagramme verwenden, bei denen Zellen gleicher Größe eine ähnliche Fläche und Höhe aufweisen. Daher werden die Singlets diagonal gruppiert und von Dubletten getrennt.
HINWEIS: FSC ist ungefähr proportional zur Zellgröße. Die Spannungsimpulse sind definiert als FSC-H, die Intensität des Signals, FSC-Breite, die die Zellgröße und die Dauer des Signals widerspiegelt, und FSC-A, das H × W ist. Daher basiert das Gating für Singlets auf der Erkennung von Disproportionen zwischen H, W und A, die durch Dubletten verursacht werden. - Bereiten Sie ein Histogramm der Anzahl der Ereignisse gegen das interessierende Fluorophor vor.
- Bereiten Sie ein Punkt-Blot-Diagramm der Vorwärtsstreuung (FSC) im Vergleich zur Seitenstreuung (SCC) vor, um die gewünschte Grundgesamtheit auszuwählen. Definieren Sie das erste Gatter, indem Sie die gewünschte Grundgesamtheit (P1) in einem Vorwärts- und Seitenstreudichtediagramm identifizieren und auswählen. Schließen Sie die Trümmer aus, die die Population mit dem niedrigsten Vorwärtsstreusignal darstellen.
- Stellen Sie vor Beginn der Durchflusszytometrie sicher, dass das Erfassungs-Dashboard zur Steuerung der Probenerfassung, der Inspektor und das Zytometer zur Anpassung der Spannungsparameter sowie das Arbeitsblatt mit allen Diagrammen geöffnet sind.
HINWEIS: Zur Durchführung der Analyse werden mindestens 100 μL einer 10 ×10 4-Zell-Suspension in einem Durchflusszytometrieröhrchen benötigt. Insbesondere bei geringeren Vitrinen kann eine Propidiumiodidfärbung durchgeführt werden, um die lebensfähige Zellpopulationauszuwählen 21,22. - Um die erste Probe auszuführen, stellen Sie auf der linken Seite des Bildschirms sicher, dass der Pfeil, der auf das Röhrchen zeigt, grün ist. Wenn dieser Pfeil nicht grün ist, klicken Sie auf den Pfeil, um ihn grün zu machen.
- Übertragen Sie jede Probe mit einer Pipette von der schwarzen 96-Well-Platte in ein Durchflusszytometrieröhrchen. Führen Sie die unbehandelte, ungefärbte Kontrollprobe bei niedriger Geschwindigkeit aus.
- Wählen Sie im Erfassungsdashboard eine geeignete Anzahl von Ereignissen aus, die aufgezeichnet werden sollen (30.000), ändern Sie die Durchflussrate in niedrig und klicken Sie auf Daten erfassen.
- Stellen Sie die Spannung für die FSC- und SCC-Parameter ein. Stellen Sie sicher, dass die Zellpopulation innerhalb des Punktdiagramms zentralisiert ist und dass keine Zellen eine der beiden Achsen des Diagramms berühren, um zu vermeiden, dass die interessierenden Zellen verloren gehen.
- Erhöhen Sie die Erfassungsgeschwindigkeit auf mittel oder hoch, um die Proben schneller zu analysieren, aber nicht mehr als 200 Ereignisse/s zu überschreiten. Klicken Sie dann auf Daten aufzeichnen.
- Führen Sie die Gating für P1 (Abbildung 4A) und die Einzelzellpopulation (Abbildung 4B) durch. Konstruieren Sie das Histogramm der Ereignisse gegen das verwendete Fluorochrom und wählen Sie P1 basierend auf den Daten (in diesem Fall Allophycocyanin (APC)) (Abbildung 4C).
- Nachdem Sie die Spannung eingestellt, angegessen und die Daten aufgezeichnet haben, nehmen Sie die Probe heraus und klicken Sie auf Next Tube.
- Legen Sie die nächste Probe ein, und wiederholen Sie die Aufzeichnungsdaten für alle Kontroll- und Testproben (Abbildung 3).
- Sobald alle Daten gesammelt sind, waschen Sie das Durchflusszytometer, indem Sie drei Röhrchen mit 50% Bleichmittel, FACS-Spülung und Reinstwasser jeweils 5 Minuten lang mit einer hohen Durchflussrate laufen lassen.
- Klicken Sie dann im Dropdown-Menü Zytometer auf Fluidics Shut Down.
- Bevor Sie die Software schließen und den Computer und den Computer ausschalten, exportieren Sie die Ergebnisse als .fcs-Dateien auf ein USB-Laufwerk, um sie wie folgt zu übertragen und zu analysieren:
- Drücken Sie in der Analysesoftware die Taste NEU , um ein neues Dokument und Fenster für die Analyse zu erstellen. Ziehen Sie die Beispieldateien in das neue Fenster.
- Doppelklicken Sie, um die ungefärbte Probe zu öffnen. Wählen Sie die P1-Population aus, doppelklicken Sie auf die P1-Population, um ein FSC-H- versus FSC-A-Diagramm zu erstellen, und gate Sie die Einzelzellpopulation.
- Doppelklicken Sie auf die einzelnen Zellen, um ein Histogramm der Ereignisse gegen das verwendete Fluorochrom zu erstellen.
- Wählen Sie im ursprünglichen Fenster P1 und einzelne Zellen aus und ziehen Sie sie auf Alle Stichproben , sodass alle Proben jetzt den gleichen Gating enthalten.
- Klicken Sie auf die Schaltfläche Layout-Editor , um das Fenster Layouts zu öffnen. Ziehen Sie zwei Stichproben (Kontrolle und Test) übereinander, um ein Overlay-Histogramm zu erstellen.
Ergebnisse
Ein wichtiger Aspekt der Entdeckung und Entwicklung neuer Wirkstoffe ist die Sicherstellung der Selektivität und Spezifität des Wirkstoffkandidaten. Dies bedeutet, dass der Wirkstoffkandidat in der Lage sein sollte, zwischen verschiedenen Zellen zu unterscheiden und nur die interessierende Zellpopulation zu beeinflussen (Selektivität). Die Selektivität wird anhand von Zelllinien untersucht, die sich in der Expression des interessierenden Proteins unterscheiden. In dieser Studie wurden MDA-MB-231 und HEK 293T Zelllinien als EpCAM-positive und -negative Zellen ausgewählt. Die Spezifität ist eine weitere Determinante, die zeigt, dass das interessierende Protein nur auf einen einzigen Medikamentenkandidaten reagiert. Hier wurde unter Verwendung eines EpCAM-unverbindlichen Aptamers, TENN, gezeigt, dass nur TEPP an EpCAM angeschlossen ist. In EpCAM-positiven Zellen (MDA-MB-231) zeigt die Überlagerung der Histogramme, die mit TEPP und TENN behandelte Zellen darstellen, dass die TEPP-behandelten Zellen im Vergleich zu den TENN-behandelten Zellen nach rechts verschoben sind. Dies zeigt die Bindung des Aptamers TEPP an das interessierende Protein EpCAM (Abbildung 4D). In Negativkontrollzellen von HEK 293T spiegelt die Überlagerung von Histogrammen von TEPP und TENN keine Verschiebung wider (Abbildung 4E). Dies bedeutet, dass in EpCAM-exprimierenden Zellen TEPP als EpCAM-Aptamer an seinen Rezeptor gebunden ist und außerdem keine Bindung in EpCAM-negativen Zellen beobachtet wurde. Diese Ergebnisse bestätigen die Selektivität und Spezifität des entwickelten Aptamers.
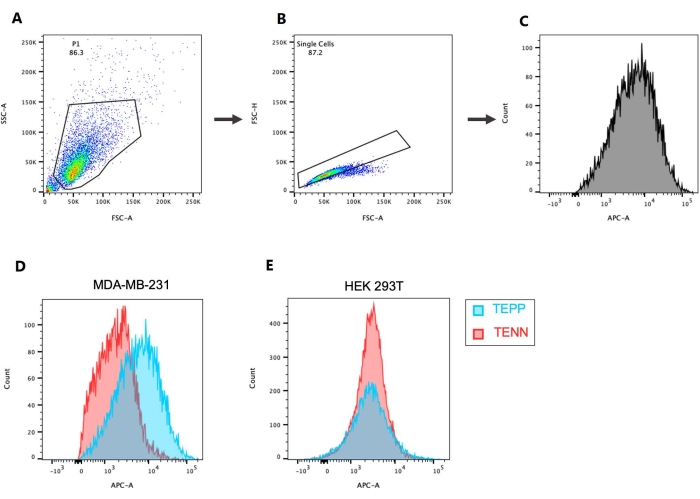
Abbildung 4: Gating und Histogramme, die die Bindung der Zellen an das Aptamer zeigen. (A) Auswahl der Zellpopulation, (B) der einzelnen Zellen und (C) des Histogramms der an das Aptamer gebundenen Zellen (200 nM). Die Bindung von EpCAM-Aptamer (TEPP) mit einem nicht-EpCAM-bindenden Aptamer (TENN) in (D) EpCAM+ MDA-MB-231-Zellen und (E) EpCAM-HEK 293T-Zellen wird verglichen. Abkürzungen: EpCAM = epitheliales zelluläres Adhäsionsmolekül; FSC-A = Vorwärts-Streu-Peak-Fläche; SSC-A = Seitenstreu-Peak-Fläche; FSC-H = Vorwärts-Streu-Peak-Höhe; APC = Allophycocyanin. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Diskussion
Die größte Herausforderung bei der Entwicklung neuer Aptamere ist das Fehlen von Standardrichtlinien, die für verschiedene Schritte dieses Prozesses gelten. McKeague et al. haben kürzlich einige der damit verbundenen Herausforderungen aufgezeigt, die zu unklaren Darstellungen von Daten in Publikationen und zum Versagen führen, die Forschung zu replizieren. Sie schlugen grundlegende Richtlinien vor, die für die Berücksichtigung bei der Charakterisierung von Aptameren erforderlich sind19. Ein Aptamerbindungsassay ist ein kritischer Schritt beim Screening und/oder der Charakterisierung von Aptameren23, der von Forschern auf diesem Gebiet weit verbreitet ist. Da es keine einheitliche Richtlinie zur Darstellung des Schritt-für-Schritt-Protokolls gibt, wird im begleitenden Videoprotokoll eine Durchflusszytometrie-Methode demonstriert, die üblicherweise zur Untersuchung der Aptamer-Protein-Bindung verwendet wird.
Es gibt mehrere Methoden, die die Interaktion des Aptamers und seines Ziels messen. Die Durchflusszytometrie ist eine dieser Methoden. Weitere Beispiele sind Fluoreszenzpolarisation, Oberflächenplasmonresonanz, Kapillarelektrophorese und isotherme Titrationskalorimetrie24. Die Wahl der richtigen Methode hängt von der Anwendung des Aptamers ab. Es ist jedoch wichtig zu wissen, dass jede Methode ihre Grenzen hat und dass die Anwendung mehrerer Assays für die Charakterisierung von niedermolekularen Aptameren vorteilhafter ist24. Die hier beschriebene Methode hat mehrere Vorteile. Es ist eine der zuverlässigsten, präzisesten und genauesten Methoden und ist auch schnell und kostengünstig. Die durchflusszytometrische Analyse der Aptamerbindung kann in verschiedenen Schritten der Aptamerentwicklung angewendet werden, einschließlich Kandidatenscreening, Trunkierung und Optimierung, Charakterisierung und Validierung.
Bei der Fluoreszenzmarkierung besteht die Möglichkeit, entweder die intrinsische Fluoreszenz des Ziels zu markieren oder zu verwenden oder den Aptamer24 zu markieren. Nach den Erfahrungen der Autoren ist die Verwendung von markierten Aptameren zuverlässig und einfach einzurichten. Aptamere können an jedem Ende (3' oder 5')25 mit einem Fluorophor markiert werden; Das Ende mit weniger Guanin ist jedoch günstiger, da Guanin die Fluoreszenz und deren Nachweis löschen kann26. Ein Nachteil dieser Methode ist, dass das Aptamer an ein Fluorophor markiert werden sollte. Obwohl diese Methode die Bindung des Aptamers an das spezifische Protein widerspiegelt, zeigt sie nicht den Ort der Interaktionsstelle an. Daher könnten weitere Studien, wie die Fluoreszenzmikroskopie, erforderlich sein, um den Aptamer-Protein-Interaktionsort25 zu bestätigen.
Es gibt einige kritische Anmerkungen, die bei der Durchführung dieses Assays zu berücksichtigen sind. Es ist wichtig zu berücksichtigen, dass jedes Aptamer spezifische Anforderungen an die Handhabung und das Falten hat. Dazu gehören die Wahl der Rekonstitutions- und Verdünnungspuffer und die Faltbedingungen. Die Rekonstitution von lyophilisierten Aptameren erfolgt in gereinigtem sterilem Pyrogen- und RNase-freiem Reinstwasser. Dies dient dazu, die Konzentration von Ionen um die Nukleotide zu minimieren. Die Ionenkonzentration beeinflusst stark die Bildung von 2D-Strukturen von Aptameren und deren Affinität und Stabilität. Daher sollte für die weitere Rekonstitution des Aptamerbestandes auf die korrekte Herstellung anderer Puffer und metallkationischer Lösungen wieMgCl225,27 geachtet werden. Die optimale Konzentration der metallischen kationischen Lösung schirmt die negative Ladung des Aptamers noch ab, hemmt nicht die Wechselwirkung des Aptamers mit seinem Ziel.
Da das Aptamer ein Potenzial für eine unspezifische, zielferne Bindung hat, spielt die Anwendung von Blockierpuffer eine entscheidende Rolle im Bindungsassay. Neben dem negativ geladenen BSA, das üblicherweise für Antikörper verwendet wird, wird hier auch Lachsspermien-DNA oder tRNA benötigt. Diese Mischung blockiert die positiv geladenen Proteine und Nukleinsäurebindungsstellen. Dieses Blockierungsstadium ist besonders wichtig für die Selektivität und Spezifität von Aptameren gegenüber Krebszellen, aufgrund ihrer negativen Ladung im Vergleich zu neutralen und positiv geladenen normalen Zellen28. Darüber hinaus ist die 3D-Faltung von Aptameren von anderen Faktoren wie der Temperatur abhängig. Die Bedeutung von Bedingungen, die die 3D-Bildung des Aptamers beeinflussen, einschließlich der Wahl der Röhre und der Dauer der PCR-Phasen, wurde untersucht25. Darüber hinaus sollte die Inkubationszeit des Aptamers mit dem Target für jedes spezifische Aptamer optimiert werden. Die Inkubationstemperatur ist ebenfalls sehr wichtig. Die Aufrechterhaltung einer Temperatur von 4 °C ist besonders kritisch für Zelloberflächenproteine, die leicht internalisiert werden können, wie z.B. EpCAM25.
Der andere wichtige Faktor in diesem Assay ist die Anwendung geeigneter Kontrollen. Krebszellen, die das Zielprotein entweder exprimieren oder nicht, sollten verwendet werden, um die Selektivität des Aptamers zu bewerten. In jedem Experiment ist zusätzlich zum interessierenden Aptamer ein Negativkontrollaptamer (eine Zufallssequenz oder eine verschlüsselte Sequenz) erforderlich, um die Spezifität des Aptamers zu zeigen. Idealerweise sollte diese Kontrolle eine ähnliche Länge von Nukleotiden haben und eine ähnliche Faltung wie das Aptamer durchlaufen. In diesem Experiment wurde eine Negativkontrolle (TENN), ein Aptamer mit einer ähnlichen Struktur wie TEPP, das eine geringe Bindungsaffinität zu EpCAM aufweist, verwendet10.
Das hier vorgestellte Protokoll ist ein qualitativer Assay und kann weiter für die quantitative Beurteilung der Bindungsaffinität und die Bestimmung der Dissoziationskonstante (Kd)29,30,31 verwendet werden. Es ist jedoch wichtig, die Reproduzierbarkeit der Ergebnisse anhand technischer und biologischer Replikate nachzuweisen. Um dies zu erreichen, ist es sehr wichtig, die wichtigsten Determinanten zu berücksichtigen, um diesen Assay ordnungsgemäß durchzuführen. Dies könnte unter anderem die Verwendung einer geringen Durchgangszahl von mykoplasmenfreien Zellen, die unter ihren optimalen Bedingungen richtig gezüchtet werden, die Verwendung einer konstanten Anzahl von Zellen, die in jedem Replikat mit dem Aptamer exponiert werden sollen, die Anwendung geeigneter Temperatur- und Faltungsbedingungen, die Aufrechterhaltung ähnlicher experimenteller Bedingungen sowohl für das Aptamer als auch für die Kontrolle, Aufrechterhaltung einer minimalen Exposition der Aptamere gegenüber Umgebungslicht, unter Verwendung einer optimierten Aptamerzell-Expositionszeit und -temperatur, Aufrechterhaltung einer Temperatur von 4 °C für die Zellen, einschließlich Vorkühlung der Zentrifuge, der 96-Well-Platte und der Durchflusszytometrieröhrchen, und genaue Vorbereitung der Puffer mit einer Ionenkonzentration, die den angegebenen Konzentrationen so nahe wie möglich kommt.
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Die Autoren würdigen die SEED-Finanzierung des Institute for Mental and Physical Health and Clinical Translation (IMPACT), das Programm "Alfred Deakin Postdoctoral Research Fellowship" an der Deakin University und das "Australian Government Research Training Program Scholarship".
Materialien
| Name | Company | Catalog Number | Comments |
| 1.5 mL microcentrifuge tubes with attached lid | Sigma-Aldrich | T6649 | |
| 15 mL CellStar blue screw cap, conical bottom tube | Greiner Bio One | 188271 | |
| 5 mL serological pipettes | Greiner Bio One | 606180 | |
| BD FACSCanto II Flow Becton Dickinson Cytometer | Becton Dickinson | N/A | |
| BD FACSDiva V9.0 | BD Biosciences | N/A | |
| Bovine Serum Albumin (BSA), Lyophilized powder | Sigma-AldrichTM | A7906-50G | |
| Bright-line Hemocytometer | Sigma-Aldrich | Z359629 | |
| Dulbecco’s Modified Eagle Medium (DMEM) High Glucose Media Powder | Life Technologies | 12100046 | |
| Dulbecco’s Phosphate- Buffered Saline (DPBS) | Life Technologies | 21300025 | |
| FlowJo, LLC 10.8.1 | BD Biosciences | N/A | |
| Foetal Bovine Serum (FBS) | Bovogen | SFBS-F | |
| HEK293T | American Type Culture Collection | ACS-4500 | |
| Heracell 150i CO2 Incubator | Thermo Fisher Scientific | N/A | |
| Heraeus Megafuge 16R Centrifuge | Thermo Fisher Scientific | N/A | |
| Magnesium Chloride (MgCl2) | Sigma-Aldrich | M8266 | |
| MDA-MB-231 | American Type Culture Collection | CRM-HTB-26 | |
| Microplate, PS, 96 well, F-bottom (Chimney well), Black | Greiner Bio One | 655076 | |
| MiniAmp Thermal Cycler | Thermo Fisher Scientific | A37834 | |
| Phosphate-Buffered Saline (PBS) tablets | Life Technologies | 18912014 | |
| Pyrogen- and RNase-free ultrapure water | Milli-Q | ||
| T75 Cell Culture flask | Cellstar | 658170 | |
| TENN | Integrated DNA Technologies | N/A | 5′-GC GCG TGCA CGC GC TA ACG GA TTCCTTT TCC GT-3 |
| TEPP | Integrated DNA Technologies | N/A | 5′-GC GCG GTAC CGC GC TA ACG GA GGTTGCG TCC GT-3′ |
| Transfer RNA (tRNA) | Sigma-Aldrich | R8508-5X1ML | |
| Trypan Blue Solution | Life Technologies | 15250061 | |
| Trypsin-EDTA | Gibco | 15400054 |
Referenzen
- Cancer. World Health Organization Available from: https://www.who.int/news-room/fact-sheets/detail/cancer#:~:text=Cancer%20is%20a%20leading%20cause.and%20rectum%20and%20prostate%20cancers (2022)
- Liu, J. K. H. The history of monoclonal antibody development - Progress, remaining challenges and future innovations. Annals of Medicine and Surgery. 3 (4), 113-116 (2014).
- Nakhjavani, M., Shigdar, S. Future of PD-1/PD-L1 axis modulation for the treatment of triple-negative breast cancer. Pharmacological Research. 175, 106019 (2022).
- Bukari, B., Samarasinghe, R. M., Noibanchong, J., Shigdar, S. L. Non-invasive delivery of therapeutics into the brain: the potential of aptamers for targeted delivery. Biomedicines. 8 (5), 120 (2020).
- Wu, X., Chen, J., Wu, M., Zhao, J. X. Aptamers: active targeting ligands for cancer diagnosis and therapy. Theranostics. 5 (4), 322 (2015).
- Feng, X., et al. The aptamer functionalized nanocomposite used for prostate cancer diagnosis and therapy. Journal of Nanomaterials. 2022, (2022).
- Huang, J., et al. Advances in aptamer-based biomarker discovery. Frontiers in Cell and Developmental Biology. 9, 571 (2021).
- Ashrafuzzaman, M. Aptamers as both drugs and drug-carriers. BioMed Research International. 2014, (2014).
- Nakhjavani, M., Samarasinghe, R. M., Shigdar, S. Triple-negative breast cancer brain metastasis: an update on druggable targets, current clinical trials, and future treatment options. Drug Discovery Today. , (2022).
- Macdonald, J., et al. Development of a bifunctional aptamer targeting the transferrin receptor and epithelial cell adhesion molecule (EpCAM) for the treatment of brain cancer metastases. ACS Chemical Neuroscience. 8 (4), 777-784 (2017).
- Macdonald, J., et al. Bifunctional aptamer-doxorubicin conjugate crosses the blood-brain barrier and selectively delivers its payload to EpCAM-positive tumor cells. Nucleic Acid Therapeutics. 30 (2), 117-128 (2020).
- Shigdar, S., Agnello, L., Fedele, M., Camorani, S., Cerchia, L. Profiling cancer cells by cell-SELEX: use of aptamers for discovery of actionable biomarkers and therapeutic applications thereof. Pharmaceutics. 14 (1), 28 (2021).
- Rahimizadeh, K., et al. Development of cell-specific aptamers: recent advances and insight into the selection procedures. Molecules. 22 (12), 2070 (2017).
- Chen, M., et al. Development of cell-SELEX technology and its application in cancer diagnosis and therapy. International Journal of Molecular Sciences. 17 (12), 2079 (2016).
- Shigdar, S., et al. The use of sensitive chemical antibodies for diagnosis: detection of low levels of EpCAM in breast cancer. PLoS One. 8 (2), 57613 (2013).
- Ni, J., et al. Role of the EpCAM (CD326) in prostate cancer metastasis and progression. Cancer and Metastasis Reviews. 31 (3), 779-791 (2012).
- Ni, J., et al. Epithelial cell adhesion molecule (EpCAM) is associated with prostate cancer metastasis and chemo/radioresistance via the PI3K/Akt/mTOR signaling pathway. The International Journal of Biochemistry & Cell Biology. 45 (12), 2736-2748 (2013).
- McKeague, M., Kruse, P. F., Patterson, M. K., et al. . Tissue Culture. , 395-397 (1973).
- McKeague, M., et al. The minimum aptamer publication standards (MAPS guidelines) for de novo aptamer selection. Aptamers. 6, 10-18 (2022).
- Schoofs, G., Van Hout, A., D'huys, T., Schols, D., Van Loy, T. A flow cytometry-based assay to identify compounds that disrupt binding of fluorescently-labeled CXC Chemokine ligand 12 to CXC Chemokine receptor 4. Journal of Visualized Experiments. (133), e57271 (2018).
- McKinnon, K. M. Flow cytometry: an overview. Current Protocols in Immunology. 120 (1), 1-11 (2018).
- Kamiloglu, S., Sari, G., Ozdal, T., Capanoglu, E. Guidelines for cell viability assays. Food Frontiers. 1 (3), 332-349 (2020).
- Ruscito, A., DeRosa, M. C. Small-molecule binding aptamers: Selection strategies, characterization, and applications. Frontiers in Chemistry. 4, 14 (2016).
- McKeague, M., et al. Comprehensive analytical comparison of strategies used for small molecule aptamer evaluation. Analytical Chemistry. 87 (17), 8608-8612 (2015).
- Henri, J., Bayat, N., Macdonald, J., Shigdar, S. A guide to using nucleic acid aptamers in cell based assays. Aptamers. 23, (2019).
- Mao, H., et al. The mechanism and regularity of quenching the effect of bases on fluorophores: the base-quenched probe method. Analyst. 143 (14), 3292-3301 (2018).
- McKeague, M., et al. Analysis of in vitro aptamer selection parameters. Journal of Molecular Evolution. 81 (5), 150-161 (2015).
- Chen, B., et al. Targeting negative surface charges of cancer cells by multifunctional nanoprobes. Theranostics. 6 (11), 1887 (2016).
- Shigdar, S., et al. RNA aptamers targeting cancer stem cell marker CD133. Cancer Letters. 330 (1), 84-95 (2013).
- Amraee, M., Oloomi, M., Yavari, A., Bouzari, S. DNA aptamer identification and characterization for E. coli O157 detection using cell based SELEX method. Analytical Biochemistry. 536, 36-44 (2017).
- Yu, X. -. X., et al. Selection and characterization of a novel DNA aptamer, Apt-07S specific to hepatocellular carcinoma cells. Drug Design, Development and Therapy. 14, 1535 (2020).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten